高特异性生产麦芽三糖的麦芽三糖淀粉酶突变体的制作方法
本发明涉及高特异性生产麦芽三糖的麦芽三糖淀粉酶突变体,属于基因工程和酶工程领域。
背景技术:
麦芽三糖(maltotriose,α-d-glc-(1→4)-α-d-glc-(1→4)-d-glc,简称g3)属于一种功能性低聚糖,由三个葡萄糖单元经α-(1→4)糖苷键连接而成。其分子式为c18h32o16,相对分子质量为504.438,密度为1.8±0.1g·cm-3,熔点为132-135℃,常温下为白色固体粉末。g3具有多种优异的物理化学和生理学性质,其是可消化的营养甜味品,具有非致龋、低甜度、低粘度、低热量、抗结晶和低渗透压的特点,可用于改变冷冻食品的冷冻温度,并控制热加工食品中美拉德反应引起的褐变量。g3提供高保湿能力,防止过度干燥和低水分活度,便于控制微生物污染,具有强稳定性,是淀粉回生的强抑制剂。g3在食品、饮料、化工、医药等行业应用广泛。市场上的g3产品大都来自日本。日本食品化工公司的“富士低聚g3”、日本玉米淀粉公司的“新型麦芽低聚糖”、日本三菱化学食品株式会社的“线性寡糖origo”、群荣化学工业株式会社的“puretose”,主要成分都为g3。
麦芽三糖淀粉酶(ec3.2.1.116,maltotrioseformingα-amylase,简称amya)由基因tfa编码,是一种具有形成麦芽三糖能力的α-淀粉酶。amya以淀粉或糊精为底物,通过内切或外切作用水解糖链中的α-(1→4)糖苷键,产生麦芽三糖及少量副产物。该种酶的发现为g3的制备提供了新思路。
目前g3的制备工艺分为化学法和酶法两种。化学法以普鲁兰多糖为起始原料,通过乙酰解、脱乙酰基两个步骤即可制备g3。在两步反应中,需要用到浓硫酸、石油醚、甲醇和甲醇钠等高危试剂,虽然步骤简短、操作简单,但是底物成本高、环境危害大。酶法作为一种绿色节能高效安全的替代方法可显著降低生产成本、提高产品品质、满足大众消费需求,是推动麦芽三糖产业可持续发展的原动力。因为所用酶的不同,酶法制备又分为两种。酶法制备因为所用酶的不同又分为两种。第一种酶法是通过普鲁兰酶水解普鲁兰多糖,可连续化、自动化制备医药级的g3(纯度99%以上),但是这种方法存在着底物成本高的问题。与普鲁兰多糖相比,淀粉是一种丰富的天然可再生资源,是许多重要粮食作物(如大米、玉米、小麦、水稻、马铃薯和木薯)的种子、根或块茎中的主要储存碳水化合物,更加经济实惠。麦芽糊精是淀粉糊化后添加α-淀粉酶的液化产物。因α-淀粉酶的作用,麦芽糊精的聚合度低,溶解度大,有利于其他酶进行作用。第二种方法就是以amya作为主要用酶,以淀粉或麦芽糊精等廉价原料作为底物生产g3。通常而言,在反应体系中还需添加普鲁兰酶进行脱支作用。但是目前amya存在着副产物葡萄糖和麦芽糖多的问题,增加了分离纯化的难度和成本。因此,通过分子改造等手段获得高g3特异性的amya,可提高g3产量,简化生产工艺,降低生产成本。
技术实现要素:
为了解决目前存在的问题,本发明提供了麦芽三糖淀粉酶突变体。本发明所使用的来源于thermobifidafuscantu22的麦芽三糖淀粉酶tfamya在酶转化反应初期就产生了g1-g3的产物,属于一种内切酶,这降低了tfamya生产g3时对底物的得率,同时也增加了分离纯化的难度。因此,进一步提高该酶的产g3能力与特异性有利于g3生产成本的降低与规模化生产。
本发明提供了一种高特异性生产麦芽三糖的麦芽三糖淀粉酶突变体,是相对于亲本tfamya在羧基末端截短,或截短的突变体氨基酸的第47位、第101位、第103位、第153位任一位点的氨基酸发生突变。
在本发明的一种实施方式中,所述亲本tfamya的氨基酸序列如seqidno.1所示。
在本发明的一种实施方式中,所述突变体为以下(a)~(e)任一:
(a)将亲本的第451-572位氨基酸截去,氨基酸序列如seqidno.4所示;
(b)将序列如seqidno.4所示氨基酸的第47位的亮氨酸突变为赖氨酸或精氨酸,分别将突变体命名为l47k、l47r;
(c)将序列如seqidno.4所示氨基酸的第101位的甘氨酸突变为丝氨酸、苏氨酸、天冬氨酸、天冬酰胺、谷氨酸、或谷氨酰胺,分别将突变体命名为g101s、g101t、g101d、g101n、g101e、g101q;
(d)将序列如seqidno.4所示氨基酸的第103位的甘氨酸突变为组氨酸,将突变体命名为g103h;
(e)将序列如seqidno.4所示氨基酸的第153位的甘氨酸突变为丝氨酸、天冬氨酸、天冬酰胺、谷氨酸、谷氨酰胺或组氨酸,分别将突变体命名为g153s、g153d、g153n、g153e、g153q、g153h。
本发明提供了编码所述突变体的基因。
本发明提供了携带所述基因的重组表达载体,所述表达载体为pet系列、duet系列、pgex系列、phy300、phy300plk、ppic3k或ppic9k系列中的任意一种。
本发明提供了表达所述突变体,或携带所述基因的微生物细胞。
在本发明的一种实施方式中,所述微生物细胞为原核细胞或真核细胞。
本发明提供了一种提高麦芽三糖生产特异性的方法,所述方法以糊精为底物,以所述突变体为催化剂,生产麦芽三糖。
在本发明的一种实施方式中,所述糊精以大米、玉米、小麦、水稻、马铃薯、番薯、葛根和/或木薯为原料。
本发明还保护所述突变体,或所述基因,或所述重组表达载体,或权利要求6或7所述微生物细胞,或提高麦芽三糖生产特异性的方法在制备麦芽三糖中的应用。
本发明的有益效果:本发明通过对tfamya进行截短突变及定点突变,获得了多种突变体,麦芽三糖淀粉酶的活力增强;产物中麦芽三糖的比例提高,有利于简化麦芽三糖的后续纯化工艺与实现规模化生产,并且能显著降低麦芽三糖的生产成本,具备广泛的工业化应用前景。
附图说明
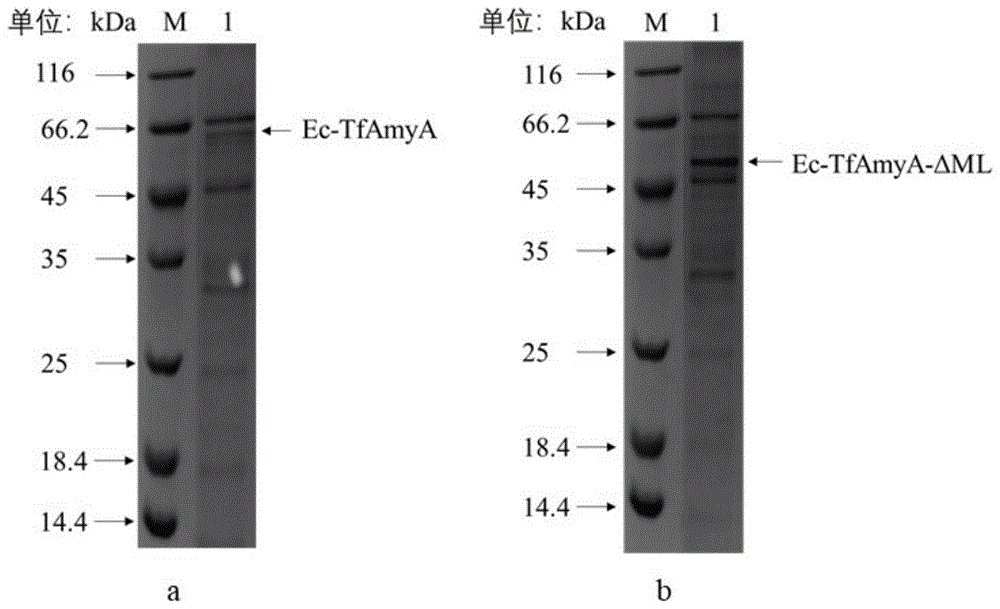
图1为截短前后的sds-page图;a为野生型,b为截短后的突变体。
具体实施方式
下面结合具体实施方式对本发明的技术方案做进一步说明,但本发明所保护范围不局限于此。
实施例中所述的培养基配方如下:
lb培养基(g/l):蛋白胨10,酵母提取物5,nacl10。
tb培养基(g/l):蛋白胨10,酵母粉24,甘油5,k2hpo4·3h2o16.43,kh2po42.31。
实施例中所述的普鲁兰酶为任何一种具有淀粉脱支效果的脱支酶。
实施例中g3含量的检测方法:
将酶转化的样品沸水浴10min,以12000r·min-1离心10min,再用终浓度50%(体积比)的乙腈共沉淀,放置2h后,以12000r·min-1离心10min,取上清液,用注射器过0.22μm滤膜。使用乙腈:水(74:26)的比例配制流动相,使用agilent1200hplc色谱仪和hypersilaps2氨基柱检测样品,设定柱温为40℃,流速为0.8ml·min-1。根据吸收峰面积和麦芽三糖标准品峰面积计算得到麦芽三糖的生成量。
实施例1:截短体酶的制备
1)截短体与野生型载体构建
①根据来源于thermobifidafuscantu22的麦芽三糖淀粉酶的氨基酸序列及核苷酸序列(核苷酸序列如seqidno.2所示),将截短后的基因片段tftfa-δml(核苷酸序列如seqidno.3所示),与载体pet20b(+)(表达载体上有启动基因表达的信号肽)利用一步克隆酶exnaseⅱ(南京诺唯赞生物科技有限公司)进行连接,构建重组质粒pet20b(+)-tftfa-δml。
②将核苷酸序列如seqidno.2所示的基因片段利用上述同样的方法与载体pet20b(+)连接,构建得到重组质粒pet20b(+)-tftfa。
2)截短体及野生酶的的表达与纯化
①将重组质粒pet20b(+)-tftfa-δml转化至宿主大肠杆菌bl21(de3)感受态细胞,在37℃下培养12-14h至长出单克隆,挑取单克隆,进行测序验证;将验证正确的阳性转化子接入lb液体培养基(含100μg/ml氨苄青霉素),在37℃、200rpm,培养8~10h,按5%接种量将种子发酵液接到tb液体培养基(含100μg/ml氨苄青霉素和7.5g/l甘氨酸);大肠杆菌在37℃摇床培养至od600=0.6~0.8,加入终浓度为0.134mm的iptg诱导胞外表达,并在25℃摇床继续培养发酵48小时后,将发酵液于4℃、10000g离心15min,去除菌体,收集上清液并纯化,纯化后的截短体酶。
②将重组质粒pet20b(+)-tftfa以步骤①相同的方法转化至宿主大肠杆菌bl21(de3)感受态细胞,并进行培养、诱导,得到纯化后的野生型酶。
实施例2:突变体酶的制备
1)定点突变
以质粒pet20b(+)-tftfa-δml为模板,利用合成的突变引物,进行pcr,构建突变体。
表1引物
pcr体系:2×phantamaxmastermix25μl、模板1μl、上游引物2μl、下游引物2μl、ddh2o20μl。pcr参数:94℃预变性4min;进入pcr循环:98℃变性10s,55℃退火30s,72℃延伸(时长根据扩增片段长度计算,1000bp·min-1),循环25-30次;最后72℃10min,4℃保温。pcr产物经dpni(fermentas公司)消化,转化大肠杆菌jm109感受态细胞,转化的感受态细胞在lb固体培养基(含100μg/ml氨苄青霉素)培养过夜后,挑单克隆于lb液体培养基(含100μg/ml氨苄青霉素)中培养,后提取质粒,并进行测序。
2)突变体酶的表达与纯化
将测序正确突变的质粒转化表达宿主大肠杆菌bl21(de3)感受态细胞,挑取转入表达宿主大肠杆菌bl21(de3)的单克隆于lb液体培养基(含100μg/ml氨苄青霉素)37℃,200rpm,培养8~10h,按5%接种量将种子发酵液接到tb液体培养基(含100μg/ml氨苄青霉素、7.5g·l-1甘氨酸);大肠杆菌在37℃摇床培养至od600=0.6~0.8,加入0.134mm终浓度的iptg诱导胞外表达,并在25℃摇床继续培养发酵48小时后,将发酵液于4℃、10000g离心15min除菌体,收集上清液并纯化,分别得到突变体酶样品。
实施例3:麦芽三糖淀粉酶的酶活测定
酶活测定采用dns法,体系如下。
底物配制:1%质量浓度的可溶性淀粉悬浮于相应的buffer,加热搅拌糊化。
反应体系:空白组:1ml底物(2g/100ml可溶性淀粉)+1mlbuffer。
对照组:1ml底物+0.9mlbuffer+0.1ml稀释适当倍数的酶液。
提前加好底物和buffer成分至具塞试管中,放置于恒温水浴锅温育10min。将酶稀释至适当倍数,加入对照组反应,精确计时10min。10min后,加入3mldns,所有具塞试管同时放置于沸水锅中,精确计时7min,迅速拿出置于冰浴盆降温。待降至低温后,加入10mlh2o混匀。利用540nm分光光度计测得相应去空白的吸光值abs540,代入标准曲线计算酶活。
标准曲线:以不同浓度的葡萄糖按照上述体系可测得abs540与还原糖浓度之间的关系,线性回归方程即为标准曲线。
酶活定义:每分钟释放1μmol还原糖所需的酶量定义为一个酶活力单位(u)。
25℃摇床继续培养发酵48小时后,收集上清液,测野生酶及突变体酶摇瓶酶活结果如表1所示。截短体tfamya-δml酶活变化不大,除l47,g103的突变酶活提高外,g101位点和g153的突变大部分酶活降低或略有降低。
表2突变体与野生型的摇瓶酶活对照表
实施例4:hplc方法分析麦芽三糖生成量
配制de值5-7的5%质量浓度的麦芽糊精溶液(ph5.5),分别加入一定量的麦芽三糖淀粉酶野生酶及突变酶,加酶量为60u·g-1底物,普鲁兰酶以加酶量32u·g-1底物添加至反应体系中,置于转速为150rpm、温度为55℃的水浴摇床中,反应11h。取样,沸水浴煮10min将酶灭活,离心取上清,即可得到产物溶液。取500μl产物溶液与乙腈1:1混合,室温放置2h,使大分子量的糊精或极限糊精沉淀,随后以12000rpm离心20min,将上清液用注射器过0.22μm滤膜,得到能进行hplc分析的样品。使用乙腈:水(74:26)的比例配制流动相,使用agilent1200hplc色谱仪和hypersilaps2氨基柱检测样品,设定柱温为40℃,流速为0.8ml·min-1。根据吸收峰面积和g3标准品峰面积计算得到g3的生成量。
野生酶及突变体酶酶转化分析结果如表2所示。截短体tfamya-δml提高了g3产率,同时g3的比率也有小幅度提高。除g103的突变g103v和g103p外,其他l47,g101,g103和g153的突变使酶转化产物的g3比率明显提高。单突变体提高了g3的特异性,使产物中其他糖减少,主产物g3增多。比率指产物中g3的质量浓度与总产物的质量浓度比。产率指产物的g3质量浓度与底物的质量浓度比。
表3突变体与野生型的g3产率、比率对照表
虽然本发明己以较佳实施例公开如上,但其并非用以限定本发明,任何熟悉此技术的人,在不脱离本发明的精神和范围内,都可做各种的改动与修饰,因此本发明的保护范围应该以权利要求书所界定的为准。
sequencelisting
<110>江南大学
<120>高特异性生产麦芽三糖的麦芽三糖淀粉酶突变体
<160>38
<170>patentinversion3.3
<210>1
<211>572
<212>prt
<213>thermobifidafusca
<400>1
alaproserglyasnargaspvalilevalhisleupheglntrparg
151015
trplysserilealaaspglucysargthrthrleuglyprohisgly
202530
pheglyalavalglnvalserproproglngluhisvalvalleupro
354045
alagluasptyrprotrptrpglnasptyrglnprovalsertyrlys
505560
leuaspglnthrargargglyserargalaasppheileaspmetval
65707580
asnthrcysargglualaglyvallysiletyrvalaspalavalile
859095
asnhismetthrglythrglyseralaglyalaglyproglyserala
100105110
glysersertyrserlystyrasptyrproglyiletyrglnsergln
115120125
asppheasnaspcysargargaspilethrasntrpasnasplystrp
130135140
gluvalglnhiscysgluleuvalglyleualaaspleulysthrser
145150155160
serprotyrvalglnaspargilealaalatyrleuasngluleuile
165170175
aspleuglyvalalaglypheargileaspalaalalyshisilepro
180185190
gluglyaspleuglnalaileleuserargleulysasnvalhispro
195200205
alatrpglyglyglylysprotyrilepheglngluvalilealaasp
210215220
serthrileserthrglysertyrthrhisleuglyservalthrglu
225230235240
pheglntyrhisargaspileserhisalaphealaasnglyasnile
245250255
alahisleuthrglyleuglyserglyleuthrproserasplysala
260265270
valvalphevalvalasnhisaspthrglnargtyrgluproileleu
275280285
thrhisthraspglyalaargtyraspleualaglnlysphemetleu
290295300
alahisprotyrglythrprolysvalmetsersertyrthrtrpser
305310315320
glyaspasplysalaglypropromethisseraspglythrthrarg
325330335
prothraspcysseralaaspargtrpleucysgluhisargalaval
340345350
alaglymetvalglyphehisasnalavalalaglyglnglyilegly
355360365
seralavalthraspglyasnglyargleualaphealaargglyser
370375380
alaglytyralaalapheasnalathrasnthralatrpthrargthr
385390395400
phethrthrserleuproaspglyvaltyrcysaspvalalaasngly
405410415
thrphevalaspglyvalcysaspglyprosertyrglnvalsergly
420425430
glylysphethralathrvalproalaasnglyalavalalaleuhis
435440445
valglualaproglysercysglyproaspglycysglythrpropro
450455460
glyglyglyaspaspcysthrthrvalthralaargphehisalathr
465470475480
valthrthrtrptyrglyglngluvalalavalvalglyserilepro
485490495
gluleuglysertrpglnproalaglnglyvalargleuargthrasp
500505510
serglythrtyrprovaltrpserglyalavalaspleuproalagly
515520525
valglypheglutyrlystyrvallysleulysproaspglythrval
530535540
glutrpgluglnglyglyasnargilealathrvalaspaspsergly
545550555560
glyglycysserglnasnphetyraspsertrparg
565570
<210>2
<211>1716
<212>dna
<213>thermobifidafusca
<400>2
gccccgagtggtaatcgcgatgttattgttcatctgtttcagtggcgctggaaaagcatt60
gcagatgaatgtcgtaccaccttaggtccgcatggttttggtgcagttcaggtgtctccg120
ccgcaggaacatgttgtgctgccggccgaagattatccgtggtggcaggattatcagccg180
gtgagctataaattagatcagacccgtcgcggctcacgcgcagattttattgatatggtt240
aatacctgtcgcgaagcaggtgttaaaatttatgtggatgccgttattaatcacatgacc300
ggaaccggctcagcaggcgcaggtcctggctcagccggctcaagctatagtaaatatgat360
tatccgggcatttatcagtcacaggattttaatgattgtcgtcgcgatattaccaattgg420
aatgataaatgggaagttcagcattgtgaattagtgggtttagcagacctgaaaacctct480
agtccgtatgttcaggatcgcattgccgcctatctgaatgaactgattgatctgggcgtt540
gcaggctttcgtattgatgccgccaaacatattccggaaggcgatttacaggccattctg600
tctcgtctgaaaaatgttcatccggcctggggcggcggtaaaccgtatatttttcaggaa660
gttattgcagattctaccatttcaaccggctcttatacccatctgggtagcgtgaccgaa720
tttcagtatcatcgcgatattagtcatgcctttgccaatggcaatattgcacatctgacc780
ggcttaggtagcggtctgaccccgtcagataaagcagttgtgtttgttgttaatcatgat840
acccagcgctatgaaccgattctgacccataccgatggtgcacgctatgatttagcacag900
aaatttatgctggcccatccgtatggaacaccgaaagttatgtcgtcttatacctggagt960
ggcgatgataaagcaggtccgccgatgcatagcgatggcaccacccgcccgaccgattgt1020
agtgcagatcgctggctgtgtgaacatcgcgcagttgccggcatggtgggttttcataat1080
gccgttgcaggccagggcattggtagtgcagtgaccgatggtaatggtcgcttagccttt1140
gcacgcggtagcgcaggctatgcagcctttaatgcaaccaataccgcctggacccgcacc1200
tttaccacctcactgccggatggtgtgtattgtgatgttgccaatggcacctttgtggat1260
ggcgtgtgtgatggtccgagctatcaggtgagcggcggcaaatttaccgcaaccgttccg1320
gccaatggtgccgttgccttacatgttgaagcgcctggttcttgtggtccggatggctgt1380
ggcaccccgccgggcggtggcgatgattgtaccaccgtgaccgcacgctttcatgcaacc1440
gtgaccacctggtatggccaggaagttgccgttgtgggctcaattccggaactgggttct1500
tggcagccggcccagggcgtgcgcttacgtaccgatagcggtacatatccggtttggagc1560
ggtgcagtggatctgccggcaggcgtgggttttgaatataaatatgttaaactgaaaccg1620
gatggcaccgttgaatgggaacagggcggtaatcgtattgcaaccgtggatgatagcggc1680
ggtggctgtagtcagaatttttatgattcttggcgt1716
<210>3
<211>1350
<212>dna
<213>人工序列
<400>3
gccccgagtggtaatcgcgatgttattgttcatctgtttcagtggcgctggaaaagcatt60
gcagatgaatgtcgtaccaccttaggtccgcatggttttggtgcagttcaggtgtctccg120
ccgcaggaacatgttgtgctgccggccgaagattatccgtggtggcaggattatcagccg180
gtgagctataaattagatcagacccgtcgcggctcacgcgcagattttattgatatggtt240
aatacctgtcgcgaagcaggtgttaaaatttatgtggatgccgttattaatcacatgacc300
ggaaccggctcagcaggcgcaggtcctggctcagccggctcaagctatagtaaatatgat360
tatccgggcatttatcagtcacaggattttaatgattgtcgtcgcgatattaccaattgg420
aatgataaatgggaagttcagcattgtgaattagtgggtttagcagacctgaaaacctct480
agtccgtatgttcaggatcgcattgccgcctatctgaatgaactgattgatctgggcgtt540
gcaggctttcgtattgatgccgccaaacatattccggaaggcgatttacaggccattctg600
tctcgtctgaaaaatgttcatccggcctggggcggcggtaaaccgtatatttttcaggaa660
gttattgcagattctaccatttcaaccggctcttatacccatctgggtagcgtgaccgaa720
tttcagtatcatcgcgatattagtcatgcctttgccaatggcaatattgcacatctgacc780
ggcttaggtagcggtctgaccccgtcagataaagcagttgtgtttgttgttaatcatgat840
acccagcgctatgaaccgattctgacccataccgatggtgcacgctatgatttagcacag900
aaatttatgctggcccatccgtatggaacaccgaaagttatgtcgtcttatacctggagt960
ggcgatgataaagcaggtccgccgatgcatagcgatggcaccacccgcccgaccgattgt1020
agtgcagatcgctggctgtgtgaacatcgcgcagttgccggcatggtgggttttcataat1080
gccgttgcaggccagggcattggtagtgcagtgaccgatggtaatggtcgcttagccttt1140
gcacgcggtagcgcaggctatgcagcctttaatgcaaccaataccgcctggacccgcacc1200
tttaccacctcactgccggatggtgtgtattgtgatgttgccaatggcacctttgtggat1260
ggcgtgtgtgatggtccgagctatcaggtgagcggcggcaaatttaccgcaaccgttccg1320
gccaatggtgccgttgccttacatgttgaa1350
<210>4
<211>450
<212>prt
<213>人工序列
<400>4
alaproserglyasnargaspvalilevalhisleupheglntrparg
151015
trplysserilealaaspglucysargthrthrleuglyprohisgly
202530
pheglyalavalglnvalserproproglngluhisvalvalleupro
354045
alagluasptyrprotrptrpglnasptyrglnprovalsertyrlys
505560
leuaspglnthrargargglyserargalaasppheileaspmetval
65707580
asnthrcysargglualaglyvallysiletyrvalaspalavalile
859095
asnhismetthrglythrglyseralaglyalaglyproglyserala
100105110
glysersertyrserlystyrasptyrproglyiletyrglnsergln
115120125
asppheasnaspcysargargaspilethrasntrpasnasplystrp
130135140
gluvalglnhiscysgluleuvalglyleualaaspleulysthrser
145150155160
serprotyrvalglnaspargilealaalatyrleuasngluleuile
165170175
aspleuglyvalalaglypheargileaspalaalalyshisilepro
180185190
gluglyaspleuglnalaileleuserargleulysasnvalhispro
195200205
alatrpglyglyglylysprotyrilepheglngluvalilealaasp
210215220
serthrileserthrglysertyrthrhisleuglyservalthrglu
225230235240
pheglntyrhisargaspileserhisalaphealaasnglyasnile
245250255
alahisleuthrglyleuglyserglyleuthrproserasplysala
260265270
valvalphevalvalasnhisaspthrglnargtyrgluproileleu
275280285
thrhisthraspglyalaargtyraspleualaglnlysphemetleu
290295300
alahisprotyrglythrprolysvalmetsersertyrthrtrpser
305310315320
glyaspasplysalaglypropromethisseraspglythrthrarg
325330335
prothraspcysseralaaspargtrpleucysgluhisargalaval
340345350
alaglymetvalglyphehisasnalavalalaglyglnglyilegly
355360365
seralavalthraspglyasnglyargleualaphealaargglyser
370375380
alaglytyralaalapheasnalathrasnthralatrpthrargthr
385390395400
phethrthrserleuproaspglyvaltyrcysaspvalalaasngly
405410415
thrphevalaspglyvalcysaspglyprosertyrglnvalsergly
420425430
glylysphethralathrvalproalaasnglyalavalalaleuhis
435440445
valglu
450
<210>5
<211>36
<212>dna
<213>人工序列
<400>5
aacatgttgtgaaaccggccgaagattatccgtggt36
<210>6
<211>33
<212>dna
<213>人工序列
<400>6
tcttcggccggtttcacaacatgttcctgcggc33
<210>7
<211>34
<212>dna
<213>人工序列
<400>7
aacatgttgtgcgtccggccgaagattatccgtg34
<210>8
<211>33
<212>dna
<213>人工序列
<400>8
tcttcggccggacgcacaacatgttcctgcggc33
<210>9
<211>29
<212>dna
<213>人工序列
<400>9
tcacatgaccagcaccggctcagcaggcg29
<210>10
<211>33
<212>dna
<213>人工序列
<400>10
tgctgagccggtgctggtcatgtgattaataac33
<210>11
<211>30
<212>dna
<213>人工序列
<400>11
tcacatgaccaccaccggctcagcaggcgc30
<210>12
<211>33
<212>dna
<213>人工序列
<400>12
tgctgagccggtggtggtcatgtgattaataac33
<210>13
<211>29
<212>dna
<213>人工序列
<400>13
tcacatgaccgataccggctcagcaggcg29
<210>14
<211>36
<212>dna
<213>人工序列
<400>14
tgctgagccggtatcggtcatgtgattaataacggc36
<210>15
<211>32
<212>dna
<213>人工序列
<400>15
tcacatgaccaataccggctcagcaggcgcag32
<210>16
<211>37
<212>dna
<213>人工序列
<400>16
ctgctgagccggtattggtcatgtgattaataacggc37
<210>17
<211>26
<212>dna
<213>人工序列
<400>17
tcacatgaccgaaaccggctcagcag26
<210>18
<211>33
<212>dna
<213>人工序列
<400>18
tgctgagccggtttcggtcatgtgattaataac33
<210>19
<211>32
<212>dna
<213>人工序列
<400>19
tcacatgacccagaccggctcagcaggcgcag32
<210>20
<211>36
<212>dna
<213>人工序列
<400>20
tgctgagccggtctgggtcatgtgattaataacggc36
<210>21
<211>27
<212>dna
<213>人工序列
<400>21
accggaaccccttcagcaggcgcaggt27
<210>22
<211>34
<212>dna
<213>人工序列
<400>22
gcgcctgctgaaggggttccggtcatgtgattaa34
<210>23
<211>25
<212>dna
<213>人工序列
<400>23
accggaaccgtttcagcaggcgcag25
<210>24
<211>24
<212>dna
<213>人工序列
<400>24
gcgcctgctgaaacggttccggtc24
<210>25
<211>26
<212>dna
<213>人工序列
<400>25
gaccggaacccactcagcaggcgcag26
<210>26
<211>30
<212>dna
<213>人工序列
<400>26
cgcctgctgagtgggttccggtcatgtgat30
<210>27
<211>41
<212>dna
<213>人工序列
<400>27
gtgaattagtggaattagcagacctgaaaacctctagtccg41
<210>28
<211>39
<212>dna
<213>人工序列
<400>28
ggtctgctaattccactaattcacaatgctgaacttccc39
<210>29
<211>41
<212>dna
<213>人工序列
<400>29
gtgaattagtgcagttagcagacctgaaaacctctagtccg41
<210>30
<211>41
<212>dna
<213>人工序列
<400>30
caggtctgctaactgcactaattcacaatgctgaacttccc41
<210>31
<211>40
<212>dna
<213>人工序列
<400>31
tgaattagtggacttagcagacctgaaaacctctagtccg40
<210>32
<211>35
<212>dna
<213>人工序列
<400>32
ggtctgctaagtccactaattcacaatgctgaact35
<210>33
<211>40
<212>dna
<213>人工序列
<400>33
tgaattagtgaatttagcagacctgaaaacctctagtccg40
<210>34
<211>35
<212>dna
<213>人工序列
<400>34
ggtctgctaaattcactaattcacaatgctgaact35
<210>35
<211>40
<212>dna
<213>人工序列
<400>35
tgaattagtgagtttagcagacctgaaaacctctagtccg40
<210>36
<211>34
<212>dna
<213>人工序列
<400>36
ggtctgctaaactcactaattcacaatgctgaac34
<210>37
<211>41
<212>dna
<213>人工序列
<400>37
gtgaattagtgcatttagcagacctgaaaacctctagtccg41
<210>38
<211>40
<212>dna
<213>人工序列
<400>38
aggtctgctaaatgcactaattcacaatgctgaacttccc40
- 还没有人留言评论。精彩留言会获得点赞!