联产1,3-丙二醇和D-1,2,4-丁三醇的方法与流程
本发明涉及联产1,3-丙二醇和d-1,2,4-丁三醇的方法,属于发酵技术领域以及生物技术领域。
背景技术:
1,3-丙二醇(1,3-propanediol,1,3-pdo)为无色、无臭,具有咸味和吸湿性的黏稠液体。在聚氨酯行业中,它常用作聚酯多元醇的原料、聚醚多元醇的起始剂和聚氨酯的扩链剂等;在有机化工行业中,它是重要的单体和中间体,最主要的用途是作为聚合物单体合成聚对苯二甲酸丙二醇酯(ptt)。因此,1,3-丙二醇的市场容量巨大。
目前,工业上常通过将可生产1,3-丙二醇的肺炎克雷伯菌(klebsiellapneumoniae)接种至含有甘油的发酵培养基中进行发酵以生产1,3-丙二醇。但是,肺炎克雷伯菌以甘油作为碳源生产1,3-丙二醇时还原力不足,这直接导致了甘油的转化率低下以及1,3-丙二醇的产量低下(具体可见参考文献:付晓萌.利用木糖途径对克雷伯氏菌合成1,3-丙二醇代谢中nad(p)h-nad(p)的研究[d].[硕士学位论文].无锡:江南大学生物工程学院,2015.)。
研究表明,木糖的加入可有效增加肺炎克雷伯菌以甘油作为碳源生产1,3-丙二醇时的还原力,进而提高肺炎克雷伯菌以甘油作为碳源生产1,3-丙二醇时的甘油转化率和1,3-丙二醇产量(具体可见参考文献:陈琳,王领民,金平,等.甘油和木糖共发酵生产1,3-丙二醇的研究[j].化学工程,2013,41(07):5-8.)。但是,木糖的价格偏高,并且,木糖在1,3-丙二醇生产过程中的转化率低下,这严重限制了木糖在1,3-丙二醇生产中的应用。
因此,急需找到一种提高木糖在1,3-丙二醇生产过程中的转化率的方法。
技术实现要素:
[技术问题]
本发明要解决的技术问题是提供一种木糖转化率高的生产1,3-丙二醇的方法。
[技术方案]
为解决本发明的技术问题,本发明提供了一种重组克雷伯氏菌,所述重组克雷伯氏菌以克雷伯氏菌(klebsiella)为宿主,表达编码木糖脱氢酶的基因xdh、编码2-酮异戊酸脱羧酶的基因kivd以及编码木糖酸脱水酶的基因yjhg。
在本发明的一种实施方式中,所述克雷伯氏菌为肺炎克雷伯菌(klebsiellapneumoniae)。
在本发明的一种实施方式中,所述木糖脱氢酶来源于新月柄杆菌(caulobactercrescentus)。
在本发明的一种实施方式中,所述木糖脱氢酶的氨基酸序列如seqidno.1所示。
在本发明的一种实施方式中,所述编码木糖脱氢酶的基因xdh的核苷酸序列如seqidno.2所示。
在本发明的一种实施方式中,所述2-酮异戊酸脱羧酶来源于乳酸乳球菌(lactococcuslactis)。
在本发明的一种实施方式中,所述2-酮异戊酸脱羧酶的氨基酸序列如seqidno.3所示。
在本发明的一种实施方式中,所述编码2-酮异戊酸脱羧酶的基因kivd的核苷酸序列如seqidno.4所示。
在本发明的一种实施方式中,所述木糖酸脱水酶来源于大肠杆菌(escherichiacoli)。
在本发明的一种实施方式中,所述木糖酸脱水酶的氨基酸序列如seqidno.5所示。
在本发明的一种实施方式中,所述编码木糖酸脱水酶的基因yjhg的核苷酸序列如seqidno.6所示。
在本发明的一种实施方式中,所述重组克雷伯氏菌以克雷伯氏菌(klebsiella)为宿主,以petac质粒为表达载体,表达编码木糖脱氢酶的基因xdh、编码2-酮异戊酸脱羧酶的基因kivd以及编码木糖酸脱水酶的基因yjhg。
本发明还提供了一种联产1,3-丙二醇和d-1,2,4-丁三醇的方法,所述方法为先将上述重组克雷伯氏菌接种至含有甘油和木糖的发酵培养基中进行发酵,获得发酵液,然后将发酵液进行提取,获得1,3-丙二醇和d-1,2,4-丁三醇。
在本发明的一种实施方式中,所述发酵的温度为35~40℃。
在本发明的一种实施方式中,所述发酵的温度为37℃。
在本发明的一种实施方式中,所述发酵的转速为170~200rpm。
在本发明的一种实施方式中,所述发酵的转速为180rpm。
在本发明的一种实施方式中,所述发酵培养基的成分包含葡萄糖10g/l、木糖30g/l、甘油20g/l、酵母粉7.5g/l、氯化钠15g/l、蛋白胨15g/l、mgso42g/l、(nh4)2so42g/l、kh2po47.5g/l、feso4·7h2o0.005g/l、vb125g/l以及微量元素溶液100μl/l;所述微量元素溶液的成分包含zncl20.7g/l、mncl2·4h2o1g/l、h3bo30.6g/l、cocl2·6h2o2g/l、cucl20.2g/l、nicl2·6h2o0.25g/l以及na2moo4·2h2o0.35g/l。
本发明还提供了上述重组克雷伯氏菌或上述方法在生产1,3-丙二醇和d-1,2,4-丁三醇方面的应用。
[有益效果]
(1)本发明提供了一种可高效转化甘油和木糖联产1,3-丙二醇和d-1,2,4-丁三醇的重组克雷伯氏菌,此重组克雷伯氏菌以克雷伯氏菌为宿主,表达编码木糖脱氢酶的基因xdh、编码2-酮异戊酸脱羧酶的基因kivd以及编码木糖酸脱水酶的基因yjhg;将此重组克雷伯氏菌接种至含有甘油和木糖的发酵培养基中发酵48h,即可使发酵液中1,3-丙二醇的产量高达18.1g/l、d-1,2,4-丁三醇的产量高达3.3g/l,此时,发酵液中甘油的转化率高达45.3%、木糖的转化率高达12.3%。
(2)本发明提供了一种高效转化甘油和木糖联产1,3-丙二醇和d-1,2,4-丁三醇的方法,此方法为将表达编码木糖脱氢酶的基因xdh、编码2-酮异戊酸脱羧酶的基因kivd以及编码木糖酸脱水酶的基因yjhg的重组克雷伯氏菌接种至含有甘油和木糖的发酵培养基中进行发酵;利用此方法发酵48h,即可使发酵液中1,3-丙二醇的产量高达18.1g/l、d-1,2,4-丁三醇的产量高达3.3g/l,此时,发酵液中甘油的转化率高达45.3%、木糖的转化率高达12.3%;并且,由于d-1,2,4-丁三醇价值较高,利用此方法转化甘油和木糖联产1,3-丙二醇和d-1,2,4-丁三醇的价效高达12.31元/l,远高于单独合成1,3-丙二醇,利润较高。
附图说明
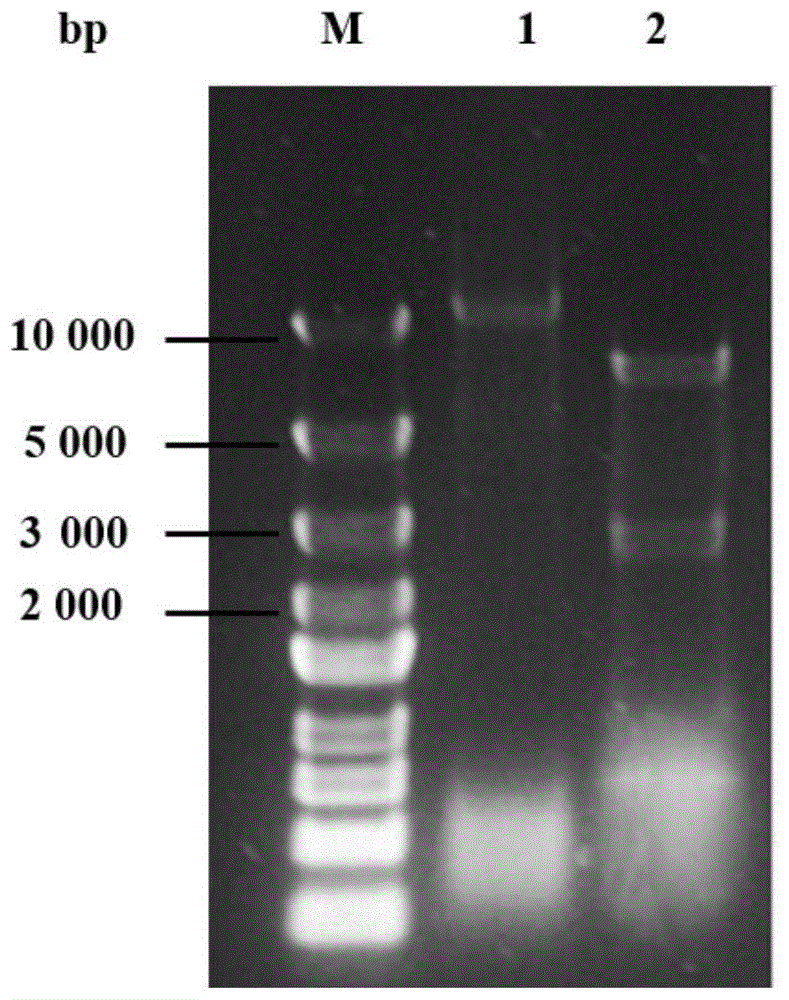
图1:重组质粒petac-yjhg-tac-kivd-tac-xdh的酶切酶切验证结果;其中,m:2503dnamarker;1:xbaⅰ单酶切产物;2:xbaⅰ和ncoⅰ双酶切产物。
图2:重组质粒petac-yjhg-tac-kivd-tac-xdh的质粒图谱。
图3:不同肺炎克雷伯菌发酵获得的发酵液中生物量的变化。
图4:不同肺炎克雷伯菌发酵获得的发酵液中甘油和1,3-丙二醇含量的变化。
图5:不同肺炎克雷伯菌发酵获得的发酵液中木糖和d-1,2,4-丁三醇含量的变化。
图6:不同肺炎克雷伯菌发酵获得的发酵液中2,3-丁二醇含量的变化。
图7:不同肺炎克雷伯菌发酵获得的发酵液中琥珀酸含量的变化。
图8:不同肺炎克雷伯菌发酵获得的发酵液中乳酸含量的变化。
图9:不同肺炎克雷伯菌发酵获得的发酵液中乙酸含量的变化。
图10:不同肺炎克雷伯菌发酵获得的发酵液中乙醇含量的变化。
具体实施方式
下述实施例中涉及的大肠杆菌(escherichiacoli)w3110购自上海北诺生物科技有限公司;下述实施例中涉及的肺炎克雷伯菌(klebsiellapneumoniae)ji22914购自标准品信息网,产品编号为:ji22914。
下述实施例中涉及的培养基如下:
lb液体培养基:蛋白胨10g/l、酵母膏5g/l、nacl10g/l。
lb固体培养基:蛋白胨10g/l、酵母膏5g/l、nacl10g/l、琼脂15g/l。
发酵培养基:葡萄糖10g/l、木糖30g/l、甘油20g/l、酵母粉7.5g/l、氯化钠15g/l、蛋白胨15g/l、mgso42g/l、(nh4)2so42g/l、kh2po47.5g/l、feso4·7h2o0.005g/l、vb125g/l以及微量元素溶液100μl/l;所述微量元素溶液的成分包含zncl20.7g/l、mncl2·4h2o1g/l、h3bo30.6g/l、cocl2·6h2o2g/l、cucl20.2g/l、nicl2·6h2o0.25g/l以及na2moo4·2h2o0.35g/l。
下述实施例中涉及的检测方法如下:
发酵液中生物量的测定:通过紫外分光光度计检测发酵液的od600值,细胞干重(dcw)与测得的od600值的对应关系公式为0.36g/l=1od600。
发酵液中甘油、木糖、1,3-pdo、bt、2,3-丁二醇、乙酸、琥珀酸、乙醇以及乳酸含量的测定:将发酵液经10000g离心15min后,取500μl上清液经0.22μm水系微孔滤膜处理后进行液相检测;色谱柱为aminexhpx-87h(300mm×7.8mm,9μm);流动相为5mmol·l-1h2so4;柱温为60℃;流速为0.6ml·min-1;进样体积为20μl;检测器为紫外检测器和示差折光检测器。
实施例1:重组肺炎克雷伯菌的构建
具体步骤如下:
合成来源于大肠杆菌(escherichiacoli)的编码木糖酸脱水酶的基因yjhg(seqidno.6);将合成得到的编码2-酮异戊酸脱羧酶的基因kivd与重组质粒petac-kivd-tac-xdh(记载于文献“jingpy,caox,luxy,etc.modificationofanengineeredescherichiacolibyacombinedstrategyofdeletingbranchpathway,fine-tuningxyloseisomeraseexpression,andsubstitutingdecarboxylasetoimprove1,2,4-butanetriolproduction[j].journalofbioscienceandbioengineering,2018,126(5):547-552.”中,tac启动子的核苷酸序列如seqidno.7所示)经限制性内切酶xbai和ncoi酶切后进行连接,得到连接产物;将连接产物转化大肠杆菌(escherichiacoli)bl21,得到转化产物;将转化产物涂布在lb固体培养基(含有50μg·ml-1卡那霉素)上,于37℃恒温培养箱中倒置培养8~12h,得到转化子;挑取转化子接种至lb液体培养基中,于37℃、120~180rpm的条件下摇瓶培养8~12h后提取质粒进行酶切验证(验证结果见图1)以及测序验证,验证正确即获得重组质粒petac-yjhg-tac-kivd-tac-xdh(质粒图谱见图2);将获得的重组质粒petac-yjhg-tac-kivd-tac-xdh电击转化肺炎克雷伯菌(klebsiellapneumoniae)ji22914,得到转化产物;将转化产物涂布于lb固体培养基(含有10μg·ml-1卡那霉素),于37℃恒温培养箱中倒置培养36h,得到转化子;挑取转化子接种至lb液体培养基中,于37℃恒温培养箱中、120~180rpm的条件下摇瓶培养36h后提取质粒进行酶切验证以及测序验证,验证正确即获得重组肺炎克雷伯菌ji22914/petac-yjhg-tac-kivd-tac-xdh。
实施例2:1,3-丙二醇和d-1,2,4-丁三醇的联产
具体步骤如下:
以肺炎克雷伯菌(klebsiellapneumoniae)ji22914为对照,将实施例1获得的重组肺炎克雷伯菌ji22914/petac-yjhg-tac-kivd-tac-xdh的单菌落划线接种于lb固体培养基中,于37℃培养12~14h,形成单菌落;挑取单菌落接种到lb液体培养基中,37℃、100rpm下培养12h,得到一级种子液;按照1%(v/v)的接种量将一级种子液转接到新的lb液体培养基中,37℃、100rpm下培养8h,得到二级种子液;按照4%(v/v)的接种量将二级种子液转接到发酵培养基中,37℃、150rpm下震荡培养48h,得到发酵液。
检测发酵液中的生物量、甘油含量、1,3-丙二醇含量、木糖含量、d-1,2,4-丁三醇含量、2,3-丁二醇含量、琥珀酸酯含量、乳酸盐含量以及乙醇含量变化,检测结果见图3-10。计算发酵液中甘油的转化率,并且,计算发酵液中木糖的转化率;
其中,甘油的转化率=(1,3-丙二醇浓度/甘油浓度)×100%;
木糖的转化率=(d-1,2,4-丁三醇浓度/木糖浓度)×100%。
由图3-10可知,发酵48h时,重组肺炎克雷伯菌ji22914/petac-yjhg-tac-kivd-tac-xdh发酵获得的发酵液中1,3-丙二醇的产量高达18.1g/l、d-1,2,4-丁三醇的产量高达3.3g/l、甘油的转化率高达45.3%、木糖的转化率高达12.3%,而肺炎克雷伯菌(klebsiellapneumoniae)ji22914发酵获得的发酵液中1,3-丙二醇的产量仅有16g/l、d-1,2,4-丁三醇的产量为0g/l、甘油的转化率仅有40%、木糖的转化率为0%。可见,重组肺炎克雷伯菌ji22914/petac-yjhg-tac-kivd-tac-xdh可高效转化甘油和木糖联产1,3-丙二醇和d-1,2,4-丁三醇,而肺炎克雷伯菌(klebsiellapneumoniae)ji22914无此能力。
甘油的市值为6元/kg、木糖的市值为178元/kg、1,3-丙二醇的市值为20元/kg、d-1,2,4-丁三醇的市值为3720元/kg,根据产物价值计算利用重组肺炎克雷伯菌ji22914/petac-yjhg-tac-kivd-tac-xdh转化甘油和木糖联产1,3-丙二醇和d-1,2,4-丁三醇的价效,计算结果为12.31元/l,可见,利用重组肺炎克雷伯菌ji22914/petac-yjhg-tac-kivd-tac-xdh转化甘油和木糖联产1,3-丙二醇和d-1,2,4-丁三醇的利润较高。
虽然本发明已以较佳实施例公开如上,但其并非用以限定本发明,任何熟悉此技术的人,在不脱离本发明的精神和范围内,都可做各种的改动与修饰,因此本发明的保护范围应该以权利要求书所界定的为准。
序列表
<110>江南大学
<120>联产1,3-丙二醇和d-1,2,4-丁三醇的方法
<160>7
<170>patentinversion3.3
<210>1
<211>249
<212>prt
<213>新月柄杆菌
<400>1
metserseralailetyrproglnleulysglylysargvalvalile
151015
thrglyglyglyserglyileglyalaglyleuvalalaglypheval
202530
argglnglyalagluvalilepheleuaspilevalaspalaaspser
354045
glnalaleuvalalagluleuserlysasnalaalailealaproval
505560
tyrlysargcysaspleumetaspileaspalaleulysalavalphe
65707580
alagluileglyaspvalaspvalleuvalasnasnalaglyasnasp
859095
asparghisserleuseraspleuthrproglutyrtrpaspasnarg
100105110
ileglyvalasnleuarghismetvalphealaalaglnalavalala
115120125
aspglymetarglysargglyglyglyalaileileasnpheglyser
130135140
ilesertrphisleuglyleugluaspleuvalleutyrgluthrala
145150155160
lysalaglyilegluglymetserargalaleualaarggluleugly
165170175
proaspaspileargvalthrcysvalvalproglyasnvallysthr
180185190
lysargglnglulystrptyrthrprogluglyglualagluileval
195200205
lysalaglncysleulysglyargileleuproasphisvalalaser
210215220
leuvalleupheleualaseraspaspalaserleucysthrglyhis
225230235240
glutyrtrpileaspalaglytrparg
245
<210>2
<211>750
<212>dna
<213>新月柄杆菌
<400>2
atgtcctcagccatctatccccagctgaagggcaagcgcgtcgtcatcaccggcggcggt60
tcgggcatcggcgcgggcctcgtcgccggcttcgtgcgccagggcgccgaggtgatcttc120
ctcgacatcgtcgacgcggactcccaggccctggtcgccgagttgtcgaagaacgcggcc180
atcgcgcccgtctacaagcgctgcgacctgatggacatcgacgcgctgaaggcggtcttc240
gccgagatcggcgacgtcgacgttctggtcaacaacgccggcaacgacgaccgtcacagc300
ctgtcggacctgacgcccgaatactgggacaaccgcatcggcgtgaacctgcgccacatg360
gtgttcgcggcccaggccgtcgcggacggcatgaggaagcgcggcggcggcgccatcatc420
aacttcggctcgatcagctggcacctgggcctggaggacctcgtcctctacgaaaccgcc480
aaggccggcatcgagggcatgtcccgggcgctggcccgcgagctgggtcccgacgacatc540
cgcgtcacctgcgtggtgccaggcaacgtcaagaccaagcgccaggagaagtggtacacg600
ccggaaggcgaggccgagatcgtcaaggcccagtgcctgaagggccgcatcctgcccgat660
cacgtcgcttcgctggtgctgttcctggcctcggacgacgcctcgctctgcaccggtcac720
gagtactggatcgacgccggctggcgttga750
<210>3
<211>548
<212>prt
<213>乳酸乳球菌
<400>3
mettyrthrvalglyasptyrleuleuaspargleuhisgluleugly
151015
ileglugluilepheglyvalproglyasptyrasnleuglnpheleu
202530
aspglnileileserhislysaspmetlystrpvalglyasnalaasn
354045
gluleuasnalasertyrmetalaaspglytyralaargthrlyslys
505560
alaalaalapheleuthrthrpheglyvalglygluleuseralaval
65707580
asnglyleualaglysertyralagluasnleuprovalvalgluile
859095
valglyserprothrserlysvalglnasngluglylysphevalhis
100105110
histhrleualaaspglyaspphelyshisphemetlysmethisglu
115120125
provalthralaalaargthrleuleuthralagluasnalathrval
130135140
gluileaspargvalleuseralaleuleulysgluarglysproval
145150155160
tyrileasnleuprovalaspvalalaalaalalysalaglulyspro
165170175
serleuproleulyslysgluasnserthrserasnthrseraspgln
180185190
gluileleuasnlysileglngluserleulysasnalalyslyspro
195200205
ilevalilethrglyhisgluileileserpheglyleuglulysthr
210215220
valthrglnpheileserlysthrlysleuproilethrthrleuasn
225230235240
pheglylysserservalaspglualaleuproserpheleuglyile
245250255
tyrasnglythrleusergluproasnleulysgluphevalgluser
260265270
alaasppheileleumetleuglyvallysleuthraspserserthr
275280285
glyalaphethrhishisleuasngluasnlysmetileserleuasn
290295300
ileaspgluglylysilepheasngluargileglnasnpheaspphe
305310315320
gluserleuileserserleuleuaspleusergluileglutyrlys
325330335
glylystyrileasplyslysglngluaspphevalproserasnala
340345350
leuleuserglnaspargleutrpglnalavalgluasnleuthrgln
355360365
serasngluthrilevalalagluglnglythrserphepheglyala
370375380
serserilepheleulysserlysserhispheileglyglnproleu
385390395400
trpglyserileglytyrthrpheproalaalaleuglyserglnile
405410415
alaasplysgluserarghisleuleupheileglyaspglyserleu
420425430
glnleuthrvalglngluleuglyleualaileargglulysileasn
435440445
proilecyspheileileasnasnaspglytyrthrvalgluargglu
450455460
ilehisglyproasnglnsertyrasnaspilepromettrpasntyr
465470475480
serlysleuprogluserpheglyalathrgluaspargvalvalser
485490495
lysilevalargthrgluasngluphevalservalmetlysgluala
500505510
glnalaaspproasnargmettyrtrpilegluleuileleualalys
515520525
gluglyalaprolysvalleulyslysmetglylysleuphealaglu
530535540
glnasnlysser
545
<210>4
<211>1647
<212>dna
<213>乳酸乳球菌
<400>4
atgtatacagtaggagattacctattagaccgattacacgagttaggaattgaagaaatt60
tttggagtccctggagactataacttacaatttttagatcaaattatttcccacaaggat120
atgaaatgggtcggaaatgctaatgaattaaatgcttcatatatggctgatggctatgct180
cgtactaaaaaagctgccgcatttcttacaacctttggagtaggtgaattgagtgcagtt240
aatggattagcaggaagttacgccgaaaatttaccagtagtagaaatagtgggatcacct300
acatcaaaagttcaaaatgaaggaaaatttgttcatcatacgctggctgacggtgatttt360
aaacactttatgaaaatgcacgaacctgttacagcagctcgaactttactgacagcagaa420
aatgcaaccgttgaaattgaccgagtactttctgcactattaaaagaaagaaaacctgtc480
tatatcaacttaccagttgatgttgctgctgcaaaagcagagaaaccctcactccctttg540
aaaaaggaaaactcaacttcaaatacaagtgaccaagaaattttgaacaaaattcaagaa600
agcttgaaaaatgccaaaaaaccaatcgtgattacaggacatgaaataattagttttggc660
ttagaaaaaacagtcactcaatttatttcaaagacaaaactacctattacgacattaaac720
tttggtaaaagttcagttgatgaagccctcccttcatttttaggaatctataatggtaca780
ctctcagagcctaatcttaaagaattcgtggaatcagccgacttcatcttgatgcttgga840
gttaaactcacagactcttcaacaggagccttcactcatcatttaaatgaaaataaaatg900
atttcactgaatatagatgaaggaaaaatatttaacgaaagaatccaaaattttgatttt960
gaatccctcatctcctctctcttagacctaagcgaaatagaatacaaaggaaaatatatc1020
gataaaaagcaagaagactttgttccatcaaatgcgcttttatcacaagaccgcctatgg1080
caagcagttgaaaacctaactcaaagcaatgaaacaatcgttgctgaacaagggacatca1140
ttctttggcgcttcatcaattttcttaaaatcaaagagtcattttattggtcaaccctta1200
tggggatcaattggatatacattcccagcagcattaggaagccaaattgcagataaagaa1260
agcagacaccttttatttattggtgatggttcacttcaacttacagtgcaagaattagga1320
ttagcaatcagagaaaaaattaatccaatttgctttattatcaataatgatggttataca1380
gtcgaaagagaaattcatggaccaaatcaaagctacaatgatattccaatgtggaattac1440
tcaaaattaccagaatcgtttggagcaacagaagatcgagtagtctcaaaaatcgttaga1500
actgaaaatgaatttgtgtctgtcatgaaagaagctcaagcagatccaaatagaatgtac1560
tggattgagttaattttggcaaaagaaggtgcaccaaaagtactgaaaaaaatgggcaaa1620
ctatttgctgaacaaaataaatcataa1647
<210>5
<211>655
<212>prt
<213>大肠杆菌
<400>5
metservalargasnilephealaaspgluserhisaspiletyrthr
151015
valargthrhisalaaspglyproaspglygluleuproleuthrala
202530
glumetleuileasnargproserglyaspleupheglymetthrmet
354045
asnalaglymetglytrpserproaspgluleuaspargaspglyile
505560
leuleuleuserthrleuglyglyleuargglyalaaspglylyspro
65707580
valalaleualaleuhisglnglyhistyrgluleuaspileglnmet
859095
lysalaalaalagluvalilelysalaasnhisalaleuprotyrala
100105110
valtyrvalseraspprocysaspglyargthrglnglythrthrgly
115120125
metpheaspserleuprotyrargasnaspalasermetvalmetarg
130135140
argleuileargserleuproaspalalysalavalileglyvalala
145150155160
sercysasplysglyleuproalathrmetmetalaleualaalagln
165170175
hisasnilealathrvalleuvalproglyglyalathrleuproala
180185190
lysaspglygluaspasnglylysvalglnthrileglyalaargphe
195200205
alaasnglygluleuserleuglnaspalaargargalaglycyslys
210215220
alacysalaserserglyglyglycysglnpheleuglythralagly
225230235240
thrserglnvalvalalagluglyleuglyleualaileprohisser
245250255
alaleualaproserglygluprovaltrparggluilealaargala
260265270
seralaargalaalaleuasnleuserglnlysglyilethrthrarg
275280285
gluileleuthrasplysalailegluasnalametthrvalhisala
290295300
alapheglyglyserthrasnleuleuleuhisileproalaileala
305310315320
hisglnalaglycyshisileprothrvalaspasptrpileargile
325330335
asnlysargvalproargleuvalservalleuproasnglyproval
340345350
tyrhisprothrvalasnalaphemetalaglyglyvalprogluval
355360365
metleuhisleuargserleuglyleuleuhisgluaspvalmetthr
370375380
valthrglyserthrleulysgluasnleuasptrptrpgluhisser
385390395400
gluargargglnargphelysglnleuleuleuaspglngluglnile
405410415
asnalaaspgluvalilemetserproglnglnalalysalaarggly
420425430
leuthrserthrilethrpheprovalglyasnilealaproglugly
435440445
servalilelysserthralaileaspprosermetileaspglugln
450455460
glyiletyrtyrhislysglyvalalalysvaltyrleuserglulys
465470475480
seralailetyraspilelyshisasplysilelysalaglyaspile
485490495
leuvalileileglyvalglyproserglythrglymetglugluthr
500505510
tyrglnvalthrseralaleulyshisleusertyrglylyshisval
515520525
serleuilethraspalaargpheserglyvalserthrglyalacys
530535540
ileglyhisvalglyproglualaleualaglyglyproileglylys
545550555560
leuargthrglyaspleuilegluilelysileaspcysarggluleu
565570575
hisglygluvalasnpheleuglythrargseraspgluglnleupro
580585590
serglngluglualathralaileleuasnalaargproserhisgln
595600605
aspleuleuproaspprogluleuproaspaspthrargleutrpala
610615620
metleuglnalavalserglyglythrtrpthrglycysiletyrasp
625630635640
valasnlysileglyalaalaleuargaspphemetasnlysasn
645650655
<210>6
<211>1968
<212>dna
<213>大肠杆菌
<400>6
atgtctgttcgcaatatttttgctgacgagagccacgatatttacaccgtcagaacgcac60
gccgatggcccggacggcgaactcccattaaccgcagagatgcttatcaaccgcccgagc120
ggggatctgttcggtatgaccatgaatgccggaatgggttggtctccggacgagctggat180
cgggacggtattttactgctcagtacactcggtggcttacgcggcgcagacggtaaaccc240
gtggcgctggcgttgcaccaggggcattacgaactggacatccagatgaaagcggcggcc300
gaggttattaaagccaaccatgccctgccctatgccgtgtacgtctccgatccttgtgac360
gggcgtactcagggtacaacggggatgtttgattcgctaccataccgaaatgacgcatcg420
atggtaatgcgccgccttattcgctctctgcccgacgcgaaagcagttattggtgtggcg480
agttgcgataaggggcttccggccaccatgatggcactcgccgcgcagcacaacatcgca540
accgtgctggtccccggcggcgcgacgctgcccgcaaaggatggagaagacaacggcaag600
gtgcaaaccattggcgcacgcttcgccaatggcgaattatctctacaggacgcacgccgt660
gcgggctgtaaagcctgtgcctcttccggcggcggctgtcaatttttgggcactgccggg720
acatctcaggtggtggccgaaggattgggactggcaatcccacattcagccctggcccct780
tccggtgagcctgtgtggcgggagatcgccagagcttccgcgcgagctgcgctgaacctg840
agtcaaaaaggcatcaccacccgggaaattctcaccgataaagcgatagagaatgcgatg900
acggtccatgccgcgttcggtggttcaacaaacctgctgttacacatcccggcaattgct960
caccaggcaggttgccatatcccgaccgttgatgactggatccgcatcaacaagcgcgtg1020
ccccgactggtgagcgtactgcctaatggcccggtttatcatccaacggtcaatgccttt1080
atggcaggtggtgtgccggaagtcatgttgcatctgcgcagcctcggattgttgcatgaa1140
gacgttatgacggttaccggcagcacgctgaaagaaaacctcgactggtgggagcactcc1200
gaacggcgtcagcggttcaagcaactcctgctcgatcaggaacaaatcaacgctgacgaa1260
gtgatcatgtctccgcagcaagcaaaagcgcgcggattaacctcaactatcaccttcccg1320
gtgggcaatattgcgccagaaggttcggtgatcaaatccaccgccattgacccctcgatg1380
attgatgagcaaggtatctattaccataaaggtgtggcgaaggtttatctgtccgagaaa1440
agtgcgatttacgatatcaaacatgacaagatcaaggcgggcgatattctggtcattatt1500
ggcgttggaccttcaggtacagggatggaagaaacctaccaggttaccagtgccctgaag1560
catctgtcatacggtaagcatgtttcgttaatcaccgatgcacgtttctcgggcgtttct1620
actggcgcgtgcatcggccatgtggggccagaagcgctggccggaggccccatcggtaaa1680
ttacgcaccggggatttaattgaaattaaaattgattgtcgcgagcttcacggcgaagtc1740
aatttcctcggaacccgtagcgatgaacaattaccttcacaggaggaggcaactgcaata1800
ttaaatgccagacccagccatcaggatttacttcccgatcctgaattgccagatgatacc1860
cggctatgggcaatgcttcaggccgtgagtggtgggacatggaccggttgtatttatgat1920
gtaaacaaaattggcgcggctttgcgcgattttatgaataaaaactga1968
<210>7
<211>253
<212>dna
<213>人工序列
<400>7
ggagcttatcgactgcacggtgcaccaatgcttctggcgtcaggcagccatcggaagctg60
tggtatggctgtgcaggtcgtaaatcactgcataattcgtgtcgctcaaggcgcactccc120
gttctggataatgttttttgcgccgacatcataacggttctggcaaatattctgaaatga180
gctgttgacaattaatcatcggctcgtataatgtgtggaattgtgagcggataacaattt240
cacacaggaaaca253
- 还没有人留言评论。精彩留言会获得点赞!