核酸扩增方法及其应用与流程
本公开涉及核酸扩增方法,尤其是利用切刻内切酶的恒温核酸扩增方法。本公开还涉及该核酸扩增方法在细菌或病毒检测方面的应用。
背景技术:
核酸扩增技术在生化分析、分子诊断、食品安全等领域具有重要意义。近几年,核酸扩增技术飞速发展,尤其是由dr.mullis于1983年发明的聚合酶链式反应(polymerasechainreaction,pcr)技术已是目前应用最为广泛的核酸扩增技术。pcr技术有三个基本步骤,即:变性、退火和延伸,这使得pcr扩增需要反复升降温过程,从而对仪器设备要求十分严苛,同时pcr技术也需要熟练的专业技术人员在实验室中进行操作,这些都限制了pcr技术的广泛应用。近些年新开发出的恒温核酸扩增技术克服了pcr技术的一些缺陷,例如其不需要反复升降温,降低了对仪器设备的要求,缩短了反应时间。目前,国内外已报道了多种恒温核酸扩增技术,其中以重组酶聚合酶扩增(recombinasepolymeraseamplification,rpa)、解旋酶依赖性恒温dna扩增(helicase-dependentisothermaldnaamplification,hda)、环介导恒温扩增(loop-mediatedisothermalamplification,lamp)、滚环扩增(rollingcircleamplification,rca)和指数扩增反应(exponentialamplificationreaction,expar)等为代表。但是,这些恒温核酸扩增技术也还存在扩增速度慢、扩增片段短、成本较高或操作不够简便等方面的问题。
技术实现要素:
为了克服上述问题,在一方面,本公开提供了一种核酸扩增方法,其包括向扩增反应体系中加入以下组分:
1)包括待扩增靶片段的靶核酸,所述待扩增靶片段从5’端至3’端依次包括5’端序列、中间序列和3’端序列;
2)切刻内切酶;
3)dna聚合酶,其具有链置换活性,缺乏5’→3’外切酶活性;以及
4)核心引物对,所述核心引物对包括:
a)正向核心引物,其从5’端至3’端依次包括正向核心引物第一序列、正向核心引物第二序列和正向核心引物第三序列,其中所述正向核心引物第一序列和所述正向核心引物第二序列的连接处被设计为所述切刻内切酶的切割位点;所述正向核心引物第三序列的核苷酸序列与所述待扩增片段的5’端序列相同;和
b)反向核心引物,其从5’端至3’端依次包括反向核心引物第一序列和反向核心引物第二序列,所述反向核心引物第一序列的核苷酸序列与所述正向核心引物第二序列相同或者为所述正向核心引物第二序列的3’端序列的一部分;所述反向核心引物第二序列的核苷酸序列与所述待扩增片段的3’端序列反向互补。
在一些实施方案中,所述方法还包括向所述扩增反应体系中加入置换引物对,所述置换引物对包括:
a)正向置换引物,其核苷酸序列与所述靶核酸中所述待扩增靶片段的上游序列的一部分相同;和
b)反向置换引物,其核苷酸序列与所述靶核酸中所述待扩增靶片段的下游序列的一部分反向互补。
在一些实施方案中,所述方法在恒温情况下进行。
在一些实施方案中,所述待扩增靶片段的长度为20至1000个碱基。优选地,所述待扩增靶片段的长度为40至400个碱基。
在一些实施方案中,所述方法生成的扩增产物包括双链dna分子和茎环结构dna分子。
在一些实施方案中,所述双链dna分子的一条链从5’端到3’端依次包括:
所述正向核心引物第二序列;
所述待扩增靶片段;以及
所述正向核心引物第二序列的反向互补序列。
在一些实施方案中,所述茎环结构dna分子包括:
所述正向核心引物第二序列;
所述待扩增靶片段或其互补序列;以及
所述正向核心引物第二序列的反向互补序列;
其中所述正向核心引物第二序列和所述正向核心引物第二序列的反向互补序列形成茎部,所述待扩增靶片段或其互补序列形成环部。
在一些实施方案中,所述茎环结构dna分子的tm值不低于所述扩增反应体系的反应温度。
在一些实施方案中,所述正向核心引物与所述靶核酸的互补链结合形成的双链的tm值不小于所述正向置换引物与所述靶核酸的互补链结合形成的双链的tm值,并且所述反向核心引物与所述靶核酸链结合形成的双链的tm值不小于所述反向置换引物与所述靶核酸链结合形成的双链的tm值。
在一些实施方案中,所述正向核心引物第一序列与其互补链形成的双链的tm值不小于正向核心引物第二序列与其互补链形成的双链的tm值,并且这二者都不小于所述扩增反应体系的反应温度。
在一些实施方案中,所述方法还包括向所述反应体系中加入核酸染料或荧光探针,用于监测扩增反应进展。
在一些实施方案中,所述荧光探针为dna分子信标、pna分子信标或双链探针。
在一些实施方案中,所述双链探针包括dna长链和dna短链,所述dna短链与所述dna长链的3’端序列或5’端序列互补,并且在所述dna长链和dna短链的两互补末端上分别连接有荧光分子和淬灭分子。
在一些实施方案中,所述荧光探针包括与所述茎环结构产物dna分子的环部互补的序列。
在一些实施方案中,所述靶核酸在所述待扩增靶片段的上游具有所述切刻内切酶的识别位点。
在一些实施方案中,所述靶核酸为单链或双链dna。
在一些实施方案中,所述靶核酸为rna,并且所述dna聚合酶具有反转录酶活性。
另一方面,本公开提供了一种检测样品中是否存在靶核酸的方法,包括以本公开的扩增所述靶核酸的特异核苷酸序列,其中能够获得所述特异核苷酸序列的扩增产物表明所述样品中存在所述靶核酸。
另一方面,本公开提供了一种检测样品中是否存在细菌或病毒的方法,包括:
1)从所述样品中提取核酸;以及
2)以本公开的方法扩增所述细菌或病毒的特异核苷酸序列,
其中能够获得所述特异核苷酸序列的扩增产物表明所述样品中存在所述细菌或病毒。
本文提供的核酸扩增方法利用特别设计的引物对,联合dna聚合酶和切刻内切酶,在恒温条件下即可实现靶核酸的指数扩增,操作简便,反应快速,灵敏度高。扩增产物为包括待扩增片段的双链dna分子和单链茎环结构dna分子,能够与多种检测方式联合使用,方便地应用于疾病诊断或病原体的检测。
附图说明
图1为本发明的核酸扩增方法的优选实施方案的扩增原理示意图。
图2为利用本发明的扩增方法扩增肺炎支原体核酸片段的一个实施例的扩增过程曲线图。
图3为利用本发明的扩增方法扩增肺炎支原体核酸片段的另一个实施例的扩增过程曲线图。该实施例不使用置换引物。
图4为利用本发明的扩增方法扩增肺炎支原体核酸片段的另一个实施例的扩增过程曲线图。该实施例采用双链dna探针进行扩增过程的监测。
图5为利用本发明的扩增方法扩增乙肝病毒核酸的一个实施例的扩增过程曲线图。
图6为利用本发明的扩增方法扩增甲型h1n1流感病毒核酸的一个实施例的扩增过程曲线图。
图7为对于特定序列利用软件预测的二级结构示意图。
具体实施方式
除非另有说明,本文使用的技术和科学术语具有本领域技术人员所通常理解的含义。除非另有说明,本文采用的分子生物学、基因工程等基本操作为本领域技术人员所公知的常规生物学技术。除非另有说明,本发明所使用的试验材料可从普通生化试剂公司购得。
用在本文时,术语“待扩增靶片段”指准备采用本公开提供的扩增方法对其进行扩增的目的核苷酸序列,其通常为更长的核酸分子(即靶核酸)的一部分。该靶核酸可以为双链dna分子、单链dna分子或单链rna分子。出于方便描述扩增反应的目的,在下文中将双链dna分子的一条链(例如图1中双链分子的上方链)称为靶核酸,将与其互补结合另一条链称为其互补链。对于单链dna分子和rna分子,靶核酸可以是这些单链dna分子和rna分子本身,或者是其互补链。同样出于方便描述的目的,还将待扩增靶片段从5’端至3’端分为三段:5’端序列、中间序列和3’端序列,其中5’端序列和3’端序列或它们的互补链可以与扩增引物结合。中间序列的长度可以为数个到数千个碱基长度。或者在一些情况下,5’端序列和3’端序列直接连接,即不存在中间序列。
用在本文时,术语“上游序列”指核酸链中位于特定核苷酸序列5’端方向上核苷酸序列,其用于表示核苷酸序列在核酸链中的相对位置关系。例如,当提到待扩增靶片段的“上游序列”时,表明该上游序列在靶核酸中处于该靶核酸的5’末端到该待扩增靶片段的5’末端之间。相应地,将核酸链中位于特定核苷酸序列3’端方向上核苷酸序列称为“下游序列”。
用在本文时,术语“切刻内切酶”指一种核酸内切酶,与常用的核酸内切酶在dna分子双链上都形成切口不同,其仅在识别位点附近于双链dna分子的一条链上形成切口。这些酶可以在市场上从供应商购得。
用在本文时,术语“核心引物”指用于和待扩增靶片段互补结合的核苷酸片段。核心引物成对使用,其中能够与待扩增靶片段的互补链的3’端序列结合的核心引物称为“正向核心引物”,其具有与待扩增片段的5’端序列相同的核苷酸序列;能够与待扩增靶片段的3’端序列结合的核心引物称为“反向核心引物”,其具有与待扩增片段的互补链的5’端序列相同的核苷酸序列。本公开中使用的正向核心引物从5’端至3’端依次包括正向核心引物第一序列、正向核心引物第二序列和正向核心引物第三序列,其中正向核心引物第三序列用于与待扩增靶片段的互补链的3’端序列结合,而正向核心引物第一序列和正向核心引物第二序列被设计为它们的连接处为某一切刻内切酶的切割位点。当正向核心引物第一序列和正向核心引物第二序列处于与它们的互补链结合的双链dna分子形式时,该切刻内切酶可以识别正向核心引物第一序列内的、正向核心引物第二序列内的或者它们的结合而形成的切刻内切酶识别位点,并且在正向核心引物第一序列和正向核心引物第二序列的连接处产生切口。本公开中使用的反向核心引物从5’端至3’端依次包括反向核心引物第一序列和反向核心引物第二序列,其中反向核心引物第二序列用于与待扩增靶片段的3’端序列结合,反向核心引物第一序列的核苷酸序列被设计为与正向核心引物第二序列相同或者为正向核心引物第二序列的3’端序列的一部分。
用在本文时,术语“置换引物”指能够与靶核酸中待扩增靶片段或其互补链的上游序列的一部分结合的引物。置换引物通常成对使用,其中用于与靶核酸中待扩增靶片段的互补链的下游序列的一部分结合的置换引物称为正向置换引物,其具有与靶核酸中待扩增靶片段的上游序列的一部分相同的核苷酸序列;用于与靶核酸中待扩增靶片段的下游序列的一部分结合的置换引物称为反向置换引物,其具有与靶核酸中待扩增靶片段的互补链的上游序列的一部分相同的核苷酸序列。在本公开的扩增方法中,置换引物的使用有利于在扩增反应初期使由核心引物扩增的延伸链与模板链脱离。在一些实施方案中,可以不使用置换引物,但这有可能会延长扩增反应时间。除此之外,还可以考虑仅使用正向置换引物或者反向置换引物。
用在本文时,术语“互补”指核苷酸之间的配对结合,包括核苷酸a与t(或u)以及c与g的结合。“互补链”指在一条核酸链基础上通过这种配对方式给出的另一条链。
提及靶核酸、细菌或病毒核酸时,术语“特异核苷酸序列”指能够用于将该靶核酸、细菌或病毒核酸与其它核酸分子区分开的核苷酸序列,尤其是考虑其中用于与核心引物结合的序列。一般地,可以选择该靶核酸、或细菌或病毒核酸的特有序列作为“特异核苷酸序列”。
提及核酸链时,术语“茎环结构”指dna单链自身通过5’端碱基与3’端碱基反向互补配对而形成的二级结构。通过碱基配对形成的双链部分为“茎部”,配对碱基之间的序列则形成“环部”。在某些情况下,该茎环结构还可以具有末端突出。
用在本文时,术语“tm值”指dna分子的双螺旋结构解离一半时的温度。就引物与模板的结合而言,tm值一般指在模板过量情况下有50%的引物与模板配对结合,而另外50%引物处于解离状态时的温度。对于茎环结构的单链dna分子而言,tm值指50%的单链dna分子为茎环结构形式而另外50%的单链dna分子为线性形式时的温度。一般地,其gc含量越高,tm值越大。tm值还与溶液中离子强度、ph值、是否存在变性剂、以及dna分子的长度等有关。本公开所使用的核心引物和置换引物以及茎环结构的tm值可以使用nupack(http://www.nupack.org/partition/new)进行二级结构模拟及tm值估算,以及使用quikfold(http://unafold.rna.albany.edu/?q=dinamelt/quickfold)进行tm值估算。本公开中提到的茎环结构的优选形式为只存在一种稳定状态并且其tm值高于反应温度的结构。例如,对于以下序列(下文实施例1中扩增过程产生的中间产物(对应图1中茎环结构v)):5’-cgtagtgtagagagtcacatacgcattcatcgagtgtctgctacacctttgcgttacgccggtatgacctcgccgggcgcgccttatacgacctcgatttttcgaagttaaacccgcaaacgcccacgtagcagacactcgatg-3’(seqidno:1),利用quikfold对其进行二级结构模拟及tm值估算,在na+离子浓度为100mm,mg2+离子浓度为4mm和反应温度为65℃的条件下,测试仅得到一种结构状态(见图7),并且其tm=72.9℃,高于反应温度65℃。
本发明核酸扩增反应原理
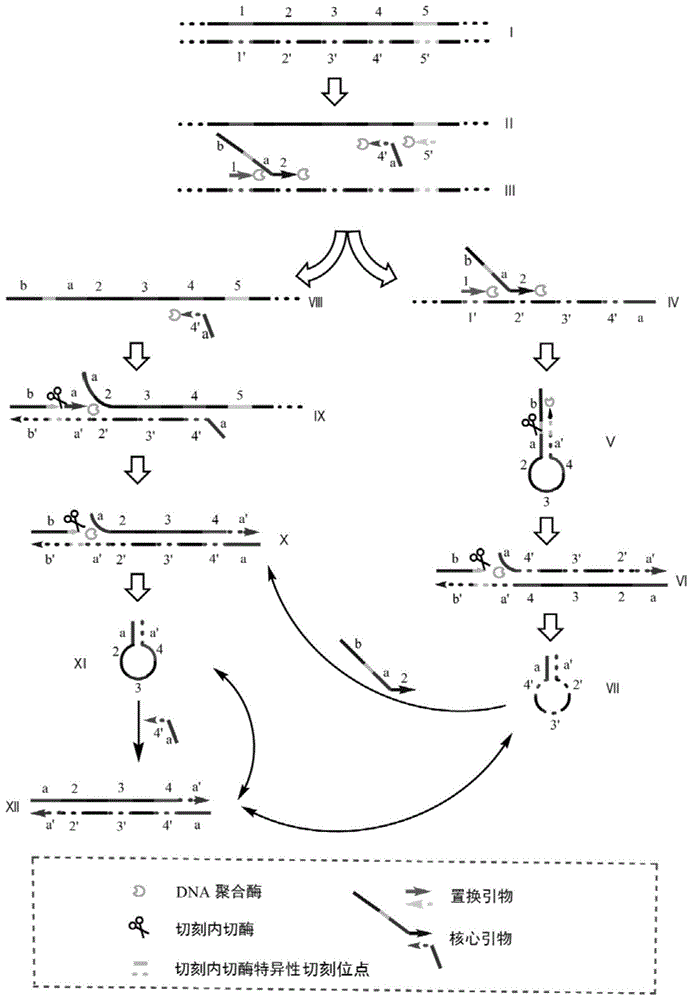
现结合图1来详细说明本发明的恒温扩增方法。为了方便阐述,在图中以阿拉伯数字和小写字母代表单链核酸序列,并且以加撇(’)的和不加撇相应数字或字母表示互补序列,例如序列1’为序列1的互补序列,序列a为序列a’的互补序列。以1、2、3、4、和5分别代表靶核酸(i)的正链(ii)连续序列,而加撇的数字1’、2’、3’、4’、和5’分别代表相应的互补负链(iii)连续序列。靶核酸中待扩增靶片段为序列2、3、和4(或序列2’、3’、和4’)。
本发明的核酸扩增方法中使用两组引物。一组为核心引物,其包括正向核心引物和反向核心引物。如图1中所示,正向核心引物3’端为序列2,其与双链dna分子(i)上的负链(iii)序列2’互补;5’端为序列b;序列b和序列2之间为序列a,并且序列b和序列a的邻接区域在双链情况下形成切刻内切酶识别序列,使得切刻内切酶可以在序列b和序列a的连接处生成切口。负向核心引物从5’端至3’端依次为序列a和序列4’,序列4’与双链dna分子(i)上的正链(ii)序列4互补。另一组引物为置换引物,其包括正向置换引物和反向置换引物。正向置换引物包括序列1,反向置换引物包括序列5’。
扩增反应的初始步骤为各种引物与靶核酸上的互补序列结合。通过调整反应温度、利用解旋酶或者重组酶或者利用dna呼吸作用使局部靶核苷酸序列双链dna打开,从而使引物与靶核酸上的互补序列结合,随后通过具有链置换活性的dna聚合酶聚合延伸而生成双链dna分子。下面先描述反向核心引物与靶核酸正链(ii)结合并聚合延伸的情况。反向核心引物通过序列4’与靶核酸正链(ii)的序列4结合,在dna聚合酶作用下聚合延伸。反向置换引物与靶核酸正链(ii)的序列5结合,在dna聚合酶作用下聚合延伸。就反向核心引物和反向置换引物的聚合延伸反应而言,反向置换引物的结合序列(序列5)处于反向核心引物的结合序列(序列4)的上游,所以反向置换引物的聚合延伸可以将反向核心引物聚合延伸形成的单链置换下来。该置换下来的单链(iv)包括序列a-4’-3’-2’-1’…(从5’端到3’端),可以参与后续描述的反应步骤(参见图1中分叉箭头右侧及以下部分)。正向核心引物通过序列2与该剥离下来的单链(iv)的序列2’结合,并在dna聚合酶作用下聚合延伸。正向置换引物与该剥离下来的单链(iv)的序列1’结合,并在dna聚合酶作用下聚合延伸,将正向核心引物聚合延伸形成的单链(b-a-2-3-4-a’(从5’端到3’端))置换下来。该置换下来的单链的3’端序列a’与靠近5’端的序列a互补,可以形成带有5’端突出末端(即序列b)的茎环结构(v)。该茎环结构(v)的3’端可以在dna聚合酶作用下以序列b为模板延伸补齐,形成能够被切刻内切酶识别的双链。随后,切刻内切酶在茎环结构(v)的序列b和序列a之间产生切口,在dna聚合酶作用下,序列b的3’末端按顺序以茎部的序列a’,环部的序列4、3、2,以及茎部的序列a为模板进行延伸,形成双链dna分子(vi)。该双链dna分子(vi)上形成的切刻酶识别序列又可被切刻酶识别。之后,在切刻酶和dna聚合酶的作用下,反复在双链dna分子(vi)的序列b和序列a之间产生切口,并在切口的3’末端延伸形成新的延伸链,不断地置换双链dna分子上的旧链(相对于正在合成的延伸链而言)。被置换下的dna单链具有a-4’-3’-2’-a’(从5’端到3’端)的序列,其末端的序列a和序列a’能够反向互补配对而形成茎环结构(vii)。茎环结构(vii)可与正向核心引物互补配对,在dna聚合酶作用下以茎环结构(vii)的序列为模板聚合延伸,同时茎环结构(vii)的3’末端也可以在聚合酶作用下以正向核心引物的序列b为模板聚合延伸。这样形成的双链dna分子(x)也带有切刻酶识别序列,在切刻酶和dna聚合酶的作用下,切口处新合成的延伸链不断置换双链上的旧链。被置换下的旧链具有a-2-3-4-a’(从5’端到3’端)的序列,其通过末端的序列a和序列a’反向互补而形成茎环结构(xi)。茎环结构(xi)可以与反向核心引物互补配对,在dna聚合酶作用下形成双链dna分子(xii)。在扩增反应体系中,茎环结构(vii)可以与茎环结构(xi)互补配对,也形成扩增产物双链dna分子(xii)。在扩增反应条件下,茎环结构(vii)和(xi)通常与扩增产物双链dna分子(xii)处于动态平衡中。
对于正向核心引物与负链(iii)结合并聚合延伸的情况,描述如下。正向核心引物通过序列2与负链(iii)的序列2’结合,在dna聚合酶作用下聚合延伸。正向置换引物与负链(iii)的序列1’结合,在dna聚合酶作用下聚合延伸。该正向置换引物的聚合延伸可以将正向核心引物聚合延伸形成的单链置换下来。该置换下来的单链(viii)包括序列b-a-2-3-4-5…(从5’端到3’端),可以参与后续描述的步骤(参见图1中分叉箭头左侧及以下部分)。反向核心引物通过序列4’与该置换下来的单链(viii)的序列4结合,并在dna聚合酶作用下聚合延伸,形成的不完全互补的双链产物(ix),该双链上具有切刻酶识别位点,在切刻酶作用下在序列b和序列a之间产生切口。序列b的3’端在dna聚合酶作用下聚合延伸,同时将旧链a-2-3-4-5…剥离下来,生成具有完全互补序列的双链dna分子(x)。该双链dna分子(x)随后可参与上文描述过的剩余扩增过程。
由于反应过程中可以分别从双链dna分子(x)和双链dna分子(vi)不断产生茎环结构(xi)和(vii),并且扩增反应产物(xii)也处于与茎环结构(xi)和(vii)的动态平衡中,导致反应体系中实际上存在有越来越多的模板链,从而实现了对靶核酸的高效快速扩增。
在一些实施方案中,可以不使用置换引物。置换引物参与本发明核酸扩增方法的起始阶段的反应过程。在一些反应条件下,例如,通过调整反应体系的离子强度、反应温度、向反应体系中添加能够稳定单链dna分子的蛋白等,核心引物的延伸链能够相对容易地从模板链脱落,从而参与本发明扩增反应的后续扩增步骤。
在一些实施方案中,负向核心引物的序列a可以为正向核心引物的序列a的一部分,即这两个片段不一定等长,例如负向核心引物的序列a可以仅为正向核酸引物的序列a的3’端部分序列,只要能够在扩增反应体系中形成茎环结构(v、vii和xi)即可。这是由于反应体系中存在的dna聚合酶可以以正向核心引物的序列a为模板将不足的部分补齐。
在一些优选的实施方案中,茎环结构的tm值不小于反应温度,从而保证反应过程中茎环结构的顺利形成。在负向核心引物的序列a可以仅为正向核心引物的序列a的3’端部分序列时,考虑所形成的带有突出末端的茎环结构的tm值也不小于反应温度。
在一些优选的实施方案中,正向核心引物与靶核酸的互补链结合形成的双链的tm值不小于正向置换引物与靶核酸的互补链结合形成的双链的tm值,反向核心引物与靶核酸结合形成的双链的tm值不小于反向置换引物与靶核酸结合形成的双链的tm值,从而使得核心引物能够先于置换引物结合于模板序列上进行扩增延伸。
在一些优选的实施方案中,序列b与b’形成的双链dna分子的tm值不小于序列a与a’形成的双链dna分子的tm值,并且都高于扩增体系的反应温度。b与b’形成的双链结构稳定时,切刻内切酶在序列b和序列a之间产生切口后,序列b不易从其配对链解离脱落,从而能够进行后续的链延伸;a与a’形成的双链dna的tm值低于b与b’形成的双链dna的tm值有利于链置换,而高于反应温度,主要是考虑到茎环结构的稳定性。
在一些实施方案中,可以向扩增反应体系中加入荧光染料或荧光探针,用于监测反应进展情况。常用的荧光染料的例子包括sybrgreen和evagreen核酸染料。用于本发明扩增方法的荧光探针例如可以为:dna分子信标(molecularbeacon)或pna(肽核酸)分子信标,优选其环部包含一段与核酸扩增产物茎环结构的环部(或其一部分)互补的片段;或双链dna探针,其由长短两条互补链构成,长链与扩增产物茎环结构的环部(或其一部分)完全互补,短链与长链的3’端或5’端序列互补,荧光分子和淬灭分子分别结合在长链和短链的两互补末端上(如实施例中所采用的探针)。与上述分子信标相比,该双链探针的灵敏性和特异性均有提高。另外,这些探针序列能够方便地与扩增产物中茎环结构的环部单链杂交,有利于提高检测的灵敏度和特异性。
本公开还考虑,如果在靶核酸内待扩增靶片段的上游也具有上述切刻内切酶的识别位点(例如通过特意引入该切刻内切酶识别位点),则在扩增反应开始阶段,切刻内切酶即会不断识别并切割靶核酸,在dna聚合酶作用下不断形成包括待扩增靶序列的dna单链,该dna单链可直接参与本发明核酸扩增的主要过程,从而达到更为高效快速地实现核酸扩增的目的。
从上文描述的扩增过程可知,作为扩增模板的核酸分子可以是单链或双链,并且在具有逆转录活性的dna聚合酶情况下可以以rna作为模板链。
对本领域技术人员而言显而易见的是,扩增反应体系中还可以包括扩增反应通常使用的其它试剂,例如dddntps、多种金属离子、各种添加剂,等等。除此以外,一些添加剂,例如甜菜碱、甲酰胺、二甲基亚砜、二硫苏糖醇、脯氨酸、聚乙二醇、牛血清白蛋白等,可以用来优化本发明的扩增条件,从而提高核酸扩增反应的反应速度、扩增效率、特异性、灵敏度等。例如,甜菜碱能够提高扩增的效率和特异性,保护酶活;甲酰胺能够提高反应的特异性;二甲基亚砜能够降低非特异性扩增。
本发明的核酸扩增方法可以根据实际需求选择反应温度、dna聚合酶和切刻内切酶以及其他组分。例如,反应温度为65℃时,可利用dna呼吸作用,使核心引物与靶核酸的互补序列区域形成的变性泡结合,随后在dna聚合酶和切刻内切酶作用下达到对靶核酸扩增的目的;反应温度为37℃时,可以利用重组酶或解旋酶及单链结合蛋白打开双链,实现最初的核心引物与靶核酸的互补结合和聚合延伸,随后依靠dna聚合酶和切刻内切酶的联合作用实现对靶核酸的快速扩增。
下面将结合具体的实施例对本发明作更详细的解释说明,使本发明的目的、技术方案及效果更为清楚明确。以下实施例仅作为示例说明,对本发明保护范围不构成任何限定。
实施例1.扩增肺炎支原体核酸片段,利用核酸染料监测扩增过程
本实施例将合成的含肺炎支原体核酸片段的质粒作为靶核酸,利用核心引物对、置换引物对、dna聚合酶、切刻内切酶等组分进行恒温扩增,通过检测反应体系中evagreen核酸染料的荧光信号来监测扩增过程。所合成的肺炎支原体核酸片段的序列为:
ggactcaccgtagtgggacacttcacaagtaccaccacgacgctcaagcgccagcaatttagctacacccgccctgacgaggtcgcgctgcgccacaccaatgccatcaacccgcgcttaaccccgtgaacgtatcgtaacacgagcttttcctccctccccctcacgggtgaaaatcccggggcgtgggccttagtgcgcgacaacagcgctaagggcatcactgccggcagtggcagtcaacaaaccacgtatgatcccacccgaaccgaagcggctttgaccgcatcaaccacctttgcgttacgccggtatgacctcgccgggcgcgccttatacgacctcgatttttcgaagttaaacccgcaaacgcccacgcgcgaccaaaccgggcagatcacctttaacccctttggcggctttggtttgagtggggctgcaccccaacagtgaaacgaggtcaaaaacaaggtccccgtcgaggtggcgcaagacccctccaatccttatcggtttgccgttttactcgtgccgcgtagcgtggtgtactatgagcagttgca(seqidno:2)。
为了方便阅读,对该核酸片段(以及下文的其他核酸片段)的一些序列用下划线或黑体进行了标识,具体见下文结合核心引物序列和置换引物序列进行的说明。
向扩增反应体系分别加入浓度为0.1ng/μl的包含肺炎支原体核酸片段的puc57质粒1μl,或者加入双蒸水(ddh2o)1μl作为无模板对照(notemplatecontrol,ntc)。该扩增反应体系还包括50mmtris-hcl,100mmnacl,10mm(nh4)2so4,2mmmgso4,2mmmgac2,100μg/mlbsa,0.1%tritonx-100(ph8.0@25℃),6mmdntps,正向核心引物和反向核心引物各1μm,正向置换引物和反向置换引物各0.5μm,0.5×evagreen核酸染料,4ubstdna聚合酶,8unb.bsmi切刻内切酶,用ddh2o补足成10μl。
所用的正向核心引物的序列为:
让扩增反应在65℃进行30min,利用宏石实时荧光定量pcr仪每分钟检测一次荧光信号,结果显示在图2中。图2中
对扩增产物进行测序,测序结果表明扩增产物的序列为:
实施例2.不使用置换引物扩增肺炎支原体核酸片段,利用核酸染料监测扩增过程
重复实施例1的扩增反应,区别是不添加置换引物,反应结果显示在图3中。其中,
由图3可知,不使用置换引物时,扩增反应仍可以进行,只是在时间上延长了几分钟。对扩增产物进行测序,得到的测序结果与实施例1相同。
实施例3.扩增肺炎支原体核酸片段,利用双链荧光探针监测扩增过程
重复实施例1的扩增反应,以0.1μm的双链荧光探针代替核酸染料来监测反应过程。
所用的探针为:
fam-5′aaaaatcgaggtcgtataaggcgcgcccggcgag-3′-p(seqidno:8)
bhq1-3′-tttttagctccagcatattccgc-5′(seqidno:9),
其中长链序列与肺炎支原体核酸片段(seqidno:1)中以黑体显示的序列反向互补。
让反应在65℃进行45分钟,利用宏石实时荧光定量pcr仪每分钟检测一次荧光信号,结果显示在图4中。其中,
实施例4.检测乙肝病毒
采用病毒核酸提取试剂盒(qiagen)对含有乙肝病毒的血清样品进行核酸提取,以不含乙肝病毒的血清样品作为阴性对照。其中该乙肝病毒靶核酸序列为:
5’-ttgttgacaagaatcctcacaataccacagagtctaga
向扩增反应体系添加提取的乙肝病毒dna1μl(经qubit测定dna浓度为16.3ng/μl),以添加不含乙肝病毒的提取液1μl作为阴性对照。
该扩增反应体系还包括50mmtris-hcl,75mmkac,10mmmgac2,10mmdtt,100μg/mlbsa,(ph7.9@25℃),6mmdntps,正向和反向核心引物各1μm,正向和反向置换引物各0.5μm,0.5μmpna分子信标探针,4ubsudna聚合酶,8unb.bsssi切刻内切酶,用ddh2o补足成10μl。
所用的正向核心引物序列为:
反应在37℃进行40分钟,利用宏石实时荧光定量pcr仪每分钟检测一次荧光信号,结果显示在图5中。其中
实施例5.检测甲型h1n1流感病毒
采用病毒rna提取试剂盒(qiaampviralrnaminikit,qiagen)对甲型h1n1流感样品进行核酸提取。采用不含甲型h1n1流感病毒的样品作为阴性对照。其中甲型h1n1流感病毒核酸序列为:5’-aauucuccaguauucaauuacaacaaggcaaccaaacgacuuacaguucuuggaaaggaugcaggugcauugacugaagauccagaugaaggcacaucugggguggagucugcuguccugagaggauuucucauuuuaggcaaagaagacaagagauauggcccagcauuaagcaucaaugaacugagcaaucuug-3’(seqidno:17)
向扩增反应体系分别加入提取的甲型h1n1流感病毒rna1μl(经qubit测定rna浓度为4.68ng/μl)和不含甲型h1n1流感病毒的提取液1μl。
扩增反应体系还包括20mmtris-hcl,75mmkcl,10mm(nh4)2so4,4mmmgso4,100μg/mlbsa,0.1%tween-20(ph8.0@25℃),6mmdntps,正向核心引物和反向核心引物各1μm,正向和反向置换引物各0.5μm,0.1μmdna分子信标,6ubst3.0dna聚合酶,8unb.bsrdi切刻内切酶,用ddh2o补足成10μl。
所用的正向核心引物的序列为:
让反应在65℃进行40分钟,利用宏石实时荧光定量pcr仪每分钟检测一次荧光信号,结果如图6所示。其中
序列表
<110>北京艾克伦医疗科技有限公司
<120>核酸扩增方法及其应用
<130>18755ci
<160>23
<170>siposequencelisting1.0
<210>1
<211>144
<212>dna
<213>artificialsequence
<400>1
cgtagtgtagagagtcacatacgcattcatcgagtgtctgctacacctttgcgttacgcc60
ggtatgacctcgccgggcgcgccttatacgacctcgatttttcgaagttaaacccgcaaa120
cgcccacgtagcagacactcgatg144
<210>2
<211>563
<212>dna
<213>artificialsequence
<400>2
ggactcaccgtagtgggacacttcacaagtaccaccacgacgctcaagcgccagcaattt60
agctacacccgccctgacgaggtcgcgctgcgccacaccaatgccatcaacccgcgctta120
accccgtgaacgtatcgtaacacgagcttttcctccctccccctcacgggtgaaaatccc180
ggggcgtgggccttagtgcgcgacaacagcgctaagggcatcactgccggcagtggcagt240
caacaaaccacgtatgatcccacccgaaccgaagcggctttgaccgcatcaaccaccttt300
gcgttacgccggtatgacctcgccgggcgcgccttatacgacctcgatttttcgaagtta360
aacccgcaaacgcccacgcgcgaccaaaccgggcagatcacctttaacccctttggcggc420
tttggtttgagtggggctgcaccccaacagtgaaacgaggtcaaaaacaaggtccccgtc480
gaggtggcgcaagacccctccaatccttatcggtttgccgttttactcgtgccgcgtagc540
gtggtgtactatgagcagttgca563
<210>3
<211>68
<212>dna
<213>artificialsequence
<400>3
cgtagtgtagagagtcacatacgcattcatcgagtgtctgctacacctttgcgttacgcc60
ggtatgac68
<210>4
<211>41
<212>dna
<213>artificialsequence
<400>4
catcgagtgtctgctacgtgggcgtttgcgggtttaacttc41
<210>5
<211>20
<212>dna
<213>artificialsequence
<400>5
cgaaccgaagcggctttgac20
<210>6
<211>19
<212>dna
<213>artificialsequence
<400>6
atctgcccggtttggtcgc19
<210>7
<211>125
<212>dna
<213>artificialsequence
<400>7
cattcatcgagtgtctgctacacctttgcgttacgccggtatgacctcgccgggcgcgcc60
ttatacgacctcgatttttcgaagttaaacccgcaaacgcccacgtagcagacactcgat120
gaatg125
<210>8
<211>34
<212>dna
<213>artificialsequence
<400>8
aaaaatcgaggtcgtataaggcgcgcccggcgag34
<210>9
<211>23
<212>dna
<213>artificialsequence
<400>9
tttttagctccagcatattccgc23
<210>10
<211>380
<212>dna
<213>hepatitisbvirus
<400>10
ttgttgacaagaatcctcacaataccacagagtctagactcgtggtggacttctctcaat60
tttctagggggagcacccacgtgtcctggccaaaattcgcagtccccaacctccaatcac120
tcaccaacctcttgtcctccaatttgtcctggctatcgctggatgtgtctgcggcgtttt180
atcatattcctcttcatcctgctgctatgcctcatcttcttgttggttcttctggactac240
caaggtatgttgcccgtttgtcctctacttccaggaacatcaactaccagcacgggacca300
tgcaagacctgcacgattcctgctcaaggaacctctatgtttccctcttgttgctgtaca360
aaaccttcggacggaaactg380
<210>11
<211>46
<212>dna
<213>artificialsequence
<400>11
cagtcacattcacacctcgtggttcagccgttttatcatattcctc46
<210>12
<211>29
<212>dna
<213>artificialsequence
<400>12
tcgtggttcagcaacataccttggtagtc29
<210>13
<211>16
<212>dna
<213>artificialsequence
<400>13
tatcgctggatgtgtc16
<210>14
<211>16
<212>dna
<213>artificialsequence
<400>14
tgttcctggaagtaga16
<210>15
<211>22
<212>dna
<213>artificialsequence
<400>15
cactcttcttgttggttcagtg22
<210>16
<211>101
<212>dna
<213>artificialsequence
<400>16
tcgtggttcagccgttttatcatattcctcttcatcctgctgctatgcctcatcttcttg60
ttggttcttctggactaccaaggtatgttgctgaaccacga101
<210>17
<211>196
<212>rna
<213>influenzaavirus
<400>17
aauucuccaguauucaauuacaacaaggcaaccaaacgacuuacaguucuuggaaaggau60
gcaggugcauugacugaagauccagaugaaggcacaucugggguggagucugcuguccug120
agaggauuucucauuuuaggcaaagaagacaagagauauggcccagcauuaagcaucaau180
gaacugagcaaucuug196
<210>18
<211>69
<212>dna
<213>artificialsequence
<400>18
cgtagtgtagagagtcacataccattgccatcgagtgtctgctagtgcattgactgaaga60
tccagatga69
<210>19
<211>39
<212>dna
<213>artificialsequence
<400>19
catcgagtgtctgctagctgggccatatctcttgtcttc39
<210>20
<211>25
<212>dna
<213>artificialsequence
<400>20
aattctccagtattcaattacaaca25
<210>21
<211>21
<212>dna
<213>artificialsequence
<400>21
caagattgctcagttcattga21
<210>22
<211>59
<212>dna
<213>artificialsequence
<400>22
ccgagtgatctcctctcaggacagcagactccaccccagatgtgccggagatcactcgg59
<210>23
<211>147
<212>dna
<213>artificialsequence
<400>23
cattgccatcgagtgtctgctagtgcattgactgaagatccagatgaaggcacatctggg60
gtggagtctgctgtcctgagaggatttctcattttaggcaaagaagacaagagatatggc120
ccagctagcagacactcgatggcaatg147
- 还没有人留言评论。精彩留言会获得点赞!