一种斑节对虾PmGFPT2基因及其应用的制作方法
本发明属于分子生物学技术领域,具体涉及一种斑节对虾pmgfpt2基因及其应用。
背景技术:
己糖胺通路(hbp)是葡萄糖代谢途径之一,是所有生物普遍存在的代谢途径,葡萄糖进入细胞后,首先转化为6-磷酸葡萄糖(fru6p),然后谷氨酰胺6-磷酸果糖酰胺转移酶(glutamine:fructose-6-phosphateaminotransferase2,gfpt2)将氨和fru6p合成为6-磷酸葡糖胺(glcn6p),再经由中间代谢环节,最终产物为二磷酸尿嘧啶n-乙酰氨基葡萄糖(udp-glcnac)。gfpt2是氨基己糖生物合成限速酶,也被称为葡糖胺-6-磷酸合成酶(glucosamine-6-phosphatesynthase),主要参与几丁质的合成和蛋白质的糖基化,udp-glcnac是细菌和真菌细胞壁的主要成分,是蛋白质糖基化的重要底物,也是几丁质的活性前体,几丁质是节肢动物的表皮和中肠围食膜(pm)的主要成分,是形成哺乳动物的糖蛋白、粘多糖基础,在葡萄糖调节中发挥着重要的作用。与gfpt2作用相反的葡糖胺-6-磷酸脱氨酶(glucosamine-6pdeaminase)可以催化glcn6p的分解代谢,在细胞内这两种活动必须分开进行,避免形成无效循环。
对谷氨酰胺6-磷酸果糖酰胺转移酶的研究热点主要集中在其在ⅱ型糖尿病和结核病中发挥的作用及将其作为靶标进行抗病新药物的研制。gfpt2的活性增强表达量升高被认为导致高血糖症的原因,乐军等人的研究发现,葡糖胺-6-磷酸合成酶表达量的升高与结核分枝菌耐药性增强显著相关。gfpt2在甲壳动物中的研究很少,而其催化合成6-磷酸葡糖胺的过程中有氨氮的参与,对gfpt2在养殖水环境中氨氮浓度升高条件下是否参与氨氮胁迫应答以及发挥的作用等方面的研究较少。
技术实现要素:
本发明的目的在于提供一种斑节对虾pmgfpt2基因及其编码蛋白。
本发明的目的还在于提供上述基因或编码蛋白在提高斑节对虾耐氨氮方面的应用。
本发明的最后一个目的在于提供一种斑节对虾pmgfpt2基因氨氮相关的snp位点的筛选方法。
本发明的上述第一个目的是通过以下技术方案来实现的:一种斑节对虾pmgfpt2基因,所述基因的碱基序列如seqidno:2所示。
所述的斑节对虾pmgfpt2基因的编码蛋白,所述编码蛋白的氨基酸序列如seqidno:3所示。
所述pmgfpt2基因的cdna全长为3399bp,包括开放阅读框orf2097bp,5’端非编码区(utr)65bp,3’端非编码区(utr)1237bp,pmgfpt2基因的orf推测包含698个氨基酸,分子量为78.3101kda,理论等电点为6.31。
所述斑节对虾pmgfpt2基因的克隆方法,包括以下步骤:首先提取斑节对虾不同离体组织的总rna,合成cdna第一条链,用做race模板,并将斑节对虾不同离体组织的总rna反转录,用作荧光定量pcr样品;然后根据斑节对虾cdna文库中的est序列,设计pmgfpt2基因orf的引物和race引物,即得到斑节对虾pmgfpt2基因的全长。
所述pmgfpt2基因orf的引物包括qpcr引物pmgfpt2和内参引物pmef-1α,其中所述qpcr引物pmgfpt2包括pmgfpt2-qf和pmgfpt2-qr,其碱基序列如seqidno:4和seqidno:5所示,所述内参引物pmef-1α包括pmef-1α-qf和pmef-1α-qr,其碱基序列如seqidno:6和seqidno:7所示;所述race引物包括pmgfpt2-5’gsp1、pmgfpt2-5’gsp2、pmgfpt2-3’gsp1和pmgfpt2-3’gsp2,其碱基序列分别如seqidno:8~11所示。
本发明的上述第二个目的是通过以下技术方案来实现的:上述的基因或上述基因的编码蛋白在提高斑节对虾耐氨氮方面的应用。
本发明的上述最后一个目的是通过以下技术方案来实现的:一种斑节对虾pmgfpt2基因氨氮相关的snp位点的筛选方法,包括以下步骤:
(1)样品选择:选择非洲野生群体,泰国野生群体和中国三亚野生群体作为snp位点筛选样品,选择耐氨氮极端群体以及氨氮敏感群体作为snp分型样品,并选取标粗池中混养的各家系幼虾为实验材料,使用不同颜色的荧光在虾尾进行标记以区分各家系;
(2)斑节对虾pmgfpt2基因组序列的扩增:通过race技术克隆得到的斑节对虾pmgfpt2基因的cdna,然后使用cdna序列设计特异性引物,使用特异性引物进行pcr扩增,得到斑节对虾pmgfpt2基因组序列;
(3)重测序与snp位点的初筛:提取非洲野生群体、泰国野生群体、三亚野生群体与耐氨氮极端群体的dna,采用步骤(2)中的特异性引物使用直接测序法初步筛选pmgfpt2上的snp位点;
(4)斑节对虾pmgfpt2基因的snp位点的分型:将挑选好的snp位点使用直接测序法进行snp位点的分型,分型使用的样品为耐氨氮极端群体以及氨氮敏感群体;
(5)pmgfpt2基因组序列的扩增,重测序及snp位点的分型:以混合的斑节对虾dna为模板,通过扩增得到pmgfpt2基因组序列,以非洲、泰国、三亚三个野生群体的dna为模板,通过直接测序法在得到的斑节对虾pmgfpt2基因区域2879bp上共筛选得到12个snp位点,其中4个snp位于外显子上,8个位于内含子上;
(6)斑节对虾pmgfpt2基因与抗氨氮性状关联分析:在扩增得到的斑节对虾pmgfpt2基因上筛选得到的12个snp中,选取外显子上的4个snp位点进行分型,并且在两个耐氨氮极端群体中与耐氨氮性状进行关联分析,结果表明,在seqidno:1所示序列的第1557位存在t-c突变,该突变与耐氨氮相关。
其中步骤(1)中所述的特异性引物包括gfpt2-qf1、gfpt2-qr1、gfpt2-hf1和gfpt2-hr1,其碱基序列分别如seqidno:12~15所示。
本发明的还提供了一种斑节对虾pmgfpt2基因耐氨氮相关的snp位点在斑节对虾育种方面的应用,所述的snp位点与斑节对虾耐氨氮相关,与所述斑节对虾pmgfpt2基因紧密连锁,位于seqidno:1所示斑节对虾gfpt2参考基因组的第1557上,该位点突变为t/c,所述应用指采用所述snp位点筛选在位于斑节对虾pmgfpt2参考基因组第1557的位点上具有t/c的斑节对虾。
本发明具有以下优点:
(1)本发明使用race技术首次克隆了斑节对虾pmgfpt2基因,对其进行了生物信息学分析和组织表达分析,使用qpcr技术检测了其在96h急性氨氮胁迫后的表达变化趋势,并对其变化规律进行了分析和探讨,通过不同微生物刺激实验分析探讨其在免疫调节过程中发挥的作用,综合验证分析了pmgfpt2基因在氨氮胁迫响应和免疫刺激中的重要作用;
(2)本发明可以准确地从斑节对虾基因组dna中,快速克隆获得pmgfpt2基因的全长,通过在氨氮胁迫和多种菌刺激下定量特征,鉴别基因在氨氮响应和免疫应答中发挥的调节作用;通过直接测序方法流程能够便捷的检测对虾个体中pmgfpt2基因存在的突变(snp位点),通过检测到snp的类型鉴别对虾对氨氮的耐受性强弱,用于筛选育种个体,为高效的开展标记辅助选择育种提供关键的分子标记。
附图说明
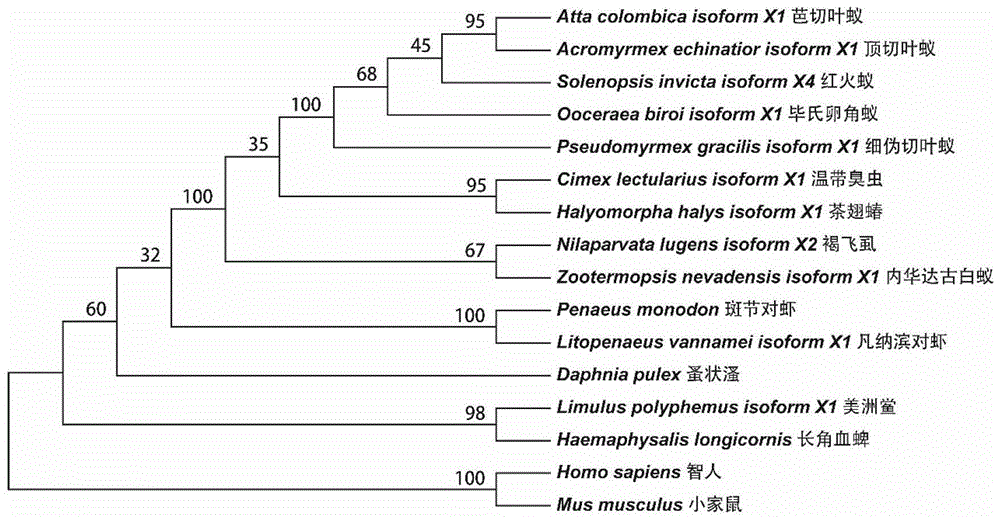
图1是实施例1中pmgfpt2基因的nj进化树;
图2是实施例1中pmgfpt2的和其他物种gfpt2氨基酸多重序列比对;
图3是实施例1中pmgfpt2在各组织中的表达,muscle:肌肉,testis:精巢,spermatophore:精荚,heart:心脏,ventralnerves腹神经:,lymph:淋巴,eyestalknerve:眼柄神经,brainnerves:脑,epidermis:表皮,hepatopancreas:肝胰腺,intestine:肠,gill:鳃,stomach:胃;
图4是实施例1中急性氨氮胁迫后pmgfpt2在肝胰腺(a)中的相对表达量;
图5是实施例1急性氨氮胁迫后pmgfpt2在鳃(b)中的相对表达量;
图6是实施例1中不同微生物刺激后pmgfpt2在肝胰腺hepatopancreas(a)和鳃gill(b)中的相对表达量;
图7是实施例1中斑节对虾dna1%凝胶电泳图;
图8是实施例1中斑节对虾pmgfpt2基因扩增片段1%凝胶电泳图;
图9是实施例1中斑节对虾pmgfpt2基因snp位点示意图。
具体实施方式
下面结合具体实施例进一步说明本发明的应用方法。下述实施例和附图仅用于示例性说明,不能理解为对本发明的限制。除非特别说明,下述实施例中使用的试剂原料为常规市购或商业途径获得的生试剂原料,使用的实验仪器均为实验室常规仪器,除非特别说明,下述实施例中使用的方法和设备为本领域常规使用的方法和设备。
实施例1
一、实验过程
1.1、实验材料
实验所用的斑节对虾均来自于南海水产研究所深圳基地。实验用虾在循环水池中暂养3d后使用,盐度为30,水温(25±1)℃。选取雌、雄各三尾健康的对虾进行解剖,分别取肝胰腺(hepatopancreas)、鳃(gill)、肠(intestine)、肌肉(muscle)、胃(stomach)、淋巴(lymph)、眼柄神经(eyestalknerve)、脑神经(brainnerves)、腹神经(ventralnerves)和精巢(testis)等组织用作各组织cdna模板的制备。选取体长为7~8cm,体质量为6~7g的幼虾用于急性氨氮胁迫实验。选取体长为6~7cm,体质量为5~6g的幼虾用于细菌感染实验。斑节对虾急性氨氮胁迫实验和微生物刺激实验在南海水产研究所深圳试验基地进行。
1.2、总rna的提取与cdna的合成
按照magen说明书提取斑节对虾不同组织的总rna,用nanodrop2000超微量分光光度计检测rna的浓度和纯度,用1.2%琼脂糖凝胶电泳检测rna条带的完整度。用于做race模板的样品按照primescriptⅱ1ststrandcdnasynthesiskit(takara)试剂盒合成cdna第一条链。用做荧光定量pcr的样品,分别取各组织1μg总rna,按照primescriptrtreagentkitwithgdnaeraser试剂盒进行反转录。所得cdna经ef-1α引物检测后-80°保存备用。
1.3、斑节对虾pmgfpt2基因的克隆
根据本实验室构建的斑节对虾cdna文库中的est序列,使用primer5.0软件设计验证pmgfpt2基因orf的引物和race引物(表1),得到pmgfpt2基因的全长。
表1本实施例中采用的引物和引物序列
总反应体系为25μl:2.5μlextaqbuffer,1μlextaq,2.0uldntp、1μlcdna,1μl的上游引物和1μl的下游引物,双蒸水补足至25μl。
pcr程序如下:
1.4、斑节对虾pmgfpt2基因序列分析
利用ncbi(https://www.ncbi.nlm.nih.gov/)的blast工具将斑节对虾pellino与其他物种pellino蛋白进行同源性比对,用signalp4.1yi(http://www.cbs.dtu.dk/services/signalp)预测pellino家族基因的信号肽;用cdsearch(https://www.ncbi.nlm.nih.gov/structure/cdd/wrpsb.cgi)分析蛋白结构域;用computepi/mw工具(http://web.expasy.org/compute_pi)预测蛋白的分子量和等电点;用clustalx1.83进行多重序列比对,并用texmaker作图;使用mega6.0的邻接法(neighbor-joining,nj)构建系统进化树。
1.5、斑节对虾gfpt2基因在不同组织的表达
选取健康的斑节对虾雌、雄各3尾进行解剖取样,提取不同组织的总rna,以其反转录产物为模板,以pmpellino的基因全长为基础设计荧光定量的特异性引物(表1),以ef-1α为内参基因,以双蒸水代替模板作阴性对照,按照tbgreen说明书进行荧光定量pcr检测,每个样品和内参均设置3个重复。采用相对δct法(2-δδct法)分析pmpellino基因在斑节对虾各组织中的相对表达量。
1.6、急性氨氮胁迫
通过预实验求出斑节对虾96小时急性氨氮胁迫的半致死浓度lc50以及安全浓度sc。该预试验设置7个氨氮浓度梯度,分别为0,20,40,60,80,100,120mg·l-1。每个梯度选取混合家系大小均匀的30尾虾随机放置在7个黑桶中。每个黑桶中加入200l过滤海水及两个气头。预试验期间不投喂饲料,每隔固定时间记录死亡情况至96小时实验结束,捞取死虾,每天换全部海水。实验结束后通过直线内插法计算半致死浓度和安全浓度。
正式实验分为2组,低浓度胁迫组和高浓度胁迫组,以第0小时的样品为对照组,使用过滤后的养殖海水。低浓度胁迫组的胁迫浓度为安全浓度;高浓度胁迫组的胁迫浓度为96小时半致死浓度。每组设3个重复,每个重复30尾虾。实验所用的黑桶和斑节对虾选取方法与预试验相同。实验通过添加氯化铵(nh4cl)控制各实验组的氨氮浓度,每隔2小时记录死亡情况并捞取死虾,每24小时更换全部海水。于胁迫后第0,第3,第6,第12,第24,第48,第72和第96小时进行取样。取样时每组选取3尾活力较好的个体,取肝、鳃、肠等组织保存在rnalater(ambion)中,-80°保存样品。
1.7、不同微生物刺激
为了研究不同微生物刺激对斑节对虾糖原合成酶基因的激活作用,实验选取了健康的斑节对虾进行了微生物刺激。根据实验室关于金黄葡萄球菌、哈维氏弧菌、鳗弧菌对于斑节对虾96h半致死浓度分别为:1.5×109cfu/ml、2.0×108cfu/ml和3.5×108cfu/ml。实验将200尾斑节对虾平均分为4组:1)注射金黄色葡萄球菌(革兰氏阳性菌);2)注射哈维氏弧菌(革兰氏阴性菌);3)注射鳗弧菌(革兰氏阴性菌);4)注射pbs作为对照组。实验组分别注射100μl已经稀释好备用浓度为1×108cfu/ml的菌液,对照组注射100μlpbs。刺激后分别在第0、第3、第6、第9、第12、第24、第48和第72小时取对虾的肝胰腺、鳃等组织,rnalater样品放入-80℃保存,提取总rna后合成cdna模板进行qpcr实验。以ef-1α作为内参基因。
1.8、统计学分析
使用spss23.0对定量结果进行单因素方差分析(one-wayanova),结果以“平均值±标准差(x±sd)”表示。分析差异显著性(turkey),p<0.05为差异显著,p<0.01为差异极显著。若组间存在差异,则用turkey方法进行多重比较分析。
二、实验结果
2.1pmgfpt2的cdna序列特征
通过race技术克隆得到pmgfpt2基因的cdna全长。pmgfpt2基因的cdna全长为3399bp(如seqidno:1所示),包括开放阅读框orf2097bp(seqidno:2所示),5’端非编码区(utr)65bp,3’端非编码区(utr)1237bp,pmgfpt2基因的orf推测包含698个氨基酸(seqidno:3所示),分子量为78.3101kda,理论等电点为6.31。pmgfpt2无信号肽,属于glms超家族(1-698aa),包含位于n端的谷氨酰胺转氨酶gfat类结构域(2-297aa)以及糖异构酶结构域sis(379-508aa)。
具体如下:
2.2同源性和系统进化树分析
使用mega6.0软件构建斑节对虾与其他物种的pmgfpt2的系统进化树(图1)。从系统树可以看出,斑节对虾与凡纳滨对虾的gfpt2在进化关系上最为接近,它们先聚为一枝后再与其他无脊椎动物聚合。
斑节对虾pmgfpt2编码的氨基酸序列在blastx上与其他物种的比对结果表明pmgfpt2蛋白具有较高的同源性。将其他物种的氨基酸序列与斑节对虾的pmgfpt2进行多重比对分析,使用clustalx与texshade软件制作多重序列比对图,见图2。
2.3、pmgfpt2在不同组织的表达分析
以ef-1α为内参,使用qpcr技术检测pmgfpt2在各组织中的表达情况。结果显示,pmgfpt2在各组织中的表达具有一定的差异性(图3)。pmgfpt2在斑节对虾各组织中都有表达,其中在胃中的表达量最高,在鳃、肠、肝胰腺中的表达量较高,在表皮、脑神经、眼柄神经、淋巴、腹神经、心脏中的表达量中等,而在精荚、精巢和肌肉中的表达量较低,其中pmgfpt2在胃中的表达量是肌肉中的30倍(p<0.05)。
2.4急性氨氮胁迫下pmgfpt2的表达变化
使用rt-qpcr技术检测pmgfpt2基因在96小时急性氨氮胁迫后的表达变化趋势(图4-图5)。
在肝组织中,高浓度组pmgfpt2的表达量显著上升,在第3小时与对照组0小时有极显著差异(p<0.01),且在第12小时达到了峰值(p<0.01),在第12小时至第96小时之间表达量呈下降趋势,且第96小时与对照组0小时无显著差异。低浓度组pmgfpt2的表达量显著上调(p<0.01),表达模式有周期性,在第12小时,第24小时,第72小时分别达到峰值,且第96小时与对照组有极显著差异(p<0.01)。
在鳃组织中,低浓度组的pmgfpt2表达量均高于0h对照组,表达模式与肝组织的低浓度组相似,呈现周期性调节趋势,分别在第12小时及第72小时达到峰值(p<0.01),72小时后呈下降趋势,且第96小时与0小时对照组无显著差异。而高浓度组中,pmgfpt2的表达被显著抑制,第3小时、第48小时、第72小时以及第96小时均与0h对照组有极显著差异(p<0.01)。
2.5微生物刺激下pmgfpt2基因的表达变化
以注射磷酸缓冲液pbs组作为对照组,以ef-1α作为内参基因,使用qpcr技术检测斑节对虾gfpt2基因在不同微生物刺激后的表达变化,pmgfpt2的表达量变化趋势如图6所示。由图可知,在哈维氏弧菌刺激后,pmgfpt2在肝胰腺组织中的表达量在第0小时至第24小时与对照组无显著差异,而在第48小时、第72小时的表达量均显著低于对照组(p<0.01),第48小时的表达量是对照组的0.24倍。而在鳃组织中,pmgfpt2基因的表达量在第0小时至第12小时均与对照组无显著差异,而在第24小时,第72小时的表达均显著上调,且与对照组有极显著差异(p<0.01),在第24小时到达峰值且其表达量是对照组的5.78倍。
3、讨论
3.1斑节对虾pmgfpt2基因的克隆和序列分析
本发明首次克隆了斑节对虾谷氨酰胺-6-磷酸果糖酰胺转移酶基因(pmgfpt2),将pmgfpt2在blast中与其它物种的gfpt2比对可以发现,pmgfpt2具有较高的同源性。斑节对虾pmgfpt2与凡纳滨对虾gfpt2的蛋白相似度高达99.43%,与其他物种相比也有较高的保守性。谷氨酰胺6-磷酸果糖酰胺转移酶属于酰胺转移酶家族,pmgfpt2具有位于n端的谷氨酰胺转氨酶gfat类结构域(2-297aa)以及糖异构酶结构域sis(379-508aa)。位于n端的谷氨酰胺酶区域能催化谷氨酰胺水解为氨和谷氨酸,而糖异构酶结构则可以利用氨将6-磷酸葡萄糖合成为6-磷酸葡糖胺。gfpt2在进行氨基转移时不接受外来的氮源,只能将自身水解谷氨酰胺产生的氨与果糖胺上的c-2位点结合。这是gfpt2与其他酰胺转氨酶的不同之处,可能与它的四级结构有关。
3.2斑节对虾pmgfpt2基因在不同胁迫条件下的表达分析
肝胰腺是甲壳动物的主要氨氮解毒代谢器官,其中含有大量的酶与蛋白质。对氨氮的反应较为灵敏。鳃是大多数水生动物体内的氨氮排泄器官,在氨氮代谢过程中发挥重要作用,当甲壳动物体内氨氮浓度过高时,机体会通过降低鳃组织对nh3的通透性,增加对nh4+的排出来进行调节。由图4-图5可知,斑节对虾pmgfpt2基因在肝胰腺、鳃组织中的具有较高的表达量。推测pmgfpt2基因可能参与氨氮胁迫应答中并发挥一定的作用。
已有的研究表明,甲壳动物在氨氮胁迫条件下,可以通过代谢途径将体内多余的氨转化为无毒害作用的谷氨酰胺、谷氨酸等进行氨氮解毒。急性氨氮胁迫后,pmgfpt2在肝胰腺中的表达量在不同浓度组中都显著上调,与陈劲松等研究的斑节对虾氨氮代谢通路上的关键基因谷氨酰胺合成酶的表达模式相似。虽然,pmgfpt2不能利用外来氮源,但其合成6-磷酸葡糖胺所需的氨来自谷氨酰胺,推测其可以通过降低体内谷氨酰胺的含量来加速机体中氨向谷氨酰胺的转化,以此降低体内氨氮的毒性。在鳃组织中,低浓度(6.5mg/l)的氨氮胁迫显著激活了pmgfpt2的表达,而高浓度组(65mg/l)中,pmgfpt2的表达被显著抑制。有研究表明,在氨氮胁迫下甲壳动物的鳃组织会受到损伤,其正常功能会受到影响。推测高浓度氨氮对鳃组织造成了一定的损伤,导致pmgfpt2的表达被抑制。
pmgfpt2催化形成的最终产物udp-glcnac是细菌和真菌细胞壁的组成部分,近年来以其作为分子靶标进行抗生素新药物的研制成为研究热点。gfpt2是hbp通路的限速酶,调节着葡萄糖与在体内的流量和能量平衡。在甲壳动物中,gfpt2由对微生物的免疫应答的研究依然空白。由不同微生物刺激下pmgfpt2的表达变化结果可知,在肝胰腺中,革兰氏阴性菌对其表达有一定的抑制作用。在鳃组织中,哈维氏弧菌对其有一定的激活作用。在革兰氏阳性菌金黄葡萄球菌的刺激下,pmgfpt2在肝胰腺和鳃中的表达量均显著上升。表明,pmgfpt2可能在斑节对虾免疫应答中发挥一定的作用。
在鳗弧菌刺激后,pmgfpt2在第3小时至第72小时内在肝胰腺中的表达量均低于pbs对照组,且在第12小时、第24小时、第48小时、第72小时的表达量与对照组有极显著差异(p<0.01),可见鳗弧菌对pmgfpt2在肝胰腺中的表达具有显著的抑制作用。而在鳃组织中,pmgfpt2在第0小时至第72小时内的表达量与对照组均无显著差异。
在金黄色葡萄球菌刺激后,pmgfpt2在肝胰腺中的表达被激活,在第3小时达到第一个峰值,且与对照组有极显著差异(p<0.01),为对照组的5.28倍,其后呈先下降后上升趋势,在第24小时与第48小时的表达量均与对照组有极显著差异(p<0.01),且在第48小时达到第二个峰值,为对照组的1.69倍。在第6小时、第72小时的表达量均显著高于对照组(p<0.05)。在鳃组织中,pmgfpt2表达同样被显著激活,在第3小时、第6小时、第24小时、第48小时和第72小时的表达量与对照组有极显著差异(p<0.01),在第6小时达到第一个峰值,其表达量为对照组的14.25倍,在第48小时达到第二个峰值,其表达量为对照组的9.86倍。
4斑节对虾谷氨酰胺6-磷酸果糖转氨酶基因snp位点的开发
4.1、实验材料的选择
通过候选基因法筛选snp的样品是来自不同地理位置的斑节对虾野生群体,主要包括非洲野生群体,泰国野生群体和中国三亚野生群体。在非洲南部莫桑比克附近海域采集非洲野生群体样本,在泰国普吉岛海域采集泰国野生群体样本,在中国海南三亚海域采集三亚野生群体。
本实施例使用对氨氮具有不同耐受性的极端群体作为实验材料进行snp分型。氨氮极端群体通过氨氮胁迫实验进行,实验所用氨氮浓度为96h急性氨氮胁迫的半致死浓度。通过添加氯化铵的方式来控制氨氮浓度。氨氮胁迫实验在南海水产研究所深圳试验基地4号车间进行。通过对选育的多个混合家系进行96h急性氨氮胁迫实验,根据死亡时间判断氨氮耐受力的强弱,选取耐氨氮能力最强的前150尾作为氨氮耐受群体,选取耐氨氮能力最弱的后150尾作为氨氮敏感群体。
实验选取标粗池中混养的各家系8-10cm的幼虾为实验材料,使用不同颜色的荧光在虾尾进行标记以区分各家系。将32个家系的虾均匀投放于4个水泥池中,设两个平行,每个平行每个家系各50尾虾,盐度为30,水温(25±1)℃。每24小时换一次海水,每隔3小时统计一次死虾数量,捞取死虾,通过识别荧光标记颜色识别家系。实验结束时实验中所有家系的斑节对虾全部死亡,选取最先死亡的150尾虾作为氨氮敏感群体,最后死亡的150尾虾为氨氮耐受群体,取肌肉样品保存在dna中。
4.2、斑节对虾谷氨酰胺果糖6-磷酸转氨酶基因组序列的扩增
通过race技术克隆得到的斑节对虾谷氨酰胺6-磷酸果糖转氨酶基因(pmgfpt2)的cdna为3399bp。通过在ncbi的gene中查询其他物种的gfpt2基因的生物学信息,推测pmgfpt2在斑节对虾中可能的基因长度和内含子与外显子的结构分布,然后使用pmpmgfpt2的cdna序列设计引物,采集健康的斑节对虾提取dna作为扩增pmgfpt2的内含子序列的模板,通过扩增得到pmgfpt2基因组序列。使用特异性引物进行pcr扩增,扩增pmgfpt2基因组序列所用引物如表2,之后进行直接测序,将测序结果与cdna进行比对拼接。
表2扩增pmgfpt2实验中所用的引物序列
4.3重测序与snp位点的初筛
首先进行非洲野生群体、泰国野生群体、三亚野生群体与耐氨氮极端群体的dna提取,其中三种野生群体各20尾,耐氨氮极端群体各60尾。dna提取按照上海magen公司的dna提取试剂盒进行样品模板的提取,使用nanodrop2000超微量核酸定量分析系统检测dna的纯度与浓度,通过1.2%琼脂糖凝胶电泳验证pcr产物的完整性(图7)。
使用直接测序法初步筛选pmgfpt2上的snp位点。在所提取的非洲、泰国、三亚三个斑节对虾野生群体的dna中样品中每种选取15个作为模板,使用扩增pmgfpt2所用的引物来进行扩增重测序,将pcr产物送到北京睿博兴科生物技术有限公司进行测序,之后将45个样本的测序结果用sequencher4.1.4软件进行比对以得到snp位点。
4.4、斑节对虾谷氨酰胺果糖6-磷酸转氨酶snp位点的分型
将挑选好的snp位点送到广州擎科生物技术有限公司使用直接测序法进行snp位点的分型,分型使用的样品为耐氨氮极端群体60个以及氨氮敏感群体60个。实验步骤包括引物的设计与合成、模板质检、pcr扩增、扩增产物纯化、延伸反应、延伸产物纯化、测序仪检测与数据分析等。
4.5pmgfpt2基因组序列的扩增,重测序及snp位点的分型
以混合的斑节对虾dna为模板,通过扩增得到pmgfpt2基因组序列的7个外显子和6个完整内含子,总共4180bp。以非洲、泰国、三亚三个野生群体的dna为模板,通过直接测序法在得到的斑节对虾pmgfpt2基因区域2879bp上共筛选得到12个snp位点,其中4个snp位于外显子上,8个位于内含子上,部分扩增序列如图8。斑节对虾gfpt2基因snp位点示意图如图9所示。
表3位于斑节对虾gfpt2基因外显子上的snp
4.6、斑节对虾pmgfpt2基因与抗氨氮性状关联分析
在扩增得到的斑节对虾pmgfpt2基因上筛选得到的12个snp中,选取外显子上的4个snp位点进行分型,并且在两个耐氨氮极端群体中与耐氨氮性状进行关联分析。结果如表4所示。
表4斑节对虾gfpt2基因snp位点与耐氨氮性状关联分析
4.7结果分析
本实施例采用直接测学法在部分斑节对虾谷氨酰胺6-磷酸果糖转氨酶上筛选得到12个snp位点,且每个snp位点的最小等位基因频率都大于1%。已知同义突变的位点编码的氨基酸不会有变化,在外显子上的4个snp位点中有3个位点是为同义突变,一个位点pmgfpt2-1501为缺失突变。研究表明,在基因组的编码区内,由于生物在进化过程中具有的自身消除有害突变的机制,同义突变远多于非同义突变。非同义突变会导致氨基酸的改变,进而导致蛋白组成的变化而影响其功能,也因此非同义snp突变是研究的热点,被认为是高效突变位点,许多与生物的生长、抗逆和免疫等性状相关联的非同义snp位点被开发出来。本实施例用于关联分析所使用的群体是两个对氨氮耐受性不同的极端群体,通过氨氮急性胁迫实验选出最先死亡的氨氮敏感群体60尾及最后死亡的耐氨氮群体60尾。关联分析的结果表明,在fisher检验模型中,pmgfpt2-1557位点的t突变在耐氨氮组与不耐氨氮组有显著差异(p=0.0309),可以作为耐氨氮新品系选育的候选位点。
本发明不局限于上述特定的实施方案范围内,上述实施方案仅仅是为了能够对本发明的使用过程进行详细地说明,而且有相等功能的生产方法和技术细节也属于本发明内容的一部分。事实上,本领域技术人员根据前文的描述,就能够根据各自需要找到不同的调整方案,这些调整都应在本文所附的权利要求书的范围内。
序列表
<110>中国水产科学研究院南海水产研究所
<120>一种斑节对虾pmgfpt2基因及其应用
<160>15
<170>siposequencelisting1.0
<210>1
<211>3399
<212>dna
<213>谷氨酰胺6-磷酸果糖酰胺转移酶(glutamine:fructose-6-phosphateaminotransferase2)
<400>1
ctggacaggctcgagcgactagaatcgatttggactgtgggaaatctttcgttacattac60
caaccatgtgtggaatcttcgcctacctgaactacctgacccccaagcgccgcgaggaga120
tcatcgagcttctggtgcgaggactggagaggctcgagtaccgcggctacgactccgcag180
gtattggcatcgactccgtggacggtggcgtgtgcctcatcaagcagacgggcaaggtca240
agtgcctccgcgacaagatcgaggcccagaaggagaagctcgccttcgaaaaggaacatg300
aggtccacatcggcatcgcccacacccgctgggccacgcacggcgtcccctccgacgtga360
actcccacccgcagcgctccggcgaggggaacgccttcgtggtcgtgcataacggaatta420
ttaccaactacaaggacgtgaagcagttcctcatccagaagggtcacaagttcgagtccg480
agacagacactgaggtgatcgccaagctgaccgagcaccttcaccacctgcaccccacct540
actccttcagggaactcgtcgagcaggtggtgcaacagctggaaggagcctttgctcttt600
gcttcaagagcaagcacttcccaggagaggtcgtggccacccgccggggatctccccttc660
tcgtgggcatcaaaactaagaccaagttggccacagaccacattcccgttctctacagca720
aggactcccccgagaaggttgaactgaaagacttagaagcacttcctaaatcagacaacc780
aagatcatcgtccccatgccaacgacatccccgcaacaggctttaaccgcacacagtcca840
ccacagaattcttacccttgggcgatgacagggaagtggagtacttctttgcctctgacg900
cttcagccatcattgagcacacagatcgtgtcatctaccttctggatgatgatgtatcca960
gtgtagacacagaaggtgttctgaagatccatcgtctcaaacgctctctggatgacacac1020
atgcccgtgaaatcacaaccctcaagatggagatccaacagatcatgaagggcaacttct1080
cctccttcatgcagaaagaaatctttgagcagcctgagtctgtagttaacaccatgagag1140
gacgcattaactttgaccgagagactgttgtcttgggaggcatcaaggattacatcagtg1200
aaattcgccgttgtcgccgtttgctacttattgcttgcggaacatcataccactcagcaa1260
ttgcaacaaggcagttgttggaggagctcactgaactccctgtaatggttgatttggcct1320
cagacttccttgaccgcaacacacctatcttcagagatgatgtgtgcttctttatttcac1380
aatctggagaaactgcagatactctcatggctctcagatactgtaaacaacgtggtgccc1440
tgattgtaggaattactaacacagtcggctcttctatctgccgagagtctcactgtggag1500
tgcatattaatgctggacctgagattggtgttgcttccacaaaggcctacacttcccaaa1560
tgatttcccttgttatgtttgcccttatgatctcagaggatcgtatctcagcacagcccc1620
gccgttcagaaattattcagggactgcacaagcttccagagcagatcaaggaggtgctga1680
gtctggattcaaaaatccaggagatcagtaaagaagtatttgagaagaaatccttgttgg1740
ttcttggtcgaggctacaactatgcaacctgccttgaaggggctctgaaaatcaaggaaa1800
tcacatacatgcacagtgaaggtattcttgctggagaactcaagcatggtccccttgcct1860
tagttgacagaggcatgcccatcatcatgatttgcacacgtgacaatgtatttgtgaaat1920
gcatgaatgcactccagcaagttacagctcgtgaaggaaggccaattctcatctgccaca1980
agggtgatgaggaaccacagaagtatggttctagtgttcttgaggtccccaaaacagttg2040
actgtcttcaaggtctcctcacagtcattccgatgcagctactttcctaccacattgctg2100
tcctcaggggctgcaatgtagattgtccacgaaatttggcaaagtccgttactgtagaat2160
aaaattgtcttgtcaatatcctgcattcttcctgtgctcatctcattgttcttatattga2220
tccacaacagttattgttggtgaatgaattatagctgtaatatttgtatttttcaaggta2280
ttagaggaaatcttatatattagggttgggaaaatttaggcaccaaatatattgttgcag2340
gaaagaattttaaggtgagccagatggtatgtaagtttccatctgtgattgtttgatgct2400
aacccttatctaggttataatctgaatgatactaagctctctggcttctctccattccta2460
cagtatttctacccctttttgagcttgtaaatttttggctcccagttgctcactgacctg2520
agatgaattctaggattaatgtatctatttcatcctaagtgatggaatagattttctttt2580
aaaatttacagtaatgaacaagatttgattttatgacttatctaagtgtcagtgttgagt2640
tctttgccttgtctgatttagtaatgaaaatgacttagtgagcgagattctgtaaagtat2700
attgtttaatggtaaaacattttcagctcactaacatctcatggttgaatctgagggtcc2760
ttgtatatatgtatttattgttaagatctttttgatcaggcctcaacttgtggcttccaa2820
attgttgaaattaatgaaagtttataaatagagacagttgaattgagtgaaatttgttaa2880
cccaacactgatattcatctctgatttttcttcacaatttccttacagtctatcagttat2940
tcagataatctcacattttcctaaaagcaatactttcattcctgttagatattatctact3000
tgcactttgttttgtatatgatttgcacagaggatgtacaaaatattaaaaatgctacat3060
tataattcatgacagaaaaaatacattccctaaattgacattgaggaaaataagaaacta3120
taatacatacataaatatcctatgtattgtgcatagtttacagatgtatttgccccagtg3180
tcagtgtcagtgtctctgctgagtctttgtataaatacatttttgttattggttccttgg3240
aaaaaggaaccattataaaaagagttggaattaaaacctgcattatcatactcttggagt3300
attactattccccttcatgccatatgatttaggcagtaaaactgattgcacacaaggtaa3360
tttagaaacaaaatgaaaaagagagatcggaagagcgtc3399
<210>2
<211>2097
<212>dna
<213>谷氨酰胺6-磷酸果糖酰胺转移酶(glutamine:fructose-6-phosphateaminotransferase2)
<400>2
atgtgtggaatcttcgcctacctgaactacctgacccccaagcgccgcgaggagatcatc60
gagcttctggtgcgaggactggagaggctcgagtaccgcggctacgactccgcaggtatt120
ggcatcgactccgtggacggtggcgtgtgcctcatcaagcagacgggcaaggtcaagtgc180
ctccgcgacaagatcgaggcccagaaggagaagctcgccttcgaaaaggaacatgaggtc240
cacatcggcatcgcccacacccgctgggccacgcacggcgtcccctccgacgtgaactcc300
cacccgcagcgctccggcgaggggaacgccttcgtggtcgtgcataacggaattattacc360
aactacaaggacgtgaagcagttcctcatccagaagggtcacaagttcgagtccgagaca420
gacactgaggtgatcgccaagctgaccgagcaccttcaccacctgcaccccacctactcc480
ttcagggaactcgtcgagcaggtggtgcaacagctggaaggagcctttgctctttgcttc540
aagagcaagcacttcccaggagaggtcgtggccacccgccggggatctccccttctcgtg600
ggcatcaaaactaagaccaagttggccacagaccacattcccgttctctacagcaaggac660
tcccccgagaaggttgaactgaaagacttagaagcacttcctaaatcagacaaccaagat720
catcgtccccatgccaacgacatccccgcaacaggctttaaccgcacacagtccaccaca780
gaattcttacccttgggcgatgacagggaagtggagtacttctttgcctctgacgcttca840
gccatcattgagcacacagatcgtgtcatctaccttctggatgatgatgtatccagtgta900
gacacagaaggtgttctgaagatccatcgtctcaaacgctctctggatgacacacatgcc960
cgtgaaatcacaaccctcaagatggagatccaacagatcatgaagggcaacttctcctcc1020
ttcatgcagaaagaaatctttgagcagcctgagtctgtagttaacaccatgagaggacgc1080
attaactttgaccgagagactgttgtcttgggaggcatcaaggattacatcagtgaaatt1140
cgccgttgtcgccgtttgctacttattgcttgcggaacatcataccactcagcaattgca1200
acaaggcagttgttggaggagctcactgaactccctgtaatggttgatttggcctcagac1260
ttccttgaccgcaacacacctatcttcagagatgatgtgtgcttctttatttcacaatct1320
ggagaaactgcagatactctcatggctctcagatactgtaaacaacgtggtgccctgatt1380
gtaggaattactaacacagtcggctcttctatctgccgagagtctcactgtggagtgcat1440
attaatgctggacctgagattggtgttgcttccacaaaggcctacacttcccaaatgatt1500
tcccttgttatgtttgcccttatgatctcagaggatcgtatctcagcacagccccgccgt1560
tcagaaattattcagggactgcacaagcttccagagcagatcaaggaggtgctgagtctg1620
gattcaaaaatccaggagatcagtaaagaagtatttgagaagaaatccttgttggttctt1680
ggtcgaggctacaactatgcaacctgccttgaaggggctctgaaaatcaaggaaatcaca1740
tacatgcacagtgaaggtattcttgctggagaactcaagcatggtccccttgccttagtt1800
gacagaggcatgcccatcatcatgatttgcacacgtgacaatgtatttgtgaaatgcatg1860
aatgcactccagcaagttacagctcgtgaaggaaggccaattctcatctgccacaagggt1920
gatgaggaaccacagaagtatggttctagtgttcttgaggtccccaaaacagttgactgt1980
cttcaaggtctcctcacagtcattccgatgcagctactttcctaccacattgctgtcctc2040
aggggctgcaatgtagattgtccacgaaatttggcaaagtccgttactgtagaataa2097
<210>3
<211>698
<212>prt
<213>谷氨酰胺6-磷酸果糖酰胺转移酶(glutamine:fructose-6-phosphateaminotransferase2)
<400>3
metcysglyilephealatyrleuasntyrleuthrprolysargarg
151015
glugluileilegluleuleuvalargglyleugluargleuglutyr
202530
argglytyraspseralaglyileglyileaspservalaspglygly
354045
valcysleuilelysglnthrglylysvallyscysleuargasplys
505560
ileglualaglnlysglulysleualapheglulysgluhisgluval
65707580
hisileglyilealahisthrargtrpalathrhisglyvalproser
859095
aspvalasnserhisproglnargserglygluglyasnalapheval
100105110
valvalhisasnglyileilethrasntyrlysaspvallysglnphe
115120125
leuileglnlysglyhislyspheglusergluthraspthrgluval
130135140
ilealalysleuthrgluhisleuhishisleuhisprothrtyrser
145150155160
phearggluleuvalgluglnvalvalglnglnleugluglyalaphe
165170175
alaleucysphelysserlyshispheproglygluvalvalalathr
180185190
argargglyserproleuleuvalglyilelysthrlysthrlysleu
195200205
alathrasphisileprovalleutyrserlysaspserproglulys
210215220
valgluleulysaspleuglualaleuprolysseraspasnglnasp
225230235240
hisargprohisalaasnaspileproalathrglypheasnargthr
245250255
glnserthrthrglupheleuproleuglyaspasparggluvalglu
260265270
tyrphephealaseraspalaseralaileilegluhisthrasparg
275280285
valiletyrleuleuaspaspaspvalserservalaspthrglugly
290295300
valleulysilehisargleulysargserleuaspaspthrhisala
305310315320
arggluilethrthrleulysmetgluileglnglnilemetlysgly
325330335
asnpheserserphemetglnlysgluilephegluglnprogluser
340345350
valvalasnthrmetargglyargileasnpheasparggluthrval
355360365
valleuglyglyilelysasptyrilesergluileargargcysarg
370375380
argleuleuleuilealacysglythrsertyrhisseralaileala
385390395400
thrargglnleuleuglugluleuthrgluleuprovalmetvalasp
405410415
leualaserasppheleuaspargasnthrproilepheargaspasp
420425430
valcysphepheileserglnserglygluthralaaspthrleumet
435440445
alaleuargtyrcyslysglnargglyalaleuilevalglyilethr
450455460
asnthrvalglyserserilecysarggluserhiscysglyvalhis
465470475480
ileasnalaglyprogluileglyvalalaserthrlysalatyrthr
485490495
serglnmetileserleuvalmetphealaleumetilesergluasp
500505510
argileseralaglnproargargsergluileileglnglyleuhis
515520525
lysleuprogluglnilelysgluvalleuserleuaspserlysile
530535540
glngluileserlysgluvalpheglulyslysserleuleuvalleu
545550555560
glyargglytyrasntyralathrcysleugluglyalaleulysile
565570575
lysgluilethrtyrmethissergluglyileleualaglygluleu
580585590
lyshisglyproleualaleuvalaspargglymetproileilemet
595600605
ilecysthrargaspasnvalphevallyscysmetasnalaleugln
610615620
glnvalthralaarggluglyargproileleuilecyshislysgly
625630635640
aspglugluproglnlystyrglyserservalleugluvalprolys
645650655
thrvalaspcysleuglnglyleuleuthrvalileprometglnleu
660665670
leusertyrhisilealavalleuargglycysasnvalaspcyspro
675680685
argasnleualalysservalthrvalglu
690695
<210>4
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>4
cctgagattggtgttgcttcc21
<210>5
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>5
gggctgtgctgagatacga19
<210>6
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>6
attggaggtattggaacagtgcc23
<210>7
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>7
tcggtaagagcttcgtggtgc21
<210>8
<211>29
<212>dna
<213>人工序列(artificialsequence)
<400>8
gtggtatgatgttccgcaagcaataagta29
<210>9
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>9
ggaggcacttgaccttgcccgtc23
<210>10
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>10
cagcacagccccgccgttca20
<210>11
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>11
attgctgtcctcaggggctgcaa23
<210>12
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>12
tccgagacagacactgaggtg21
<210>13
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>13
atgatgttccgcaagcaataa21
<210>14
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>14
atggagatccaacagatcatgaag24
<210>15
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>15
ttattctacagtaacggactttgc24
- 还没有人留言评论。精彩留言会获得点赞!