一种细胞大分子锚定的DNA步移索引成像方法与流程
本发明属于分析化学、化学生物学及细胞成像领域,涉及一种细胞大分子锚定的dna步移索引成像方法。
背景技术:
细胞环境中某些分子或组分的空间分布和相互关系是生命过程的基础。染色质化学修饰已被证明在生物体中起着调控基因表达、基因组复制和染色质结构的关键作用。5-羟甲基胞嘧啶(5-hydroxymethylcytosine,5-hmc)、5-羟甲基尿嘧啶(5-hydroxymethyluracil,5hmu)和5-醛基尿嘧啶(5-formyluracil,5fu)等是哺乳动物基因组中常见的染色质化学修饰,通常与包括癌症在内的各种疾病有关。组蛋白可以进行多种翻译后修饰,特定的组蛋白修饰与活跃或沉默的基因组区域密切相关,因此其在染色质状态和基因调控中起重要作用。
在特定基因组位点或目标大分子(如修饰后的组蛋白)周围会同时存在多种染色质修饰已经被广泛证实,这暗示了他们之间存在潜在的互作关系或者空间分关系,研究这些关系将有助于阐明生物过程中的复杂表观遗传。然而现存的方法很难实现细胞纳米环境中不同生物分子的空间关系分析。
显微成像是常用的染色质化学修饰检测方法,它可以实现单个细胞内目标分子的空间定位,揭示其依赖细胞类型的分布特征以及显著的细胞异质性。现存的dna邻近连接方法主要是通过dna序列连接两种相邻的蛋白、基因和化学修饰。但是,单个的一对一组合在检测时会完全消耗掉探针,这使得其难以探究多种相邻染色质修饰的组合模式。综上,单细胞纳米环境内多种染色质化学修饰的同时成像分析依然是细胞成像分析领域的重大挑战。
技术实现要素:
为了克服上述现有技术的缺点,本发明的目的在于开发一种细胞大分子锚定的dna步移索引成像方法(cellularmacromolecules-tethereddnawalkingindexing,cell-talking),结合dna纳米技术,通过酶切核酸及链置换反应以驱动dna步移探针,从而实现dna编码探针的组装-解组装循环,实现多种目标物同时检测。该方法克服了无法进行多种染色质化学修饰的检测、难以区分相邻修饰分子的技术缺点。
为了达到上述目的,本发明采用以下技术方案予以实现:
本发明公开了一种细胞大分子锚定的dna步移索引成像方法,在cell-talking方法中,dna步移探针可以连续切除邻近的dna编码探针,使这些编码探针产生新的3′-oh,随后通过滚换扩增实现荧光信号放大,最终,通过cell-talking方法实现细胞纳米微环境中大分子锚定的dna步移索引的成像分析。
其中,cell-talking方法在细胞中实施时,主要包括以下步骤:
1)细胞进行固定、透性处理后,通过一抗识别组蛋白,二抗-dna交联探针识别一抗;
2)以5hmu、5hmc以及5fu的识别标记顺序,通过特异性酶识别和点击化学反应分别将对应的dna编码探针连接到三种化学修饰分子上;
3)杂交dna步移探针(已提前与walkingblocker步移阻挡探针探针杂交并形成双链);
4)加入内切酶引发dna步移探针连续切除邻近的dna编码探针,使这些编码探针产生新的3′-oh,随后通过滚换扩增实现荧光信号放大;
5)最后通过激光共聚焦荧光显微镜进行观察和分析。
与现有技术相比,本发明具有以下有益效果:
1、本发明所述的cell-talking方法,设计了dna步移探针和多种dna编码探针,这些dna编码探针的末端序列带有硫代磷酸和索核酸修饰,该修饰能够抑制核酸被外切酶切除,无法进行3′聚合延伸。当通过酶切核酸及链置换反应驱动dna步移探针,连续切除了邻近的dna编码探针,这些编码探针才会产生新的3′-oh,进行下一步的引物延伸反应。
2、本发明所述的cell-talking方法,可实现单细胞单分子成像。
3、本发明所述的cell-talking方法,反应体系简单,反应效率高,可实现细胞纳米环境内大分子锚定的多种染色质化学修饰分子的同时、快速、准确成像。
4、本发明所述的cell-talking方法,用于研究单细胞纳米环境内大分子锚定的多种染色质化学修饰分子,发现其在不同的翻译后修饰的组蛋白的邻近分布有所区别。
附图说明
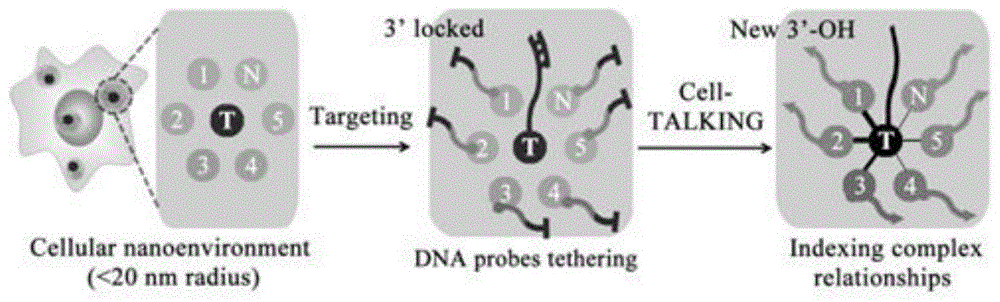
图1为本发明的方法原理概述图;
图2为本发明方法成像细胞大分子锚定的dna步移索引的具体步骤图;
图3为玻璃板上cell-talking方法的验证;其中,a为cell-talking方法可同时实现两种目标物的检测证明,左图为示意图,右图为荧光成像图;b为cell-talking方法可同时实现三种目标物的检测证明,左图为示意图,右图为荧光成像图;
图4为探索单细胞内不同纳米环境下的染色质修饰;其中,a为五种翻译后修饰的组蛋白邻近的多种染色质化学修饰的细胞成像图,标尺为5μm;b为五种翻译后修饰的组蛋白邻近的多种染色质化学修饰的点统计;c不同点的叠加组合分析,其中右侧的曲线图为左侧细胞成像内箭头上的荧光谱线;d单细胞中潜在的复杂组合修饰研究。
具体实施方式
为了使本技术领域的人员更好地理解本发明方案,下面将结合本发明实施例中的附图,对本发明实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例仅仅是本发明一部分的实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都应当属于本发明保护的范围。
需要说明的是,本发明的说明书和权利要求书及上述附图中的术语“第一”、“第二”等是用于区别类似的对象,而不必用于描述特定的顺序或先后次序。应该理解这样使用的数据在适当情况下可以互换,以便这里描述的本发明的实施例能够以除了在这里图示或描述的那些以外的顺序实施。此外,术语“包括”和“具有”以及他们的任何变形,意图在于覆盖不排他的包含,例如,包含了一系列步骤或单元的过程、方法、系统、产品或设备不必限于清楚地列出的那些步骤或单元,而是可包括没有清楚地列出的或对于这些过程、方法、产品或设备固有的其它步骤或单元。
下面结合附图对本发明做进一步详细描述:
参见图1和图2,为本发明的原理图和步骤图,图中灰色球表示核小体,棕色小旗子表示翻译后修饰的组蛋白,红色圆表示5hmu,紫色方框表示5hmc,绿色三角形表示5fu。本发明以细胞大分子锚定的dna步移索引为研究对象,通过cell-talking方法实现细胞纳米环境内大分子锚定的多种染色质化学修饰分子的同时单细胞单分子成像。
所述的cell-talking,包括以下三部分:
第一部分,通过一抗识别组蛋白,二抗-dna交联探针识别结合一抗;
第二部分,以5hmu、5hmc以及5fu的识别标记顺序,通过特异性酶识别和点击化学反应分别将对应的dna编码探针连接到三种化学修饰分子上;
其中,通过5-hmudk酶将带有炔基修饰的atp结合在5hmu位点上,随后通过点击化学反应标记编码探针-5hmu;通过t4β-gt酶将带有叠氮修饰的二磷酸尿苷葡糖结合在5hmc位点上,随后通过点击化学反应标记编码探针-5hmc;通过硼氢化钠将5fu还原为5hmu,通过5-hmudk酶将带有炔基修饰的atp结合在已被还原的5fu位点上,随后通过点击化学反应标记编码探针-5fu;
第三部分,杂交dna步移探针(已提前与walkingblocker步移阻挡探针探针杂交并形成双链)。随后加入内切酶引发dna步移探针连续切除邻近的dna编码探针,使这些编码探针产生新的3些编码探,最后通过滚换扩增实现荧光信号放大。
涉及序列如下表1所示:
表1:
‘p'和‘*’分别代表末端基团和硫代磷酸修饰。
具体地,本发明公开的细胞大分子锚定的dna步移索引成像方法,包括以下操作:
1)通过一抗识别组蛋白,二抗-dna交联探针识别结合一抗;
2)以5hmu、5hmc以及5fu的识别标记顺序,通过特异性酶识别和点击化学反应分别将对应的dna编码探针连接到三种化学修饰分子上;
3)杂交dna步移探针(已提前与walkingblocker步移阻挡探针探针杂交并形成双链);
4)加入内切酶引发dna步移探针连续切除邻近的dna编码探针,使这些编码探针产生新的3码探针,.
5)通过滚换扩增实现荧光信号放大。
6)通过激光共聚焦荧光显微镜进行观察,实现细胞纳米环境内大分子锚定的多种染色质化学修饰分子的同时成像。
实施例1利用cell-talking方法实现细胞纳米环境内大分子锚定的多种染色质化学修饰分子的同时成像。
以mcf-10a人乳腺细胞系为基本模型,用4%(质量/体积)多聚甲醛室温固定细胞10分钟,pbs清洗细胞3次后,用0.5%(体积/体积)tritonx-100在室温下对细胞进行透性处理5分钟;pbs清洗细胞3次后,细胞与quickblock性封闭缓冲液在室温下孵育1小时后,加入识别翻译后修饰的组蛋白的一抗,于4℃孵育过夜。pbst清洗细胞3次后,加入二抗-dna交联探针,室温孵育1小时。
用pbst洗涤细胞3次后,我们遵循标记顺序(先5hmu,然后5hmc,最后5fu)以区分细胞中这三种dna修饰。对于典型的5hmu磷酸化反应,将细胞与20u5-hmudk和500μm炔基修饰的atp在1xcutsmart缓冲液中于37℃孵育2小时。然后加入含有250nm编码探针-5hmu,1mm硫酸铜(ii),0.5μg/ml鲱鱼精dna和100mm抗坏血酸钠的溶液,在室温下避光反应1小时。仔细洗涤后,进行5hmc的糖基化反应。将细胞与含有50μm叠氮修饰的二磷酸尿苷葡糖和5ut4β-gt的20μlt4磷酸尿苷葡糖有多种在37℃下孵育2小时。在100nm编码探针-5hmc和0.5μg/ml鲱鱼精dna的存在下,在37℃下进行无铜点击反应60分钟。随后,避光室温下用1mg/ml硼氢化钠将5fu还原为5hmu。洗涤3次后,通过上述的磷酸化和点击反应,用编码探针-5fu标记新产生的5hmu。pbs清洗细胞3次后,将已制备好的dna步移探针(已提前与步移阻挡探针探针杂交并形成双链)添加到细胞中,其可以被二抗-dna交联探针捕获。
经过上述标记反应后,进行了内切酶辅助的dna步移反应、环型探针杂交反应以及滚换扩增。为了进行dna步移反应,将包含5unt.bbvci内切酶的20μl1xcutsmart缓冲液添加到细胞样品中,并在37℃下孵育2小时。在此反应过程中,每个步移探针分子被等温释放以识别附近的任何编码探针,使编码探针产生新的3'-oh末端。然后加入三个环化挂锁(每个200nm),被具有新的3'-oh的编码探针捕获,在37℃下与细胞孵育3小时;2xssc清洗细胞3次后,加入10μl1xphi29缓冲液,该缓冲液中包含5uphi29聚合酶、2.5mmdntps和0.5μlbsa(10mg/ml),于37℃孵育2小时。洗涤后,加入20μl杂交混合液,杂交混合液中包括200nmfam-探针、200nmcy3-探针、200nmcy5-探针和20%甲酰胺,在37℃下培养30分钟;2xssc清洗细胞3次后,加入20μldapi工作液染细胞核,在室温下培养10分钟,最终利用激光共聚焦荧光显微镜对细胞纳米环境内大分子锚定的多种染色质化学修饰分子进行同时成像分析。
如图1所示,因细胞内纳米微环境小于20nm,探测纳米微环境非常困难。为了解决以上问题,基于dna纳米技术,我们设计了dna步移探针和多种dna编码探针,这些dna编码探针的末端序列带有硫代磷酸和索核酸修饰,该修饰能够抑制核酸被外切酶切除,无法进行3′聚合延伸。当通过酶切核酸及链置换反应驱动dna步移探针,连续切除了邻近的dna编码探针,这些编码探针才会产生新的3′-oh,进行下一步的引物延伸反应。由于染色质上的修饰碱基的含量非常低,因此通过滚换扩增方法实现荧光信号放大。
首先,我们在玻璃板上验证cell-talking方法的可行性(此处用的dna序列为玻璃板实验的序列),由图3中a所示,1对2索引表示1种dna步移探针同时切除2种dna编码探针,从荧光成像图可看出,两种荧光信号完全叠加,说明我们设计的cell-talking方法可实现1对2的同时成像分析。由图3中b所示,1对3索引表示1种dna步移探针同时切除3种dna编码探针,从荧光成像图可看出,两种荧光信号完全叠加,说明我们设计的cell-talking方法可实现1对3的同时成像分析。
进一步地,实现cell-talking方法在单细胞内的有效运行。由图4中a所示,五种翻译后修饰的组蛋白邻近的多种染色质化学修饰的细胞成像图上均出现了三种不同目标物点(红色代表5hmu、绿色代表5fu和蓝色代表5hmc),该结果说明cell-talking方法可实现胞内纳米环境下的不同染色质化学修饰的同时检测。随后我们对这五种组蛋白上邻近的3种修饰的个数及分布规律进行了统计学研究(图4中b,c和d),发现它们不尽相同。
以上内容仅为说明本发明的技术思想,不能以此限定本发明的保护范围,凡是按照本发明提出的技术思想,在技术方案基础上所做的任何改动,均落入本发明权利要求书的保护范围之内。
序列表
<110>西安交通大学
<120>一种细胞大分子锚定的dna步移索引成像方法
<160>21
<170>siposequencelisting1.0
<210>1
<211>58
<212>dna
<213>人工序列(artificialsequence)
<400>1
agtcagagtcagagtgtagacagtaaaaaaaaaaaaaaaaaaaactgctgaggcacat58
<210>2
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>2
tatgtgcctcagcagagt18
<210>3
<211>36
<212>dna
<213>人工序列(artificialsequence)
<400>3
actgtctacactctgactctgactaaaaaaaaaaaa36
<210>4
<211>55
<212>dna
<213>人工序列(artificialsequence)
<400>4
atcctcgtaaatcctcatcaatcatctatgtgcctcagcagaggagaatcacnag55
<210>5
<211>55
<212>dna
<213>人工序列(artificialsequence)
<400>5
ggagtgcagcaaacgggaagagtctttatgtgcctcagcagaggagaatcacnag55
<210>6
<211>55
<212>dna
<213>人工序列(artificialsequence)
<400>6
gctatgtttcttgaggagggcagcaatatgtgcctcagcagaggagaatcacnag55
<210>7
<211>56
<212>dna
<213>人工序列(artificialsequence)
<400>7
gaggatttactcgacagagcttactcacagccagcatcacaaggtcgatgattgat56
<210>8
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>8
gtaaatcctcatcaatcatc20
<210>9
<211>55
<212>dna
<213>人工序列(artificialsequence)
<400>9
ccgtttgctgtggcctgagccttcctcggtacggtctgtaaggtcaagactcttc55
<210>10
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>10
cagcaaacgggaagagtctt20
<210>11
<211>55
<212>dna
<213>人工序列(artificialsequence)
<400>11
cctcaagaaatcgacgcgtataataatgctgggctctagtaggtcttgctgccct55
<210>12
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>12
tttcttgaggagggcagcaa20
<210>13
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>13
agagcttactcacagccagcatcaca26
<210>14
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>14
tgagccttcctcggtacggtctgta25
<210>15
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>15
gcgtataataatgctgggctctagt25
<210>16
<211>102
<212>dna
<213>人工序列(artificialsequence)
<400>16
actcggaaggagccatgccagacattcctgtccgaagtcccggggaacgtaactcatgtc60
aaggtacactctgactctgactcaacgattataacagttttt102
<210>17
<211>77
<212>dna
<213>人工序列(artificialsequence)
<400>17
agtcagagtcagagtgtagacagtaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaa60
aaactgctgaggcacat77
<210>18
<211>70
<212>dna
<213>人工序列(artificialsequence)
<400>18
aaactgttataatcgttgtcctcgtaaatcctcatcaatcatctatgtgcctcagcagag60
gagaatcacg70
<210>19
<211>70
<212>dna
<213>人工序列(artificialsequence)
<400>19
ccttgacatgagttacgtgagtgcagcaaacgggaagagtctttatgtgcctcagcagag60
gagaatcacg70
<210>20
<211>70
<212>dna
<213>人工序列(artificialsequence)
<400>20
tccccgggacttcggacactatgtttcttgaggagggcagcaatatgtgcctcagcagag60
gagaatcacg70
<210>21
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>21
ggaatgtctggcatggctccttccgagt28
- 还没有人留言评论。精彩留言会获得点赞!