一种定点敲除水稻OsAUR2基因的sgRNA的oligoDNA组的制作方法
发明属于植物基因工程领域。具体的说,本发明涉及一种基于crispr-cas9技术定点敲除水稻osaur2基因的sgrna的oligodna组及应用。
背景技术:
aurora激酶存在于所有真核生物,在进化上非常保守,是有丝分裂的关键决定因子。aurora激酶是丝氨酸/苏氨酸激酶家族成员,酵母中只有一个aurora激酶,而多细胞真核生物至少有两个功能不同成员。aurora激酶是细胞调节有丝分裂必需的激酶,特别是染色体分离过程。此外,它们还参与了减数分裂过程。目前,aurora激酶在动物细胞中的功能研究较为清楚,尤其是人类细胞。人类细胞中有三个aurora激酶成员:auroraa,aurorab和aurorac。aurora激酶的缺失可能导致细胞分裂的失败损害胚胎发育,而在许多癌症中aurora激酶的表达量上调。越来越多的研究已经证明抑制aurora激酶可以增强化疗。在过去的几十年里,一系列aurora激酶抑制剂(akis)有效地抑制了许多癌症的发展和生长。这表明极光激酶可能是一种新的治疗靶点。
相比之下,植物中aurora激酶的功能研究才刚刚开始。然而,通过遗传学、细胞生物学和生物化学方法已经发现植物中aurora激酶在形成(不对称)分裂、染色质修饰和维持基因组的稳定性发挥重要作用,但不同aurora激酶存在一定功能差异性。拟南芥中有三个aurora激酶:ataur1、ataur2和ataur3。ataur1和ataur2为α-aurora,功能类似。而ataur3为β-aurora,定位与功能与其它两个成员都有不同。ataur3定位于分生组织细胞间期的细胞核,而分裂期定位于着丝粒,可能在染色体分离发挥作用。by2细胞过表达ataur3导致异常的细胞分裂模式,包括分裂方向异常,纺锤丝形成存在缺陷等。
有丝分裂是真核生物最重要的生命活动,而水稻是我国主要的粮食作物,因此研究水稻中aurora激酶的功能对水稻正常发育,确保产量以及抗逆都具有重要意义。水稻中aurora激酶只有两个成员,α-aurora和β-aurora各一个,分别是osaur1和osaur2。目前为止,对水稻osaur2的功能研究几乎空白,而osaur2对应的突变体也并无发现。因此,敲除水稻中osaur2基因获得其突变体对分析其功能以及阐明水稻有丝分裂机制具有重要的作用。本发明通过设计sgrna构建基因编辑载体、转基因技术和crispr-cas9技术获得转基因植株、测序分析获得osaur2基因编辑(敲除)的植株及序列编辑情况。
技术实现要素:
本发明的目的在于提供一种敲除水稻aurora激酶基因osaur2的方法,提供了一种基于crispr-cas9技术敲除水稻osaur2的sgrna以及用于敲除水稻osaur2基因的载体。
使用crispr/cas9技术靶向敲除水稻osaur2的方法,其特征在于,包括如下步骤:
a)选择osaur2基因编码区第36至55核酸序列作为crispr/cas9系统的靶序列(seqidno.1):gatcggcaagtacatcggcg;
根据靶序列设计两条oligodna:
osaur2-f1(seqidno.2):caggatcggcaagtacatcggcg
osaur2-r1(seqidno.3):aaccgccgatgtacttgccgatc;
b)将oligodna组osaur2-f1和osaur2-r1混合,通过退火反应形成二聚体结构,然后与载体片段vk005进行连接,构建得到含有水稻osaur2基因靶序列的质粒vk005-osaur2;
c)用含有vk005-osaur2质粒的根癌农杆菌eha105侵染水稻的愈伤组织,通过潮霉素筛选,再生获得转基因水稻植株,并用hptⅱ基因特异引物进行转基因鉴定;
d)利用sq-osaur2-f(seqidno.4)和sq-osaur2-r(seqidno.5)对靶序列附近序列进行扩增,扩增基因组片段进行测序,鉴定osaur2编辑情况并筛选敲除植株;
sq-osaur2-f(seqidno.4):ccatatccattccacccagtgcggc
sq-osaur2-r(seqidno.5):gctcacagtagcagcggtgcgctg
本发明的有益效果是基因编辑载体vk005-osaur2转化水稻后,即可实现高效、快速、特异对水稻osaur2基因进行靶向编辑和敲除,可直接用此做研究材料来探讨osaur2基因的功能和作用机理,为在生产实践上更好利用osaur2基因进行遗传改良奠定基础。
附图说明
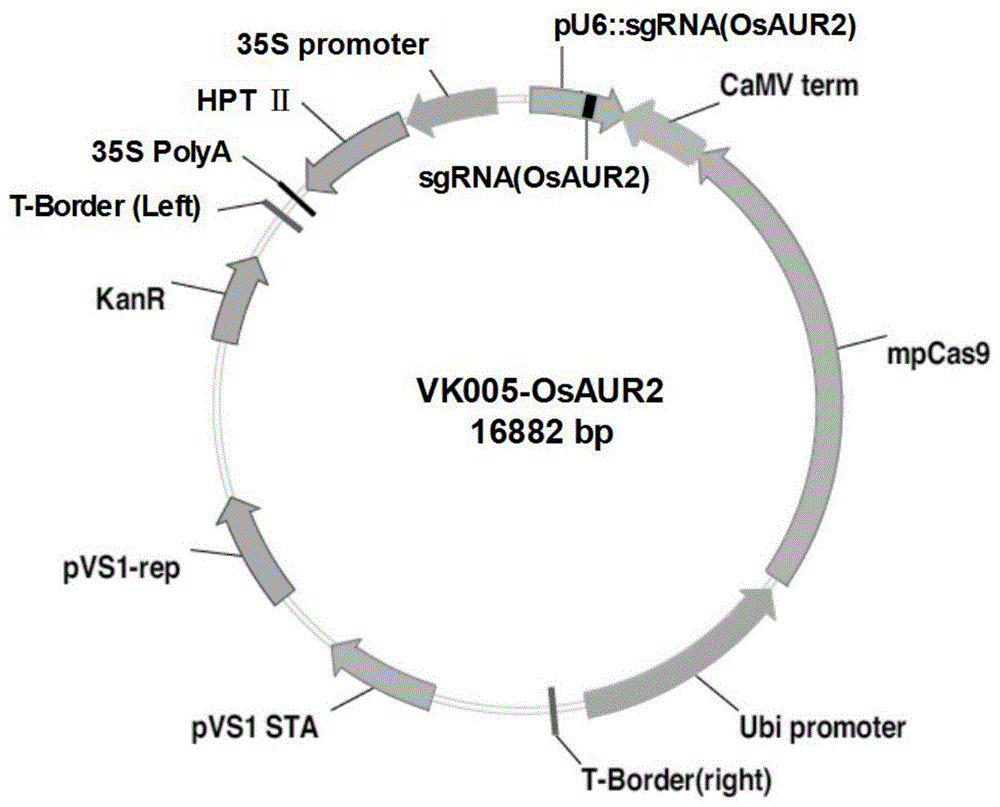
图1osaur2基因编辑载体vk005-osaur2结构示意图。
图2vk005-osaur2转基因鉴定图。
图3转基因阳性植株靶位点附近区域pcr产物。
图4转基因2号株系osaur2基因编辑情况。
图5转基因4号株系osaur2基因编辑情况。
图6转基因9号株系osaur2基因编辑情况。
图7转基因22号株系osaur2基因编辑情况。
具体实施方式
下面结合具体实施例对本发明进行进一步描述。这些描述并不是对本发明内容作进一步的限定,以下实施例中的若未特别说明,所用的技术手段为本领域技术人员所熟知的常规手段。
下述实施例中所用的材料、试剂等,如无特殊说明,均可从商业途径得到。
实施例1、水稻osaur2基因sgrna序列的设计与合成
水稻osaur2基因的编码区序列如seqidno.7所示。
本实施例crispr/cas9编辑靶序列长度为20bp,位于osaur2编码区第一外显子的第36至55碱基位,编辑的靶序列为seqidno.1:gatcggcaagtacatcggcg,该序列在水稻基因组上特异,脱靶概率极低。
根据靶序列合成两条oligodna:
osaur2-f1(seqidno.2):caggatcggcaagtacatcggcg
osaur2-r1(seqidno.3):aaccgccgatgtacttgccgatc;
实施例2、osaur2编辑载体vk005-osaur2构建
将合成的osaur2-f1和osaur2-r1加水溶解至10μm,按下列反应体系混合后,95℃加热3分钟,自然冷却退火使osaur2-f1和osaur2-r1形成二聚体结构,然后与vk005载体片段进行连接,通过热激法将连接产物转化大肠杆菌,挑选单菌落至lb液体培养基(+kan)培养12小时,测序。挑选测序正确的细菌提取质粒dna,获得含有水稻osaur2基因靶序列的编辑载体vk005-osaur2,载体图见图1。
实施例3、vk005-osaur2载体农杆菌转化
用冻融法将vk005-osaur2质粒转化如农杆菌,将10μl质粒dna加入200μl农杆菌eha105感受态中,冰浴30min,液氮冷冻3min,37℃水浴5min,加入1mlyeb培养基,28℃摇培3-4h。6000rpm,室温离心1min,弃上清,加入200μlyeb培养基重悬菌体,涂于yeb固体培养基上(+利福平),28℃培养3天。挑单菌落鉴定。
实施例4、水稻转化
本实施选用日本晴作为受体进行农杆菌转化。选用300粒左右日本晴种子,去壳后用75%的乙醇浸泡1分钟,倒掉75%乙醇后用次氯酸钠溶液消毒30分钟,用无菌水洗6次,用灭菌纱布吸干水分后将种子种到含2,4-d(2mg/l)的nb培养基上26℃避光培养2周,挑选生长旺盛的愈伤用作转化的受体。用含有编辑载体(vk005-osaur2)的eha105菌株制备的工程菌液侵染水稻愈伤,在黑暗、25℃条件下共培养3天后,在含有50mg/lhygromycin的筛选培养基上光照培养14天左右(光照强度为13200lx,温度为32℃)。将预分化的愈伤转至分化培养基上在光照条件下(光照强度为13200lx,温度为32℃)培养一个月左右得到抗性转基因植株。用1/2ms培养基生根壮苗获得t0代植株,移入田间种植。
实施例5、osaur2基因编辑情况分析
取t0代植株叶片提取dna,根据潮霉素基因序列设计引物hpt-f和hpt-r,确定阳性转基因植株,见图2。然后用引物sq-osaur2-f和sq-osaur2-r扩增阳性转基因植株(图3),通过测序分析osaur2基因靶位点附近序列(图4-7为部分被编辑植株的测序结果),总体结果统计见表1。潮霉素基因与靶位点附近序列扩增引物序列为:
sq-osaur2-f(seqidno.4):ccatatccattccacccagtgcggc
sq-osaur2-r(seqidno.5):gctcacagtagcagcggtgcgctg
hpt-f(seqidno.6):acggtgtcgtccatcacagtttgcc
hpt-r(seqidno.7):ttccggaagtgcttgacattgggga
表1.osaur2基因编辑情况分析
以上所述仅为本发明较佳的实施例,并非因此限制本发明的实施方式及保护范围,对于本领域技术人员而言,应当能够意识到凡运用本发明说明书及图示内容所作出的等同替换和显而易见的变化所得到的方案,均应当包含在本发明的保护范围内。
序列表
<110>江西农业大学
<120>一种定点敲除水稻osaur2基因的sgrna的oligodna组
<160>8
<170>siposequencelisting1.0
<210>1
<211>20
<212>dna
<213>oryzasativa
<400>1
gatcggcaagtacatcggcg20
<210>2
<211>23
<212>dna
<213>人工序列
<400>2
caggatcggcaagtacatcggcg23
<210>3
<211>23
<212>dna
<213>人工序列
<400>3
aaccgccgatgtacttgccgatc23
<210>4
<211>25
<212>dna
<213>人工序列
<400>4
ccatatccattccacccagtgcggc25
<210>5
<211>24
<212>dna
<213>人工序列
<400>5
gctcacagtagcagcggtgcgctg24
<210>6
<211>25
<212>dna
<213>人工序列
<400>6
acggtgtcgtccatcacagtttgcc25
<210>7
<211>25
<212>dna
<213>人工序列
<400>7
ttccggaagtgcttgacattgggga25
<210>8
<211>930
<212>dna
<213>oryzasativa
<400>8
atggagaagccggagtggagcatggacgacttcgagatcggcaagtacatcggcgagggc60
aagttcgggaaggtctacctcgcccgcgagaagcagagcggctacgtggtggcgctcaag120
gtgacattcaaggcgaagctggacaagtacaggttccacgcgcacctgcggcgggagatc180
gagatccagcacggcctcgaccaccccaacgtgctccgtctcttcgcctggttccacgac240
gccgagcgcgtcgtcctcgtgctcgagtacgccgcccgtggggagctttacaagctcctc300
cgcaccgtccgccgcttctctgagcgcaccgctgctacttatgttgccagccttgccggt360
gcactagcatactgtcataagaagcaagttatccatcgggacatcaagccagagaattta420
ctccttgatattgagggccgacttaaaattgcagattttggatgggcggttagatcaaat480
gctaaacgacacacactttgtggtacaattgattacctggcacctgaaatgatagagaaa540
aaggctcatgaccatgctgttgacaactggactttaggaatcttgtgctatgagttctta600
tatgggtcaccaccttttgaagcagcagaacaggatgatacattgaggaggatagttaaa660
gtggatttgtcgttcccctcaactccttatgtttctgcagatgctaaggatctcatttgc720
aaggtaaaatttgtggtattgattatacctattgtatatgttacactaaaatgggttgaa780
cttttgagttttgagcttcgagttcgtatgcagcttttggtgaaggattcaaacaagaga840
ctttctcttgatgacataatgaaacatccatggattgtgaagaatgcagacccatcaggg900
agttgtagtgaccaaaaggctcgcacataa930
- 还没有人留言评论。精彩留言会获得点赞!