促进蛋白胞外表达的信号肽的制作方法
本发明涉及促进蛋白胞外表达的信号肽,属于基因工程及酶工程技术领域。
背景技术:
普鲁兰酶(pullulanase,ec3.2.1.41)是一种被广泛使用的淀粉脱支酶,它可以水解淀粉多糖如普鲁兰多糖、支链淀粉、α-糊精、β-糊精、糖原和相关多糖中的α-1,6糖苷键。普鲁兰酶已经被广泛的应用于乙醇燃料、环糊精、抗性淀粉、麦芽三糖糖浆等产品的生产中。因为其水解多糖的性质,普鲁兰酶的表达体系最好能在食品级表达系统中构建。
枯草芽孢杆菌在食品工业、抗体生产和畜牧业中均具有重要的应用。但是,目前在枯草芽孢杆菌中普鲁兰酶的表达量很低,而且需要检测细胞生长状态,在合适的时机添加昂贵的蔗糖诱导剂,因此有必要进一步优化普鲁兰酶的产量。在目前的研究中主要是通过添加信号肽或对普鲁兰酶进行突变,以期能够提高酶的胞外表达量。对于普鲁兰酶本身的突变,虽然可以提高其胞外的表达量,但是常会使其本身的催化性质发生改变;而信号肽的添加虽然可提高酶的胞外表达量,但是其提高量仍然有限。
因此,需要提供一种新的方法来保证普鲁兰酶本身性质不发生改变的同时,又可提高其胞外表达量。
技术实现要素:
为了解决上述存在的问题,本发明选择了枯草芽孢杆菌中的信号肽ggt、ywea、yola,并对这些信号肽n端编码区(ncs)进行同义突变,这样既能够确保蛋白翻译不受影响的情况,又筛选出能显著提高蛋白表达量的信号肽,为蛋白的高效表达提供了一种新方法。
本发明提供了一类信号肽,所述信号肽的核苷酸序列如seqidno.1~3所示任一。
在本发明的一种实施方式中,所述信号肽的核苷酸序列分别是信号肽ggt、ywea和yola的同义突变序列。
在本发明的一种实施方式中,信号肽ggt的野生型序列如seqidno.8所示。
在本发明的一种实施方式中,信号肽ywea的野生型序列如seqidno.9所示。
在本发明的一种实施方式中,信号肽yola的野生型序列如seqidno.10所示。
本发明提供了含有信号肽ggt、ywea和yola的同义突变序列的重组质粒。
在本发明的一种实施方式中,信号肽ggt的野生型序列如seqidno.8所示。
在本发明的一种实施方式中,信号肽ywea的野生型序列如seqidno.9所示。
在本发明的一种实施方式中,信号肽yola的野生型序列如seqidno.10所示。
在本发明的一种实施方式中,所述重组质粒上含有lytr启动子,lytr启动子的核苷酸序列如seqidno.4所示。
在本发明的一种实施方式中,所述重组质粒以p43nmk为出发载体。
本发明提供了含有核苷酸序列如seqidno.1~3所示的信号肽,或所述重组质粒的重组菌。
在本发明的一种实施方式中,所述重组菌以枯草芽孢杆菌wb600为宿主。
本发明提供一种提高蛋白表达量的方法,利用所述含有核苷酸序列如seqidno.1~3所示的信号肽的生产蛋白。
在本发明的一种实施方式中,发酵培养基为tb培养基,在30~40℃、200~250rpm培养20~30小时。
本发明提供一种提高枯草芽孢杆菌胞外蛋白表达量的方法,在编码蛋白的核苷酸序列的n端添加核苷酸序列如seqidno.1~3所示的信号肽。
在本发明的一种实施方式中,在目的蛋白的n端添加核苷酸序列如seqidno.1~3所示的信号肽,并构建重组质粒,将重组质粒导入枯草芽孢杆菌获得重组菌,再利用此重组菌生产胞外蛋白。
在本发明的一种实施方式中,所述目的蛋白包括任意枯草芽孢杆菌分泌至胞外的蛋白。
在本发明的一种实施方式中,所述目的蛋白包括但不限于普鲁兰酶和/或sfgfp。
在本发明的一种实施方式中,所述普鲁兰酶的ncbi登录号为amq67157,根据枯草芽孢杆菌密码子偏好性进行优化,氨基酸序列如seqidno.5所示,核苷酸序列如seqidno.6所示。
在本发明的一种实施方式中,所述sfgfp的核苷酸序列如seqidno.7所示。
本发明还提供一种提高酶活普鲁兰酶表达量的方法,将核苷酸序列如seqidno.1~3所示的信号肽添加至普鲁兰酶的n端。
在本发的一种实施方式中,将核苷酸序列如seqidno.1~3所示的信号肽添加至普鲁兰酶的n端构建得到重组质粒,接着将重组质粒转入宿主中得到重组菌,再将重组菌在培养体系中发酵生产普鲁兰酶。
在本发的一种实施方式中,将核苷酸序列如seqidno.1~3所示的信号肽插入编码普鲁兰酶的核苷酸序列的起始密码子atg后。
在本发的一种实施方式中,所述培养体系中含有蛋白胨10~20g/l,酵母提取物20~30g/l,甘油1~5ml/l。
本发明还保护核苷酸序列如seqidno.1~3所示的信号肽,或所述重组质粒,所述重组菌在提高胞外蛋白表达量中的应用。
本发明还保护所述提高蛋白表达量的方法,或所述提高枯草芽孢杆菌胞外蛋白表达量的方法在提高胞外蛋白表达量中的应用。
本发明的有益效果:
本发明通过同义突变信号肽所得的核苷酸序列,分别将其融合在普鲁兰酶的n端,并转入枯草芽孢杆菌中进行表达,可显著提高和降低普鲁兰酶的胞外酶活产量。在这里,我们对筛选到的三条能促进普鲁兰酶分泌表达的信号肽(ggt,ywea,yola),在n端前十个氨基酸进行同义突变,通过荧光蛋白的表征,在原有基础上,可进一步显著提高普鲁兰酶的胞外酶活,相比优化前提高了145%、59%和104%。
附图说明
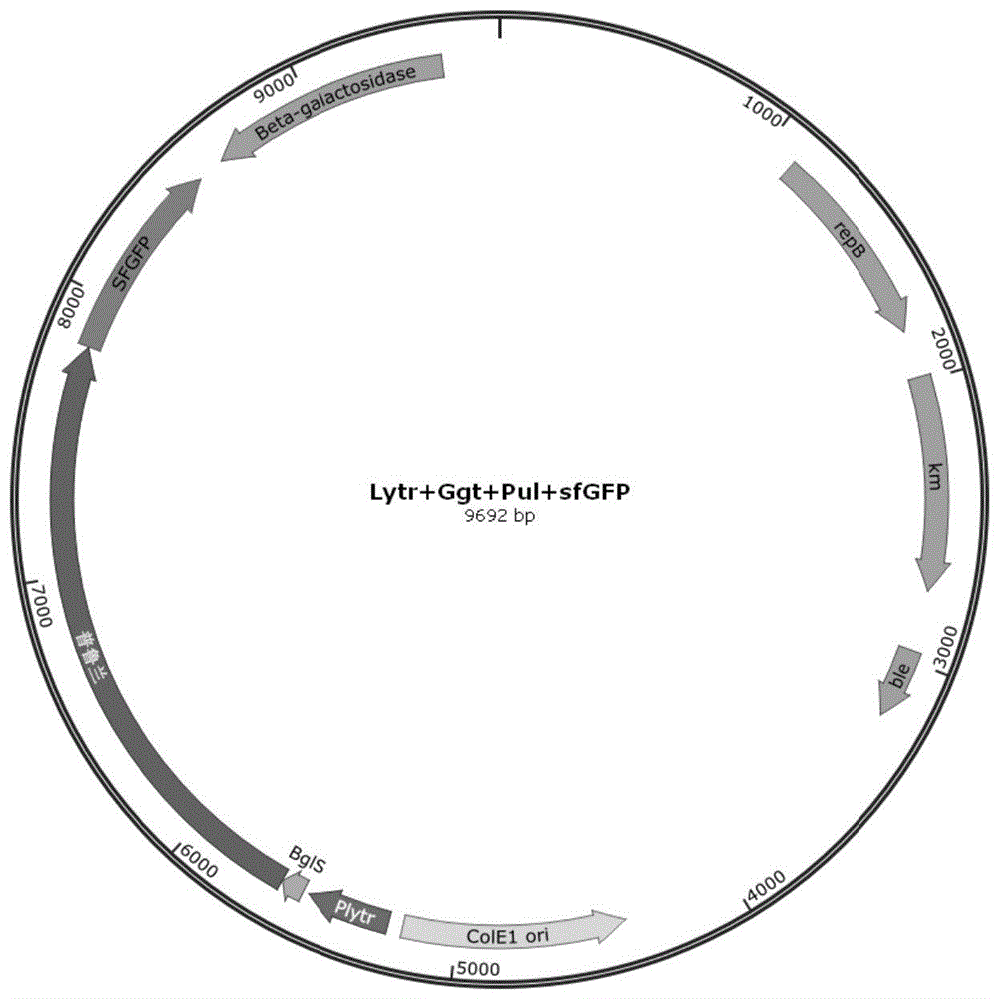
图1为信号肽和sfgfp分别融合在普鲁兰酶n端和c端的表达质粒图谱(以ggt为例)。
图2为信号肽融合在普鲁兰酶n端的表达质粒图谱(以ggt为例)。
图3为野生型与ncs改造菌株分泌的普鲁兰酶胞外酶活。
具体实施方式
1、培养基
种子培养基(g/l):蛋白胨10,酵母提取物5,氯化钠5;
发酵培养基(tb培养基):将下列组分溶解在0.9l水中:蛋白胨12g,酵母提取物24g,甘油4ml;各组分溶解后高压灭菌;冷却到60℃,再加100ml灭菌的0.17mol/l的k2hpo4、0.72mol/l的k2hpo4溶液(2.31g的k2hpo4和12.54g的k2hpo4溶在足量的水中,使终体积为100ml;0.22μm的滤膜过滤除菌)。
2、培养方法
种子培养:挑取工程菌单菌落接入装液量为25ml的三角瓶(250ml)中,培养温度37℃,摇床转速200r/min,培养12h;
发酵培养:按4%的接种量接入装液量为25ml的三角瓶(250ml)中,培养温度37℃,发酵48h。
3、绿色荧光蛋白表达量及生物量测定
在96孔板中假如200μl稀释后的发酵液,使用cytation3细胞成像微孔板检测仪(美国伯腾仪器有限公司),绿色荧光激发波长:480nm,绿色荧光发射波长:520nm,细胞生长od吸收波长:600nm。
4、普鲁兰酶酶活测定方法
将1ml1mg/100ml的普鲁兰多糖底物和0.9ml100mmph4.5乙酸-乙酸钠缓冲液混合均匀,置于60℃水浴锅内预热10min,加入普鲁兰酶液0.1ml,反应10min后,加入3mldns显色液,然后于沸水浴中煮7min,置于冰水中终止显色反应,再加10ml去离子水,混匀,在540nm下测定吸光值。
单位时间内生成1μmol还原糖的酶量定义为一个酶活力单位。
实施例1:构建ggt信号肽ncs同义突变文库
为了促进普鲁兰酶的高效表达,将先前实验室表征过的内源性转录水平最高的枯草芽孢杆菌转录lytr启动子(序列信息见seqidno.4所示)通过一步克隆试剂盒(购自南京诺唯赞生物科技有限公司公司)连接至p43nmk质粒,构建得到重组质粒,将重组质粒转入e.colijm109中,将菌液涂布于含有50μg/ml氨苄霉素抗性的lb平板,37℃培养至长出单克隆,挑取单克隆经培养后提取质粒测序验证,得到质粒p43nmk-lytr;利用相同的一步克隆方法,将普鲁兰酶基因(核苷酸序列如seqidno.6所示)通过一步克隆试剂盒融合至lytr的下游,构建得到重组质粒p43nmk-lytr-pul;利用相同的一步克隆方法,将sfgfp荧光蛋白(核苷酸序列如seqidno.7所示)连接至普鲁兰酶的c端,将ggt信号肽野生型(核苷酸序列如seqidno.8所示)连接至普鲁兰酶的n端,构建得到重组质粒p43nmk-ggt-pul。
以p43nmk-ggt-pul为模板,使用简并引物(核苷酸序列如seqidno.11和seqidno.12所示),pcr获得ggt的n端前30位碱基的同义突变文库(同义突变重组质粒),即前30位氨基酸序列不变,核苷酸序列改变。
实施例2:构建ywea信号肽ncs同义突变文库
具体实施方式同实施例1,区别在于,在得到p43nmk-lytr-pul后,利用一步克隆法,分别将sfgfp荧光蛋白连接至普鲁兰酶的c端,将ywea信号肽野生型(核苷酸序列如seqidno.9所示)连接至普鲁兰酶的n端,构建得到重组质粒p43nmk-ywea-pul。
以p43nmk-ywea-pul为模板,使用简并引物(核苷酸序列如seqidno.11和seqidno.13所示),pcr获得ywea的n端前30位碱基的同义突变文库(同义突变重组质粒),即前30位氨基酸序列不变,核苷酸序列改变。
实施例3:构建yola信号肽ncs同义突变文库
具体实施方式同实施例1,区别在于,在得到p43nmk-lytr-pul后,利用一步克隆法,分别将sfgfp荧光蛋白连接至普鲁兰酶的c端,将yola信号肽野生型(核苷酸序列如seqidno.10所示)连接至普鲁兰酶的n端,构建得到重组质粒p43nmk-yola-pul。
以p43nmk-yola-pul为模板,使用简并引物(核苷酸序列如seqidno.11和seqidno.14所示),pcr获得yola的n端前30位碱基的同义突变文库(同义突变重组质粒),即前30位氨基酸序列不变,核苷酸序列改变。
实施例4:ncs同义突变文库的筛选
将实施例1~3中构建得到的同义突变重组质粒分别转化至表达宿主枯草芽孢杆菌wb600中,将转化液涂布于50μg/ml卡纳霉素抗性的lb平板,在37℃下培养至长出单克隆,挑取单克隆至含有200μllb培养基的96浅孔板,培养种子液8小时;
接着,按照4ml/100ml的接种量接种至含有50μg/ml卡纳霉素抗性的800μltb培养基的96深孔板,培养24小时,得到发酵液;
然后将发酵液迅速置于冰上冷冻,离心后,去除上清,将100mm的pbs缓冲液(ph7.2)稀释至合适倍数后,通过酶标仪测定荧光值(激发光480,吸收光520)以及od600。
定义相对荧光强度rfi=荧光值/od,根据rfi值的高低,选择相对荧光强度最高的细胞送至上海生工公司测序。
经测序鉴定后,ggt信号肽在n端成功实现同义突变,突变的rfi值最高的核苷酸序列如seqidno.1所示;ywea信号肽在n端成功实现同义突变,突变的rfi值最高核苷酸序列如seqidno.2所示;yola信号肽在n端成功实现同义突变,突变的rfi最高的核苷酸序列如seqidno.3。
实施例5:同义突变信号肽在生产普鲁兰酶中的应用
分别选择ggt、ywea、yola经序列鉴定无误后的rfi值最高的变体质粒,使用引物(核苷酸序列如seqidno.15和16所示)通过pcr去除sfgfp荧光蛋白,以减少质粒片段大小从而增强质粒稳定性。将去除了sfgfp荧光蛋白后得到的质粒分别转化到枯草芽孢杆菌wb600中,并将其接种至含有50μg/ml卡纳霉素抗性的20mllb培养基的250ml摇瓶中,37℃、220rpm发酵8小时后,使得od600达到4以上,按照4ml/100ml的比例接种到含有50μg/ml卡纳霉素抗性25mltb培养基的250ml摇瓶中,在37℃、250rpm发酵30小时,发酵结束后测定普鲁兰酶的胞外酶活,结果如图3和表1所示:在通过ncs改造之后,由信号肽ggt,ywea,yola诱导的的普鲁兰酶酶活显著得到提高,分别较野生型提高了145%、59%和104%。
表1普鲁兰酶胞外酶活(u/ml)
虽然本发明已以较佳实施例公开如上,但其并非用以限定本发明,任何熟悉此技术的人,在不脱离本发明的精神和范围内,都可做各种的改动与修饰,因此本发明的保护范围应该以权利要求书所界定的为准。
sequencelisting
<110>吉林中粮生化有限公司
江南大学
<120>促进蛋白胞外表达的信号肽
<160>16
<170>patentinversion3.3
<210>1
<211>81
<212>dna
<213>人工序列
<400>1
aaaagaacttggaatgtatgtttaactgcactgcttagtgttctgttagtcgctggaagt60
gtcccttttcacgcggaagct81
<210>2
<211>87
<212>dna
<213>人工序列
<400>2
ctaaaaagaacatcatttgtatcatctttattcatcagttcagctgttttactatcaatc60
ttacttccttcgggccaagctcatgca87
<210>3
<211>81
<212>dna
<213>人工序列
<400>3
aaaaaaagaattacttattctctacttgctcttctagcagttgttgctttcgctttcact60
gattcatcaaaagcaaaagcg81
<210>4
<211>320
<212>dna
<213>人工序列
<400>4
ctaaccctacataagtaccttcttttgtttcaatgttactgtctggcgatacatcttcac60
cttgactcttttgactattaaccccgcaacccgaaagaagcaatataaagaacagtaaag120
caataaattttttcatttttttcacctcattatattttatcgtcaacctattttatattt180
taaagaaaaattaagaaacaatgaaacttttttttataaaaaacgactattttaggattt240
cattcttgtattaaatagagttgtatttattggaaatttaactcataatgaaagtaattt300
aaaggaggtgaaatgtacac320
<210>5
<211>724
<212>prt
<213>人工序列
<400>5
aspalaalalysproalavalserasnalatyrleuaspalaserasn
151015
glnvalleuvallysleuserglnproleuthrleuglygluglyala
202530
serglyphethrvalhisaspaspthralaasnlysaspileproval
354045
thrservallysaspalaserleuglyglnvalgluserglyvallys
505560
thraspleuvalthrvalthrleuglygluaspproaspvalserhis
65707580
thrleuserileglnthraspglytyrglnalalysglnvalilepro
859095
argasnvalleuasnserserglntyrtyrtyrserglyaspaspleu
100105110
glyasnthrtyrthrglnlysalathrthrphelysvaltrpalapro
115120125
thrserthrglnvalasnvalleuleutyraspseralathrglyser
130135140
valthrlysilevalprometthralaserglyhisglyvaltrpglu
145150155160
alathrvalasnglnasnleugluasntrptyrtyrmettyrgluval
165170175
thrglyglnglyserthrargthralavalaspprotyralathrala
180185190
ilealaproasnglythrargglymetilevalaspleualalysthr
195200205
aspproalaglytrpasnserasplyshisilethrprolysasnile
210215220
gluaspgluvaliletyrglumethisvalargasppheserileasp
225230235240
proasnserglymetlysasnlysglylystyrleualaleuthrglu
245250255
lysglythrlysglyproaspasnvallysthrglyileaspserleu
260265270
lysglnleuglyilethrhisvalglnleumetprovalphealaser
275280285
asnservalaspgluthraspprothrglntyrasntrpglytyrasp
290295300
proargasntyraspvalprogluglyglntyralathrasnalaasn
305310315320
glyasnalaargilelysgluphelysglumetvalleuserleuhis
325330335
arggluhisileglyvalasnmetaspvalvaltyrasnhisthrphe
340345350
alathrglnileserasppheasplysilevalproglutyrtyrtyr
355360365
argthraspaspalaglyasntyrthrasnglyserglythrglyasn
370375380
gluilealaalagluargprometvalglnlyspheileileaspser
385390395400
leulystyrtrpvalasnglutyrhisileaspglypheargpheasp
405410415
leumetalaleuleuglylysaspthrmetserlysalaalaserglu
420425430
leuhisalaileasnproglyilealaleutyrglygluprotrpthr
435440445
glyglythrseralaleuproaspaspglnleuleuthrlysglyala
450455460
glnlysglymetglyvalalavalpheasnaspasnleuargasnala
465470475480
leuaspglyasnvalpheaspserseralaglnglyphealathrgly
485490495
alathrglyleuthraspalailelysasnglyvalgluglyserile
500505510
asnaspphethrserserproglygluthrileasntyrvalthrser
515520525
hisaspasntyrthrleutrpasplysilealaleuserasnproasn
530535540
aspserglualaaspargilelysmetaspgluleualaglnalaval
545550555560
valmetthrserglnglyvalprophemetglnglyglygluglumet
565570575
leuargthrlysglyglyasnaspasnsertyrasnalaglyaspala
580585590
valasnglupheasptrpserarglysalaglntyrproaspvalphe
595600605
asntyrtyrserglyleuilehisleuargleuasphisproalaphe
610615620
argmetthrthralaasngluileasnserhisleuglnpheleuasn
625630635640
serprogluasnthrvalalatyrgluleuthrasphisvalasnlys
645650655
asplystrpglyasnileilevalvaltyrasnproasnlysthrval
660665670
alathrileasnleuproserglylystrpalaileasnalathrser
675680685
glylysvalglygluserthrleuglyglnalagluglyservalgln
690695700
valproglyilesermetmetileleuhisglngluvalserproasp
705710715720
hisglylyslys
<210>6
<211>2172
<212>dna
<213>人工序列
<400>6
gatgctgctaaaccagcagtttctaacgcttaccttgacgcttctaaccaagttttagtt60
aaattatctcaaccattaacattaggtgaaggtgcttctggtttcactgtacatgatgac120
actgctaacaaagacatcccagtaacatctgtaaaagacgcttctttaggtcaagttgaa180
tcaggtgtaaaaactgaccttgttactgttactttaggcgaagatccagatgtatctcac240
actttatctatccaaacagacggttaccaagctaaacaagtaatcccacgtaacgtactt300
aactcttctcaatattactattctggtgatgatttaggaaacacatacacacaaaaagct360
actactttcaaagtttgggctcctacatctactcaagttaacgtattgttatacgattct420
gctacaggtagcgttacaaaaatcgttccaatgacggcttcaggtcacggtgtttgggag480
gctactgttaaccaaaacttagaaaactggtactacatgtacgaagtaactggtcaaggt540
tctacacgcactgctgttgatccttacgctactgctatcgctccaaacggtacacgcggc600
atgatcgtagatttagctaaaactgacccagcaggttggaactctgataaacacattact660
ccaaaaaacattgaagatgaagttatctacgaaatgcacgtacgtgatttctctatcgat720
ccaaactcaggtatgaaaaacaaaggtaaatacttagctctaactgaaaaaggcactaaa780
ggtcctgataacgttaaaacaggtatcgactctcttaagcaattaggtattacacatgtt840
caattaatgccagttttcgcatctaactcagttgacgaaactgatccaacacaatacaac900
tggggttacgacccacgtaactacgatgtaccagaaggtcaatatgcaactaacgctaac960
ggtaacgcacgtattaaagaattcaaagaaatggttttatcactacaccgtgagcacatc1020
ggtgttaacatggacgttgtttacaaccacacgttcgctactcaaatctctgacttcgat1080
aaaattgttccagagtactattaccgcactgacgacgcaggtaactacactaacggttct1140
ggtactggtaacgaaattgctgcagaacgtcctatggtgcaaaaattcatcatcgatagc1200
cttaaatactgggttaacgaataccacattgacggcttccgtttcgacttaatggcttta1260
cttggtaaagacacaatgtctaaggctgcttctgagttacatgctatcaacccaggtatt1320
gctttatatggcgaaccttggactggtggtacaagcgctcttcctgacgaccaactttta1380
actaaaggtgcacaaaaaggcatgggagtagctgtattcaacgataaccttcgtaacgca1440
ttagacggaaacgttttcgattcttctgctcaaggattcgcaacaggagctacaggtctg1500
actgatgctattaaaaacggagttgaaggatcaatcaacgatttcacttcttctcctggc1560
gaaacaattaactacgttacatcacacgataactacactctttgggacaaaatcgctttg1620
tctaaccctaacgactctgaagcagatcgcatcaaaatggatgagcttgctcaagctgtt1680
gttatgacttctcaaggtgtacctttcatgcaaggtggtgaagaaatgttacgcactaaa1740
ggtggtaacgataacagctataacgcgggtgatgctgtaaacgaattcgactggtctcgt1800
aaagctcaataccctgacgttttcaactactactcaggtttaatccaccttcgtcttgac1860
catccagctttccgtatgacaacagctaacgaaatcaactctcaccttcaattccttaac1920
tcacctgaaaacacagtagcttacgaacttactgaccacgtaaacaaagataaatggggt1980
aacattatcgttgtttacaaccctaacaagactgtagcaactatcaacttaccatctggt2040
aaatgggctatcaacgcaactagcggtaaagtaggtgaatctacattaggtcaagctgaa2100
ggatctgtacaagttcctggtatttctatgatgatccttcaccaagaagtttctccagat2160
cacggtaaaaaa2172
<210>7
<211>717
<212>dna
<213>人工序列
<400>7
gtgagcaagggcgaggagctgttcaccggggtggtgcccatcctggtcgagctggacggc60
gacgtaaacggccacaagttcagcgtgagaggcgagggcgagggcgatgccaccaatggc120
aagctgaccctgaagttcatctgcaccaccggcaagctgcccgtgccctggcccaccctc180
gtgaccaccctgacctacggcgtgcagtgcttcagccgctaccccgaccacatgaagcgc240
cacgacttcttcaagtccgccatgcccgaaggctacgtccaggagcgcaccatcagtttc300
aaggacgacggcacatacaagacccgcgccgaggtgaagttcgagggcgacaccctggtg360
aaccgcatcgagctgaagggcatcgacttcaaggaggacggcaacatcctggggcacaag420
ctggagtacaacttcaacagccacaacgtctatatcacggccgacaagcagaagaacggc480
atcaaggccaacttcaagatccgccacaacgtggaggacggcagcgtgcagctcgccgac540
cactaccagcagaacacccccatcggcgacggccccgtgctgctgcccgacaaccactac600
ctgagcacccagtccgtgctgagcaaagaccccaacgagaagcgcgatcacatggtcctg660
ctggagttcgtgaccgccgccgggatcactcacggcatggacgagctgtacaagtaa717
<210>8
<211>81
<212>dna
<213>人工序列
<400>8
aaaagaacgtggaacgtctgtttaacagctctgcttagtgttctgttagtcgctggaagt60
gtcccttttcacgcggaagct81
<210>9
<211>87
<212>dna
<213>人工序列
<400>9
ctaaaaagaacttcattcgtatcttcattattcatcagttcagctgttttactatcaatc60
ttacttccttcgggccaagctcatgca87
<210>10
<211>81
<212>dna
<213>人工序列
<400>10
aagaagagaattacatattcactgcttgctcttctagcagttgttgctttcgctttcact60
gattcatcaaaagcaaaagcg81
<210>11
<211>36
<212>dna
<213>人工序列
<400>11
gtgtacatttcacctcctttaaattactttcattat36
<210>12
<211>64
<212>dna
<213>人工序列
<220>
<221>misc_feature
<222>(12)..(12)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(21)..(21)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(30)..(30)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(33)..(33)
<223>nisa,c,g,ort
<400>12
atgaaragracntggaaygtntgyttracngcnctgcttagtgttctgttagtcgctgga60
agtg64
<210>13
<211>61
<212>dna
<213>人工序列
<220>
<221>misc_feature
<222>(6)..(6)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(15)..(15)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(21)..(21)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(24)..(24)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(27)..(27)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(33)..(33)
<223>nisa,c,g,ort
<400>13
atgcgnaayttracnaaracntcnctnttrctngccggcttatgcacagcggcccaaatg60
g61
<210>14
<211>61
<212>dna
<213>人工序列
<220>
<221>misc_feature
<222>(21)..(21)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(30)..(30)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(33)..(33)
<223>nisa,c,g,ort
<400>14
atgaayathaaraarttygcnaarcargcnacngtattaacctttactaccgcactgctg60
g61
<210>15
<211>44
<212>dna
<213>人工序列
<400>15
cggtaaaaaataatgagattatcaaaaaggatcttcacctagat44
<210>16
<211>40
<212>dna
<213>人工序列
<400>16
gataatctcattattttttaccgtgatctggagaaacttc40
- 还没有人留言评论。精彩留言会获得点赞!