低钾条件下影响水稻苗期根数的QTLqRN5a及其应用的制作方法
本发明涉及分子生物学和植物遗传育种领域,具体地说,涉及低钾条件下影响水稻苗期根数的qtlqrn5a及其应用。
背景技术:
水稻(oryzasatival.)是世界上最重要的粮食作物之一,尤其是在亚洲,是粮食安全的主要支柱。钾(k)是一种必需的矿质元素,是细胞膨胀的主要渗透介质,参与植物生长发育所需的许多生理过程(leighandjones1984;amtmannetal.2005)。钾还参与许多细胞和组织过程,如蛋白质合成、碳水化合物代谢、酶激活、气孔调节和离子稳态(clarksonandhanson1980;amtmannetal.2005)。此外,钾是限制农业生态系统中植物生长和发育的主要营养元素之一(rengelanddamon2008)。在作物中,水稻是对钾需求量最大的作物之一,其对钾的需求量是氮的1.2倍和磷的7倍(dobermannetal.1998)。
目前种植的水稻品种和杂交水稻的肥料利用率较低,营养元素的利用效率至关重要。发展中国家的农民通常使用较少的钾肥,因为其成本高,这会导致土壤中的钾缺乏(hoaetal.2006;andrist-rangeletal.2007)。大部分可耕种土地缺乏钾,包括中国约75%的水稻种植区(rengelanddamon2008)。钾缺乏是限制作物产量的主要因素,因为它被土壤颗粒强烈吸附和固定,也是土壤中的低流动性养分(amtmannetal.2005;rengelanddamon2008)。因此,施用钾肥是农民增产的有效途径。但是,高施肥量和低钾利用效率增加了农民的生产成本和环境污染(laegreidetal.1999)。
作物钾利用效率的遗传改良(即使用耐低钾品种)已成为农业可持续发展的一个良好策略,同时也降低了农民的生产成本,并将生产损失降至最低(pettigrew2008)。植物的钾利用效率受根系吸钾能力的控制。通过优化根系结构,可以提高植物根系的钾吸收能力。不同物种间以及同一物种不同品种(基因型)的钾获取和利用效率不同(glassandperley1980;petterssonandjensen1983)。植物对养分的吸收和利用取决于植物根系结构(
植物耐低钾性是一个复杂的数量性状,基因型与环境关系密切。数量性状基因座(qtl)分析是揭示数量性状相关基因的有效途径(patersonetal.1988)。在大多数情况下,传统的qtl定位群体是双亲群体,如f2、重组自交系(rils)或双单倍体(dh)。在这些群体中,由于背景复杂,可能无法确定效应较小的qtl。相比之下,染色体片段置换系(cssls)或近等基因系(nils)等高级定位群体更适合qtl鉴定(nadeauandfrankel2000)。在这些群体中,每一个品系都带有一个小片段,提供了一个更为均匀的遗传背景,从而增加了找到效应较小qtl的可能性(tanksleyandnelson1996,septiningsihetal.2003)。此外,来自于目标cssl或nil与轮回亲本进一步回交产生的次级f2或f3群体,是进一步精细定位qtl的更好选择(yano2001)。
前人的研究利用dh、bil或ril定位群体发现了几个在低钾条件下和苗期性状相关的qtl(wuetal.1998;miyamotoetal.2012;fangetal.2015;wangetal.2015)。在不同生长条件下的不同根系性状中,很少有qtl被精细定位,同时也没有在缺钾条件下水稻根数qtl的报道。obara等(2010)在水培系统中,在水稻苗期鉴定出根长qtlqrl6.1,并将其缩小到水稻第6染色体上337kb的基因组区域。anis等(2019)在低氮条件下,将控制水稻苗期根干重的主要qtlrdwn6xb被缩小到水稻6号染色体上的52.3kb区域。
迄今为止,共有53个控制水稻根系生长发育的基因被鉴定和克隆(mengetal.2019)。大多数已鉴定的基因在生长素或细胞分裂素信号中起作用,并参与根系发育过程。第一个与根发育相关的基因是arl1/crl1(inukaietal.2005;liuetal.2005),它编码一个lob结构域蛋白。随后,osiaa13/23、osaux1和ospin2等调控水稻根系伸长和不定根形成的基因也陆续被克隆(nietal.2011;kitomietal.2012;yuetal.2015;zhaoetal.2015;inahashietal.2018)。近年来,在水稻中发现了编码与冠根形成有关的sbp结构域蛋白的osspl3(shaoetal.2019)。此外,在水稻中还发现了27种高亲和力k+(hak)转运蛋白(yangetal.2009),在低钾条件下,对钾离子运输、植物生长发育起着重要作用。在不同供钾水平下,水稻根中存在3种钾离子转运体,oshak1/5/21(yangetal.2014;chenetal.2015b;shenetal.2015)。这些发现大大提高了人们对钾利用效率和钾缺乏耐受机制的认识,但具体机制尚未完全阐明。
技术实现要素:
本发明的目的是提供低钾条件下影响水稻苗期根数的qtlqrn5a及其应用。
本发明利用以中恢9308(低钾敏感型)为受体,协青早b(低钾耐受型)为供体构建得到的cssl35(染色体片段置换系,用“协青早b”5号染色体上的7853948~17620241bp替代“中恢9308”5号染色体上的7854125~17620455bp)与zh9308回交所构建的bc5f2:3群体为材料,进行了水稻苗期缺钾条件下根系性状的qtl定位。
为了实现本发明目的,第一方面,本发明提供低钾条件下影响水稻苗期根数的qtlqrn5a,所述qrn5a位于水稻5号染色体indel标记a99和a139之间48.8-kb的区间内。其中,indel标记a99和a139见表1。
以上物理位置所基于的水稻基因组版本号为riceannotationprojectdatabase(rap-db,https://rapdb.dna.affrc.go.jp/)和ricegenomeannotationprojectdatabase(rgap,http://rice.plantbiology.msu.edu/)。
上述qtlqrn5a位点包含3个可能的候选基因os05g0346700、loc_os05g27980和loc_os05g28000,所述的3个候选基因在中恢9308(zh9308)和协青早b(xqzb)中的cds序列分别如seqidno:1-6所示,启动子序列分别如seqidno:7-12所示。其中,os05g0346700在中恢9308和协青早b中的cds序列分别如seqidno:1-2所示,loc_os05g27980在中恢9308和协青早b中的cds序列分别如seqidno:3-4所示,loc_os05g28000在中恢9308和协青早b中的cds序列分别如seqidno:5-6所示;os05g0346700的启动子序列在中恢9308和协青早b中的cds序列分别如seqidno:7-8所示,loc_os05g27980的启动子序列在中恢9308和协青早b中的cds序列分别如seqidno:9-10所示,loc_os05g28000的启动子序列在中恢9308和协青早b中的cds序列分别如seqidno:11-12所示。
在之前研究中,从一个cssl群体中鉴定了几个与苗期性状相关的qtl,这些qtl来自于供体亲本协青早b(xqzb)和轮回亲本中恢9308(zh9308)。在已鉴定的qtl中,在bc5f2群体中验证了qrn5a在5号染色体长臂上的作用。为进一步阐明qrn5a(在lk条件下增加rn)的分子基础,我们通过将携带该基因座的cssl35与zh9308杂交,构建了一个较大的次级f2:3(bc5f2:3)群体,并最终用纯合交换单株将qrn5a基因定位到indel标记a99和a139之间48.8kb的区段上,并对该区段中7个假定候选基因进行了分析,其中3个基因(os05g0346700、loc_os05g27980和loc_os05g28000)在双亲之间的编码区域中具有非同义的单核苷酸多态性。分析结果表明,loc_os05g27980编码一个lateralorganboundaries域包含蛋白,是根数低钾耐受性的正调控因子,是qrn5a最可能的候选基因。此外,通过qrt-pcr检测,在低钾条件下,qrn5a促进根组织中osiaa23基因的表达,并抑制根组织中oshak5基因的表达,以促进cssl35的根数。
第二方面,本发明提供与所述qrn5a紧密连锁的分子标记,包括15个snp标记snp1~snp15和3个indel标记indel1~indel3。
其中,snp1位于水稻基因os05g0346700cds序列第142bp,多态性为a/g;snp2位于水稻基因loc_os05g27980cds序列第442bp,多态性为g/t;snp3位于水稻基因loc_os05g28000cds序列第347bp,多态性为a/g;snp4位于水稻基因loc_os05g28000cds序列第409bp,多态性为t/c;
snp5~snp13分别位于水稻基因os05g0346700atg起始密码子上游第-115、-186、-194、-405、-436、-589、-599、-714和-770bp,多态性分别为c/t、a/g、a/g、t/c、g/a、t/c、g/a、g/a和c/t;snp14位于水稻基因loc_os05g27980atg起始密码子上游第-1054bp,多态性为t/a;snp15位于水稻基因loc_os05g28000atg起始密码子上游第-586bp,多态性为a/g;
indel1位于水稻基因os05g0346700atg起始密码子上游第-515~-500bp,多态性为tatggcaggattgtcc/-;
indel2位于水稻基因loc_os05g27980atg起始密码子上游第-4~-5bp,多态性为--/ag;
indel3位于水稻基因loc_os05g27980atg起始密码子上游第-1109bp,多态性为-/a。
第三方面,本发明提供用于扩增上述分子标记的引物,扩增各分子标记的引物如下:
扩增snp1的正向引物和反向引物序列分别为seqidno:13和14;
扩增snp2的正向引物和反向引物序列分别为seqidno:15和16;
扩增snp3和snp4的正向引物和反向引物序列分别为seqidno:17和18;
扩增snp5~snp13和indel1的正向引物和反向引物序列分别为seqidno:19和20、seqidno:21和22以及seqidno:23和24;
扩增snp14、indel2和indel3的正向引物和反向引物序列分别为seqidno:25和26、seqidno:27和28以及seqidno:29和30;
扩增snp15的正向引物和反向引物序列分别为seqidno:31和32、seqidno:33和34以及seqidno:35和36。
第四方面,本发明提供上述分子标记或其检测试剂的以下任一应用:
(1)用于低钾条件下水稻苗期根系性状的鉴定;
(2)用于耐低钾胁迫水稻材料的早期预测;
(3)用于鉴定低钾条件下影响水稻苗期根数的qtlqrn5a位点;
(4)用于水稻基因os05g0346700、loc_os05g27980和loc_os05g28000的基因分型;
(5)用于耐低钾水稻种质资源鉴定或分子标记辅助育种。
第五方面,本发明提供源自籼稻品种‘协青早b’的qtl位点qrn5a的以下任一应用:
(1)调控植物根系生长发育;
(2)调控植物苗期低钾条件下根系性状;
(3)植物品种改良;
(4)制备转基因植物;
(5)促进水稻根组织中osiaa23基因的表达;
(6)抑制水稻根组织中oshak5基因的表达。
所述调控为正调控。
所述根系性状为根数。
优选地,所述植物为禾本科植物,更优选水稻。
第六方面,本发明提供源自籼稻品种‘协青早b’的水稻基因os05g0346700、loc_os05g27980和loc_os05g28000:
水稻基因os05g0346700的cds序列为:
i)seqidno:2所示的核苷酸序列;
ii)seqidno:2所示的核苷酸序列经取代、缺失和/或增加一个或多个核苷酸且表达相同功能蛋白质的核苷酸序列;
iii)在严格条件下与seqidno:2所示序列杂交且表达相同功能蛋白质的核苷酸序列,所述严格条件为在含0.1%sds的0.1×sspe或含0.1%sds的0.1×ssc溶液中,在65℃下杂交,并用该溶液洗膜;或
iv)与i)、ii)或iii)的核苷酸序列具有90%以上同源性且表达相同功能蛋白质的核苷酸序列。
水稻基因os05g0346700的启动子序列如seqidno:8所示。
水稻基因loc_os05g27980的cds序列为:
a)seqidno:4所示的核苷酸序列;
b)seqidno:4所示的核苷酸序列经取代、缺失和/或增加一个或多个核苷酸且表达相同功能蛋白质的核苷酸序列;
c)在严格条件下与seqidno:4所示序列杂交且表达相同功能蛋白质的核苷酸序列,所述严格条件为在含0.1%sds的0.1×sspe或含0.1%sds的0.1×ssc溶液中,在65℃下杂交,并用该溶液洗膜;或
d)与a)、b)或c)的核苷酸序列具有90%以上同源性且表达相同功能蛋白质的核苷酸序列。
水稻基因loc_os05g27980的启动子序列如seqidno:10所示。
水稻基因loc_os05g28000的cds序列为:
a)seqidno:6所示的核苷酸序列;
b)seqidno:6所示的核苷酸序列经取代、缺失和/或增加一个或多个核苷酸且表达相同功能蛋白质的核苷酸序列;
c)在严格条件下与seqidno:6所示序列杂交且表达相同功能蛋白质的核苷酸序列,所述严格条件为在含0.1%sds的0.1×sspe或含0.1%sds的0.1×ssc溶液中,在65℃下杂交,并用该溶液洗膜;或
d)与a)、b)或c)的核苷酸序列具有90%以上同源性且表达相同功能蛋白质的核苷酸序列。
水稻基因loc_os05g28000的启动子序列如seqidno:12所示。
第七方面,本发明提供含有所述水稻基因os05g0346700、loc_os05g27980和/或loc_os05g28000的生物材料,所述生物材料包括但不限于重组dna、表达盒、转座子、质粒载体、噬菌体载体、病毒载体、工程菌或非可再生的植物部分。
第八方面,本发明提供水稻基因os05g0346700、loc_os05g27980和/或loc_os05g28000,或者含有所述基因的生物材料的以下任一应用:
(1)调控植物根系生长发育;
(2)调控植物苗期低钾条件下根系性状;
(3)植物品种改良;
(4)制备转基因植物;
(5)促进水稻根组织中osiaa23基因的表达;
(6)抑制水稻根组织中oshak5基因的表达。
所述调控为正调控。
所述根系性状为根数。
优选地,所述植物为禾本科植物,更优选水稻。
第九方面,本发明提供一种促进水稻苗期低钾条件下根系生长发育的方法,包括:
1)使水稻包含基因os05g0346700、loc_os05g27980和/或loc_os05g28000;或
2)使水稻过表达基因os05g0346700、loc_os05g27980和/或loc_os05g28000。
其中,基因os05g0346700、loc_os05g27980和loc_os05g28000的cds序列分别如seqidno:2、4、6所示,它们的启动子序列分别如seqidno:8、10、12所示。
前述方法包括但不限于转基因、杂交、回交、自交或无性繁殖。
本发明中,所述低钾条件是指培养环境中的供钾(k+)水平为5mg/l。
借由上述技术方案,本发明至少具有下列优点及有益效果:
本发明首次发现了低钾条件下影响水稻苗期根数的qtlqrn5a,本发明提供的源自籼稻品种‘协青早b’的qtl位点qrn5a对培育耐低钾水稻品种的分子标记辅助选择具有重要意义,为最终克隆水稻根系耐低钾胁迫基因座,改善缺钾条件下的水稻根系结构奠定了坚实的基础。
附图说明
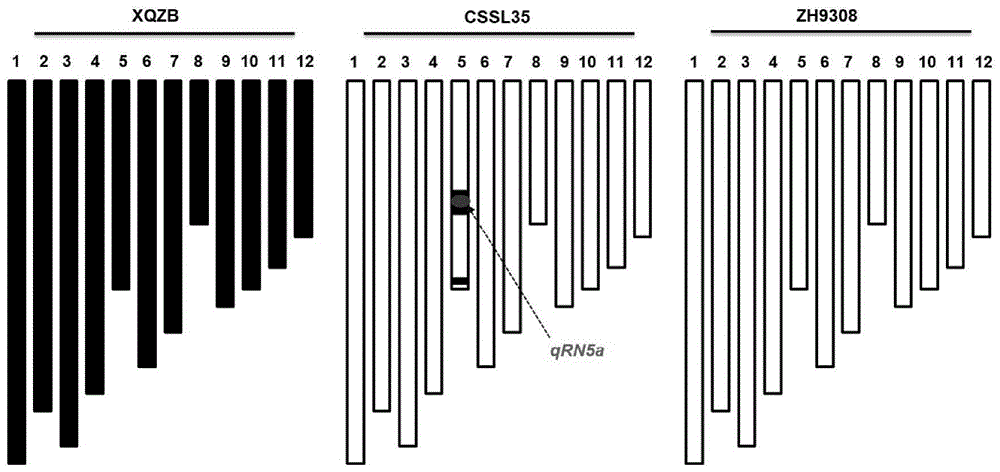
图1为本发明较佳实施例中xqzb、cssl35和zh9308的遗传背景。在cssl35染色体图谱中,黑色区域表示zh9308背景中的xqzb等位基因,红色区域表示主要qrn5a位点。
图2为本发明较佳实施例中水稻导入系cssl35和轮回亲本zh9308的根系表型。其中,a为两个亲本在lk和nk下的根系表型。b-d为两个亲本在lk和nk下的其他根部表型,包括根数(b)、根长(c)和根干重(d)。用12株植物测定生长参数,数据用平均值±标准差表示。*,**和***分别表示5%,1%和0.1%水平上的显著性。
图3为本发明较佳实施例中qrn5a在5号染色体上的精细定位。其中,a为基于75个cssls的qrn5a遗传连锁图谱。b为验证bc5f2群体中的qrn5a位点。c为使用2962个单株精细定位qrn5a区间的高分辨率连锁图。d为用于精细定位的11个纯合重组家系和两亲本(zh9308和cssl35)的基因型和表型。白条和黑条分别表示zh9308和cssl35的标记基因型;rn、rl和rdw表示为平均值±标准差。肩标字母(a,b,c)表明重组体与双亲相比在性状上存在显著差异。e为7个预测靶区基因。f为候选基因的结构和突变位点;黑匣子代表外显子。
图4为本发明较佳实施例中三个候选基因在35日龄苗lk条件下的zh9308和cssl35的地上部(a)和根部(b)组织中的表达模式。数据表示为平均值±标准差,并根据3个技术重复和3个生物重复进行计算。**和***分别在1%和0.1%水平上表示显著性。
图5为本发明较佳实施例中目的候选基因loc_os05g27980在35天苗期的两个亲本在nk和lk条件下的表达(a),以及纯合重组系在lk条件下的表达(b)。数据以平均值±标准差表示,并根据3个技术重复和2个生物重复进行计算。**和***分别表示1%和0.1%的显著性。
图6为本发明较佳实施例中lk处理后14日龄幼苗根组织两个亲本中loc_os05g27980(a)、osiaa23(b)、osaux1(c)、osspl3(d)、oscrl1(e)、osiaa13(f)、ospin2(g)、osdro1(h)和k转运体基因osakt1(i)、oshak1(j)、oshak5(k)和oshak21(1)的节律表达。数据(以平均值±标准差表示)由3个qrt-pcr技术重复和3个生物学重复计算得出。
具体实施方式
本发明中涉及的术语:
lk:低钾(lowpotassium),k+浓度为5mg/l。
nk:正常钾(normalpotassium),k+浓度为87mg/l。
rn:根数(rootnumber)。
rl:根长(rootlength)。
rdw:根干重(rootdryweight)。
以下实施例用于说明本发明,但不用来限制本发明的范围。若未特别指明,实施例均按照常规实验条件,如sambrook等分子克隆实验手册(sambrookj&russelldw,molecularcloning:alaboratorymanual,2001),或按照制造厂商说明书建议的条件。
实施例1qrn5a的表型
在之前研究中,利用具有zh9308遗传背景且携带来自xqzb的第5染色体片段的染色体片段置换系cssl35为起始材料,利用cssl35和受体亲本zh9308进行回交,构建了次级f2(bc5f2)群体,将控制水稻根数的qtlqrn5a定位于第5染色体长臂上标记ind78和rm18472之间。进一步发现,cssl35是以zh9308为遗传背景,在qrn5a区域及第5染色体长臂末端插入xqzb染色体片段,并且在插入片段中不存在其他根数qtl(图1)。两亲本的表型结果表明,zh9308和cssl35的表型在根数(rn)、根长(rl)和根干重(rdw)方面存在显著差异,并且cssl35在nk和lk下的根数高于zh9308(图2,a和b),根长和根干重也高于zh9308(图2,c和d)。以上结果表明,qrn5a是控制水稻苗期lk条件下根数的qtl,是一个稳定的位点,可以精细定位和克隆。
实施例2qrn5a的精细定位
qrn5a位点先前被定位在5号染色体长臂上标记ind78到rm18472之间的1023kb基因组区域(图3,a和b)。为进一步缩小qrn5a的位置,我们构建了一个较大的f2:3(bc5f2:3)群体,其中包含从cssl35和zh9308杂交获得的2962个植株个体。然后利用新开发的具有ind78到rm18472的双亲之间多态性的indel标记筛选群体(图3c),并使用纯合重组植株精细定位qrn5a。
利用13个标记对bc5f2:3群体的2962个单株进行基因分型,从bc5f2:3群体中发现118个交换单株,根据基因型将纯合交换单株分为11组(图3d)。利用纯合交换单株,将每个重组家系的平均表型值与zh9308或cssl35进行比较,以定位qrn5a。l8(其中,l为重组家系,8为重组家系的编号)的根数显著高于zh9308,而l3的基因型与l8相反,l3的平均根数与cssl35显著不同。因此,qrn5a被限定在标记a26的上游侧(图3d)。同样,l5和l10将qrn5a划定到标记a145的下游区域。在另一个实例中,与含有qrn5azh9308等位基因的l4和l9相比,含有qrn5axqzb等位基因的l8表现出更高的根数。相反,l4和l9与cssl35差异显著,与zh9308相似。通过定位其他重组家系,qrn5a最终缩小到第5染色体上标记a99和a139之间的48.8kb基因组区域(图3d)。在标记a99和a139之间含有qrn5axqzb等位基因的纯合重组体的根数均显著高于zh9308。精细定位所用引物见表1。
表1用于扩增精细定位qrn5a的标记的引物序列
实施例3qrn5a的候选基因分析
利用日本晴基因组序列做参考,将qrn5a定位在5号染色体长臂上标记a99和a139之间48.8kb的物理位置(www.gramene.org/)(图3c)。根据水稻注释项目数据库(rap-db,https://rapdb.dna.affrc.go.jp/)以及水稻基因组注释项目数据库(rgap,http://rice.plantbiology.msu.edu/),qrn5a定位包括7个预测基因,分别是os05g0346700、loc_os05g27980、loc_os05g27990、loc_os05g28000、loc_os05g28010、loc_os05g28020和loc_os05g28040(图3e,表2)。根据预测结果,这7个基因中没有一个与增加根数相关。因此,qrn5a可能是一个lk条件下增加根数的新qtl。
表2qrn5a候选基因在双亲间的突变
然后对候选基因进行了测序,由于loc_os05g27990编码转座子蛋白,未对其进行测序。对其余6个基因在两亲本间的序列比较显示,os05g0346700的编码序列(cds)中有一个非同义snp,loc_os05g27980中有一个非同义snp,loc_os05g2800中有两个非同义snp(表3,图3f)。在另外三个基因(loc_os05g28010、loc_os05g28020和loc_os05g28040)中,两亲本间没有差异。我们还对在双亲之间有snps的三个基因的启动子区进行了测序,os05g0346700在双亲之间有9个snp和1个indel,loc_os05g27980有1个snp和2个indel,loc_os05g28000有1个snp。这三个基因的编码区和启动子区在两亲本间的序列分别如seqidno:1-12所示。用于对qrn5a候选基因cds区测序的引物如表4所示,用于对qrn5a在双亲间有差异基因的启动子区测序的引物表5所示。
表3qrn5a候选基因在双亲间有差异基因的核苷酸及氨基酸差异
表4用于对qrn5a候选基因cds区测序的引物
表5用于对qrn5a在双亲间有差异基因的启动子区测序的引物
实施例4qrn5a候选基因的表达模式分析
为进一步确定qrn5a的候选基因,首先搜索ricexpro数据库,研究三个具有非同义snps的候选基因(os05g0346700、loc_os05g27980和loc_os05g28000)的表达模式。结果发现只有loc_os05g27980在水稻根中表达,另外两个位点在水稻根组织中没有表达(http://ricexpro.dna.affrc.go.jp/rxp_0007/gene-search.php)。因此,loc_os05g27980可能在根组织中表达,并在lk条件下诱导根的生长。
然后对这三个基因进行qrt-pcr检测。我们分析了在lk条件下生长的35天幼苗的两个亲本的叶片和根中的基因表达模式。在叶片中,我们发现os05g0346700和loc_os05g27980在zh9308中的表达都比cssl35高一些,loc_os05g28000的转录在双亲之间没有显著差异(图4a)。在根中,两个亲本的os05g0346700和loc_os05g28000的转录水平相似(图4b)。然而,与zh9308相比,loc_os05g27980在cssl35中的转录显著提高(图4b),这一结果在根中24小时节律表达分析中得到进一步证实(图5a)。综上,loc_os05g27980在两个亲本的根之间的表达差异最大。
我们还观察了目标候选基因(loc_os05g27980)在nk和lk下生长35天幼苗的两个亲本的根组织中的表达模式(图5a),以及在lk条件下生长35天幼苗的根组织中的纯合重组系(l1~l11)的表达模式(图5b)。结果发现,在nk和lk条件下,在cssl35中loc_os05g27980的表达高于zh9308(图5a),并且携带xqzb等位基因(l5~l8)的交换单株中的qrn5a表达水平显著高于携带zh9308等位基因的交换单株(图5b)。
实施例5qrn5a与其它根系生长调控基因及k转运蛋白的关系
loc_os05g27980在促进缺钾条件下根系形成中的作用及其在根组织中的表达差异表明,它是qrn5a最可能的候选基因,qrn5a参与了水稻根系的生长调节。为了研究qrn5a对根生长调节基因和k转运蛋白的影响,我们采用qrt-pcr技术比较了7个与根系结构相关的基因(oscrl1、osdro1、ospin2、osaux1、osspl3、osiaa13和osiaa23)和4个k转运蛋白基因(oshak1、oshak5、oshak21和osakt1)在lk条件下14天幼苗的双亲根组织中的表达水平。
osiaa23在cssl35的根组织中的表达水平在各阶段都比zh9308高,并且表现出与loc_os05g27980相似的表达模式(图6,a和b)。根据此前研究,qrn5a的xqzb等位基因对zh9308等位基因表现为显性,以上结果表明,qrn5a促进osiaa23在根组织中的表达,并且osiaa23基因的上调可能对cssl35的根系形成起重要作用。ni等人(2011)观察到osiaa23直接作用于根系发育。相反,在lk条件下,与zh9308相比,oshak5在cssl35根部的表达下调,这表明oshak5的表达被qrn5a抑制(图6k)。此外,其他根生长调节基因(osaux1、osspl3、oscrl1、osiaa13、ospin2和osdro1)和k转运蛋白基因(osakt1、oshak1和oshak21)在两个亲本之间的表达水平没有显著差异(图6,c-j和1),表明qrn5a不影响它们的转录。综上所述,基于基因表达的结果,我们可以推测qrn5a是根数的正调节因子,上调osiaa23的表达,下调oshak5的表达,促进lk条件下cssl35的根系形成。
虽然,上文中已经用一般性说明及具体实施方案对本发明作了详尽的描述,但在本发明基础上,可以对之做一些修改或改进,这对本领域技术人员而言是显而易见的。因此,在不偏离本发明精神的基础上所做的这些修改或改进,均属于本发明要求保护的范围。
参考文献:
[1]amtmanna,hammondjp,armengaudp,whitepj(2005)nutrientsensingandsignallinginplants:potassiumandphosphorus.advbotres43:209-257.
[2]andrist-rangely,edwardsa,hilliers,
[3]anisgb,zhangy,islama,zhangy,caoy,wuw,caol,chengs(2019)rdwn6xb,amajorquantitativetraitlocuspositivelyenhancesrootsystemarchitectureundernitrogendeficiencyinrice.bmcplantbiol19(1):12.
[4]cheng,huq,luol,yangt,zhangs,huy,yul,xug(2015b)ricepotassiumtransporteroshak1isessentialformaintainingpotassium-mediatedgrowthandfunctionsinsalttoleranceoverlowandhighpotassiumconcentrationranges.plantcellenviron38:2747-2765.
[5]clarksondt,hansonjb(1980)themineralnutritionofhigherplants.annurevplantphysiol31(1):239-298.
[6]dobermanna,cassmank,mamarilc,sheehyj(1998)managementofphosphorus,potassium,andsulfurinintensive,irrigatedlowlandrice.fieldcropres56(1-2):113-138.
[7]dorlodots,forsterb,pagèsl,pricea,tuberosar,drayex(2007)rootsystemarchitecture:opportunitiesandconstraintsforgeneticimprovementofcrops.trendsplantsci12(10):474-481.
[8]fangy,wuw,zhangx,jiangh,luw,panj,huj,guol,zengd,xued(2015)identificationofquantitativetraitlociassociatedwithtolerancetolowpotassiumandrelatedionsconcentrationsatseedlingstageinrice(oryzasatival.)plantgrowthregul77(2):157-166.
[9]glassad,perleyje(1980)varietaldifferencesinpotassiumuptakebybarley.plantphysiol65(1):160-164.
[10]hoanm,janssenbh,oenemao,dobermanna(2006)comparisonofpartialandcompletesoilkbudgetsunderintensivericecroppinginthemekongdelta,vietnam.agrecosystenviron116(1-2):l21-131.
[11]
[12]inahashih,shelleyij,yamauchit,nishiuchis,nosakamt,matsunamim,ogawaa,noday,inukaiy(2018)ospin2,whichencodesamemberoftheauxineffluxcarrierproteins,isinvolvedinrootelongationgrowthandlateralrootformationpatternsviatheregulationofauxindistributioninrice.physiolplant164(2):216-225.
[13]inukaiy,sakamotot,ueguchi-tanakam,shibatay,gomik,umemurai,hasegaway,ashikarim,kitanoh,matsuokam(2005)crownrootlessl,whichisessentialforcrownrootformationinrice,isatargetofanauxinresponsefactorinauxinsignaling.plantcell17:1387-1396.
[14]kellermeierf,chardonf,amtmanna(2013)naturalvariationofarabidopsisrootarchitecturerevealscomplementingadaptivestrategiestopotassiumstarvation.plantphysiol161(3):1421-1432.
[15]kitomiy,inahashih,takehisah,satoy,inukaiy(2012)osiaa13-mediatedauxinsignalingisinvolvedinlateralrootinitiationinrice.plantsci190:116-122.
[16]
[17]leighra,jonesrw(1984)ahypothesisrelatingcriticalpotassiumconcentrationsforgrowthtothedistributionandfunctionsofthisionintheplantcell.newphytol97(1):1-13.
[18]liuhj,wangsf,yux,yuj,hexw,zhangsl,shouhx,wup(2005)arl1,alob-domainproteinrequiredforadventitiousrootformationinrice.plantj.43:47-56.
[19]mengf,xiangd,zhuj,liy,maoc(2019)molecularmechanismsofrootdevelopmentinrice.rice,12:1.
[20]miyamotot,ochiaik,takeshitas,matoht(2012)identificationofquantitativetraitlociassociatedwithshootsodiumaccumulationunderlowpotassiumconditionsinriceplants.soilsciplantnutr58(6):728-736.
[21]nadeaujh,frankelwn(2000)theroadsfromphenotypicvariationtogenediscovery:mutagenesisversusqtls.natgenet25(4):381.
[22]nij,wangg,zhuz,zhangh,wuy,wup(2011)osiaa23-mediatedauxinsignalingdefinespostembryonicmaintenanceofqcinrice.plantj.68,433-442.
[23]obaram,tamuraw,ebitanit,yanom,satot,yamayat(2010)fine-mappingofqrl6.1,amajorqtlforrootlengthofriceseedlingsgrownunderawiderangeofnh4+concentrationsinhydroponicconditions.theorapplgenet121(3):535-547.
[24]patersonah,landeres,hewittjd,petersons,lincolnse,tanksleysd(1988)resolutionofquantitativetraitsintomendelianfactorsbyusingacompletelinkagemapofrestrictionfragmentlengthpolymorphisms.nature335(6192):721.
[25]petterssons,jensenp(1983)variationamongspeciesandvarietiesinuptakeandutilizationofpotassium.plantsoil72(2-3):231-237.
[26]pettigrewwt(2008)potassiuminfluencesonyieldandqualityproductionformaize,wheat,soybeanandcotton.physiolplant133(4):670-681.
[27]rengelz,damonpm(2008)cropsandgenotypesdifferinefficiencyofpotassiumuptakeanduse.physiolplant133(4):624-636.
[28]septiningsihe,trijatmikok,moeljopawiros,mccouchs(2003)identificationofquantitativetraitlociforgrainqualityinanadvancedbackcrosspopulationderivedfromtheoryzasativavarietyir64andthewildrelativeo.rufipogon.theorapplgenet107(8):1433-1441.
[29]shaoy,zhouhz,wuy,zhangh,linj,jiangx,heq,zhuj,liy,yuh,maoc(2019)osspl3,ansbp-domainprotein,regulatescrownrootdevelopmentinrice.plantcell31(6),1257-1275.
[30]sheny,shenl,shenz,jingw,geh,zhaoj,zhangw(2015)thepotassiumtransporteroshak21functionsinthemaintenanceofionhomeostasisandtolerancetosaltstressinrice.plantcellenviron38:2766-2779.
[31]shinr(2014)strategiesforimprovingpotassiumuseefficiencyinplants.molcells37(8):575-584.
[32]tanksleys,nelsonj(1996)advancedbackcrossqtlanalysis:amethodforthesimultaneousdiscoveryandtransferofvaluableqtlsfromunadaptedgermplasmintoelitebreedinglines.theorapplgenet92(2):191-203.
[33]wangg,luw,chenh,zhangx,xued(2015)seedlingscreeningofricegermplasmresourceswithlowpotassiumtolerance.j.hangzhounorm.univ1:44_48.
[34]wup,nij,luoa(1998)qtlsunderlyingricetolerancetolow-potassiumstressinriceseedlings.cropsci38(6):1458-1462.
[35]yangzf,gaoqs,suncs,liwj,gusl,xucw(2009)molecularevolutionandfunctionaldivergenceofhakpotassiumtransportergenefamilyinrice(oryzasatival.).jgenetgenomics36(3):161-72.
[36]yangt,zhangs,huy,wuf,huq,cheng,caij,wut,morann,yul,xug(2014)theroleofapotassiumtransporteroshak5inpotassiumacquisitionandtransportfromrootstoshootsinriceatlowpotassiumsupplylevels.plantphysiol166:945-959.
[37]yuc,su,c,shenc,wangs,liuf,liuy,chenyl,lic,qianq,aryalb,geislerm,jiangd,qiyh(2015).theauxintransporter,osaux1,isinvolvedinprimaryrootandroothairelongationandincdstressresponsesinrice(oryzasatival.).plantj.83(5),818-830.
[38]zhaoh,mat,wangx,dengy,mah,zhangr,zhaoj(2015)osaux1controlslateralrootinitiationinrice(oryzasatival.).p1antcellenviron38(1):2208-2222.
序列表
<110>中国水稻研究所
<120>低钾条件下影响水稻苗期根数的qtlqrn5a及其应用
<130>pi202010654
<160>36
<170>siposequencelisting1.0
<210>1
<211>288
<212>dna
<213>水稻(oryzasativa)
<400>1
atggtgttcggccccgccgccgtgcagaggtacggccccgacatgacgctgcgggaggca60
ctggacgggaggggagacatctaccggacgctgctgtgggaggccacggcggtgctcctc120
aacgcctactacaacactcccagcgtcgcagccgccgccgccaccgcgctcgcccgccga180
cgccgcctcgccccgccgcagcgcgggagcaggagggcgtcagcggcaggatggcggctt240
atagggacattgacgcggtcgtggctgctggggaagaagaacatgtag288
<210>2
<211>288
<212>dna
<213>水稻(oryzasativa)
<400>2
atggtgttcggccccgccgccgtgcagaggtacggccccgacatgacgctgcgggaggca60
ctggacgggaggggagacatctaccggacgctgctgtgggaggccacggcggtgctcctc120
aacgcctactacaacactcccggcgtcgcagccgccgccgccaccgcgctcgcccgccga180
cgccgcctcgccccgccgcagcgcgggagcaggagggcgtcagcggcaggatggcggctt240
atagggacattgacgcggtcgtggctgctggggaagaagaacatgtag288
<210>3
<211>603
<212>dna
<213>水稻(oryzasativa)
<400>3
atgggcggtgggtcgccgtgcgcgtcgtgcaagctgctgcggcggcggtgcaccaaggac60
tgcatcttcgcgcccttcttccccgccgacgacccccacaagttcgccatcgtccacaag120
gtcttcggcgccagcaatgtcagcaagatgctccaggagctgccggcgcagcagcgaggc180
gacgcggtgagcagcctggtgtacgaggcgaacgcccggatgcgggaccccgtctacggc240
tgcgtcggggccatctccttcctccagaaccaggtgtcgcagctgcagatgcagctcgcc300
gtcgcgcaggccgagatcctctgcatccagatgcagcaccgcgacgacggcgccgcccac360
cagctggacgccgccgccgaccaccacccgctgctggatcagcagcagcaacagcagatg420
gtcgtggacgccgccgacgcggccgcctccttcctcgtgcagaacggtggcggaggcgga480
ggccccgcggcgcagctgataagcggctacggctcgccggcggccggcggcggcggtggc540
catggcgtggtgcactacgcggcggcgcaggagcatctcaagagggagtcgctctggacg600
tag603
<210>4
<211>603
<212>dna
<213>水稻(oryzasativa)
<400>4
atgggcggtgggtcgccgtgcgcgtcgtgcaagctgctgcggcggcggtgcaccaaggac60
tgcatcttcgcgcccttcttccccgccgacgacccccacaagttcgccatcgtccacaag120
gtcttcggcgccagcaatgtcagcaagatgctccaggagctgccggcgcagcagcgaggc180
gacgcggtgagcagcctggtgtacgaggcgaacgcccggatgcgggaccccgtctacggc240
tgcgtcggggccatctccttcctccagaaccaggtgtcgcagctgcagatgcagctcgcc300
gtcgcgcaggccgagatcctctgcatccagatgcagcaccgcgacgacggcgccgcccac360
cagctggacgccgccgccgaccaccacccgctgctggatcagcagcagcaacagcagatg420
gtcgtggacgccgccgacgcgtccgcctccttcctcgtgcagaacggtggcggaggcgga480
ggccccgcggcgcagctgataagcggctacggctcgccggcggccggcggcggcggtggc540
catggcgtggtgcactacgcggcggcgcaggagcatctcaagagggagtcgctctggacg600
tag603
<210>5
<211>474
<212>dna
<213>水稻(oryzasativa)
<400>5
atggaaggaggcgacaagggggtaggggaggaggtcgtcgccattgaggaggaggttggt60
gcccatgagcacctatggtcccttgtgcttgctagcagctccacctccgccttgatgtcg120
gtgtggcggatgccgagcacaagaaggttggggaagatgtcagcgcgcggggcgaaggtg180
gtcgatgttgaggaacaacttctagtgaccaaggtagaggcggtggaggaacatgacact240
gaggacatagatccagcttggctagaatgggaaccaccaccaaaactcataggtggtgcg300
gctagtgacaaagaagtggctgtagctgcccatggcttgccactccagtcaccggtggtg360
ggagagggtcaccggggcgagggagagaaggaaattgtcgatagcgcgtgcgaagaagaa420
gataaggttggggaagacaagataaatggacagggtattttggtccatatataa474
<210>6
<211>474
<212>dna
<213>水稻(oryzasativa)
<400>6
atggaaggaggcgacaagggggtaggggaggaggtcgtcgccattgaggaggaggttggt60
gcccatgagcacctatggtcccttgtgcttgctagcagctccacctccgccttgatgtcg120
gtgtggcggatgccgagcacaagaaggttggggaagatgtcagcgcgcggggcgaaggtg180
gtcgatgttgaggaacaacttctagtgaccaaggtagaggcggtggaggaacatgacact240
gaggacatagatccagcttggctagaatgggaaccaccaccaaaactcataggtggtgcg300
gctagtgacaaagaagtggctgtagctgcccatggcttgccactccggtcaccggtggtg360
ggagagggtcaccggggcgagggagagaaggaaattgtcgatagcgcgcgcgaagaagaa420
gataaggttggggaagacaagataaatggacagggtattttggtccatatataa474
<210>7
<211>934
<212>dna
<213>水稻(oryzasativa)
<400>7
cagcgtttgacaggacttctagaaattataaaaacgaaacacaatgagacaataaattct60
aaaaactacaagatctaattttaaatggttacaaggagaatgaatagaattagtggtaga120
tcgagcaagcaaataaataatgatatgacagaagtaaggggtagcagctggtgtgacttt180
taaaaactatataattagaaatacggggatgataaggtttgatctttcaaagtcttaaga240
caacgagatagctatttaataaattttagagtaattttaagtaaaatcatactgaaaaat300
atatgattttgtttgggggctagccgcgcaattgcgcggggcacctagcaagtttgttag360
aagtagaaaataaactattcaatggttttttatacttgtggactatggcaggattgtcct420
atggcaggattgtccgtttgcatttggaaaaagtacactgaaggtccctcaacttgtcat480
cgagttacaaaatcatccgcaaaccgcaaaaccggatacaacgcatccctcaacttataa540
aaccagtgcaaatccttcagcggttttgaccctggttttggccgacgtggcagctaagtc600
agcgtgggacccacgcgggccccacatgttaggatggcacgtcagcctctcccttcctct660
cctttcctccccacgtgactgtgatcgcggcgccgctgaggtgaaggcggcggtcgatgc720
gcgtggcggcgccgacgcccacgccaccacggccagtgctgggcgggttcgagaaggagc780
aggaggacgtcgcgctcgccccctcctcctctccgactccaatcatcgcgccgcccccct840
gtcctctccaactccccctttcccgcccctctggcgccgcaggcctcgccgcctgccgcc900
catctggcgccgctcaccctcgccgcctgtggcc934
<210>8
<211>918
<212>dna
<213>水稻(oryzasativa)
<400>8
cagcgtttgacaggacttctagaaattataaaaacgaaacacaatgagacaataaattct60
aaaaactacaagatctaattttaaatggttacaaggagaatgaatagaattagtggtaga120
tcgagcaagcaaataaataatgatatgacagaagtaaggggtagtagctggtgtgacttt180
taaaaactatataattagaaatacggggatgataaggtttaatctttcaaagtcttaaga240
caacgagatagctatttaataaattttagagtaattttaagtaaaatcatactgaaaaat300
atatgattttgtttgggggctagccgcgcaattgcacggggcacccagcaagtttgttag360
aagtagaaaataaactattcaatggttttttatacttgtggactatggcaggattgtccg420
tttgcatttggaaaaagtacactgaaggtccctcaacttgtcatcgagttacaaaatcat480
ccacaaaccgcaaaaccggatacaacgcatcccccaacttataaaaccagtgcaaatcct540
tcagcggttttgaccctggttttggccgacgtggcagctaagtcagcgtgggacccacgc600
gggccccacatgttaggatggcacgtcagcctctcccttcctctcctttcctccccacgt660
gactgtgatcgcggcgccgctgaggtgaaggcggcggtcgatgcgcgtggcggcgccgac720
gcccgcgccaccgcggccagtgctgggcgggttcgagaaggagcaggaggacgtcgcgct780
cgccccctcctcctctccgactctaatcatcgcgccgcccccctgtcctctccaactccc840
cctttcccgcccctctggcgccgcaggcctcgccgcctgccgcccatctggcgccgctca900
ccctcgccgcctgtggcc918
<210>9
<211>1417
<212>dna
<213>水稻(oryzasativa)
<400>9
tcatgttctggtagaactagtagaaggtgaggcctaactaaacttgaggtgatatattaa60
tacagtttgtttgtatatgctcatccttaccatgcattataaaaaaaacaaatctaggta120
aaacataaaaatacatcttgattaatccagaaagttgagaaactaaaaatttaagttgca180
ttaaatttacatatattaaaaaattctagcctatatatgtggcaaaagatatatcatgaa240
aaacattggtctcgggagaagggaattctggagggtccttgccacagcagcaaactcata300
aaaaaaaactatatatcatatatatttgtatagttaattagcactactgcagtctgcagc360
ttctcgcacgtacccttggtatcacgcgacggggaggagtaaactctacactgctccatc420
gattctttgcttcaagtccacgatatactatagtaatttgtttgcatccctagctagtac480
acataattaatccagaatattaattataactagtctgttttatctttccttaattatata540
tctttcaccgatcacctctattattccatatagcacgagtcaaatccgaccatcgatcac600
atgcatgaatgcatatggtgcatgctgcaaccaattaatcctcctgtcgtcgatcgcaaa660
gaaattattgatcaaaatctgacaggaattaatggtttggcgtttgtactgataagattc720
ttccttagctaattgacggtgactttgatgatgcccttgtgtccaatggaaacatcgcac780
ctgtgcccatctccatagcttggttaatctctaatttaactaattagagccgatctgaaa840
ccgtactagtcccctttgtcttgcagctgtcagtgacagattatatgggagaaagcgcta900
gcatgtagtatcgttcagattaagttaggtttactttttaccctgttaccagattaactt960
ttgttagtttttattaattaacctctctctcctggtcaggggggttggcatctcttgaga1020
agctaaccaacagcagcaagtgattaattacccctctcgcctcgtgtctcaacaccaggc1080
aggcatgccttgtcgtcgtcgttgtcctccgttaattagtttctactataccctaccgtg1140
tctgtgtgtacatgtgtcgctgtttaggttataaatatagctcgtttcacactctcatgg1200
cgattccttcatattcttaagctcaagctgatcaagtgtgtactagctagtaattagtcc1260
tatcctatatactatagtagctgggttgctcggctcgccggagatctccggccggctgcc1320
ggcggcggtgagccggtgacactaatcacatatcgatcgtacacgcgcgcgtattacgtg1380
tgtgaaggaacattagatagatagagagagagagggg1417
<210>10
<211>1420
<212>dna
<213>水稻(oryzasativa)
<400>10
tcatgttctggtagaactagtagaaggtgaggcctaactaaacttgaggtgatatattaa60
tacagtttgtttgtatatgctcatccttaccatgcattataaaaaaaacaaatctaggta120
aaacataaaaatacatcttgattaatccagaaagttgagaaactaaaaatttaagttgca180
ttaaatttacatatattaaaaaattctagcctatatatgtggcaaaagatatatcatgaa240
aaacattggtctcgggagaagggaattctggagggtccttgccacagcagcaaactcata300
aaaaaaaaactatatatcatatatatttgtatagttaattagcactactgcagtctgcag360
cttcacgcacgtacccttggtatcacgcgacggggaggagtaaactctacactgctccat420
cgattctttgcttcaagtccacgatatactatagtaatttgtttgcatccctagctagta480
cacataattaatccagaatattaattataactagtctgttttatctttccttaattatat540
atctttcaccgatcacctctattattccatatagcacgagtcaaatccgaccatcgatca600
catgcatgaatgcatatggtgcatgctgcaaccaattaatcctcctgtcgtcgatcgcaa660
agaaattattgatcaaaatctgacaggaattaatggtttggcgtttgtactgataagatt720
cttccttagctaattgacggtgactttgatgatgcccttgtgtccaatggaaacatcgca780
cctgtgcccatctccatagcttggttaatctctaatttaactaattagagccgatctgaa840
accgtactagtcccctttgtcttgcagctgtcagtgacagattatatgggagaaagcgct900
agcatgtagtatcgttcagattaagttaggtttactttttaccctgttaccagattaact960
tttgttagtttttattaattaacctctctctcctggtcaggggggttggcatctcttgag1020
aagctaaccaacagcagcaagtgattaattacccctctcgcctcgtgtctcaacaccagg1080
caggcatgccttgtcgtcgtcgttgtcctccgttaattagtttctactataccctaccgt1140
gtctgtgtgtacatgtgtcgctgtttaggttataaatatagctcgtttcacactctcatg1200
gcgattccttcatattcttaagctcaagctgatcaagtgtgtactagctagtaattagtc1260
ctatcctatatactatagtagctgggttgctcggctcgccggagatctccggccggctgc1320
cggcggcggtgagccggtgacactaatcacatatcgatcgtacacgcgcgcgtattacgt1380
gtgtgaaggaacattagatagatagagagagagagagggg1420
<210>11
<211>795
<212>dna
<213>水稻(oryzasativa)
<400>11
tcatattagaaagatatcattactattcacctatttagaaccataccattacaattcttt60
ctttatgctagaaaaaatgtccgtacattgcaacaggtgaatgctattttaatcttatta120
ttgttatacggtttagctataatcaaattcaccgtgggaattcagttggatatttttttt180
agaaaatcatgagctgcaattaggaatccaactttcatctcaagttagcatgtgagttat240
tttaaagagatttcttatacgactccttttgtatttccaaaagcaaacgaatttaaaatc300
tgactcaaacaagtatctgtatttccaaaagtgaacaaacttaaaaacccgactcaaata360
cggatgatgtaccaaaataccggtaaaaacatcttcaattttttataatagtagagataa420
tatatgacattttgtacaccttggccgctattggacccacgagcatgcaattgtattttt480
gaatcacccaaaatgcccctgcaacatatttatctccaccagctaacttgtgggccccac540
gcccttcctcgggatgagctgctcgaggcacgacaaccactgtgagctcctccgtgatgg600
acatccaaaagagtgtggtgtagctaaacgatgaaagagagcttctccaacgcagggttg660
tattggaggtcaaggtccttggccttgatgtgggtgcgaaagatgtcgagctcggtgagg720
ttggatgaagatgtcagcgcgagggcggaggtagtcggtgtcaggaaatgacttcccgag780
atcaagatagaggcg795
<210>12
<211>795
<212>dna
<213>水稻(oryzasativa)
<400>12
tcatattagaaagatatcattactattcacctatttagaaccataccattacaattcttt60
ctttatgctagaaaaaatgtccgtacattgcaacaggtgaatgctattttaatcttatta120
ttgttatacggtttagctataatcaaattcaccgtgggaattcagttggatatttttttt180
agaaaatcatgagctgcaattaggaatccgactttcatctcaagttagcatgtgagttat240
tttaaagagatttcttatacgactccttttgtatttccaaaagcaaacgaatttaaaatc300
tgactcaaacaagtatctgtatttccaaaagtgaacaaacttaaaaacccgactcaaata360
cggatgatgtaccaaaataccggtaaaaacatcttcaattttttataatagtagagataa420
tatatgacattttgtacaccttggccgctattggacccacgagcatgcaattgtattttt480
gaatcacccaaaatgcccctgcaacatatttatctccaccagctaacttgtgggccccac540
gcccttcctcgggatgagctgctcgaggcacgacaaccactgtgagctcctccgtgatgg600
acatccaaaagagtgtggtgtagctaaacgatgaaagagagcttctccaacgcagggttg660
tattggaggtcaaggtccttggccttgatgtgggtgcgaaagatgtcgagctcggtgagg720
ttggatgaagatgtcagcgcgagggcggaggtagtcggtgtcaggaaatgacttcccgag780
atcaagatagaggcg795
<210>13
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>13
ctcctctgtcctctcccaag20
<210>14
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>14
acccagcaagtttgttagaagt22
<210>15
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>15
tacacgtacgccaagctaac20
<210>16
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>16
cactctcatggcgattcctt20
<210>17
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>17
ctcggtgaggttggatgaag20
<210>18
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>18
ttcgaagggtgtcgttatgc20
<210>19
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>19
aaatgcaaacggacaatcctg21
<210>20
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>20
ctgaccgcaaagcaaaacaa20
<210>21
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>21
cgtaaaggagtaatggcagcg21
<210>22
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>22
gcgatgattggagtcggaga20
<210>23
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>23
aatcatccgcaaaccgca18
<210>24
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>24
agtaggcgttgaggagcacc20
<210>25
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>25
gtgattagtgtcaccggctc20
<210>26
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>26
agaagggaattctggagggt20
<210>27
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>27
ccctctcgcctcgtgtctc19
<210>28
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>28
gtggacgatggcgaacttg19
<210>29
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>29
tgaatgatctactttaaactgggtca26
<210>30
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>30
gcatcatcaaagtcaccgtcaa22
<210>31
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>31
ctgtaagacgcggtacgttt20
<210>32
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>32
cgcctctaccttggtcacta20
<210>33
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>33
atactgtaagacgcggtacg20
<210>34
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>34
tcttccccaaccttcttgtg20
<210>35
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>35
tgccattacaattctttctttatgc25
<210>36
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>36
tcgtgggtccaatagcgg18
- 还没有人留言评论。精彩留言会获得点赞!