用于普通小麦常规种品种纯度鉴定的特异性SNP位点组合以及鉴定方法与流程
本发明涉及品种纯度鉴定领域,具体涉及用于普通小麦常规种品种纯度鉴定的特异性snp位点组合以及鉴定方法。
背景技术:
小麦是我国重要的粮食作物之一,小麦种子质量优劣直接关系到农民的切身利益和国家粮食安全,在种子生产、加工、贮藏、销售等各环节进行种子质量监控,对确保我国粮食安全、保护育种家和农民的切身经济利益具有重要的社会和经济意义。近年来,随着我国种植业粮食结构的调整和市场经济的发展,小麦精播用种量显著降低,生产上对种子质量和品种纯度提出了更高的要求。品种纯度是衡量种子质量的重要指标之一,目前有关国家标准所规定的鉴定方法为形态鉴定法、苯酚染色法、氢氧化钠测定法、醇溶蛋白法以及田间小区种植鉴定法。形态鉴定法、苯酚染色法、氢氧化钠测定法和醇溶蛋白法等具有速度快、费用低、操作简单等优点,但区分能力非常有限,降低了鉴定结果的准确性。田间种植鉴定占地面积大,费时费工,易受环境因素的影响,难以区别表型相近的个体。综上所述,以上方法均已不能满足准确和快速鉴定的需要,给种子质量监管带来了巨大的困难。
随着分子生物学的发展,以ssr和snp标记为代表的dna指纹检测技术因具备共显性、高多态性、在基因组上均匀分布、高准确性、较好的重复性、较高通量、易实现自动化等优点,可快速准确地鉴别非典型个体,实现种子质量准确有效的监管。这两种标记已相继被国际植物新品种保护联盟(upov)、国际种子联盟(isf)和国际种子检验协会(ista)推荐作为作物品种鉴定和指纹数据库构建的优选标记。目前ssr标记法在品种鉴定中的研究与应用已基本趋向成熟,但其具有不易实现数据整合和通量低等缺陷。snp标记为二等位基因,数据统计简单,同时,国内外大公司相继推出了各种snp高通量检测平台,能很好地弥补snp标记的技术缺陷。现阶段高通量snp检测方法主要有dna芯片、定点测序和竞争性等位基因特异性pcr(kompetitiveallelespecificpcr,kasp)技术等。芯片技术为位点高通量检测技术,成本较高,适于少量样本大量位点的检测;定点测序技术成本高、检测时间长、适于大量样品指纹数据库构建;kasp技术为样本高通量检测技术,可对snps进行精准的双等位基因判断,具有高通量、高质量、准确、低成本、操作简单、灵活等优点,适于大量样本少量位点的检测,尤其适合品种纯度检测技术的要求。
技术实现要素:
针对现有技术存在的问题,本发明的目的是提供一组基于kasp技术开发的用于普通小麦常规种品种纯度鉴定的特异性snp位点组合,基于所述特异性snp位点组合,可实现对小麦常规种品种纯度的高通量snp检测。
本发明的再一目的是提供小麦常规种品种纯度鉴定方法。
本发明的用于小麦常规种品种纯度鉴定的特异性snp位点组合包括小麦基因组的24个snp位点,编号为
表1.用于小麦常规种纯度鉴定的专用snp位点
所述
本发明的特异性snp位点组合中的各个snp位点分布于小麦基因组的21条染色体上,每条染色体上snp位点数目为1~2个,共24个snp位点。基于这套专用snp位点组合,可实现对小麦常规种品种纯度的高通量snp分型检测,准确性高、重复稳定性好、效率高、方便快速。
本发明还提供用于扩增所述特异性snp位点的引物组合,包括上述24个snp位点的引物组,编号为
wsnp01p的正向引物1的序列为:5’ggagaacaaagcagaaaagctgcaa3’,
wsnp01p的正向引物2的序列为:5’gagaacaaagcagaaaagctgcag3’,
wsnp01p的反向引物的序列为:5’gtgaacattcaggatatttgtgctgcata3’;
wsnp02p的正向引物1的序列为:5’ggcaactctaacaaacacactcc3’,
wsnp02p的正向引物2的序列为:5’agggcaactctaacaaacacactct3’,
wsnp02p的反向引物的序列为:5’gacgaactcggcgggaccattt3’;
wsnp03p的正向引物1的序列为:5’aacacattcagagaatatgactaggg3’,
wsnp03p的正向引物2的序列为:5’ataacacattcagagaatatgactagga3’,
wsnp03p的反向引物的序列为:5’gcctgagcgggcgtttggtaa3’;
wsnp04p的正向引物1的序列为:5’cctccctcccgacctctg3’,
wsnp04p的正向引物2的序列为:5’ccctccctcccgacctcta3’,
wsnp04p的反向引物的序列为:5’ctagttgggccgaggcagttgaa3’;
wsnp05p的正向引物1的序列为:5’agttgaaaacaccgaaccagcga3’,
wsnp05p的正向引物2的序列为:5’gttgaaaacaccgaaccagcgg3’,
wsnp05p的反向引物的序列为:5’ggtgagagcaaggatcccaatctat3’;
wsnp06p的正向引物1的序列为:5’catgagatcctaaatcagagtcgca3’,
wsnp06p的正向引物2的序列为:5’atgagatcctaaatcagagtcgcg3’,
wsnp06p的反向引物的序列为:5’ggtcaagttttctggaccccataattatt3’;
wsnp07p的正向引物1的序列为:5’cgattgaatctcaagcgaacaaacg3’,
wsnp07p的正向引物2的序列为:5’gcgattgaatctcaagcgaacaaaca3’,
wsnp07p的反向引物的序列为:5’tgtagacatctagcaaccccctgaa3’;
wsnp08p的正向引物1的序列为:5’ggtaagctttgcctagctttcctat3’,
wsnp08p的正向引物2的序列为:5’gtaagctttgcctagctttcctac3’,
wsnp08p的反向引物的序列为:5’gtttgtccaactaagatagagtatgggaa3’;
wsnp09p的正向引物1的序列为:5’gtaagggacacacctccatccat3’,
wsnp09p的正向引物2的序列为:5’aagggacacacctccatccag3’,
wsnp09p的反向引物的序列为:5’atagctagattgtcatctggccaaacaaa3’;
wsnp10p的正向引物1的序列为:5’gataacagcgttttcctgacgaga3’,
wsnp10p的正向引物2的序列为:5’ataacagcgttttcctgacgagg3’,
wsnp10p的反向引物的序列为:5’ccatccccaaccataggaccgat3’;
wsnp11p的正向引物1的序列为:5’agattcctggagctgcgtttgc3’,
wsnp11p的正向引物2的序列为:5’agattcctggagctgcgtttgg3’,
wsnp11p的反向引物的序列为:5’agttgaatttggcggacgtgaggtt3’;
wsnp12p的正向引物1的序列为:5’cacagggataccgaaacatgga3’,
wsnp12p的正向引物2的序列为:5’cacagggataccgaaacatggg3’,
wsnp12p的反向引物的序列为:5’ttgttaccgattttgttctcttttgtcgtt3’;
wsnp13p的正向引物1的序列为:5’ggagatctgctagagacgcct3’,
wsnp13p的正向引物2的序列为:5’ggagatctgctagagacgccg3’,
wsnp13p的反向引物的序列为:5’ggatgccatgccatgagttagtgtt3’;
wsnp14p的正向引物1的序列为:5’gcgttcgaattaaggagagaatcag3’,
wsnp14p的正向引物2的序列为:5’ggcgttcgaattaaggagagaatcaa3’,
wsnp14p的反向引物的序列为:5’cagtgtcgtaggctggtcgctt3’;
wsnp15p的正向引物1的序列为:5’caatcatattgttttggcgaggagc3’,
wsnp15p的正向引物2的序列为:5’aacaatcatattgttttggcgaggagt3’,
wsnp15p的反向引物的序列为:5’cagagctgaaccccactggtgta3’;
wsnp16p的正向引物1的序列为:5’aggctgagcaagagttcttctg3’,
wsnp16p的正向引物2的序列为:5’gtaggctgagcaagagttcttcta3’,
wsnp16p的反向引物的序列为:5’tattcttctgctcgacgtcgcactt3’;
wsnp17p的正向引物1的序列为:5’ccttcctctccgtgatcgtcat3’,
wsnp17p的正向引物2的序列为:5’cttcctctccgtgatcgtcag3’,
wsnp17p的反向引物的序列为:5’ggtttgccggctagaccacgta3’;
wsnp18p的正向引物1的序列为:5’gaacaaattagagatgctattaagtggg3’,
wsnp18p的正向引物2的序列为:5’gaacaaattagagatgctattaagtgga3’,
wsnp18p的反向引物的序列为:5’ccaagttatcgtggcgtacaagacat3’;
wsnp19p的正向引物1的序列为:5’catctcttcacggtacatcaactcat3’,
wsnp19p的正向引物2的序列为:5’atctcttcacggtacatcaactcac3’,
wsnp19p的反向引物的序列为:5’gtggccatccgacagaaaaagtattaatt3’;
wsnp20p的正向引物1的序列为:5’atgggcctgttgaactagccac3’,
wsnp20p的正向引物2的序列为:5’catgggcctgttgaactagccat3’,
wsnp20p的反向引物的序列为:5’ggagtaaatcgcatagaaccaccataaat3’;
wsnp21p的正向引物1的序列为:5’cgcacgggactctgtccaca3’,
wsnp21p的正向引物2的序列为:5’gcacgggactctgtccacc3’,
wsnp21p的反向引物的序列为:5’ggactgtctagttgttgcaatcgcat3’;
wsnp22p的正向引物1的序列为:5’gccaaaagcccaacacagagga3’,
wsnp22p的正向引物2的序列为:5’ccaaaagcccaacacagaggc3’,
wsnp22p的反向引物的序列为:5’gcttcaagctgtggatcgctatcta3’;
wsnp23p的正向引物1的序列为:5’cagggaaagatcattaacgtcaacc3’,
wsnp23p的正向引物2的序列为:5’gcagggaaagatcattaacgtcaact3’,
wsnp23p的反向引物的序列为:5’gtcccgacttcgaattaatgaagccat3’;
wsnp24p的正向引物1的序列为:5’agggaattcttctacttttctaaaccg3’,
wsnp24p的正向引物2的序列为:5’tagggaattcttctacttttctaaacca3’,
wsnp24p的反向引物的序列为:5’gttcagtgttgggtccgtctgtaaa3’;
本发明还提供用于鉴定小麦常规种品种纯度的试剂盒,包含上述引物组合的混合液或者粉末,各个引物组由同一引物组的正向引物1、正向引物2和反向引物构成。
根据本发明的用于鉴定小麦常规种品种纯度的试剂盒,其中,两条正向引物的5’端分别带有不同的序列标签,所述序列标签的核苷酸序列互不相同,并且与小麦基因组序列不同源。
根据本发明的用于鉴定小麦常规种品种纯度的试剂盒,其中,两条正向和反向引物的摩尔比为2:2:5。
根据本发明的用于鉴定小麦常规种品种纯度的试剂盒,其中,所述试剂盒还包含pcr预混液,所述pcr预混液含有荧光探针和淬灭探针,所述荧光探针的核苷酸序列与序列标签的核苷酸序列一致,其5’端连接荧光基;所述淬灭探针的核苷酸序列与所述序列标签的核苷酸序列反向互补,其3’端连接淬灭基团。
根据本发明的鉴定小麦常规种品种纯度的方法包括以下步骤:
(1)提取待测小麦品种的dna;
(2)采用kasp技术进行基因分型:以步骤(1)中的dna为模板,加入上述所述snp引物组合中的每一组引物混合液和pcr预混液,进行pcr扩增;采用荧光检测仪检测pcr扩增产物的荧光信号,根据荧光信号的颜色获得待测小麦品种在所述引物组合对应的snp标记位点的基因型;
(3)品种纯度计算:统计分析步骤(2)获得的待测样品个体的基因型,鉴别非典型个体,计算品种纯度。
根据本发明的鉴定小麦常规种纯度的方法,其中,在步骤(3)中,首先判断每个snp位点是否为非纯合位点,将非纯合snp位点排除后,若某一待测小麦个体基因型在2个及以上snp位点上不同于其他多数待测个体,则判断定所述待测小麦个体为非典型个体。
根据本发明的鉴定小麦常规种品种纯度的方法,其中,在步骤(3)中,当某个位点在待测样品群体中具有两种来自双亲的等位变异,且随机分布在不同待测小麦个体中时,判定该位点为非纯合snp位点。
本发明的技术方案的优点和益处:
1、本发明提供的24个特异性snp位点是经过多样品多次试验确定的,重复性和稳定性好,便于推广应用;
2、本发明提供的特异性snp位点组合,对1433份小麦审定品种识别效率可达到95.67%,分辨力高,在确保品种纯度鉴定结果准确性的基础上,大大降低了检测成本;
3、本发明提供的用于鉴定小麦常规种品种纯度的方法,解决了非纯合snp位点对非典型个体准确判断的干扰,确保了品种纯度鉴定结果的准确性;
4、本发明提供的用于鉴定小麦常规种品种纯度的特异性snp位点组合以及鉴定方法,仅需将本发明的特异性snp引物混合液和通用的pcr预混液加入到含有dna样本的pcr微孔反应板中,进行pcr扩增,采用荧光检测仪分析pcr产物即可,1人半天即可完成1个品种的纯度检测。本方法具有操作简单、方便、高通量、准确、高效、低成本、节约人力、物力等优点。
附图说明
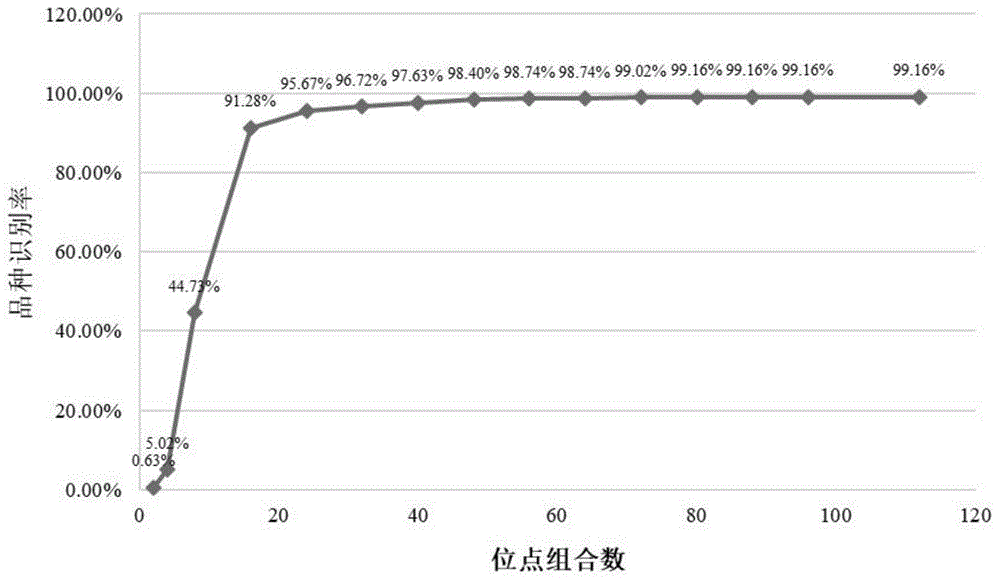
图1显示不同snp位点组合数目的品种识别率;
图2显示24个snp位点基因分型图。
具体实施方式
以下的实施例便于更好地理解本发明,但并不限定本发明。以下实施例中所用的实验材料或试剂,如无特殊说明,均为自常规生化试剂商店购买得到的。以下实施例中使用的pcr预混液购自英国lgc公司,货号为kbs-1016-003
实施例1、基于kasp技术开发的24个用于小麦常规种品种纯度鉴定的专用snp位点
以189份具有广泛遗传代表性的样品为材料,筛选出420个分型质量高(phr类型)、缺失率小于10%、最小等位变异频率(maf)大于0.3、连锁不平衡值(ld)小于0.8、物理距离大于2m、在染色体上均匀分布的snp位点,获取位点侧翼序列,设计并合成kasp引物。
利用kasp引物对上述189份样品中的95份样品进行基因分型,筛选出282个自动分型效果好、缺失率小的snp位点。
以我国1433份小麦审定标准样品为材料评价282个snp位点,筛选出112个重复性和稳定性好、缺失率小的snp位点。基于1433份小麦标准样品112个snp位点的指纹数据,确定24个snp最优位点组合,该组合对小麦品种识别效率可达到95.67%,既满足检测效率、准确率的要求,又显著降低了检测成本,操作方便。
所述24个snp位点组合不能区分的少量品种均有高度系谱相关性或者为实质性派生品种。
本发明的24个snp位点的基因分型结果如图2所示,各个引物组在待测品种个体样本中可以得到很好的分型效果。
本发明的24个专用snp位点的多态性信息见表3。
表224个专用snp位点的多态性信息
根据24个snp位点的侧翼序列,针对每个snp位点,在snp位点上游设计两条正向引物,下游设计一条反向引物。设计得到的24个snp位点的引物序列信息见表3。wsnp01p~wsnp24p引物组分别用于检测wsnp01~wsnp24标记。
所述24个专用snp位点的maf和pic值是根据1433份小麦审定品种计算出来的。
表3用于pcr扩增专用snp标记的引物组
实施例2、利用本发明的特异性snp标记组合检测待测小麦常规品种“京花10号”的纯度
检测方法如下:
1、dna制备
随机挑取待测品种“京花10号”种子95粒,提取dna。保证提取的dna数量和质量符合pcr扩增的要求,dna无降解,溶液的紫外光吸光度od260与od280的比值宜介于1.70~2.0之间,dna的浓度在30ng/μl以上,dna总量至少为2ug。将每粒种子的dna统一稀释至30ng/μl,4℃保存备用。
2、采用kasp技术进行基因分型
选择实施例1中24个snp位点的特异性引物(编号为wsnp01p~wsnp24p)进行检测。正向引物1和正向引物2的5’端分别加上序列标签a和序列标签b
序列标签a:gaaggtgaccaagttcatgct;
序列标签b:gaaggtcggagtcaacggatt;
所述序列标签a和序列标签b适用于英国lgc公司kaspmastermix(pcr预混液)。所述引物合成后,用10mmph8.3的tris-hcl溶解三条引物至100μm,分别取18μl的两条正向引物溶液,45μl的反向引物溶液和69μl10mmph8.3的tris-hcl溶液,混合成150ul引物混合液备用;
kaspmastermix(pcr预混液)包含了荧光探针a、荧光探针b、淬灭探针a和淬灭探针b、rox内参染料,kleartaqdna聚合酶,dntp和mgcl2,由英国lgc公司提供。
所述荧光探针a带有的荧光基团a为fam,荧光信号具体为蓝色;所述荧光探针b带有的荧光基团b为hex,荧光信号具体为红色;所述淬灭探针带有的淬灭基团为bhq。
基因分型检测步骤如下:
(1)将95粒种子dna和ddh2o(阴性对照,ntc)分别分装到384孔板96个pcr微孔中;
(2)向每个孔中加入引物混合液和通用的kaspmastermix,封板,离心1min后,按照如下反应体系和反应条件进行水浴pcr:
所述pcr的反应体系为:30ng/μl模板dna1.5μl;pcr预混液1.5μl;引物混合液0.042μl,其中两条正向引物的终浓度均为0.504nm,反向引物的终浓度为1.26nm。
所述pcr的反应条件为:94℃预变性15min;94℃变性20s,61~55℃退火延伸60s,每个循环的退火延伸降低0.6℃,共10个循环;94℃变性20s,55℃退火延伸60s,26-36个循环。
(3)完成步骤(2)后,待pcr扩增产物温度降至40℃以下时使用qpcr仪或者带有fret功能的酶标仪进行荧光信号读取,根据荧光信号颜色获得待测样品95个个体在编号为wsnp01~wsnp24的snp标记位点的基因分型结果(见表4-1和表4-2)。
需要说明的是,若pcr扩增结束后荧光信号弱,影响数据分析,需要增加循环数,直至结果满意为止。每增加一次循环的程序为94℃20s,57℃60s,3个循环,每增加一次循环后扫描。
表4-1
表4-2
3、品种纯度计算
(1)统计分析待测样品95个个体24个snp位点的基因型,首先判断24个snp位点是否为非纯合位点。
所述非纯合snp位点的特点是该位点在待测样品群体中具有两种来自双亲的等位变异,且随机分布在不同待测小麦个体中时,判定该位点为非纯合snp位点。如表4所示,“京花10号”的95个个体在位点wsnp20上分别携带来自父母本的基因型或父母本的杂合基因型,即有55个个体携带aa基因型、39个个体携带gg基因型,1个个体携带ag基因型,a和g两种等位变异随机分布在不同待测小麦个体中,应判为非纯合snp位点。
(2)将非纯合snp位点排除后,若某一待测个体中存在2个及以上位点不同于其他多数待测个体,则判定所述待测小麦个体为非典型个体。如表4所示,排除非纯合位点wsnp20后,93个个体在其余23个snp位点的基因型一致,为正常株;编号为25的个体在wsnp02、wsnp10、wsnp13、wsnp15、wsnp18和wsnp19等8个snp位点上不同于其他93个个体,判断为非典型个体;编号为81的个体在wsnp02、wsnp07、wsnp10、wsnp15、wsnp18和wsnp19等8个snp位点上不同于其他93个个体,判断为非典型个体。
(3)根据步骤(2)中非典型个体数目和检测试样总数计算品种纯度。
所述品种纯度用正常个体数目(检测试样总数减去非典型个体数目)占检测试样总数的百分率表示。所述待测品种“京花10号”的品种纯度为:
(95-2)/95*100%=97.9%。
序列表
<110>北京市农林科学院
<120>用于普通小麦常规种品种纯度鉴定的特异性snp位点组合以及鉴定方法
<160>30
<170>siposequencelisting1.0
<210>1
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>1
ggagaacaaagcagaaaagctgcaa25
<210>2
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>2
gagaacaaagcagaaaagctgcag24
<210>3
<211>29
<212>dna
<213>人工序列(artificialsequence)
<400>3
gtgaacattcaggatatttgtgctgcata29
<210>4
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>4
ggcaactctaacaaacacactcc23
<210>5
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>5
agggcaactctaacaaacacactct25
<210>6
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>6
gacgaactcggcgggaccattt22
<210>7
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>7
aacacattcagagaatatgactaggg26
<210>8
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>8
ataacacattcagagaatatgactagga28
<210>9
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>9
gcctgagcgggcgtttggtaa21
<210>10
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>10
cctccctcccgacctctg18
<210>11
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>11
ccctccctcccgacctcta19
<210>12
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>12
ctagttgggccgaggcagttgaa23
<210>13
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>13
agttgaaaacaccgaaccagcga23
<210>14
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>14
gttgaaaacaccgaaccagcgg22
<210>15
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>15
ggtgagagcaaggatcccaatctat25
<210>16
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>16
catgagatcctaaatcagagtcgca25
<210>17
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>17
atgagatcctaaatcagagtcgcg24
<210>18
<211>29
<212>dna
<213>人工序列(artificialsequence)
<400>18
ggtcaagttttctggaccccataattatt29
<210>19
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>19
cgattgaatctcaagcgaacaaacg25
<210>20
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>20
gcgattgaatctcaagcgaacaaaca26
<210>21
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>21
tgtagacatctagcaaccccctgaa25
<210>22
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>22
ggtaagctttgcctagctttcctat25
<210>23
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>23
gtaagctttgcctagctttcctac24
<210>24
<211>29
<212>dna
<213>人工序列(artificialsequence)
<400>24
gtttgtccaactaagatagagtatgggaa29
<210>25
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>25
gtaagggacacacctccatccat23
<210>26
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>26
aagggacacacctccatccag21
<210>27
<211>29
<212>dna
<213>人工序列(artificialsequence)
<400>27
atagctagattgtcatctggccaaacaaa29
<210>28
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>28
gataacagcgttttcctgacgaga24
<210>29
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>29
ataacagcgttttcctgacgagg23
<210>30
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>30
ccatccccaaccataggaccgat23
- 还没有人留言评论。精彩留言会获得点赞!
- 一种在鱼类生殖系细胞中特异性表达基因的3’UTR区序列及其应用的制造方法与工艺
- 一种氯霉素分子印迹聚合物的制备方法与制造工艺
- 含有与前列腺抗原特异性结合短肽的噬菌体及其应用的制造方法与工艺
- 一种多级肝靶向智能化纳米递药系统的制备方法和应用与制造工艺
- 体外检测肿瘤新生抗原特异性T细胞的方法、试剂盒以及肿瘤疫苗与制造工艺
- 双特异性T细胞活化性抗原结合分子的制造方法与工艺
- 一种特异性识别及灵敏检测人血清蛋白的荧光试剂合成方法及应用与制造工艺
- 运用核酸适配体与溶菌酶特异性识别并形成G四链体进行荧光显现潜指纹的方法与制造工艺
- 双芘修饰苝酰亚胺衍生物荧光探针及其合成方法和应用与制造工艺
- 一种脂质体疫苗制剂及其用途和生产IgG抗体的方法与制造工艺
- 与CD56分子特异性结合的多肽及其应用的制造方法与工艺
- 用于治疗HBV感染和相关病症的三特异性结合分子的制造方法与工艺
- 对磺胺类药物具有特异性的固相微萃取纤维的制备方法与流程
- 避免信号干扰的肌原纤维蛋白丝氨酸磷酸化水平的测定方法与流程
- 一种检测猪初乳中抗PEDV特异性IgA抗体的夹心ELISA试剂盒的制造方法与工艺
- 用于施用CD19xCD3双特异性抗体的给药方案的制造方法与工艺
- 双特异性抗‑VEGF/抗‑ANG‑2抗体及其在治疗眼血管疾病中的应用的制造方法与工艺
- 特异性人EWS‑FLI1融合蛋白抗体的制备方法及在制备尤文肉瘤诊断抗体试剂的应用与制造工艺
- 活化人CD40的抗体和针对人PD‑L1的抗体的组合疗法的制造方法与工艺
- 双特异性四价抗体及其制造和使用方法与制造工艺