功能化的dCas9修饰蛋白及其在检测核酸中的应用的制作方法
本发明涉及生物工程技术领域,具体涉及两种功能化的dcas9修饰蛋白及其在检测核酸中的应用。
背景技术:
核酸检测可为生命科学、公共安全、医疗诊断和相关基础研究提供重要依据。目前,核酸检测作为一项重要的分子诊断实践不断发展。已经开发出诸如印迹杂交、聚合酶链式反应(pcr)、杂交捕获、基因芯片等技术。然而现有的多数技术在性能指标上具有偏重性,检测需繁琐的样品/试剂处理,容易产生假阳/阴结果。因此,设计开发便捷特异性高的核酸检测手段就显得尤为重要。
crispr/cas是原核生物进化出的一类抵抗外来病毒入侵的免疫系统。这类工程编辑系统利用酶把一段作为引导工具的小rna(sgrna)切入dna或rna,能在特定的核酸序列处切断,具有很高的特异性。目前这种基因“魔剪”已经扩展到几乎所有的基因组目标,并被探索应用于生物传感新兴方向。
crispr/cas9复合物是一种新的dna靶向编辑工具,其包括一个cas9蛋白和一条引导rna(sgrna),其中,sgrna负责识别外源dna并与之杂交,而cas9含有在氨基末端的ruvc和蛋白质中部的hnh两个独特的活性位点,在双链dna剪切中发挥作用。目前crispr/cas9被广泛应用基因编辑和调控,还没有充分的探讨用于核酸检测领域。
dcas9蛋白是cas9蛋白的突变体,即cas9内切酶的ruvc1和hnh两个核酸酶活性区域同时发生突变,导致cas9蛋白的内切酶活性全部消失,只保留由sgrna引导进入基因组的能力。专利cn107574226a通过将cas9的酶切位点定点突变,使得其酶切功能失活,能够仅应用其精确的靶向功能,实现核酸检测的技术,而避免了cas9酶切功能对待检测基因进行剪切等不良作用。但在核酸的实际检测的过程中,由于cas9的脱靶效应、样品成分复杂以及ru(bpy)32+强正电性都会导致核酸的非特异性结合捕获,使定量不准,容易报告假阳、阴结果。
技术实现要素:
针对上述现有技术,本发明的目的是提供一种功能化的dcas9修饰蛋白及其在检测核酸中的应用。本发明分别用生物素和碱性磷酸酶标记dcas9蛋白,得到功能化的dcas9修饰蛋白;然后在此基础上,利用固相磁性微球与crispr指导的“夹心识别”构建一种检测核酸的化学发光方法,可实现体系中0.42fmol核酸(3.6kbp)的检出。
为实现上述目的,本发明采用如下技术方案:
本发明的第一方面,提供功能化的dcas9修饰蛋白,包括:dcas9-biotin和dcas9-ap;
所述dcas9-biotin由如下方法制备而成:
将dcas9蛋白液用ph=8.0的pbs缓冲液超滤浓缩处理,使dcas9蛋白的终浓度大于或等于2mg/ml,得到dcas9蛋白溶液;向dcas9蛋白溶液中加入ez-link-nhs-biotin母液,混合均匀,室温反应30-60分钟,加入甘氨酸终止反应,以ph=8.0的pbs缓冲液为流动相过除盐柱,除去未反应小分子,收集修饰蛋白,超滤浓缩,得到dcas9-biotin;
所述dcas9-ap由如下方法制备而成:
将dcas9蛋白液用ph=8.0的pbs缓冲液超滤浓缩处理,使dcas9蛋白的终浓度大于或等于2mg/ml,得到dcas9蛋白溶液;向dcas9蛋白溶液中加入traut’s试剂,混合均匀,20-30℃条件下反应80-100min,反应结束后超滤除盐,得到第一溶液;
向碱性磷酸酶(ap)溶液中加入smcc试剂,混合均匀,20-30℃条件下反应80-100min,反应结束后超滤除盐,得到第二溶液;
将第一溶液和第二溶液按体积比1:1混合,4℃条件下反应12-24h,制备得到dcas9-ap。
优选的,所述dcas9蛋白的氨基酸序列如seqidno.2所示。
本发明的第二方面,提供上述功能化的dcas9修饰蛋白在如下(1)或(2)中的应用:
(1)检测核酸;
(2)制备检测核酸的试剂。
优选的,所述核酸为dna。
本发明的第三方面,提供一种检测核酸的试剂,所述试剂以上述功能化的dcas9修饰蛋白为有效成分。
进一步的,所述试剂还包含:sgrna1、sgrna2、清洗液和反应液。
优选的,所述sgrna1的序列如seqidno.3所示;所述sgrna2的序列如seqidno.4所示。
优选的,所述清洗液为添加有0.1~0.5mol/lnacl、0.1~0.01%bsa、0.01~0.05%tritonx-100、1~5mmol/mledta、0.01~0.05%叠氮钠、0.02-0.05%proclin300、0.1~0.01%变性鲑鱼精dna的0.1mpbs,ph6.0~8.0。
优选的,所述反应液中含有1m二乙醇胺、5mmmgcl2、0.2mkcl和20mm碱性磷酸酶底物amppd,反应液的ph为10.3。
本发明的第四方面,提供一种由dcas9介导特异性夹心识别dna检测核酸的方法,利用上述试剂进行检测,具体包括以下步骤:
(1)将dcas9-biotin与固相磁性微球复合,得到dcas9包被的固相磁性微球;将dcas9包被的固相磁性微球用清洗液稀释至10-30μg/ml;向稀释后的dcas9包被的固相磁性微球中加入sgrna1,孵育,得到r1&sgrna1;
(2)将dcas9-ap用清洗液稀释至50-150μg/ml;向稀释后的dcas9-ap中加入sgrna2,孵育,得到r2&sgrna2;
(3)将系列浓度梯度的dna标准品分别与r2&sgrna2混合,37℃条件下孵育10-20min,反应结束后再加入r1&sgrna1,37℃条件下孵育10-20min,磁性分离磁球,弃去上清,磁球用清洗液进行清洗,清洗后的磁球用清洗液重悬,检测其发光值,构建发光值与dna标准品浓度的工作曲线,利用构建的工作曲线对待测样品进行核酸检测。
本发明的有益效果:
(1)本发明调取化脓链球菌(streptococcuspyogene)的crispr/cas9基因,利用分子生物学方法得到具有核酸结合而不具备切割能力的dcas9;本发明在dcas9基础上修饰得到两种功能化蛋白dcas9-biotin和dcas9-ap,在此基础上构建一种新型的检测核酸的化学发光方法;其中,通过固相修饰的dcas9-biotin对dna进行特异性捕获,通过碱性磷酸酶标记的dcas9进行定量检测,可实现体系中0.42fmol核酸(3.6kbp)的检出;dcas9-biotin和dcas9-ap的夹心识别更提高检测体系的特异性,为进一步降低核酸检测假阳/假阴结果提供有效手段。
(2)为配合功能化蛋白dcas9-biotin和dcas9-ap的核酸检测,本发明在检测体系中还开发设计了清洗液和反应液,其中,清洗液能有效减少核酸、dcas9功能化蛋白的非特异结合,稳定蛋白,延长试剂效期,增加体系分散性;反应液则是提供反应底物,用于发光检测。
附图说明
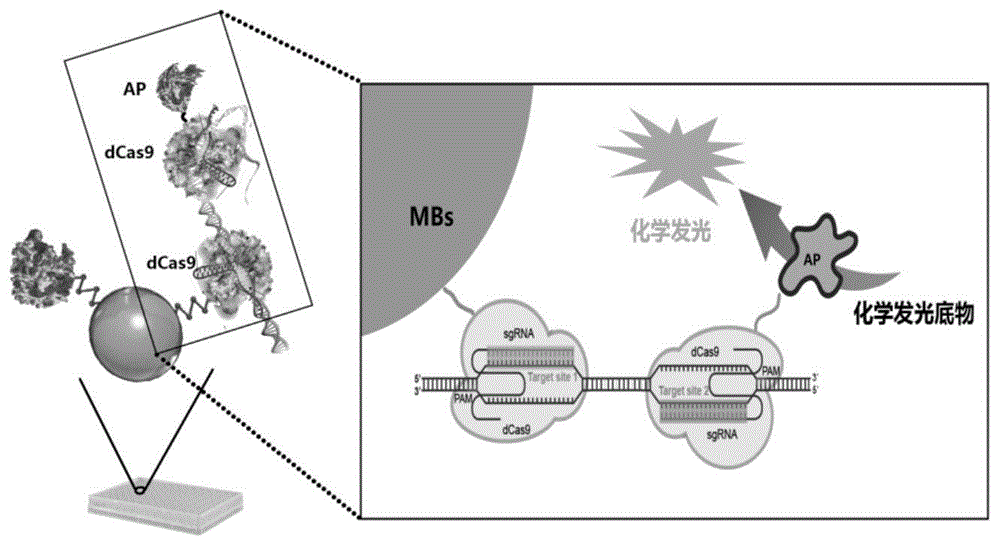
图1:本发明的检测原理示意图;dcas9-biotin&sgrna1和dcas9-ap&sgrna2与dna形成夹心复合物,使得dcas9-ap被捕获到dcas9-biotin修饰的磁性微球(mbs)表面,dcas9-ap催化发光底物amppd发光,其发光值与与dna成正比,以此实现对核酸的检测。
图2:最终纯化后dcas9的sds-page蛋白电泳图;其中,m道是marker,2道为纯净的dcas9样本。
图3:本发明制备的dcas-biotin及dcas9-ap的sds-page蛋白电泳图;其中,1道为生物素修饰的dcas-biotin样本,2道为ap样本,3道为ap修饰的dcas-ap样本,m道是marker。
图4:本发明所述dcas9-biotin与dcas9-ap与dna结合的凝胶阻滞实验;其中,m道是marker,1道为dcas9-biotin+sgrna1+dna1,2道为dcas9-biotin+dna1,3道为空白道,4道为dcas9-ap+sgrna1+dna1,5道为dcas9-ap+dna1。
图5:本发明所述的dcas9功能蛋白夹心识别检测核酸化学发光方法的标准曲线。
图6:不同溶液孵育的电泳图;图中,道1marker;道2为3.6kbpdna;道3为3.6kbpdna与dcas9-biotin包被的磁性微球(不含sgrna)在本发明的清洗液中孵育2h后的上清;道4为3.6kbpdna与dcas9-biotin包被的磁性微球(不含sgrna)在pbs溶液中孵育2h后的上清。由图可以看出,本发明的清洗液液能有效减少dna的非特异性吸附。
具体实施方式
应该指出,以下详细说明都是例示性的,旨在对本申请提供进一步的说明。除非另有指明,本文使用的所有技术和科学术语具有与本申请所属技术领域的普通技术人员通常理解的相同含义。
术语说明:
室温:本文中的“室温”,表示的温度范围为15-30℃。
正如背景技术部分所介绍的,目前crispr/cas9被广泛应用基因编辑和调控,但还没有充分的探讨用于核酸检测领域。
基于此,本发明的目的在于提供一种通过crispr“夹心识别”检测核酸的方法。本发明所涉及的dcas9酶,其野生型来自化脓链球菌(streptococcuspyogene),命名为spcas9,详见参考文献:genomeengineeringusingcrispr-cas9system,congl,zhangf,methodsinmolecularbiology(clifton,n.j.)1239(2015):197。cas9蛋白源自脓链球菌(streptococcuspyogene)基因,dcas9是以cas9为模板将其核酸酶结构域进行突变(hnh结构域中h840a,ruvc结构域中d10a),使其失去切割dna的能力,但在sgrna的指导下仍能以相同的精确度靶向和结合dna。其氨基酸序列如seqidno.2所示。
将编码dcas9的基因序列优化后(优化后的基因序列如seqidno.1所示)连接到表达载体pet16b上获得重组表达质粒,并转化到大肠杆菌e.colibl21(de3)中得到表达dcas9的工程菌。工程菌进行表达所得产物为可溶性蛋白,通过超声破碎、镍离子亲和柱、疏水层析和离子交换层析进行分离纯化得到纯净的目标,即dcas9。
本发明所述的功能化dcas9包含生物素修饰的dcas9和碱性磷酸酶偶联的dcas9两种,获得的方法如下:
(1)生物素修饰的dcas9(dcas9-biotin)制备方法:
(i)将1mldcas9(1-2mg)用pbs(ph=8.0)缓冲液稀释至5ml,用50kda分子量超滤筒超滤浓缩至1ml;
(ii)重复上述步骤2次,目的为更换缓冲液,提高修饰效率;
(iii)取14-27μlez-link-nhs-biotin母液加入到dcas9蛋白溶液中,均匀混合,室温反应30-60分钟;
(iv)加入甘氨酸终止反应,以pbs(ph=8.0)缓冲液稀为流动相过除盐柱除去未反应小分子,收集修饰蛋白,超滤浓缩,得到dcas9-biotin。
(2)dcas9-碱性磷酸酶(dcas9-ap)的制备方法:
(i)dcas9200μg,补加pbs(ph=8.0)至体积为400μl,运用超滤管(50k)超滤三次,8000rpm离心5min,每次补加pbs(ph=8.0)至体积为400μl,最后浓缩成体积为100μl的dcas9溶液(浓度为2mg/ml),使dcas9的buffer更换为pbs(ph=8.0)。
(ii)取已分装好的traut’s试剂(母液浓度为10mg/ml)用pbs(ph=8.0)稀释10倍后取3.7μl(20x)加入到100μl的dcas9溶液中,涡旋振荡使其充分混匀,25℃下反应90min,期间每10min涡旋混匀一次。
(iii)除盐:以pbs(ph=8.0)缓冲液稀为流动相过除盐柱除去未反应小分子,收集修饰蛋白,超滤浓缩。最后浓缩成体积为100μl的dcas9溶液。
(iv)预处理ap:取ap600μg,补加conjugationbuffer(pbs100mmph=7.2,nacl150mm,edta1~5mm)至体积为400μl,运用超滤管(10k)超滤三次,8000rpm离心5min,每次补加conjugationbuffer至体积为400μl,最后浓缩成体积为100μl的ap溶液(浓度为6mg/ml)。
(v)取已分装好的smcc试剂(母液浓度为3.7mg/ml)10μl(20x)加入到100μl的ap溶液中,涡旋振荡使其充分混匀,25℃下反应90min,期间每10min涡旋混匀一次。
(vi)除盐:以pbs(ph=7.2)缓冲液稀为流动相过除盐柱除去未反应小分子,收集修饰蛋白,超滤浓缩。最后浓缩成体积为100μl的ap溶液。
将上述步骤(iii)制备的100μl的dcas9溶液与步骤(vi)制备的100μl的ap溶液混合,涡旋混匀,置于4℃过夜反应,第二天上午补加300μl的保存液(ph=7.2)重悬,4℃保存。
基于上述制备的生物素修饰的dcas9和碱性磷酸酶偶联的dcas9,本发明开发设计了一种由dcas9介导特异性夹心识别dna检测核酸的化学发光方法,其检测原理如图1所示。检测试剂包含r1、r2、sgrna1、sgrna2、清洗液和反应液六项,其中r1、r2分别是由dcas9-biotin修饰的磁性固相微粒和dcas9-ap偶联蛋白配置的溶液,而sgrna1、sgrna2为针对待检测目标所设计的指导rna。通过sgrna1与修饰于磁性微粒上的dcas9形成rnp复合物,对待测dna进行识别捕获,而dcas9-ap在sgrna2指导下识别结合待测dna,形成类似于免疫检测的“夹心”复合物,最终通过夹心吸附上的ap催化特异性底物amppd进行发光检测,待测dna片段与发光值成正相关。
本发明所述的由dcas9介导特异性夹心识别dna检测核酸的操作方法如下:
1)将制备的生物素修饰的dcas9(dcas9-biotin)30~100μg与1.0mg的链霉亲和素修饰磁性微球混合,在室温条件下孵育30min,洗涤,采用r1溶剂进行重悬,得到dcas9-biotin修饰的磁性固相微粒;作为r1。以碱性磷酸酶偶联的dcas9作为r2。
注:磁性微球为invitrogen出售的dynabeadstmmyonetmstreptavidinc1,配基类型为链霉亲和素,平均直径为1.0μm。
2)如有需要,可用清洗液将r1、r2稀释至合适浓度(r1:10~30μg/ml(以磁性固相微粒计)、r2:50~150μg/ml)。
3)根据待测物向r1、r2中加入对应指导rna进行孵育结合;具体为:向r1中加入sgrna1、r2中加入sgrna2,37℃孵育15min,分别得到r1&sgrna1和r2&sgrna2。
4)将待测样本与50μl的r2&sgrna2混合均匀,37℃下孵育15min,每5min涡旋振荡一次;
5)上述反应结束后,加入r1&sgrna1(磁球包被的dcas9rnp)50μl,混合均匀,37℃下孵育15min,每5min涡旋振荡一次;
6)磁性分离磁球,弃去上清;
7)用400μl清洗液重悬洗涤磁球,磁性分离磁球,弃去上清;
8)重复上步骤两次;
清洗后磁球用50μl清洗液重悬,转移至96孔板中,加入150μl反应液,立即混匀上机检测。
为进一步的提高检测效果,本发明还对r1溶剂、清洗液和反应液的组成进行了优化。其中,r1溶剂用于悬浮着dcas9-biotin修饰的磁性固相微粒,优化后的r1溶剂的组成为:添加有0.1~0.5mol/lnacl、0.1~0.01%bsa、5~100μg/ml肝素钠盐、0.01~0.05%tritonx-100、0.01~0.05%叠氮钠、0.02-0.05%proclin300、0.1~0.01%变性鲑鱼精dna的50mmtris-hcl(ph6.0~8.0)。叠氮钠、proclin300作为常用抑菌剂能有效抑菌,肝素能够竞争消除dna与dcas9(不含sgrna)的非特异结合,因此,采用优化后的r1溶剂能有效减少核酸与dcas9的非特异结合,稳定蛋白,延长试剂效期。
清洗液主要用于清洗固相微球,也可在测试时用于稀释r1、r2。优化后的清洗液的组成为:添加有0.1~0.5mol/lnacl、0.1~0.01%bsa、0.01~0.05%tritonx-100、1~5mmol/mledta、0.01~0.05%叠氮钠、0.02-0.05%proclin300、0.1~0.01%变性鲑鱼精dna的0.1mpbs(ph6.0~8.0)。经优化后的清洗液能有效减少r2、核酸在微球上的非特异性吸附,增加微球分散性,稳定蛋白,延长试剂效期(图6)。
反应液用于发光检测,反应液中含有1m二乙醇胺、5mmmgcl2、0.2mkcl和20mm底物amppd,反应液的ph为10.3。
综上,目前已有利用dcas9酶切功能失活但靶向功能保留的特点实现核酸检测的报道(专利cn107574226a)。但在核酸的实际检测的过程中,由于cas9的脱靶效应、样品成分复杂以及ru(bpy)32+强正电性都会导致核酸的非特异性结合捕获,使定量不准,容易报告假阳、阴结果。
针对以上问题,本发明引入两种dcas9功能化蛋白,在两种sgrna引导下,通过dcas9-biotin对待测核酸片段进行识别捕获,而dcas9-ap则对待测核酸片段另一处进行识别。与之相比,本发明具有如下技术优点:1.由于需要两处共40bp序列的特异性识别,该检测方法较其他专利所述的20bp片段长度的识别具有更高的特异性,能有效排除复杂样品所带来的潜在脱靶效应;2.由于通过dcas9-biotin对待测核酸进行捕获,免去对待测核酸进行生物素标记的处理,检测更便捷;3.与带强正电的ru(bpy)32+相比,碱性磷酸酶是一种反应活性高、生物相容性好、电荷分布适中的功能酶,不会与负电性较强的核酸进行非特异相互作用。同时碱性磷酸酶在各种检测体系中应用更为广泛,本发明所述的体系可在酶标仪等小型仪器上进行检测报告;4.本发明还提供了相应试剂能有效减少核酸非特异结合,稳定蛋白,延长试剂效期,增加体系分散性。利于本发明所述的检测体系在核酸检测上的实际应用。
为了使得本领域技术人员能够更加清楚地了解本申请的技术方案,以下将结合具体的实施例详细说明本申请的技术方案。如果实施例中未注明的实验具体条件,通常按照常规条件,或者按照试剂公司所推荐的条件;下述实施例中所用的试剂、耗材等,如无特殊说明,均可通过商业途径获得。
实施例1:(e.coli)功能化dcas9-biotin蛋白的制备
(1)dcas9表达载体的构建
根据cas9基因序列对cas9进行基因合成及密码子优化,并突变两个氨基酸h840a和d10a,在5’端引入ndeⅰ酶切位点(catatg),3’端引入xhoⅰ酶切位点(ctcgag)。目的基因与克隆载体puc57相连形成重组质粒puc57-dcas9。目的基因序列如seqidno.1所示。将重组质粒puc57-dcas9用ndeⅰ和xhoⅰ双酶切,与经过ndeⅰ和xhoⅰ双酶切的载体pet16b的骨架片段进行16℃过夜连接,获得重组载体pet16b-dcas9,测序验证。
酶切体系如下(20μl):
连接体系如下(20μl):
(2)dcas9的表达、纯化
将pet16b-dcas9表达载体转化进入e.coilbl21(ed3)感受态细胞中,操作如下:将-80℃冰箱保存的感受态细胞置于冰中融化5min。加入表达载体pet16b-dcas92μl,轻轻混匀后冰上放置30min。42℃水浴锅中放置90s后,立即于冰中放置2min。每管加入800μl预温的lb培养基,37℃培养45min(160r/min)。4000rpm离心1min后,取沉淀涂布平板,将平板倒置于37℃恒温培养箱中培养过夜,培养形成单一菌落。
挑取上步所得单一菌落至含有100μg/ml氨苄的lb培养基中(20ml),37℃、180r/min振荡培养过夜,获得种子液。将种子液以1%(v/v)的比例接种量接种至2l含有100μg/ml氨苄的lb培养基中,37℃、180r/min振荡培养至od600=0.6-0.8,然后加入iptg至终浓度为0.2mmol/l,18℃、180r/min继续振荡培养18h,离心收集菌体-80摄氏度冻存。
菌体中纯化dcas9方法如下:配置缓冲液a(ph7.5tris-hcl)的配方:取24.2gtris-base置于1l烧杯中,加入约800ml去离子水充分搅拌溶解,用浓hcl调ph至7.5,将溶液用容量瓶定容至1l,4℃保存。使用时稀释10倍。缓冲液b(ph8.0tris-hcl)的配方:取24.2gtris-base置于1l烧杯中,加入约800ml去离子水充分搅拌溶解,用浓hcl调ph至8.0,将溶液用容量瓶定容至1l,4℃保存。使用时稀释10倍。
取冻存工程菌重悬于缓冲液a中,浓度为1g/ml,置于冰上超声破碎20min(分2次),当菌液由粘稠状态变为透亮即可,4℃、12000r/min离心30min,收集上清蛋白液,对上清液中可溶的dcas9进行ni柱亲和纯化,所得洗脱液加入氯化钠至终浓度1m,经phenylhp疏水层析纯化,用含有氯化钠浓度为1.5m-0m的缓冲液a线性洗脱,page电泳验证,收集并合并目的蛋白,将收集的蛋白液用缓冲液b透析过夜除盐;将透析后的蛋白液进行highs阳离子交换层析,用含nacl浓度为0m-0.5m的缓冲液b线性洗脱,page电泳验证,收集并合并目的蛋白,将收集的蛋白液用缓冲液b透析过夜除盐;将透析后的蛋白液再次进行highs阳离子交换层析,用含nacl浓度为0.1m-1m的缓冲液b线性洗脱,收集并合并目的蛋白,用缓冲液b透析除盐后,即得到本发明所述的dcas9蛋白,其sds-page电泳结果如图2所示,其氨基酸序列如seqidno.2所示。
(3)dcas9-biotin、dcas9-ap的制备
将收集到的酶蛋白液用100mmpbs(ph8.0)超滤浓缩处理,使蛋白终浓度大于2mg/ml,取14-27μlez-link-nhs-biotin母液加入到dcas9蛋白溶液中,均匀混合,室温反应30-60分钟,加入甘氨酸终止反应,以pbs(ph=8.0)缓冲液稀为流动相过除盐柱除去未反应小分子,收集修饰蛋白,超滤浓缩,得到dcas-biotin,其sds-page电泳结果如图3中泳道1所示。
同样的,将收集到的酶蛋白液用100mm、ph8.0的pbs超滤浓缩处理,使蛋白终浓度大于2mg/ml,取trauts试剂(10mg/ml)用pbs(ph=8.0)稀释10倍后取3.7μl(20x)加入到100μl的dcas9溶液中,涡旋振荡使其充分混匀,25℃下反应90min,期间每10min涡旋混匀一次。超滤除盐,用smcc预处理待连接的ap,最后浓缩成体积为100μl的ap溶液(浓度为6mg/ml)。将上述100μl的dcas9溶液与100μl的ap溶液混合,涡旋混匀,置于4℃反应16h,获得dcas9-ap,其sds-page电泳结果如图3中泳道3所示。
实施例2:功能化dcas9的特征
功能化dcas9(dcas9-biotin和dcas9-ap)可在rna指导下与序列特异性双链dna片段结合。本发明通过设计合成指导rna(sgrna1、sgrna2,其rna序列如seqidno.3、seqidno.4所示),可以指导dcas9与dna1(dna1序列如seqidno.5所示)特异性结合,我们用凝胶电泳验证了sgrna1能够指导功能化dcas9蛋白与dna1结合,由于功能化dcas9在sgrna指导下可与dna1特异性结合形成复合物,非变性电泳下其迁移率要小于游离的dna1,表现为dcas9-sgrna-dna1条带比未形成复合物的dna1条带靠上。
凝胶阻滞电泳方法如下
1)5%非变性page胶的配置
2)电泳条件:0.5xtbe缓冲液作为电泳液,4℃,120v,1h。
凝胶阻滞电泳结果如图4所示。
实施例3:应用dcas9功能蛋白夹心识别检测核酸(化学发光方法)
1)配置清洗液、反应液:清洗液为0.1mpbs(ph6.0~8.0)含nacl(0.1~0.5mol/l)、bsa(0.1~0.01%)、tritonx-100(0.01~0.05%)、edta(1~5mmol/ml)还含有叠氮钠(0.01~0.05%)、proclin300(0.02-0.05%)、变性鲑鱼精dna(0.1~0.01%);反应液中含有1m二乙醇胺、5mmmgcl2、0.2mkcl和20mm底物amppd,反应液的ph为10.3。
2)配置dna标准品:s0:0ng/ml,s1:11.25ng/ml,s2:22.5ng/ml,s3:45ng/ml,s4:90ng/ml。dna标准品的核苷酸序列如seqidno.5所示,长度为3.6kbp。上述配制的dna标准品换算成摩尔浓度分别为:0pm、4.725pm、9.45pm、18.9pm和37.8pm。
3)将dcas9-biotin修饰的磁性固相微粒(mbs-dcas9)用清洗液稀释到10~30μg/ml,将dcas9-ap用清洗液稀释到50~150μg/ml,以蛋白含量计。
检测操作方法如下:向mbs-dcas9加入sgrna1,向dcas9-ap中加入sgrna2,37℃孵育15min;将dna标准品(s0~s4)50μl分别与50μl的dcas9-aprnp混合均匀,37℃下孵育15min,每5min涡旋振荡一次;上述反应结束后,加入mbs-dcas9rnp50μl,混合均匀,37℃下孵育15min,每5min涡旋振荡一次;磁性分离磁球,弃去上清;用400μl清洗液重悬洗涤磁球,磁性分离磁球,弃去上清;重复洗涤步骤两次;清洗后磁球用50μl清洗液重悬,转移至96孔板中,加入150μl反应液立即混匀上机检测。dcas9-biotin&sgrna1和dcas9-ap&sgrna2与dna形成夹心复合物,使得dcas9-ap被捕获到dcas9-biotin修饰的磁性微球(mbs)表面,dcas9-ap催化发光底物amppd发光,其发光值与与dna成正比,此化学发光方法的标准曲线如图5所示。
经测算,采用本发明的方法可实现体系中0.42fmol核酸(3.6kbp)的检出。
以上所述仅为本申请的优选实施例而已,并不用于限制本申请,对于本领域的技术人员来说,本申请可以有各种更改和变化。凡在本申请的精神和原则之内,所作的任何修改、等同替换、改进等,均应包含在本申请的保护范围之内。
sequencelisting
<110>济南国科医工科技发展有限公司
<120>功能化的dcas9修饰蛋白及其在检测核酸中的应用
<130>2020
<160>5
<170>patentinversion3.5
<210>1
<211>4124
<212>dna
<213>人工序列
<400>1
atgcatcatcatcatcatcacgtggataagaaatactcaataggcttagctatcggcaca60
aatagcgtcggatgggcggtgatcactgatgaatataaggttccgtctaaaaagttcaag120
gttctgggaaatacagaccgccacagtatcaaaaaaaatcttataggggctcttttattt180
gacagtggagagacagcggaagcgactcgtctcaaacggacagctcgtagaaggtataca240
cgtcggaagaatcgtatttgttatctacaggagattttttcaaatgagatggcgaaagta300
gatgatagtttctttcatcgacttgaagagtcttttttggtggaagaagacaagaagcat360
gaacgtcatcctatttttggaaatatagtagatgaagttgcttatcatgagaaatatcca420
actatctatcatctgcgaaaaaaattggtagattctactgataaagcggatttgcgctta480
atctatttggccttagcgcatatgattaagtttcgtggtcattttttgattgagggagat540
ttaaatcctgataatagtgatgtggacaaactatttatccagttggtacaaacctacaat600
caattatttgaagaaaaccctattaacgcaagtggagtagatgctaaagcgattctttct660
gcacgattgagtaaatcaagacgattagaaaatctcattgctcagctccccggtgagaag720
aaaaatggcttatttgggaatctcattgctttgtcattgggtttgacccctaattttaaa780
tcaaattttgatttggcagaagatgctaaattacagctttcaaaagatacttacgatgat840
gatttagataatttattggcgcaaattggagatcaatatgctgatttgtttttggcagct900
aagaatttatcagatgctattttactttcagatatcctaagagtaaatactgaaataact960
aaggctcccctatcagcttcaatgattaaacgctacgatgaacatcatcaagacttgact1020
cttttaaaagctttagttcgacaacaacttccagaaaagtataaagaaatcttttttgat1080
caatcaaaaaacggatatgcaggttatattgatgggggagctagccaagaagaattttat1140
aaatttatcaaaccaattttagaaaaaatggatggtactgaggaattattggtgaaacta1200
aatcgtgaagatttgctgcgcaagcaacggacctttgacaacggctctattccccatcaa1260
attcacttgggtgagctgcatgctattttgagaagacaagaagacttttatccattttta1320
aaagacaatcgtgagaagattgaaaaaatcttgacttttcgaattccttattatgttggt1380
ccattggcgcgtggcaatagtcgttttgcatggatgactcggaagtctgaagaaacaatt1440
accccatggaattttgaagaagttgtcgataaaggtgcttcagctcaatcatttattgaa1500
cgcatgacaaactttgataaaaatcttccaaatgaaaaagtactaccaaaacatagtttg1560
ctttatgagtattttacggtttataacgaattgacaaaggtcaaatatgttactgaagga1620
atgcgaaaaccagcatttctttcaggtgaacagaagaaagccattgttgatttactcttc1680
aaaacaaatcgaaaagtaaccgttaagcaattaaaagaagattatttcaaaaaaatagaa1740
tgttttgatagtgttgaaatttcaggagttgaagatagatttaatgcttcattaggtacc1800
taccatgatttgctaaaaattattaaagataaagattttttggataatgaagaaaatgaa1860
gatatcttagaggatattgttttaacattgaccttatttgaagatagggagatgattgag1920
gaaagacttaaaacatatgctcacctctttgatgataaggtgatgaaacagcttaaacgt1980
cgccgttatactggttggggacgtttgtctcgaaaattgattaatggtattagggataag2040
caatctggcaaaacaatattagattttttgaaatcagatggttttgccaatcgcaatttt2100
atgcagctgatccatgatgatagtttgacatttaaagaagacattcaaaaagcacaagtg2160
tctggacaaggcgatagtttacatgaacatattgcaaatttagctggtagccctgctatt2220
aaaaaaggtattttacagactgtaaaagttgttgatgaattggtcaaagtaatggggcgg2280
cataagccagaaaatatcgttattgaaatggcacgtgaaaatcagacaactcaaaagggc2340
cagaaaaattcgcgagagcgtatgaaacgaatcgaagaaggtatcaaagaattaggaagt2400
cagattcttaaagagcatcctgttgaaaatactcaattgcaaaatgaaaagctctatctc2460
tattatctccaaaatggaagagacatgtatgtggaccaagaattagatattaatcgttta2520
agtgattatgatgtcgatgccattgttccacaaagtttccttaaagacgattcaatagac2580
aataaggtcttaacgcgttctgataaaaatcgtggtaaatcggataacgttccaagtgaa2640
gaagtagtcaaaaagatgaaaaactattggagacaacttctaaacgccaagttaatcact2700
caacgtaagtttgataatttaacgaaagctgaacgtggaggtttgagtgaacttgataaa2760
gctggttttatcaaacgccaattggttgaaactcgccaaatcactaagcatgtggcacaa2820
attttggatagtcgcatgaatactaaatacgatgaaaatgataaacttattcgagaggtt2880
aaagtgattaccttaaaatctaaattagtttctgacttccgaaaagatttccaattctat2940
aaagtacgtgagattaacaattaccatcatgcccatgatgcgtatctaaatgccgtcgtt3000
ggaactgctttgattaagaaatatccaaaacttgaatcggagtttgtctatggtgattat3060
aaagtttatgatgttcgtaaaatgattgctaagtctgagcaagaaataggcaaagcaacc3120
gcaaaatatttcttttactctaatatcatgaacttcttcaaaacagaaattacacttgca3180
aatggagagattcgcaaacgccctctaatcgaaactaatggggaaactggagaaattgtc3240
tgggataaagggcgagattttgccacagtgcgcaaagtattgtccatgccccaagtcaat3300
attgtcaagaaaacagaagtacagacaggcggattctccaaggagtcaattttaccaaaa3360
agaaattcggacaagcttattgctcgtaaaaaagactgggatccaaaaaaatatggtggt3420
tttgatagtccaacggtagcttattcagtcctagtggttgctaaggtggaaaaagggaaa3480
tcgaagaagttaaaatccgttaaagagttactagggatcacaattatggaaagaagttcc3540
tttgaaaaaaatccgattgactttttagaagctaaaggatataaggaagttaaaaaagac3600
ttaatcattaaactacctaaatatagtctttttgagttagaaaacggtcgtaaacggatg3660
ctggctagtgccggagaattacaaaaaggaaatgagctggctctgccaagcaaatatgtg3720
aattttttatatttagctagtcattatgaaaagttgaagggtagtccagaagataacgaa3780
caaaaacaattgtttgtggagcagcataagcattatttagatgagattattgagcaaatc3840
agtgaattttctaagcgtgttattttagcagatgccaatttagataaagttcttagtgca3900
tataacaaacatagagacaaaccaatacgtgaacaagcagaaaatattattcatttattt3960
acgttgacgaatcttggagctcccgctgcttttaaatattttgatacaacaattgatcgt4020
aaacgatatacgtctacaaaagaagttttagatgccactcttatccatcaatccatcact4080
ggtctttatgaaacacgcattgatttgagtcagctaggaggtga4124
<210>2
<211>1375
<212>prt
<213>人工序列
<400>2
methishishishishishisvalasplyslystyrserileglyleu
151015
alaileglythrasnservalglytrpalavalilethraspglutyr
202530
lysvalproserlyslysphelysvalleuglyasnthrasparghis
354045
serilelyslysasnleuileglyalaleuleupheaspserglyglu
505560
thralaglualathrargleulysargthralaargargargtyrthr
65707580
argarglysasnargilecystyrleuglngluilepheserasnglu
859095
metalalysvalaspaspserphephehisargleuglugluserphe
100105110
leuvalglugluasplyslyshisgluarghisproilepheglyasn
115120125
ilevalaspgluvalalatyrhisglulystyrprothriletyrhis
130135140
leuarglyslysleuvalaspserthrasplysalaaspleuargleu
145150155160
iletyrleualaleualahismetilelyspheargglyhispheleu
165170175
ilegluglyaspleuasnproaspasnseraspvalasplysleuphe
180185190
ileglnleuvalglnthrtyrasnglnleupheglugluasnproile
195200205
asnalaserglyvalaspalalysalaileleuseralaargleuser
210215220
lysserargargleugluasnleuilealaglnleuproglyglulys
225230235240
lysasnglyleupheglyasnleuilealaleuserleuglyleuthr
245250255
proasnphelysserasnpheaspleualagluaspalalysleugln
260265270
leuserlysaspthrtyraspaspaspleuaspasnleuleualagln
275280285
ileglyaspglntyralaaspleupheleualaalalysasnleuser
290295300
aspalaileleuleuseraspileleuargvalasnthrgluilethr
305310315320
lysalaproleuseralasermetilelysargtyraspgluhishis
325330335
glnaspleuthrleuleulysalaleuvalargglnglnleuproglu
340345350
lystyrlysgluilephepheaspglnserlysasnglytyralagly
355360365
tyrileaspglyglyalaserglnglugluphetyrlyspheilelys
370375380
proileleuglulysmetaspglythrglugluleuleuvallysleu
385390395400
asnarggluaspleuleuarglysglnargthrpheaspasnglyser
405410415
ileprohisglnilehisleuglygluleuhisalaileleuargarg
420425430
glngluaspphetyrpropheleulysaspasnargglulysileglu
435440445
lysileleuthrpheargileprotyrtyrvalglyproleualaarg
450455460
glyasnserargphealatrpmetthrarglysserglugluthrile
465470475480
thrprotrpasnpheglugluvalvalasplysglyalaseralagln
485490495
serpheilegluargmetthrasnpheasplysasnleuproasnglu
500505510
lysvalleuprolyshisserleuleutyrglutyrphethrvaltyr
515520525
asngluleuthrlysvallystyrvalthrgluglymetarglyspro
530535540
alapheleuserglygluglnlyslysalailevalaspleuleuphe
545550555560
lysthrasnarglysvalthrvallysglnleulysgluasptyrphe
565570575
lyslysileglucyspheaspservalgluileserglyvalgluasp
580585590
argpheasnalaserleuglythrtyrhisaspleuleulysileile
595600605
lysasplysasppheleuaspasnglugluasngluaspileleuglu
610615620
aspilevalleuthrleuthrleuphegluaspargglumetileglu
625630635640
gluargleulysthrtyralahisleupheaspasplysvalmetlys
645650655
glnleulysargargargtyrthrglytrpglyargleuserarglys
660665670
leuileasnglyileargasplysglnserglylysthrileleuasp
675680685
pheleulysseraspglyphealaasnargasnphemetglnleuile
690695700
hisaspaspserleuthrphelysgluaspileglnlysalaglnval
705710715720
serglyglnglyaspserleuhisgluhisilealaasnleualagly
725730735
serproalailelyslysglyileleuglnthrvallysvalvalasp
740745750
gluleuvallysvalmetglyarghislysprogluasnilevalile
755760765
glumetalaarggluasnglnthrthrglnlysglyglnlysasnser
770775780
arggluargmetlysargileglugluglyilelysgluleuglyser
785790795800
glnileleulysgluhisprovalgluasnthrglnleuglnasnglu
805810815
lysleutyrleutyrtyrleuglnasnglyargaspmettyrvalasp
820825830
glngluleuaspileasnargleuserasptyraspvalaspalaile
835840845
valproglnserpheleulysaspaspserileaspasnlysvalleu
850855860
thrargserasplysasnargglylysseraspasnvalproserglu
865870875880
gluvalvallyslysmetlysasntyrtrpargglnleuleuasnala
885890895
lysleuilethrglnarglyspheaspasnleuthrlysalagluarg
900905910
glyglyleusergluleuasplysalaglypheilelysargglnleu
915920925
valgluthrargglnilethrlyshisvalalaglnileleuaspser
930935940
argmetasnthrlystyraspgluasnasplysleuilearggluval
945950955960
lysvalilethrleulysserlysleuvalseraspphearglysasp
965970975
pheglnphetyrlysvalarggluileasnasntyrhishisalahis
980985990
aspalatyrleuasnalavalvalglythralaleuilelyslystyr
99510001005
prolysleuglusergluphevaltyrglyasptyrlysvaltyr
101010151020
aspvalarglysmetilealalyssergluglngluileglylys
102510301035
alathralalystyrphephetyrserasnilemetasnphephe
104010451050
lysthrgluilethrleualaasnglygluilearglysargpro
105510601065
leuilegluthrasnglygluthrglygluilevaltrpasplys
107010751080
glyargaspphealathrvalarglysvalleusermetprogln
108510901095
valasnilevallyslysthrgluvalglnthrglyglypheser
110011051110
lysgluserileleuprolysargasnserasplysleuileala
111511201125
arglyslysasptrpaspprolyslystyrglyglypheaspser
113011351140
prothrvalalatyrservalleuvalvalalalysvalglulys
114511501155
glylysserlyslysleulysservallysgluleuleuglyile
116011651170
thrilemetgluargserserpheglulysasnproileaspphe
117511801185
leuglualalysglytyrlysgluvallyslysaspleuileile
119011951200
lysleuprolystyrserleuphegluleugluasnglyarglys
120512101215
argmetleualaseralaglygluleuglnlysglyasngluleu
122012251230
alaleuproserlystyrvalasnpheleutyrleualaserhis
123512401245
tyrglulysleulysglyserprogluaspasngluglnlysgln
125012551260
leuphevalgluglnhislyshistyrleuaspgluileileglu
126512701275
glnileserglupheserlysargvalileleualaaspalaasn
128012851290
leuasplysvalleuseralatyrasnlyshisargasplyspro
129513001305
ilearggluglnalagluasnileilehisleuphethrleuthr
131013151320
asnleuglyalaproalaalaphelystyrpheaspthrthrile
132513301335
asparglysargtyrthrserthrlysgluvalleuaspalathr
134013451350
leuilehisglnserilethrglyleutyrgluthrargileasp
135513601365
leuserglnleuglyglyasp
13701375
<210>3
<211>135
<212>rna
<213>sgrna1
<400>3
ggauccuaauacgacucacuauagguguccgggcuuuugucacguuuaagagcuaugcug60
gaaacagcauagcaaguuuaaauaaggcuaguccguuaucaacuugaaaaaguggcaccg120
agucggugcuuuuuu135
<210>4
<211>135
<212>rna
<213>sgrna2
<400>4
ggauccuaauacgacucacuauagcuggcgugggcgugaucagguuuaagagcuaugcug60
gaaacagcauagcaaguuuaaauaaggcuaguccguuaucaacuugaaaaaguggcaccg120
agucggugcuuuuuu135
<210>5
<211>3627
<212>dna
<213>3.6kbpdna切割底物序列
<400>5
tccgtaagatgcttttctgtgactggtgagtactcaaccaagtcattctgagaatagtgt60
atgcggcgaccgagttgctcttgcccggcgtcaacacgggataataccgcgccacatagc120
agaactttaaaagtgctcatcattggaaaacgttcttcggggcgaaaactctcaaggatc180
ttaccgctgttgagatccagttcgatgtaacccactcgtgcacccaactgatcttcagca240
tcttttactttcaccagcgtttctgggtgagcaaaaacaggaaggcaaaatgccgcaaaa300
aagggaataagggcgacacggaaatgttgaatactcatactcttcctttttcaatattat360
tgaagcatttatcagggttattgtctcatgagcggatacatatttgaatgtatttagaaa420
aataaacaaataggggttccgcgcacatttccccgaaaagtgccacctgacgtctaagaa480
accattattatcatgacattaacctataaaaataggcgtatcacgaggccctttcgtctt540
caagaattctcatgtttgacagcttatcatcgataagctttaatgcggtagtttatcaca600
gttaaattgctaacgcagtcaggcaccgtgtatgaaatctaacaatgcgctcatcgtcat660
cctcggcaccgtcaccctggatgctgtaggcataggcttggttatgccggtactgccggg720
cctcttgcgggatatccggatatagttcctcctttcagcaaaaaacccctcaagacccgt780
ttagaggccccaaggggttatgctagttattgctcagcggtggcagcagccaactcagct840
tcctttcgggctttgttagcagccggatgaaacaaagcactattgcactggcactcttac900
cgttactgtttacccctgtgacaaaagcccggacaccagaaatgcctgttctggaaaacc960
gggctgctcagggcgatattactgcacccggcggtgctcgccgtttaacgggtgatcaga1020
ctgccgctctgcgtgattctcttagcgataaacctgcaaaaaatattattttgctgattg1080
gcgatgggatgggggactcggaaattactgccgcacgtaattatgccgaaggtgcgggcg1140
gcttttttaaaggtatagatgccttaccgcttaccgggcaatacactcactatgcgctga1200
ataaaaaaaccggcaaaccggactacgtcaccgactcggctgcatcagcaaccgcctggt1260
caaccggtgtcaaaacctataacggcgcgctgggcgtcgatattcacgaaaaagatcacc1320
caacgattctggaaatggcaaaagccgcaggtctggcgaccggtaacgtttctaccgcag1380
agttgcaggatgccacgcccgctgcgctggtggcacatgtgacctcgcgcaaatgctacg1440
gtccgagcgcgaccagtgaaaaatgtccgggtaacgctctggaaaaaggcggaaaaggat1500
cgattaccgaacagctgcttaacgctcgtgccgacgttacgcttggcggcggcgcaaaaa1560
cctttgctgaaacggcaaccgctggtgaatggcagggaaaaacgctgcgtgaacaggcac1620
aggcgcgtggttatcagttggtgagcgatgctgcctcactgaattcggtgacggaagcga1680
atcagcaaaaacccctgcttggcctgtttgctgacggcaatatgccagtgcgctggctag1740
gaccgaaagcaacgtaccatggcaatatcgataagcccgcagtcacctgtacgccaaatc1800
cgcaacgtaatgacagtgtaccaaccctggcgcagatgaccgacaaagccattgaattgt1860
tgagtaaaaatgagaaaggctttttcctgcaagttgaaggtgcgtcaatcgataaacagg1920
atcatgctgcgaatccttgtgggcaaattggcgagacggtcgatctcgatgaagccgtac1980
aacgggcgctggaattcgctaaaaaggagggtaacacgctggtcatagtcaccgctgatc2040
acgcccacgccagccagattgttgcgccggataccaaagctccgggcctcacccaggcgc2100
taaataccaaagatggcgcagtgatggtgatgagttacgggaactccgaagaggattcac2160
aagaacataccggcagtcagttgcgtattgcggcgtatggcccgcatgccgccaatgttg2220
ttggactgaccgaccagaccgatctcttctacaccatgaaagccgctctggggctgaaat2280
aattcgatatggccgctgctgtgatgatgatgatgatgatgatgatgatggcccatggta2340
tatctccttcttaaagttaaacaaaattatttctagaggggaattgttatccgctcacaa2400
ttcccctatagtgagtcgtattaatttcgcgggatcgagatctcgatcctctacgccgga2460
cgcatcgtggccggcatcaccggcgccacaggtgcggttgctggcgcctatatcgccgac2520
atcaccgatggggaagatcgggctcgccacttcgggctcatgagcgcttgtttcggcgtg2580
ggtatggtggcaggccccgtggccgggggactgttgggcgccatctccttgcatgcacca2640
ttccttgcggcggcggtgctcaacggcctcaacctactactgggctgcttcctaatgcag2700
gagtcgcataagggagagcgtcgagatcccggacaccatcgaatggcgcaaaacctttcg2760
cggtatggcatgatagcgcccggaagagagtcaattcagggtggtgaatgtgaaaccagt2820
aacgttatacgatgtcgcagagtatgccggtgtctcttatcagaccgtttcccgcgtggt2880
gaaccaggccagccacgtttctgcgaaaacgcgggaaaaagtggaagcggcgatggcgga2940
gctgaattacattcccaaccgcgtggcacaacaactggcgggcaaacagtcgttgctgat3000
tggcgttgccacctccagtctggccctgcacgcgccgtcgcaaattgtcgcggcgattaa3060
atctcgcgccgatcaactgggtgccagcgtggtggtgtcgatggtagaacgaagcggcgt3120
cgaagcctgtaaagcggcggtgcacaatcttctcgcgcaacgcgtcagtgggctgatcat3180
taactatccgctggatgaccaggatgccattgctgtggaagctgcctgcactaatgttcc3240
ggcgttatttcttgatgtctctgaccagacacccatcaacagtattattttctcccatga3300
agacggtacgcgactgggcgtggagcatctggtcgcattgggtcaccagcaaatcgcgct3360
gttagcgggcccattaagttctgtctcggcgcgtctgcgtctggctggctggcataaata3420
tctcactcgcaatcaaattcagccgatagcggaacgggaaggcgactggagtgccatgtc3480
cggttttcaacaaaccatgcaaatgctgaatgagggcatcgttcccactgcgatgctggt3540
tgccaacgatcagatggcgctgggcgcaatgcgcgccattaccgagtccgggctgcgcgt3600
tggtgcggatatctcggtagtgggata3627
- 还没有人留言评论。精彩留言会获得点赞!