用于同时检测多种基因突变类型的探针组、基因芯片及其应用的制作方法
本发明属于基因检测技术领域,更具体而言本发明涉及用于同时检测多种基因突变类型的探针组及其应用。
背景技术:
根据世界卫生组织(who)统计数据,全世界约有60%的人死于癌症、糖尿病、心血管疾病、慢性呼吸系统疾病这4大类疾病,而癌症则是最主要的死因之一。2015年中国恶性肿瘤发病约392.9万人,死亡约233.8万人。平均每天超过1万人被确诊为癌症,每分钟有7.5个人被确诊为癌症。且近十几年来恶性肿瘤的发病死亡均呈持续上升态势,每年恶性肿瘤所致的医疗花费超过2200亿(chen,w.,etal.,cancerstatisticsinchina,2015.cacancerjclin,2016.66(2):p.115-32)。
肿瘤的发生和发展是一个多因素、多步骤、多基因参与的复杂过程。肿瘤基因学研究揭示了近140个驱动基因,常见基因有:egfr、kras、eml4-alk、braf、pi3kca、akt1、mek1、met、her2、ret、nras和ntrk1等,这些基因分布于12条信号通路,分别调节细胞增殖、细胞凋亡以及细胞分化等(vogelstein,b.,etal.,cancergenomelandscapes.science,2013.339(6127):p.1546-58)。
2015年年初,美国总统奥巴马在国情咨文演讲中正式启动了“精准医疗计划”,当前的肿瘤治疗正逐渐从对“症”治疗向对“基因”治疗转变,通过对相关驱动基因的突变检测,针对性选择合适的靶向药物,从而实现个体化医疗,使患者达到最佳的治疗效果。截止2016年6月,美国食品药品监督管理局(fda,u.s.foodanddrugadministration)批准了72个靶向抗肿瘤药物。然而针对肿瘤驱动基因的检测却并非易事。一方面,分子靶点涉及的类型复杂,包括点突变、短片断插入缺失、拷贝数变异和融合基因等,相关靶点的突变类型的漏检将对患者靶向用药指导大打折扣。另一方面,晚期肿瘤患者的组织样本往往获取困难,而ctdna(cell-freecirculatingtumordna)检测虽然无创,但由于存在大量正常组织游离dna,使得ctdna含量通常在1%~0.01%超低频之间,精准检测难度较大。目前对于驱动基因的检测,最常用的技术方法是rt-pcr技术,该方法虽简单快捷,但存在只能检测已知的突变位点、检测的突变类型不全、灵敏度低(1%左右)等局限性。目前行业公认的基因突变检测标准是sanger测序法,这类技术操作复杂、通量较低、灵敏度低(10%)、具有较高的漏检风险,难以满足大量肿瘤患者的检测需求。
因此,本领域需要一种全面、高灵敏度、高通量、并且针对多种突变类型同时精准检测的新技术,以有效补充和完善现有的肿瘤个体化诊疗。
技术实现要素:
如上所述,本领域需要一种全面、高灵敏度、高通量、并且针对多种突变类型同时精准检测的新技术。
因此,在第一方面,本发明提供了用于同时检测多种基因突变类型的探针组,所述探针组包括针对靶向用药相关基因的探针,其中所述靶向用药相关基因包括egfr、braf、kras、her2、met、alk、ret、ros1、ntrk1、ntrk2、ntrk3,其上的基因突变包括:
1)对于egfr基因:g719c、g719s、g719a、a698t、v765m、f712s、l703p、a702s、g724s、e709、e709g、e709k、s720f、e709v、s752_i759del、l747_s752del、l747_t751del、e746_a750del、e746_a750del、e746_s752>d、l747_e749del、l747_t751>s、e746_e749del、k745_e749del、l747_t751del、s752_i759del、t751_i759del、l747_p753>s、e746_s752del、e746_t751del、e746_t751>a、e746_t751>v、e746_s752>v、l747_t751>p、l747_a750>p、l747_p753>s、l747_t751del、e746_s752>a、k745_e746ins、c797s、v769l、s768i、t790m、r776c、v765m、s784p、g796a、v786m、a767v、c775y、f795s、r776h、g796s、d770n、h773r、g796d、l792p、s784f、g779f、v774m、h773l、d770>gy、d770_n771insg、h773_v774insh、l858r、l861q、h870y、h870r、e868g、a871t、g874s、l858l、g863d、g873e、a864t、a871g、a859t、l858r、l861r、l858m;
2)对于braf基因:a598_t599insv、a598v、d594g、d594n、d594v、e586e、e586k、f583f、f595l、f595s、g596d、g596r、g606e、h608r、i582m、i592m、i592v、k601e、k601n、l584f、l584l、l597l、l597q、l597r、l597s、l597v、n581s、p213s、s605f、s605g、s605n、t599_v600inst、t599_v600instt、t599i、v600_k601>e、v600a、v600d、v600e、v600g、v600k、v600l、v600m、v600q、v600r、w604g;
3)对于kras基因:g10_a11insg、g12g13insg、l19f、g13v、v14i、g12d、l19f、g13_v14insg、g12y、g12i、v8v、a11p、a11v、g12f、g12l、g12v、g12c、g12s、g12r、g12e、g12v、g12d、g12a、g12g、g13c、g13s、g13r、g13d、g13a、g13v、g13g、g13g、g15s、a18d、q22k、g13e、a11_g12insga、a59g、a59t、a59e、q61k、q61e、q61p、q61r、q61l、q61h、t58i、a146t、a146v、a146p、k117n;
4)对于her2基因:d769h、l755s、i767m、l755p、g776>vc、v777l、g776v、g778_s779insg、v777a、v842i、h878y;
5)对于met基因:e168d、t1010i、h1112y、h1124d、h1112l、l1130l、n1118y、h1112r、m1268t、y1248c、y1253d、14号外显子跳跃突变;
6)对于alk基因:eml4-alk;
7)对于ret基因:ncoa4-ret、prkar1a-ret、pcm1-ret、ktn1-ret、kif5b-ret、hook3-ret、golga5-ret、ccdc6-ret;
8)对于ros1基因:tpm3-ros1、slc34a2-ros1、sdc4-ros1、lrig3-ros1、gopc-ros1、ezr-ros1、cd74-ros1;
9)对于ntrk1/2/3基因:tpm3-ntrk1、lmna-ntrk1、nacc2-ntrk2、qki-ntrk2、etv6-ntrk3。
在第二方面,本发明提供了一种用于同时检测多种基因突变类型的基因芯片,所述基因芯片包括本发明第一方面的探针组。
在第三方面,本发明提供了一种用于检测多种基因突变类型的方法,所述方法包括以下步骤:
1)探针设计:设计并合成本发明第一方面的探针组;
2)dna提取和打断:提取待检测样本的dna并将其打断为200-250bp的dna片段;
3)文库构建:利用打断的dna构建样本文库;
4)杂交捕获:将步骤3)的样本文库与步骤1)的探针组相接触以进行杂交捕获;
5)上机测序:对步骤4)得到的捕获序列进行测序以产生测序数据;
6)信息分析:对步骤5)得到的测序数据进行基因突变信息分析和注释。
在第四方面,本发明提供了一种用于同时检测多种基因突变类型的试剂盒,所述试剂盒包括本发明第一方面的探针组或本发明第二方面的基因芯片。
在第五方面,本发明提供了本发明第一方面的探针组或本发明第二方面的基因芯片在制备用于同时检测一个或者多个基因突变类型的检测试剂中的用途。
本发明的有益效果是:
(1)本发明可以实现一次性同时检测包括egfr、kras、braf、c-met、her2、alk、ros1、ret和ntrk1/2/3等102种肿瘤驱动基因的基因突变,包括点突变、短片段插入缺失、拷贝数变异和基因融合;
(2)本发明可以检测探针覆盖区域的已知突变,同时还可以发现新的未知变异;
(3)本发明可以一次性检测相关稀有热点变异——met基因的14号外显子跳跃突变、bcl2l11(bim)2号内含子缺失多态性相关区域以及ntrk1/2/3热点融合突变;
(4)针对6个特定融合断点区域的基因-alk、ros1、ret、ntrk1、ntrk2、ntrk3,本发明对其进行了三倍高探针丰度覆盖,因此可以更灵敏地(0.5%)检出其已知热点融合和未知融合变异;
(5)本发明适用于石蜡包埋组织或石蜡切片,新鲜病理组织,血浆、胸水、腹水、脑脊液等多种样本类型;
(6)本发明提供一种可以同时提供靶向用药和化疗用药指导的检测方法。
附图说明
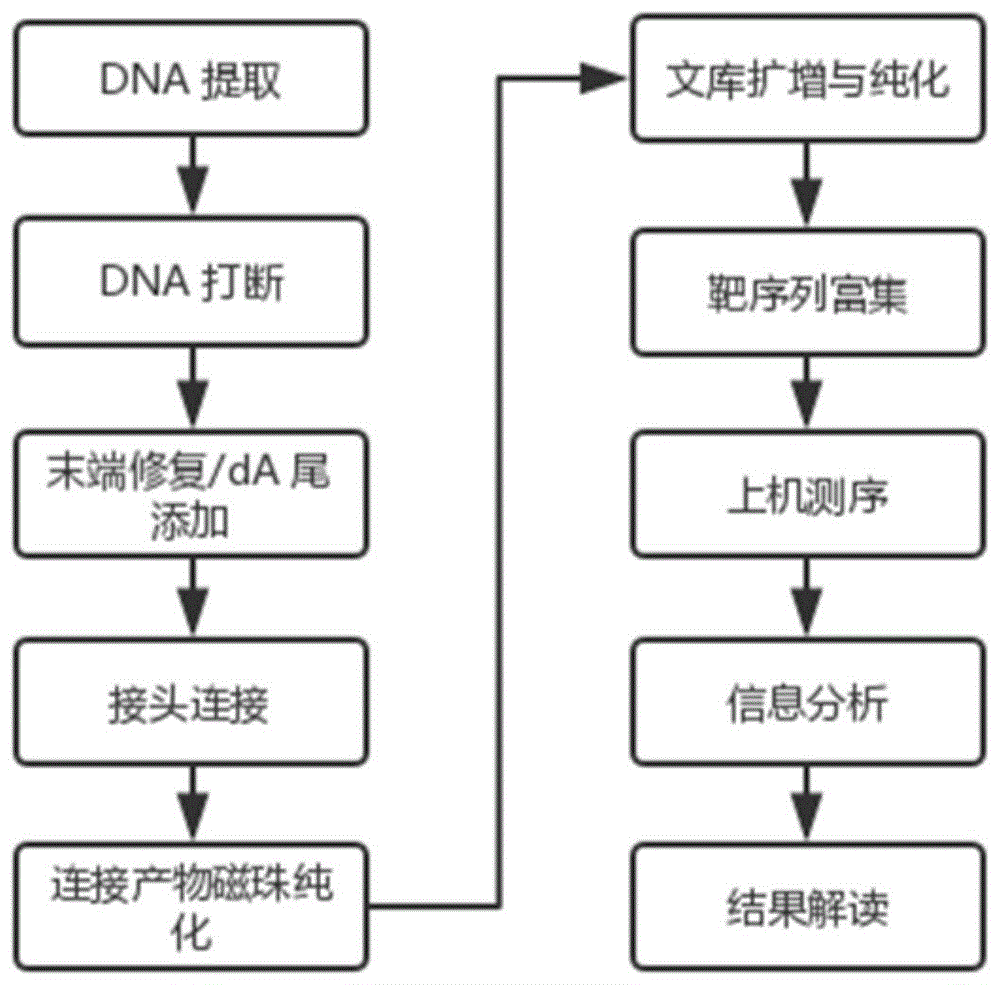
图1为本发明的实施流程图。
具体实施方式
为更好地说明本发明的目的、技术方案和优点,下面将结合附图和具体实施例对本发明进一步说明。本领域技术人员应当理解,此处所描述的具体实施例仅用以解释本发明,并不用于限定本发明。所描述的实施方案仅仅是本发明的一部分实施方案,而不是全部的实施方案。基于本发明中的实施方案,本领域普通技术人员可以获得的所有其他实施方案,并且它们都属于本发明保护的范围。
在本发明说明书和权利要求书中,提及基因序列时,本领域技术人员应当理解,该基因序列实际包括互补双链的任意一条或者两条。为了方便之目的,在本说明书和权利要求书中,虽然多数情况下只给出了一条链,但实际上也公开了与之互补的另一条链。例如提及一条探针序列,实际上包括该序列以及其互补序列。例如,提及seqidno:1,实际包括其互补序列。本领域技术人员还可以理解,利用一条链可以检测另一条链,反之亦然。
本发明中的基因序列包括dna形式或rna形式,在公开其中一种的情况下,意味着另一种也被公开。例如提及一条探针序列,实际也包括相应的rna序列。
如上所述,本领域需要一种全面、高灵敏度、高通量、并且针对多种突变类型同时精准检测的新技术。
本发明提供了用于同时检测多种基因突变类型的一组探针。本发明探针的设计长度为60-150nt,例如120nt,经过精密计算可达到100%目标区域覆盖以及至少60%以上的高特异性捕获效果。设计的探针由相关合成公司如idt或罗氏完成合成。将样品文库采用本发明设计的富集探针进行杂交捕获,根据建库时采用的不同测序平台的接头序列(illumina平台或华大智造测序平台),选择对应测序平台进行pe150测序,获得的测序数据,之后进行单样品测序信息分析流程,获得有效的靶向用药相关基因的突变以及化疗用药相关基因的碱基型,从而进行个体化用药指导。
在本发明中,针对靶向用药相关基因的探针的设计基于tcga、icgc、cosmic等数据库和相关文献参考,涵盖常见肿瘤例如肺癌、结肠直肠癌、胃癌、乳腺癌、肾癌、胰腺癌、卵巢癌、子宫内膜癌、甲状腺癌、子宫颈癌、食管癌和肝癌等的热点肿瘤驱动基因的全面突变位点,包括点突变、短片段插入缺失、拷贝数变异和融合基因,总共5727个热点突变。
因此,在第一方面,本发明提供了一种用于同时检测多种基因突变类型的探针组,所述探针组包括针对靶向用药相关基因的探针,其中所述靶向用药相关基因包括egfr、braf、kras、her2、met、alk、ret、ros1、ntrk1、ntrk2、ntrk3,其上的基因突变包括:
1)对于egfr基因:g719c、g719s、g719a、a698t、v765m、f712s、l703p、a702s、g724s、e709、e709g、e709k、s720f、e709v、s752_i759del、l747_s752del、l747_t751del、e746_a750del、e746_a750del、e746_s752>d、l747_e749del、l747_t751>s、e746_e749del、k745_e749del、l747_t751del、s752_1759del、t751_i759del、l747_p753>s、e746_s752del、e746_t751del、e746_t751>a、e746_t751>v、e746_s752>v、l747_t751>p、l747_a750>p、l747_p753>s、l747_t751del、e746_s752>a、k745_e746ins、c797s、v769l、s768i、t790m、r776c、v765m、s784p、g796a、v786m、a767v、c775y、f795s、r776h、g796s、d770n、h773r、g796d、l792p、s784f、g779f、v774m、h773l、d770>gy、d770_n771insg、h773_v774insh、l858r、l861q、h870y、h870r、e868g、a871t、g874s、l858l、g863d、g873e、a864t、a871g、a859t、l858r、l861r、l858m;
2)对于braf基因:a598_t599insv、a598v、d594g、d594n、d594v、e586e、e586k、f583f、f595l、f595s、g596d、g596r、g606e、h608r、i582m、i592m、i592v、k601e、k601n、l584f、l584l、l597l、l597q、l597r、l597s、l597v、n581s、p213s、s605f、s605g、s605n、t599_v600inst、t599_v600instt、t599i、v600_k601>e、v600a、v600d、v600e、v600g、v600k、v600l、v600m、v600q、v600r、w604g;
3)对于kras基因:g10_a11insg、g12g13insg、l19f、g13v、v14i、g12d、l19f、g13_v14insg、g12y、g12i、v8v、a11p、a11v、g12f、g12l、g12v、g12c、g12s、g12r、g12e、g12v、g12d、g12a、g12g、g13c、g13s、g13r、g13d、g13a、g13v、g13g、g13g、g15s、a18d、q22k、g13e、a11_g12insga、a59g、a59t、a59e、q61k、q61e、q61p、q61r、q61l、q61h、t58i、a146t、a146v、a146p、k117n;
4)对于her2基因:d769h、l755s、i767m、l755p、g776>vc、v777l、g776v、g778_s779insg、v777a、v842i、h878y;
5)对于met基因:e168d、t1010i、h1112y、h1124d、h1112l、l1130l、n1118y、h1112r、m1268t、y1248c、y1253d、14号外显子跳跃突变;
6)对于alk基因:eml4-alk;
7)对于ret基因:ncoa4-ret、prkar1a-ret、pcm1-ret、ktn1-ret、kif5b-ret、hook3-ret、golga5-ret、ccdc6-ret;
8)对于ros1基因:tpm3-ros1、slc34a2-ros1、sdc4-ros1、lrig3-ros1、gopc-ros1、ezr-ros1、cd74-ros1;
9)对于ntrk1/2/3基因:tpm3-ntrk1、lmna-ntrk1、nacc2-ntrk2、qki-ntrk2、etv6-ntrk3。
需要注意,在本发明中,上文所涉及到的基因位置依据cosmic数据库。但是,本领域技术人员应理解,这样的基因组位置可以对应于其他版本的基因组数据的位置,只要基因的相对位置对应上即可。此外,在本发明中,采用本领域通用表示法来表示突变。例如,突变g719c和g719s分别表示第719位的甘氨酸g突变为半胱氨酸c和丝氨酸s。
本领域技术人员可以理解,针对某个基因突变,在探针数量相对较多的情况下会获得更为准确的检测结果。但是从探针之间彼此之间可能发生的不利相互作用考虑以及从经济层面考虑,可以针对这些突变选择典型的探针组合,从而实现有效的检测。
为此,本发明人对设计得到的探针进行了筛选,发现部分探针的组合可以实现有效检测。因此,在一个实施方案中,所述探针组包括seqidno:1-16所示的探针序列。
此外,对于6个特定融合断点区域的基因-alk、ros1、ret、ntrk1、ntrk2、ntrk3,发明人对其进行了3倍高探针丰度覆盖,从而可以更灵敏(0.5%)的检出已知热点融合变异及未知融合变异。
因此,在一个实施方案中,本发明的探针组还包括seqidno:17-28所示的探针序列。
seqidno:1-16和seqidno:17-28的序列示于下表中。
除了上文所述的靶向用药相关基因外,本发明还涉及其他的靶向用药相关基因,目的在于更为有效地筛选出可能的基因突变,从而为用药提供指导。
因此,在一个实施方案中,所述靶向用药相关基因还包括:akt1、cdk4、fgfr2、map2k2、cdk6、fgfr3、pdgfra、smarca4、apc、cdkn2a、flt3、mlh1、pik3ca、smo、ar、ctnnb1、hras、msh2、pms2、src、atm、ddr2、idh1、msh6、ptch1、stk11、bcl2l11(bim)、idh2、mtor、pten、tp53、erbb2、jak2、nf1、ptpn11、tsc1、brca1、esr1、kit、nras、raf1、tsc2、brca2、fbxw7、rb1、vhl、ccnd1、fgfr1、map2k1。
本发明除了涉及靶向用药相关基因外,还涉及化疗用药基因。具体地,在一个实施方案中,所述探针组还包括针对化疗用药相关基因的探针,所述化疗用药相关基因包括:xrcc1、xpc、wt1、umps、ugt1a1、tyms、tp53、stmn1、sod2、slit1、slc29a1、slc28a3、sema3c、sele、rrm1、ptgs2、nqo1、nlgn1、mthfr、lta、htr1e、gstp1、gsto1、ggh、esr2、esr1、ercc2、ercc1、dync2h1、dpyd、dhfr、cyp3a4、cyp2d6、cyp19a1、colec10、cep72、cda、cbr3、c8orf34、areg、abcg2、abcc11、abcb1。
针对化疗用药相关基因的探针的设计基于pharmgkb、pubmed、fda等数据库和相关文献及机构,涵盖了目前主要的化疗药物如顺铂、卡铂、紫杉醇、多西紫杉醇、长春新碱、环磷酰胺、表柔比星、其他蒽环类、吉西他滨、甲氨蝶呤、依托泊苷等,总共检测47个位点。
在第二方面,本发明提供了一种用于同时检测多种基因突变类型的基因芯片,所述基因芯片包括本发明第一方面的探针组。
在第三方面,本发明提供了一种用于检测多种基因突变类型的方法,所述方法包括以下步骤:
1)探针设计:设计并合成本发明第一方面的探针组;
2)dna提取和打断:提取待检测样本的dna并将其打断为200-250bp的dna片段;
3)文库构建:利用打断的dna构建样本文库;
4)杂交捕获:将步骤3)的样本文库与步骤1)的探针组相接触以进行杂交捕获;
5)上机测序:对步骤4)得到的捕获序列进行测序以产生测序数据;
6)信息分析:对步骤5)得到的测序数据进行基因突变信息分析和注释。
如上所述,探针设计基于tcga、icgc、cosmic等数据库(对于靶向用药相关基因)、pharmgkb、pubmed、fda等数据库(对于化疗相关基因)、以及相关文献参考进行。在探针设计好之后委托相关合成公司如idt或罗氏完成合成。
dna可以从合适的来源提取。例如,在一个实施方案中,可以从石蜡包埋组织、石蜡切片、新鲜病理组织、血浆、胸水、腹水、脑脊液等体液样本中提取,但是不限于此。任何合适的样本均可用于本发明中。
dna的打断可以用例如超声波dna打断仪进行。由此,可以将dna打断为200-250bp的dna片段。
本领域技术人员可以理解,除了上文列出的具体步骤之外,还可以根据需要额外再引入一个或者多个其他步骤,例如片段纯化、文库纯化、捕获产物扩增等。
在第四方面,本发明提供了一种用于同时检测多种基因突变类型的试剂盒,所述试剂盒包括本发明第一方面的探针组或本发明第二方面的基因芯片。
可以理解,除了本发明的探针组或者基因芯片外,所述试剂盒还可以包含其它可用于检测基因突变的试剂。
例如,在一个实施方案中,所述试剂盒还可以包括:(1)dna提取试剂,例如适用于肿瘤cfdna、ffpe组织提取的试剂;(2)文库构建试剂;(3)引物和接头,具体取决于测序平台如illumina平台或者华大智造测序平台的要求;(4)核酸纯化试剂;(5)阳性质控品和阴性质控品。
本发明的试剂盒可以有效满足肿瘤个性化用药指导需求。
在第五方面,本发明提供了本发明第一方面的探针组或本发明第二方面的基因芯片在制备用于同时检测一个或者多个基因突变类型的检测试剂中的用途。
如上所述,本发明可以一次性对多达102个基因进行突变检测,从而发现已知和/或未知的突变;本发明可以一次性检测相关稀有热点变异——met基因的14号外显子跳跃突变、bcl2l11(bim)2号内含子缺失多态性相关区域以及ntrk1/2/3热点融合突变;由于对6个特定融合断点区域的基因-alk、ros1、ret、ntrk1、ntrk2、ntrk3进行了三倍探针丰度覆盖,因此本发明可以更灵敏(0.5%)地检出其已知热点融合和未知融合变异。
下面将通过以下实施例对本发明进行进一步描述。以下实施例中所使用的试验方法如无特殊说明均为常规方法;以下实施例中所使用的材料、试剂等如无特殊说明均可从商业途径得到。
实施例
1.dna提取与打断
取1例测试石蜡包埋病例组织10片-20片组织切片,按照qiaampdnaffpetissuekit(qiagen)提取试剂盒说明书,进行石蜡包埋病例组织样品的dna提取,之后采用qubit定量,要求组织dna至少大于100ng。提取后的组织dna,采用超声波dna打断仪打断至200bp-250bp的dna片段,然后按照文库构建试剂盒构建样本文库。
2.文库构建
2.1末端修复/da尾添加
于无菌1.5mlpcr管中,按照下表配置进行末端修复以及加“a”反应:
使用移液器轻轻吹打或振荡混匀,并短暂离心将反应液离心至管底,之后放入pcr仪,按照如下程序进行处理:30℃20分钟,70℃20分钟。
2.2接头连接
基于上一步的产物,按照下表配置,连接特定的mgiseq2000接头。
使用移液器轻轻吹打或振荡混匀,并短暂离心将反应液离心至管底,之后放入pcr仪,按照如下程序进行处理:20℃15分钟。
2.3连接产物磁珠纯化
对2.2步骤的产物进行片段分选与纯化,具体操作步骤如下:
1)准备工作:将dnaselectionbeads磁珠由冰箱中取出,室温平衡至少30分钟;配制80%乙醇。
2)将磁珠和80%乙醇混合,涡旋振荡或充分颠倒磁珠以保证充分混匀。
3)吸取60μldnaselectionbeads(0.6×,beads∶dna=0.6∶1)至接头连接产物中,室温孵育5分钟。
4)将pcr管短暂离心并置于磁力架中分离磁珠和液体,待溶液澄清后(约5分钟),小心移除上清。
5)始终保持pcr管置于磁力架中,加入200μl新鲜配制的80%乙醇来漂洗磁珠,室温孵育30秒后,小心移除上清。
6)重复步骤5)总计漂洗两次。
7)始终保持pcr管置于磁力架中,开盖空气干燥磁珠至刚刚出现龟裂(不超过5分钟)。
8)将pcr管从磁力架中取出,加入21μlddh2o,涡旋振荡或使用移液器轻轻吹打至充分混匀,室温静置5分钟。
9)将pcr管短暂离心并置于磁力架中静置,待溶液澄清后(约5分钟),小心移取20μl上清至新pcr管中,切勿触碰磁珠。
2.4文库扩增与纯化
于无菌0.2mlpcr管中,按照下表配置pcr反应体系,对接头连接产物进行pcr扩增富集。
使用移液器轻轻吹打或振荡混匀,并短暂离心将反应液离心至管底,之后放入pcr仪进行pcr,pcr反应条件如下:
扩增完成的产物使用dnaselectionbeads(0.9×,beads∶dna=0.9∶1)进行片段分选与纯化。纯化产物需进行qubit-br定量和2100质控。通常合格的文库产物要求浓度大于20ng/μl,目标片段主峰约380bp左右。
3.靶序列富集与上机测序
3.1在扩增文库质控合格后,采用本发明设计的探针组进行杂交捕获,参照芯片制造商(idt)提供的说明书。最后洗脱回溶21μlddh2o带杂交洗脱磁珠。
3.2杂交捕获产物的扩增
3.3先除去上一步磁珠,然后进行磁珠纯化,最后回溶25μlddh2o,进行qc及上机。
3.4采用mgiseq2000测序仪进行pe150上机测序,测序实验操作按照制造商提供的操作说明书进行上机测序操作。
4.信息分析及结果整理
数据下机后,基于barcode序列拆分检测样本,同时进行低质量数据删除。具体参数设置有:过滤掉序列头尾质量值低于20的片段;低于40bp的短序列丢弃不用;过滤占比>30%n碱基片段以及接头序列片段等。
根据模板片段位置形态(uid),进行聚簇标记duplicate,同时通过聚簇情况对测序引入的错误碱基进行纠正和碱基质量值修饰,之后使用bwamen进行重比对,使用gatk进行realignment和recalibration。
用mutect、varscan、somvar流程callsomaticsnv变异;用gatk、varscan、somvar流程callsomaticindel变异;用contra.py流程callcnv变异;用somvar流程callsv,最后注释变异的功能、reads支持数、变异频率、氨基酸变异及cosmic中的变异标注等。
获得的变异结果还需基于相关条件过滤,从而得到最终有意义的相关突变,具体标准有:a.过滤掉不在外显子(exonic)区、剪接(splicing)区、外显子和剪接区的突变;b.过滤掉同义突变;c.过滤掉位于重复区域的质量值低于30的变异;d.过滤掉1000g,exac,esp6500数据库中大于0.01的多态性突变。
5.检测结果分析:
5.1探针性能评估(qc):
通常情况下,30%x覆盖率大于90%表明覆盖情况很好。本发明的检测结果达到96.07%,这表明检测的目标区间均有效覆盖。
通常情况下,探针捕获效率越高,表明探针的特异性越好,更能有效降低测序成本。本发明的捕获效率达到65.47%,均高于行业大多数组织捕获效率50%。这预示探针特异性好。
5.2变异检测结果
本发明检测靶向基因3个变异结果与已知的变异类型和频率结果完全一致,详情见表3,化疗47个位点碱基型与已知碱基型也完全一致。
表3.靶向基因变异结果详情
5.3结果解读
基于靶向基因egfrl858r&e746a750del变异,预示该患者可使用egfr-tkis药物敏感,如厄洛替尼、奥希替尼、达可替尼、吉非替尼、埃克替尼、阿法替尼。基于靶向基因alk融合突变,预示该患者可考虑使用克唑替尼、艾乐替尼、色瑞替尼、布加替尼、劳拉替尼等alk抑制剂。
基于47个化疗检测位点信息,计算显示患者对嘧啶类似物卡培他滨、氟尿嘧啶、替加氟、替吉奥、吉西他滨、雷替曲塞疗效较好,但具体用药还需结合临床谨慎使用。
序列表
<110>武汉弘康健基因诊断技术有限公司
<120>用于同时检测多种基因突变类型的探针组、基因芯片及其应用
<130>cf200420s
<160>28
<170>siposequencelisting1.0
<210>1
<211>100
<212>dna
<213>人工序列(artificialsequence)
<400>1
gaaacatcctggtagctgaggggcggaagatgaagatttcggatttcggcttgtcccgag60
atgtttatgaagaggattcctacgtgaagaggagccaggt100
<210>2
<211>100
<212>dna
<213>人工序列(artificialsequence)
<400>2
gttaggtgggacagtacagcttccctccagccccaggttacccctgtcgtgtggctcctt60
ctttgctataggattattctttttggtgggtttctctgta100
<210>3
<211>100
<212>dna
<213>人工序列(artificialsequence)
<400>3
ggagaatagcaggtgagtgagttcccctctcgccgctccagcatcatggggacctgacaa60
agtcccactctcccctgtgatctttgcagccagcctcgca100
<210>4
<211>100
<212>dna
<213>人工序列(artificialsequence)
<400>4
ccaaacaacgctattaatcagacccatctccatatccactgtgagtgagacaggcatagt60
tcaggccttcaggcttgccagaagggcagtaagccacttg100
<210>5
<211>100
<212>dna
<213>人工序列(artificialsequence)
<400>5
ttggtgtacagaaaaagagattaatggttaatgggcagtcttcctctatatttctctatt60
tcaatccctactccaggttccataccttccaggtcatcaa100
<210>6
<211>100
<212>dna
<213>人工序列(artificialsequence)
<400>6
gcagtaagggagtgagtgggcaactcggcgcatgaaggaggtactcctcattttcgttct60
ctctctctgtgccccagcccgttggcagacccggatcatt100
<210>7
<211>100
<212>dna
<213>人工序列(artificialsequence)
<400>7
gacaccatgcagttgcgggctgctagatctcggtgcacaaacttgttggcagcaaggtag60
gccatgccgtctgcaatctcaccagccatttggatcattt100
<210>8
<211>100
<212>dna
<213>人工序列(artificialsequence)
<400>8
acgcaactgtctagtgggccagggactggtggtcaagattggtgattttggcatgagcag60
ggatatctacagcaccgactattaccgtgtaagggtcctt100
<210>9
<211>100
<212>dna
<213>人工序列(artificialsequence)
<400>9
gctgttaatgttttttccccccttgtgaatgttttctaactttgtcttggtaattgcaat60
ttaactaggtgcggtggctactaaagttcgaaggcacgat100
<210>10
<211>100
<212>dna
<213>人工序列(artificialsequence)
<400>10
atagtcccccctacaacatactgtcatactgctgggttttcatgggtaggaaagcttgtc60
ctgaccccagcagcaaagaggtggcaggtcgctaatgaat100
<210>11
<211>100
<212>dna
<213>人工序列(artificialsequence)
<400>11
caaatgaagtctgaaaatgactatgccagtcaacacactcctcttgaccaagaagtgact60
caccccaaaagtgtcttcttctggtttgtgggtcacagtg100
<210>12
<211>100
<212>dna
<213>人工序列(artificialsequence)
<400>12
ggactctggatcccagaaggtgagaaagttaaaattcccgtcgctatcaaggaattaaga60
gaagcaacatctccgaaagccaacaaggaaatcctcgatg100
<210>13
<211>100
<212>dna
<213>人工序列(artificialsequence)
<400>13
atgcccttcggctgcctcctggactatgtccgggaacacaaagacaatattggctcccag60
tacctgctcaactggtgtgtgcagatcgcaaaggtaatca100
<210>14
<211>100
<212>dna
<213>人工序列(artificialsequence)
<400>14
attttgggctggccaaactgctgggtgcggaagagaaagaataccatgcagaaggaggca60
aagtaaggaggtggctttaggtcagccagcattttcctga100
<210>15
<211>100
<212>dna
<213>人工序列(artificialsequence)
<400>15
tccacaaaatggatccagacaactgttcaaactgatgggacccactccatcgagatttca60
ctgtagctagaccaaaatcacctatttttactgtgaggtc100
<210>16
<211>100
<212>dna
<213>人工序列(artificialsequence)
<400>16
ctgaattagctgtatcgtcaaggcactcttgcctacgccaccagctccaactaccacaag60
tttatattcagtcattttcagcaggccttataataaaaat100
<210>17
<211>100
<212>dna
<213>人工序列(artificialsequence)
<400>17
aatgcccccctccctcgagccctcccttccacatggattgaaaacaaactctatggtaga60
atttcccatgcatttactagattctagcaccgctgtcccc100
<210>18
<211>100
<212>dna
<213>人工序列(artificialsequence)
<400>18
tgctgcatttcagagaacgcctccccgagtgagctgcgagacctgctgtcagagttcaac60
gtcctgaagcaggtcaaccacccacatgtcatcaaattgt100
<210>19
<211>100
<212>dna
<213>人工序列(artificialsequence)
<400>19
aagaaaaactgcttagtaactagcagaagtgttcctaaaagagtcatacacaggcccagg60
gcagttcttgggtgggtccatccggcctccactggtgaca100
<210>20
<211>100
<212>dna
<213>人工序列(artificialsequence)
<400>20
cagcaccgtcccgtggtcacagaagcagatgaccttgtggctttcagggtccatgtgaca60
ttcgtctacctcacagtgactgcagtttagataatgctta100
<210>21
<211>100
<212>dna
<213>人工序列(artificialsequence)
<400>21
acttctccaaaggctccacttcccagcaagagacgcagagtcagtttttcccgagggaag60
gcaggaagattttcaatctcctcttgggttggaagagtac100
<210>22
<211>100
<212>dna
<213>人工序列(artificialsequence)
<400>22
ttggcttgtggcttgattgcaaagtcctttaacgtgcaactctccacaatacgcacagct60
tctgcacaccggccagatggtacaggaaggtctgctctat100
<210>23
<211>100
<212>dna
<213>人工序列(artificialsequence)
<400>23
tgctttctctcctccctcctgctgcagtctccttctcgccggtgggtgagtagcccaagg60
tggagggcaggttctgcctggtctctggagctgaggctgg100
<210>24
<211>100
<212>dna
<213>人工序列(artificialsequence)
<400>24
cccccacggctcctcgggactgcgatgcacccgggatggggccctggatagcctccacca60
cctgcccggcgcagagaacctgactgagctgtgagtgtcc100
<210>25
<211>100
<212>dna
<213>人工序列(artificialsequence)
<400>25
cttcagcgacagtgtaaggaaggaagggggaggtgacttaaggggtgagggtgctgtggg60
gagggagaatgttgagaatgaagtggtgtttctatttatg100
<210>26
<211>100
<212>dna
<213>人工序列(artificialsequence)
<400>26
aaggacagtgttgaccacctccggtttctacttctctttcgaagtttattttatgttttg60
ttgtggttttcagatttctcatggtttggatttgggaaag100
<210>27
<211>100
<212>dna
<213>人工序列(artificialsequence)
<400>27
acctgcacacaccaagagagacgcaggacctggtgacgtcacatccggccagtttccagc60
cctgtccacagacaacccccaactactgttcccatcacaa100
<210>28
<211>100
<212>dna
<213>人工序列(artificialsequence)
<400>28
gagagcaatgggagattaaaacagtatcttttgatcaaaagcagtaatgtttccccccat60
gacgcccttgaaaatgaaactcccaccttacccttcattc100
- 还没有人留言评论。精彩留言会获得点赞!