结直肠癌相关的甲基化标志物以及用于检测结直肠癌的试剂盒的制作方法
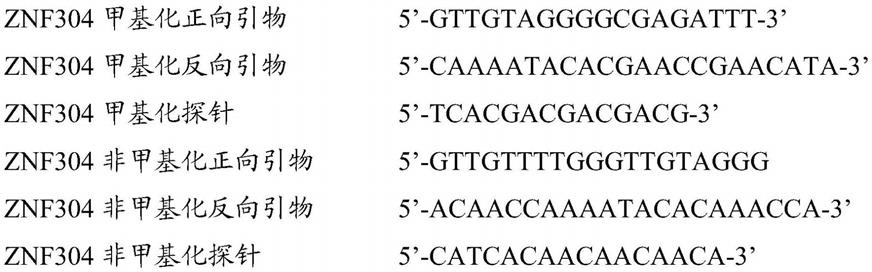
[0001]
本发明涉及一种结直肠癌相关的甲基化标志物,还涉及一种用于检测结直肠癌的试剂盒,本发明属于生物医药技术领域。
背景技术:
[0002]
结直肠癌(colorectal cancer)是常见的消化道恶性疾病,据globcan 2018预计新发病例超过180万,约占每年癌症新发病例的10%,居世界第三位,而死亡率仅次于肺癌
[1]
。据估计2019年美国将有超过14万新发病例及5万死亡病例,约占全部癌症发病和死亡的十分之一
[2]
。结直肠癌的5年生存率可达到65%以上,早期诊断并治疗的患者5年生存率可达到80%以上,而ⅳ期患者的5年生存率只有12%
[3]
。据国家癌症中心统计2000年到2011年间的数据显示,我国结直肠癌的发病率和死亡率均排在第五位,但发病率呈显著上升趋势,而男性增长速度一直高于女性
[4]
。与我国情况相反,自1990年美国的结直肠癌发病率和死亡率呈显著下降趋势,这与美国生活方式和饮食模式的转变尤其是早期筛查策略的制定密切相关
[5]
,早期筛查能够早期发现并且清除处于癌变前期或早期的结直肠病变,进而避免部分肿瘤的发生与进展
[6]
,而近年来发病率的迅速下降主要归功于结肠镜的使用,美国50岁及以上成年人的结肠镜检查使用率从2000年的21%增至2015年的60%,增长了近3倍;而50岁以下的成年人的发病率仍是呈现2%年增长率
[7]
。因此早期筛查是控制结直肠癌的有效措施。
[0003]
结直肠癌的早期筛查主要包括粪便隐血试验(fobt)和结肠镜检查等,粪便隐血试验具有简便无创等优点,但其灵敏度较低,假阳性率高,患者的依从性差[8]。结肠镜作为目前筛查,诊断和治疗结直肠癌的主流方式已经显著降低了美国欧洲等的发病及死亡率,但其有创性和不良反应,耗时,以及费用高可能会在一定程度上限制其的普及率
[9]
。并且只能提供一种静态的检查结果
[10]
。血清癌胚抗原(cea)作为一种非侵入性生物标志物具有良好的特异性,可识别隐匿性crc,但由于其敏感性低(40%至60%)而受到限制
[11]
。
[0004]
循环肿瘤dna(circulating tumor dna,ctdna)主要由原发性肿瘤,循环肿瘤细胞(ctc),微小或明显转移到癌症患者血液中的dna片段组成,ctdna大部分来源于肿瘤细胞的凋亡和坏死
[12]
。mandelp于1948年首次在正常人外周血发现游离dna(cfdna)
[13]
。1977年,leon sa等再次发现肿瘤患者血浆cfdna含量显著高于健康人,且晚期肿瘤患者的cfdna水平更高
[14]
。直到1994年,sorenson,g.d首次在胰腺癌患者血液中发现ras突变,并且和组织中保持一致
[15]
。随后有研究表明ctdna携带肿瘤特异性遗传改变,包括突变
[16]
,拷贝数变异
[17]
,同时发现ctdna还携带有癌症组织特异的表观遗传改变,肿瘤组织的特异性甲基化模式与ctdna高度相关
[18]
。因此通过对ctdna的生物学特性检测可以达到微创快速的筛查癌症并能对癌症进行实时纵向监测。
[0005]
dna甲基化是表观遗传学修饰中的一种,涉及分化和发育,衰老,肿瘤发生等。全基因组低甲基化和启动子区cpg岛高甲基化是肿瘤组织中最常见的生物学现象
[19]
。异常甲基
j.he.cancer statistics in china,2015.ca cancer j clin.2016;66(2):115-32.
[0013]
5.singh,g.k.,b.a.miller,and b.f.hankey.changing area socioeconomic patterns in u.s.cancer mortality,1950-1998:part ii
--
lung and colorectal cancers.j natl cancer inst.2002;94(12):916-25.
[0014]
6.edwards,b.k.,e.ward,b.a.kohler,c.eheman,a.g.zauber,r.n.anderson,a.jemal,m.j.schymura,i.lansdorp-vogelaar,l.c.seeff,m.van ballegooijen,s.l.goede,and l.a.ries.annual report to the nation on the status of cancer,1975-2006,featuring colorectal cancer trends and impact of interventions(risk factors,screening,and treatment)to reduce future rates.cancer.2010;116(3):544-73.
[0015]
7.national center for health,s.,health,united states,in health,united states,2015:with special feature on racial and ethnic health disparities.2016,national center for health statistics(us):hyattsville(md).
[0016]
8.ibanez-sanz,g.,m.garcia,n.mila,r.a.hubbard,c.vidal,g.binefa,l.benito,and v.moreno.false-positive results in a population-based colorectal screening program:cumulative risk from 2000 to 2017 with biennial screening.cancer epidemiol biomarkers prev.2019;28(11):1909-1916.
[0017]
9.nee,j.,r.z.chippendale,and j.d.feuerstein.screening for colon cancer in older adults:risks,benefits,and when to stop.mayo clin proc.2020;95(1):184-196.
[0018]
10.dominguez-vigil,i.g.,a.k.moreno-martinez,j.y.wang,m.h.a.roehrl,and h.a.barrera-saldana.the dawn of the liquid biopsy in the fight against cancer.oncotarget.2018;9(2):2912-2922.
[0019]
11.macdonald,j.s.carcinoembryonic antigen screening:pros and cons.semin oncol.1999;26(5):556-60.
[0020]
12.stroun,m.,j.lyautey,c.lederrey,a.olson-sand,and p.anker.about the possible origin and mechanism of circulating dna apoptosis and active dna release.clin chim acta.2001;313(1-2):139-42.
[0021]
13.mandel,p.and p.metais.les acides nucl
é
iques du plasma sanguin chez l'homme.c r seances soc biol fil.1948;142(3-4):241-3.
[0022]
14.leon,s.a.,b.shapiro,d.m.sklaroff,and m.j.yaros.free dna in the serum of cancer patients and the effect of therapy.cancer res.1977;37(3):646-50.
[0023]
15.sorenson,g.d.,d.m.pribish,f.h.valone,v.a.memoli,d.j.bzik,and s.l.yao.soluble normal and mutated dna sequences from single-copy genes in human blood.cancer epidemiol biomarkers prev.1994;3(1):67-71.
[0024]
16.medford,a.j.,r.n.gillani,and b.h.park.detection of cancer dna in early stage and metastatic breast cancer patients.methods mol biol.2018;1768:209-227.
[0025]
17.whale,a.s.,j.f.huggett,s.cowen,v.speirs,j.shaw,s.ellison,c.a.foy,and d.j.scott.comparison of microfluidic digital pcr and conventional quantitative pcr for measuring copy number variation.nucleic acids res.2012;40(11):e82.
[0026]
18.xu,r.h.,w.wei,m.krawczyk,w.wang,h.luo,k.flagg,s.yi,w.shi,q.quan,k.li,l.zheng,h.zhang,b.a.caughey,q.zhao,j.hou,r.zhang,y.xu,h.cai,g.li,r.hou,z.zhong,d.lin,x.fu,j.zhu,y.duan,m.yu,b.ying,w.zhang,j.wang,e.zhang,c.zhang,o.li,r.guo,h.carter,j.k.zhu,x.hao,and k.zhang.circulating tumour dna methylation markers for diagnosis and prognosis of hepatocellular carcinoma.nat mater.2017;16(11):1155-1161.
[0027]
19.suzuki,m.m.and a.bird.dna methylation landscapes:provocative insights from epigenomics.nat rev genet.2008;9(6):465-76.
[0028]
20.lund,a.h.and m.van lohuizen.epigenetics and cancer.genes dev.2004;18(19):2315-35.
[0029]
21.baylin,s.b.and p.a.jones.a decade of exploring the cancer epigenome-biological and translational implications.nat rev cancer.2011;11(10):726-34.
[0030]
22.laird,p.w.the power and the promise of dna methylation markers.nat rev cancer.2003;3(4):253-66.
[0031]
23.moran,s.,a.martinez-cardus,s.sayols,e.musulen,c.balana,a.estival-gonzalez,c.moutinho,h.heyn,a.diaz-lagares,m.c.de moura,g.m.stella,p.m.comoglio,m.ruiz-miro,x.matias-guiu,r.pazo-cid,a.anton,r.lopez-lopez,g.soler,f.longo,i.guerra,s.fernandez,y.assenov,c.plass,r.morales,j.carles,d.bowtell,l.mileshkin,d.sia,r.tothill,j.tabernero,j.m.llovet,and m.esteller.epigenetic profiling to classify cancer of unknown primary:a multicentre,retrospective analysis.lancet oncol.2016;17(10):1386-1395.
[0032]
24.koch,a.,s.c.joosten,z.feng,t.c.de ruijter,m.x.draht,v.melotte,k.m.smits,j.veeck,j.g.herman,l.van neste,w.van criekinge,t.de meyer,and m.van engeland.analysis of dna methylation in cancer:location revisited.nat rev clin oncol.2018;15(7):459-466.
[0033]
25.church,t.r.,m.wandell,c.lofton-day,s.j.mongin,m.burger,s.r.payne,e.castanos-velez,b.a.blumenstein,t.rosch,n.osborn,d.snover,r.w.day,and d.f.ransohoff.prospective evaluation of methylated sept9 in plasma for detection of asymptomatic colorectal cancer.gut.2014;63(2):317-25.
[0034]
26.issa,i.a.and m.noureddine.colorectal cancer screening:an updated review of the available options.world j gastroenterol.2017;23(28):5086-5096.
[0035]
27.powrozek,t.,p.krawczyk,t.kucharczyk,and j.milanowski.septin 9promoter region methylation in free circulating dna-potential role in noninvasive diagnosis of lung cancer:preliminary report.med oncol.2014;31(4):
quantitation of dna copy number.anal chem.2011;83(22):8604-10.
[0044]
36.yu,m.,k.t.carter,k.w.makar,k.vickers,c.m.ulrich,r.e.schoen,d.brenner,s.d.markowitz,and w.m.grady.methylight droplet digital pcr for detection and absolute quantification of infrequently methylated alleles.epigenetics.2015;10(9):803-9.
技术实现要素:
[0045]
为了填补目前并没有一种能够实现特异性检测结直肠癌的甲基化标志物的空白,本发明的目的之一即在于提供可用于特异性检测结直肠癌的甲基化标志物。
[0046]
本发明的目的之二在于提供一种基于上述甲基化标志物建立的可用于检测结直肠癌的试剂盒。
[0047]
为了达到上述目的,本发明采用了以下技术手段:
[0048]
本发明利用tcga等数据库分析结直肠癌组织特异性甲基化谱,筛选出肿瘤甲基化特异性标志物,主要筛选出分布在znf304,lifr及c20orf194基因上的10个cpg位点。然后使用靶向测序methyltarget技术在结直肠癌和癌旁正常组织中进行差异甲基化验证,并在组织结果的基础上提出了一种基于dna甲基化特异性定量检测技术(quantitative methylation specific pcr,qmsp)和基于数字pcr技术检测人组织/血浆dna中znf304和lifr基因甲基化的方法及所用探针与引物,和已有的基于qpcr技术的人sept 9基因甲基化检测方法相比,本发明的检测方法具有更高的特异性。此外,数字pcr能够对待检测目的片段进行绝对定量,通过计算甲基化和非甲基化阳性液滴或者拷贝数的比值而得到待测样品中目的片段的甲基化水平,能够实现高灵敏度的检测。
[0049]
因此,本发明首先提出了一种与结直肠癌相关的甲基化标志物,所述的甲基化标志物为含有至少一个cpg甲基化位点的znf304,lifr或c20orf194基因序列或其片段,或是所述的基因序列或其片段的任意组合;
[0050]
根据450k芯片的基因组注释信息,其中在c20orf194基因上存在3个cpg位点,分别为cg04125300、cg15863924以及cg02893482,cg04125300位于第20号染色体反链的第3388262核苷酸处,cg15863924位于第20号染色体反链的第3388269核苷酸处,cg02893482位于第20号染色体正链的第3388900核苷酸处;
[0051]
其中在lifr基因上存在3个cpg位点,分别为cg18174928、cg11841722以及cg12602374,cg18174928位于第5号染色体正链的第38557085核苷酸处,cg11841722位于第5号染色体反链的第38557253核苷酸处,cg12602374位于第5号染色体正链的第38557162核苷酸处;
[0052]
其中在znf304基因上存在4个cpg位点,分别为cg10157975、cg13788592、cg21627760以及cg23250910,cg10157975位于第19号染色体正链的第57862442核苷酸处,cg13788592位于第19号染色体反链的第57862612核苷酸处,cg21627760位于第19号染色体正链的第57862638核苷酸处,cg23250910位于第19号染色体反链的第57862410核苷酸处。
[0053]
进一步的,本发明还提出了用于扩增所述的结直肠癌的甲基化标志物的引物组,所述的引物组由分别用于扩增含有所述cpg位点的znf304,lifr及c20orf194基因片段的引物对组成,所述的引物序列如下所示:
[0054]
znf304_1
ꢀꢀꢀꢀꢀ
f:5
’-
aattagaatgtayggatygggtatagg-3’[0055]
r:5
’-
aaatatcrttttcccaatcttttctc-3’[0056]
znf304_2
ꢀꢀꢀꢀꢀ
f:5
’-
aygttagaagtttygtttttgtagtttagag-3’[0057]
r:5
’-
aaatacataccctttaaaaacaactataacc-3’[0058]
c20orf194
ꢀꢀꢀꢀ
f:5
’-
gattttaatagtaaaggttaggggtttt-3’[0059]
r:5
’-
ccaacrcacctccccaatac-3’[0060]
lifr
ꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
f:5
’-
gayggttttgyggggagga-3’[0061]
r:5
’-
ccccacacccracaaaaa-3’[0062]
其中,znf304_1引物对用于扩增znf304基因上存在的2个cpg位点,分别为cg13788592以及cg21627760,cg13788592位于第19号染色体反链的第57862612核苷酸处,cg21627760位于第19号染色体正链的第57862638核苷酸处;znf304_2引物对用于扩增znf304基因上存在的另外2个cpg位点,分别为cg10157975以及cg23250910,cg10157975位于第19号染色体正链的第57862442核苷酸处,cg23250910位于第19号染色体反链的第57862410核苷酸处。
[0063]
更进一步的,本发明还提出了一种用于结直肠癌相关基因甲基化检测的数字pcr试剂盒,所述试剂盒包括用于结直肠癌相关基因znf304以及lifr基因甲基化检测的ddpcr引物探针组合,所述的ddpcr引物探针组合如下1)和/或2)所示:
[0064]
1)znf304甲基化检测的ddpcr引物及探针以及znf304非甲基化检测的ddpcr引物及探针,所述的引物及探针序列如下所示:
[0065]
znf304甲基化正向引物
ꢀꢀꢀꢀꢀꢀꢀ5’-
gttgtaggggcgagattt-3’[0066]
znf304甲基化反向引物
ꢀꢀꢀꢀꢀꢀꢀ5’-
caaaatacacgaaccgaacata-3’[0067]
znf304甲基化探针
ꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ5’-
tcacgacgacgacg-3’[0068]
znf304非甲基化正向引物
ꢀꢀꢀꢀꢀ5’-
gttgttttgggttgtaggg
[0069]
znf304非甲基化反向引物
ꢀꢀꢀꢀꢀ5’-
acaaccaaaatacacaaacca-3’[0070]
znf304非甲基化探针
ꢀꢀꢀꢀꢀꢀꢀꢀꢀ5’-
catcacaacaacaaca-3’[0071]
2)lifr甲基化检测的ddpcr引物及探针以及lifr非甲基化检测的pcr引物及探针,所述的引物及探针序列如下所示:
[0072]
lifr甲基化正向引物
ꢀꢀꢀꢀꢀꢀꢀꢀꢀ5’-
gggattttgcgaattatttaaatag-3’[0073]
lifr甲基化反向引物
ꢀꢀꢀꢀꢀꢀꢀꢀꢀ5’-
caaaaaaaccaaaacgcgaa-3’[0074]
lifr甲基化探针
ꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ5’-
acgacgcctcgac-3’[0075]
lifr非甲基化正向引物
ꢀꢀꢀꢀꢀꢀ5’-
tgattgttgggattttgtga-3’[0076]
lifr非甲基化反向引物
ꢀꢀꢀꢀꢀꢀ5’-
acccaacaaaaaaaccaaaac-3’[0077]
lifr非甲基化探针
ꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ5’-
caacacctcaaccc-3’[0078]
其中,优选的,所述探针的5’端包含有荧光报告基团,所述荧光报告基团包括fam、hex、ned、rox、tet、joe、tamra、cy3、cy5中的任意一种,且甲基化探针和非甲基化探针使用不同的荧光基团,所述荧光探针的3’端包含有荧光淬灭基团,所述荧光淬灭基团包括mgb、bhq-1、bhq-2、bhq-3中的任意一种。
[0079]
其中,优选的,所述试剂盒的数字pcr反应体系和条件如下:
[0080]
数字pcr反应体系:2
×
ddpcr supermix for probes 10ul,加入引物及探针使其
终浓度分别达到引物终浓度为0.9um以及探针终浓度为0.25um,5-6ul浓度为0.002-70ng/ul的模板dna,补足ddh2o至21ul;
[0081]
数字pcr反应条件如下:
[0082]
95℃预变性10分钟,94℃变性30秒,60℃退火60秒,共进行40个循环,98℃灭活酶10分钟,之后4℃保持;升降温速度≤2.5℃/s。
[0083]
其中,优选的,所述的模板dna为待测者血液中的dna。
[0084]
其中,优选的,所述的模板dna为外周血血浆或血清中循环游离dna(circulating cellfreetumor dna,cfdna)、循环肿瘤dna(circulating tumor dna,ctdna)或循环肿瘤细胞(circulating tumor cells,ctcs)dna。
[0085]
更进一步的,本发明还提出了一种用于结直肠癌相关基因甲基化检测的qmsp试剂盒,所述试剂盒包括用于结直肠癌相关基因znf304以及lifr基因甲基化检测的qmsp引物探针组合,所述的qmsp引物探针组合如下1)和/或2)所示:
[0086]
1)znf304甲基化检测的qmsp引物及探针,所述的引物及探针序列如下所示:
[0087]
znf304甲基化正向引物
ꢀꢀꢀ5’-
cgttttttgttgttttgggtt-3’[0088]
znf304甲基化反向引物
ꢀꢀꢀ5’-
aaatacacgaaccgaacataaa-3’[0089]
znf304甲基化探针
ꢀꢀꢀꢀꢀꢀꢀ5’-
aatacgtcacgacgacgacgcca-3’[0090]
2)lifr甲基化检测的qmsp引物及探针,所述的引物及探针序列如下所示:
[0091]
lifr甲基化正向引物
ꢀꢀ5’-
ggatcgcg ttttttcgaatg-3’[0092]
lifr甲基化反向引物
ꢀꢀ5’-
acaaaaacgacgaaaacacg-3’[0093]
lifr甲基化探针
ꢀꢀꢀꢀꢀꢀ5’-
tccccgctttcctctcctcaaaacac-3’[0094]
其中,优选的,所述探针的5’端包含有荧光报告基团,所述荧光报告基团包括fam、hex、ned、rox、tet、joe、tamra、cy3、cy5中的任意一种,所述荧光探针的3’端包含有荧光淬灭基团,所述荧光淬灭基团包括mgb、bhq-1、bhq-2、bhq-3中的任意一种。
[0095]
其中,优选的,所述试剂盒的pcr反应体系和条件如下:
[0096][0097]
pcr反应扩增条件:95℃预变性10分钟,95℃变性10秒,60℃退火30秒,共进行45个循环,40℃冷却30秒,之后4℃保持。
[0098]
相较于现有技术,本发明的有益效果是:
[0099]
1、本发明通过分析甲基化450k高通量芯片数据首次发现了分布在c20orf194、znf304、lifr基因上的10个结直肠癌特异性的cpg位点,并使用靶向测序的方法在组织中对其中8个位点进行了验证;并成功建立了基于qmsp技术检测人组织znf304、lifr基因甲基化的检测方法。
[0100]
2、本发明首次在外周血dna中检测了c20orf194、znf304、lifr基因的甲基化水平,并首次使用数字pcr和methylight相结合的检测方法,通过优化数字pcr引物和探针,优化反应体系和反应条件,成功建立了基于ddpcr技术检测人znf304、lifr基因甲基化的检测方法。和已有的基于qpcr技术的人sept9基因甲基化检测方法相比,本发明的检测方法具有更高的特异度,通过计算甲基化阳性液滴数和甲基化阳性液滴及非甲基化阳性液滴的比值进而实现绝对定量,可以对结直肠癌患者znf304、lifr基因甲基化进行特异性定性和定量检测,从而实现对结直肠癌的辅助诊断。
附图说明
[0101]
图1为11种来源于tcga数据库的癌症组织及来自于geo的健康人外周血白细胞数据的平均甲基化水平;
[0102]
图2a以及图2b为c20orf194基因所包含的位点甲基化水平箱式图;
[0103]
图3a以及图3b为lifr基因所包含的位点甲基化水平箱式图;
[0104]
图4a以及图4b为znf304基因所包含的位点甲基化水平箱式图;
[0105]
图5为crc癌和癌旁组织靶向测序差异性甲基化分析结果;
[0106]
图6为crc癌、腺瘤、息肉组织和癌旁组织差异性甲基化分析结果;
[0107]
图7a和图7b分别为扩增znf304基因以及基因lifr的引物位置图;
[0108]
其中,图7a中各cpg的位置以及所对应的编号如下所示:
[0109]
[0110][0111]
其中,图7b中各cpg的位置以及所对应的编号如下所示:
[0112]
[0113][0114]
图8为znf304 qmsp扩增标准曲线;
[0115]
图9为lifr qmsp扩增标准曲线;
[0116]
图10为actb qmsp扩增标准曲线;
[0117]
图11为qmsp扩增znf304甲基化水平的箱式散点图;
[0118]
图12为qmsp扩增lifr甲基化水平的箱式散点图;
[0119]
图13为znf304数字pcr质控品检测结果;
[0120]
图14为lifr数字pcr质控品检测结果;
[0121]
图15为结直肠癌患者及健康对照血浆中znf304的甲基化水平箱式散点图;
[0122]
图16为结直肠癌患者及健康对照血浆中lifr的甲基化水平箱式散点图。
具体实施方式
[0123]
以下通过具体实施例对本发明进行详细描述,以使本领域技术人员能够容易地根据本说明书的公开内容实施本发明。以下所描述的实施例仅是本发明的一部分实施例,而不是全部。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其它实施例,都属于本发明保护的范围。
[0124]
实施例1结直肠癌的cfdna甲基化标志物的筛选及验证
[0125]
1、结直肠癌组织特异性
[0126]
本发明利用tcga等数据库分析结直肠癌组织特异性甲基化谱,筛选出肿瘤甲基化特异性标志物,筛选出主要分布在znf304,lifr及c20orf194基因上的10个cpg位点。基于tcga数据库中包括结直肠癌在内的11种癌症组织及来自于geo的健康人外周血白细胞的450k甲基化数据,表明10个cpg的结直肠癌组织特异性,平均甲基化水平见图1,三个基因所包含的位点甲基化水平箱式图见图2、图3、图4。10个cpg位点基因组信息见表1,癌症类型见表2。
[0127]
表1
[0128]
[0129][0130]
注:基因组注释信息取自450k芯片(grch37/hg19)
[0131]
表2
[0132][0133]
2、甲基化靶向测序
[0134]
组织甲基化检测方法是结合目标区域富集和二代测序技术methyltarget,凭借高深的测序深度实现多重pcr,能够准确计算每个cpg位点的甲基化水平。便于评价本发明所选取的10个cpg位点的甲基化水平,及差异甲基化水平。该技术准确性高,灵活性强,性价比优。
[0135]
本发明共纳入经病理组织学确诊原发结直肠癌患者276例,腺瘤组织8例,息肉组织10例,部分匹配的癌旁组织23例;使用酚-氯仿法和dna mini kit(qiagen,51306,德国)提取组织中的dna,具体操作按照试剂盒操作手册进行。用thermo scientific nanodrop 2000c分光光度计对提取的dna进行定性和定量检测(a260/a280在1.7-1.9之间,a260/a230>2)。提取后dna分装于-80℃保存。
[0136]
使用fast dna bisulfite kit(qiagen,59826,德国)对1-2ug原始dna进行亚硫酸氢盐处理,具体操作按照试剂盒操作手册进行。硫化后dna用nanodrop 2000c进行浓度(ng/ul)和纯度测定,硫化后dna序列中非甲基化的c被转化为u,因此大多数序列不再互补而成为单链,与rna较为相似。因此测定模式为rna,检测结果a260/280在2.0-2.4之间且a260/a230>1提示硫化后dna质量合格。硫化后dna稀释至20-25ng/ul分装后于-20℃保存。
[0137]
甲基化靶向测序实验步骤:
[0138]
1)样品质控
[0139]
硫化后dna浓度在20-25ng/ul之间,总量≥500ng,od260/280=2.0-2.4,od260/230≥1。
[0140]
2)引物设计与单位点pcr条件优化
[0141]
使用primer3或自行调整针对目标cpg位点所在区域设计高质量的测序引物。在将序列导入软件之前要将目标序列手动转换为硫化后序列,即保持目标cpg的胞嘧啶不改变而其余cpg的c转换为u;挑选能够以经重亚硫酸盐处理后的人基因组为模板,扩增获得清晰单一条带的引物用于后续实验。c20orf194基因共包括3个cpg位点,引物预试成功一个位点(3388900);lifr包含的3个cpg位点均预试成功;znf304包含4个cpg位点,分别设计在两条引物上。引物序列见表3。
[0142]
表3
[0143]
[0144][0145]
3)多重pcr引物panel优化
[0146]
将经步骤2)优化后的引物混合为多重pcr引物panel,以标准人基因组为模板进行扩增。基于毛细管电泳结果判断多重体系中每对引物是否高效、特异地进行扩增,并以此调整,优化多重pcr panel中的引物组成及浓度。
[0147]
4)样本目标片段多重pcr反应
[0148]
使用优化后的多重pcr引物panel,以硫化后dna为模板,进行多重pcr扩增。经质控后,将以同一个样品基因组dna为模板的所有多重pcr引物panel的扩增产物混合,并确保每个位点引物扩增产物的量相当,将混合后的多重pcr产物,各取2μl,稀释到60μl,作为后续index pcr步骤的模板。多重pcr反应体系见表4和扩增条件见表5,总反应体系为10ul,余下用水补齐。
[0149]
表4多重反应体系
[0150]
[0151][0152]
表5多重反应条件
[0153] 变性退火延伸保持循环数1step95℃2min
ꢀꢀꢀ1×
2step95℃20s62℃40s72℃1min 11
×
(-0.5℃/cycle)3step95℃20s64℃30s72℃1min 24
×
4step
ꢀꢀ
72℃1min 1
×
5step
ꢀꢀꢀ
4℃forever
[0154]
5)样本添加特异性标签序列
[0155]
利用带有index序列的引物,通过pcr扩增向上述步骤扩增后建立的文库末端引入和illumina平台兼容的特异性标签序列。反应采用12个循环数的pcr程序,尽可能降低pcr偏倚。indexpcr体系及扩增条件见表6,表7。总反应体系为10ul,余下用水补齐。
[0156]
表6
[0157][0158][0159]
表7
[0160] 变性退火延伸保持循环数
1step95℃2min
ꢀꢀꢀ1×
2step95℃20s60℃40s72℃1min 123step
ꢀꢀ
72℃2min 1
×
4step
ꢀꢀꢀ
4℃forever
[0161]
6)定量后上机测序
[0162]
将所有样品index pcr扩增产物等量混合,并经割胶回收获得最终的methyl target测序文库,文库的片段长度分布经agilent 2100 bioanalyzer验证。文库摩尔浓度精确定量后,最终于illumina hiseq平台,以2
×
150bp的双端测序模式进行高通量测序,获得fastq数据。
[0163]
3、甲基化靶向测序数据分析
[0164]
首先根据目标区域的基因组位置核对引物扩增的区域在参考基因组上的注释信息。其次对测序原始数据整理及质量评估,及对胞嘧啶转化效率统计以评价硫化转化效率。最后计算各cpg位点甲基化水平。
[0165]
c20orf194,znf304_1,znf304_2均设计在反链上,因此基因位点的位置相比450k注释信息增加一位。剔除胞嘧啶转化效率<98%的3个样本以保证完全硫化转化,剔除测序深度低于10
×
的42个样本以保证数据质量,剩余272例样本数据。crc癌和癌旁差异性甲基化分析结果见图5,所筛选的位点及其周围临近位点均显示癌组织甲基化水平显著高于癌旁组织,auc的范围为46.17%-89.81%,结果见表8(目标位点加粗);且数据结果表明所筛选的基因可能在腺瘤性息肉阶段甲基化水平就有升高趋势(见图6或表9)。基于外部数据和组织验证结果,说明本发明筛选的得到的10个cpg甲基化位点能够作为特异性检测结直肠癌的甲基化位点,而含有上述cpg甲基化位点的核苷酸序列可以作为特异性检测结直肠癌的甲基化标志物。
[0166]
表8 methyltarget_auc
[0167]
[0168]
[0169]
[0170]
[0171][0172]
表9 methyltarget_methylation
[0173]
[0174]
[0175][0176]
实施例2甲基化特异性荧光定量pcr(qmsp)
[0177]
使用qmsp探针法对结直肠癌和癌旁组织及外周血白细胞dna进行甲基化检测,在靶向测序结果的基础上设计引物。以经过亚硫酸氢盐处理过的全甲基化人类基因组dna为模板构建目标片段质粒,梯度稀释构建甲基化标准曲线并建立方程,以actb上无cg位点序列为内参基因,样本目的基因扩增拷贝数和内参基因扩增拷贝数的比值作为目标基因相对
甲基化水平。
[0178]
1、引物及探针设计
[0179]
针对所筛选位点设计znf304、lifr及内参基因actb的qmsp引物和探针,扩增引物包括甲基化扩增引物及内参扩增引物,qmsp探针包括甲基化探针及内参探针;内参引物设计在actb基因上无cg序列,防止对甲基化检测有干扰。探针的荧光基团如fam、vic等具体参照荧光定量pcr仪器荧光范围,猝灭基团可以选择能够增加探针退火温度的mgb基团或其他;挑选能够在阳性质控品即经过亚硫酸氢盐处理过的全甲基化人类基因组dna为模板的体系中扩增获得清晰单一光滑的“s”型扩增曲线的引物探针组合用于后续实验,而在去rna酶水或空白对照ntc中无任何荧光信号。反应引物及探针序列见表10。引物位置如图7所示。
[0180]
表10引物及探针序列
[0181]
[0182][0183]
2、dna样本的提取和制备
[0184]
本发明共纳入经病理组织学确诊原发结直肠癌(crc)患者90例,部分匹配的癌旁组织(normal)41例,结直肠癌患者外周血白细胞(crc_wb)52例,健康对照外周血白细胞(wb)50例;使用dna mini kit(qiagen,#51306,德国)提取样本中的dna,具体操作按照试剂盒操作手册进行。用thermo scientific nanodrop 2000c分光光度计对提取的dna进行定性和定量检测(a260/a280在1.7-1.9之间,a260/a230>2)。提取后dna分装于-80℃保存。
[0185]
使用fast dna bisulfite kit(qiagen,#59826,德国)对1-2ug原始dna进行亚硫酸氢盐处理,具体操作按照试剂盒操作手册进行。硫化后dna用nanodrop 2000c进行浓度(ng/ul)和纯度测定,硫化后dna序列中非甲基化的c被转化为u,因此大多数序列不再互补而成为单链,与rna较为相似。因此测定模式为rna,检测结果a260/280在2.0-2.4之间且a260/a230>1提示硫化后dna质量合格。硫化后dna稀释至20-25ng/ul分装后于-20℃保存。
[0186]
3、人znf304、lifr甲基化荧光定量pcr检测体系
[0187]
反应体系包括480probes master pcr预混液及目的基因引物探针组合和模板见表11。使用480software release 1.5.0设定pcr反应扩增条件:95℃预变性10分钟,95℃变性10秒,60℃退火30秒,共进行45个循环,40℃冷却30秒,之后4℃保持。pcr完成后使用abs quant/2nd derivative max模块进行分析,将样本cp值带
入目标基因和内参基因标准曲线,计算相对甲基化水平。
[0188]
表11
[0189][0190]
4、检测体系有效性及结果的判断标准
[0191]
(1)znf304检测体系有效性及结果的判断:
[0192]
a)有效性判定:梯度稀释质粒浓度范围为5e7-5e1 copies/ul,均有扩增信号且扩增曲线为光滑的“s”型,去rna酶水/ntc无扩增信号或无“s”型,判断有效。否则重新配置。
[0193]
b)结果判定:使用标准梯度稀释质粒的检测cp值和经对数转换的浓度构建标准曲线,标准曲线的扩增效率在1.9-2.1之间(90%-110%)。样本cp值在标准曲线范围内可纳入标准曲线计算拷贝数;样本cp大于最低浓度质粒cp值,提示人znf304基因甲基化阴性或甲基化程度低于最低检出限。znf304扩增标准曲线见图8。
[0194]
(2)lifr检测体系有效性及结果的判断:
[0195]
a)有效性判定:梯度稀释质粒浓度范围为5e7-5e1 copies/ul,均有扩增信号且扩增曲线为光滑的“s”型,去rna酶水/ntc无扩增信号或无“s”型,判断有效。否则重新配置。
[0196]
b)结果判定:使用标准梯度稀释质粒的检测cp值和经对数转换的浓度构建标准曲线,标准曲线的扩增效率在1.9-2.1之间(90%-110%)。样本cp值在标准曲线范围内可纳入标准曲线计算拷贝数;样本cp大于最低浓度质粒cp值,提示人lifr基因甲基化阴性或甲基化程度低于最低检出限。lifr扩增标准曲线见图9。
[0197]
(3)actb检测体系有效性及结果的判断:
[0198]
a)有效性判定:梯度稀释质粒浓度范围为5e7-5e1 copies/ul,其中5个连续浓度有扩增信号且扩增曲线为光滑的“s”型,去rna酶水/ntc无扩增信号或无“s”型,判断有效。否则重新配置。
[0199]
b)结果判定:使用标准梯度稀释质粒的检测cp值和经对数转换的浓度构建标准曲线,标准曲线的扩增效率在1.9-2.1之间(90%-110%)。样本cp值在标准曲线范围内可纳入标准曲线计算拷贝数;样本cp大于最低浓度质粒cp值,提示人actb基因阴性或低于最低检出限。actb扩增标准曲线见图10。
[0200]
5、检测结果及分析
[0201]
本发明共检测原发性结直肠癌90例,部分匹配的癌旁组织41例,结直肠癌患者外周血白细胞52例,健康对照外周血白细胞50例;将样本cp值带入标准曲线计算相对甲基化
水平。结果显示znf304、lifr在结直肠癌组织中呈现高甲基化状态而在癌旁及外周血白细胞中呈较低甲基化水平,表明外周血白细胞dna甲基化水平不会对后续cfdna中两基因检测有干扰,znf304、lifr箱式散点图分别见图11、图12。znf304甲基化水平检测结直肠癌组织的灵敏度为77.8%,特异度为97.6%,受试者曲线下面积为0.812;lifr甲基化水平检测结直肠癌组织的灵敏度为73.3%,特异度为90.2%,受试者曲线下面积为0.806。使用roc曲线计算两基因最佳cut-off值,分别在不同分期中计算两基因及其联合阳性率,结果见表12、表13、表14。
[0202]
表12
[0203][0204][0205]
表13
[0206]
样本lifr阳性/检测人数阳性率(%)ⅰ期5/5100%ⅱ期27/3871.1%ⅲ期29/4170.7%ⅳ期5/683.3%所有患者66/9073.3%健康对照4/419.8%(特异性90.2%)
[0207]
表14
[0208]
样本znf304或lifr阳性/检测人数阳性率(%)ⅰ期5/5100%ⅱ期34/3889.5%ⅲ期36/4187.8%ⅳ期6/6100%所有患者81/9090.0%健康对照4/419.8%(特异性90.2%)
[0209]
实施例3 methylight ddpcr检测方法以及试剂盒的建立
[0210]
本发明所采用的技术是数字pcr技术结合methylight法,通过设计特异性的人znf304、lifr候选甲基化区域的引物和探针,扩增经亚硫酸氢盐修饰并经微滴化制备的待测dna样本,根据pcr扩增的阳性微滴数及与总阳性液滴数的比值来确定待测样本中人znf304、lifr候选甲基化区域是否发生了甲基化以及甲基化水平,从而判断待测样本是否
发生结直肠癌。
[0211]
1、引物及探针设计
[0212]
针对所筛选位点设计znf304和lifr的ddpcr引物和探针,扩增引物包括甲基化序列扩增引物及非甲基化序列扩增引物,taqman探针包括甲基化探针和非甲基化探针,为连接报告荧光基团和淬灭基团的核苷酸,甲基化探针和非甲基化探针应使用不同的荧光基团如fam、vic等具体参照数字pcr机器荧光范围,猝灭基团可以选择能够增加探针退火温度的mgb基团,或者使用锁式探针;ddpcr反应引物及探针序列见表15。
[0213]
表15
[0214]
[0215][0216]
在本发明的示例性实施例中,数字pcr采用微滴pcr技术(droplet digital,pcr)。其主要特征是将数字pcr混合液加入微滴发生器,生成10000~20000个微反应液滴后进行pcr扩增。在每次的pcr布板中要同时包括阳性质控品(m)、阴性质控品(u)、去rna酶水和空白对照ntc,所谓的阳性质控品可以是经过亚硫酸氢盐处理过的全甲基化人类基因组dna,而阴性质控品可以是经过硫化的非甲基化人类基因组dna。根据质控品和空白对照设定阈值判断待测样品中是否含有发生甲基化的人znf304、lifr基因的dna目标分子,同时确定发生甲基化的人znf304、lifr的目标dna分子的数量。通过相应的已经商品化的软件(如quantasoft,bio-rad)进行数据分析。
[0217]
2、dna样本的提取和制备
[0218]
本发明检测的dna模板可以来源于提取待测者外周血血浆/血清中的循环游离dna(cfdna)、循环肿瘤dna(ctdna)或通过提取循环肿瘤细胞的dna;具体操作详述如下。
[0219]
1)待测样本cfdna/ctdna的提取
[0220]
取40例经病理组织学诊断的结直肠癌患者或13例健康对照的3-5ml血浆或血清,使用商品化的游离核酸提取试剂盒如qiaamp circulating nucleic acid kit制备dna样本,具体操作按照试剂盒操作手册进行。用50ul洗脱液将待测dna从离心柱上洗脱下来,用qubit3.0荧光定量仪对样本浓度进行测定,此系统采用专门研制的荧光染料,比传统的紫外吸光法更加灵敏,尤其适用于本发明中珍贵的微量样品。
[0221]
2)待测样本dna的硫化
[0222]
使用市面上常用的商业化试剂盒例如fast dna bisulfite kit对提取的待测dna进行亚硫酸氢盐处理,具体操作按照试剂盒操作手册进行。用20ul洗脱液洗脱待测dna,用于数字pcr检测。
[0223]
3、人znf304、lifr基因甲基化数字pcr检测体系
[0224]
(1)数字pcr反应体系:将硫化后样本分为两份,分别配置两基因的反应体系,在20ul pcr反应体系中,2
×
ddpcr supermix for probes 10ul,加入引物及探针使其终浓度分别达到引物终浓度为0.9um以及探针终浓度为0.25um,5-6ul浓度为0.002-70ng/ul的模板dna,补足ddh2o至21ul。
[0225]
(2)微滴pcr反应体系的制备与扩增:在配置好的体系中抽取20ul(上步配多体系以免产生气泡)加入微滴发生卡(bio-rad)中间一排的8个孔内,在下方的空孔中加入70ul微滴发生油,垫上胶垫,缓慢转移到微滴生成仪(bio-rad)中生成微滴,约在2分钟内生成10000-20000个微滴。将制备好的微滴吸取40ul缓慢转移到96孔板中,封好膜之后应该在30分钟内进行pcr反应,或者放于4℃冰箱4小时之内进行pcr,可在任意一台96孔pcr仪中按照设定好的反应条件进行pcr反应。
[0226]
上述pcr反应扩增条件为95℃预变性10分钟,94℃变性30秒,60℃退火60秒,共进行40个循环,98℃灭活酶10分钟,之后4℃保持;升降温速度≤2.5℃/s。
[0227]
(3)信号检测和结果判定:pcr扩增结束后,将96孔板放入微滴读取仪,按照设定好的顺序吸取每个样品的微滴并随微滴阅读油通过双色检测器(bio-rad),通过quantasoft
(version 1.7)软件分析计算出待测样本中znf304、lifr的液滴数。
[0228]
4、检测体系有效性及结果的判断标准
[0229]
(1)znf304检测体系有效性及结果的判断:
[0230]
a)阳性质控品(m):在fam(ch1)通道有信号,在vic(ch2)通道、去rna酶水/ntc无信号,则判断为阳性质控品有效。否则重新配置。
[0231]
b)阴性质控品(u):在fam(ch1)通道、去rna酶水/ntc无信号,在vic(ch2)通道有信号,则判断阴性质控品有效。否则重新配置。
[0232]
c)结果判定:根据阳性、阴性质控品、去rna酶水/ntc划定阈值线,fam(ch1)通道阳性液滴数2个及以上判定为甲基化阳性;vic(ch2)通道阳性液滴数3个及以上判定为非甲基化阳性。在本检测体系中非甲基化阳性液滴要远远多于甲基化阳性液滴,质控品检测结果见图13。
[0233]
(2)lifr检测体系有效性及结果的判断:
[0234]
a)阳性质控品(m):在fam(ch1)通道有信号,在vic(ch2)通道、去rna酶水/ntc无信号,则判断为阳性质控品有效。否则重新配置。
[0235]
b)阴性质控品(u):在fam(ch1)通道、去rna酶水/ntc无信号,在vic(ch2)通道有信号,则判断阴性质控品有效。否则重新配置。
[0236]
c)结果判定:根据阳性、阴性质控品、去rna酶水/ntc划定阈值线,fam(ch1)通道阳性液滴数2个及以上判定为甲基化阳性;vic(ch2)通道阳性液滴数2个及以上判定为非甲基化阳性,质控品检测结果见图14。
[0237]
5.检测结果及分析
[0238]
本发明共检测40例crc患者和13例健康对照血浆游离dna,根据阈值线计算每个样品的甲基化阳性液滴数和非甲基化阳性液滴数,甲基化阳性液滴数在总阳性液滴数中占的比例作为样品甲基化水平,结直肠癌患者血浆中znf304、lifr均呈现高甲基化状态见图15、图16。使用roc曲线计算两基因最佳cut-off值分别为0.003和0.002,分别在不同分期中计算两基因及其联合阳性率,结果见表16、表17、表18。
[0239]
表16
[0240]
crc患者znf304阳性/检测人数阳性率(%)ⅰ期1/250%ⅱ期5/955.6%ⅲ期11/1957.9%ⅳ期7/1070%所有患者24/4060%健康对照1/137.7%(特异性92.3%)
[0241]
表17
[0242]
crc患者lifr阳性/检测人数阳性率(%)ⅰ期0/20ⅱ期5/955.6%ⅲ期11/1957.9%ⅳ期4/1040%
所有患者21/4250.0%健康对照0/130(特异性100%)
[0243]
表18
[0244][0245]
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1