用于检测肺炎支原体的引物组、试剂盒及方法与流程
本发明属于分子生物学、体外诊断与检测领域,具体涉及一种用于检测肺炎支原体的引物组、试剂盒及方法。
背景技术:
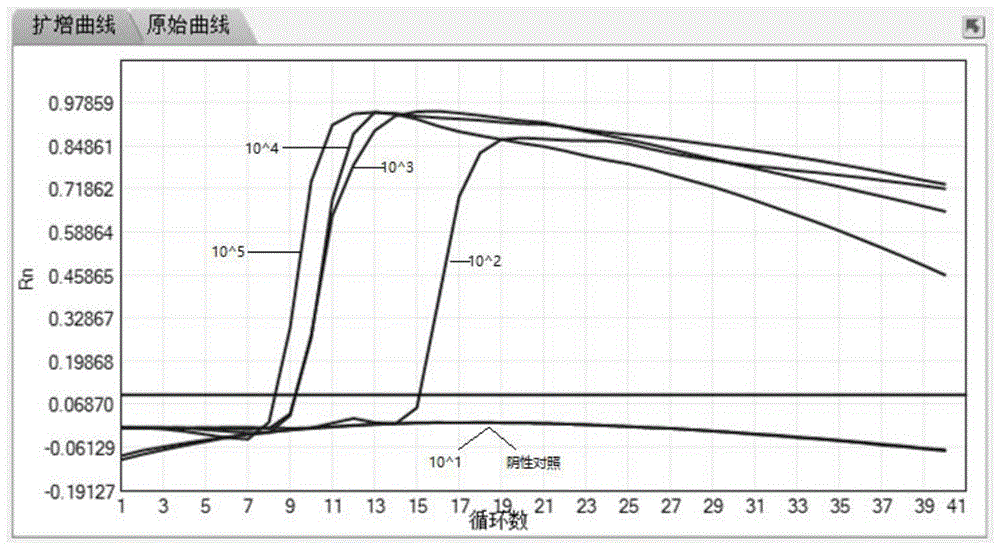
:肺炎支原体是社区获得性肺炎的主要病原体,是全球各地冬春季节的主要流行病原,具有高度的传染类,婴幼儿尤其易感,在中国人群的感染率较高,且近年感染发生率呈上升趋势。肺炎支原体感染可引起肺炎,甚至侵蚀肺外器官,严重者可导致死亡。呼吸道病原体种类很多,主要包括病毒类、细菌类、肺炎支原体、肺炎衣原体,此外还有一些属于原虫类和真菌类等等。不同病原菌的首选治疗药物和治疗方案并不相同,单就抗生素而言,因病原体差异首选有效抗生素完全不同,但不同病原体造成的病征有很大的相似度,难以区分,精准快速的区分病原体种类成为有效治疗疾病、控制疫情蔓延的关键,以争取尽早使用正确的药物,同时避免无效抗生素滥用。目前,国内肺炎支原体临床诊断,以“间接荧光法呼吸道九联检抗体检测”和“金标层析法的2-5联检抗原检测”为代表的免疫法产品为主,此外还有其他一些技术产品。从技术原理的角度,可以分为培养法、抗原免疫检测法、血清特异性抗体免疫检测法以及实时荧光定量pcr(实时荧光pcr、荧光定量pcr、real-timequantitativepcr、real-timeqpcr、qpcr、qpcr等)为代表的分子诊断法。但这些方法都有明显的技术缺陷、应用制约因素,比如,实时荧光定量pcr技术面临着核酸提取和检测操作复杂、仪器平台和操作人员要求高等一系列问题,成本高且使用场景有限。技术实现要素:针对现有技术中存在的上述不足,本发明提出一种基于lamp恒温扩增技术使用的引物组、试剂盒和方法,本发明创新性地开发无需纯化的一步法样本核酸抽提技术,免去繁琐的手工操作步骤;设计优选靶标特异性的引物组、开发优化反应配方,提高检测效率;开发优化冻干配方,制成可冷藏保存/常温运输的试剂盒,极大地拓宽了本发明产品应用场景。本发明使用的核心技术方法为环介导等温扩增技术(loop-mediatedisothermalamplification,lamp)是近年来发展起来的一种新型恒温核酸扩增方法,属于分子诊断方法的一种。lamp技术具有特异性强、反应时间短、样本耐受度高、操作便捷、硬件成本低等优点。该方法为日本荣研公司的专利技术,所涉专利包括cn100393875c、cn1222614c、cn100422323c等。lamp方法针对靶序列的8个区域设计6条特异性引物(包括上下游外引物f3和b3、上下游内引物fip和bip、上下游环引物lf和lb),利用一种具有链置换活性的bstdna聚合酶,在恒温条件保温<60min,即可完成核酸扩增反应,产生肉眼可见的反应副产物-白色焦磷酸镁沉淀,也可通过嵌入式染料进行更为精准的荧光定量。本发明人创造性地使用该技术,设计了特异性强、灵敏度高的引物组,并对反应体系中的各项条件进行了摸索。本发明通过以下技术方案实现:本发明第一方面涉及一种用于检测肺炎支原体的引物组,该引物组具有特异性强(对其他病原体没有检测活性)、灵敏度高、通用性好(种内各亚型通用)的特点。该引物组的具体序列如表1中seqidno:1至seqidno:6所示。表1引物序列表所述引物的设计过程如下所示:首先针对mp基因组进行同源性分析,识别出高度保守区域,利用优质高效的lamp引物设计软件lampdesigner1.16,设计lamp检测引物。本发明中,所述能扩增mp基因组的特异性碱基序列的引物组为genbanklr214945.1的mp基因组710397~710673bp位核酸序列的一部分或其互补链的一部分。其中,所述mp基因组共有特异性碱基序列是指仅为mp基因组所特有的,而其它微生物基因组所不包含的碱基序列。引物特异性分析:分别对上述引物组特异性进行理论分析,方法是将引物f3和b3间的序列与genbank数据库中的mp的基因组进行比对,确定检测区域,然后将检测区域序列在公共数据库资源中的非mp进行blast比对,检测引物区域与基因组序列匹配程度。若匹配程度越高,则特异性越差;若引物不能同时比对到非检测菌种菌株上,表明特异性好。结果发现,各引物组无引物比对到非mp的序列上,无法形成lamp扩增所需的引物匹配,表明各引物组的特异性均较好。引物通用性分析:分别对上述引物组通用性进行理论分析,方法是将引物f3和b3间的序列与genbank数据库中的mp的基因组进行比对,确定检测区域,然后将检测区域序列在公共数据库资源中的多个mp进行比对,检测引物区域与基因组序列的匹配程度,若引物区域完全匹配,则通用性好。结果发现,各引物组的引物区域与上述mp的多个基因组完全匹配,表明各引物组的通用性均较好。本发明第二方面涉及一种用于检测肺炎支原体的试剂盒,其包括反应体系,所述反应体系包含第一方面所述的引物组。较佳地,所述反应体系还包含dntp、bstdna聚合酶、tris-hcl、kcl、(nh4)2so4、tritonx-100、甜菜碱、mg2+、荧光染料。更佳地,所述反应体系包含:0.125-5mmol/ldntp;0.15-2u/μlbstdna聚合酶;10-100mmol/lph为7-9的tris-hcl;0-100mmol/lkcl;0-100mmol/l(nh4)2so4;0-1%tritonx-100;0-1.5mol/l甜菜碱;2-9mmol/lmg2+;0.15-0.3μmol/lmp外引物对;0.8-2.0μmol/lmp内引物对;以及0.15-0.6μmol/lmp环引物对。所述mg2+可为本领域常规,较佳地为mgso4或mgcl2。所述荧光染料可为sybrgreen、evagreen、syto-9以及syto-82中的任意一种。在本发明一具体实施方案中,所述反应体系包含:1.0mmol/ldntp;0.5u/μlbstdna聚合酶;20mmol/ltris-hcl(ph8.8);10mmol/lkcl;10mmol/l(nh4)2so4;0.1%tritonx-100;100×稀释的sybrgreen(neb);0.5mol/l甜菜碱;6mmol/lmgso4;0.2μmol/lmp外引物对;1.6μmol/lmp内引物对;0.4μmol/lmp环引物对。所述试剂盒较佳地为冻干粉的形式。较佳地,制备所述冻干粉所用保护剂为5%~20%蔗糖或海藻糖,优选10%海藻糖。作为优选的实施方案,所述冻干粉的冻干工艺为:将上述液态试剂混合物15~30ul分装至反应管中,然后置于超低温冻干机中,先将温度降至-40℃以下,抽真空,20~30小时,升华抽除水分,直到在管底形成小块状干粉,真空状态下盖盖,得到冻干后的干粉试剂管,取出后,于2~8℃冷藏保存。冻干粉形式的试剂混合物即为该试剂盒的核心成分。本发明第三方面涉及一种检测肺炎支原体的方法,所述方法包括以下步骤:(1)提取待检样品中的总dna;(2)以步骤(1)提取的总dna为模板,利用第二方面所述的试剂盒进行lamp扩增反应,得扩增产物;(3)结果检测;步骤(3)中所述检测为荧光检测、目视显色检测、浊度检测、电泳检测中的任意一种。较佳地,所述lamp扩增反应的扩增时间为20min、温度为60~65℃。作为优选的实施方案,提取待检样品病原体核酸的具体步骤为:a.取咽拭子/鼻拭子/鼻咽拭子,采样后,浸入配方优选的样本保存液中,涮洗5-10下,将浸出液80~100℃热裂解5-20分钟;b.室温静置1-2分钟冷却;c.低速离心机瞬甩,或用手甩,将液体收集至管底待用或置于2-8℃保存当天使用,长期保存置于-20℃以下,可将其作为后续lamp扩增的模板备用。第四方面本申请涉及第一方面所述的引物组或第二方面所述的试剂盒在制备用于诊断肺炎支原体的产品中的应用,所述产品适用于第三方面所述的方法。本发明实验过程,均使用精准确定核酸浓度的mp的基因组dna,或精准确定核酸浓度的与各引物组匹配的合成质粒,作为检测模板进行实验测试。所述与各引物组匹配的合成质粒合成和定量方法为:订制合成各引物组f3、b3区间的dna,并构建在质粒载体puc57上,转入大肠杆菌dh5α中,表达扩增质粒,并提取纯化质粒dna,然后采用核酸定量仪(od260/od280法),进行核酸浓度的精准测定,再根据质粒的分子量,计算拷贝数浓度,逐级稀释成10^9copies/ul~10^1copies/ul的标准品。所述基因组dna制备和定量方法为:采购atcc的mpfh株型(15531-ttr),按说明进行培养,并提取纯化病原体基因组dna,制备梯度稀释物,用qpcr方法,同时测试该基因组dna梯度稀释物和前述已定量的质粒梯度标准品,将质粒梯度标准品的浓度和起峰ct数据制备标准曲线,代入基因组dna梯度稀释物的ct数据,反算基因组dna浓度。所述质粒载体puc57、大肠杆菌dh5α为本领域常规。作为优选的实施方案,lamp扩增检测步骤为:将上述提取得到的待测样本核酸作为lamp的模板,直接加入检测管中,与上述冻干粉形式的试剂进行混合均匀,形成扩增反应体系,并置于恒温核酸扩增分析仪(例如实时荧光检测仪)的反应孔,在经历20min的恒定温度60~65℃下进行扩增,并在扩增过程中选择合适的荧光通道监控荧光信号的变化,记录实时荧光曲线。在扩增反应结束后,根据事先确定的荧光强度信号的变化率和荧光强度信号阈值对检测项目进行阴阳性结果判读,并实时显示在屏幕界面上。本发明方法中,反应体系中可用荧光染料,包括但不限于sybrgreen、evagreen、syto-9、syto-82等,使用荧光pcr仪(如abi7500)或其它恒温荧光检测仪进行检测,根据扩增曲线判断结果,有"s"型扩增曲线的为阳性,无"s"型扩增曲线的为阴性。本发明方法中,lamp扩增检测方法包括但不限于上述荧光检测,及下述目视显色检测、浊度检测或电泳检测等。所述目视显色检测,是在反应管中加入显色剂,包括但不限于钙黄绿素,或羟基萘酚蓝(hnb),等。当采用钙黄绿素或sybrgreen作为显色剂时,如反应后颜色为橙色,则待测样品为阴性;如反应后颜色为绿色,则待测样品为阳性。当采用羟基萘酚蓝作为显色剂时,如反应后颜色为紫罗兰色,则待测样品为阴性;如反应后颜色为天蓝色,则待测样品为阳性。所述显色检测,除了上述通过肉眼观察反应结果外,也可以通过检测仪器进行实时或终点检测反应结果,通过合理的设定阴性反应的阈值,当待测样品反应的结果低于或等于该阈值时,则待测样品为阴性;当待测样品反应的结果大于该阈值时,则待测样品为阳性。所述检测仪器包括但不限于荧光分光光度计、荧光定量pcr仪、恒温扩增微流控芯片核酸分析仪和等温扩增荧光检测系统等。所述浊度检测,是用肉眼观察或浊度仪检测浊度,检测管出现明显混浊,则待测样品呈阳性,含有mp;如未见混浊,则待测样品为阴性。也可以经离心后肉眼观察反应管底是否有沉淀,若反应管底有沉淀,则待测样品呈阳性;如反应管底没有沉淀,则待测样品呈阴性。所述电泳检测,优选为凝胶电泳检测法,可以是琼脂糖凝胶,也可以是聚丙烯酰胺凝胶。电泳检测结果中,如电泳图呈现特征性阶梯状条带,则待测样品呈阳性;如电泳图不呈现特征性阶梯状条带,则待测样品呈阴性。本发明的积极进步效果是:简易的样本核酸提取方法,摆脱了分子诊断样本需要大型核酸提取仪的高要求,也避免了传统手工提取试剂盒长耗时的劣势,大大增强客户使用的便捷性;恒温扩增技术,可在价格低廉的小仪器上使用,大大降低了分子诊断硬件方面的高要求和高成本;优选的引物序列,具有灵敏度高、检测准确、种内兼容各亚型(通用性)、种间排他(特异性)的优点;高效的引物组和反应液配方,反应速度快,耗时缩短至20分钟(最常用的实时荧光定量pcr技术耗时一般在40~60min),大幅提高了检测的精准度和检测速度;本发明的产品试剂盒可冷藏(2~8℃)保存、常温运输,摆脱了分子诊断试剂通常需要冷冻保存(-20℃)、冷链运输(-20℃或2~8℃)的弊端,特别适合医疗条件较差的低级别医疗检测单位使用。综上所述,该方法具有快速高效、操作简便、高特异性、高灵敏度、鉴定简便、适合现场检测等特点,能快速检测肺炎支原体dna。附图说明图1为肺炎支原体lamp扩增的灵敏度&扩增时间示意图。图2为lamp扩增体系冻干粉拍摄图。具体实施方式下面通过实施例的方式进一步说明本发明,但并不因此将本发明限制在所述的实施例范围之中。实施例1:lamp法扩增mp序列(1)扩增反应体系:1.0mmol/ldntp;0.2μmol/lmp外引物对;1.6μmol/lmp内引物对;0.4μmol/lmp环引物对;0.5u/μlbstdna聚合酶;20mmol/ltris-hcl(ph8.8);10mmol/lkcl;10mmol/l(nh4)2so4;0.1%tritonx-100;6mmol/lmgso4;0.5mol/l甜菜碱;100x稀释的sybrgreen。(2)扩增条件:置于恒温核酸扩增分析仪,65℃扩增20分钟。实施例2:lamp反应灵敏度检测目前市场上主流的分子诊断产品,技术原理多为qpcr,虽然被普遍认为灵敏度表现较好(100-500copies/反应),但受限于需要昂贵的变温荧光定量pcr仪、pcr循环升降温速度慢等弊端,推广一直受限。相比而言,等温lamp技术的特点恰好弥补了qpcr技术的弊端,但文献报道、市面上的lamp产品的灵敏度表现参差不齐,因此,lamp的推广使用瓶颈是能否通过引物序列优选、反应体系优化,确保lamp具有较高的检测灵敏度。如图1、表2,图中的s型曲线即为lamp扩增实验荧光曲线,s型意味着荧光信号增长,本质原理是随着扩增产物的增多,更多的sybrgreen等荧光染料嵌入双链产物中,相比原初的游离状态,荧光染料产生更强的发光性能,荧光信号增强。表2为图1的出峰时间数据汇总,即lamp反应经过多少分钟,荧光信号曲线开始进入s型曲线的上升阶段(noct表示lamp反应不起峰,即无扩增产物生成)。如图1、表2所示,本发明选用的引物组,检测灵敏度可达100copies/反应,优于文献报道、市面上的lamp产品的常规灵敏度表现(500-1000copies/反应),与市面上的qpcr产品的常规灵敏度表现相当。好的灵敏度表确保了临床样本检测的临床符合率,避免了检测病原体含量较低的样本时得出假阴性的结论(真阳性的病人样本,无法测出阳性结果,被误诊为阴性,即非染病)(见以下实施例9)。表2:肺炎支原体lamp扩增的灵敏度&扩增时间汇总表copies/反应起峰时间(mins)阴性对照noct10^1noct10^215.0710^39.1910^49.1710^58.23实施例3:反应耗时检测如图1、表2,及表3所示,本发明选用的引物组,20分钟内即可完成lamp扩增反应,大大优于文献报道、市面上的lamp/主流qpcr产品30-60分钟的耗时。表3:不同mp引物组的起峰时间比较实施例4:干燥的反应体系的储存稳定性和复溶效率检测如图2、表6和表7所示,本发明开发的lamp冻干工艺,干粉形态好,多种温度条件下,可稳定储存,至少可满足37℃7天、2~8℃180天、-20℃180天性能无明显损失,不影响产品的检测灵敏度和反应速度,实现冷藏保存、常温运输。实施例5:引物筛选过程mp引物设计如前文所述,本发明实验阶段,利用lampdesigner1.16软件共设计了3组引物,即表1中的引物组mp至引物组mp-3,并合成了文献报道的2套lamp引物组,即表1的引物组mp-4至引物组mp-5。各引物组分别在相同的反应体系配方中,检测相同的基因组dna梯度稀释物,通过灵敏度、反应时间两方面的性能表现,进行筛选。如表3所示,本发明选用引物组mp-引物组1表现最佳,灵敏度可达100copies/反应,反应耗时20min以内,100copies/反应及以上的反应均能正常出峰。mp-引物组4的灵敏度也可达100copies/反应,但反应耗时相对更长,mp-引物组2和mp-引物组5的灵敏度、反应时间两方面表现均不如mp-引物组1,mp-引物组3,虽然也有与mp-引物组1相近的灵敏度、反应时间表现,但阴性对照出峰,很可能是引物聚体的原因造成,极易造成假阳性结果,故排除。实施例6:酶用量筛选过程酶是lamp反应的核心物料,用量过少,反应效率低,用量过多,提高了成本。以mp优选引物组的为例,根据文献报道和酶供应商的推荐,优化筛选酶的合适用量。在相同的反应体系中,加入不同用量的酶,检测相同的基因组dna梯度稀释物,通过灵敏度、反应时间两方面的性能表现,进行筛选。本发明选用酶用量,即终浓度0.5u/ul浓度,表现最佳,灵敏度可达100copies/反应,反应耗时20min以内,所有的阳性反应均能正常出峰(如表4所示)。较低的用量0.125u/ul和0.25u/ul,灵敏度和反应耗时受到影响;较高的用量,1u/ul、2u/ul,也有相近的表现,但额外提高了成本。表4:bst酶浓度筛选实施例7:dntp浓度筛选过程dntp是dna的组成基本单位,是lamp反应的底物,反应体系中dntp浓度过低,反应效率低,浓度过高,可能提高成本、甚至与模板/引物形成配对,抑制反应。以mp优选引物组的为例,根据文献报道和酶供应商的推荐,优化筛选dntp的合适浓度。在相同的反应体系中,加入不同用量的dntp,检测相同的基因组dna梯度稀释物,通过灵敏度、反应时间两方面的性能表现,进行筛选。本发明选用dntp浓度,即终浓度1mm,表现最佳,灵敏度可达100copies/反应,反应耗时20min以内,所有的阳性反应均能正常出峰(如表5所示)。较低的用量0.25mm和0.5mm,灵敏度和反应耗时受到影响;2mm也有相近的表现,但额外提高了成本;4mm灵敏度和反应耗时表现不佳,可能的原因,如前文所述,dntp与模板/引物形成配对,抑制反应。表5:dntp浓度筛选实施例8:冻干保护剂筛选过程本发明实验阶段,根据文献报道,测试了多种分子反应试剂的冻干保护剂,如下表,通过对lamp的抑制作用、冻干后效价、干粉形态、储存稳定性等方面的检测(如表6和表7所示),最终选取了蔗糖和海藻糖作为优化冻干保护剂。并优化了使用浓度,优选为10%,浓度优化筛选过程与下表7相同,数据未展示。表6:冻干剂的筛选表7:lamp扩增体系冻干粉储存稳定性数据汇总表。实施例9:临床样本比对使用本发明产品,检测已使用市面上商售的nmpa获证产品确认过mp阴阳性的病人呼吸道临床样本,阴阳性符合率基本一致。仅一例比对系统鉴定为阳性的样本,使用本发明产品测试结果为阴性(如表8所示),但该样本使用文献报道的qpcr方法测试,结果同样为阴性(如表9所示),可能的原因是临床样本储存及冻融过程中,dna发生了降解,导致两种方法均无法成功检测。表8:lamp法对mp样本的检测结果与比对系统的比较表9:文献记载的qpcr法对mp样本的检测结果与比对系统的比较sequencelisting<110>上海奥普生物医药股份有限公司<120>用于检测肺炎支原体的引物组、试剂盒及方法<130>p20014349c<160>30<170>patentinversion3.5<210>1<211>21<212>dna<213>artificialsequence<220><223>f3_mp<400>1gtactgtgaaccacatgagtt21<210>2<211>21<212>dna<213>artificialsequence<220><223>b3_mp<400>2tagttcacattgtggtactgc21<210>3<211>42<212>dna<213>artificialsequence<220><223>fip_mp<400>3gttagggttggcagtccactttctagcttaactcagcaaccc42<210>4<211>43<212>dna<213>artificialsequence<220><223>bip_mp<400>4tgtggacaagtacactggtgccacccaaactaaggtcagtaat43<210>5<211>22<212>dna<213>artificialsequence<220><223>lf_mp<400>5cgtaagctgattgttccttgtg22<210>6<211>22<212>dna<213>artificialsequence<220><223>lb_mp<400>6ctcacctagttaacgttgacca22<210>7<211>18<212>dna<213>artificialsequence<220><223>f3_mp-2<400>7actctgtagcggtgacat18<210>8<211>20<212>dna<213>artificialsequence<220><223>b3_mp-2<400>8gtatttcaacaaggcttggg20<210>9<211>39<212>dna<213>artificialsequence<220><223>fip_mp-2<400>9tagcggatcattgctctgcgcaatgagtaccgcgaagtt39<210>10<211>39<212>dna<213>artificialsequence<220><223>bip_mp-2<400>10tggctaccttaaacggcaccggtagtgttccaagatggc39<210>11<211>20<212>dna<213>artificialsequence<220><223>lf_mp-2<400>11ccacaagaagaacaggacga20<210>12<211>18<212>dna<213>artificialsequence<220><223>lb_mp-2<400>12agcgattgaccgcatctt18<210>13<211>21<212>dna<213>artificialsequence<220><223>f3_mp-3<400>13gtactgtgaaccacatgagtt21<210>14<211>21<212>dna<213>artificialsequence<220><223>b3_mp-3<400>14tagttcacattgtggtactgc21<210>15<211>40<212>dna<213>artificialsequence<220><223>fip_mp-3<400>15gggttggcagtccactttcactagcttaactcagcaaccc40<210>16<211>43<212>dna<213>artificialsequence<220><223>bip_mp-3<400>16tgtggacaagtacactggtgccacccaaactaaggtcagtaat43<210>17<211>22<212>dna<213>artificialsequence<220><223>lf_mp-3<400>17cgtaagctgattgttccttgtg22<210>18<211>22<212>dna<213>artificialsequence<220><223>lb_mp-3<400>18ctcacctagttaacgttgacca22<210>19<211>19<212>dna<213>artificialsequence<220><223>f3_mp-4<400>19caccctcgggggcagtcag19<210>20<211>19<212>dna<213>artificialsequence<220><223>b3_mp-4<400>20ctgattgtccctgctggtc19<210>21<211>42<212>dna<213>artificialsequence<220><223>fip_mp-4<400>21ttcagagctggaggttggcttttgatgattacaggcggttcg42<210>22<211>43<212>dna<213>artificialsequence<220><223>bip_mp-4<400>22gggcggggtgaaggaatgatatttctcgtgaacttggtgtggt43<210>23<211>22<212>dna<213>artificialsequence<220><223>lf_mp-4<400>23aggcttcaagtggacaaagtga22<210>24<211>23<212>dna<213>artificialsequence<220><223>lb_mp-4<400>24gtcgagggttcttcgaggcgaac23<210>25<211>18<212>dna<213>artificialsequence<220><223>f3_mp-5<400>25atgcagtaccaccccgac18<210>26<211>20<212>dna<213>artificialsequence<220><223>b3_mp-5<400>26ccctcaccaaagacactgtt20<210>27<211>42<212>dna<213>artificialsequence<220><223>fip_mp-5<400>27cctcgtacgcctcattgacctcataagggtgagggtgagact42<210>28<211>41<212>dna<213>artificialsequence<220><223>bip_mp-5<400>28taagtgacaccgagaagcgtggacccagtttcatggaagcc41<210>29<211>20<212>dna<213>artificialsequence<220><223>lf_mp-5<400>29cgctttggccatgaagggtt20<210>30<211>25<212>dna<213>artificialsequence<220><223>lb_mp-5<400>30acttctcttcgttttgtttttggac25当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1