Keratin5-IRES-eGFP敲入的hESCs细胞系的构建及诱导分化方法
本发明属于分子生物学领域,具体涉及的keratin5-ires-egfp敲入的hescs细胞系的构建方法和应用。
背景技术:
人胚胎干细胞(humanembryonicstemcell,hescs)是早期胚胎或原始性腺中分离出来的一类细胞,它具有体外培养无限增殖、自我更新和多向分化的特性。在体外或体内环境,hescs细胞都能被诱导分化为机体几乎所有的细胞类型。
基底层角质形成细胞(basalkeratinocyte)位于表皮最底层细胞,目前认为该类细胞处于分裂活跃状态,经过分化最终变成角质层的无细胞核鳞状细胞,基底层角质形成细胞通过不断地自我更新得以维持表皮的屏障作用。而现有的技术方法难以将基底层角质形成细胞分离培养,使得对其的研究和应用受到限制。
细胞角蛋白(cytokeratin)是上皮细胞中发现的一大类细胞骨架蛋白,是构成生皮表皮、毛皮毛囊的主要蛋白质。角蛋白可保护上皮组织细胞免受损伤或压力。现已发现的角蛋白肽链超过20种,毛角蛋白和表皮角蛋白分别由不同的角蛋白肤链构成。其中角蛋白5(keratin5)发现在复层鳞状上皮细胞如舌粘膜,以及基底气管上皮细胞,基底层角质形成细胞,毛囊,皮肤的皮脂和汗腺,腔的乳腺细胞等均有表达,基底层角质形成细胞表达角蛋白14(keratin14)和角蛋白5(keratin5),因此角蛋白14和角蛋白5可作为基底层角质形成细胞的标志物。然而现有文献并没有报道相关的利用标志物对基底层角质形成细胞分离培养成功的技术。
技术实现要素:
本发明的目的是提供一种keratin5-ires-egfp敲入的hescs细胞系的构建方法及诱导分化方法,将该细胞系诱导分化为角质形成细胞后可标记keratin5阳性的表皮基底层角质形成细胞,从而通过细胞流式(flowcytometry,fcm)技术将其分选出来。
本发明解决技术问题的方案如下:
一种靶向keratin5终止密码子下游的sgrna,其具有seqidno.1所示的dna序列。
一对具有与目的基因keratin5下游断裂位点上下游相同序列的同源臂,包括与目的基因keratin5下游断裂位点上游相同序列的上游同源臂keratin5arm1,如seqidno.2所示,以及包括与目的基因keratin5下游断裂位点下游相同序列的下游同源臂keratin5arm2,如seqidno.3所示。
一种待重组入基因组靶位点的ires-egfp,其dna序列如seqidno.4所示。
一种打靶donor载体keratin5-ires-egfp-loxp-neo,其具有与目的基因keratin5下游断裂位点上游相同序列的上游同源臂keratin5arm1,以及与断裂位点下游相同序列的下游同源臂keratin5arm2,上游同源臂、下游同源臂之间是待重组入基因组靶位点的ires-egfp的dna片段,keratin5arm1的dna序列如seqidno.2所示,keratin5arm2的dna序列如seqidno.3所示,ires-egfp的dna序列如seqidno.4所示。
一种keratin5-ires-egfp敲入的hescs细胞系,其具有ires-egfp,dna序列如seqidno.4所示。
一种keratin5-ires-egfp敲入的hescs细胞系的构建方法。具体实施步骤如下:
(1)构建sgrna质粒。针对目的基因keratin5终止密码子下游设计并合成相应的sgrna,将sgrna室温退火后连入bsmbi单酶切后的pu6-efsgrna2.1scaffold载体中。筛选后获得sgrna质粒,确定最高切割活性的sgrna质粒,sgrna的dna序列如seqidno.1所示;
(2)构建靶向keratin5基因的打靶donor载体keratin5-ires-egfp-loxp-neo。包括针对目的基因keratin5断裂位点的上游设计429bp的上游同源臂keratin5arm1及下游设计386bp的下游同源臂keratin5arm2,其中上游同源臂keratin5arm1的dna序列见seqidno.2,下游同源臂的keratin5arm2的dna序列见seqidno.3。将keratin5arm1和keratin5arm2克隆至本实验室保存的donor载体中,得到带有同源臂的donor质粒,再将本实验室保存的ire-egfp连至带有同源臂的donor质粒中,得到打靶donor载体,命名为keratin5-ires-egfp-loxp-neo,其中ires-egfp的dna序列如seqidno.4所示。
(3)将sgrna质粒,cas9质粒,打靶donor载体共电转至hescs细胞,sgrna质粒、cas9质粒、打靶donor载体电转的数量比例为1:4:10。待细胞系稳定后,用1μg/ml的嘌呤霉素筛选2天,培养至细胞系稳定,通过有限稀释法将80个左右的克隆铺于96孔板中,培养半个月后,将挑取的单克隆细胞至24孔板进行培养后,收取细胞提取基因组dna,随后进行pcr测序初步筛出阳性单克隆细胞并命名为keratin5-ires-egfp#3;
(4)将初步鉴定为阳性单克隆细胞keratin5-ires-egfp#3培养至稳定后,电转的方法转入表达cre酶的质粒,pcmv-cre质粒,培养至细胞系稳定,通过有限稀释法将80个左右的克隆铺于96孔板中,培养半个月后,将挑取的单克隆细胞至24孔板进行培养后,收取细胞提取基因组dna,随后进行pcr测序筛出阳性克隆,此步骤为了删除单克隆细胞中除ires-egfp以外其它donor质粒上携带的元件,将阳性单克隆细胞命名为keratin5-ires-egfp#3-16;
一种诱导keratin5-ires-egfp敲入的hescs分化为基底层角质形成细胞的方法,
具体实施步骤如下:
在分化前3天,将得到的keratin5-ires-egfp#3-16细胞系铺在matrigel包被的六孔板中培养,其中加入的培养液为es培养基。在第0天时,将hescs细胞移至i型胶原蛋白包被的12孔板上培养,用含有1μmra和25ng/mlbmp4的dmem/f12的培养基维持培养,在第8-15天,用含有1μmra和25ng/mlbmp4的ksfm培养基培养,第15天后,采用含25ng/mlbmp4的ksfm培养基,持续培养至第30天,此时将hescs诱导成为表达绿色荧光蛋白的角质形成细胞。
一种keratin5-ires-egfp敲入的hescs细胞系在研究临床上表皮基底角质形成细胞异常的皮肤病的应用.
由于本发明构建的keratin5-ires-egfp敲入的hescs细胞系将绿色荧光蛋白报告基因定向整合至目标基因下游,通过诱导分化hescs细胞为角质形成细胞从而实现对keratin5阳性的人表皮基底角质形成细胞的标记,利用细胞流式(flowcytometry,fcm)技术将其分选出来。该细胞系的建立有利于筛选出keratin5阳性的基底层角质形成细胞,从而为临床上表皮基底层角质形成细胞异常的皮肤病研究提供细胞模型。
附图说明
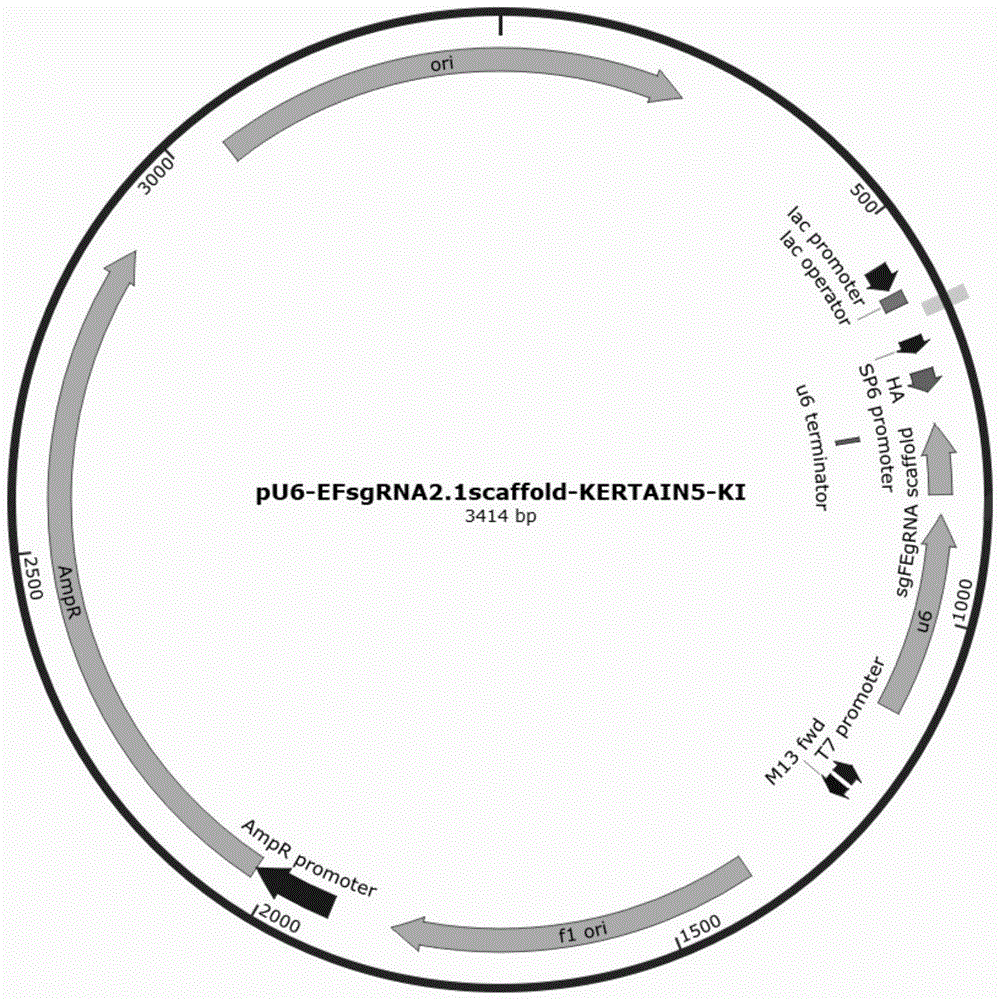
图1为sgrna质粒pu6-efsgrna2.1scaffold-keratin5-ki的示意图。
图2为打靶donor载体keratin5-ires-egfp-loxp-neo的示意图。
图3为阳性单克隆细胞keratin5-ires-egfp#3的pcr跑胶检测电泳图。
图4为阳性单克隆细胞keratin5-ires-egfp#3-16的pcr跑胶检测电泳图。
图5为阳性单克隆细胞keratin5-ires-egfp#3-16的sanger测序图。
图6为keratin5-ires-egfp敲入的hescs细胞诱导成为角质形成细胞,图中a为分化的流程示意图,b为分化后获得的es-kc的白光视野与角质形成细胞系hacat图,c为分化所得的es-kc荧光染色结果。
具体实施方式
下面将结合附图以及具体实施例来详细说明本发明,在此以本发明的示意性实施例及说明用来解释本发明,但并不作为对本发明的限定。
crispr/cas系统是目前发现存在于大多数细菌与所有的古菌中的一种后天免疫系统,其以消灭外来的质体或者噬菌体并在自身基因组中留下外来基因片段作为“记忆”。crispr/cas系统全名为常间回文重复序列丛集/常间回文重复序列丛集关联蛋白系统。crrna(crispr-derivedrna)通过碱基配对与tracrrna(trans-activatingrna)结合形成tracrrna/crrna复合物,此复合物引导核酸酶cas9蛋白在与crrna配对的序列靶位点剪切双链dna。而通过人工设计这两种rna,可以改造形成具有引导作用的sgrna(singleguiderna),足以引导cas9对dna的定点切割,然后通过细胞的非同源末端连接(non-homologousendjoining,nhej)和同源性重组(homologousrecombination,hr)两种修复途径进行修饰,从而实现基因靶向编辑。该技术称为“crispr/cas9技术”。
本发明通过crispr/cas9技术,将绿色荧光蛋白报告基因定向整合至目标基因下游,通过诱导分化hescs为角质形成细胞,实现对keratin5阳性的基底层角质形成细胞的标记,从而易于将其分选出来,具有方便快捷,直观简单的优点。
实施例1sgrna质粒的构建
针对目的基因keratin5的终止密码子下游设计并合成相应的sgrna,该项合成工作由广州睿博兴科生物技术有限公司完成,将合成的sgrna室温退火后连入bsmbi单酶切后的pu6-sgrna2.1scaffold载体(本实验室保存)中,筛选后得到sgrna质粒,pu6-efsgrna2.1scaffold-keratin5-ki,确定最高切割活性的sgrna质粒,得到seqidno.1所示包含sgrna的dna序列,sgrna质粒的结构见图1。
seqidno1:
ctagaagaggcaatctccat
实施例2打靶donor载体keratin5-ires-egfp-loxp-neo的构建
针对目的基因keratin5断裂位点的上游设计429bp的上游同源臂keratin5arm1及下游设计386bp的下游同源臂keratin5arm2,其中上游同源臂keratin5arm1的dna序列见seqidno.2,下游同源臂的keratin5arm2的dna序列见seqidno.3。将keratin5arm1和keratin5arm2克隆至本实验室保存的donor载体中,得到带有同源臂的donor质粒,再将本实验室保存的ire-egfp连至带有同源臂的donor质粒中,得到打靶donor载体,其如图2所示,命名为keratin5-ires-egfp-loxp-neo,其中ires-egfp的dna序列如seqidno.4所示。
seqidno2
ccaacaccagcagatcaaataaatgggccatgcaggatcagcctggcagatggtctcactgagtcctccctcctttccctgcagctgttgtcacaagcagtgtttcctctggatatggcagtggcagtggctatggcggtggcctcggtggaggtcttggcggcggcctcggtggaggtcttgccggaggtagcagtggaagctactactccagcagcagtgggggtgtcggcctaggtggtgggctcagtgtggggggctctggcttcagtgcaagcagtggccgagggctgggggtgggctttggcagtggcgggggtagcagctccagcgtcaaatttgtctccaccacctcctcctcccggaagagcttcaagagctaagaacctgctgcaagtcactgccttccaagtgcagcaacccagccca
seqidno3
tggagattgcctcttctaggcagttgctcaagccatgttttatccttttctggagagtagtctagaccaagccaattgcagaaccacattctttggttcccaggagagccccattcccagcccctggtctcccgtgccgcagttctatattctgcttcaaatcagccttcaggtttcccacagcatggcccctgctgacacgagaacccaaagttttcccaaatctaaatcatcaaaacagaatccccaccccaatcccaaattttgttttggttctaactacctccagaatgtgttcaataaaatgcttttataatataagctggtgtgcagaattgttttttttttctaggtttacaatcagcctagagataccatgagggtca
seqidno4
cccctctccctcccccccccctaacgttactggccgaagccgcttggaataaggccggtgtgcgtttgtctatatgttattttccaccatattgccgtcttttggcaatgtgagggcccggaaacctggccctgtcttcttgacgagcattcctaggggtctttcccctctcgccaaaggaatgcaaggtctgttgaatgtcgtgaaggaagcagttcctctggaagcttcttgaagacaaacaacgtctgtagcgaccctttgcaggcagcggaaccccccacctggcgacaggtgcctctgcggccaaaagccacgtgtataagatacacctgcaaaggcggcacaaccccagtgccacgttgtgagttggatagttgtggaaagagtcaaatggctctcctcaagcgtattcaacaaggggctgaaggatgcccagaaggtaccccattgtatgggatctgatctggggcctcggtacacatgctttacatgtgtttagtcgaggttaaaaaaacgtctaggccccccgaaccacggggacgtggttttcctttgaaaaacacgatgataatatggccacaaccatggtgagcaagggcgaggagctgttcaccggggtggtgcccatcctggtcgagctggacggcgacgtaaacggccacaagttcagcgtgtccggcgagggcgagggcgatgccacctacggcaagctgaccctgaagttcatctgcaccaccggcaagctgcccgtgccctggcccaccctcgtgaccaccctgacctacggcgtgcagtgcttcagccgctaccccgaccacatgaagcagcacgacttcttcaagtccgccatgcccgaaggctacgtccaggagcgcaccatcttcttcaaggacgacggcaactacaagacccgcgccgaggtgaagttcgagggcgacaccctggtgaaccgcatcgagctgaagggcatcgacttcaaggaggacggcaacatcctggggcacaagctggagtacaactacaacagccacaacgtctatatcatggccgacaagcagaagaacggcatcaaggtgaacttcaagatccgccacaacatcgaggacggcagcgtgcagctcgccgaccactaccagcagaacacccccatcggcgacggccccgtgctgctgcccgacaaccactacctgagcacccagtccgccctgagcaaagaccccaacgagaagcgcgatcacatggtcctgctggagttcgtgaccgccgccgggatcactctcggcatggacgagctgtacaagtaa
实施例3构建keratin5-ires-egfp敲入的hescs细胞系
将sgrna质粒,cas9质粒(购自addgene编号:#44719),打靶donor载体共电转至hescs细胞,其中sgrna,cas9质粒,打靶donor载体电转的数量比例为1:4:10。待细胞系稳定后,用1μg/ml的嘌呤霉素筛选2天,培养至细胞系稳定,通过有限稀释法将80个左右的克隆铺于96孔板中,培养半个月后,将挑取的单克隆细胞至24孔板进行培养后,收取细胞提取基因组dna,随后进行pcr测序初步筛出阳性单克隆细胞keratin5-ires-egfp#3,电泳结果见图3;
将初步鉴定为阳性单克隆细胞keratin5-ires-egfp#3培养至稳定后,电转的方法转入本实验室保存的pcmv-cre的质粒,待细胞系稳定后,通过有限稀释法将80个左右的克隆铺于96孔板中,培养半个月后,将挑取的单克隆细胞至24孔板进行培养后,收取细胞提取基因组dna,随后进行pcr测序,结果见图4,以及sanger测序,结果见图5,筛出阳性克隆,此步骤为了删除单克隆细胞中除ires-egfp以外其它donor质粒上携带的元件,将阳性单克隆细胞命名为keratin5-ires-egfp#3-16;
上述电转所用电转仪为ecm830型细胞电转仪,电转条件为300ma,4ms。
实施例4将hescs诱导成为角质形成细胞
在分化前3天,将得到的keratin5-ires-egfp#3-16细胞系铺在matrigel包被的六孔板中培养,其中加入的培养液为es培养基。在第0天时,将hescs细胞移至i型胶原蛋白包被的12孔板上培养,用含有1μmra和25ng/mlbmp4的dmem/f12的培养基维持培养,在第8-15天,用含有1μmra和25ng/mlbmp4的ksfm培养基培养,第15天后,采用含25ng/mlbmp4的ksfm的培养基,持续培养至第30天,此时将hescs诱导成为表达绿色荧光蛋白的角质形成细胞,操作条件见图6a,图6b为分化后获得的人胚胎干细分化获得的角质形成细胞系es-kc的白光视野与角质形成细胞系hacat图,人胚胎干细分化获得的角质形成细胞系分化所得的es-kc荧光染色结果见图6c。
以上对本发明实施例所提供的技术方案进行了详细介绍,本文中应用了具体个例对本发明实施例的原理以及实施方式进行了阐述,以上实施例的说明只适用于帮助理解本发明实施例的原理;同时,对于本领域的一般技术人员,依据本发明实施例,在具体实施方式以及应用范围上均会有改变之处,综上所述,本说明书内容不应理解为对本发明的限制。
序列表
<110>南方医科大学皮肤病医院(广东省皮肤病医院、广东省皮肤性病防治中心、中国麻风防治研究中心)
<120>keratin5-ires-egfp敲入的hescs细胞系的构建及诱导分化方法
<130>2020
<160>4
<170>siposequencelisting1.0
<210>1
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>1
ctagaagaggcaatctccat20
<210>2
<211>429
<212>dna
<213>人工序列(artificialsequence)
<400>2
ccaacaccagcagatcaaataaatgggccatgcaggatcagcctggcagatggtctcact60
gagtcctccctcctttccctgcagctgttgtcacaagcagtgtttcctctggatatggca120
gtggcagtggctatggcggtggcctcggtggaggtcttggcggcggcctcggtggaggtc180
ttgccggaggtagcagtggaagctactactccagcagcagtgggggtgtcggcctaggtg240
gtgggctcagtgtggggggctctggcttcagtgcaagcagtggccgagggctgggggtgg300
gctttggcagtggcgggggtagcagctccagcgtcaaatttgtctccaccacctcctcct360
cccggaagagcttcaagagctaagaacctgctgcaagtcactgccttccaagtgcagcaa420
cccagccca429
<210>3
<211>386
<212>dna
<213>人工序列(artificialsequence)
<400>3
tggagattgcctcttctaggcagttgctcaagccatgttttatccttttctggagagtag60
tctagaccaagccaattgcagaaccacattctttggttcccaggagagccccattcccag120
cccctggtctcccgtgccgcagttctatattctgcttcaaatcagccttcaggtttccca180
cagcatggcccctgctgacacgagaacccaaagttttcccaaatctaaatcatcaaaaca240
gaatccccaccccaatcccaaattttgttttggttctaactacctccagaatgtgttcaa300
taaaatgcttttataatataagctggtgtgcagaattgttttttttttctaggtttacaa360
tcagcctagagataccatgagggtca386
<210>4
<211>1307
<212>dna
<213>人工序列(artificialsequence)
<400>4
cccctctccctcccccccccctaacgttactggccgaagccgcttggaataaggccggtg60
tgcgtttgtctatatgttattttccaccatattgccgtcttttggcaatgtgagggcccg120
gaaacctggccctgtcttcttgacgagcattcctaggggtctttcccctctcgccaaagg180
aatgcaaggtctgttgaatgtcgtgaaggaagcagttcctctggaagcttcttgaagaca240
aacaacgtctgtagcgaccctttgcaggcagcggaaccccccacctggcgacaggtgcct300
ctgcggccaaaagccacgtgtataagatacacctgcaaaggcggcacaaccccagtgcca360
cgttgtgagttggatagttgtggaaagagtcaaatggctctcctcaagcgtattcaacaa420
ggggctgaaggatgcccagaaggtaccccattgtatgggatctgatctggggcctcggta480
cacatgctttacatgtgtttagtcgaggttaaaaaaacgtctaggccccccgaaccacgg540
ggacgtggttttcctttgaaaaacacgatgataatatggccacaaccatggtgagcaagg600
gcgaggagctgttcaccggggtggtgcccatcctggtcgagctggacggcgacgtaaacg660
gccacaagttcagcgtgtccggcgagggcgagggcgatgccacctacggcaagctgaccc720
tgaagttcatctgcaccaccggcaagctgcccgtgccctggcccaccctcgtgaccaccc780
tgacctacggcgtgcagtgcttcagccgctaccccgaccacatgaagcagcacgacttct840
tcaagtccgccatgcccgaaggctacgtccaggagcgcaccatcttcttcaaggacgacg900
gcaactacaagacccgcgccgaggtgaagttcgagggcgacaccctggtgaaccgcatcg960
agctgaagggcatcgacttcaaggaggacggcaacatcctggggcacaagctggagtaca1020
actacaacagccacaacgtctatatcatggccgacaagcagaagaacggcatcaaggtga1080
acttcaagatccgccacaacatcgaggacggcagcgtgcagctcgccgaccactaccagc1140
agaacacccccatcggcgacggccccgtgctgctgcccgacaaccactacctgagcaccc1200
agtccgccctgagcaaagaccccaacgagaagcgcgatcacatggtcctgctggagttcg1260
tgaccgccgccgggatcactctcggcatggacgagctgtacaagtaa1307
- 还没有人留言评论。精彩留言会获得点赞!