通过毛细管电泳(CE)DNA图谱对微卫星进行的比较分析的制作方法
通过毛细管电泳(ce)dna图谱对微卫星进行的比较分析
技术领域
1.本发明涉及一种基于毛细管电泳(ce)图谱中的偏移(ce图谱偏移)来确定至少一个微卫星不稳定性(msi)的方法,ce图谱偏移是通过至少一个微卫星的靶序列的毛细管电泳(ce)图谱(msi靶图谱)与其特定野生型序列的毛细管电泳(ce)图谱(ms野生型图谱)之间的比较来确定的。此外,本发明涵盖用于所述方法的合适的引物、包含用于成功执行所述方法的所有必要组件的试剂盒、作为系统的完全封闭的装置,即“msi modaplex分析系统”和用于诊断与炎症、癌症、炎症相关癌症和/或自身免疫疾病相关联的msi表型的方法,其中所述诊断包含用于确定如上所述的至少一种ce图谱偏移的方法。最后,本发明涉及改进的msi组用于确定和优选诊断msi肿瘤的用途,所述组由str生物标志物nr
‑
21、nr
‑
24、mono27、d2s123、d5s346、d17s250、bat
‑
25和bat
‑
26组成。
背景技术:
2.在现有技术中,公开了用于识别微卫星不稳定性(msi)的各种方法。ep1478781公开了一种评估与肿瘤相关的微卫星不稳定性的方法,其中扩增包含来自肿瘤的基因组dna的生物样品中的微卫星基因座,确定dna扩增产物的大小,然后比较肿瘤扩增产物的大小和正常扩增产物的大小。当肿瘤dna扩增产物的大小被确认为比相应的正常扩增产物短至少3bp或2bp时,评估msi。所述方法是针对生物标志物bat
‑
26、bat
‑
25、nr
‑
21、nr
‑
22、nr
‑
24和nr
‑
27公开的。所述方法的缺点是上述用于评估msi的解决方案的前提。wo02086448a2公开了一种msi检测方法及其与不同的生物标志物在肿瘤诊断中的用途。所述方法的缺点是使用者或医生必须处理使msi的诊断相对困难的残迹伪影(stutter artefact)。
技术实现要素:
3.因此申请人基于modaplex技术转而开发了一种基于实时pcr结合毛细管凝胶电泳的方法,一种克服缺点并结合更多生物标志物的新方法,用于有效确定msi,并有可能用于有效的诊断。此外,还提供一种全封闭装置。与现有技术相比的区别特征是msi的确定和所述msi的识别是基于在所述实时pcr期间获得的并且未解析为ce电泳图中的单分子峰的扩增子的迁移图谱来进行的。
4.根据本发明,不区分残迹伪影。因此,msi存在的证据不是识别的序列,也不是其大小[bp],而是在实时pcr和ce中,潜在的msi靶序列的扩增子的表现和相应的野生型序列的扩增子的表现。所述表现由在某些条件下通过毛细管的迁移来展示,并且所述迁移由相应的扩增子的图谱反映,如图1a和b所示。如果识别出图谱之间的偏移,则两种图谱的以下比较可以作为msi的证据。
[0005]
因此,本发明结合了实时pcr和毛细管电泳(与毛细管凝胶电泳同义)的优点,但没有对所获得的扩增子进行解析和测序到单个碱基。
[0006]
与现有技术相比,本发明的另一个区别特征是,在str的情况下,本发明的dna图谱包含真实str等位基因(对于杂合野生型至多2)的所有数据对(电泳分析物保留时间上的
rfu)的总和及其相应的残迹伪影。计算出的改变的str的dna图谱(靶图谱或靶modaplex图谱)只能与本文定义的野生型图谱或野生型modaplex图谱(例如,在突变情况下的野生型核酸)相比进行解释和量化。此外,现有技术已知的残迹伪影在例如图2a所示的本发明的图谱中没有被解析,使得使用者更容易确定和诊断msi。
[0007]
本发明的另一个优点是它提供了一个完全封闭的系统。pcr在限定的modaplex装置中进行,因此无需转移样品。因此,可以避免由于样品转移造成的污染。
[0008]
因为排序不是必需的,本发明的方法更快并且提供了更早的结果,为此只需要一个装置,即modaplex装置。本发明的方法避免了用于购买dna测序装置或用于样品的外部测序所投入的费用和时间。
[0009]
因此,本发明的目的是提供
[0010]
‑
快速确定msi中潜在突变的方法,其中避免测序,
[0011]
‑
封闭的自动操作系统,其中避免污染并避免各种单一(手动)步骤和手动操作时间,
[0012]
‑
用于执行上述限定方法的所有组件,所述组件还适用于一种装置,因此目的在于提供完全独立的系统,
[0013]
‑
另一个目的是提供用于不同msi的多重分析的方法、系统和装置,特别是用于研究、临床前研究和诊断,
[0014]
‑
进一步的目的是提供根据贝塞斯达组和修正的贝塞斯达组无需测序来确定msi的方法、引物、试剂盒和装置,
[0015]
‑
提供用于有效的msi确定和优选有效的msi肿瘤诊断的改进的生物标志物组和
[0016]
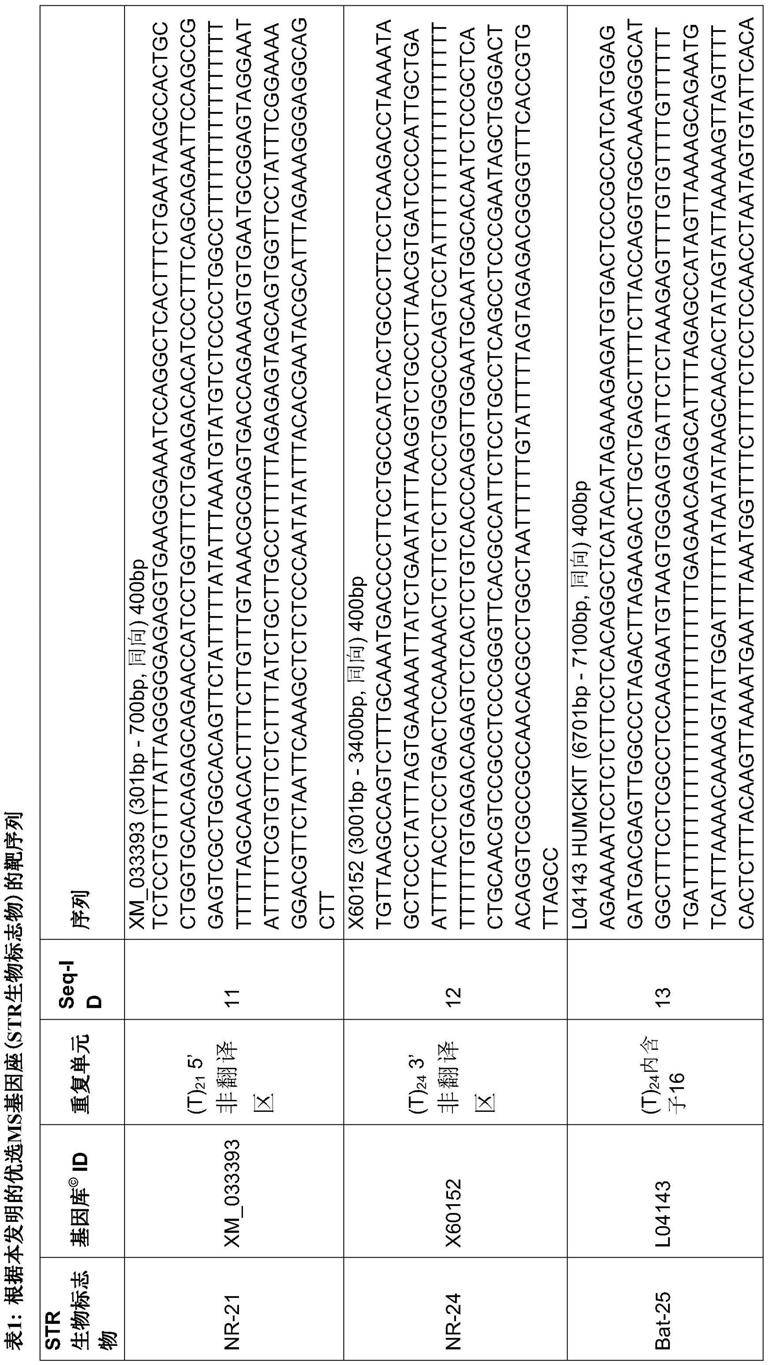
‑
用于确定与炎症、癌症、炎症相关癌症和/或自身免疫疾病相关的新str生物标志物的系统,特别是在研究和诊断中,和
[0017]
‑
易于操作的系统,包含方法、作为引物和对照的相关组件、装置和软件,以支持用于msi的重要分析和优选诊断的用途。
[0018]
因此,为了解决上述目的,本发明提供了如权利要求中所限定的并且在下面详细解释的主题。可行性由所选实施例示出,这些实施例不应被解释为限制保护范围。
[0019]
因此,根据本发明的解决方案的第一方面是一种用于基于毛细管电泳(ce)图谱的偏移(ce图谱偏移=图谱偏移)来确定至少一个微卫星不稳定性(msi)的方法,ce图谱偏移是通过至少一个微卫星的靶序列的毛细管电泳(ce)图谱(msi靶ce图谱=靶图谱)与其特定野生型序列的毛细管电泳(ce)图谱(ms野生型ce图谱=野生图谱)之间的比较来确定的,该方法包括以下步骤
[0020]
‑
通过实时pcr同时扩增至少一种靶序列和至少一种特定野生型序列,特别是代表至少一种str生物标志物,并在扩增期间标记其核酸,特别是实现带有标记或可检测的标记的扩增子,
[0021]
‑
在扩增中,实时通过毛细管电泳(ce)分离尤其是获得的扩增子的标记的核酸,尤其包括电动注射,并分别检测至少一种靶序列和至少一种野生型序列的荧光团标记的扩增子,
[0022]
‑
至少一种靶图谱和至少一种对应的野生型图谱的基于计算机程序的计算,
[0023]
‑
比较至少一个靶图谱和对应的野生型图谱,尤其为了直接比较,手动或优选地通
过合适的软件(计算机程序)自动进行获得的靶图谱和野生型图谱的叠加,和
[0024]
‑
确定靶图谱和对应的野生型图谱之间的ce图谱偏移,尤其由应用所述方法的人在视觉上进行或通过编写用于识别显着的ce图谱偏移的计算机程序进行。优选地,所述ce图谱偏移表示至少一个感兴趣的msi。
[0025]
优选地,至少一种微卫星不稳定性(msi)选自单核苷酸(bat
‑
25、bat
‑
26、nr
‑
21、nr
‑
24、nr
‑
22)、准单型(quasi
‑
monomorphic)的单核苷酸和/或二核苷酸(d5s346、d2s123和d17s250)重复。ce图谱偏移包括插入和删除。
[0026]
检测引物延伸产物(扩增子),其包含对应于每个样品特异性序列标签的可区分可检测的标记的寡核苷酸引物,其中所述分离核酸分子和所述检测掺入在扩增期间实时进行,并且其中所述检测允许在ce中实时进行所述扩增的监控。
[0027]
在一个优选的实施方案中,本发明的方法同时包括或分析至少三个反应设置,用于a)至少一种msi靶序列、b)至少一种ms野生型序列和c)校准序列(与modaplex校准品同义)。至少一个校准序列同时被扩增以定义扩增子检测的范围。
[0028]
更优选地,在本发明方法中同时应用/使用第四反应设置,该第四反应设置是如下定义的污染对照(与患者可追踪性对照同义)。本发明的污染对照,如本文所定义的,与法医学中的人类dna图谱相关并且已知。在本发明的含义内,污染对照满足了生物样品和所选str生物标志物的相应靶序列的可追踪性的功能。
[0029]
本发明将靶序列(与靶核酸序列同义)定义为待通过要求保护的技术或方法识别和/或定量的特定dna分析物。例如,靶序列可以位于用于扩增它的一对寡核苷酸引物的杂交区域之间(定义如下)。靶序列包括人类、动物和植物dna。特别地,靶序列包含对用于识别遗传分类或表征的医学适应症(疾病)的生物标志物或基因具有特异性的序列。靶序列是一种核酸序列,它特异于生物标志物,所述生物标志物例如用于在分析或诊断框架内对个体(法医、系谱学)、物种(例如病原体、转基因生物)、疾病(例如癌症)或其他生物特征的特定识别。所述靶序列包含在生物样品中或从生物样品中获得(生物样品=样本=从其自然环境中分离的并含有多核苷酸的生物材料)。
[0030]
如本文所用,野生型序列(与野生型生物材料同义)是感兴趣的生物标志物的未受影响的核酸序列。野生型序列可包含在生物样品或标准化生物库中或从其获得(在数字化野生型序列的背景下)。根据本发明的方法(与“modaplex
‑
野生型
‑
图谱”对比“modaplex
‑
野生型
‑
图谱”同义),野生型序列在此用于获得所述野生型序列(msi的生物标志物)的野生图谱作为比较“modaplex
‑
野生型
‑
ce图谱”与“modaplex
‑
靶
‑
ce图谱”二者并最后确定所述图谱之间的偏移(ce图谱偏移)的基础。在本发明的靶序列(分析物或生物标志物)中识别的偏移的情况下,可以识别并可能地定性msi(例如mss=微卫星稳定,msi
‑
l=微卫星不稳定性(msi)msi低,msi
‑
h=微卫星不稳定性msi高)。在一个特殊的实施方案中,至少一种感兴趣的生物标志物的野生型ce图谱被保存为野生型ce图谱数据库内的数字野生型ce图谱,该数据库保存在存储介质(微芯片/存储装置等)上。
[0031]
生物样品是靶序列的来源,也可以是野生型序列的来源。优选地,靶序列和野生型序列均来自同一生物供体(人、动物、植物)。生物样品转而从固体或液体活检获得或可以从固体或液体活检获得,并且包括主要或仅含有良性核酸的样本,否则是未受影响的或一般生物背景材料中与感兴趣的生物标志物不同或干扰感兴趣的生物标志物。如果生物样品是
人类或动物来源的,它可以通过显微解剖从显微镜下预先表征(病理学上)的ffpe组织或其他未受影响的固体或液体活检(例如血细胞相对于实体瘤)获得。生物样品还包括植物和植物dna的相应靶序列。
[0032]
样品包括具有生物量的任何可想到的源材料。优选地,根据本发明的样品是人类样品,其可再分为固体或液体活检的。当今人类突变诊断中最优先的实体活检是病理学中使用的福尔马林固定且石蜡包埋(ffpe)的组织。然而,本发明不限于这种固体样品类型。在恶性疾病(例如癌症)的情况下,ffpe组织以不同的比例包含疾病影响的细胞和良性细胞。两种细胞类型都可以通过显微解剖分离,后一种细胞类型可以用作参考生物材料。人细胞外循环液特别是血液、血浆、血清和/或淋巴液、消化液,尤其唾液、胃液、胰液和/或胆汁、分泌物和排泄物,尤其汗液、尿液、粪便、精液、阴道分泌物、泪液、鼻腔分泌物和/或母乳和/或另外的液体或分泌物,尤其是羊水、耵聍、脑液。液体活检还包含不同程度的来自背景和受影响细胞的核酸混合物。后者有时可以如现有技术中所述进行富集(例如通过细胞表面特异性单克隆抗体进行免疫分离)。
[0033]
生物标志物(与str生物标志物同义)定义为与生物体、病毒或生物功能相关的核酸分析物(靶序列)。生物标志物的例子是用于区分物种(例如病原体与宿主生物体)或物种的个体、品系或特征的这样的生物标志物。此外,生物标志物可能与微生物、人类、动物或植物的疾病(医学适应症)和/或生理状态相关。在人类的情况下,后者的相关性也被称为体外诊断装置的科学和/或临床证据。微卫星是对于各种医学适应症(炎症、癌症、炎症相关癌症和/或自身免疫疾病)或遗传改变有用的生物标志物。
[0034]
炎症包括由病毒、细菌或两者引起的与微生物相关的炎症。炎症还包括慢性炎症,例如胃肠道、上呼吸道和下呼吸道、哮喘、结节病、皮肤病、牛皮癣、风湿病的慢性炎症。优选地,与本文描述的医学适应症相关的单核苷酸、准单型单核苷酸和/或二核苷酸重复用作本发明方法内的生物标志物。
[0035]
微卫星(与str生物标志物同义)虽然指短序列重复(ssr)或短串联重复(str),但它是具有1
‑
10bp重复序列的dna基因组区域。通常,微卫星段可能由碱基替换(不完美的微卫星)或插入(中断的微卫星)破坏。有时,微卫星也可能由多于一个的重复类型组成(复合微卫星)(schloetterer 2000,vieira等人,2016)。它们频繁出现在所有生物体甚至病毒的蛋白质或必需rna(例如内含子或其他“沉默”基因组区域)的非编码dna区域中,并且在高等生物体或微生物菌株(>10
‑
20等位基因)中是高度多态性的。然而,其中一些,特别是具有一或两个核苷酸重复单位的影响str被描述为准单型,在限定的群体中产生一个或仅几个等位基因。多态性str已被广泛用作人工特征选择的遗传生物标志物(例如植物或动物育种;miah等人2013、merritt等人2015、zhao等人2018)、流行病学研究(klaassen 2009、chen等人2011)、法医学应用(例如遗传指纹、亲子鉴定;becker等人2007a、becker等人2007b、romos和vallone 2015)和造血干细胞移植后的嵌合分析(thiede等人2004)。此外,许多str因为它们与遗传疾病和基因调控功能的关联已被考虑作为生物标志物。在微卫星不稳定性(msi)分析的情况下,准单型str已被证明是非常有信息的(umar等人,2004年)。每个靶序列对一个定义和选定的微卫星具有特异性,而微卫星又可用作生物标志物。
[0036]
微卫星不稳定性(msi)已成为用于检测其与体细胞突变(str重复单元的丢失或增加)的关联的流行研究和诊断议题。具有单核苷酸和二核苷酸重复的str已被证明是最敏感
的,因为它们在自然界中准单型的存在。据报道,一些癌症如结直肠癌、前列腺癌、胃癌、子宫内膜癌、卵巢癌和乳腺癌与msi相关。它是由dna复制期间dna错配修复(mmr)缺陷(也称为复制错误、rer或错配修复缺陷、dmmr)引起的替代标志物,并且对转移性肿瘤的免疫检查点抑制剂(ici)治疗的功效显示出积极的预测价值(boland等人1998、boland和goel 2010、colle等人2017、svrcek等人2019)。此外,据报道,一些传染病(park等人2005)、神经系统疾病(brouwer等人2009)和肺部炎症性疾病(哮喘、慢性阻塞性肺病、结节病)和其他器官或系统性疾病(类风湿性关节炎、牛皮癣、翼状胬肉)与msi相关(samara等人,2006年)。这些识别的标志物可在医学诊断应用和/或个性化治疗中扮演重要角色,并且根据本发明用作生物标志物或str生物标志物。
[0037]
杂合性丢失(loh)是一种跨染色体事件,导致二倍体细胞的一个亲本基因组拷贝内的整个染色体生物标志物(例如基因、str或另一种遗传多态性)丢失。在野生型杂合性的情况下,种系或体细胞突变的分析结果是纯合的信号。在源自野生型和体细胞突变细胞的dna混合物的情况下,可以看到等位基因失衡(ai)。
[0038]
如本文所用,污染对照或患者可追踪性对照是至少一种额外的生物标志物,其在多重pcr中共同扩增以验证样品与个体患者的来源和一致性。此外,反应孔之间的交叉污染可以通过多个样品的平行处理进行监测。这可以通过使用患者(个体)特定的法医学条形码来实现,例如具有四核苷酸或五核苷酸重复的多态性str(becker等人2007a,becker等人2007b)、双等位基因缺失
‑
插入多态性(ep2271777b1)和/或性染色体性别特异性dna标志物,如amelxy(haas
‑
rochholz和weiler 1997)、y染色体str或y染色体功能基因(例如sry、tspy、dxys156、sts;butler和li 2014),它们通过多重pcr共同扩增,分析并存放在外部的外部数据处理系统中。替代的,可以在单个样品中加入人工dna条形码作为对照靶序列。
[0039]
如本文所用,术语扩增/扩增(或复制反应)是指产生特定多核苷酸序列的至少一个拷贝或增加特定多核苷酸序列的拷贝数或量的反应。扩增的产物也称为扩增子(=扩增产物)。例如,多核苷酸扩增可为使用聚合酶、一对寡核苷酸引物和具有确定的ph值的缓冲液和聚合酶的共底物(例如ntp或dntp、mgcl2、单价阳离子如k
+
或nh
4+
、pcr增强剂如n,n,n
‑
三甲基甘氨酸、二甲亚砜、蛋白质和核酸稳定剂,如牛血清白蛋白、聚氧乙烯(20)脱水山梨糖醇单月桂酸酯、海藻糖、1,4,5,6
‑
四氢
‑2‑
甲基
‑4‑
嘧啶羧酸以及杀生物剂例如叠氮化钠)的过程,该过程用于产生任何特定的多核苷酸序列。扩增可通过聚合酶链反应(pcr)的体外方法完成,该方法为本发明所属领域的专家所熟知的现有技术。
[0040]
多重(multiplex)描述了在一个反应设置中扩增(或复制)和确定多个靶核酸序列。多重是指至少两个,优选四个或更多个,六个或更多个,八个或更多个微卫星,它们在相同的反应设置中同时用相应的引物混合物进行分析,所述引物混合物适合特异性地扩增所有存在的靶序列(对各个微卫星(str生物标志物)具有特异性)。多重方法的例子是通过基因分型至少6个或更多,优选12个微卫星(短串联重复)的人类的基因指纹(法医学dna分析和亲子鉴定)、肿瘤中不同体细胞突变的差异诊断、通过检测特定病原体组和/或扩增核酸库(组)的器官特异性感染的阐明(例如肺、肠、性传播感染)。优选地,根据本发明的方法是多重方法,其可用于任何期望的类型的检测(等同于或类似于所述实施例)。在一个优选的实施方案中,用于诊断的方法被设计和验证为多重。
[0041]
modaplex检测系统或modaplex装置(以前也发布为iceplex或star技术;
ep1601780b1;hlousek等人,2012年)是一种自动化的高多重实时qpcr平台技术,它在集成系统解决方案中结合了pcr热循环仪和毛细管电泳(ce)装置(biotype有限公司,德累斯顿,德国)。ce的荧光检测单元包括至少一个,优选至少两个荧光激发源(气体激光器、二极管激光器、发光二极管)和至少一个合适的荧光检测通道。modaplex系统的当前光学配置由蓝色氩激光器(激发488nm)和红色发光二极管(激发最大637nm,带通
±
6.5nm)与500
‑
620nm和>644nm的两个荧光发射通道结合组成,以分别检测染料6
‑
羧基荧光素(6
‑
fam)和5'
‑
tye
tm
665(所有权属于integrated dna technologies有限公司,圣地亚哥,加州,美国)的相对荧光单位(rfu)。在两个pcr热循环间隔下,至少11倍地将标记的dna扩增子电动力地注入ce并进行定量,产生可用于核酸定量的扩增图。ce提供在单个反应管中至少多达50个生物标志物或靶核酸序列的多重扩增产物的分离。ce的计算分离能力至少≥5
‑
10bp,尽管dna扩增子的二级结构可能会进一步影响其在电泳过程中的表观运动,这是多重pcr设计必须考虑的。
[0042]
除了modaplex系统之外,目前还有其他毛细管电泳仪器可以支持本发明,尽管仅与单独的热循环仪装置组合并且仅用于定性终点检测。这些仪器的例子是thermofisher scientific有限公司(applied biosystems division,卡尔斯巴德,加州,美国)abigenetic analyzer系列仪器(例如310、3100、3500)、promega有限公司(菲奇堡,威斯康辛州,美国)spectrum和spectrum compact ce系统和agilent technologies有限公司(圣克拉拉,加州,美国)4200tapestation系统、agilent aati碎片分析仪和agilent 2100生物分析仪器。不排除其他仪器。然而,在这些情况下,多重应用的引物设计、检测标记和校准品必须适应这些仪器的技术能力特征(检测通道、电泳分离能力)。
[0043]
如本文所用,modaplex校准品是至少一种,优选两种或更多种共扩增的pcr产物,该modaplex校准品在毛细管电泳中在多重设置中的至少一个荧光通道中被检测,它们与感兴趣的分析物或生物标志物无关(无关的或人工的模板和引物)。它们通过modaplex仪器的分析软件限定了扩增子检测的范围。在本发明所述的实施例中,modaplex校准品mix 2(biotype有限公司,德累斯顿,德国)以1倍终浓度使用。它由人工模板(与人类dna无关)和相应的pcr引物对组成。在6
‑
fam和5'
‑
tye
tm
665荧光通道中,分别放大了三种不同大小的校准品。计算的扩增子大小为110bp、249bp、306bp。然而,表观长度可能有最大5bp的轻微不同,不影响检测分析。modaplex校准品还代表独立于模板的pcr对照,并且应该添加到所有样品、阴性和阳性对照孔中。替代的,pcr设置可以通过校准品以适当的终点浓度加入。在这种情况下,必须使用其他标志物作为独立于模板的pcr对照。modaplex校准品之间的区域可以再分为靶、等位基因或感兴趣的扩增子的特征调用的区域。但是,准确的扩增子大小调用是不可能的。
[0044]
如本文所用,引物是指能够退火到多核苷酸模板(选自靶序列或野生型序列)并提供3'
‑
oh端以产生核酸依赖的dna的聚合酶延伸产物的寡核苷酸分子(即dna或rna),所述dna的聚合酶延伸产物与多核苷酸模板互补。在单重扩增反应的情况下,一对寡核苷酸引物位于期望的核酸区域的侧面,特别是靶序列和野生型序列的侧面,并分别退火到正向和反向的多核苷酸链,所述寡核苷酸引物用于生成特定的扩增子。在多重反应的情况下,在一个反应设置中使用多个引物对。引物可以由天然ntp(核糖核苷酸三磷酸)或dntp(脱氧核糖核苷三磷酸)组成。然而,本领域技术人员已知多种化学修饰,尤其调节引物的退火温度和扩增反应的特异性,尤其是对于多重设置(关于核苷酸的核碱基、糖环和骨架连接的修饰;
bergstrom 2009、malyshev和romesberg 2015、anosova等人2016、houlihan等人2017年以及其中的参考资料)。为了通过modaplex毛细管电泳检测,每个引物对中的一个引物必须用仪器相容的荧光染料共价标记。此外,引物对中的一个引物可以在其5'端通过人工加尾序列进行修饰,以调节毛细管电泳中的迁移表现,从而在多重反应中明确分辨扩增子。这些尾部可以是人工核苷酸序列,优选具有高腺苷和/或胸苷含量(aa、tt、ta、at;它们对退火温度的影响较小),或pcr终止剂例如六甘醇部分(us6756205b2专利族),或例如扭曲的嵌入核酸构建块(schneider等人2012)单独或与进一步的5'端核苷酸延伸或其他化学修饰组合共价连接。
[0045]
如本文所用,标记或可检测的标记是指可用于提供可检测(优选可量化)信号的任何部分或分子。标记的核苷酸(例如dntp)或标记的寡核苷酸(引物)或多核苷酸(扩增子)是一种与可检测标记连接的核苷酸。在modaplex系统的情况下,术语连接优选包括共价键合的标记。优选地,对所需核酸(靶序列、野生型序列和/或污染对照)有特异性的引物对的一个引物在其5'端用与modplex系统的检测单元相容的荧光团共价修饰。合适的荧光染料包括荧光染料例如cy5、cy3、罗丹明和衍生物(例如德克萨斯红)、5'
‑
tye
tm
665(integrated dna technologies有限公司,圣地亚哥,美国加州)、荧光素和衍生物(例如6
‑
羧基荧光素,5
‑
溴甲基荧光素),荧光黄,iaedans,7
‑
me2n
‑
香豆素
‑4‑
乙酸酯,7
‑
oh
‑4‑
ch3‑
香豆素
‑3‑
乙酸酯,7
‑
nh2‑4‑
ch3‑
香豆素
‑3‑
乙酸酯(amca)、单溴双烷、芘三磺酸盐例如cascade blue和单溴甲基
‑
氨基双烷(见示例deluca,免疫荧光分析,in antibody as a tool,marchalonis等人,eds.,john wiley&sons有限责任公司,1982)。对于其他仪器(agilent technologies有限公司),使用选择性和非共价结合双链dna的荧光染料用作嵌入剂或小沟结合剂,如(ep0512334b1)或(sang和ren 2006)。
[0046]
在毛细管电泳(ce)技术中,残迹已被很好地表征用于pcr扩增子分析。它们尤其在str区域扩增期间通过taq dna聚合酶的滑链错位(根据复制滑移插入或删除一个或多个重复单元)而产生的。对于重复单元为1
‑
3bp的str,这些伪影的频率更高。因此,具有4或5bp重复的str是法医学应用的标准,以最小化残迹问题,同时保持扩增子大小足够小以进行法医学上的污渍分析(dna片段)。另一个ce残迹伪影是由taq dna聚合酶的独立于内在模板的核苷酸转移酶活性引起的,该酶产生3'
‑
腺嘌呤突出端。后一个问题可以在一定程度上通过延长的最终延伸pcr热循环步骤(modaplex技术不可能,因为定期注入ce)和非标记基因座特异性pcr引物的序列特异性人工5'拖尾来减少。
[0047]
本发明中定义的图谱或ce图谱是pcr扩增子的dna图谱,其在根据本发明的方法,特别是根据本文描述的modaplex技术的ce电泳图中未解析为单分子峰。在str的情况下,dna图谱包括所有数据对(电泳分析物保留时间上的rfu)、真正的str等位基因(对于杂合野生型dna最多2个)及其相应的残迹伪影的总和。改变的str的dna图谱(靶图谱或靶modaplex图谱)只能与如上定义的野生型图谱或野生型modaplex图谱(例如在突变情况下的野生型核酸)相比较来进行解释和量化。
[0048]
根据本发明的方法的另一方面是用于确定至少一个msi的自动化方法,其中至少扩增、分离和检测步骤,优选扩增、分离、检测和计算在封闭系统中自动进行。优选地,所有步骤自动执行并由软件控制和/或操作。更优选地,在输出介质上自动可视地呈现ce图谱和/或产生的ce图谱比较。输出介质包括数字数据格式的监视器和模拟格式的打印机。
[0049]
本发明的方法(也称为msi modaplex方法)由计算机程序自动控制和操作。所述程序保存在硬件内的处理器(微芯片等)上。所述硬件优选地是“msimodaplex装置”的一部分,其组合毛细管电泳部件以及计算机的所有部件并因此作为完全封闭的系统执行。优选地,msi modaplex装置满足用于体外诊断的医疗装置的所有必要规定。
[0050]
在优选的实施方案中,本发明的方法借助于“msi modaplex装置”来执行,该装置包括用于与外部“数据源”进行数字数据传输的接口(w
‑
lan、蓝牙、网络电缆、usb)。本发明含义内的“数据源”是同一网络内或第三方的另一个网络内的另一台计算机或存储介质。第三方包括生成、处理和/或存储患者相关数据的卫生组织,如健康保险公司、医院、医疗实践、研究机构、临床中心、医疗服务提供商和公共当局。
[0051]
第三方的另一个“数据源”可以提供与医学适应症相关的附加数据,针对该医学适应症,潜在的感兴趣的str生物标志物通过本发明的方法确定并且优选地通过本发明的诊断方法评估。所述医学适应症包括炎症、癌症、炎症相关癌症和/或自身免疫疾病,并且优选地与选自单核苷酸、准单型单核苷酸和/或二核苷酸重复中的至少一种str生物标志物相关(参见表2)。
[0052]
第三方的另一个“数据源”可以是lis(实验室信息系统)和/或his(医院信息系统)。因此,在用于诊断的方法中的评估考虑了来自第三方的相关数据,并基于本发明方法中识别的ce图谱偏移并基于与一般健康数据相关的附加数据来计算治疗组或建议,所述一般健康数据相关于患者的健康数据(心脏活动、其他风险)、病史以及其他生理参数和病理参数(来自成像方法(例如超声检查、x射线、正电子发射断层扫描等)、临床化学测量、其他基因分型的结果和治疗措施)。
[0053]
在一个实施方案中,用于确定的方法和诊断方法全部由一个计算机程序控制和操作。在另一个实施方案中,确定方法和诊断方法由两个不同且独立的计算机程序控制和操作,对此,从modaplex装置到另一台计算机的数据传输可为必要的(方法的不受位置限制的计算和比较)。
[0054]
在根据本发明的方法的另一方面,ms野生型序列代表未受影响的感兴趣的微卫星基因座,并且msi靶序列代表感兴趣的微卫星基因座,其由于错配修复系统中的突变而可能受到msi影响。在一个优选的实施方案中,两种序列均获自相同来源(患者、植物、动物),其中野生型序列来自来源(组织、血液、它们的混合物)的未受影响区域,该区域邻近/应该邻近/可以邻近可能受影响的、从中分离出靶序列的区域。
[0055]
上述优选实施方案中两个序列均从相同来源(人、动物、植物)获得,这在来源表现出由遗传赋予的基因倾向时是不适用的。在这种情况下,使用数字野生型序列或已经保存和可用的数字野生型ce图谱可能是感兴趣的。
[0056]
因此,在另一个实施方案中,野生型序列是数字序列或已经数字化的野生型ce图谱,前者是从真实来源(组织、血液、它们的混合物)的至少一个未受影响的区域获得的,后来被保存为数字野生型ce图谱。优选地,所述数字野生型ce图谱是从一个以上的原始来源获得的并且代表统计上显著的数字野生型ce图谱或感兴趣的特定ms基因座。优选地,数字ce野生型图谱适用于单核苷酸和准单型str生物标志物,其中所述数字ce野生型图谱基于考虑了各种野生型微卫星序列及其各种相应等位基因序列的重要群组(cohort)的验证记录。
[0057]
本质上,ms/msi ce图谱是获得的标记扩增子的dna图谱,其在ce电泳图中未解析为单分子峰,并且优选地,靶和/或ms野生型序列的相应的ce图谱包含所有真正的微卫星等位基因和相应的残迹伪影的峰区域的总和。
[0058]
在根据本发明的方法的另一方面,至少一个感兴趣的微卫星的msi靶谱图的偏移是与至少一个选择的微卫星相关的医学适应症的证据。因此,野生型序列(未受影响或未突变)和靶序列(可能有突变)的图谱的比较显示了所述序列之间的相对差异,其中差异是由细胞中的错配修复系统的突变导致的,所述细胞包括在如上定义的生物样品,从中分离靶序列。因此,在无法确定ce图谱偏移的情况下,靶序列可能不包含任何突变。替代的,所述图谱之间没有差异,因为由于遗传赋予的基因倾向,序列(靶对于野生型)是相同的。
[0059]
优选地,至少一种选择的微卫星是与炎症、癌症、炎症相关的癌症和/或自身免疫疾病相关且有特异性的str生物标志物。更优选地,特定str生物标志物选自至少一种单核苷酸、准单型单核苷酸和/或二核苷酸重复(见下表1)。
[0060]
[0061][0062]
根据著名的“贝塞斯达组”,两个单核苷酸(bat
‑
25和bat
‑
26)和三个二核苷酸(d2s123、d5s346和d17s250)重复微卫星被推荐用于检测结直肠癌中的msi,并根据哪些肿
瘤出现两个或更多不稳定标志物(如果测试更多标志物,则≥30
‑
40%)应定义为msi/msi
‑
h。根据本发明,提出了d2s123、d5s346、d17s250、bat
‑
25和/或bat
‑
26nr
‑
21、nr
‑
24、mono27的组合。
[0063]
在根据本发明的方法的另一方面,msi靶序列及其特定野生型选自包括nr
‑
21、nr
‑
22、nr
‑
24、nr
‑
27、d2s123、d5s346、d17s250、mono27、cat
‑
25、ht
‑
17、bat
‑
52、bat
‑
55、bat
‑
56、bat
‑
57、bat
‑
59、bat
‑
25和/或bat
‑
26的str生物标志物。上述生物标志物定义在表1中,每个被定义为微卫星基因座:nr
‑
21作为位于slc7a8基因5'非翻译区的21t重复;nr
‑
24作为位于锌指2基因3'非翻译区的24t重复;nr
‑
22作为位于推定跨膜前体蛋白b5基因3'非翻译区的22t重复;和/或nr
‑
27作为位于细胞凋亡蛋白1基因抑制因子的5'非翻译区的27a重复。
[0064]
对于高msi肿瘤,可能优选5种str生物标志物;bat
‑
26、bat
‑
40、mfdl5、apc和d2s123。apc和d2s123对低和高msi肿瘤的识别显示出最高的灵敏度。而bat
‑
26、bat
‑
40和mfdl5在检测高msi肿瘤方面表现出最具特异性的表现。d2s123、d5s346、d17s250、bat
‑
25和/或bat
‑
26、nr
‑
21、nr
‑
24、mono27。对于几乎所有单核苷酸重复(bat
‑
25、bat
‑
26、bat
‑
4o、52c10和soclo;表2),已知具有许多明显偏移的基因座,其中由bat
‑
26和bat
‑
4o扩增的单核苷酸重复显示最清晰的偏移。
[0065]
在根据本发明的方法的另一方面,msi靶序列及其特定ms野生型序列选自str生物标志物nr
‑
21、nr
‑
24、mono27、d2s123、d5s346、d17s250、bat
‑
25和/或bat
‑
26。本发明的方法也适用于报告msi的另一种适应症的新生物标志物。技术人员能够通过应用现有技术中的引物设计方法获得用于新生物标志物的合适引物。msi
‑
h定义为≥30
‑
40%,msi
‑
l定义为<30
‑
40%和mss=0%。
[0066]
在根据本发明的方法的另一方面,至少一个ce图谱偏移与炎症、癌症、炎症相关癌症和/或自身免疫疾病相关。对于良性疾病中的loh和msi的例子,例如光化性角化病、翼状胬肉、动脉粥样硬化、哮喘、慢性阻塞性肺病、结节病和特发性肺纤维化。对于loh和msi以及慢性炎症性疾病的例子,例如子宫内膜异位症、平滑肌瘤、子宫腺肌症、慢性溃疡性结肠炎、克罗恩病(不太常见)、结肠腺瘤性息肉、慢性胃炎、巴雷特食管、胰腺炎。对于ici通路和感染的例子,例如可导致癌症的急性或慢性感染、非癌症感染和败血症。
[0067]
本发明方法的另一个应用涉及临床前研究,其中基于ms基因座和其ms ce图谱中的可能的偏移来分析细胞毒性。
[0068]
在根据本发明的方法的另一方面,同时分析至少三个、优选至少四个或更多个不同的微卫星。一个优选的实施方案定义了一种基于毛细管电泳(ce)谱图中的偏移(ce谱图偏移=谱图偏移)确定至少一个微卫星不稳定性(msi)的多重方法,包括至少五个、六个、七个、八个、九个、10、大于或等于11、12、13、1、20或更多个各自不同str生物标志物的微卫星(表1、2)。遵循贝塞斯达指南,对str生物标志物d2s123、d5s346、d17s250、bat
‑
25和/或bat
‑
26进行分析。根据修正的贝塞斯达指南,分析str生物标志物nr
‑
21、nr
‑
24、mono27、bat
‑
25和/或bat
‑
26。在一个优选的实施方案中,分析了八种不同的str生物标志物,特别是选自d2s123、d5s346、d17s250、bat
‑
25和/或bat
‑
26 nr
‑
21、nr
‑
24、mono27。
[0069]
本发明的另一方面是通过本发明方法获得的至少一种靶序列的msi靶图谱和/或至少一种野生型序列的ms野生型图谱用于基于毛细管ce谱图的偏移确定至少一种微卫星不稳定性(msi)。优选地,对于从生物样品中识别的所选择的str生物标志物和对于患者,
msi靶图谱和/或ms野生型图谱分别是数字图谱。
[0070]
本发明的另一方面是至少一个引物对的用途,所述引物对具有对至少一个微卫星的至少一个靶序列及其特定ms野生型序列的特异性,或者适用于扩增至少一个微卫星的至少一个靶序列及其特定ms野生型序列,其中至少一个引物对的至少一个引物包含用于调节ce中迁移表现的人工加尾序列,并且该引物对中的至少一个引物包含荧光团,其中该人工加尾序列包含高腺苷和/或胸苷含量(aa、tt、ta、at),其对退火温度的影响较小,六乙二醇部分用作pcr终止剂,并且在us6756205b2中也有描述,或者,单独共价连接或与进一步的5'端核苷酸延伸结合共价连接的扭曲嵌入核酸结构块(描述于schneider等人,2021)或其他化学修饰。
[0071]
上述引物和引物对在基于毛细管ce图谱的偏移来确定至少一个微卫星不稳定性(msi)的方法、优选多重方法中用于扩增至少一种msi靶序列和/或ms野生型序列,其优选从生物样品中分离。
[0072]
在根据本发明的用途的另一方面,用于获得至少一种msi靶序列和至少一种ms野生型序列的标记的扩增子,所述扩增子可在本发明方法(优选多重方法)中检测,以基于如上所述毛细管ce图谱的偏移来确定至少一个微卫星不稳定性(msi)。
[0073]
在根据本发明的用途的另一方面,用于体外分析生物样品中包含的核酸,以基于如上所述毛细管ce图谱的偏移来确定至少一个微卫星不稳定性(msi)。
[0074]
本发明的引物使用使得技术人员能够借助如上所述的本发明方法获得msi靶图谱和ms野生型图谱。如此创建引物对使得所述引物实现在本发明的方法中的明显区分。在本发明的含义内,任何技术人员都可以通过使用现有技术方法来创建任何对如下表2中总结的至少一种str生物标志物具有特异性并适合于扩增如下表2中总结的至少一种str生物标志物的引物对。
[0075]
表2:本发明方法可应用的代表ms基因座的定义
[0076]
[0077][0078]
本发明的另一方面是对至少一个微卫星基因座(str生物标志物)的至少一个靶序列有特异性并适合扩增至少一个微卫星基因座(str生物标志物)的至少一个靶序列的引物对,所述微卫星基因座(str生物标志物)尤其选自表1,所述引物对可应用于或应用在用于基于毛细管电泳(ce)图谱的偏移(ce图谱偏移=图谱偏移)来确定至少一个微卫星不稳定性(msi)的方法中,优选本发明的方法,所述方法用于诊断与炎症、癌症、炎症相关的癌症以及/或自身免疫疾病相关的msi表型,其中本发明的引物对具有选自以下的序列
[0079]
‑
对于nr
‑
21 seq id no:1和seq id no:2,
[0080]
‑
对于nr
‑
24 seq id no:3和seq id no:4,
[0081]
‑
对于bat
‑
25 seq id no:5和seq id no:6,
[0082]
‑
对于bat
‑
26 seq id no:7和seq id no:8,和/或
[0083]
‑
对于mono27 seq id no:9和seq id no:10或上述对的任意组合。
[0084]
表3:根据本发明的引物对的序列(msi多重pcr)
[0085]
[0086][0087]
通过应用引物设计方法的状态,技术人员能够创建或设计用于本发明方法的另外的合适引物对并且其对表2的至少一种str生物标志物具有特异性并且适合于扩增表2中的至少一种str生物标志物。
[0088]
本发明的另一方面是一种用于根据如上定义的方法的分析、特别是确定至少一个ce图谱偏移的试剂盒,其中ce图谱偏移是通过至少一个微卫星的靶序列的电泳(ce)图谱(msi靶ce图谱=靶图谱)及其特定野生型序列的毛细管电泳(ce)图谱(ms野生型ce图谱=野生型图谱)之间的比较来确定的,该试剂盒(也称为“msi modaplex分析试剂盒”)包括
[0089]
‑
至少一个引物对,该引物对具有对待分析的至少一个微卫星的至少一个靶序列的特异性,并且适用于扩增待分析的至少一个微卫星的至少一个靶序列,其中至少一个引物对的每个引物对包含人工加尾序列,并且引物对中的至少一个引物包含荧光团,如上文中定义的,
[0090]
‑
至少一个校准序列(如上定义的校准品的同义词),
[0091]
‑
至少一个引物对,该引物对具有对至少一个校准序列的特异性,并适用于扩增至少一个校准序列,和
[0092]
‑
污染对照。
[0093]
在本发明的另一方面,该试剂盒包含至少一对引物,所述引物对对至少一个ms基因座具有特异性并适合扩增该至少一个ms基因座,所述至少一个ms基因座选自nr
‑
21、nr
‑
24、mono27、d2s123、d5s346、d17s250、bat
‑
25和/或bat
‑
26。优选地,根据本发明的试剂盒的引物对具有以下seq id:对于nr
‑
21为seq id no:1和seq id no:2,对于nr
‑
24为seq id no:3和seq id no:4,对于bat
‑
25为seq id no:5和seq id no:6,对于bat
‑
26为seq id no:7和seq id no:8,对于mono27为seq id no:9和seq id no:10。
[0094]
进行pcr所需的其他组分优选是上述试剂盒的一部分,并选自合适的引物混合(如上定义)、pcr缓冲液、增强剂、热稳定dna聚合酶、至少一种modaplex校准品的核酸序列的可分量,优选至少一个污染对照和无核酸水。
[0095]
对于根据本发明的成功方法,将大于或等于0.5ng至小于或等于5ng的核酸添加到反应装置中。优选地,添加2ng核酸。
[0096]
本发明的试剂盒包含用于成功的msi modaplex分析、优选msi体外诊断、至少对于str生物标志物bat
‑
25、bat
‑
26、nr
‑
21、nr
‑
24、mono27、d2s123、d5s346和/或d17s250的所有必需试剂。并且借助于modaplex装置使用并在此描述。此外,本发明的试剂盒使技术人员能
够通过使用合适的软件,优选moda
‑
ra软件获得ce图谱,该软件基于从根据本发明的方法的扩增、分离和检测中获得的原始数据计算本发明的ce图谱。
[0097]
在如上定义的本发明试剂盒的另一个实施方案中,其被设计用于确定至少一个ms基因座(str生物标志物)的靶序列中的msi偏移,该ms基因座(str生物标志物)优选与炎症、癌症、炎症相关癌症和/或自身免疫疾病相关,如上定义。
[0098]
本发明的另一方面是如上定义的引物对,或如上定义的试剂盒,用在通过自动化ce装置(优选自动化modaplex系统)进行的自动化毛细管电泳(ce)中、优选用在本文所述的用于基于毛细管电泳(ce)图谱中的偏移(ce图谱偏移=图谱偏移)来确定至少一个微卫星不稳定性(msi)的方法中或用于如下所述的诊断的方法中,所述ce图谱偏移是通过至少一个微卫星的靶序列的毛细管电泳(ce)图谱(msi靶ce图谱=靶图谱)及其特定野生型序列的毛细管电泳(ce)图谱(ms野生型ce图谱=野生型图谱)之间的比较来确定的。
[0099]
因此,本发明的另一方面是一种优选用于通过计算机程序自动控制和操作的方法,诊断与炎症、癌症、炎症相关癌症和/或自身免疫疾病相关的msi表型,其中所述方法包括确定至少一个ce图谱偏移,优选基于毛细管电泳(ce)图谱中的偏移(ce图谱偏移=图谱偏移)来确定至少一个微卫星不稳定性(msi),所述ce图谱偏移是通过至少一个微卫星的靶序列的毛细管电泳(ce)图谱(msi靶ce图谱=靶图谱)及其特定野生型序列的毛细管电泳(ce)图谱(ms野生型ce图谱=野生型图谱)之间的比较来确定的,如上所述。
[0100]
在根据本发明的诊断方法的一个实施方案中,还包括计算msi分数的步骤,该msi分数代表所选ms基因座的所识别偏移程度。
[0101]
在根据本发明的诊断方法的另一个实施方案中,该方法还包括以下步骤:
[0102]
‑
评估,尤其基于计算机程序,通过至少在从患者获得生物样品时、优选ce图谱被识别时考虑与患者的健康状况相关的进一步的患者相关数据(优选该数据代表健康状况进展)来评估识别的如上所述的ce图谱偏移和/或计算的msi分数,
[0103]
‑
生成(尤其自动和基于计算机程序的)考虑并呈现了所有考虑的数据的诊断报告(优选地在输出介质(监视器/打印机)上视觉呈现),优选地,所述诊断报告以数字方式生成和/或经相应网络系统的接口自动传输给指定的医生或其他接收者,并且优选
[0104]
‑
呈现针对炎症、癌症、炎症相关癌症和/或自身免疫疾病的预防或治疗的定制治疗计划建议,优选地这是自动生成的诊断报告的一部分。
[0105]
对于任何医学适应症的有效诊断,必须考虑相同ms基因座(str生物标志物)的不同等位基因的变异范围。这同样适用于表现出由遗传赋予的基因倾向的患者(动物或人类)。在这种情况下,无法从患者获得ms野生型序列和相应的ms野生型图谱。因此,在这种情况下,对于感兴趣的ms基因座(str生物标志物)使用数字野生型ce序列或已经保存和可用的数字野生型ce图谱可能是感兴趣的。
[0106]
优选地,所述数字野生型ce图谱是从一个以上的原始来源获得的并且代表统计上显著的数字野生型ce图谱或感兴趣的特定ms基因座。优选地,数字ce野生型图谱适用于单核苷酸和准单型str生物标志物,其中所述数字ce野生型图谱基于考虑了各种野生型微卫星序列及其各种相应等位基因序列的重要群组的验证记录(goel等人2010)。
[0107]
本发明的另一方面是一种装置,特别是“msi modaplex装置”,包括:至少一个激发源,该激发源是可变的但优选选自气体激光器、二极管激光器、发光二极管;和至少一个检
测器,用于检测荧光团标记的核酸,特别是在上述方法中获得的标记扩增子的核酸;至少一个处理器;至少一个微芯片;和至少一个接口用于数字数据传输,其中处理器包括至少一个程序,其控制和操作用于确定msi ce图谱偏移的方法和/或用于诊断msi表型的方法。
[0108]
ce的所有必要组件都存在于上述装置中,因此提供了一个完整且完全封闭的系统。优选满足用于体外诊断的医疗器械的所有必要规定。
[0109]
在优选的实施方案中,本发明的方法借助于“msimodaplex装置”来执行,该装置包括用于与外部“数据源”进行数字数据传输的接口(w
‑
lan、蓝牙、网络电缆、usb)。本发明含义内的“数据源”是同一网络内或第三方的另一个网络内的另一台计算机或存储介质。第三方包括生成、处理和/或存储患者相关数据的卫生组织,如健康保险公司、医院、医疗实践、研究机构、临床中心、医疗服务提供商和公共当局。
[0110]
第三方的另一个“数据源”可以提供与医学适应症(遗传性非息肉病结直肠癌(hnpcc)、散发性(13
‑
28%)和遗传性非息肉病(79%)crc)相关的附加数据,针对该医学适应症,潜在的感兴趣的str生物标志物通过本发明的方法确定并且优选地通过本发明的诊断方法评估。所述医学适应症包括炎症、癌症、炎症相关癌症和/或自身免疫疾病,并且优选地与选自单核苷酸、准单型单核苷酸和/或二核苷酸重复中的至少一种str生物标志物相关(参见表2)。
[0111]
第三方的另一个“数据源”可以是lis(实验室信息系统)和/或his(医院信息系统)。因此,在诊断方法中的评估考虑了来自第三方的相关数据,并基于本发明方法中识别的ce图谱偏移并基于与一般健康数据相关的附加数据来计算治疗组,所述一般健康数据相关于患者的健康数据(心脏活动、其他风险)、病史以及其他生理参数和病理参数(来自成像方法(例如超声检查、x射线、正电子发射断层扫描等)、临床化学测量、其他基因分型的结果和治疗措施)。
[0112]
在诊断方法的另一个实施方案中,另外地监测患者,其中用通信连接到数据网络的处理器从连接到数据网络的社交网络服务接收与患者相对应的非医学数据;用处理器识别从社交网络服务接收的非医疗数据中患者的健康特征;用处理器生成包括与所识别的患者健康特征相关联的健康建议的消息;并且用处理器通过数据网络将消息发送到与患者相关联的电子装置。
[0113]“modaplex装置”包括用于与外部“数据源”进行数字数据传输的接口(w
‑
lan、蓝牙、网络、usb)。这里的“数据源”是指同一网络或第三方的另一个网络内的另一台计算机或存储介质。第三方可以是生成、处理和/或存储患者相关数据的卫生组织,包括健康保险公司、医院、医疗实践、研究机构、临床中心、医疗服务提供商和公共当局。
[0114]
根据本发明的方法适用于预测(转移性)肿瘤的ici疗效功效(boland等人,1998,current clinical guidelines),适用于确定非癌症疾病的msi和loh(samara等人,2006年),适用于通过细胞培养物或实验室动物确定临床前毒性和致突变性测试(chen等人2000,beal等人2015),且适用于菌株特异性病原体图谱的传染物流行病学的分析(chen等人2011)。
具体实施方式
[0115]
预先表征为结肠癌阳性的福尔马林固定的石蜡包埋(ffpe)组织交叉的配对(疾病
和正常)获自indivumed有限公司(德国汉堡)。获得了患者的书面知情同意。使用dna ffpe tissue试剂盒(qiagen有限公司,德国希尔德)来分离dna。dna的品质、数量和浓度是用qubit
tm
3.0荧光计(thermofisher scientific,沃尔瑟姆,美国马萨诸塞)测定的,该荧光计用制造商的标准dna校准。建立五重pcr,它包含五个准单型单核苷酸str标志物nr
‑
21、nr
‑
24、bat
‑
25、bat
‑
26和mono
‑
27,如bacher等人(2004)提出的,其支持用于胃肠道肿瘤msi诊断的修正的贝塞斯达指南(umar等人,2004)。modaplex多重分析中使用的引物在表3和序列表中进行了描述。当0、1
‑
2和3
‑
5个或更多的准单型单核苷酸str标志物在视觉上被解释为分别受到影响时,修正的贝塞斯达指南将样品分别定义为mss(微卫星稳定)、msi
‑
l(不稳定低)和msi
‑
h(不稳定高)。
[0116]
多重pcr由总体积为25μl的一倍modaplex缓冲液3(包含tris/hcl缓冲液ph 8.0、250mm dntp、单价阳离子、增强剂和稳定剂;biotype有限公司,德累斯顿,de)、4mm mgcl2、1单位modaplex taq dna聚合酶t(biotype有限公司,德累斯顿,德国)、0.5
‑
1.0μm的每个引物对和2ng基因组dna组成。所有反应、样品、阴性和阳性对照进一步包括终浓度为1倍的modaplex校准品mix2。
[0117]
pcr在没有盖和没有盖加热的modaplex检测系统内进行,以允许周期性注入毛细管电泳。因此,反应孔最后用一滴矿物油覆盖以避免蒸发。
[0118]
根据制造商的说明来设置自动modaplex检测系统。使用三步(每步40秒)热循环方案,其中dna变性步骤为96
‑
98℃,dna退火步骤为54
‑
64℃,dna延伸步骤为72℃。在pcr过程中,扩增产物被电动注入并在自动毛细管电泳中分离。从pcr循环19开始到pcr循环41结束,在延伸步骤结束时每隔一个循环周期性进行电泳。收集原始数据并将其导入modaplex result analyzer(moda
‑
ra)软件,用于显示dna图谱,其随后用于由针对msi分析临床指南推荐的视觉分析。后一种软件可以自动叠加匹配的患者样本对的扩增子图谱。在图1和2中,在pcr循环39结束时str扩增子的dna图谱用虚线表示为正常组织(野生型序列),且用直通线表示匹配的疾病组织(靶序列)。需要强调的是,图1a
‑
b和2a
‑
b的绘制图并不代表相应扩增子的实际大小或长度。x轴[bp]的定义基于如上定义的校准物计算,也可以表示为相对于校准物的百分比或保留时间。因此,与校准品相比,只能估计每个扩增子的相对大小。
[0119]
根据信号强度,其他pcr循环的原始数据可用于概览或呈现单个生物标志物。该软件使得能够获得对多重pcr结果的概览的第一眼(图1,a和b)。然后可以通过单击计算机鼠标单独放大str特异性检测区域。这样做时,生物标志物图谱的相对荧光单位(rfu,y轴)被归一化,以获得弱扩增和强扩增str的可比的呈现(见图2,a和b)。这有助于根据当前临床指南进行可视化数据分析。
[0120]
如图所示,患者样本可以清楚地解释为msi
‑
h(高msi,ulmar等人,2004年),因为5个标志物中的5个可以被分析为受msi影响。
附图说明
[0121]
图1:msi多重pcr的6
‑
fam标记的(图1a)和5'
‑
tye
tm
665标记的(图1b)的生物标志物的电泳图。
[0122]
ffpe组织交叉的匹配对(疾病组织以实线表示,正常组织以虚线表示)的原始数据用modaplex result analyzer(moda
‑
ra)软件叠加,并可视化为dna图谱。用于检测生物标
fragments using a new fluorescence intercalating dye evagreen.j sep sci 29:1275
‑
80。
[0152]
c(2000).evolutionary dynamics of microsatellite dna.chromosoma 109:365
‑
71。
[0153]
schneider uv,mikkelsen nd,lindqvist a,等人(2012).improved efficiency and robustness in qpcr and multiplex end
‑
point pcr by twisted intercalating nucleic acid modified primers.plos one 7:e38451。
[0154]
svrcek m,lascols o,cohen r,等人(2019).msi/mmr
‑
deficient tumor diagnosis:which standard for screening and for diagnosis?diagnostic modalities for the colon and other sites:differences between tumors.bull cancer 106:119
‑
128。
[0155]
thiede c,m,ehninger g(2004).evaluation of str informativity for chimerism testing
‑
comparative analysis of 27str systems in 203matched related donor recipient pairs.leukemia 18:248
‑
54。
[0156]
umar a,boland cr,terdiman jp,等人(2004).revised贝塞斯达guidel ines for hereditary nonpolyposis colorectal cancer(lynch syndrome)and microsatellite instability.j natl cancer inst 96:261
‑
8。
[0157]
vieira ml,santini l,diniz al,munhoz cde f(2016).microsatellite markers:what they mean and why they are so useful.genet mol biol 39:312
‑
28。
[0158]
zhao j,li t,zhu c,等人(2018).selection and use of microsatellite markers for individual identification and meat traceability of six swine breeds in the chinese market.food sci technol int 24:292
‑
300。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1