条码化核酸用于检测和测序的方法与流程
条码化核酸用于检测和测序的方法
1.本专利申请要求2019年6月4日提交的临时文件us62857096和2019年7月19日提交的临时文件us62876455的优先权。其整体引入本文。本文中述及的所有出版物、专利和其他文件都通过引用全文纳入本文。
技术领域
2.本发明涉及用于改进的核酸检测和测序的一般方法。
背景技术:
3.本发明属于基因组学的技术领域。更具体地,本发明属于核酸测序的技术领域。核酸测序可以为很多种生物医学应用提供信息,包括诊断、预后、药物基因组学和法医生物学。测序可能涉及基础的低通量方法,包括maxam-gilbert测序(化学修饰的核苷酸)和sanger测序(链终止)法,或高通量的下一代方法,包括大规模平行焦磷酸测序、合成测序、连接测序、半导体测序和其他。对于大多数测序方法,样品(如核酸靶标)在引入测序仪器之前需要被加工。例如,样品可以被片段化、扩增或附接至标识符。独特标识符通常被用来鉴定特定样品的来源。大多数测序方法产生相对较短的测序读数,长度在几十个碱基至几百个碱基之间,并且由于测序读数长度限制,不能产生完整的单倍型定相(haplotype phase)信息。
技术实现要素:
4.一方面,本文描述的是通过加条形码标签追踪核酸片段来源的方法。所述方法包括提供多个核酸靶标和多个转座体,每个转座体包含至少一个转座子和一个转座酶;将核酸靶标和转座体一起孵育,在核酸靶标上形成链转移复合物(stc);提供多个独特条形码模板,其中每个条形码模板包含中央条形码序列,其侧接两个柄序列,其可用作引发位点、杂交位点或结合位点;隔室化具有stc的核酸靶标和条形码模板,以产生两个或更多隔室,其包含具有不同条形码序列的一个条形码模板或多于一个条形码模板和核酸靶标;在隔室中扩增条形码模板,通过打破stc将核酸靶标片段化,以形成标签化的核酸片段,并将条形码序列附接至标签化的核酸片段上,使得多个片段共有同一个或多于一个条形码序列;去除隔室并收集加条形码标签的核酸片段。
5.一方面,本文描述的是通过加条形码标签追踪核酸片段来源的方法。所述方法包括提供多个核酸靶标和多个转座体,每个转座体包含至少一个转座子和一个转座酶;将核酸靶标和转座体一起孵育,在核酸靶标上形成链转移复合物(stc);提供多个独特条形码模板,其中每个条形码模板包含中央条形码序列,其侧接两个柄序列,其可用作引发位点、杂交位点或结合位点;隔室化具有stc的核酸靶标和条形码模板,以产生两个或更多隔室,其包含具有不同条形码序列的一个条形码模板或多于一个的条形码模板和核酸靶标;通过以下方式在隔室内将条形码序列附接至核酸靶标:i)通过打破stc片段化核酸靶标以产生标签化的核酸片段;ii)用非靶标特异性(即仅转座子特异性)引物扩增标签化的核酸片段,并
同时扩增一个或多个条形码模板;iii)将条形码模板连接至标签化的核酸片段,其中多个片段共有同一个或多于一个条形码序列;去除隔室并收集加条形码标签的核酸片段。
6.一方面,本文所述的是单细胞atac-seq方法。所述方法包括提供多个核或细胞和多个转座体,每个转座体包含至少一个转座子和一个转座酶;将它们一起孵育,以在核内可及染色质上形成链转移复合物(stc);提供多个独特条形码模板,其中每个条形码模板包含中央条形码序列,其侧接两个柄序列,其可用作引发位点、杂交位点或结合位点;隔室化经处理的核或细胞和条形码模板以产生两个或更多包含具有不同条形码序列的一个条形码模板或多于一个条形码模板和核或细胞的隔室;在隔室中扩增条形码模板,通过打破stc将可及染色质片段化,以形成标签化的核酸片段,并将条形码序列附接至标签化的核酸片段上,使得多个片段共有同一个或多于一个条形码序列;去除隔室并收集加条形码标签的核酸片段;测序条形码和加条形码标签的核酸以单细胞为基础表征可及染色质区域。
7.一方面,本文所述的是单细胞atac-seq方法。所述方法包括提供多个核或细胞和多个转座体,每个转座体包含至少一个转座子和一个转座酶;将它们一起孵育,以在核内可及染色质上形成链转移复合物(stc);提供多个独特条形码模板,其中每个条形码模板包含中央条形码序列,其侧接两个柄序列,其可用作引发位点、杂交位点或结合位点;隔室化经处理的核或细胞和条形码模板以产生两个或更多包含具有不同条形码序列的一个或多于一个条形码模板和核或细胞的隔室;通过以下将条形码序列附接至隔室中的可及染色质片段:i)通过打破stc将可及染色质片段化,以形成标签化的核酸片段,ii)扩增所述标签化的核酸片段并同时扩增条形码模板;iii)将条形码模板连接至标签化的核酸片段,其中多个片段共有同一个或多于一个条形码序列;去除隔室并收集加条形码标签的核酸片段;测序条形码和加条形码标签的核酸以单细胞为基础表征可及染色质区域。
8.一方面,本文描述的是条码化单细胞基因组的方法。所述方法包括提供多个核或细胞,并对其进行处理,以通过变性染色质上的蛋白质同时细胞单元保持完整使dna从染色质暴露出来;提供多个转座体,每个转座体包含至少一个转座子和一个转座酶;将经过处理的核或细胞和转座体一起孵育,以在核或细胞中的双链核酸上形成链转移复合物(stc);提供多个独特条形码模板,其中每个条形码模板包含中央条形码序列,其侧接两个柄序列,其可用作引发位点、杂交位点或结合位点;隔室化经处理的核或细胞和条形码模板以产生两个或更多包含具有不同条形码序列的一个或多个条形码模板和核或细胞的隔室;在隔室中扩增条形码模板,打破stc以形成标签化的核酸片段,并将条形码序列附接至标签化的核酸片段上,使得多个片段共有同一条形码序列;去除隔室并收集加条形码标签的核酸片段。
9.一方面,本文描述的是条码化单细胞基因组的方法。所述方法包括提供多个核或细胞,并处理核或细胞,以通过变性染色质上的蛋白质同时保持细胞单元完整使dna从染色质暴露出来;提供多个转座体,每个转座体包含至少一个转座子和一个转座酶;将经处理的核或细胞与转座体一起孵育,以在核或细胞中的双链核酸上形成链转移复合物(stc);提供多个独特条形码模板,其中每个条形码模板包含中央条形码序列,其侧接两个柄序列,其可用作引发位点、杂交位点或结合位点;隔室化经处理的核和条形码模板以产生两个或更多包含具有不同条形码序列的一个或多于一个条形码模板和核或细胞的隔室;通过以下将条形码序列附接至隔室中的所述核或细胞中的所述基因组dna:i)打破stc以形成标签化的核酸片段;ii)扩增所述标签化的核酸片段,同时扩增条形码模板;iii)将条形码模板连接至
标签化的核酸片段,其中多个片段共有同一个或多于一个条形码序列;去除隔室并收集加条形码标签的核酸片段。
10.一方面,本文所述的是用于单细胞靶向测序的方法。所述方法包括提供多个细胞或核,提供多个独特条形码模板,其中每个条形码模板包含中央条形码序列,其侧接两个柄序列,其可用作引发位点、杂交位点或结合位点,以及提供多个靶特异性引物,其中所述靶特异性引物能够附接至条形码模板;隔室化细胞或核、条形码模板和靶特异性引物以产生两个或更多包含所述细胞或核、具有不同条形码序列的一个或多于一个条形码模板、和靶特异性引物的隔室;在隔室中扩增条形码模板,将条形码序列附接至靶特异性引物,用靶特异性引物引发靶基因组区域以生成附接有条形码的靶片段,使得多个附接有条形码的靶片段共有同一个或多个条形码序列;去除隔室并收集附接有条形码的靶片段;并测序条形码和加条形码标签的核酸,以基于单细胞表征靶向区域。
11.一方面,本文所述的是用于单细胞靶向测序的方法。所述方法包括提供多个细胞或核,提供多个独特条形码模板,其中每个条形码模板包含中央条形码序列,其侧接两个柄序列,其可用作引发位点、杂交位点或结合位点,以及提供多个靶特异性引物,其中所述靶特异性引物能够附接至条形码模板;隔室化细胞或核、条形码模板和靶特异性引物以产生两个或更多包含具有不同条形码序列的一个或多于一个条形码模板、所述细胞或核和靶特异性引物的隔室;通过以下将条形码序列附接至隔室中的靶向核酸片段:i)打破细胞或核膜以释放核酸;ii)扩增核酸靶标并同时扩增条形码模板;iii)将条形码模板连接至所述扩增的核酸靶标,其中多个核酸靶标共有同一个或多于一个条形码序列;去除隔室并收集附接有条形码的靶片段;并测序条形码和条码化加标签的核酸,以基于单细胞表征靶向区域。
12.一方面,本文所述的是用于单细胞rna测序的方法。所述方法包括提供多个细胞或核,提供多个独特条形码模板,其中每个条形码模板包含中央条形码序列,其侧接两个柄序列,其可用作引发位点、杂交位点或结合位点,提供逆转录酶和提供多个引物,其中引物能够作为引物用于cdna合成,或用于条形码模板扩增,或用于与cdna引发,或用于其组合;隔室化细胞和/或核、条形码模板、逆转录酶和引物以产生两个或更多包含具有不同条形码序列的一个或多于一个条形码模板、细胞和/或核、逆转录酶和引物的隔室;在隔室中,裂解细胞和/或核,产生cdna,扩增条形码模板,将所述条形码序列附接至cdna片段或从cdna产生的片段,使得多个附接有条形码的片段共有同一个或多个条形码序列;去除隔室并收集附接有条形码的片段;并测序条形码和条码化加标签的核酸,以基于单细胞表征cdna概况(cdna profile)。一方面,在隔室化之前,细胞或核用逆转录酶处理,用于cdna合成。
13.附图简要说明
14.图1说明了一种用隔室化反应利用转座体和条形码模板进行核酸条码化的方法。bc表示条形码。
15.图2说明了一种将克隆扩增的条形码模板附接至标签化的核酸片段的方法。bc表示条形码。在隔室中,a)条形码模板直接附接至标签化的片段;b)条形码模板通过接头寡核苷酸间接附接至标签化的片段;c)条形码模板和标签化的片段都被扩增且条形码模板附接至标签化的片段;d)具有不同条形码序列的条形码模板和标签化的片段被扩增,且条形码模板附接至标签化的片段。
16.图3说明了一种用隔室化反应利用转座体加标签的核和条形码模板进行单细胞
atac-seq文库制备的方法。
17.图4说明了一种用隔室化反应利用转座体加标签的醇固定的核和条形码模板进行单细胞全基因组条码化的方法。
18.图5说明了使用条码化核酸片段和靶特异性引物组富集靶向区域的方法。
19.图6说明了条码化单细胞可以显著改进体细胞突变的检测能力,具有用于个体细胞鉴定和用独特分子鉴定(umi)的测序错误校正的组合能力。
20.图7说明了在隔室内单细胞核酸条码化反应用于靶向测序。
21.图8说明了在液滴中通过一个或多个条形码模板和标签化的片段的双扩增和将扩增的条形码模板附接至标签化的片段的克隆条码化反应。
22.图9说明了同一条形码读数1读数距离与下一个读数1比对的测序读数计数直方图,以证明连接读数(linked-read)特征
23.图10显示了经净化的单细胞atac-seq文库的tapestation高灵敏度d1000屏幕录像谱(tapestation high sensitivity d1000 screen tape profile)。
24.图11显示了单细胞atac-seq实验的cell ranger分析报告的屏幕截图。
25.所有图中的转座酶都是作为基于mua转座系统的转座体中的四聚体说明的。
具体实施方式
26.大多数市售测序技术的测序读数长度有限。第二代高通量测序技术只能测序几百个碱基,很少达到上千个碱基。然而,基因的核酸序列可以跨越从几千碱基到几十千和几百千碱基,这意味着几十千碱基的测序读数长度对于成功确定所有基因的单倍型是必要的。
27.同时,尽管个体细胞是不同的,现今大多数测序都是一次从许多细胞中提取的dna或rna的批量测序。通过使用细胞群的平均分子或表型测量来代表个体细胞的行为,结论可能会被大多数细胞群的表达谱或过度表达的异常值所偏离;而且我们将没有鉴定来自个体细胞的所有独特模式的灵敏度,所述模式可能是细胞在给定的位置和时间的独特功能行为。此外,由于来自正常细胞或组织的高背景野生型信号的存在,目前检测非常低频的体细胞突变的能力有限,这大大限制了早期肿瘤检测的能力。然而,随着鉴定每个单细胞能力的提高,我们将能够通过单细胞水平的基因分型将突变的肿瘤细胞与野生型细胞分开。这将几乎完全消除正常细胞产生的野生型背景信号,使体细胞突变检测与种系突变检测一样容易。
28.先前已经描述过tn5转座体和mua转座体在体外同时片段化dna并以高频率引入衔接子,为下一代dna测序创建测序文库(adey等2010,caruccio等2011,和kavanagh等2013)。由于dna的片段化,这些特定的方案去除了任何定相或相邻性信息。在这些方案中,dna与转座体反应后,需要进行柱纯化、热处理步骤、蛋白酶处理或与sds溶液或edta溶液孵育,以从链转移复合物(stc)中释放转座酶,使得dna被标签化到片段。已知mua转座体在攻击dna靶标时可以形成非常稳定的stc(surette等1987,mizuuchi等1992,savilahti等1995,burton和baker 2003,au等2004)。在转座反应期间,对于tn5转座体也观察到了类似的稳定性(amini等2014)。
29.本发明利用了stc的稳定性和通过隔室扩增产生的克隆条形码,并提供了在单细胞中对核酸靶标亚片段和/或条形码核酸进行独特条形码化的方法。
30.本文所用术语“衔接子”是指核酸序列,其可以包含引物结合序列、条形码、接头序列、与接头序列互补的序列、捕获序列、与捕获序列互补的序列、限制性位点、亲和部分、独特分子标识符及其组合。
[0031]“条形码模板”,其包含一侧末端侧接至少一个柄序列或两侧末端侧接两个柄序列的条形码序列。条形码序列的长度范围为4个碱基到100个碱基。柄序列可用作杂交或退火的结合位点、扩增期间的引发位点或测序引物或转座酶的结合位点。此外,条形码序列可以选自已知核苷酸序列库,或从随机合成的核苷酸序列中随机选择。
[0032]
本文所用术语“转座酶”是指能够进行转座的功能性核酸蛋白质复合物的组分并介导转座的蛋白质,包括但不限于tn、mu、ty和tc转座酶。术语“转座酶”也指来自逆转录转座子(retrotransposon)或逆转录病毒来源的整合酶。它还指野生型蛋白、突变型蛋白和带标签的融合蛋白,如gst标签、his标签等及其组合。
[0033]
如本文所用,术语“转座子”是指被转座酶或整合酶识别并且是能够转座的功能性核酸-蛋白质复合物的基本组分的核酸区段。它们与转座酶一起形成转座体并进行转座反应。它指的是野生型和突变型转座子。
[0034]
如本文所用,“可转座的dna”是指包含至少一个转座子单元的核酸区段。它还可以包含亲和部分、非天然核苷酸和其他修饰。可转座的dna中除转座子序列外的序列可以包含衔接子序列。
[0035]
本文所用的术语“转座体”是指由转座酶与转座子非共价结合形成的稳定的核酸和蛋白质复合物。它可以包含相同或不同的单体单元的多聚体单元。
[0036]
如本文所用,“转座子接合链”是指通过转座酶在插入位点接合至靶核酸的双链转座子dna的链。
[0037]
如本文所用,“转座子互补链”是指双链转座子dna中转座子接合链的互补链。
[0038]
如本文所用,“链转移复合物(stc)”是指转座子插入其中的转座体及其靶核酸的核酸-蛋白质复合物,其中转座子连接链的3
′
端与其靶核酸共价连接。它是一种非常稳定的核酸和蛋白质复合物形式,可在体外抵抗极热和高盐(burton和baker,2003)。
[0039]
本文所用的“链转移反应”是指核酸和转座体之间的反应,其中形成稳定的链转移复合物。
[0040]
如本文所用,“反应容器”是指具有连续开放空间以容纳液体的物质;其选自下组:管、孔、板、多孔板中的孔、载玻片、载玻片上的点、液滴、管道、通道、瓶子、腔室和流动池。
[0041]
如本文所用,“标签化的片段”是指在与转座体进行链转移反应后用至少一个转座子末端加标签的核酸片段。
[0042]
将带有链转移复合物的核酸和条形码模板包封在油包水乳液液滴中
[0043]
本发明提供了一种将带有stc的核酸靶标与条形码模板包封在油包水乳液液滴中,并进一步产生加条形码标签的核酸片段的方法。
[0044]
核酸靶标与转座体(101)反应并形成稳定的链转移复合物(102)同时保持核酸靶标的相邻性(图1)。核酸靶标是双链的。在一些实施方式中,它们是双链dna。在一些实施方式中,它们是dna和rna杂交体。在一个反应容器中链转移反应发生在多个核酸靶标。在一些实施方式中,使用一种类型的转座体;在其他实施方式中,同时或顺序地使用多于一种类型的转座体。带有stc(102)的核酸靶标在溶液中与多个条形码模板(103)混合。在一些实施方
式中,每个条形码模板具有独特条形码序列且彼此不同。在一些实施方式中,大多数条形码模板各自具有独特条形码序列且彼此不同。任何大于50%的可以被认为是大多数。优选地,大于90%的条形码模板各自具有独特条形码序列且彼此不同。转座体中至少一个可转座dna能够直接杂交(图2a)或通过接头和/或引物间接杂交(图2b)至条形码模板的一端。其他酶和底物,例如dna聚合酶、dntp和引物也以水性溶液的形式提供在同一反应容器中。在一些实施方式中,引物用于扩增条形码模板。在一些实施方式中,引物可用于扩增标签化的核酸靶片段。扩增包括指数扩增和线性扩增。在一些实施方式中,可以使用不同的引物来平行扩增条形码模板和标签化的核酸靶片段(图2c),然后两组扩增产物能够通过两个内部引物之间共有的同源性(图2c,208和209)或通过能够将条形码模板和标签化的片段桥接在一起的附加接头合并成一体。产生油包水乳液液滴(104)。在一些实施方式中,在一个液滴中具有stc的一个到几个核酸靶标与一个条形码模板混合。此处可以基于泊松分布对乳液液滴和条形码模板进行适当的滴定。在一些实施方式中,具有不同条形码序列的多于一个条形码模板可用于乳液液滴中。它将显著增加乳液液滴中条形码的存在和具有阳性产物的液滴数量,从而将显著增加反应产率。在一些实施方式中,当条形码模板和标签化的片段两者在将条形码序列附接至标签化的片段之前被扩增时,如果不同的条形码随机地附接至标签化的片段的扩增拷贝上,同一乳液液滴中具有不同条形码序列的多于一个条形码模板可能根本不会影响核酸靶标的真实表现(图2d)。通过这种方式,几乎每个乳液液滴都将包含至少一个条形码模板,其将可用于当靶标也存在于液滴中时将条形码附接至核酸靶标上。这使得获得几乎100%含有任何可用于反应的核酸靶标的液滴成为可行的。乳液液滴的直径为5μm至200μm,优选5μm至50μm。热处理后,例如在60℃至75℃处理约5-10分钟,转座酶将从stc中释放出来,核酸靶标会断裂成更小的标签化的片段。当仍在油包水液滴中时,dna聚合酶将填补转座反应过程中留下的间隙。进行乳液扩增以扩增液滴中的条形码模板。扩增的条形码模板将直接(图2a)或间接(图2b)地杂交至标签化的片段,并在扩增反应期间将条形码序列附接至片段(105、201和202)。在一些实施方式中,在乳液反应期间,独特分子标识符(umi)被添加到条形码模板中。在一些实施方式中,umi被整合为图2中的接头(203)或引物(209和212)。在一些实施方式中,umi作为转座体中可转座dna的部分被引入标签化的片段。乳液扩增反应后,乳液液滴被去污剂、醇、有机化学物、高盐或其组合打破。收集水相溶液。在一些实施方式中,使用一种或多种生物素化引物,以便可以用链霉亲和素珠容易地拉出扩增的条码化片段。在一些实施方式中,一种或多种生物素化dntp被用于乳液扩增。在一些实施方式中,在乳液扩增期间,具有样品特异性条形码的引物被用于乳液液滴,使得来自不同样品反应的乳液扩增产物可以被汇集在一起用于最终扩增或衔接子修饰,以制作测序读数文库。
[0045]
在一些实施方式中,核酸靶标是全基因组dna。该条码化方法可被用于从头测序以产生长程测序信息、全基因组单倍型定相和结构变体检测。在一些实施方式中,核酸靶标是dna片段、cdna、dna/rna杂交体或由杂交捕获、引物延伸或pcr扩增所捕获的dna部分。该条码化方法将能够在这些分子中定相变体。
[0046]
将转座酶加标签的核和条形码模板包封在油包水乳液液滴中
[0047]
本发明提供了一种在链转移反应后的核和条形码模板包封于油包水乳液液滴中,并进一步产生加条形码标签的核酸片段用于单细胞级分析的方法。
[0048]
atac-seq(用测序检测转座酶可及的染色质)作为评估全基因组染色质可及性的现有技术分子生物学工具受到越来越多的欢迎(buenrostro等,2013)。atac-seq通过用超活的(hyperactive)突变tn5转座酶(其将测序衔接子整合到基因组的开放区域)给开放染色质加标签鉴定可及染色质区域。加标签的dna片段被纯化,通过pcr扩增并测序。然后测序读数被用于推断可及性增加的区域以及映射转录因子结合位点的区域和核小体位置。虽然天然野生型转座酶的活性水平较低,但atac-seq采用突变的超活转座酶(reznikoff等,2008),其已成功适用于高效鉴定开放染色质和鉴定整个基因组的调控元件。此外,单细胞atac-seq是为了分开单核并单独地进行atac-seq反应(buenrostro等,2015)。更高通量的单细胞atac-seq使用组合细胞标引以测量数千个个体细胞的染色质可及性。单细胞atac-seq能够鉴定细胞类型和状态用于发育谱系追踪。atac-seq可能将是综合性表观遗传学工作流程的关键组分。
[0049]
本发明使用乳液方法以包封经转座酶处理的核和独特条形码模板,然后克隆扩增在乳液液滴内的条形码模板并将克隆扩增的条形码附接至标签化的可及dna片段(图3)。这种条码化方法为单细胞atac-seq分析提供了高通量和低成本的细胞标引。
[0050]
在一些实施方式中,从细胞或组织样品中收集核(302)并与转座酶孵育以形成stc(304),然后在批量反应中与多个不同的条形码模板混合(图3)。在一些实施方式中,完整细胞被用转座酶直接处理,而无需核分离。在一些实施方式中,转座体包含突变的超活tn5转座酶。在一些实施方式中,转座体包含mua转座酶。其他酶和底物,例如dna聚合酶、dntp和引物也以水性溶液的形式提供在相同的批量反应中。产生油包水乳液液滴。在一些实施方式中,通过基于泊松分布的有限滴定或分区,大多数液滴中存在一个核和一个条形码模板(307)。10x基因组学单细胞atac-seq方法使用条码化珠(gem),其包含在乳液液滴中作为具有相同的条形码序列的条形码模板发挥功能的大量寡核苷酸的拷贝。本发明是为了仅包封条形码模板的单拷贝而无需附接至任何物理运载体。在一些实施方式中,在乳液液滴中具有不同条形码序列的多于一个条形码模板被靶向,以使得几乎所有液滴包含至少一个条形码模板,以增加核捕获率。乳液液滴的直径为10μm至200μm,优选20μm至100μm。热处理后,例如在60℃至75℃处理约5-10分钟,转座酶将从stc中释放出来,核酸靶标会断裂成更小的加标签的片段。当仍在油包水液滴中时,dna聚合酶将在加标签的片段上填补转座反应过程中留下的间隙。在乳液pcr变性步骤期间将打破核膜,并进行乳液扩增以在液滴中扩增条形码模板。扩增的条形码模板能够直接或间接地杂交至标签化的片段,并在扩增反应期间将条形码序列附接至片段。在一些实施方式中,首先条码化模板和标签化的片段二者平行地被扩增,然后合并在一起以形成条码化标签化的片段,如图2c和2d。乳液扩增反应后,乳液液滴被去污剂、醇、有机溶液、高盐或其组合打破。收集水相溶液。在一些实施方式中,使用一种或多种生物素化引物或一种或多种生物素化dntp,以便可以用链霉亲和素珠容易地拉出扩增的条码化片段。从这些条码化片段中制备的测序文库将是单细胞atac-seq文库。
[0051]
除了单细胞atac-seq应用之外,本发明还提供了一种具有恰当修改的单细胞全基因组测序方法。其使用乳液方法以包封经转座酶处理的固定的核和独特条形码模板,并克隆扩增在乳液液滴内的条形码模板并将该条形码附接至标签化的基因组dna片段(图4)。
[0052]
在一些实施方式中,从细胞或组织样品中收集核(402)并用醇基固定剂固定。醇基固定剂或其他固定剂将能够变性核内蛋白质但保持核酸完整。通过这种方法,其能够从染
色质上暴露所有的基因组dna。在一些实施方式中,固定的细胞或组织样品被直接用于该过程,无需核分离,包括缺乏核的原核细胞的情况。洗去固定溶液后,用转座体处理细胞核以在基因组dna上形成stc(405),然后在批量反应中与多个不同的条形码模板混合。其他酶和底物,例如dna聚合酶、dntp和引物也以水性溶液的形式提供在相同的批量反应中。产生油包水乳液液滴。在一些实施方式中,通过基于泊松分布的有限滴定或分区,液滴中存在一个核和一个条形码模板(408)。在一些实施方式中,在乳液液滴中具有不同条形码序列的多于一个条形码模板被靶向,以使得几乎所有液滴包含至少一个条形码模板,以增加核或细胞捕获率。乳液液滴的直径为10μm至200μm,优选20μm至100μm。热处理后,例如在60℃到75℃处理约5-10分钟,转座酶将从stc中释放出来,核酸靶标会断裂成更小的标签化片段。当仍在油包水液滴中时,dna聚合酶将填补转座反应过程中留下的间隙。在乳液扩增期间将打破核膜。进行乳液扩增以扩增液滴中的条形码模板。扩增的条形码模板能够直接或间接地杂交至标签化片段,并在扩增反应期间将条形码序列附接至片段。在一些实施方式中,首先条码化模板和标签化的片段二者平行地被扩增,然后合并在一起以形成条码化标签化片段,如图2c和2d。乳液扩增反应后,乳液液滴被去污剂、醇、有机试剂、高盐或其组合打破。收集水相溶液。在一些实施方式中,使用一种或多种生物素化引物或一种或多种生物素化dntp,以便可以用链霉亲和素珠容易地拉出扩增的条码化片段。在一些实施方式中,从这些条码化片段制备的文库可以直接用于单细胞全基因组测序和单细胞cnv分析。在一些实施方式中,从这些条码化片段制备的文库可以被用于全外显子或更小靶向区域的进一步靶向捕获以进行靶向测序(图5)。
[0053]
这种单细胞靶向测序的一个优点是它对低频率变体检测具有高得多的灵敏度,例如,体细胞突变检测(图6)。由于能够独特地条码化个体细胞,我们可以在单细胞水平上检测任何突变,这将有效地消除来自周围细胞的背景噪音。这实现了用于检测极低频率体细胞突变的极高灵敏度,而这正是早期癌症检测所需要的。图6说明了在单细胞水平上基因分型的能力。有细胞含有突变的等位基因a(601),但在同一个样品中,有许多野生型细胞含有正常的等位基因t(602)。在乳液反应期间,独特分子标识符(umi)被添加。在一些实施方式中,umi被整合为图2中的接头(203)或引物(209和212)。在一些实施方式中,umi作为转座体中可转座dna的部分被引入标签化的片段。通过在单细胞条码化和测序期间纳入分子特异性umi,测序读数可以首先基于其细胞id被分组,并且对于每个细胞,我们能够基于umi鉴定测序错误,并容易地进行正确的变体识别。这种方法可以应用于循环肿瘤细胞、组织活检样本或组织切片。组织和/或切片包括新鲜冷冻组织/切片和福尔马林固定的石蜡包埋组织/切片。
[0054]
将细胞、条形码模板和靶特异性引物包封在油包水乳液液滴中
[0055]
本发明提供了用于单分子靶向测序的高通量方法。分离的细胞或核(702)被与独特条形码模板(703)和第一组靶特异性引物(704)包封在乳液液滴中(图7)。在一些实施方式中,在包封反应之前,细胞或分离的核被预处理。预处理可以是用固定剂孵育、原位酶促反应、杂交或这些处理的组合。其他酶和底物,例如dna聚合酶、dntp和共同引物(common primer)也以水性溶液的形式提供。油包水乳液液滴(701)在这样的条件下产生,即通过基于泊松分布的有限滴定或分区,液滴中存在一个细胞或一个核和一个条形码模板。乳液液滴的直径为10μm至200μm,优选20μm至100μm。在乳液扩增期间打破细胞膜或核膜,并将基因
组dna释放到乳液液滴中。进行乳液扩增以扩增条形码模板并将靶特异性引物附接至液滴中的条形码模板。在3’端具有靶特异性序列的单链扩增条形码模板(705)能够杂交至基因组dna靶标并在扩增反应期间制造靶向区域的拷贝。在一些实施方式中,在乳液液滴产生期间,第二组靶特异性引物(706)被包含在水性溶液中。乳液扩增反应后,靶标的加条形码标签的扩增子(707)将被产生,可用于测序文库制备和测序分析。在一些实施方式中,为了减少在扩增期间产生的引物二聚体,可以使用含有dutp的引物并在乳液扩增后与用udg/ape1/exoi处理组合。清理引物二聚体后,测序文库衔接子可以通过连接添加。
[0056]
在一些实施方式中,细胞或核被处理并与逆转录酶反应用于原位cdna合成,然后用乳液液滴包封。在一些实施方式中,逆转录酶和作为第一组引物的cdna引物可以被包含于乳液反应中。在一些实施方式中,cdna引物在3’端具有多聚t序列;在一些实施方式中,cdna引物在3’端具有ggg;在一些实施方式中,edna引物在3’端具有靶特异性引物。在乳液反应的早期,逆转录酶会从单细胞或核中的mrna生成cdna或部分cdna。条码化反应将如前所述进行,但使用cdna作为输入dna。使用用于逆转录或cdna引发的不同引物,该方法可以被修改用于单细胞转录组分析、单细胞rna-seq分析、单细胞靶向-测序(target-seq)应用和免疫组库分析。
[0057]
在一些实施方式中,具有不同条形码序列的多于一个条形码模板可存在于乳液液滴中以增加细胞捕获率。
[0058]
通过测序对转录组和表位进行细胞标引(cite-seq)是一种多模式单细胞表型分析方法,它使用dna条码化抗体将蛋白质的检测转换为定量的、可测序的读取数。抗体结合的寡核苷酸作为合成转录物,在大多数大规模基于寡核苷酸-dt的单细胞rna-seq文库制备方案中被捕获(stoeckius等,2017)。对于我们上述的方法,当cdna引物是多聚t型设计时,cite-seq型文库将能够高效地产生。
[0059]
产生油包水乳液的方法有很多种,例如,通过涡旋、均质化、过滤、吹打、通过微流体装置将水和油合并等。在一些实施方式中,本发明的乳化方法是在微管或孔中用移液管混合水性溶液和油,以便于设置和放大样品制备程序。乳液液滴大小可以通过混合速度和移液管尖端的孔口大小来控制。混合速度范围为20μl/s至1000μl/s时,可以产生适当大小的乳液液滴。
[0060]
虽然本发明所述的隔室化方法是油包水乳液,但其他方法也是可行的。某些类型的脂质体,例如直径为1-200um的巨型单层脂质体囊泡(guv),已显示出非常高的热稳定性,并且能够在其外壳内进行pcr扩增(kurihara等2011,laouini等2012)。在一些实施方式中,本发明中用于隔室生成的乳液液滴可以被guv代替。在一些实施方式中,用于隔室生成的乳液液滴可以用微孔、微阵列、微量滴定板或其他物理分离的隔室化方法代替。
[0061]
尽管本发明已经就实施方式进行了解释,但应该理解,在不背离本文所述的本发明的精神和范围的情况下,可以进行许多其他可能的修改和变化。
[0062]
此外,一般来说就本文所描述的过程、系统、方法等而言,应当理解,尽管此类过程等的步骤被描述为按照一定的顺序发生,但此类过程可以按照本文所描述的顺序以外的顺序来实施所述步骤。还应理解的是,某些步骤可以同时进行,可以增加其他步骤,或者可以省略此处描述的某些步骤。换言之,本文对过程的描述是为了说明某些实施方式而提供的,而不应该被解释为限制所要求保护的发明。
[0063]
此外,应理解,上述描述的目的是说明性的,而不是限制性的。除所提供的实施例外,对于本领域的技术人员来说,许多实施方式和应用在阅读上述描述后将是显而易见的。在确定本发明的范围时,不应参照上述描述,而应参照所附的权利要求,以及这些权利要求所赋予的等同物的全部范围。在本文所讨论的技术中将会出现未来的发展,预期所公开的系统和方法将被纳入这种未来的实施方式中。总之,应理解,本发明是能够修改和变化的,并且只受以下权利要求的限制。
[0064]
最后,本技术中使用的所有定义的术语旨在给予其与本文提供的定义一致的最广泛的合理解释。权利要求书中使用的所有未定义的术语,除非本文中有明确的相反指示,将按照本领域技术人员理解的普通含义给予其最广泛的合理解释。具体来说,单数冠词例如“一个”、“一种”、“所述”等应被理解为叙述一个或多个所述要素,除非权利要求明确表明相反的限制。
[0065]
实施例
[0066]
实施例1.在液滴中条码化长片段以产生连接读数
[0067]
本实施例描述了在液滴中条码化dna片段以产生连接读数的方法。
[0068]
1ng大肠杆菌dh1ob基因组dna(图8,806)通过与野生型转座体和突变的mua转座体孵育被链转移(807),同时使用来自tell-seq wgs文库试剂盒1(通用测序技术公司(universal sequencing technology),加利福尼亚州卡尔斯巴德)的1μl的条码化酶(野生型mua转座体)和1μl加标签酶(突变的mua转座体),在1x有辅因子的反应缓冲液中以20μl反应体积在37℃下进行15分钟以形成链转移复合物(stc,802)。在0.2ml pcr管中,取1μl的stc反应混合物加入到10μl的扩增水性溶液中,其含有1x pfu聚合酶缓冲液、dntp、条形码模板码1.2
[0069]
(5
’‑
caagcagaagacggcatacgagatnnnatnnnncannnncgnnntggtcatgtggagacgctgggacag-3’,801)、引物[p7(5
’‑
caagcagaagacggcatacgagat-3’,803)、t25(5
′‑
ctgtcccagcgtctccacatgacca-3
′
,804)、tsmu(5
′‑
gctgggacaggtcacttttcgtgcgccgcttca-3
′
,808)、bio-mp5(5
′‑
生物素-acactctttccctacattaactgca-3
′
,809)]和pfu dna聚合酶。加入90μl的在矿物油(西格玛奥德里奇公司(sigma-aldrich),密苏里州圣路易斯)中的7%abil em90(赢创公司(evonik corporation),弗吉尼亚州里士满)。将p200移液器设置为70μl,并在30秒内通过上下吹打30次以混合溶液。将50μl混合物转移到另一个0.2ml pcr管中并加入50μl 7%abil em90的矿物油溶液。通过在15秒内上下吹打15次混合溶液。如下进行扩增:72℃持续2分钟,94℃持续30秒,(94℃持续20秒,55℃持续1分钟,72℃持续1分钟)21个循环,(94℃持续30秒,35℃持续1分钟,72℃持续2分钟)12个循环,72℃持续3分钟,在4℃保持。在pcr结束时,加入100μl打破缓冲液(100mm nacl、10mm tris-hcl、ph 7.5、0.2%sds、15%异丙醇)并在室温下孵育10分钟。以5,000g离心管10分钟以分离油和水性溶液。从顶层除去油。在结合缓冲液中,将70μl水性溶液转移到0.5ml低结合dna管中并加入35μl myone
tm
链霉亲和素t1珠(生命技术公司(life technologies),加利福尼亚州卡尔斯巴德)。在室温下旋转孵育15分钟。用珠洗涤缓冲液洗涤珠三次。将珠重悬于15μl 0.02%tween-20中。使用带有p7引物的pfu dna聚合酶和来自tell-seq文库多重引物(1-8)试剂盒(通用测序技术公司(universal sequencing technology),加利福尼亚州卡尔斯巴德)的多重引物之一,在40μl总体积中使用5μl珠进行pcr扩增。如下进行pcr扩增:94℃持续30秒,(94℃持
续20秒,58℃持续1分钟,72℃持续1分钟)6个循环,72℃持续3分钟,在4℃保持。pcr扩增后,用0.9x ampure xp珠清理文库产物并定量用于测序。测试了条形码模板分子与乳液液滴的不同比率。实施例中使用了3比1的比率,以确保大约95%的液滴包含至少一个条形码模板。
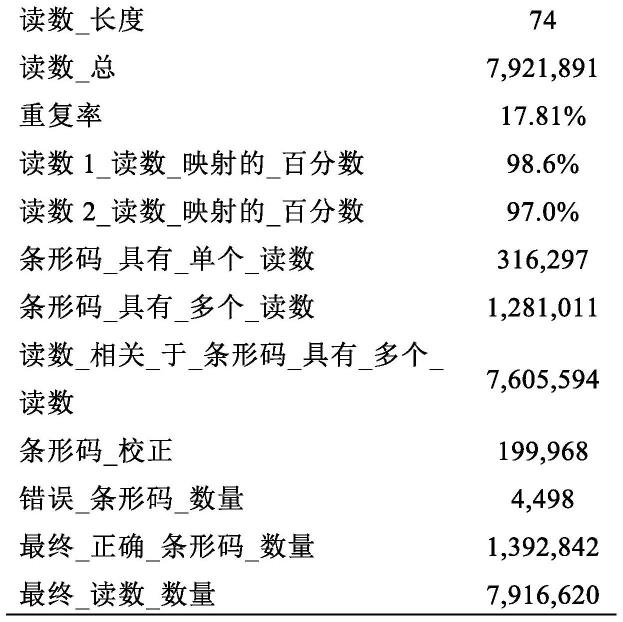
[0070]
该文库在miseq系统上以2x74配对末端运行进行测序。实验中使用的条形码模板包含20个碱基的条形码序列,并作为标引1读数被测序。表1显示了测序运行的总结。读数1和读数2的映射率分别为98.6%和97.0%。共鉴定了1,392,842个条形码。
[0071]
表1.来自2x74配对末端miseq运行的大肠杆菌文库的测序统计数据
[0072][0073][0074]
为了检验条码化反应对于加标签的片段是否是克隆性的,我们生成了一个读数距离图(图9),其为共有相同条形码序列的那些r1读数的下一个比对读数距离的读数1读数计数直方图。如果条码化反应对于加标签的片段确实是克隆性的,则会有许多相同的条码化读数彼此相邻,距离短(通常小于50kb),其将显示为连接读数群;而来自不同基因组dna片段的相同条码化读数在远端读数群中会显示出很大的距离(通常大于100kb)。图9显示了对于该大肠杆菌文库的非常好的克隆性条码化反应。我们使用turingassembler进一步从头组装这些连接读数,turingassembler是一个连接读数组装器,并且我们获得了4,591,903bp的n50重叠群(contig)大小,这非常接近大肠杆菌dh10b基因组的完整大小(4,686,137bp),具有非常好的组装精度(表2)。
[0075]
表2.使用turingassembler从头组装的quast结果与大肠杆菌dh10b基因组参考(4,686,137bp)比较
[0076][0077][0078]
实施例2.单细胞atac-seq
[0079]
k562细胞(atcc,弗吉尼亚州马纳萨斯)被培养于含有10%fbs(生命技术公司
(life technologies),加利福尼亚州卡尔斯巴德)、1∶100mem非必需氨基酸(生命技术公司(life technologies),加利福尼亚州卡尔斯巴德)、1∶100青霉素/链霉素(生命技术公司(life technologies),加利福尼亚州卡尔斯巴德)、1∶100 glutamax(生命技术公司(life technologies),加利福尼亚州卡尔斯巴德)和1∶1000 bme(生命技术公司(life technologies),加利福尼亚州卡尔斯巴德)的dmem培养基(生命技术公司(life technologies),加利福尼亚州卡尔斯巴德)中。当细胞浓度达到约500,000/ml时,将150万个细胞加入到1.5ml蛋白低结合离心管中,并以300xg离心3分钟。去除上清液,将沉淀重新悬浮在1ml的1x pbs中。然后将细胞以300xg再次离心3分钟。细胞沉淀被重悬于150μl冰冷的裂解缓冲液(10mm nacl、10mm tris ph 7.4、3mm mgcl2、0.01%洋地黄素、0.1%tween和o.1%np40)中。用设置为100μl的p200移液器混合细胞5次并置于冰上3分钟。孵育3分钟后,用设置为100μl的移液器混合细胞10次。
[0080]
850μl的洗涤缓冲液(10mm nacl、10mm tris ph 7.4、3mm mgcl2、0.1%tween)被加入并用设置为850μtl的p1000移液器混合5次。将核以400xg离心3分钟并重悬于1ml洗涤缓冲液中。通过0.4μm flowmi过滤器过滤核以除去任何团块,然后以400xg离心3分钟。将核沉淀重悬于20μl的洗涤缓冲液中。2μl核在98μl中稀释并计数两次以获得精确的细胞计数。将最终浓度调整为25,000核/μl,并将核保持在冰上。
[0081]
5μm tn5me转座体被组装,使用ez-tn5
tm
转座酶(lucigen,威斯康星州米德尔顿)和预退火的tn5meds-a和tn5meds-b寡核苷酸(picelli等2014)。在20μl反应缓冲液(最终10%dmf、10mm tris ph7.5和5mm mgcl2、0.33x pbs、0.1%tween、0.01%洋地黄素)中,通过用0.35μm tn5me转座体处理50,000 k562核,进行链转移反应。在热循环仪上在37℃孵育该混合物1小时。反应后,在核重悬缓冲液(10mm nacl、10mm tris ph 7.4、3mm mgcl2)中,核被稀释到终浓度为500核/μl。
[0082]
在0.2ml pcr管中,1,000个加标签的核被用于20μl的扩增混合物中,其包含pfu dna聚合酶、dntp、引物[tn5-bc-r(5
’‑
tctccgagcccacgagac-3’)、tn5-r2-f28(5
’‑
tgggctcggagatgtgtataagagacag-3’)、p7(5
’‑
caagcagaagacggcatacgagat-3’)和tn5-r1-s(5
’‑
tcgtcggcagcgtcagatgt-3’)]、条形码模板码1.3(5
’‑
gaagacggcatacgagatnnnatnnnncannnncgnnngtctcgtgggctcggaga-3’)。矿物油(西格玛奥德里奇公司(sigma-aldrich),密苏里州圣路易斯)中的80μl的油混合物[7%abil em90(赢创公司(evonik corporation),弗吉尼亚州里士满)被加入到20μl扩增混合物的顶部。条形码模板数量与预期液滴数量的靶向比率为3比1,以使大约95%的液滴包含至少一个条形码模板。将p200移液器设置为70μl,并通过45秒内上下吹打30次以及30秒内额外吹打15次以混合溶液。进行以下pcr程序:72℃持续5分钟,95℃持续30秒,(95℃持续15秒,58℃持续30秒,和72℃持续20秒)20个循环,(95℃持续20秒,40℃持续2分钟,和72℃持续30秒)5个循环,72℃持续2分钟,20℃持续1分钟,和在4℃保持。
[0083]
液滴扩增后,较大的液滴沉降到底部,在顶部留下较小的液滴和油。顶部的50μl被除去并丢弃,而不会干扰沉降液滴的底层。向乳液中加入50μl打破溶液(100mm nacl、10mm tris-hcl,ph 7.5、0.2%sds、15%异丙醇)并混合10次。将乳液在10k微型离心机上离心8分钟除去并丢弃另外的10-15μl顶部油层,确保不要除去任何底部水性层。慢慢地,从底部取出60μl底部水性溶液并置于新试管中,同时注意不要吸出顶层的任何残余油。向水性溶液
中加入72μl ampure xp珠,进行1.2x珠净化。将混合物在室温下孵育5分钟,然后置于磁体上2-3分钟(或直到澄清)。去除澄清的上清并使用200μl新鲜制备的80%乙醇进行两次洗涤。洗过的珠子被重新悬浮在33μl的低te缓冲液中。取出30μl并置于新的pcr管中。15μl清理后的产物被用于在40μl混合物中进行最终pcr扩增,该混合物为1x phusion热启动ii高保真pcr主混合物,其含有p7引物和来自tell-seq文库多重引物(1-8)试剂盒(通用测序技术公司(universal sequencing technology),加利福尼亚州卡尔斯巴德)的多重引物之一,以产生illumina测序文库。进行以下pcr程序:95℃持续30秒,(95℃持续20秒,63℃持续30分钟,72℃持续30秒)5个循环,72℃持续2分钟,和在4℃保持。通过向pcr产物中加入48μl ampure xp珠,进行1.2x ampure xp珠净化。将混合物在室温下孵育5分钟,然后置于磁体上2-3分钟(或直到澄清)。去除澄清的上清并使用200μl新鲜制备的80%乙醇进行两次洗涤。洗过的珠子被重新悬浮在25μl的低te缓冲液中。取出23μl并转移到新的pcr管中。使用tapestation上的高灵敏度d1000屏幕录像对最终文库进行量化(图10)。该文库在nextseq 500上测序。总计产生了25,123,635个测序读数对。98.5%的读数对包含有效的条形码。使用cell ranger v1.2.0进一步分析鉴定了1,109个细胞(图11),每个细胞有2,954个片段中位数。拐点图证明了清晰的单细胞行为。
[0084]
参考文献
[0085]
adey a.等2010.genome biol.11,r119。
[0086]
amini s.等2014.nature genetics,46(12):1343-1349。
[0087]
au,t.等2004.embo j.,23:3408-3420。
[0088]
buenrostro j.d.等2013.nature methods,10(12):1213-1218。
[0089]
buenrostro,j.d.等2015.nature,523:486-490。
[0090]
burton b.m.和baker t.a.2003.chemistry&biology 10:463-472。
[0091]
caruccio n.2011.methods mol.biol.733:241-255。
[0092]
kavanagh i,kiiskinen l.l.和haakana h.2013.美国专利申请公开us2013/0023423。
[0093]
kurihara k.等2011.nat.chem.3:775-781。
[0094]
laouini a.等2012.colloid sci.biotechnol.1:147-168。
[0095]
mizuuchi m.,baker t.a.和mizuuchi k.1992.cell 70,303-311。
[0096]
picelli s.等2014.genome research 24,2033-2040。
[0097]
savilahti h.,p.a.rice和k.mizuuchi.1995.embo j.14:4893-4903。
[0098]
stoeckius m.,等2017.nature methods 14:865-868。
[0099]
surette m.,buch s.j.和chaconas g.1987.cell 70:303-311。
[0100]
reznikoffw.s.2008.annual review of genetics 42(1):269-286。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1