一种稻瘟病穗颈瘟抗性鉴定菌系筛选方法及鉴定方法
本发明涉及稻瘟病抗性鉴定领域,具体涉及一种稻瘟病穗颈瘟抗性鉴定菌系筛选方法及鉴定方法。
背景技术:
水稻稻瘟病是一种世界性的重要真菌病害,病害流行的年份严重影响水稻产量和稻米品质,严重地区甚至颗粒无收。选育和利用抗病水稻品种是防治稻瘟病的最有效措施,但稻瘟病菌生理小种不断变化且变异较快,育成的抗病水稻品种通常种植数年后极易丧失抗性。因此,稻瘟病抗性鉴定工作对水稻品种的抗病安全布局来说是一项极其重要的工作,只有在水稻育种源头明确水稻品种的抗性,才能在实际生产中选择抗性好的品种,减少稻瘟病的发生造成损失。
稻瘟病抗性鉴定主要有田间自然诱发鉴定与室内鉴定两种方法,田间自然诱发鉴定由于受当年气候等环境因素的影响往往不能够得到准确的水稻品种抗性结果,相对而言,室内稻瘟病鉴定可以人为的控制温度,湿度等稻瘟病发病条件能够充分保证抗性鉴定结果的准确性,但室内稻瘟病鉴定的一个关键因素是稻瘟病菌的选择,鉴定菌株必须具有某一地区菌群的代表性才能真实的反应水稻品种在该地区田间种植后能够表现的抗性水平。如何有效的筛选具有代表性的鉴定菌系是稻瘟病抗性鉴定特别是穗颈瘟抗性鉴定成功与否的关键所在。
传统的稻瘟病抗性鉴定菌株筛选是通过苗期接种某些水稻品种后的抗感反应型来确定的,该方法具有一定的可行性,但接种结果容易受人员操作影响、且较耗时耗力;另外,由于该方法筛选的抗性鉴定菌株是通过水稻苗期接种结果确定的,是否对穗颈瘟抗性具有较好的鉴定能力却无法确定,没有证据表明水稻对苗(叶)瘟的抗性与穗颈瘟的具有绝对相关性。
技术实现要素:
因此,本发明要解决的技术问题在于克服现有技术中的稻瘟病抗性鉴定菌株筛选容易受人员操作影响,耗时耗力,造成对穗颈瘟抗性鉴定结果不够准确的缺陷,从而提供一种本方法通过分子测定某一地区的稻瘟病菌群致病基因与无毒基因情况,根据测定结果进行聚类划分成不同的遗传宗谱,从不同的宗谱中筛选一定的菌株组成代表该地区稻瘟病菌群的抗性鉴定菌系,可以很好的为该地区的水稻抗病育种与抗病品种筛选提供有效的助力。该方法具有高效、准确、省时省力的特点,对水稻稻瘟病特别是穗颈瘟的抗性鉴定有很重要的应用潜力。
为此,本发明提供了如下的技术方案:
一种稻瘟病穗颈瘟抗性鉴定菌系筛选方法,包括:
s1、采集稻瘟病标本,然后进行稻瘟病菌单孢菌的分离、保存;
s2、提取所述的稻瘟病菌单孢菌的dna,以所述dna为模板,以稻瘟病菌无毒基因与致病基因设计引物,进行pcr扩增,对稻瘟病菌无毒基因与致病基因进行测定;稻瘟病菌致病基因为mpg1、mps1、magb、cpka、mnh1、mgyca1、mgs11和mgatg5;稻瘟病菌无毒基因为avr-pia、avr-pit、avr-pik、avr-pita、avr-pizt、avr-co39、ace1和pre1;
s3、对稻瘟病菌无毒基因与致病基因测定结果进行聚类分析,根据pcr扩增条带的结果,有条带记为“1”,无条带记为“0”,构建基于“0-1”数据库,应用ntsys-pc数据处理系统计算遗传距离,以菌株dna扩增条带的位置相似水平为基准,用最长距离法对菌株群体遗传多样性进行聚类分析,将菌株归类成不同的组,每一个组称为一个遗传宗谱,对遗传宗谱进行编号i;
s4、设定稻瘟病穗颈瘟抗性鉴定菌系总菌株数n,计算每个遗传宗谱的稻瘟病菌株占所有菌株的百分比mi,依据所述百分比的大小,由小到大依次计算对应遗传宗谱中随机挑选菌株数,在编号为i的遗传宗谱中随机挑选菌株pi=(n-w已挑选总和)×mi个,pi值取大于0整数,w已挑选总和表示编号1-编号i-1的遗传宗谱随机挑选菌株数之和,最后挑选出的菌株作为稻瘟病穗颈瘟抗性鉴定菌系。
可选的,在s1步骤中,将采集的水稻穗颈部稻瘟病标本剪成5-8寸,浸水18-24h,然后用水冲洗2-3次,再用灭菌水冲洗1次,置于保湿培养皿中,于28℃恒温培养箱内暗培养2d,观察到标样上有灰色或灰绿色的孢子后,采用眉毛挑单孢法,将病部置于显微镜下挑取单个孢子在pda斜面培养基中培养,纯培养后转接到放有灭菌滤纸片的pda培养基培养,待滤纸片长满菌丝挑出放在在无菌的羊皮纸袋中,置于-80℃超低温冰箱保存。
可选的,在s2步骤中,提取dna的方法包括:将保存的稻瘟病菌株活化后,挑取菌丝接种到pd液体培养基中,置于120r/min摇床上28℃培养3~6d,将长出的菌丝体用灭菌纱布过滤收集、烘干并保存于-20℃备用;将收集的菌丝体分装于2ml的离心管中,加2颗直径为5mm灭菌碳钢珠后迅速加65℃预热的ctab提取液,然后进行磨样,最后提取基因组dna,保存于-20℃冰箱备用。
可选的,在s2步骤中,对稻瘟病菌无毒基因与致病基因设计的引物如seqidno.1-seqidno.32所示。
可选的,在s2步骤中,所述pcr扩增的体系如下:
dna模板1μl(≤200ng);
正向引物,浓度为0.2~0.4μmol/l,1μl;
反向引物,浓度为0.2~0.4μmol/l,1μl;
dntp,浓度2.5mmol/l,0.5μl;
taqdna聚合酶,浓度为5000u/ml,0.2μl;
10×buffer,含mg2+,2μl;
ddh2o,14.3μl。
可选的,在s2步骤中,所述pcr扩增的反应程序:95℃预变性5min;94℃变性30s,55~57℃退火1min,72℃延伸60~90s,共35个循环;最后72℃延伸10min。
可选的,在s3步骤中,以相似系数0.75进行分组。
本发明提供了一种稻瘟病穗颈瘟抗性鉴定方法,利用所述的稻瘟病穗颈瘟抗性鉴定菌系筛选方法中筛选出的稻瘟病穗颈瘟抗性鉴定菌系鉴定。
可选的,采用离体穗段点滴接种法鉴定。
可选的,选取待测水稻品种按大田正常栽培,待长至孕穗破口初期穗长露出1~2cm时从田间每一品种的小区内剪取稻穗20个,然后剪取长约7~8cm长的穗段,穗颈节上部2cm,下部5~6cm,然后将穗段放入双层过滤纸的培养皿内,每皿加2ml的无菌水浸润,在每根穗段的穗颈节位点及节下4cm处各滴入5μl的稻瘟病穗颈瘟抗性鉴定菌系的孢子悬浮液,每根穗段滴入一个抗性鉴定菌株的孢子悬浮液,将培养皿放入26℃的光照培养箱中,并用透明的薄膜包被,以保持培养皿内的湿度,光暗交替培养10-12天,离体接种培养12天后,检查各培养皿穗颈的发病情况,分级标准如下:0级:无病斑;1级:病斑长度不超过5mm;3级:病斑长度6~15mm;5级:病斑长度超过16mm,但病斑间不相连;7级:病斑相互联结,≥50%且<80%穗段发病;9级:病斑相互联结,80%以上穗段发病;统计分级并计算平均病级;
平均病级小于1为抗病;平均病级>1且≤3为中抗;平均病级>3,且≤5为中感;平均病级>5且≤7为感病;平均病级>7为高感;计算抗性频率%=抗病菌株数/总菌株数×100%。
本发明技术方案,具有如下优点
1.本发明提供的一种稻瘟病穗颈瘟抗性鉴定菌系筛选方法,其特征在于,包括:
s1、采集稻瘟病标本,然后进行稻瘟病菌单孢菌的分离、保存;
s2、提取所述的稻瘟病菌单孢菌的dna,以所述dna为模板,以稻瘟病菌无毒基因与致病基因设计引物,进行pcr扩增,对稻瘟病菌无毒基因与致病基因进行测定;稻瘟病菌致病基因为mpg1、mps1、magb、cpka、mnh1、mgyca1、mgs11和mgatg5;稻瘟病菌无毒基因为avr-pia、avr-pit、avr-pik、avr-pita、avr-pizt、avr-co39、ace1和pre1;
s3、对稻瘟病菌无毒基因与致病基因测定结果进行聚类分析,根据pcr扩增条带的结果,有条带记为“1”,无条带记为“0”,构建基于“0-1”数据库,应用ntsys-pc数据处理系统计算遗传距离,以菌株dna扩增条带的位置相似水平为基准,用最长距离法对菌株群体遗传多样性进行聚类分析,将菌株归类成不同的组,每一个组称为一个遗传宗谱,对遗传宗谱进行编号i;
s4、设定稻瘟病穗颈瘟抗性鉴定菌系总菌株数n,计算每个遗传宗谱的稻瘟病菌株占所有菌株的百分比mi,依据所述百分比的大小,由小到大依次计算对应遗传宗谱中随机挑选菌株数,在编号为i的遗传宗谱中随机挑选菌株pi=(n-w已挑选总和)×mi个,pi值取大于0整数,w已挑选总和表示编号1到编号i-1的遗传宗谱随机挑选菌株数之和,最后挑选出的菌株作为稻瘟病穗颈瘟抗性鉴定菌系;
上述方法通过分子测定某一地区的稻瘟病菌群致病基因与无毒基因情况,根据测定结果进行聚类划分成不同的遗传宗谱,从不同的宗谱中筛选一定的菌株组成代表该地区稻瘟病菌群的抗性鉴定菌系,可以很好的为该地区的水稻抗病育种与抗病品种筛选提供有效的助力。该方法具有高效、准确、省时省力的特点,对水稻稻瘟病特别是穗颈瘟的抗性鉴定有很重要的应用潜力。
附图说明
为了更清楚地说明本发明具体实施方式或现有技术中的技术方案,下面将对具体实施方式或现有技术描述中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图是本发明的一些实施方式,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他的附图。
图1是本发明实施例1中的pcr扩增条带的结果;
图2是本发明实施例1中稻瘟病菌致病基因与无毒基因分子检测结果聚类结果;
图2-a是本发明实施例1中稻瘟病菌致病基因与无毒基因分子检测结果聚类结果的局部放大图;
图2-b是本发明实施例1中稻瘟病菌致病基因与无毒基因分子检测结果聚类结果的局部放大图;
图2-c是本发明实施例1中稻瘟病菌致病基因与无毒基因分子检测结果聚类结果的局部放大图;
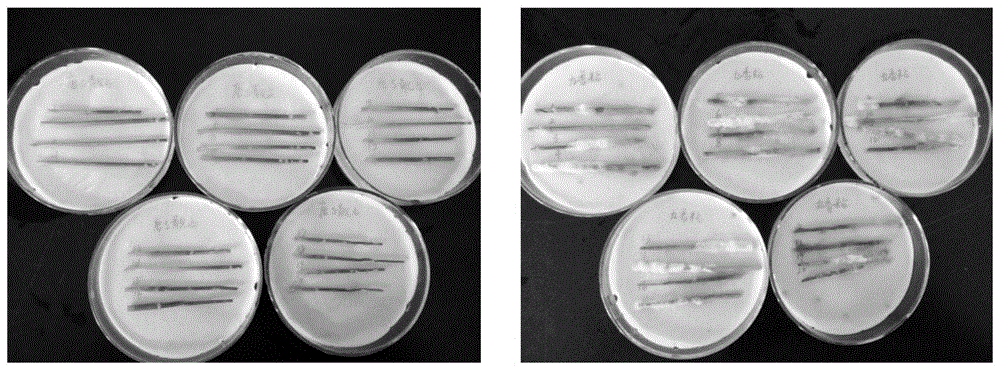
图3是本发明实施例2中室内离体抗性鉴定菌系穗颈瘟抗性鉴定结果。
具体实施方式
提供下述实施例是为了更好地进一步理解本发明,并不局限于所述最佳实施方式,不对本发明的内容和保护范围构成限制,任何人在本发明的启示下或是将本发明与其他现有技术的特征进行组合而得出的任何与本发明相同或相近似的产品,均落在本发明的保护范围之内。
实施例中未注明具体实验步骤或条件者,按照本领域内的文献所描述的常规实验步骤的操作或条件即可进行。所用试剂或仪器未注明生产厂商者,均为可以通过市购获得的常规试剂产品。
实施例1一种稻瘟病穗颈瘟抗性鉴定菌系筛选
本实施例提供了一种稻瘟病穗颈瘟抗性鉴定菌系筛选方法,包括:
(1)稻瘟病标本的采集
在某一地区种植高感品种丽江新团黑谷,在半黄熟期采集稻瘟病发病标样用牛皮纸袋装好,记录好标样采集地点、时间、寄主品种、采集人等信息。
(2)稻瘟病菌单孢菌的分离、保存
取采集的水稻穗颈部稻瘟病标样剪成5寸-8寸,浸水18~24h,待充分吸水后,用水冲洗2~3次,再用灭菌水冲洗一次,放入垫以u形玻棒的保湿培养皿中,置于28℃恒温培养箱内暗培养2d,观察到标样上有灰色或灰绿色的孢子后,采用眉毛挑单孢法,将病部置于显微镜下挑取单个孢子在pda斜面培养基中培养。纯培养后转接到放有灭菌滤纸片的pda培养基培养,待滤纸片长满菌丝挑出在无菌的羊皮纸袋中,置于-80℃超低温冰箱保存,在本实施例中从同一地区分离保存到189个稻瘟病菌单孢菌株,编号为wy1~wy189。
(3)稻瘟病菌dna的提取
将分离保存好的稻瘟病菌株活化后,挑取菌丝接种到pd液体培养基中,置于120r/min摇床上28℃培养3~6d,将长出的菌丝体用灭菌纱布过滤收集、烘干并保存于-20℃备用。之后将收集的菌丝体分装于2ml的eppendorf管中,加2颗5mm灭菌碳钢珠后迅速加65℃预热的ctab提取液,应用全自动样品快速研磨仪进行磨样,按照天根植物基因组提取试剂盒tiangen(dp350)说明书提取方法对病菌基因组dna进行提取,保存于-20℃冰箱备用。
(4)稻瘟病菌无毒基因与致病基因测定
选用已公开报道且克隆的8个稻瘟病菌致病基因与8个无毒基因作为检测目的基因,8个稻瘟病菌致病基因为mpg1、mps1、magb、cpka、mnh1、mgyca1、mgs11、mgatg5,8个稻瘟病菌无毒基因为avr-pia、avr-pit、avr-pik、avr-pita、avr-pizt、avr-co39、ace1、pre1,根据以上基因序列设计特异性引物标记(表1,序列表中seqidno.1-32所示)。以步骤(3)中获得的基因组dna为模板,利用上述引物分别对稻瘟病菌无毒基因与致病基因进行pcr扩增。pcr总反应体系为20μl,包括dna模板1μl(≤200ng)、正向引物(0.2~0.4μmol/l)1μl、反向引物(0.2~0.4μmol/l)1μl、2.5mmol/ldntp0.5μl、taqdna聚合酶(浓度为5000u/ml)0.2μl、10×buffer(含mg2+)2μl,ddh2o14.3μl。pcr反应程序:95℃预变性5min;94℃变性30s,55~57℃(按引物设定温度,见表1)退火1min,72℃延伸60~90s(根据片段大小而定,见表1),共35个循环;最后72℃延伸10min。获得pcr产物保存于4℃冰箱,用1%琼脂糖凝胶电泳检测,最后在凝胶成像系统紫外灯下拍照保存,部分结果见图1。
表1稻瘟病菌致病基因与无毒基因分子检测引物序列
(5)稻瘟病菌无毒基因与致病基因分子测定结果的聚类分析
根据pcr扩增条带的结果(图1),有条带则记为“1”,无条带则记为“0”,构建基于ssr的“0-1”数据库;应用ntsys-pc数据处理系统计算遗传距离,以菌株dna扩增条带的位置相似水平为基准,用最长距离法对菌株群体遗传多样性进行聚类分析。通过聚类可以将菌株归类成不同的密切相关的组,每一个组称为一个遗传宗谱,不同的相似系数可以划分成不同数量的遗传宗谱(图2、图2-a、图2-b、图2-c)。本实施例中分离供试的189个稻瘟病菌株在相似系数0.75时可分成7个遗传宗谱(见表2)。
表2遗传相似系数0.75水平下稻瘟病菌的遗传宗谱聚类结果
(6)稻瘟病抗性鉴定菌系的确定
一般确定稻瘟病抗性鉴定菌系总菌株数为20个比较合适,因此,在本实施例中设定稻瘟病抗性鉴定菌系总菌株数为20个,再根据每个宗谱的稻瘟病菌株占所有菌株的百分比作为菌系中各宗谱菌株所占的百分比随机挑选菌株,依据所述百分比的大小,由小到大依次计算对应遗传宗谱中随机挑选菌株数,各遗传宗谱的菌株百分比见表3,依据所述百分比的由小到大依次计算l5、l7(或l7、l5)、l2、l3、l6(或l6、l3)、l1、l4。如果鉴定菌系总菌株数与宗谱菌株数百分比相乘小于1则只挑选1个作为鉴定菌株,其他宗谱按照剩余鉴定菌株数与其占比确定筛选鉴定菌株数,计算如下:
l5=20×0.53%<1,故遗传宗谱l5鉴定菌系株数只挑选1个作为鉴定菌株;
l7=19×0.53%<1,故遗传宗谱l7鉴定菌系株数只挑选1个作为鉴定菌株;
l2=18×3.17%<1,故遗传宗谱l2鉴定菌系株数只挑选1个作为鉴定菌株;
l3=17×5.29%<1,故遗传宗谱l3鉴定菌系株数只挑选1个作为鉴定菌株;
l6=16×5.29%<1,故遗传宗谱l6鉴定菌系株数只挑选1个作为鉴定菌株;
l1=15×21.69%=3.2535,四舍五入取整数等于3,故遗传宗谱l1鉴定菌系株数挑选3个作为鉴定菌株;
l4=20-1-1-1-1-1-3=12,故遗传宗谱l4鉴定菌系株数挑选12个作为鉴定菌株。综上,本实施例中确定的20个稻瘟病菌株组成的抗性鉴定菌系包括l1宗谱3个、l2宗谱1个、l3宗谱1个、l4宗谱12个、l5宗谱1个、l6宗谱1个、l7宗谱1个(表3)。
表3稻瘟病抗性鉴定菌系的筛选结果
实施例2抗性鉴定菌系室内稻瘟病鉴定结果的验证
利用实施例1中筛选到的20个抗性鉴定菌系采用离体穗段点滴接种法鉴定。选取不同抗性的10个水稻育种中间材料按大田正常栽培,待长至孕穗破口初期从田间每一品种的小区内剪取穗长露出1~2cm的稻穗20个,带回实验室,剪取长约7~8cm长的穗段(穗颈节上部2cm,下部5~6cm),然后将4根穗段放入双层过滤纸的培养皿内(每皿加2ml的无菌水),用移液器在每根穗段的穗颈节位点及节下约4cm处各滴人5μl的抗性鉴定菌系孢子悬浮液(每根穗段滴入一个抗性鉴定菌株孢子悬浮液)。将培养皿放入26℃的光照培养箱中,并用透明的薄膜包被,以保持培养皿内的湿度。光暗交替培养10-12天。离体接种培养12天后,检查各培养皿穗颈的发病情况(部分检测结果如图3所示,左图为唐s靓占,右图为九香粘),分级标准如下:0级:无病斑;1级:病斑长度不超过5mm;3级:病斑长度6~15mm;5级:病斑长度超过16mm,但病斑间不相连;7级:病斑相互联结,≥50%且<80%的穗段发病;9级:病斑相互联结,80%以上穗段发病。将平均病级小于1的定为抗病;大于1,小于等于3的定为中抗;大于3,小于等于5的定为中感;大于5,小于等于7的定为感病;大于7的定为高感。抗性频率(%)=(抗病菌株数/总菌株数)×100%。
田间自然诱发鉴定将10个供试水稻品种按正常田间操作种植在抗性鉴定菌系来源地区大田,种植过程中不施杀菌剂,待长至半黄熟期时随机调查每个品种31根穗,计算平均病级(按nyt2646-2014水稻品种试验稻瘟病抗性鉴定与评价技术规程)以及病穗率。病穗率(%)=(发病穗数/总穗数)×100%。
通过两种方法对10个水稻品种稻瘟病抗性鉴定结果显示(表4、图3),利用本方法筛选的抗性鉴定菌系进行的稻瘟病离体鉴定结果与田间自然诱发鉴定结果表现为一致,能够很好的对水稻品种对稻瘟病(穗颈瘟)的抗性水平进行鉴定,该方法能够节省大量的人力物力,且不受环境影响,准确度更高。
表4抗性鉴定菌系室内鉴定与田间自然诱发鉴定的结果比对验证
显然,上述实施例仅仅是为清楚地说明所作的举例,而并非对实施方式的限定。对于所属领域的普通技术人员来说,在上述说明的基础上还可以做出其它不同形式的变化或变动。这里无需也无法对所有的实施方式予以穷举。而由此所引伸出的显而易见的变化或变动仍处于本发明创造的保护范围之中。
序列表
<110>江西省农业科学院植物保护研究所
<120>一种稻瘟病穗颈瘟抗性鉴定菌系筛选方法及鉴定方法
<160>32
<170>siposequencelisting1.0
<210>1
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>1
gccactctttagactccca19
<210>2
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>2
gctccctcacagaaactcc19
<210>3
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>3
gcatcgcttgtattaccc18
<210>4
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>4
cgacatcctaccctactca19
<210>5
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>5
ggcagcaccctattgatt18
<210>6
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>6
ccagatgacccagaagttta20
<210>7
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>7
actgcaacctggcaatcta19
<210>8
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>8
ttccgctgtctttagttcc19
<210>9
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>9
atgtggatccgcccagctacga22
<210>10
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>10
tcataggttaagttccatcttc22
<210>11
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>11
caaggttgtgttttggaccgc21
<210>12
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>12
ggcatttgcttcttgtttccca22
<210>13
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>13
atgatggaaccaaccaaggaga22
<210>14
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>14
gtaaataagcttatcgtcgtaa22
<210>15
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>15
atggcttcgccgcgccgatc20
<210>16
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>16
taatggcacgacggttaaac20
<210>17
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>17
tctcgggaagctgattgagt20
<210>18
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>18
ctaacggccggctaacaaac20
<210>19
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>19
gaatcgcagcacttcccact20
<210>20
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>20
ataactgtccgccgctcgt19
<210>21
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>21
actttgggaactgtcgctgtc21
<210>22
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>22
agctctaacaggttccagcatc22
<210>23
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>23
cgacccgtttccgcctttatt21
<210>24
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>24
tccctccattccaacactaacg22
<210>25
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>25
aaaccagggcagccaaaga19
<210>26
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>26
attcccaatcgagccaacg19
<210>27
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>27
aattgcataatcgctgcgat20
<210>28
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>28
gtcaagctcagaactttgtt20
<210>29
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>29
cccagagttggcgatgatgc20
<210>30
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>30
atgtggcggtgacagaggac20
<210>31
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>31
tcgtgagggttcctatctgc20
<210>32
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>32
gacctttatcggatgcgtgt20
- 还没有人留言评论。精彩留言会获得点赞!