小鼠表观遗传基因敲除筛选文库及其构建方法
本发明涉及小鼠表观遗传基因敲除筛选文库及其构建方法,属于基因生物技术领域。
背景技术:
crispr/cas9基因敲除技术是目前应用的最广泛最高效的基因编辑技术,并在此技术基础上发展出多种基因编辑方法,包括单碱基突变、基于crispr/cas9技术的同源重组等方法,在基因治疗、药物筛选等领域得到广泛的应用。
crispr/cas9基本原理为在sgrna的靶向作用下,cas9蛋白可以识别并结合到目的基因的编辑位置,将基因组切开导致双链dna断裂,引起细胞内的dna损伤修复。在细胞内主要的dna损伤修复途径为非同源末端连接(non-homologousendjoining,nhej),这种修复方式会导致目的基因的基因组上的插入或者缺失(indel),使目的基因无法正常的转录翻译,从而导致基因敲除。
目前现有的敲除筛选文库存在的缺陷有:1、主要是针对整个基因组基因的敲除文库,包括人类和小鼠的全基因组敲除文库,并没有设计获得专门针对表观遗传因子的敲除文库;2、不能很好的和单细胞测序技术结合起来,需要进行多次单细胞测序才能得到整个文库的敲除测序结果;3、目前的敲除筛选文库是所有敲除质粒的混合文库,在扩增过程中每个基因敲除质粒的扩增效率不同,导致后续的敲除筛选结果不够客观;4、目前的敲除筛选文库是单质粒表达系统,相对于双质粒之系统来说,在文库构建过程中单质粒系统的文库构建更为复杂。
技术实现要素:
(一)要解决的技术问题
为了解决现有技术的上述问题,本发明提供小鼠表观遗传基因敲除筛选文库,是专门针对小鼠表观遗传因子的敲除文库。
(二)技术方案
为了达到上述目的,本发明采用的主要技术方案包括:
小鼠表观遗传基因敲除筛选文库,其包括靶向表观遗传相关基因的sgrna表达质粒,靶向表观遗传相关基因包括赖氨酸乙酰转移酶基因、组蛋白去乙酰酶基因、组蛋白赖氨酸甲基转移酶基因、组蛋白赖氨酸去甲基化酶基因、蛋白质精氨酸甲基转移酶基因、组蛋白识别、dna甲基化相关基因、rna甲基化相关基因、染色质重塑复合物基因、染色质组装及组蛋白伴侣基因。
如上所述的小鼠表观遗传基因敲除筛选文库,优选地,所述赖氨酸乙酰转移酶基因的sgrna序列如seqidno.1-36所示,组蛋白去乙酰酶基因的sgrna序列如seqidno.37-89所示,组蛋白赖氨酸甲基转移酶基因的sgrna序列如seqidno.90-167所示,组蛋白赖氨酸去甲基化酶基因的sgrna序列如seqidno.168-257所示,蛋白质精氨酸甲基转移酶基因的sgrna序列如seqidno.258-291所示,组蛋白识别基因的sgrna序列如seqidno.292-330所示,dna甲基化相关基因的sgrna序列如seqidno.331-384所示,rna甲基化相关基因的sgrna序列如seqidno.385-474所示,染色质重塑复合物基因的sgrna序列如seqidno.475-648所示,染色质组装及组蛋白伴侣基因的sgrna序列如seqidno.649-695所示。
如上所述小鼠表观基因敲除筛选文库的构建方法,其包括如下步骤:
s1、针对靶向基因(小鼠表观遗传相关基因)的蛋白编码区的外显子设计sgrna序列;设计时遵循的原则如下:(1)设计的sgrna的靶向位点一般要位于基因蛋白编码区的第一、第二外显子上或者第三外显子;(2)设计时sgrna的设计评分要尽可能的高;
s2、分别合成设计好的sgrna序列以及sgrna序列的互补序列,并进行退火;
s3、对sgrna表达质粒进行酶切,获得sgrna表达质粒酶切产物;
s4、将sgrna表达质粒酶切产物与步骤s2中的退火产物,使用t4dna连接酶,进行连接;
s5、随后将连接产物进行转化,并挑取单克隆进行摇菌测序;
s6、将测序结果正确的菌液进行大量扩增,从而获得了单个表观因子sgrna表达质粒,构成的小鼠表观因子基因敲除筛选文库。
如上所述小鼠表观基因敲除筛选文库的构建方法,优选地,在步骤s3中,采用内切酶bsmb1进行酶切,在步骤s4中,退火的程序为37℃30min,95℃5min,rampto25℃5℃/5min,25℃5min,4℃+∞。
如上所述小鼠表观基因敲除筛选文库的构建方法,优选地,还包括对文库的验证,所述验证包括对于构建的sgrna表达质粒本身的验证和对与设计的sgrna是否可以正常引导spcas9蛋白对目的基因的设计位点进行切割。
如上所述小鼠表观基因敲除筛选文库的构建方法,优选地,所述对于构建的sgrna表达质粒本身的验证包括将小鼠表观因子敲除筛选文库进行慢病毒包装,随后感染小鼠成肌细胞c2c12,感染后12h后换液加入嘌呤霉素筛选,筛选40~48h后收取细胞并提基因组,对慢病毒质粒的5’ltr与3’ltr之间的序列设计引物,进行pcr扩增,对扩增产物进行高通量测序,测序结果中的序列与文库sgrna序列进行比对。
如上所述小鼠表观基因敲除筛选文库的构建方法,优选地,所述pcr扩增所设计引物序列如seqidno.696和seqidno.697所示。其中,seqidno.696:atttcccatgattccttca,seqidno.697:gactcggtgccactttttc。
如上所述小鼠表观基因敲除筛选文库的构建方法,优选地,对目的基因的设计位点进行切割是根据sgrna靶点位置附近的基因组序列设计引物进行pcr,将pcr产物进行纯化,随后对纯化后的pcr产物进行重新的变性退火,随后依次添加17μl重新变性退火的产物、1μlt7e1酶以及2μl10×nebbuffer2组成t7e1酶切体系,在37℃条件下酶切过夜,对酶切的产物进行琼脂糖凝胶电泳,检测观察到三条条带的出现,说明所设计的sgrna都能很好的引导spcas9对靶点序列进行切割。
(三)有益效果
本发明的有益效果是:
本发明提供的小鼠表观遗传基因敲除筛选文库,为双质粒表达系统,质粒相对较小,构建比较容易操作;同时筛选文库仅仅针对表观遗传基因,文库相对较小,进行单细胞测序相对容易进行数据分析。
本发明提供的小鼠表观遗传基因敲除筛选文库,采用了慢病毒骨架质粒,在难以表达的原代细胞以及细胞系中都可以较稳定高效的表达,提高了基因的敲除效率,进而扩展了该文库的使用范围,在众多的免疫、肿瘤、衰老、疾病、发育模型中都可以运用。同时文库为单独质粒形态存储,可以根据自己的需要构建不同的文库类型。在文库质粒扩增过程中,不会面临混合文库扩增过程中出现质粒正态分布式扩增,导致针对每个基因的质粒出现比例不均一的情况。
附图说明
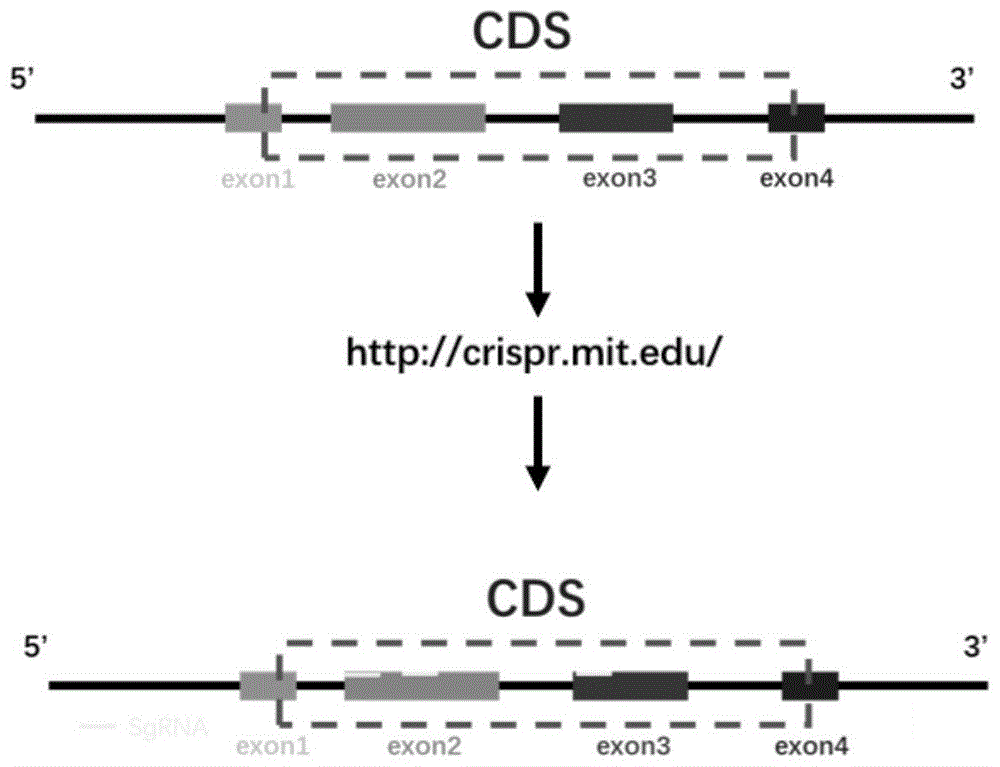
图1为确定sgrna的序列及最终靶点位置的示意图;
图2为双质粒系统的质粒结构示意图;
图3为评分低于75分的基因示意图;
图4为t7e1酶切检测原理示意图;
图5为采用酶t7e1酶切后的琼脂糖凝胶电泳结果。
具体实施方式
为了更好的解释本发明,以便于理解,下面结合附图,通过具体实施方式,对本发明作详细描述。
实施例1
对sgrna的设计,综合考虑靶点位置以及sgrna在线设计评分两方面来确定sgrna的序列及最终靶点位置。本发明中选择靶向基因的即表观遗传相关基因蛋白质编码区(codingsequence,cds)的第一、第二或者第三外显子作为靶向位点,随后在网站http://crispr.mit.edu/进行在线设计,最终根据设计序列评分确定sgrna序列。示意图如1所示,具体包括1、选择cds区位置靠前的外显子(exon1、exon2或者exon3)作为潜在的靶向位点位置;2、http://crispr.mit.edu/进行在线设计;3、结合sgrna靶向序列位置以及在线设计sgrna评分,通过实验确定最终sgrna序列。
最后确定最终选择了针对小鼠表观遗传相关的共计234个基因的敲除筛选文库,sgrna共计695条。根据功能分类,将这234个基因共分为10类,包括赖氨酸乙酰转移酶(lysineactyltransferases)、组蛋白去乙酰酶(histonedeactylases)、组蛋白赖氨酸甲基转移酶(histonelysinemethyltransferase)、组蛋白赖氨酸去甲基化酶(histonelysinedemethylases)、蛋白质精氨酸甲基转移酶(proteinargininemethyltransferases)、组蛋白识别(histonereader)、dna甲基化相关(dnamethylationrelated)、rna甲基化相关(rnamethylationrelated)、染色质重塑复合物(chromatinremodelingcomplexes)以及染色质组装及组蛋白伴侣(chromatinassemblyandhistonechaperone),其各功能基因具体信息见下表1和2,设计获得sgrna详细的序列信息及靶向位置见表3。
表1
表2
表3详细的序列信息及靶向位置
采用双质粒系统构建小鼠表观基因敲除筛选文库,质粒addgene编号为:52962、52963,质粒结构如图2。双质粒系统即为spcas9蛋白的表达与sgrna的表达分别为单独的质粒,且该双质粒系统的质粒骨架为慢病毒骨架,这为原代细胞等一些转染效率低的细胞的筛选提供了较好的选择。
质粒构建的体系步骤如下:
(1)sgrna表达质粒酶切:sgrna表达质粒(52963)6μl/10μg,内切酶bsmb12μl,内切酶buffer2μl,ddh2o10μl共计20μl,55℃反应过夜,获得sgrna表达质粒的酶切产物。
(2)sgrna引物序列退火:分别合成设计好的sgrna序列(见表3中seqidno.1-695所示序列)以及sgrna序列的互补序列,正向引物(指表3中的设计的sgrna序列,浓度为10μmol)5μl,反向引物(表3中sgrna序列的互补序列,浓度为10μmol)5μl,10×pcrbuffer5μlh2o35μl,共计50μl体系;
反应程序如下:37℃30min,95℃5min,rampto25℃5℃/5min,25℃5min,4℃+∞。,获得dna双链之后才能和酶切的载体连接,
(3)连接:步骤(1)获得sgrna表达质粒的酶切产物0.5μl,sgrna引物退火产物5μl,t4dna连接酶0.5μl,10×buffer3μl,h2o21μl混合后,16℃连接过夜。
随后将连接产物进行转化,转化到感受态transstbl3,(产品为全式金公司,货号为cd521),挑取单克隆并进行测序验证,选择测序结果正确的菌落进行大量扩增,从而获得了单个表观因子sgrna表达质粒构成的小鼠表观基因敲除筛选敲除文库。
对于构建好的小鼠表观因子敲除筛选文库,需要对文库的质量进行验证,以保证所构建的文库是成功可用的,文库在后续的筛选过程中是不会因为spcas9对靶基因的切割效率成为筛选的限速步骤。对于文库的验证主要包括两个方面:
1、对于构建的sgrna表达质粒本身的验证,即sgrna慢病毒表达质粒是否可以正常的进行慢病毒包装以及能否正常的整合到细胞的基因组上;2、对与设计的sgrna是否可以正常引导spcas9蛋白对目的基因的设计位点进行切割,在sgrna序列设计过程中,每一条sgrna序列设计都会有评分,所以我们对sgrna序列进行的随机挑选与固定挑选相结合,随机挑选部分sgrna序列以及将评分低于75分的序列挑选出来进行验证。
首先,将小鼠表观因子敲除筛选文库进行慢病毒包装,慢病毒的包装及纯化步骤如下:
(1)hek293t细胞准备:因为慢病毒包装过程对细胞依赖性很强,所以需要选择来源明确且生长状态良好的hek293t细胞,将复苏的细胞用支原体抑制剂biomyc-3(购自biologicalindustries公司);使用方法为在细胞培养过程中按照1:100的比例加入到培养基中。处理两代,保证细胞状态,进行文库病毒包装准备10个10cm培养皿的细胞。
(2)转染包装质粒:按照阳离子介导的细胞转染方法进行转染,具体为①转染前接种适宜密度的hek293t细胞;
②细胞密度达到70%~90%时进行转染,转染前1h将更换培养基;
③吸取400μl0.9%的灭菌生理盐水于1.5mlep管中,加入8μg待转染质粒;其中,待转染质粒为按质量比例文库质粒(即上述构建好的文库质粒):pspax2:pmd2.g=4:3:1混合的质粒;
④再次吸取400μl0.9%的灭菌生理盐水于1.5mlep管中,加入8μlvigofect转染试剂,混匀后室温静置5min;
⑤将第④步稀释的vigofect逐滴加入到第③步中的质粒中,室温放置15min;
⑥将静置后的混合液均匀滴加到细胞培养基中并混匀;
⑦转染6~8h后含有10%胎牛血清的dmem培养基。
其中,病毒包装质粒的比例为文库质粒:pspax2:pmd2.g=4:3:1的质量比例转染hek293t细胞,转染6~8h之后更换培养基,根据需要感染的细胞选择合适的培养基换液,c2c12为含有10%血清的dmem培养基。(pspax2:addgene官网编号为12260;pmd2.g:addgene官网编号为12259)
(3)病毒收取:转染36~48h之后可以收取细胞培养上清,用0.45μm滤器过滤去掉细胞碎片,并用millipore柱子浓缩病毒液,分装之后-80℃长期保存。(0.45μm滤器为pallcorporation公司产品,货号为4612;浓缩柱子为millipore公司产品,货号为acs510024)
将得到的浓缩病毒感染细胞:
(1)药物浓度筛选:将c2c12细胞以低密度接种在六孔板种,待贴壁生长之后,设置筛选标记药物嘌呤霉素按浓度梯度为0、2、4、6、8、10μg/ml浓度梯度加入到六孔板种,加入24h后观察细胞生存状况,选择细胞接近90%死亡时的浓度为2μg/ml嘌呤霉素。
(2)慢病毒感染细胞:复苏需要构建细胞系c2c12细胞,待生长状态良好时,接种细胞接种贴壁后密度达到30%~40%的细胞在6cm培养皿中,细胞贴壁之后,更换为含有终浓度为8μg/mlpolybrene的培养基,并加入20μl左右的浓缩病毒液,感染6~8h之后换为新鲜正常的培养基。
(3)药物筛选:感染36h后加入含有选定筛选药物浓度即嘌呤霉素2μg/ml培养基进行筛选,至不会有死细胞出现。在筛选过程中细胞密度过高时要进行传代处理,保证筛选过程中细胞密度不超过80%。整个细胞系构建过程中实验废弃物都需要高压处理。
随后收取细胞并提取基因组,因为sgrna表达质粒为慢病毒骨架质粒,所以质粒的5’ltr与3’ltr之间的序列会整合插入到细胞基因组中。选择对这段序列设计引物,上游引物的序列如seqidno.696:atttcccatgattccttca,下游引物的序列如seqidno.697:gactcggtgccactttttc(所有文库都用此引物对)进行pcr扩增,对需要提取基因组进行pcr扩增,所述pcr扩增条件为94℃预变性10min;94℃变性15s,55℃退火30s,72℃延伸1kb/min,25~30个循环;72℃延伸10min进行检测。得到含有sgrna序列的长约300bp的pcr产物,并对pcr产物进行建库测序。将测序结果中的序列与文库sgrna序列进行比对,经过比对后显示,设计的695条sgrna中有693条可以测序比对出结果,说明构建好的所有质粒可以很好的进行慢病毒包装并且能够整合到细胞的基因组中,也即文库中的质粒本身是没有问题的。
文库病毒感染c2c12后,进行基因组pcr,随后建库测序,将测序结果与文库中的sgrna序列进行比对,发现695条sgrna序列可以比对出693条,说明质粒本身都是可以进行慢病毒包装以及可以将序列整合到细胞基因组中。
同时建立c2c12稳转表达spcas9蛋白的细胞系,在后续的验证实验中都使用了c2c12-cas9细胞系进行验证。通过随机挑选以及将评分低于75分的sgrna序列选择出来,见图3所示,包括kdm4d、jmjd4、chaf1b、rbbp8、mina、hdac6、hat1、hdac1、ruvbl1以及chaf1b。将这些基因的sgrna表达质粒分别进行慢病毒包装,随后感染c2c12-cas9-3细胞系,进行嘌呤霉素筛选后提取细胞基因组,进行t7e1酶切检测,来验证设计的sgrna序列是否可以引导spcas9蛋白对靶点位置的基因组进行切割。t7e1酶切的基本原理,如图4所示,就是根据各sgrna靶点位置附近的基因组序列设计引物进行pcr,pcr扩增条件为94℃预变性10min;94℃变性15s,50~65℃退火30s,72℃延伸1kb/min,25~30个循环;72℃延伸10min进行检测。随后对pcr产物进行重新的变性退火,即金属浴95℃10min,随后关闭金属浴自然冷却至室温,随后依次添加17μl重新变性退火的产物、1μlt7e1酶以及2μl10×nebbuffer2组成t7e1酶切体系,在37℃条件下酶切过夜,对酶切的产物进行琼脂糖凝胶电泳,检测观察到三条条带的出现,说明所设计的sgrna都可以很好的引导spcas9对靶点序列进行切割。需要注意的是设计的引物要保证切割位点不在pcr序列的中央位置,保证t7e1酶切割后产生两条长度不一样的片段以便于电泳区分。如果这个靶点位置被spcas9蛋白切割断裂,必然会引起细胞内对断裂的基因组的修复,包括nhej和hr,修复的过程中会产生细胞dna序列的插入或者缺失从而导致dna序列发生改变。所以重新变性退火后必然会有错配的序列产生,使用专门识别dna错配序列的酶t7e1可以在错配的位点进行切割,导致dna双链断裂从而形成两条dna链,通过琼脂糖凝胶电泳可以检测观察到三条条带的出现。实验结果如图5表明,所选择进行验证的基因都可以观察到三条带的出现,说明不论sgrna的评分高低,本发明所设计的sgrna都可以很好的引导spcas9对靶点序列进行切割。
通过随机挑选以及将评分低于75分的sgrna表达质粒挑选出来(图3),按照t7e1酶切的原理(图4)对这些质粒进行验证,结果显示(图5)所验证的质粒都可以有效的引导spcas9蛋白对靶基因进行切割。
通过以上两个方面对小鼠表观基因敲除筛选文库的验证,说明整个文库sgrna表达质粒可以很好的包装成慢病毒并且整合到细胞的基因组上;同时对于sgrna文库的设计,不同评分的sgrna都可以很好的靶向目的基因并引导spcas9对目的序列的切割。
以上所述,仅是本发明的较佳实施例而已,并非是对本发明做其它形式的限制,任何本领域技术人员可以利用上述公开的技术内容加以变更或改型为等同变化的等效实施例。但是凡是未脱离本发明技术方案内容,依据本发明的技术实质对以上实施例所作的任何简单修改、等同变化与改型,仍属于本发明技术方案的保护范围。
- 还没有人留言评论。精彩留言会获得点赞!