一种人类及哺乳动物细胞表达载体、表达系统及其构建方法和应用
本发明属于基因工程技术领域,具体涉及一种人类及哺乳动物细胞表达载体、表达系统及其构建方法和应用。
背景技术:
目前,全球治疗性重组蛋白70%以上都是用人类及哺乳动物细胞表达系统生产的,表达载体是重组蛋白生产的重要的因素之一。当前人类及哺乳动物细胞表达系统中,一个亟需解决的问题就是转基因沉默现象(transgenesilencing)。外源基因在宿主中表达受到抑制的现象称之为转基因沉默。转基因沉默的发生与转基因整合到宿主细胞基因中的位置有关,即表现出“位置效应(positioneffect)”。此外,转基因表达的强度受载体dna序列的影响,例如,在细胞的扩大培养过程中,启动子的甲基化会导致表达的逐渐衰弱。适当地选用启动子,优化启动子和不同调控元件的组合,能提高重组蛋白的表达,阻止启动子的甲基化,增加表达的稳定性。因此,研究用于哺乳动物细胞表达系统高效稳定表达的载体,将极大地促进治疗性重组蛋白的研发和生产,具有重要的理论和实践意义。
当前,随着转基因技术的广泛应用,转基因沉默、基因表达水平低下或不能稳定表达已成为转基因动植物急需解决的关键问题。为了获得稳定表达的转基因株,人们在克服转基因沉默方面进行了许多有益的尝试,比如在构建表达载体时选用强启动子、使用增强子、在转基因操作时进行去甲基化处理等。而利用核基质附着区(matrixattachmentregion,mar)来提高转基因表达是近年来发展起来的克服外源基因沉默的一种有效方法。
mar是与核骨架(或核基质)特异结合的dna序列,属于非编码序列,长度为300bp到几千bp,富含at碱基对,位于基因两侧的非编码区。作为真核生物的一种新的顺式作用元件,目前关于mar能提高外源基因表达水平,降低转基因沉默的报道已有很多。
前期的实验结果证实,mar能提高转基因在稳定转染的中国仓鼠卵巢(chinesehamsterovary,cho)细胞的表达水平(wangty,yangr,qinc,etal.enhancedexpressionoftransgeneinchocellsusingmatrixattachmentregion[j].cellbiolint,2008,32:1279-1283.),并且不同插入位点的mar对转基因表达的影响不同,在mar和启动子之间插入不同长度的egfp,其对转基因表达的影响也不同(zhangjh,wangxy,wangty,etal.distanceeffectofmatrixattachmentregionsontransgeneexpressioninstablytransfectedchinesehamsterovarycells[j].biotechnollett,2014,36(10):1937-1943.)。公布号cn103642838b公开了一种人类及哺乳动物细胞表达载体,该载体上仅插入人β-珠蛋白mar序列(770bp)和egfp(分别为350、750bp),虽然能够介导外源基因在哺乳动物细胞中表达,但是仅用了其中一段mar,并不是mar序列的全长,并且外源片段来自egfp,其序列本身对转基因表达的影响可能会引起某些干扰。目前,mar在提高转基因表达水平方面尚不十分理想,与此同时,关于表达载体的位置、距离效应适配性的研究尚未报道。
技术实现要素:
本发明的目的在于提供一种可高效表达目的基因的人类及哺乳动物细胞表达载体。
本发明的第二个目的在于提供上述表达载体的人类及哺乳动物细胞表达系统及其构建方法。
本发明的第三个目的在于提供上述哺乳动物表达表达载体及表达系统的应用。
为了实现上述目的,本发明采用以下技术方案:
一种人类及哺乳动物细胞表达载体,该载体包含核基质附着区序列和间隔子dna序列;所述核基质附着区序列为mar1-68,其核苷酸序列如seqidno.1所示,位于启动子的5′端和/或polya的3′端;所述间隔子dna序列的核苷酸序列如seqidno.2所示,位于启动子上游,连接核基质附着区序列与启动子。
优选的,所述载体是以piresneo3为骨架的改造载体。
优选的,所述载体的启动子为cmv启动子。
进一步的,所述载体包含一个或多个编码一种或多种重组蛋白的基因。
所述表达载体可按照基因工程领域的常规方法构建。
一种人类及哺乳动物细胞表达系统,包含上述表达载体。
一种人类及哺乳动物细胞表达系统的构建方法,通过将目的基因克隆到上述表达载体,转染宿主细胞构建而成。
优选的,所述宿主细胞为cho-k1,cho-s或cho-dg44细胞。
本发明还提供了上述人类及哺乳动物细胞表达载体、人类及哺乳动物细胞表达系统在提高重组蛋白表达水平方面的应用。
本发明取得的有益效果:
本发明提供的人类及哺乳动物细胞表达载体,通过在启动子的上游和polya的下游插入mar1-68序列,同时,在mar序列与启动子之间插入一段长度为500bp的间隔子dna序列,构建获得mar1-68-spacer-cmv-目的基因-polya-mar1-68载体。该载体表达盒的两侧同时含mar1-68序列,且插入了间隔子dna序列,本发明采用的spacer片段为中性dna序列,不能提高转基因表达水平(leyd,harraghyn,lefournv,etal.marelementsandtransposonsforimprovedtransgeneintegrationandexpression[j].plosone,2013,8(4):e62784.),从而能够真正地探讨距离效应对转基因表达的影响。试验证明,包含上述表达载体的人类及哺乳动物细胞表达系统能够显著提高所携带的目的基因表达水平。因此,本发明提供的哺乳动物表达载体和表达系统能够克服转基因沉默,促进外源基因在宿主细胞中高水平稳定表达,可用于重组蛋白的生产。
附图说明
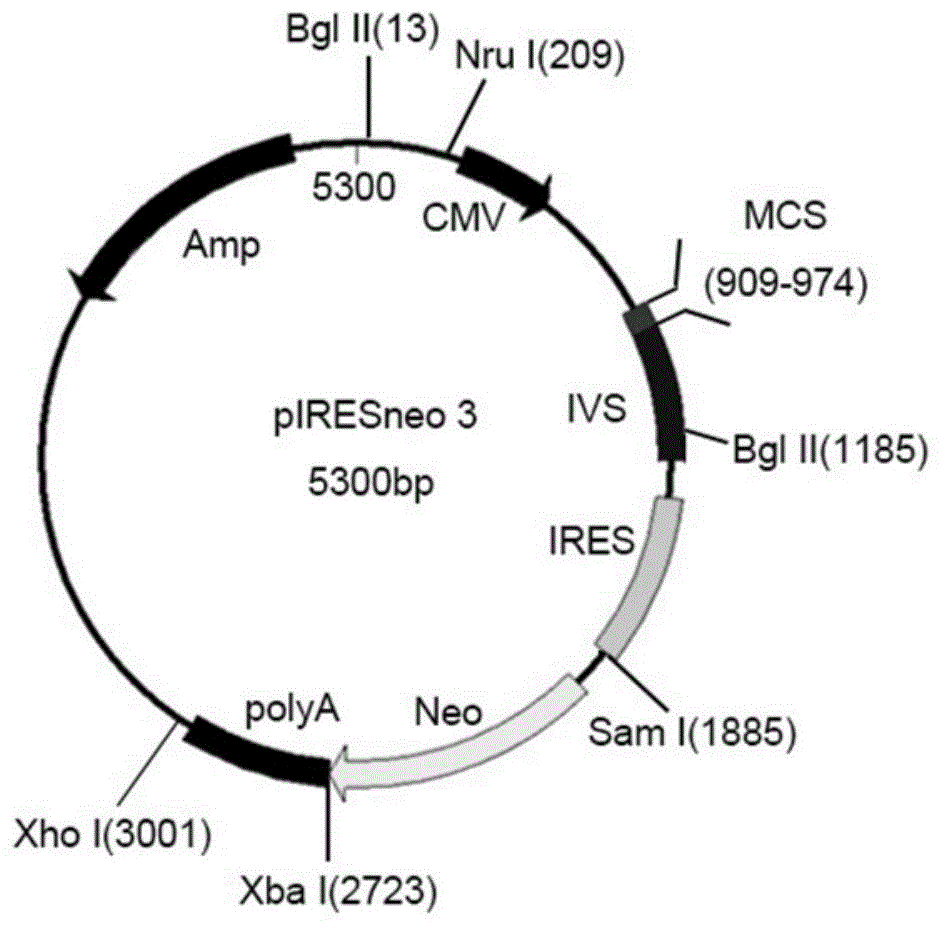
图1是piresneo3载体的物理图谱;
图2是表达载体pires-cmv-egfp的物理图谱;
图3是表达载体pires-mar-cmv-egfp-polya的物理图谱;
图4是表达载体pires-cmv-egfp-polya-mar的物理图谱;
图5是表达载体pires-mar-cmv-egfp-polya-mar的物理图谱;
图6是表达载体pires-mar-spacer-cmv-egfp-polya的物理图谱;
图7是表达载体pires-spacer-cmv-egfp-polya-mar的物理图谱;
图8是表达载体pires-mar-spacer-cmv-egfp-polya-mar的物理图谱;
图9是表达载体pires-cmv-epo的物理图谱;
图10是表达载体pires-mar-cmv-epo-polya的物理图谱;
图11是表达载体pires-cmv-epo-polya-mar的物理图谱;
图12是表达载体pires-mar-cmv-epo-polya-mar的物理图谱;
图13是表达载体pires-mar-spacer-cmv-epo-polya的物理图谱;
图14是表达载体pires-spacer-cmv-epo-polya-mar的物理图谱;
图15是表达载体pires-mar-spacer-cmv-epo-polya-mar的物理图谱;
图16是实施例3中不同表达载体稳定转染cho细胞后egfp的表达水平对比图;
图中,a为不同表达载体转染cho细胞流式测得的egfp表达值,b为含mar表达载体提高egfp表达水平的倍数;
图17是实施例3中不同表达载体稳定转染cho细胞后epo蛋白表达水平的elisa检测结果。
具体实施方式
下面结合具体实施方式对本发明作进一步描述,但本发明的保护范围并不仅限于此;本发明实施例中所使用的大肠杆菌(escherichiacoli)jm109、所使用的细胞系、质粒载体和试剂、工具酶均为市售商品。piresneo3质粒载体购自于clontech生物公司。
实施例1表达盒不同位置含mar的表达载体构建
1.1egfp的pcr扩增
参照enhancedgreenfluorescentprotein(egfp)基因片段序列(genbank登录号为no:u55763.1,第613-1329位碱基)设计如下引物:p1:5′-ccggatatcatggtgagcaagggc-3′;p2:5′-cgcgctagctcacttgtacagctc-3′,引物的5′端分别引入ecorv和nhei酶切位点。以提取的pegfp-c1质粒为模板,采用常规pcr方法进行扩增。pcr体系及如下:
pcr程序如下:95℃3min,94℃40s,60℃30s,72℃40s,30个循环,72℃3min。琼脂糖凝胶电泳回收扩增产物,送生物公司测序验证,结果表明,扩增出的dna片段与genbank公开的egfp序列完全一致。
1.2pires-cmv-egfp表达载体的构建
用ecorv/nheⅰ双酶切egfp的pcr扩增产物(经测序验证正确的序列),同时用ecorv/nheⅰ双酶切piresneo3质粒(图1),采用常规的酶切方法,37℃3h。琼脂糖凝胶电泳鉴定酶切结果,凝胶回收egfp片段和piresneo3线形质粒;酶切回收后的egfp片段和piresneo3线形质粒按1:5(摩尔比)进行连接,16℃连接过夜;然后将连接产物加入到e.colijm109菌株感受态细胞悬液中,进行转化,将200μl菌液接种在含有氨苄青霉素lb平板上,37℃培养过夜,挑取单菌落继代培养,并进行重组质粒的双酶切(ecorv/nheⅰ)验证,选取酶切验证正确的质粒,进行测序验证,将目的基因序列完全正确的载体命名为pires-cmv-egfp(质粒图谱如图2所示)。
1.3pires-mar-cmv-egfp-polya表达载体的构建
1.3.1pcr扩增目的片段
根据mar1-68(genbank登录号为no:ef694965.1)的序列,设计如下引物:p3:5′-gtcacgcgttcgactctagattat-3′;p4:5′-agcttcgaactctagatgtagtac-3′,引物的5′端分别引入mlui/bstbi酶切位点。以提取的人外周血基因组dna为模板,pcr扩增得到人mar1-68。pcr体系同前,pcr条件如下:95℃3min,94℃40s,60-56℃30s,72℃40s,每个退火温度4个循环,最后55℃,30个循环,72℃3min。琼脂糖凝胶电泳回收扩增产物,送生物公司测序验证,结果表明,扩增出的dna片段与genbank公开的mar1-68序列完全一致。
1.3.2pires-mar-cmv-egfp-polya载体的构建
用mlui/bstbi双酶切mar1-68序列的pcr扩增产物(经测序验证正确的序列),同时用mlui/bstbi双酶切pires-egfp质粒,采用常规的酶切方法,37℃3h。琼脂糖凝胶电泳鉴定酶切结果,凝胶回收mar序列片段和pires-egfp线形质粒;酶切回收后的mar片段及pires-egfp线性质粒dna按1:5(摩尔比)进行连接,16℃连接过夜;然后将连接产物加入到e.colijm109菌株感受态细胞悬液中,进行转化,将200μl菌液接种在含有氨苄青霉素lb平板上,37℃培养过夜,挑取单菌落继代培养,并进行重组质粒的双酶切(mlui/bstbi)验证,选取酶切验证正确的质粒,进行测序验证,将目的基因序列完全正确的载体命名为pires-mar-cmv-egfp-polya(质粒图谱如图3所示)。
1.4pires-cmv-egfp-polya-mar表达载体的构建
1.4.1pcr扩增目的片段
根据mar1-68(genbank登录号为no:ef694970.1)的序列,设计如下引物:p5:5′-gcggccgctcgactctagattat-3′;p6:5′-ggcgcgccctctagatgtagtac-3′,引物的5′端分别引入noti/asci酶切位点。以提取的人外周血基因组dna为模板,pcr扩增得到人mar1-68。pcr体系同前,pcr条件如下:95℃3min,94℃40s,60-56℃30s,72℃40s,每个退火温度4个循环,最后55℃,30个循环,72℃3min。琼脂糖凝胶电泳回收扩增产物,送生物公司测序验证,结果表明,扩增出的dna片段与genbank公开的mar1-68序列完全一致。
1.4.2pires-cmv-egfp-polya-mar表达载体的构建
用noti/asci双酶切mar1-68序列的pcr扩增产物(经测序验证正确的序列),同时用noti/asci双酶切pires-egfp质粒,采用常规的酶切方法,37℃3h。琼脂糖凝胶电泳鉴定酶切结果,凝胶回收mar序列片段和pires-egfp线形质粒;酶切回收后的mar片段及pires-egfp线性质粒dna按1:5(摩尔比)进行连接,16℃连接过夜;然后将连接产物加入到e.colijm109菌株感受态细胞悬液中,进行转化,将200μl菌液接种在含有氨苄青霉素lb平板上,37℃培养过夜,挑取单菌落继代培养,并进行重组质粒的双酶切(noti/asci)验证,选取酶切验证正确的质粒,进行测序验证,将目的基因序列完全正确的载体命名为pires-cmv-egfp-polya-mar(质粒图谱如图4所示)。
1.5pires-mar-cmv-egfp-polya-mar表达载体的构建
用noti/asci双酶切mar1-68序列的pcr扩增产物(经测序验证正确的序列),同时用noti/asci双酶切pires-mar-cmv-egfp-polya质粒,采用常规的酶切方法,37℃3h。琼脂糖凝胶电泳鉴定酶切结果,凝胶回收mar序列片段和pires-mar-cmv-egfp-polya线形质粒;酶切回收后的mar片段及pires-mar-cmv-egfp-polya线性质粒dna按1:5(摩尔比)进行连接,16℃连接过夜;然后将连接产物加入到e.colijm109菌株感受态细胞悬液中,进行转化,将200μl菌液接种在含有氨苄青霉素lb平板上,37℃培养过夜,挑取单菌落继代培养,并进行重组质粒的双酶切(noti/asci)验证,选取酶切验证正确的质粒,进行测序验证,将目的基因序列完全正确的载体命名为pires-mar-cmv-egfp-polya-mar(质粒图谱如图5所示)。
实施例2含mar和间隔子dna序列的表达载体构建
2.1合成spacerdna片段
参照spacerdna片段序列(ncbirefseq:nm_011682.4),首先合成一段长度为500bp的spacerdna,基因合成由北京中美泰克生物公司完成。
2.2构建含距离片段的表达载体
采用无缝克隆的方法,将500bp的spacerdna片段,分别构建到pires-mar-cmv-egfp-polya,pires-cmv-egfp-polya-mar和pires-mar-cmv-egfp-polya-mar载体的cmv表达盒上游,选取酶切验证正确的质粒,进行测序验证,分别构建含不同位置的mar1-68与距离片段的载体,载体分别命名为pires-mar-spacer-cmv-egfp-polya,pires-spacer-cmv-egfp-polya-mar和pires-mar-spacer-cmv-egfp-polya-mar(质粒图谱如图6~图8所示)。
实施例3含epo,mar和间隔子dna序列的载体构建
(1)含有促红细胞生成素(epo)的pires-cmv-epo载体构建
根据人的epo基因cdna序列(ncbirefseq:nm_000799.4)设计pcr引物p7和p8,为了实现定向克隆,引物的5′端分别引入ecorv/nheⅰ酶切位点,引物序列如下:p7:5′-ccggatatcatgggggtgcacgaa-3′;p8:5′-cgcgctagctcatctgtcccctgt-3′。以提取的人外周血基因组dna为模板,pcr扩增得到人epo。pcr体系同前,pcr条件如下:95℃3min,94℃40s,60-56℃30s,72℃40s,每个退火温度4个循环,最后55℃,30个循环,72℃3min。琼脂糖凝胶电泳回收扩增产物,送生物公司测序验证,结果表明,扩增出的dna片段与genbank公开的epo序列完全一致。
用ecorv/nheⅰ双酶切epo基因的pcr扩增产物(经测序验证正确的序列),同时用ecorv/nheⅰ双酶切pires-cmv-egfp载体,采用常规的酶切方法,37℃3h。琼脂糖凝胶电泳鉴定酶切结果,凝胶回收epo基因序列片段和pires-cmv-egfp线形质粒;酶切回收后的epo基因cdna序列片段及pires-cmv-egfp线性质粒dna按1:5(摩尔比)进行连接,16℃连接过夜;然后将连接产物加入到e.colijm109菌株感受态细胞悬液中,进行转化,接种在含有氨苄青霉素lb平板上,37℃培养过夜,挑取单菌落继代培养,用ecorv/nheⅰ双酶切重组质粒载体,选取酶切验证正确的质粒进行测序验证,将目的基因序列完全正确的载体命名为pires-cmv-epo(质粒图谱如图9所示)。
(2)含epo和mar的表达载体构建
采用分子克隆技术,分别将pires-mar-cmv-egfp-polya,pires-cmv-egfp-polya-mar和pires-mar-cmv-egfp-polya-mar表达载体中的egfp替换为epo,进而构建分别含epo和mar的载体,载体分别命名为pires-mar-cmv-epo-polya,pires-cmv-epo-polya-mar和pires-mar-cmv-epo-polya-mar(质粒图谱如图10~图12所示)。
(3)含epo,mar和间隔子dna序列的表达载体构建
采用分子克隆技术,分别将pires-mar-spacer-cmv-egfp-polya,pires-spacer-cmv-egfp-polya-mar和pires-mar-spacer-cmv-egfp-polya-mar表达载体中的egfp替换为epo,进而构建分别含epo,mar和间隔子dna序列的载体,载体分别命名为pires-mar-spacer-cmv-epo-polya,pires-spacer-cmv-epo-polya-mar和pires-mar-spacer-cmv-epo-polya-mar(质粒图谱如图13~图15所示)。
试验例1
利用本发明的表达载体在中国仓鼠卵巢(chinesehamsterovary,cho)细胞中表达egfp:
(1)不同表达载体转染cho细胞表达系统的建立
选择生长状态良好的cho细胞接种到6孔培养板上,待铺板密度达到约80%时进行转染。具体操作步骤如下:将10μllipofectamine2000添加到240μl无血清dmem/f12培养基中,在37℃孵箱中静置5min,将无血清的dmem/f12培养基与250μl(5μg)表达载体混合,37℃孵箱中静置20min;同时用pbs将6孔培养板上的细胞清洗三遍,加入2ml无血清的dmem/f12细胞培养基。根据实验设计转染共分为7组:pires-cmv-egfp(不含mar的对照载体),pires-mar-cmv-egfp-polya,pires-cmv-egfp-polya-mar,pires-mar-cmv-egfp-polya-mar,pires-mar-spacer-cmv-egfp-polya,pires-spacer-cmv-egfp-polya-mar,pires-mar-spacer-cmv-egfp-polya-mar。然后,将脂质体与各组质粒dna的混合液轻轻滴入孔中,尽快轻轻摇晃培养平板使其混匀;放入5%co2的细胞培养箱中,37℃培养6h后,将无血清的dmem/f12培养基更换成dmem/f12完全培养基,放入细胞培养箱内继续培养。
(2)不同表达载体稳定转染cho细胞egfp基因表达情况
转染48h后,在转染孔中加入600μg/ml的g418药物,每48h换成新鲜dmem/f12完全培养基,从第五天开始细胞大量死亡。筛选两周后将g418浓度调整到维持浓度300μg/ml继续培养。药物筛选完成后,在细胞形成稳定细胞池时(约14天),收集细胞进行流式细胞检测,分析mar和距离片段对报告基因egfp表达的影响。
结果如图16所示,图中,a为不同表达载体转染cho细胞流式测得的egfp表达值,b为含mar表达载体提高egfp表达水平的倍数。结果可见,表达盒两侧同时含mar和间隔子dna序列的载体peam(f/f)+500bpspacer(pires-mar-spacer-cmv-egfp-polya-mar)的egfp表达量最高,为不含mar的对照载体的2.95倍;polya下游含mar和间隔子dna序列的载体peam3(f)+500bpspacer(pires-spacer-cmv-egfp-polya-mar)的egfp表达量是不含mar的对照载体的1.65倍,启动子上游含mar和间隔子dna序列的载体peam5(f)+500bpspacer(pires-mar-spacer-cmv-egfp-polya)的egfp表达量是不含mar的对照载体的1.7倍;其它双侧含mar的载体peam(f/f)(pires-mar-cmv-egfp-polya-mar)或单侧含mar的载体peam5(f)(pires-mar-cmv-egfp-polya)和peam3(f)(pires-cmv-egfp-polya-mar)均能提高egfp的表达水平,提高倍数没有对应的同时含mar和间隔子dna序列的载体明显(p<0.05,差异显著),表明启动子上游插入的间隔子dna序列对上游启动子的5′端和下游polya的3′端的mar1-68序列对提高转基因表达具有明显的促进作用,特别是当表达盒两侧同时含mar1-68序列且插入了间隔子dna序列时(peam(f/f)+500bpspacer),间隔子dna序列产生的距离效应与两侧的mar1-68序列协同作用,能够显著提高目的基因表达。
试验例2
利用本发明的表达载体在cho细胞中表达epo基因:
(1)不同表达载体转染cho细胞表达系统的建立
选择生长状态良好的cho细胞接种到6孔培养板上,待铺板密度达到约80%时进行转染。具体操作步骤如下:将10μllipofectamine2000添加到240μl无血清opti-mem培养基中,在37℃孵箱中静置5min,将无血清的opti-mem培养基与250μl(5μg)表达载体混合,37℃孵箱中静置20min;同时用pbs将6孔培养板上的细胞清洗三遍,加入2ml无血清的opti-mem细胞培养基。根据实验设计转染共分为7组:pires-cmv-epo(不含mar的对照载体),pires-mar-cmv-epo-polya,pires-cmv-epo-polya-mar,pires-mar-cmv-epo-polya-mar,pires-mar-spacer-cmv-epo-polya,pires-spacer-cmv-epo-polya-mar,pires-mar-spacer-cmv-epo-polya-mar。然后,将脂质体与各组质粒dna的混合液轻轻滴入孔中,尽快轻轻摇晃培养平板使其混匀;放入5%co2的细胞培养箱中,37℃培养6h后,将无血清的opti-mem培养基更换成opti-mem完全培养基,放入细胞培养箱内继续培养。48h后在转染孔中加入600μg/ml的g418药物,每48h换成新鲜opti-mem完全培养基,从第五天开始细胞大量死亡。筛选两周后将g418浓度调整到维持浓度300μg/ml继续培养。药物筛选完成后,在细胞形成稳定细胞池时(约14天),换成无血清的培养基(cdoptichotmmedium,购自gibco公司)。
(2)不同表达载体稳定转染cho细胞epo基因表达情况
继续悬浮培养6天后取细胞上清做elisa检测。
elisa分析结果如图17所示,所有含spacer的表达载体均能不同程度提高epo的表达水平,其中表达盒两侧同时含mar和间隔子dna序列的载体peam(f/f)+500bpspacer(pires-mar-spacer-cmv-epo-polya-mar)的epo表达量最高,为不含mar的对照载体的3倍;启动子上游含mar和间隔子dna序列的载体peam5(f)+500bpspacer(pires-mar-spacer-cmv-epo-polya)的epo表达量是不含mar的对照载体的2倍,polya下游含mar和间隔子dna序列的载体peam3(f)+500bpspacer(pires-spacer-cmv-epo-polya-mar)的epo表达量是不含mar的对照载体的1.5倍,不同表达载体转染的细胞epo含量差异有统计学意义;而其他仅含mar的载体均能提高epo的表达水平,但没有同时含mar和距离片段的表达载体明显。
<110>新乡医学院
<120>一种人类及哺乳动物细胞表达载体、表达系统及其构建方法和应用
<160>10
<170>patentinversion3.5
<210>1
<211>3614
<212>dna
<213>homosapiens
<221>mar1-68
<400>1
tcgactctagattataccaacctcataaaataagagcatatataaaagcaaatgctctta60
tcttgcagatccctgaactgaggaggcaagatcagtttggcagttgaagcagctggaatc120
tgcaattcagagaatctaagaaaagacaaccctgaagagagagacccagaaacctagcag180
gagtttctccaaacattcaaggctgagggataaatgttacatgcacagggtgagcctcca240
gaggcttgtccattagcaactgctacagtttcattatctcagggatcacagattgtgcta300
cctattgcctaccatctgaaaacagttgcttcctatatttcatccagtttaatatttatt360
taaaccaagaaggttaatctggcaccagctattccgttgtgagtggatgtgaaagtacca420
attccattctgttttactattaactatcctttgccttaatatgtatcagtaggtggcttg480
ttgctaggaaatattaaatgaatggcatgtttcataggttgtgtttaaagttgttttttg540
agttaaatctttctttaataatactttctgatgtcaaaaacacttagaagtcatggtgtt600
gaacatctatatagggttggatctaaaatagcttcttaacctttcctaaccactgttttt660
gtttgtttgtttttaactaagcatccagtttgggaaattctgaattaggggaatcataaa720
aggtttcattttagctgggccacataaggaaagtaagatatcaaattgtaaaaatcgtta780
agaacttctatcccatctgaagtgtgggttaggtgcctcttctctgtgctcccttaacat840
cctattttatctgtatatatatatattcttccaaatatccatgggaaaaaaaatctgatc900
ataaaaatattttaggctgggagtggtggctcacgcctgtaatcccagcactttgggagg960
ctgaggtgggcggatcatgaggtcaagagatcgagaccatcctgaccaatatggtgaaac1020
cccatctctactaaagatacaaaactattagctggacgtggtggcacgtgcctgtagtcc1080
cagctactcgggaggctgaggcaggagaacggcttgaacccaggaggtggaggttgcagt1140
gagctgagatcgcgccactgcactccagcctgggcgacagagcgagactctgtctcaaaa1200
aaaaaatatatatatatatatatatacacatatatatataaaatatatatatatacacac1260
atatatatataaaatatatatatatacacacatatatataaaatatatatatatacacac1320
atatatataaaatatatatatacacacatatatataaaatatatatatacacacatatat1380
ataaaatatatatatacacacatatatataaaatatatatatacacacatatatataaaa1440
tatatatatacacacatatatataaaatatatatatacacacatatatataaaatatata1500
tatacacacatatatataaaatatatatatacacacatatatataaaatatatatataca1560
cacatatatataaaatatatatatacacacatatataaaatatatatatacacacatata1620
taaaatatatatatacacatatatataaaatatatatatacacatatatataaaatatat1680
atacacacatatatataaaatatatatatacacacatatatataaaatatatatatacac1740
atatatataaaatatatatatacacatatatataaaatatatatatatacacatatatat1800
aaaatatatatacacacatatatataaagtatatatatacacacatatatataaaatata1860
tatatacacatatatataaaatatatatatacacatatatataaaatatatatatacaca1920
tatatataaaaatatatatatatattttttaaaatattccaattgtctcactttgtggat1980
gagaaaaagaagtagttagaggtcaagtaacttggcctacatcttttctcaagattgtaa2040
actcctagtgagcaataaccacatcttcattttctttgtataaaacaagaaagtttagca2100
tgaaaaaggtactcaattacaaatgtgttggattgaattgaagacccttggaaggggatt2160
ttgtacctgaggatctctttcttttggccatattgttcaatggacaaaatttagccttcg2220
aaggcaggccgatttgaggttaatactacctttaccacttgatagctatgtgaccttggc2280
catgtggtttcaacagtctgaacctcattttctctgtgtatgtgtggtcctccttacaag2340
tttgtgaaaaatgtgaagtccttagccatgatagcccaatataacaggctaaatgataat2400
aggtttatgttcttttcctttatattctcagataagcactgtccaagtttgaggtgtttt2460
gaggtctcgcctgatttggattgtttgagtttatgctattctttgaattctttgagctgt2520
tctgaagcagtgtatcatgaacaaaaacatccccagttcagtccaaacccctggttacat2580
atcattcttatgccatgttataaccagtttgagagtgttccctctgttattgcatttaag2640
tttcagcctcacacagaaattcagcagccaatttctaagccctaagcataaaatctgggg2700
tgggggggggggatggcctgaagagcagcattatgaatagcaccattataattaatgatc2760
tctcaggaagatttacaatcacaggtagcagataaaacaaatagtactgcttctgcactt2820
cccctccttttattcgctatgaaattttatgggaaatcagtccagtgaaaaatgtaagct2880
cttaatctttcccagaaatcctacctcatttgatgaatactttgagggaatgaattagag2940
catttttttcttttatagtctacttcgcatttacgaagtgaggacggtagcttaggctgc3000
ctggccaactgatgagaaggtcagaggcatttttagagacctctgttgtctttcattcat3060
gttcattttccacaaggcaagtaatttccaacaaatcagtgtcttcattagtaataagat3120
tattaacaacaataatagtcatagtaactattcagtgagagtccattatatatcaggcat3180
tctacaaggtactttatatacatctgagtaaacctcacacaattctacagggaggtattt3240
ctatccccatttaacaaataaggaaacgaagtccaagtaaattaacttgcccaaggtcac3300
acagatagtacctggcagaacaggaatttaaacctaaatttgtccaactccaaaagcagc3360
cttctatttgttataaatgctgcctctcattatcacatattttattattaacaacaacaa3420
acataccaattagcttaagatacaatacaaccagataatcatgatgacaacagtaattgt3480
tatactattataataaaatagatgttttgtatgttactataatcttgaatttgaatagaa3540
atttgcatttctgaaagcatgttcctgtcatctaatatgattctgtatctattaaaatag3600
tactacatctagag3614
<210>2
<211>500
<212>dna
<213>人工序列
<221>间隔子dna序列
<400>2
ccaagtatggggaccttgaagccaggcctgatgatgggcagaacgaattcagtgacatca60
ttaagtccagatctgatgaacacaatgatgtacagaagaaaacctttaccaaatggataa120
acgctcgattttccaagagtgggaaaccacccatcagtgatatgttctcagacctcaaag180
atgggagaaagctcttggatcttctcgaaggcctcacaggaacatcattgccaaaggaac240
gtggttccacaagggtgcatgccttaaacaatgtcaaccgagtgctacaggttttacatc300
agaacaatgtggacttggtgaatattggaggcacggacattgtggatggaaatcccaagc360
tgactttagggttactctggagcatcattctgcactggcaggtgaaggatgtcatgaaag420
atatcatgtcagacctgcagcagacaaacagcgagaagatcctgctgagctgggtgcggc480
agaccaccaggccctacagt500
<210>3
<211>24
<212>dna
<213>人工序列
<221>p1
<400>3
ccggatatcatggtgagcaagggc24
<210>4
<211>24
<212>dna
<213>人工序列
<221>p2
<400>4
cgcgctagctcacttgtacagctc24
<210>5
<211>24
<212>dna
<213>人工序列
<221>p3
<400>5
gtcacgcgttcgactctagattat24
<210>6
<211>24
<212>dna
<213>人工序列
<221>p4
<400>6
agcttcgaactctagatgtagtac24
<210>7
<211>23
<212>dna
<213>人工序列
<221>p5
<400>7
gcggccgctcgactctagattat23
<210>8
<211>23
<212>dna
<213>人工序列
<221>p6
<400>8
ggcgcgccctctagatgtagtac23
<210>9
<211>24
<212>dna
<213>人工序列
<221>p7
<400>9
ccggatatcatgggggtgcacgaa24
<210>10
<211>24
<212>dna
<213>人工序列
<221>p8
<400>10
cgcgctagctcatctgtcccctgt24
- 还没有人留言评论。精彩留言会获得点赞!