GII.17型诺如病毒全人源中和性单链抗体的制备及鉴定方法
本发明涉及生物医学领域,特别是涉及一种gii.17型诺如病毒全人源中和性单链抗体的制备及鉴定方法。
背景技术:
诺如病毒(norovirus,nov)是引起全球各年龄段人群急性胃肠炎(acutegastroenteritis,age)暴发流行及散发的主要病原体,由nov引起的age占全球age发病总数的近五分之一。由于nov的低感染剂量及传播途径的多样化,导致nov能够在医院、学校、餐厅等半封闭的环境中迅速传播。在全球范围内,nov感染每年可致约6.99亿age病例和22万人死亡。在中国,nov一年可引起80万人感染age,年均发病率为6.0/100人年,特别是小于5岁的儿童,年均发病率约为15.6/100人年。由此可见,nov的暴发流行及散发是我国及世界范围内的一个重要公共卫生问题。
nov属杯状病毒科,是单股正链小rna病毒,直径为27~38nm,基因组全长为7.4~7.7kb。基于vp1氨基酸序列的多样性,可将nov分为gi~gx共10个基因组,49个基因型。10个基因组中gi、gii和giv基因组可感染人类。gii.4nov是自1996年以来暴发流行及散发最主要的基因型,占所有nov相关胃肠炎暴发的70%-80%。然而2014年以来,中国、日本等亚洲国家均报道了由gii.p7-gii.17新变异株引起的nov相关胃肠炎暴发。在2014-2015年间,这一变异株在nov暴发中占76.9%,并取代gii.4/2012sydney株,成为我国2014~2015和2015~2016nov流行季的主要流行株。
病毒受体是公认的引发病毒感染宿主细胞的起始步骤和关键因素之一,病原体往往为了适应不同的受体表型选择性进化。研究发现,人类组织血型抗原(histo-bloodinggroupantigen,hbga)是nov的病毒受体或病毒附着因子,是研究nov易感性和致病性的重要因素。
目前,nov缺乏成熟的细胞培养系统及小动物感染模型,其疫苗和特异性抗病毒药物研究进展仍较缓慢。在过去30年中gii.4nov一直是全球范围内的优势流行株,因此,现阶段nov疫苗和特异性抗体药物的开发及研制主要集中在gii.4,然而gii.4抗体免疫对与gii.17感染不存在交叉保护作用。gii.17感染造成急性胃肠炎的问题仍然没有被解决,继续危害人类健康。
因此,针对现有技术不足,提供一种gii.17型诺如病毒全人源中和性单链抗体制备及鉴定方法料以克服现有技术不足甚为必要。
技术实现要素:
本发明的目的在于避免现有技术的不足之处而提供一种gii.17型诺如病毒全人源中和性单链抗体的制备方法,制备的gii.17nov全人源单链抗体的有效库容大,且重组率和多样性好,为nov的临床防治及相关疫苗的研制奠定了基础。
本发明的目的通过以下技术措施实现:
提供一种gii.17型诺如病毒全人源中和性单链抗体的制备方法,包括下列步骤:
s1:构建初级全人源噬菌体单链抗体库。
具体是,采集gii.17nov目标对象恢复期外周血,利用密度梯度离心法分离出pbmc,利用trozil法提取pbmc的总rna,逆转录合成cdna。
设计合成人抗体基因的引物,并以cdna为模板,扩增抗体重链可变区基因vh和轻链可变区基因vl,重叠延伸pcr拼接vh和vl形成scfv基因。
线性化噬菌粒载体pcantab-5e,构建重组质粒pcantab-5e-scfv,将重组质粒pcantab-5e-scfv电转化至e.colitg1感受态细胞后加入辅助噬菌体m13k07培养,得到全人源噬菌体单链抗体库,并挑取抗体库中单克隆做菌液pcr且对阳性克隆进行测序分析。
s2:对初级全人源噬菌体单链抗体库进行淘筛及特异性鉴定。
具体是,以gii.17novp颗粒为固相抗原,对全人源噬菌体单链抗体库进行富集性筛选获得与gii.17nov结合的噬菌体并扩增,得到特异性gii.17nov噬菌体单链抗体库。
将特异性gii.17nov噬菌体单链抗体库铺到氨苄抗性琼脂板上挑取单克隆,补加辅助噬菌体m13k07拯救,离心制备噬菌体上清,用phage-elisa鉴定噬菌体上清的特异性,并选取阳性克隆进行测序分析。
s3:制备gii.17nov全人源单链抗体。
具体是,设计原核表达引物,以步骤s2所得阳性噬菌体上清的菌液为模板,扩增scfv片段,使用限制性内切酶bamhi和noti双酶切载体pgex-4t-1,并将scfv片段重组克隆至载体pgex-4t-1上,将产物转化至e.colit0p10感受态细胞中,挑取单克隆做菌液pcr,鉴定重组质粒是否插入目的片段,并对阳性克隆进行测序分析后提取获得pgex-scfv重组质粒。
利用大肠杆菌表达系统表达pgex-scfv重组质粒,然后用gst-resin亲和层析柱纯化,加入凝血酶去除融合蛋白gst-scfv的蛋白标签gst后获得scfv,即gii.17nov全人源中和性单链抗体。
优选的,步骤s1中gii.17型nov目标对象恢复期外周血来自由gii.17nov引起的急性胃肠炎暴发目标对象。
优选的,步骤s1中人抗体基因的引物包括6对vh家族基因、用于连接抗体重轻链的氨基酸序列为ggggsggggsggggs的两条互补连接肽、12对vl家族基因以及sfii和noti酶切位点的scfv上下游引物。
优选的,步骤s1中构建重组质粒pcantab5e-scfv的具体过程为,分别使用限制性内切酶sfii和noti对scfv基因和噬菌粒载体pcantab5e双酶切,然后用t4连接酶连接构建形成重组质粒pcantab5e-scfv。
辅助噬菌体m13k07与e.colitg1感受态细胞的moi=100:1,辅助噬菌体m13k07的滴度为10~12pfu/ml。
优选的,步骤s2中富集性筛选具体为将gii.17novp颗粒包被免疫管,吸附噬菌体单链抗体库,然后采用甘氨酸-盐酸洗脱法进行富集性淘选,重复进行4轮吸附-洗涤-洗脱-感染-拯救的过程,获得与gii.17novp颗粒结合的噬菌体,然后将与gii.17novp颗粒结合的噬菌体洗脱下来用于扩增。
优选的,步骤s2中用phage-elisa鉴定噬菌体上清的特异性的具体过程为将gii.17novp颗粒、作为对照抗原的轮状病毒vp8*和作为空白对照pbs包被于96孔酶标板,以噬菌体上清为一抗,同时以无噬菌体上清的培养基为阴性对照,hrp-鼠抗m13作为二抗,用phage-elisa鉴定特异性,若包被p颗粒的od450值大于包被vp8*对应孔2倍以上,则判定为阳性噬菌体上清。
优选的,步骤s3中所述原核表达引物根据步骤s2中阳性克隆测序分析结果设计,并在上下游引物中分别引入载体pgex-4t-1的两末端同源序列、bamhi酶切位点和noti酶切位点。
优选的,步骤s4中利用大肠杆菌表达系统表达pgex-scfv重组质粒的具体过程为将插入目的片段的重组质粒转化至e.colibl21感受态细胞中,挑取单克隆放置于37℃下以220rpm的转速振荡培养12h~16h,然后按体积比为1:100的比例转接至新鲜lb液体培养基中,在37℃下以220rpm振荡培养至细菌对数期,加入iptg于22℃以200rpm转速振荡12h诱导表达。
将诱导表达获得的菌液于4℃以5000rpm的转速离心12min,弃上清,收集菌体,然后用pbs重悬菌体,并将菌液于冰浴中进行超声破碎,然后将菌液放置于4℃下以12000rpm的转度离心75min。
本发明同时还提供一种gii.17型诺如病毒全人源中和性单链抗体的鉴定方法,用于鉴定gii.17型诺如病毒全人源中和性单链抗体的制备方法制备的scfv。
优选的,所述鉴定步骤包括:
特异性鉴定,具体为将scfv包被于96孔酶标板,同时以gst蛋白为阴性对照,pbs为空白对照,封闭后加入gii.17novp颗粒,以鼠抗gii.17血清为一抗,hrp-羊抗鼠igg为二抗,elisa鉴定scfv的特异性,若实验孔的od450大于阴性组的2倍以上,则判定scfv具有特异性。
中和性鉴定,具体为包被分泌型b型唾液于96孔酶标板,封闭后加入gii.17novp颗粒与scfv的混合液,同时阳性对照用未加scfv的p颗粒,阴性对照用确定没有阻断能力的血浆和p颗粒的混合液,空白对照用1%脱脂奶粉-pbs代替scfv和p颗粒的混合液,并以小鼠抗gii.17血清为一抗,hrp-羊抗鼠igg为二抗,体外hbga受体阻断实验鉴定scfv的中和性,测定各孔的od450值,若阻断率>50%,则判定scfv具有阻断能力。
本发明的一种gii.17型诺如病毒全人源中和性单链抗体的制备方法,包括以下步骤:s1:构建初级全人源噬菌体单链抗体库,利用gii.17nov目标对象恢复期外周血得到pbmc,逆转得到cdna,用人抗体基因的引物从中扩增出抗体重轻链,再将两者拼接形成scfv基因,构建出重组质粒pcantab-5e-scfv电转化后培养获得全人源噬菌体单链抗体库。s2:对全人源噬菌体单链抗体库进行淘筛及特异性鉴定,以gii.17novp颗粒为固相抗原,用甘氨酸-盐酸洗脱法对抗体库进行富集淘筛,制备得到特异性gii.17nov噬菌体单链抗体库,离心制备噬菌体上清,用phage-elisa鉴定噬菌体上清的特异性。s3:制备gii.17nov全人源单链抗体,设计原核表达引物,以步骤s2所得阳性噬菌体上清的菌液为模板,扩增scfv片段,构建pgex-scfv重组质粒,利用大肠杆菌表达系统表达pgex-scfv重组质粒,然后用gst-resin亲和层析柱纯化,加入凝血酶去除融合蛋白gst-scfv的蛋白标签gst后获得scfv。该方法制备出的抗体的有效库容大且重组率和多样性好,对nov抗原表位的研究及相关抗体药物研发具有重要意义。
附图说明
利用附图对本发明作进一步的说明,但附图中的内容不构成对本发明的任何限制。
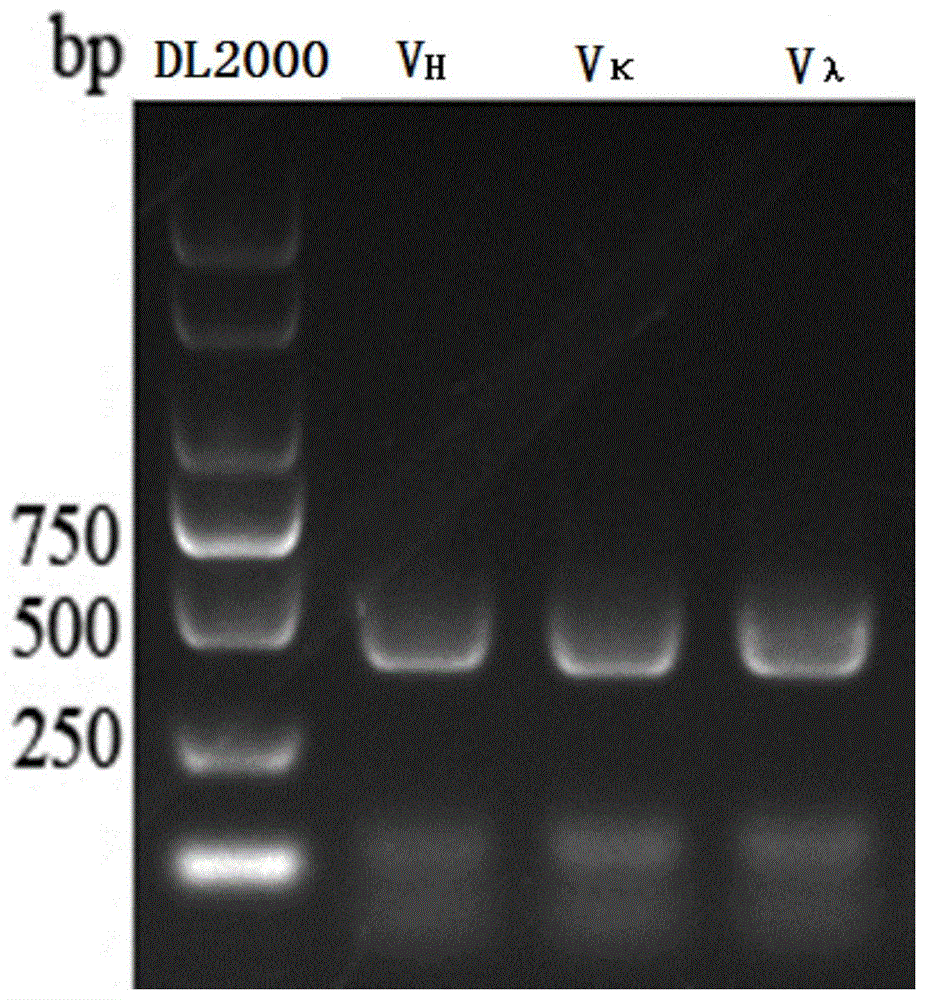
图1是本发明实施例2中vh和vl基因pcr产物琼脂糖凝胶电泳图。
图2是本发明实施例2中scfv片段pcr产物琼脂糖凝胶电泳图。
图3是本发明实施例2中噬菌体抗体库菌落pcr产物琼脂糖凝胶电泳图。
图4是本发明实施例2中噬菌体阳性克隆的phage-elisa鉴定柱状图。
图5是本发明实施例2中scfv高保真酶扩增产物琼脂糖凝胶电泳图,其中m指代dl5000,1~9指代scfv的扩增产物。
图6是本发明实施例2中全人源单链抗体scfv的sds-page图,其中m指代蛋白marker,1~9分别指代4a01、6d09、5a03、5f01、5f08、6a09、1a06、1h04和3f08。
图7是本发明实施例4中scfv特异性的elisa鉴定柱状图。
图8是本发明实施例4中scfv阻断gii.17pp-hbga结合能力的elisa鉴定柱状图。
具体实施方式
结合以下实施例对本发明作进一步说明。
实施例1。
一种gii.17型诺如病毒全人源中和性单链抗体的制备方法,包括以下步骤:
s1:构建初级全人源噬菌体单链抗体库。
具体是,采集gii.17nov目标对象恢复期外周血,利用密度梯度离心法分离出pbmc,利用trozil法提取pbmc的总rna,逆转录合成cdna。gii.17型nov目标对象恢复期外周血来自由gii.17nov引起的急性胃肠炎暴发的目标对象。
设计合成人抗体基因的引物,并以cdna为模板,扩增抗体重链可变区基因vh和轻链可变区基因vl,重叠延伸pcr拼接vh和vl形成scfv基因。人抗体基因的引物包括6对vh家族基因、用于连接抗体重轻链的氨基酸序列为ggggsggggsggggs的两条互补连接肽、12对vl家族基因以及sfii和noti酶切位点的scfv上下游引物。
使用限制性内切酶sfii和noti对scfv基因和噬菌粒载体pcantab-5e双酶切,然后用t4连接酶连接构建形成重组质粒pcantab-5e-scfv,将重组质粒pcantab-5e-scfv电转化至e.colitg1感受态细胞后加入辅助噬菌体m13k07培养,得到全人源噬菌体单链抗体库并挑取抗体库中单克隆做菌液pcr且对阳性克隆进行测序分析。其中,辅助噬菌体m13k07与e.colitg1感受态细胞的moi=100:1,辅助噬菌体m13k07的滴度约为1012cfu/ml。全人源噬菌体单链抗体具有亲和力强、特异性高的特点,且不存在种属异源性。本实施例中将含有初级全人源噬菌体单链抗体库的菌液稀释涂布2×yt-a平板,挑选单克隆做菌液pcr,以分析scfv基因是否成功插入到pcantab-5e上,鉴定抗体库的重组率。将阳性克隆送测序,以分析抗体库的多样性。
s2:对初级全人源噬菌体单链抗体库进行淘筛及特异性鉴定。
具体是,以gii.17novp颗粒为固相抗原,对全人源噬菌体单链抗体库进行富集性筛选获得与gii.17novp颗粒结合的噬菌体并扩增,得到特异性gii.17nov噬菌体单链抗体库。富集性筛选具体为将gii.17novp颗粒包被免疫管,吸附噬菌体单链抗体库,然后采用甘氨酸-盐酸洗脱法进行富集性淘选,重复进行4轮吸附-洗涤-洗脱-感染-拯救的过程,使得与gii.17novp颗粒结合的噬菌体有效富集,具有高效、快捷、经济等优势,所得到的抗体库也易于进行高通量筛选。
将第4轮洗脱后的产出铺到氨苄抗性琼脂板上挑取单克隆,补加辅助噬菌体m13k07拯救,离心制备噬菌体上清,用phage-elisa鉴定噬菌体上清的特异性,并选取阳性克隆进行测序分析。用phage-elisa鉴定噬菌体上清的特异性的具体过程为将gii.17novp颗粒、作为对照抗原的轮状病毒vp8*和作为空白对照pbs包被于96孔酶标板,以噬菌体上清为一抗,同时以无噬菌体上清的培养基为阴性对照,hrp-鼠抗m13作为二抗,phage-elisa鉴定特异性,若包被p颗粒的od450值大于包被vp8*对应孔2倍以上,则判定为阳性噬菌体上清。
s3:制备gii.17nov全人源单链抗体。
具体是,设计原核表达引物,以步骤s2所得阳性噬菌体上清的菌液为模板,扩增scfv片段,使用限制性内切酶bamhi和noti双酶切载体pgex-4t-1,并将scfv片段重组克隆至载体pgex-4t-1上,将产物转化至e.colit0p10感受态细胞中,挑取单克隆做菌液pcr,鉴定重组质粒是否插入目的片段,并对阳性克隆进行测序分析后提取获得pgex-scfv重组质粒。原核表达引物根据步骤s2中阳性克隆测序分析结果设计,并在上下游引物中分别引入载体pgex-4t-1的两末端同源序列、bamhi酶切位点和noti酶切位点。
利用大肠杆菌表达系统表达pgex-scfv重组质粒,然后用gst-resin亲和层析柱纯化,加入凝血酶去除融合蛋白gst-scfv的蛋白标签gst后获得scfv,即gii.17nov全人源中和性单链抗体。利用大肠杆菌表达系统表达pgex-scfv重组质粒的具体过程为将插入目的片段的重组质粒转化至e.colibl21感受态细胞中,挑取单克隆放置于37℃下以220rpm的转速振荡培养12h~16h,然后按体积比为1:100的比例转接至新鲜lb液体培养基中,在37℃下以220rpm振荡培养至细菌对数期,加入iptg于22℃以200rpm转速振荡12h诱导表达。
将诱导表达获得的菌液于4℃以5000rpm的转速离心12min,弃上清,收集菌体,然后用pbs重悬菌体,并将菌液于冰浴中进行超声破碎,然后将菌液放置于4℃下以12000rpm的转度离心75min。
该gii.17型诺如病毒全人源中和性单链抗体的制备方法,能够制备得到gii.17nov全人源单链抗体,为诺如病毒感染病的防治和疫苗研究奠定了基础,制备出的抗体亲和力强,特异性高,且制备过程具有快捷、经济和高效的优势。
实施例2。
一种gii.17型诺如病毒全人源中和性单链抗体的制备方法,其它特征与实施例1相同,不同之处在于,包括以下步骤:
s1:构建初级全人源噬菌体单链抗体库。
(1)采集gii.17nov患者恢复期外周血13份,用密度梯度离心法分离pbmc,用trizol法提前pbmc的总rna。具体过程如下:
分离pbmc:用ficoll-paquetmplusmedia淋巴细胞分离液利用密度梯度离心法从nov患者恢复期外周血中分离pbmc和血浆。将采集的5ml外周血缓慢加入5ml淋巴细胞分离液中,室温400g离心30min。吸取第一层血浆于-80℃保存,取第二层乳白色单个核细胞于新的离心管中,用10mlpbs重悬洗涤,1500rpm离心10min,弃上清,重复2次。取少许细胞稀释,在细胞计数板上计数,每107个细胞加1ml的trizol并于-80℃保存。
提取总rna:取出冻存的含有裂解细胞的trizol,室温融化平衡5min。每1ml的trizol试剂加200μl氯仿,涡旋震荡混匀,使氯仿和trizol不分层,室温孵育5min。取12000g于4℃离心15min,混合物分离为红色下层(酚-氯仿相)、中间相以及上层的无色水相。rna存在于水相,水相约600μl转移至rnase-free离心管中,加入等体积的异丙醇,颠倒混匀30s,室温静置10min,取12000g于4℃离心10min,弃上清,干燥rna沉淀,加入50μlrnase-freeddh2o溶解rna。
(2)rt-pcr合成cdna第一链。具体过程如下:
以提取的总rna为模板,用特异性引物或随机引物合成cdna第一链。在冰上配制以下逆转录反应体系,配置过程为:先加入rna4μl,随机引物或特异性引物1μl,rnase-freeddh2o7μl,于65℃孵育5min后置于冰上2min,再加入ribolockrnaseinhibitor(20u/μl)1μl,10mmdntpsmix2μl,revertaidm-mulvrt(200u/μl)1μl,于25℃反应5min,于42℃反应60min,于70℃反应5min。反应结束后于-80℃保存。
(3)pcr扩增vh和vl片段。具体过程如下:
如表1所示,设计的引物中共包括了6对vh家族基因,用于连接抗体重、轻链的两条互补连接肽、12对vl家族基因以及包含了酶切位点(sfii和noti)的scfv上下游引物。以cdna第一链为模板,以等比例混合后的抗体重、轻链的上下游引物分别扩增vh和vl片段。pcr反应体系为:
表1噬菌体单链抗体库的引物汇总表
(4)重叠延伸pcr构建与扩增scfv片段
按照正常人抗体胚系可变区基因频率,其中轻链kappa亚型占60%,轻链lambda链占40%,将轻链2亚型可变区基因以该比例混合作为下一步pcr模板(vl)。以等量的vh和vl片段互为模板和引物,通过重叠延伸pcr反应,将重链、轻链随机拼接形成scfv片段。反应体系为:10×sabuffer5μl,50×dntpsmix(10mm)1μl,vh片段(100ng)xμl,vl片段(100ng)xμl,50×advantage2polymerasemix1μl,ddh2oxμl,总体积为50μl。pcr反应程序:95℃变性2min;然后95℃变性1min,68℃退火1min,68℃延伸2min,进行7个循环。pcr反应结束后,每管再加入预配含scfv双向引物(5’sfii与3’noti)的混合体系:10×sabuffer5μl,50×dntpsmix(10mm)1μl,5’sfii(10μm)4μl,3’noti(10μm)4μl,50×advantage2polymerasemix1μl,ddh2o37μl。pcr反应程序:95℃变性2min,95℃变性1min,68℃退火2min,进行35个循环。pcr产物经1.2%琼脂糖凝胶电泳分离目的片段,切取750bp左右的scfv片段,按照promega胶回收试剂盒说明书进行纯化回收。
(5)双酶切scfv片段和噬菌粒载体pcantab-5e
用限制性内切酶noti和sfii双酶切scfv片段,反应体系为:10×greenbuffer5μl,noti2.5μl,scfv(3μg)xμl,ddh2oxμl,总体积50μl。用限制性内切酶noti和sfii双酶切pcantab-5e,反应体系为:10×greenbuffer5μl,noti2.5μl,pcantab-5e(3μg)xμl,ddh2oxμl,总体积50μl。以上体系经37℃反应5h后,加入2μlsfii,混匀后离心,50℃反应5h。程序结束后,用promega胶回收试剂盒纯化回收双酶切后的scfv片段和pcantab-5e。
(6)构建表达载体pcantab-5e-scfv
将酶切并纯化回收后的scfv片段和pcantab-5e以摩尔比为3:1的比率连接,t4连接酶16℃反应16h,反应体系如下:10×t4ligasebuffer5μl,t4dnaligase2.5μl,pcantab-5e(300ng)xμl,scfv(150ng)xμl,ddh2oxμl,总体积50μl。程序结束后,用promega胶回收试剂盒纯化回收连接产物。
(7)构建噬菌体单链抗体库。具体过程如下:
a.取出一管tg1电感受态细胞300μl于冰上融化,加入240ngscfv-pcantab-5e连接产物,轻柔混匀;b.将上述混合物缓慢加入至预冷的电转杯(0.2cm),2.5kv电击;c.立即加入1ml37℃预热的soc培养液,轻柔冲洗,转移至细菌培养瓶,补加19mlsoc培养基,37℃180rpm45min;d.取10μl菌液,梯度稀释后铺氨苄抗性琼脂板,30℃过夜培养,计菌落数算库容,库容约为2.4×107cfu/ml,并挑取单菌落做菌落pcr,以鉴定抗体库的重组率及多样性;e.以moi=100:1的比例向剩余菌液中加入辅助噬菌体m13k07(2×1011cfu/ml),并加入氨苄青霉素,37℃静止孵育30min,37℃180rpm1h;f.1000g离心10min,弃上清,将菌液重悬于100ml2×yt-ak液体培养基中,37℃250rpm振荡培养过夜;g.次日,4℃1000g离心15min,收集上清。加10mlpeg/nacl至上清,混匀后冰上孵育30min;h.4℃10000g离心30min,弃上清,所得沉淀即为初级噬菌体单链抗体库,用5ml1%bsa重悬沉淀;i.4℃10000g离心5min,取上清。梯度稀释后,分别加入200μl的新鲜对数期tg1,室温孵育15min后,铺氨苄抗性琼脂板,过夜培养后数克隆数,计算重组噬菌体滴度。
s2:对全人源噬菌体单链抗体库进行淘筛及特异性鉴定。
(1)噬菌体单链抗体库的淘筛。具体过程如下:
a.包被:用pbs将gii.17novp颗粒稀释至终浓度为5μg/ml,加1ml至免疫管,4℃孵育过夜;b.封闭:用pbs洗2次后,用5%脱脂奶加满免疫管,37℃封闭2h,弃去封闭液,用pbs洗2次;c.抗体库的吸附:加入1ml噬菌体抗体库,37℃孵育2h,期间振荡数次;d.洗涤:弃上清,加入2ml0.05%pbst,上下颠倒30次后弃去,此为洗一遍,第一轮重复洗9遍,以后每轮重复洗15遍;e.直接感染:加1ml对数期tg1,37℃孵育15min。将菌液转移至培养瓶中,补加2×yt培养基7ml;f.甘氨酸-盐酸洗脱后感染:加1mlph2.20.1m甘氨酸-盐酸洗脱液,37℃孵育10min,期间振荡数次。加入17μl2mtris中和液快速混匀,再加入1ml对数期tg1,37℃孵育15min。将菌液转移至上述培养瓶中。取10μl菌液(即稀释1000倍)铺氨苄抗性琼脂板,隔日计算产出量;g.扩增:将培养瓶于37℃,220rpm振荡培养1h,补加90ml2×yt-a培养基;h.拯救:培养至细菌对数期,加入2ml辅助噬菌体m13k07。30℃220rpm振荡培养过夜;i.peg/nacl沉淀噬菌体:次日收集菌液,4℃8000rpm离心15min。取上清,加入1/5体积的peg/nacl,充分混匀后,冰浴30min。4℃8000rpm离心30min。弃上清,加2ml1%bsa重悬沉淀。4℃10000g离心5min,取上清;j.测滴度:取10μl上一步获得的噬菌体上清,梯度稀释后感染200μl对数期tg1,铺氨苄抗性琼脂板。隔日计数并计算滴度,确定下一轮的噬菌体抗体的投入量。“吸附-洗涤-洗脱-感染-拯救”的过程重复4轮,富集与gii.17p颗粒特异性结合的噬菌体。噬菌体单链抗体库的4轮淘筛结果如表2所示。
表2噬菌体抗体库的4轮淘筛结果
(2)gii.17nov特异性噬菌体单链抗体的鉴定。本步骤使用phage-elisa进行,具体过程如下:
a.重组scfv噬菌体单克隆的拯救:在第4轮洗脱后铺的氨苄抗性琼脂板上挑取单克隆于1.5ml2×yt-a液体培养基中,37℃200rpm8h,每管补加30μlm13k07,30℃200rpm振荡培养过夜;b.包被:用pbs将gii.17p颗粒和轮状病毒vp8*(对照抗原)稀释至终浓度为0.1μg/ml,100μl/孔,包被96孔酶标板,4℃孵育过夜;c.次日,将过夜菌液于室温1000rpm离心5min,取上清备用;d.封闭:0.05%pbst洗2次,加入5%脱脂奶300μl/孔,37℃2h;e.一抗:0.05%pbst洗5次,加入噬菌体上清100μl/孔,37℃2h;f.二抗:0.05%pbst洗5次,加入hrp-鼠抗m13抗体(1:5000)100μl/孔,37℃1h;g.显色:0.05%pbst洗5次后,加入tmb100μl/孔,避光显色10min;h.终止显色:加入2m磷酸100μl/孔。测定各孔的od450值,若包被p颗粒的od450大于包被vp8*对应孔的2倍以上,则判定为阳性噬菌体上清,并将阳性克隆送测序分析。噬菌体阳性克隆的phage-elisa鉴定结果如图3所示。
s3:制备gii.17nov全人源单链抗体。
(1)高保真酶扩增scfv片段。具体过程如下:
根据实施例2的步骤(2)中阳性克隆的测序结果,分别设计scfv原核表达克隆引物,scfv原核表达克隆引物如表3所示。在scfv上下游引物的5’端引入线性化载体两末端同源序列,且在上游引物插入bamhi酶切位点,下游引物插入noti酶切位点。以阳性菌液为模板,用对应的引物分别扩增scfv片段。pcr反应体系为:阳性克隆(菌液)0.5μl,上游引物(10mm)1μl,下游引物(10mm)1μl,2×phantamaxmastermix10μl,ddh2o7.5μl。反应程序:95℃变性5min;然后95℃变性30s,60℃退火30s,72℃延伸30s,进行30个循环;72℃再延伸10min;最后于4℃保存。pcr完成后,1.2%琼脂糖凝胶电泳,切取750bp左右的scfv片段,按照promega胶回收试剂盒说明书进行纯化回收。琼脂糖凝胶电泳结果如图4所示。
表3scfv原核表达克隆引物
(2)双酶切载体pgex-4t-1。具体过程如下:
用限制性内切酶bamhi和noti双酶切pgex-4t-1,使其线性化,反应体系为:10×greenbuffer5μl,bamhi2μl,noti1μl,pgex-4t-1(3μg)xμl,ddh2oxμl,总体积50μl。37℃反应5h,切取5kb左右的载体片段,按照promega胶回收试剂盒说明书进行纯化回收。
(3)构建原核表达载体pgex-scfv。具体过程如下:
将scfv重组克隆至线性化载体pgex-4t-1,反应体系为:5×ceiibuffer4μl,pgex-4t-1(0.03pmol)xμl,scfv(0.06pmol)xμl,
(4)表达和纯化单链抗体scfv。具体过程如下:
将重组质粒pgex-scfv转化至e.colibl21感受态细胞中,菌液涂于氨苄抗性的琼脂板,37℃培养过夜。次日,挑取单菌落于5mllb-a液体培养基中,37℃220rpm12-16h。将菌液以1:100的比例接种于新鲜lb液体培养基中,37℃200rpm震荡培养至细菌对数期,加入iptg至终浓度为0.4mm,22℃200rpm震荡培养12-16h。次日,将菌液于4℃5000rpm离心12min,弃上清,用pbs充分重悬菌体,并将菌液于冰浴中对其进行超声破碎。超声后于4℃12000rpm离心75min,将上清液和gst-resin室温结合1h。用pbs洗去杂蛋白后,加入凝血酶于22℃酶切过夜。次日收集滤液即获得抗体scfv,纯化后的样品进行sds-page电泳检测,检测结果如图5所示。
对上述整个制备过程进行分析:从图1和图2可看出,成功扩增出vh和vl片段,大小均在300-500bp之间,并成功拼接scfv片段,大小约750bp。在噬菌体抗体库中随机选取单克隆做菌落pcr,从图3可以看出,该抗体库的重组率良好,并将阳性克隆送测序,经分析各序列之间互不相同,说明该抗体库的多样性良好。从表2可以看出,以gii.17novp颗粒为固相抗原,对噬菌体抗体库进行了4轮富集,噬菌体的产出率从第一轮的2.8×10-7提升至第四轮的2.9×10-5,说明抗gii.17nov特异性噬菌体单链抗体得到了有效富集。从图4可看出,通过phage-elisa鉴定,表明有11株序列不同的与gii.17novp颗粒特异性结合的单链抗体scfv。其中,图4中的control代表无噬菌体上清的培养基,为阴性对照。从图5和图6可看出,在与gii.17具有良好亲和力的阳性克隆中成功扩增出scfv,并在大肠杆菌系统中成功表达,可见9个约27kda的目的蛋白,与scfv大小相符,成功获得9株全人源单链抗体scfv。该gii.17型诺如病毒全人源中和性单链抗体的制备方法,获得的抗体库有效库容大,且重组率和多样性好。另外,该方法制备的抗体具有中和能力,能够阻断gii.17nov与hbga受体的结合,为后续中和抗原表位的筛选及抗体药物研发具有重要意义。
实施例3。
一种gii.17型诺如病毒全人源中和性单链抗体的鉴定方法,用于鉴定gii.17型诺如病毒全人源中和性单链抗体的制备方法制备的scfv。鉴定步骤包括:
特异性鉴定,具体为将scfv包被于96孔酶标板,同时以gst蛋白为阴性对照,pbs为空白对照,封闭后加入gii.17novp颗粒,以鼠抗gii.17血清为一抗,hrp-羊抗鼠igg为二抗,elisa鉴定scfv的特异性。
中和性鉴定,具体为包被分泌型b型唾液于96孔酶标板,封闭后加入gii.17novp颗粒与scfv的混合液,同时阳性对照用未加scfv的p颗粒,阴性对照用确定没有阻断能力的血浆和p颗粒的混合液,空白对照用1%脱脂奶粉-pbs代替scfv和p颗粒的混合液,并以小鼠抗gii.17血清为一抗,hrp-羊抗鼠igg为二抗,体外hbga受体阻断实验鉴定scfv的中和性。
该gii.17型诺如病毒全人源中和性单链抗体的鉴定方法,能够鉴定出gii.17nov全人源单链抗体的特异性和中和性。
实施例4。
一种gii.17型诺如病毒全人源中和性单链抗体的鉴定方法,其它特征与实施例3相同,不同之处在于:本实施例的鉴定过程具体为:
(1)鉴定scfv抗体的特异性
用pbs将scfv抗体稀释至终浓度为1μg/ml,100μl/孔,包被96孔酶标板,同时以gst蛋白为阴性对照,4℃过夜;0.05%pbst洗板1次,加入5%脱脂奶,300μl/孔,37℃1h;0.05%pbst洗板1次,加入0.02μg/mlgii.17novp颗粒,100μl/孔,37℃1h;0.05%pbst洗板5次,加入鼠抗gii.17血清(1:5000),100μl/孔,37℃1h;0.05%pbst洗板5次,加入hrp-羊抗鼠igg(1:5000),100μl/孔,37℃1h;0.05%pbst洗板5次,加入tmb,100μl/孔,避光显色10min;加入2m磷酸终止反应,100μl/孔,测定各孔的od450值。od450是指各样本显色后在主波长450nm处的吸光值,是elisa常用的指标。当实验孔的od450大于阴性样本od450的2倍及以上时,则判定为该scfv抗体实验样本为阳性。
(2)鉴定scfv抗体的中和能力
用hbga受体结合实验确定所用gii.17novp颗粒的浓度,并用结合力强和稳定性高的分泌型b型抗原作为hbga受体与梯度稀释的gii.17novp颗粒结合,使其od450在0.7-1.3之间。用pbs将b型唾液稀释至终浓度为1:1000,100μl/孔,包被96孔酶标板,4℃孵育过夜;0.05%pbst洗板1次,加入5%脱脂奶,300μl/孔,同时将gii.17novp颗粒与梯度稀释的scfv混匀后,阳性对照用未加scfv的p颗粒、阴性对照用确定没有阻断能力的血浆代替scfv、空白对照用1%脱脂奶代替scfv和p颗粒的混合液,37℃1h;0.05%pbst洗板1次,将上一步的混合液分别加入酶标板中,37℃1h;0.05%pbst洗板5次,加入鼠抗gii.17血清(1:5000),100μl/孔,37℃1h;0.05%pbst洗板5次,加入hrp-羊抗鼠igg(1:5000),100μl/孔,37℃1h;0.05%pbst洗板5次,加入tmb,100μl/孔,避光显色10min;加入2m磷酸终止反应,100μl/孔,测定各孔的od450值,当阻断率>50%时,则判定该scfv抗体实验样本具有中和能力。
鉴定结果如图7和图8所示,从图7和图8可看出,9株抗体能与gii.17novp颗粒结合,均具有良好反应性,且有7株能明显阻断gii.17novp颗粒与hbga受体的结合,是具有中和能力的全人源单链抗体。其中,图7中gst是阴性对照,图8中control是阴性对照,是没有阻断能力的血浆和p颗粒的混合液。
该gii.17型诺如病毒全人源中和性单链抗体的鉴定方法,能够鉴定出gii.17nov全人源单链抗体的特异性和中和性,且鉴定方法快捷准确。
最后应当说明的是,以上实施例仅用以说明本发明的技术方案而非对本发明保护范围的限制,尽管参照较佳实施例对本发明作了详细说明,本领域的普通技术人员应当理解,可以对本发明的技术方案进行修改或者等同替换,而不脱离本发明技术方案的实质和范围。
序列表
<110>南方医科大学
<120>gii.17型诺如病毒全人源中和性单链抗体的制备及鉴定方法
<130>gzzrh0504-20-1-1003
<160>49
<170>siposequencelisting1.0
<210>1
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>1
gcggcccagccggccatggcccaggtgcagctggtgcagtctgg44
<210>2
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>2
gcggcccagccggccatggcccaggtcaacttaagggagtctgg44
<210>3
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>3
gcggcccagccggccatggccgaggtgcagctggtggagtctgg44
<210>4
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>4
gcggcccagccggccatggcccaggtgcagctgcaggagtcggg44
<210>5
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>5
gcggcccagccggccatggccgaggtgcagctgttgcagtctgc44
<210>6
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>6
gcggcccagccggccatggcccaggtacagctgcagcagtcagg44
<210>7
<211>57
<212>dna
<213>人工序列(artificialsequence)
<400>7
gccagaaccacctccgcctgaaccgcctccacctgaggagacggtgaccagggtgcc57
<210>8
<211>57
<212>dna
<213>人工序列(artificialsequence)
<400>8
gccagaaccacctccgcctgaaccgcctccacctgaagagacggtgaccattgtccc57
<210>9
<211>57
<212>dna
<213>人工序列(artificialsequence)
<400>9
gccagaaccacctccgcctgaaccgcctccacctgaggagacggtgaccagggttcc57
<210>10
<211>57
<212>dna
<213>人工序列(artificialsequence)
<400>10
gccagaaccacctccgcctgaaccgcctccacctgaggagacggtgaccgtggtccc57
<210>11
<211>56
<212>dna
<213>人工序列(artificialsequence)
<400>11
tcaggcggaggtggttctggcagtggcggatcggacatccagatgacccagtctcc56
<210>12
<211>56
<212>dna
<213>人工序列(artificialsequence)
<400>12
tcaggcggaggtggttctggcagtggcggatcggatgttgtgatgactcagtctcc56
<210>13
<211>56
<212>dna
<213>人工序列(artificialsequence)
<400>13
tcaggcggaggtggttctggcagtggcggatcggaaattgtgttgacgcagtctcc56
<210>14
<211>56
<212>dna
<213>人工序列(artificialsequence)
<400>14
tcaggcggaggtggttctggcagtggcggatcggacatcgtgatgacccagtctcc56
<210>15
<211>56
<212>dna
<213>人工序列(artificialsequence)
<400>15
tcaggcggaggtggttctggcagtggcggatcggaaacgacactcacgcagtctcc56
<210>16
<211>56
<212>dna
<213>人工序列(artificialsequence)
<400>16
tcaggcggaggtggttctggcagtggcggatcggaaattgtgctgactcagtctcc56
<210>17
<211>48
<212>dna
<213>人工序列(artificialsequence)
<400>17
gagtcattctcgacttgcggccgcacgtttgatttccaccttggtccc48
<210>18
<211>48
<212>dna
<213>人工序列(artificialsequence)
<400>18
gagtcattctcgacttgcggccgcacgtttgatctccagcttggtccc48
<210>19
<211>48
<212>dna
<213>人工序列(artificialsequence)
<400>19
gagtcattctcgacttgcggccgcacgtttgatatccactttggtccc48
<210>20
<211>48
<212>dna
<213>人工序列(artificialsequence)
<400>20
gagtcattctcgacttgcggccgcacgtttgatctccaccttggtccc48
<210>21
<211>48
<212>dna
<213>人工序列(artificialsequence)
<400>21
gagtcattctcgacttgcggccgcacgtttaatctccagtcgtgtccc48
<210>22
<211>56
<212>dna
<213>人工序列(artificialsequence)
<400>22
tcaggcggaggtggttctggcagtggcggatcgcagtctgtgttgacgcagccgcc56
<210>23
<211>56
<212>dna
<213>人工序列(artificialsequence)
<400>23
tcaggcggaggtggttctggcagtggcggatcgcagtctgccctgactcagcctgc56
<210>24
<211>56
<212>dna
<213>人工序列(artificialsequence)
<400>24
tcaggcggaggtggttctggcagtggcggatcgtcctatgtgctgactcagccacc56
<210>25
<211>56
<212>dna
<213>人工序列(artificialsequence)
<400>25
tcaggcggaggtggttctggcagtggcggatcgtcttctgagctgactcaggaccc56
<210>26
<211>56
<212>dna
<213>人工序列(artificialsequence)
<400>26
tcaggcggaggtggttctggcagtggcggatcgcacgttatactgactcaaccgcc56
<210>27
<211>56
<212>dna
<213>人工序列(artificialsequence)
<400>27
tcaggcggaggtggttctggcagtggcggatcgcaggctgtgctcactcagccgtc56
<210>28
<211>56
<212>dna
<213>人工序列(artificialsequence)
<400>28
tcaggcggaggtggttctggcagtggcggatcgaattttatgctgactcagcccca56
<210>29
<211>48
<212>dna
<213>人工序列(artificialsequence)
<400>29
gagtcattctcgacttgcggccgcacctaggacggtgaccttggtccc48
<210>30
<211>48
<212>dna
<213>人工序列(artificialsequence)
<400>30
gagtcattctcgacttgcggccgcacctaggacggtcagcttggtccc48
<210>31
<211>48
<212>dna
<213>人工序列(artificialsequence)
<400>31
gagtcattctcgacttgcggccgcacctaaaacggtgagctgggtccc48
<210>32
<211>33
<212>dna
<213>人工序列(artificialsequence)
<400>32
gtcctcgcaactgcggcccagccggccatggcc33
<210>33
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>33
gagtcattctcgacttgcggccgcac26
<210>34
<211>753
<212>dna
<213>人工序列(artificialsequence)
<400>34
gaggtgcagctggtggagtctggggctgaggtgaagaggcctggggcctcagtgaaggtc60
tcctgcaaggcttctggttacacctttaccagctatggtatcagctgggtgcgacaggcc120
cctggacaagggcttgagtggatgggatggatcagcgcttacaatggtaacacaaactat180
gcacagaagctccagggcagagtcaccatgaccacagacacatccacgagcacagcctac240
atggagctgaggagcctgagatctgacgacacggccgtgtattactgtgcgagagagcct300
ttttcggggtggctacgattggtgtttgactactggggccagggaaccctggtcaccgtc360
tcctcaggtggaggcggttcaggcggaggtggttctggaggtggcggatcggacatcgtg420
atgacccagtctccactctccctgcccgtcacccttggacagccggcctccatctcctgc480
agatctagtcaaagcctcgtacacagtgatggaaacacctacttgaattggtttcagcag540
aggccaggccaatctccaaggcgcctaatttataaggtttctaaccgggactctggggtc600
ccagacagattcagcggcagtgggtcaggcactgatttcacactgaaaatcagcagggtg660
gaggctgaggatgttggggtttattactgcatgcaaggtacacactggcctccgcatact720
tttggccaggggaccaaggtggaaatcaaacgt753
<210>35
<211>738
<212>dna
<213>人工序列(artificialsequence)
<400>35
gaggtgcagctggtggagtctgggggaggcttggtccagcctggggtgtacctgagactc60
tcctgttcagcctctcgattcaccttcagtaactatgctatgcactgggtccgccaggct120
ccagggaagggactggaatatgtttcaactattagtagtaatggcgatagcacatacttc180
gcagactcagtgaagggcagattcaccatctccagagacaattccaagaacactctgtat240
cttcgaatgagcagtctgagaactgaggacacggctatatattactgtgtgaaagatagg300
aagttagcagtgactggtactggtgcttttgatatctggggccaagggacaatggtcacc360
gtctcttcaggtggaggcggttcaggcggaggtggttctggaggtggcggatcggacatc420
gtgatgacccagtctccgtccaccctgtctgcatctgtaggagacagagttgtcatcact480
tgccgggcaagtcaggccattataaatgatttcggctggtatcagctgaaaccagggaaa540
gcccctaacctcctgatctatgctgcatccattttgcaaagtggggtcccatcaaggttc600
agtggcagtggatctgggacagacttcactctcaccatcaccggtctgcagcctgaagat660
tttgcaacttactactgtcaacagagttacaatagtctttggactttcggccaagggacc720
aaagtggatatcaaacgt738
<210>36
<211>744
<212>dna
<213>人工序列(artificialsequence)
<400>36
gaggtgcagctggtggagtctgggggaggcgtggtccagcctgggaggtccctgagactc60
tcctgtgcagcctctggattcaccttcagtagctatgctatgcactgggtccgccaggct120
ccaggcaaggggctggagtgggtggcagttatatcatatgatggaagtaataaatactac180
gcagactccgtgaagggccgattcaccatctccagagacaattccaagaacacgctgtat240
ctgcaaatgaacagcctgagagctgaggacacggccgtatattactgtgcgagagtacgg300
agtatgactacagtaacctactactactactacggtatggacgtctggggccaagggaca360
atggtcaccgtctcttcaggtggaggcggttcaggcggaggtggttctggaggtggcgga420
tcggaaattgtgctgactcagtctccatcttccgtgtctgcatctgtaggagacagagtc480
accctcacttgtcgggcgagtcaggatattagtaattggttagcctggtatcagcagaga540
ccagggaaagcccctagactcttgatctttgctgcatctactttgcaaagtggggtccca600
tccaggttcagcggcagtggatctggggcggatttcactctcaccatcagcagcctgcaa660
cctgatgattttgcaacttattactgccaacattattatagttattctggtttcggccct720
gggaccaaagtggaaatcaaacgt744
<210>37
<211>720
<212>dna
<213>人工序列(artificialsequence)
<400>37
caggtacagctgcagcagtcagggggaggcttggtacagcctggcaggtccctgagactc60
tcctgtgcagcctctggattcacctttgatgattatgccatgcactgggtccggcaagct120
ccagggaagggcctggagtgggtctcaggtattagttggaatagtggtagcataggctat180
gcggactctgtgaagggccgattcaccatctccagagacaacgccaagaactccctgtat240
ctgcaaatgaacagtctgagagctgaggacacggccttgtattaccgacccaaatagcat300
tggctggttcttgactactggggccagggcaccctggtcaccgtctcctcaggtggaggc360
ggttcaggcggaggtggttctggaggtggcggatcggatgttgtgatgactcactctcca420
tcctccctgtctgcatctgtaggagacagagtcaccatcacttgccgggcgagtcagggc480
attagcaattatttagcctggtatcagcagaaaccaggggaagttcctaaggtcctgatc540
tatgctgcatccactttgcaatcaggggtcccatctcgattcagcggcagtggatctggg600
acagatttcactctcatcatcagcagcctgcagcctgaagatgttggaacttattactgt660
caaaagtacagcaatgccccattcactctcggccctgggaccaacgtggaaatcaaacgt720
<210>38
<211>732
<212>dna
<213>人工序列(artificialsequence)
<400>38
caggtacagctgcagcagtcaggcccaggactggtgaagtcttcgaggaccctgtccctc60
agttgcattgtctctggtggctccatcagcagcaattactactgggcctggatccgccag120
cccccagggaagggactggagtggattgggagtatctcttatagtgggagcacctactac180
aactcgtccctcaagaatcgtgtcaccatatttatagacacgtccaaaaaccagttctcc240
ctgaagctgaagtctttgaccgccgcagacacggctgtgtattattgtgcgagacggttc300
gtctttggtcgggggctcgggttcgacccctggggccagggcaccctggtcaccgtctcc360
tcaggtggaggcggttcaggcggaggtggttctggaggtggcggatcggaaattgtgatg420
acccagtctccatcttctgtgtctgcatctgtaggtgacagagtcaccatcacttgtcgg480
gcgagtcagggtattggcagctggttagcctggtatcagcagaaaccagggcaagcccct540
aagctcctgatttatgctgcatccagtttgcaaagtggggtcccgtcaaggttcagcggc600
agtggatctgggacagatttcactctcactatcaccagcctgcagcctgaagattttgca660
acttactattgtcaacaggctaacagtttcccattcactttcggccctgggaccaaagtg720
gatatcaaacgt732
<210>39
<211>768
<212>dna
<213>人工序列(artificialsequence)
<400>39
gaggtgcagctggtggagtctgggtctgaggtgaagaagcctgggtcctcggtgaaggtc60
tcctgcaaggcttctggaggcaccttcagcagctatgctatcagctgggtgcgacaggcc120
cctggacaagggcttgagtggatgggaaggatcatccctatccttggtatagcaaactac180
gcacagaagttccagggcagagtcacgattaccgcggacaaatccacgagcacagcctac240
atggagctgagcagcctgagatctgaggacacggccgtgtattactgtgcgagagatcgc300
ccgtattactatggttcggggagttccccactcgcttattactactactactactacatg360
gacgtctggggcaaagggacaatggtcaccgtctcttcaggtggaggcggttcaggcgga420
ggtggttctggaggtggcggatcggaaattgtgctgactcagtctccatcttccgtgtct480
gcatctgtaggagacagagtcaccatcgcttgtcgggcgagtcagggtattagcagctgg540
ttagcctggtaccagcagaaaccagggaaagcccctaacctcctgatctatgctgcatcc600
actctgcaaactggggtcccatcaaggttcagcggcagtggatctgggacagattacact660
ctcaccatcagcagcctgcagcctgaagatcttgcaacttacttttgtcaacaggctagc720
agtttccccttcactttcggcggagggaccaaagtggatatcaaacgt768
<210>40
<211>762
<212>dna
<213>人工序列(artificialsequence)
<400>40
caggtgcagctgcaggagtcgggcccaggactggtgaagccttcacagaccctgtccctc60
acctgcactgtctctggtggctccatcaacagtggttcttactattggagctggatccgg120
cagcccgccgggaagggactggagtggattgggcgtatctttaccagtgggagcaccaac180
tacaacccctccctcaagagtcgagtcaccatttcactagacaagttcgagaaccagttc240
tccctgaagttgagctctgtgaccgccgcagacacggccgtgtattactgtgcgagagga300
cgccccgtggatccagctctggggtactggtacttcgatctctggggccgtggcaccctg360
gtcaccgtctcctcaggtggaggcggttcaggcggaggtggttctggaggtggcggatcg420
gaaactgtgctgactcagtctccagactccctggctgtgtctctgggcgagagggccacc480
atcaactgcaagtccagccagagcgttttatacagctccaacaataagaactacttagct540
tggtaccagcagaaaccaggacagcctcctaagctgctcatttactgggcatctacccgg600
gaatccggggtccctgaccgattcagtggcagcgggtctgggacagatttcactctcacc660
atcagcagcctgcaggctgaagatgtggcagtttattactgtcagcaatattataatact720
cctccgacgttcggccaagggaccaaggtggaaatcaaacgt762
<210>41
<211>735
<212>dna
<213>人工序列(artificialsequence)
<400>41
caggtgcagctgcaggagtcgggcccaggactggtgaaggcttcggagaccctgtccctc60
acgtgcattgtccctggtggttccatcaagaattacttctggacctggattcggctgccc120
ccagggaaggggctggaatgggttgggtctgtcgatcacgatggcaataccgactacaac180
ccctccctcaagagtcgagtcaccatatcgctagacacgtccaggagccagttctccctg240
aaggtatactctgtgaccgctgcggacacggccgtgtattactgtgcgagaggccacccg300
ccggggagttcgctcgccctcgcctttgactcctggggccagggcaccctggtcaccgtc360
tcctcaggtggaggcggttcaggcggaggtggttctggaggtggcggatcggacatcgtg420
atgacccagtctccatcctccctgtctgcatctgtaggagacagagtcaccatcacttgc480
cgggcaagtcagagcattagcagctatttaaattggtatcagcagaaaccagggaaagcc540
cctaagctcctgatctatgctgcatccagtttgcaaagtggggtcccatcaaggttcagt600
ggcagtggatctgggacagatttcactctcaccatcagcagtctgcaacctgaagatttt660
gcaacttactactgtcaacagagttacagtaccccgtggacgttcggccaagggaccaaa720
gtggatatcaaacgt735
<210>42
<211>747
<212>dna
<213>人工序列(artificialsequence)
<400>42
caggtgcagctggtgtagtctggggcagaggtgaaaaagcccggggagtctctgaagatc60
tcctgtaagggttctggatacaactttgccaactactggatcggctgggtgcgccagatg120
cccgggagaggcctggagtggatgggaattatctatcctggtgactctgacaccagatac180
agcccgtcctccctaggccaggtcaccatctcagccgacaagtctatcggcacctccttc240
ctgcagtggaacaccctgaagccctcggacgccgccatgtattactgtgcgagagcacca300
gttctccagttactgtatcctgactcttttgatctgtggggccaaggcaccctggtcacc360
gtctcctcaggtggaggcggttcaggcggaggtggttctggaggtggcggatcggatgtt420
gtgatgactcagtctccctcagcgtctgggacccccgggcagcgggtcaccatctcttct480
tctggaagcaactccaacatcggaagtaattctgtacactggtaccagcacctcccagga540
acggcccccaaactcctcatctataatactaatcagcgaccctcaggggtccctgaccga600
ttctctgcctccaagtctggcacctcagcctccctggccatcagtggcctccagtctgaa660
gatgaggctgattattactgtgcagcatgggatgacagcctgggtgactatgtcttcgga720
cctgggaccaaggtggaaatcaaacgt747
<210>43
<211>741
<212>dna
<213>人工序列(artificialsequence)
<400>43
gaggtgcagctggtggagtctgggggaggcgttgtccaccctgggaggtccctgagactc60
tcctgtgcagcctctggattcaccttcaatagttatggcatgcactgggtccgccagtct120
ccaggcaaggggctggagtgggtggcagtcatatcatatgatggcactgatcaatactat180
gcggactccgtgaagggccgattcaccatctccagagagaattccaagaactcactgtat240
ctgcaaatgaacagcctgagagccgaagacacggctctttattactgtgcgagagatagg300
gattactatagtagtagttatcgatactactttgactactggggcctgggaaccctggtc360
accgtctcctcaggtggaggcggttcaggcggaggtggttctggaggtggcggatcggac420
atccagatgacccagtctccatctttcctgtctgcatctgagggagacagagtcaccatc480
acttgccgggcaagtcagagcataagggattacttaaattggtatcagcagaaaccaggg540
aaagcccctaaactcctgatctacactgcatccagtttgcaaagtggggtcccatcaagg600
ttcagtggcagtggatctgggaccgatttcactctcaccatcagcagtctgcaacctgaa660
gattttgcaacttactactgtcaccaggcttacatctcccctcctactttcggcgggggg720
accaaagtggatatcaaacgt741
<210>44
<211>729
<212>dna
<213>人工序列(artificialsequence)
<400>44
gaggtgcagctggtggagtctggcccaggactggtgaagccttcggagaccctgtccctc60
acctgcactgtctctggtggctccatcagtagttactactggagctggatccggcagccc120
ccagggaagggactggagtggattgggtatatctattacactgggagtcccgactacaat180
ccctccctcaagagtcgagtcgccatctcaatagacacgtccaagaaccagttctccctg240
aagctgacctctgtgaccgccgctgacacggccgtgtattactgtgcgagacggacgctt300
atggcaggagctcggaacttcgatctctggggccgtggcaccctggtcaccgtctcctca360
ggtggaggcggttcaggcggaggtggttctggaggtggcggatcggacatccagatgacc420
cagtctccaccctccctgtctgcttctgttggagacagagtcaccatcacttgccaggcg480
agtcaacacattagcaagtatttgaattggtatcaacaaaagccagggaaagcccctaaa540
ctcctgatctacgacgcatccactttggaaacaggggtcccatcaaggttcagtggaagt600
ggatctgggacagattttactttcaccatcagcagcctgcagcctgaagatattgcaaca660
tattactgtcaacagtatgataatctccctttcactttcggccctgggaccaaggtggaa720
atcaaacgt729
<210>45
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>45
tcggatctggttccgcgtggatccgaggtgcagctggtggagtc44
<210>46
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>46
tcggatctggttccgcgtggatcccaggtgcagctgcaggagtc44
<210>47
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>47
tcggatctggttccgcgtggatcccaggtacagctgcagcagtc44
<210>48
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>48
tcagtcagtcacgatgcggccgcacgtttgatttccaccttggtc45
<210>49
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>49
tcagtcagtcacgatgcggccgcacgtttgatatccactttggtc45
- 还没有人留言评论。精彩留言会获得点赞!