一种特异性识别棒曲霉毒素的优化适配体及其应用与一种试剂盒
本发明属于食品安全生物技术领域,尤其是涉及一种特异性识别棒曲霉毒素的优化适配体及其应用与一种试剂盒。
背景技术:
棒曲霉毒素是水果和蔬菜中最常见的霉菌毒素之一。它在酸性条件下稳定,但在碱性和发酵条件下易失活和降解。棒曲霉毒素具有免疫,细胞和生理毒性。目前,棒曲霉素的检测方法主要有传统的薄层色谱法、气相色谱法、液相色谱法和串联质谱法。这些方法不可避免地会出现假阳性,并且检测仪器成本高、试剂消耗量大,故而难以广泛推广。除此之外,免疫学方法灵敏度高、应用范围广,但对于棒曲霉毒素这种免疫原性低的小分子毒素,面临着抗体制备相对困难,检测过程相对复杂的缺陷。
技术实现要素:
本申请针对现有技术的不足,本发明提供了一种特异性识别棒曲霉毒素的优化适配体及其应用与一种试剂盒。本发明利用适配体的二级结构对原始序列进行剪裁优化,从而建立一种快速灵敏检测棒曲霉毒素的方法。
本发明的技术方案如下:
一种特异性识别棒曲霉毒素的优化适配体,所述优化适配体为p-30,其核苷酸序列如seqidno.1所示。
进一步的,所述优化适配体的5’端或者3’端经修饰物修饰。
进一步的,所述修饰物包括氨基、巯基或羧基。
一种特异性识别棒曲霉毒素的优化适配体在检测棒曲霉毒素中的应用。
进一步的,所述应用的方法为:将金纳米簇修饰的适配体与棒曲霉毒素混合孵育,之后加入二氧化锰纳米片,反应结束后测反应液荧光信号。
进一步的,所述应用方法还包括:反应结束后反应液中加入dnaseⅰ内切酶。
进一步的,所述金纳米簇修饰的适配体的制备方法如下:
将l-脯氨酸加入到煮沸的haucl4·3h2o溶液中搅拌5-20min后,将反应混合液冷却至室温得到auncs,然后加入edc与nhs活化羧基基团,之后将其与5’端或者3’端经修饰物修饰的适配体在25-37℃条件下震荡孵育10h以上,得到金纳米簇修饰的适配体。
进一步的,所述适配体核苷酸序列如seqidno.1所示。
一种棒曲霉毒素检测试剂盒,包括所述特异性识别棒曲霉毒素的优化适配体、二氧化锰纳米片与dnaseⅰ内切酶。
进一步的,所述优化适配体的5’端或者3’端经修饰物修饰。
本发明方法以棒曲霉毒素为靶标,在selex筛选获得的原始适配体序列(序列见seqidno.2)的基础上,根据二级结构进行序列剪裁,获得一条可与棒曲霉毒素以高亲和性、高特异性结合的适配体p-30。在此基础上,我们设计出一种新型的棒曲霉毒素检测方法,该方法基于“金纳米簇(auncs)-二氧化锰纳米片(mno2nfs)的荧光共振能量转移”原理,当棒曲霉毒素存在时,由于auncs荧光修饰的适配体p-30与棒曲霉毒素结合,使得p-30从mno2nfs表面解离,auncs荧光恢复。为了进一步增强荧光信号,利用dnaseⅰ对核苷酸链的酶切作用,对体系的荧光信号进行放大。
本发明有益的技术效果在于:
(1)与抗体相比,适配体具有可在体外筛选、筛选周期短、合成方便、易标记各种功能基团和报告分子、性质稳定可长期保存使用等优势;
(2)该适配体序列是基于原始序列的二级结构,通过剪裁获得的亲和力和特异性均较强的适配体序列,能够特异性识别棒曲霉毒素;
(3)与筛选获得的原始棒曲霉毒素适配体相比,该适配体序列具有更高的亲和力、良好的特异性以及较低的合成成本,能够更灵敏高效地检测出食品样品中的棒曲霉毒素。
附图说明
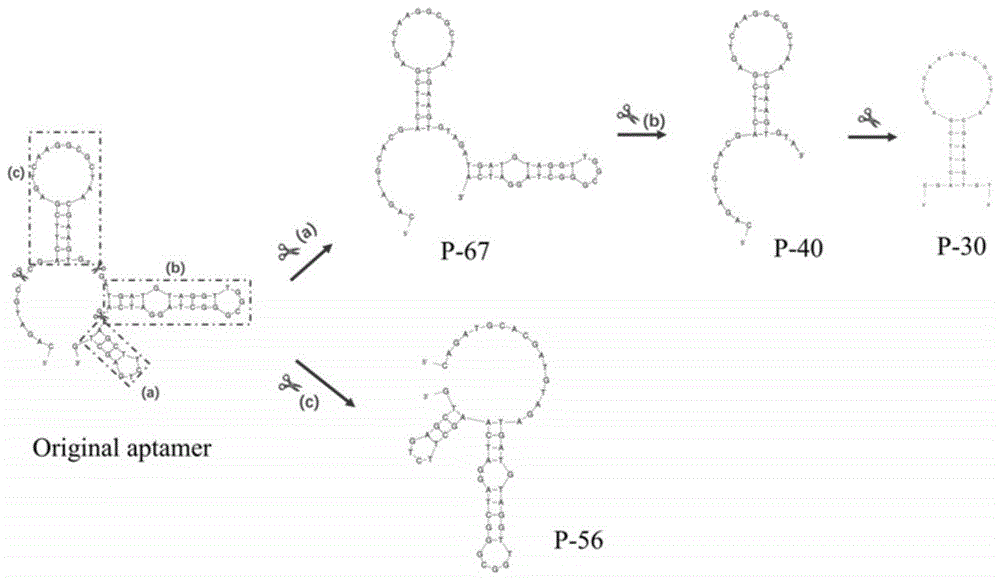
图1是本发明基于二级结构的适配体序列截短优化流程图;
图2是本发明mfold拟合的适配体原始序列的二级结构(a);原始序列亲和力曲线(b);
图3是本发明mfold拟合的适配体截短序列p-30的二级结构(a);截短序列p-30亲和力曲线(b);
图4是本发明实施例2中适配体p-30对棒曲酶毒素的特异性测试试验结果;
图5是本发明基于金纳米簇-二氧化锰纳米片的荧光法检测棒曲霉毒素的原理图;
图6是本发明实施例4棒曲霉毒素检测标准曲线图。
具体实施方式
下面结合附图和实施例,对本发明进行具体描述。
实施例1棒曲霉毒素适配体序列的定向剪裁
棒曲霉毒素原始适配体(图2)是通过selex筛选获得的棒曲霉毒素特异性适配体,使用sybrgreenⅰ(sg)荧光染料法测得与靶标结合的kd值为123.60±30.35nm,特异性良好,其序列中含有80个碱基,包括两端引物区40个碱基和中间随机区40个碱基。其中,sg荧光法测定kd值的具体操作为:将适配体与sg(1×)混合,95℃变性10min,冷却至室温20min。然后将适配体与棒曲霉毒素在25℃下孵育结合1h。孵育结束后,各取150μl体系置于酶标仪中测定荧光强度值。空白组不加棒曲霉毒素(靶标)。通过荧光强度的大小反映适配体与靶标结合力的强弱。运用graphpadprism软件做出亲和力曲线并计算kd值。
根据图1所示的截短流程图,截去茎环结构(a)得到截短序列p-67,其中,kd值为110.70±67.39nm,略低于原始序列。然后截去(b)得到p-40的kd值陡降,说明茎环结构(b)会造成空间位阻。因此,得出关键结合域应该位于最大的茎环结构(c)。保留(a)(b)结构并单独截去(c)后,所得序列p-56对靶标无亲和力,说明结构(c)在适配体和靶标结合过程中起着至关重要作用(图3)。此外,移去结构(c)两端的四个游离碱基所得的p-26亲和力略有下降,从而确定最优的截短序列为p-30。p-30的kd值为52.95±19.27nm,较原始序列的亲和力有所提高。
表1适配体亲和力数据
实施例2特异性试验
(1)材料制备:
所有玻璃器皿均用王水(hno3/hcl=1:3,v/v)浸泡,并用超纯水清洗。
auncs的合成:将15ml的l-脯氨酸(4m)快速添加到haucl4·3h2o的沸腾溶液(15ml,6mm)中。剧烈搅拌10min后,除去热源,并将混合物冷却至室温,所得产物即为auncs。
对auncs表面的羧基进行活化以连接氨基修饰的适配体:将40μl的edc(0.4m)和nhs(0.2m)加入到100μl的auncs溶液中,37℃下孵育1h。随后添加5’nh2修饰的适体(10μl,10m),并补充pbs至总体积250μl,混合物在37℃、200rpm条件下孵育10h以上。
mno2nfs的合成:将1mlbsa(10mg/ml)加入醋酸锰溶液(100ml,2×10-3m)中,室温孵育1h,用1mnaoh调节ph至7.4。反应7h后,mno2nfs产物在6000rpm下离心5min,沉淀洗3次后用超纯水重悬,并于4℃保存。
(2)特异性检测:
将100nm适配体与sg(1×)混合,95℃变性后冷却至室温。取5ng/ml的棒曲霉毒素与50ng/ml其他的常见毒素(ota,zen,afb1,t-2,don),在25℃下孵育1h。反应结束后,测定每种毒素体系的荧光强度值。如图4所示,只有棒曲霉毒素的荧光信号有明显响应,说明所优化的适配体序列p-30对棒曲霉毒素的特异性良好。
实施例3棒曲霉毒素的检测
(1)材料制备:所有玻璃器皿均用王水(hno3/hcl=1:3,v/v)浸泡,并用超纯水清洗。
auncs的合成:将15ml的l-脯氨酸(4m)快速添加到haucl4·3h2o的沸腾溶液(15ml,6mm)中。剧烈搅拌10min后,除去热源,并将混合物冷却至室温,所得产物即为auncs。
对auncs表面的羧基进行活化以连接氨基修饰的适配体。将40μl的edc(0.4m)和nhs(0.2m)加入到100μl的auncs溶液中,37℃下孵育1h。随后添加5’nh2修饰的适体(10μl,10m),并补充pbs至总体积250μl,混合物在37℃、200rpm条件下孵育12h。
mno2nfs的合成:将1mlbsa(10mg/ml)加入醋酸锰溶液(100ml,2×10-3m)中,室温孵育1h,用1mnaoh调节ph至7.4。反应7h后,mno2nfs产物在6000rpm下离心5min,沉淀洗3次后用超纯水重悬,并于4℃保存。
(2)标准曲线的制备:检测原理图如说明书附图5所示,检测体系包括auncs、mno2nfs、dnaseⅰ和auncs修饰的p-30。当棒曲霉毒素存在时,由于p-30与棒曲霉毒素的结合,使得p-30从mno2nfs表面解离,auncs的荧光恢复。溶液中的dnaseⅰ水解适配体链,使得棒曲霉毒素游离而与体系中其他的适配体结合,溶液中的荧光信号进一步增强。首先,auncs修饰的适配体被mno2nfs淬灭10-20min,然后分别选取1-100ng/ml范围内的10个浓度的棒曲霉毒素,加到auncs-mno2nfs的体系中反应1h以上,最后加入dnaseⅰ酶反应10-30min,测定各体系荧光最终恢复值,以棒曲霉毒素浓度为横坐标、荧光强度值为纵坐标,绘制棒曲霉毒素的标准曲线(图6)。说明本发明的检测方法,棒曲霉毒素浓度与反应液的荧光强度值呈现一定线性关系。
实施例4实际样品中棒曲霉毒素的检测
将不同浓度的棒曲霉素浓度(加标浓度为10、25和50ng/ml)添加到苹果汁中,然后将样品与乙酸乙酯溶液(1:1)涡旋混合,并在10000rpm下离心5min。将上清液经氮气干燥后,用pbs缓冲液溶解,得到预处理之后的果汁样品。然后,果汁样品与auncs修饰的适配体孵育1h以上,加入mno2nfs淬灭10-20min,最后加入dnaseⅰ酶反应10-30min,测定荧光最终恢复值。采用基于dnaseⅰ酶切扩增信号的auncs-mno2nfs荧光法测定实际样本中棒曲霉毒素的加标回收率,结果如表2所示。
表2果汁样品中棒曲霉毒素的检测及加标回收率
sequencelisting
<110>江南大学
<120>一种特异性识别棒曲霉毒素的优化适配体及其应用与一种试剂盒
<130>2
<160>2
<170>patentinversion3.3
<210>1
<211>30
<212>dna
<213>人工合成
<400>1
cgacttcgagtcaaggcgctaacgaagtgt30
<210>2
<211>80
<212>dna
<213>人工合成
<400>2
cagatgcacgacttcgagtcaaggcgctaacgaagtgtagatgatgtaggttggcgggct60
aggatcaagcttctgagctg80
- 还没有人留言评论。精彩留言会获得点赞!