一种检测肉瘤融合基因突变的引物组合物、试剂盒及应用的制作方法
本申请涉及基因检测
技术领域:
,特别是涉及一种检测肉瘤融合基因突变的引物组合物、试剂盒及应用。
背景技术:
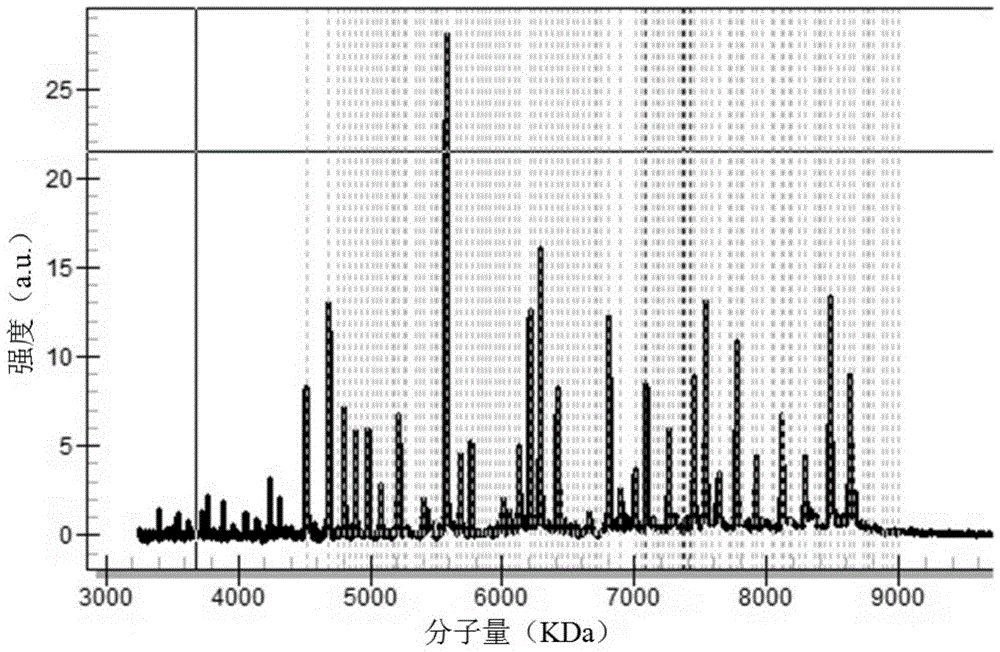
:软组织肉瘤是一种间叶性肿瘤,约60%发生于肢体部位,19%发生于躯干部位,其他部位还有腹部、颈部等。软组织肉瘤发病占成人肿瘤的1%,但是占儿童恶性肿瘤的15%,是儿童最常见的实体肿瘤之一。根据who的数据,目前肉瘤有100多种不同的亚型,表现出组织学的多样性;同时其生物学行为也是多变的,从惰性,高度侵袭到远处转移。易复发是软组织肉瘤的特征之一。目前,对软组织肉瘤的临床诊断和分型是结合临床、影像学、病理等多方面综合评估。组织病理学是软组织肉瘤诊断的金标准,随着对肉瘤的分子机制的深入研究发现,一些肉瘤亚型往往含有特定的融合基因突变,比如90%粘液性/圆细胞脂肪瘤中含有fus-ddit3的融合突变,5%为ewsr1-ddit3融合突变。针对这些融合突变的检测则可以辅助肉瘤的分子分型。对于融合基因的检测,目前主要通过荧光原位杂交技术(fluorescenceinsituhybridization,fish)和实时荧光定量pcr(realtimepcr,rt-pcr)。其中,fish作为融合基因检测的金标准具有灵敏度高、特异型好的优点;但是一次仅能检测一种类型的融合基因,操作流程复杂,成本高、通量低。rt-pcr虽然具有较高的检测通量,但是依然无法同时多种类型的基因,不利于通过融合基因的全面检测以辅助肉瘤的分子分型。此外还有相关研究通过高通量捕获测序技术对肉瘤融合基因及其伴侣基因进行捕获测序,该方法能够同时检测多个融合基因,但是该方法流程复杂、周期长且成本高,难以满足临床样本的检测需求。技术实现要素:本申请的目的是提供一种新的检测肉瘤融合基因突变的引物组合物、试剂盒及应用。本申请采用了以下技术方案:本申请的第一方面公开了一种检测肉瘤融合基因突变的引物组合物,该引物组合物包括检测38种融合突变的特异性引物对和相应的特异性单碱基延伸引物;其中,38种融合突变包括:bcor-ccnb3、bcor-itd、bcor-maml3、cic-foxo4、ewsr1-nfatc2、ewsr1-wt1、aspl-tfe3、ewsr1-erg、ewsr1-fli1、ntrk1重排、serpine1–fosb、fus-creb3l1、fus-creb3l2、ywhae-nutm2a、ewsr1-nfatc1、hey1-ncoa2、ss18-ssx1、ss18-ssx4、fus-ddit3、ewsr1-pbx1、ewsr1-znf444、ewsr1-ddit3、col1a1-pdgfb、wwtr1-camta1、yap1-tfe3、ewsr1-creb1、etv6-ntrk3、pax3-foxo1、pax3-ncoa1、pax7-foxo1、fus-atf1、ewsr1-atf1、atic-alk、cic-dux4、jazf1-suz12、nab2-stat6、myh9–usp6、plag1融合;检测38种融合突变的特异性引物对,其正向引物依序为seqidno.1至seqidno.38所示序列,其反向引物依序为seqidno.40至seqidno.77所示序列,相应的特异性单碱基延伸引物依序为seqidno.79至seqidno.116所示序列。需要说明的是,本申请中,检测38种融合突变的特异性引物对的正向引物依序为seqidno.1至seqidno.38所示序列,是指这38条正向引物依序为检测bcor-ccnb3、bcor-itd、bcor-maml3、cic-foxo4、ewsr1-nfatc2、ewsr1-wt1、aspl-tfe3、ewsr1-erg、ewsr1-fli1、ntrk1重排、serpine1–fosb、fus-creb3l1、fus-creb3l2、ywhae-nutm2a、ewsr1-nfatc1、hey1-ncoa2、ss18-ssx1、ss18-ssx4、fus-ddit3、ewsr1-pbx1、ewsr1-znf444、ewsr1-ddit3、col1a1-pdgfb、wwtr1-camta1、yap1-tfe3、ewsr1-creb1、etv6-ntrk3、pax3-foxo1、pax3-ncoa1、pax7-foxo1、fus-atf1、ewsr1-atf1、atic-alk、cic-dux4、jazf1-suz12、nab2-stat6、myh9–usp6、plag1融合的正向引物;反向引物依序为seqidno.40至seqidno.77所示序列,同样是指这38条反向引物依序为检测以上顺序的38种融合突变的反向引物;相应的特异性单碱基延伸引物依序为seqidno.79至seqidno.116所示序列,同样是指这38条特异性单碱基延伸引物依序为检测以上顺序的38种融合突变的特异性单碱基延伸引物,并且与相应的正向引物和反向引物对应。例如,对于融合突变bcor-ccnb3,其特异性引物对的正向引物为seqidno.1所示序列,反向引物为seqidno.40所示序列,相应的特异性单碱基延伸引物为seqidno.79所示序列;对于融合突变bcor-itd,正向引物为seqidno.2所示序列,反向引物为seqidno.41所示序列,特异性单碱基延伸引物为seqidno.80所示序列;对于融合突变bcor-maml3,正向引物为seqidno.3所示序列,反向引物为seqidno.42所示序列,特异性单碱基延伸引物为seqidno.81所示序列;以此类推。还需要说明的是,本申请引物组合物,使用时,特异性引物对以提取自待测肉瘤组织样本总rna的逆转录产物为模板进行pcr扩增;然后,采用特异性单碱基延伸引物对pcr扩增产物进行单碱基延伸;通过检测单碱基延伸产物在特定分子量大小位置的信号有无来判断对应的融合突变类型。本申请的引物组合物能对28种肉瘤亚型相对应的38种肉瘤融合基因突变进行检测,覆盖范围大,适用于初诊肉瘤及肉瘤诊断困难的患者的辅助诊断分型,为肉瘤的治疗方案制定和预后评估提供参考依据。本申请引物组合物,在rna层面进行融合突变检测,显著提高了检测准确性,避免了在dna的断裂重连过程中,融合断点的差异造成的漏检。本申请引物组合物,只需在1个反应孔中进行多重pcr扩增即可,操作简单方便,检测周期短,能更好满足临床样本检测的使用需求。本申请的一种实现方式中,本申请的引物组合物还包括检测内参基因的内参特异性引物对和相应的内参特异性单碱基延伸引物;内参基因为gapdh,内参特异性引物对的正向引物为seqidno.39所示序列,反向引物为seqidno.78所示序列,内参特异性单碱基延伸引物为seqidno.117所示序列。需要说明的是,内参基因的设置是为了验证检测过程或者样本是否存在错误;可以理解,除了本申请采用的gapdh内参基因以外,不排除还可以采用其他内参基因,至于具体的内参特异性引物对和相应的内参特异性单碱基延伸引物,只要能与本申请的检测38种融合突变的特异性引物对和相应的特异性单碱基延伸引物一起进行多重pcr扩增或单碱基延伸即可。本申请的一种实现方式中,正向引物和反向引物的上游各有8-14nt的标签序列。本申请的一种实现方式中,正向引物和反向引物的上游各有10nt的seqidno.118所示序列的标签序列。本申请的第二方面公开了一种检测肉瘤融合基因突变的试剂盒,该试剂盒中含有本申请的引物组合物。本申请的一种实现方式中,试剂盒中还含有逆转录引物,该逆转录引物用于对提取自肉瘤组织样本的总rna进行逆转录。本申请的一种实现方式中,逆转录引物包括6-8nt的随机引物和poly(t)引物。本申请的一种实现方式中,试剂盒中还含有逆转录酶。本申请的一种实现方式中,试剂盒中还含有虾碱性磷酸酶,用于消化dntps,使dntps脱去焦磷酸基团。本申请一种实现方式中,试剂盒还含有阳离子交换树脂,用于去除反应液中的金属离子。需要说明的是,本申请的试剂盒,其关键在于,其中含有本申请的引物组合物;至于其他组分,例如逆转录引物、逆转录酶、虾碱性磷酸酶、阳离子交换树脂等都可以根据需求选择加入试剂盒中,或者自行另外购买;在此不作具体限定。本申请的第三方面公开了本申请的引物组合物,或者本申请的试剂盒,在非诊断治疗目的的肉瘤亚型检测、肉瘤治疗药物研发或预后评估中的应用。本申请第四方面公开了一种肉瘤融合基因突变检测方法,包括以下步骤,对提取自待测肉瘤组织样本的总rna进行逆转录;采用权利要求1-4任一项所述的引物组合物中的所述检测38种融合突变的特异性引物对,对逆转录产物进行扩增;使用虾碱性磷酸酶对扩增产物进行处理,消化扩增反应中剩余的dntps,使得dntps脱去焦磷酸基团;采用权利要求1-4任一项所述的引物组合物中的所述相应的特异性单碱基延伸引物,对虾碱性磷酸酶的消化产物进行单碱基延伸,获得单碱基延伸产物;使用阳离子交换树脂对单碱基延伸产物进行纯化;采用基质辅助激光解吸电离飞行时间质谱技术检测纯化产物,通过单碱基延伸产物在特定分子量大小位置的信号来判断对应的肉瘤融合基因突变类型。需要说明的是,本申请的肉瘤融合基因突变检测方法,其直接目的在于获得肉瘤亚型的分类结果,该分类结果只是为肉瘤的治疗方案制定和预后评估提供中间参考依据,并不能直接用于疾病的诊断或治疗。本申请的有益效果在于:本申请检测肉瘤融合基因突变的引物组合物,能够对28种肉瘤亚型对应的38种肉瘤融合基因突变进行检测,覆盖范围大,适用于初诊及辅助分型。并且,本申请的引物组合物,是通过对提取的组织总rna进行融合突变转录本的定性检测来判断肉瘤亚型,通过在rna层面进行融合突变检测,显著提高了检测的准确性,避免了在dna的断裂重连过程中,融合断点的差异造成的漏检。本申请的引物组合物,只需要在1个反应中进行多重pcr扩增即可,操作简单方便,检测周期短,能够更好的满足临床样本检测的使用需求。附图说明图1和图2是本申请实施例一中阴性样本的检测结果图;图3至图11是本申请实施例二中9例已知融合突变类型的商业标准品及临床样本的检测结果图。具体实施方式现有的肉瘤融合基因突变检测方法,主要是通过高通量捕获测序技术,实现同时检测多个融合基因;但是,高通量捕获测序流程复杂、周期长、成本高,难以满足临床使用需求。有鉴于此,本申请创造性的提出,基质辅助激光解吸电离飞行时间质谱技术(maldi-tof-ms)对肉瘤组织中总rna的逆转录产物进行融合产物的检测。具体的,本申请选取了与包括脂肪母细胞瘤、肺泡性软组织肉瘤等28种肉瘤亚型相对应的38种融合突变类型,设计了特异性引物扩增富集在rna层面的融合突变转录本,经单碱基延伸及核酸质谱检测后对融合突变转录本进行定性检测,从而判断待测肉瘤组织样本中发生的是哪种融合突变,进而判断肉瘤的组织亚型,为肉瘤的治疗方案制定和预后评估提供参考依据。在以上研究的基础上,本申请创造性的研发了一种检测肉瘤融合基因突变的引物组合物,该引物组合物包括检测38种融合突变的特异性引物对和相应的特异性单碱基延伸引物;检测38种融合突变的特异性引物对,其正向引物依序为seqidno.1至seqidno.38所示序列,其反向引物依序为seqidno.40至seqidno.77所示序列,相应的特异性单碱基延伸引物依序为seqidno.79至seqidno.116所示序列。进一步的,本申请引物组合物还包括检测内参基因gapdh的内参特异性引物对和相应的内参特异性单碱基延伸引物;内参特异性引物对正向引物为seqidno.39所示序列,反向引物为seqidno.78所示序列,内参特异性单碱基延伸引物为seqidno.117所示序列。本申请使用基质辅助激光解吸电离飞行时间质谱技术对肉瘤组织中总rna的逆转录产物进行融合产物检测,该方法单次能检测覆盖的融合转录本产物可达40个,而常用的rt-pcr技术单次反应只能覆盖1-4个融合产物的检测,相比来说本申请覆盖更全面,单个融合产物的检测成本显著降低。而相比于使用基于高通量测序技术的rna测序方法来说,本申请是基于单碱基延伸的pcr扩增技术,流程简单、对rna的质量要求低,可适用于ffpe样本。本申请的引物组合、试剂盒及肉瘤融合基因突变检测方法,相对于现有的肉瘤融合基因突变检测,具有以下优点:(a)本申请针对38种融合突变类型,设计特异性引物扩增富集在rna层面的融合突变转录本,然后经单碱基延伸及核酸质谱检测,对融合突变转录本进行定性检测,判断待测肉瘤组织样本中发生的是哪种融合突变,进而判断肉瘤的组织亚型,为肉瘤的治疗方案制定和预后评估提供参考依据。(b)本申请针对检测肉瘤组织样本,通过对提取的组织总rna进行融合突变转录本的定性检测来判断肉瘤亚型;通过在rna层面进行融合突变检测能够显著提高检测的准确性,避免在dna的断裂重连过程中,融合断点的差异造成的漏检。(c)本申请覆盖了28种肉瘤亚型对应的38种融合突变,覆盖范围较大,适用于初诊肉瘤及肉瘤诊断困难的患者的辅助诊断分型。(d)本申请对所覆盖的28种肉瘤亚型对应的38种融合突变以及一个内参基因共设计了39组117条扩增和延伸引物,所有引物可用于1个反应孔中进行多重pcr扩增,操作简单,检测周期较短,能够更好的满足临床样本检测的使用需求。下面通过具体实施例对本申请作进一步详细说明。以下实施例仅对本申请进行进一步说明,不应理解为对本申请的限制。在以下的实施方式中,很多细节描述是为了使得本申请能被更好的理解。本领域技术人员可以毫不费力的认识到,其中部分特征在不同情况下可以省略,或可以由其他试剂盒、材料、方法所替代。在某些情况下,本申请相关的一些操作并没有在说明书中显示或描述,这是为了避免本申请的核心部分被过多的描述所淹没,而对于本领域技术人员而言,详细描述这些相关操作并不是必要的,根据说明书中的描述以及本领域的一般技术知识即可完整了解相关操作。以下实施例中,所用试剂或仪器未注明生产厂商者,均为可以通过市场购买获得的常规产品。本申请的编号例如,“seqidno.”、“第一方面”、“第二方面”、“第三方面”、“(a)”、“(b)”、“(c)”等,仅用于区分所描述的对象,不具有任何顺序或技术含义。实施例一本例基于单碱基延伸的多重pcr技术结合基质辅助激光解吸电离飞行时间质谱技术(maldi-tof-ms,后简称核酸质谱技术),实现肉瘤融合基因突变的高通量检测。本例选取了与包括脂肪母细胞瘤、肺泡性软组织肉瘤等28种肉瘤亚型相应的38种融合突变类型,设计特异性引物扩增富集在rna层面的融合突变转录本,经单碱基延伸及核酸质谱检测后对融合突变转录本进行定性检测,从而判断待测肉瘤组织样本中发生的是哪种融合突变,进而判断肉瘤的组织亚型。并针对内参基因设计相应的扩增引物和单碱基延伸引物。具体如下:(1)选取具有与肉瘤分子亚型对应相关性的融合突变类型,使用agena在线设计软件设计特异性扩增引物和延伸引物。38种融合突变检测的特异性引物对和相应的特异性单碱基延伸引物,以及内参基因的内参特异性引物对和相应的内参特异性单碱基延伸引物,见表1。肉瘤分子亚型与融合突变的对应关系见表2。其中每对特异性引物的扩增产物长度约为100-220bp;延伸引物的单碱基延伸产物的分子量为3500kda至9000kda。表1扩增引物和延伸引物序列表2肉瘤分子亚型与融合突变的对应关系表1所示的特异性引物对和内参特异性引物对中,正向引物和反向引物的上游各有8-14nt的标签序列,本例具体的标签长度为10nt,序列为seqidno.118所示序列。seqidno.118:5’-acgttggatg-3’。(2)使用健康人的外周血并提取总rna,取1μg总rna进行逆转录获得cdna。本例采用6-8nt的随机引物及poly(t)引物进行逆转录,逆转录的反应条件为,25℃5min、42℃30min、85℃5min。(3)使用seqidno.1至seqidno.78的特异性引物对1μl的逆转录产物进行扩增,对可能存在的融合突变转录本进行扩增富集。特异性扩增(pre-pcr)的反应体系如下:h2o(hplc级)1.8μl、10×pcrbuffer0.5μl、mgcl2(25mm)0.4μl、dntpmix(25mmeach)0.2μl、primermix(500nmeach)1μl、(5u/μl)0.1μl、逆转录产物1μl,总计5μl。特异性扩增反应条件为:94℃预变性2min;然后进入45个循环:94℃20sec、56℃30sec、72℃1min;循环结束后,72℃延伸1min。(4)特异性扩增完成后,使用虾碱性磷酸酶(sap)对扩增产物进行处理以消化扩增反应中剩余的dntp,使得dntp脱去焦磷酸基团;具体的,待扩增完成后打开扩增产物反应管,每管分别加入1.53μl的h2o,0.17μl的10×sapbuffer和0.3μl的sapenzyme(1.7u/μl),加入后吸打混匀,总体积为7μl。sap消化的反应条件为,37℃40min;85℃5min;最后4℃待机。(5)sap消化完成后,使用seqidno.79至seqidno.117的延伸引物对sap消化产物进行单碱基延伸,获得单碱基延伸产物;具体的,待sap消化完成后打开反应管,每管分别加入0.619μl的h2o,0.2μl的iplexbufferplus(10×),0.2μl的iplexterminationmix,0.94μl的iplexextendprimermix,0.041μl的iplexenzyme,总体积9μl,吸打混匀。单碱基延伸反应条件为,94℃预变性30sec;然后进入50个循环:94℃5sec、52℃5sec、80℃5min;循环结束后,72℃延伸3min。(6)使用阳离子交换树脂对单碱基延伸产物进行纯化,目的是去除反应液中的镁离子、锰离子、铁离子等;具体的,延伸反应结束后,打开反应管加入18μl的h2o并加入阳离子交换树脂,加入后封闭反应管置于静音混匀器上,室温,沿着长轴360°旋转,旋转产物板40-60min。混旋结束后产物4000rpm离心5min。(7)纯化产物可用于核酸质谱上机检测,通过在单碱基延伸产物在特定分子量大小位置的信号有无来判断对应的融合突变类型;本例具体的,按照agenamassarray点样及上机流程取上清液点样上机。上机结果如图1和图2所示,图中,横坐标为单碱基延伸产物的相对分子量大小,单位是kda;纵坐标是信号强度,信号强度越强,则说明目标产物拷贝数越高。图1和图2的结果显示,对于提取自健康人外周血的阴性样本,本例的引物组合物及其延伸引物的信号强度较为均匀,未见非特异扩增及非特异延伸产物的发生。实施例二本例采用实施例一相同的特异性引物对和相应的特异性单碱基延伸引物,以及相同的内参基因扩增引物和单碱基延伸引物,对9例已知融合突变类型的商业标准品及临床样本的rna进行肉瘤融合基因突变检测,待测样本的相关信息如表3所示。表39例已知融合突变类型的样本样本编号融合形式融合信息突变类型cbp20137rewsr1-wt1chr22:29683123/chr11:128675261融合cbp20128rewsr1-fli1chr11:118353210/chr9:20365742融合fa20061953fus-creb3l13.1%融合fa20061465fus-ddit37.5%融合fa20070162col1a1-pdgfb8.9%融合fa20061162ewsr1-creb114.1%融合fa20053065pax7-foxo15.3%融合fa20072357cic-dux45.5%融合fa20062522myh9–usp614.5%融合逆转录、pre-pcr、sap、ep、阳离子交换树脂、上机测试等都与实施例一相同。按照实施例一相同的方式点样上机,结果如图3至11所示。图3至11依序为样本cbp20137r、cbp20128r、fa20061953、fa20061465、fa20070162、fa20061162、fa20053065、fa20072357和fa20062522的测试结果。图3的结果显示,各样本在目标产物处均有产物信号,说明了对应融合突变的存在。以cbp20137r样本为例,样本中已知含有ewsr1-wt1融合突变,当使用本例引物组合物进行pre-pcr扩增时,位于ewsr1的上游特异性引物和wt1的下游特异性引物能扩增出目标融合产物,而其他融合引物组因缺乏目标模板则无法扩增。经过sap消化剩余dntps后,加入延伸引物进行单碱基延伸,延伸引物是与pre-pcr产物匹配的已知序列和分子量的单链dna,如图3中所示,根据碱基互补原则,延伸出的单碱基为c,延伸产物的分子量为5050kda,当在分子量是5050kda位置发现产物信号时,则可判定是目标产物信号,进而判断对应融合突变存在。对其他融合突变的判断也是如此,若在多个目标产物信号位置有信号检出,则说明存在多个不同的融合突变。若是在非目标产物预定分子量大小的地方产生信号,则判断为无意义的非特异扩增,不纳入结果判读。以上实施例的结果显示,本申请的肉瘤融合基因突变检测引物组合物,能对28种肉瘤亚型对应的38种肉瘤融合基因突变进行检测,通过在rna层面进行融合突变检测,能提高检测准确性,避免在dna的断裂重连过程中,融合断点的差异造成的漏检。本申请的引物组合物,只需要在1个反应中进行多重pcr扩增、进行单碱基延伸,最后进行核酸质谱检测即可,操作简单方便,检测周期短,能够更好的满足临床样本检测的使用需求。以上内容是结合具体的实施方式对本申请所作的进一步详细说明,不能认定本申请的具体实施只局限于这些说明。对于本申请所属
技术领域:
的普通技术人员来说,在不脱离本申请构思的前提下,还可以做出若干简单推演或替换。sequencelisting<110>深圳乐土生物科技有限公司<120>一种检测肉瘤融合基因突变的引物组合物、试剂盒及应用<130>21i31477<160>118<170>patentinversion3.3<210>1<211>22<212>dna<213>人工序列<400>1aggagctgttagatctggtgga22<210>2<211>20<212>dna<213>人工序列<400>2gacctggaagccttcaaccc20<210>3<211>19<212>dna<213>人工序列<400>3gtcctcccgcatatttcgc19<210>4<211>21<212>dna<213>人工序列<400>4aagaggaagaactccacggac21<210>5<211>22<212>dna<213>人工序列<400>5tgggtgtttatgggcaggagtc22<210>6<211>21<212>dna<213>人工序列<400>6ccaagtcaatatagccaacag21<210>7<211>20<212>dna<213>人工序列<400>7ccatggcggccccggcaggc20<210>8<211>26<212>dna<213>人工序列<400>8tcctacagccaagctccaagccaata26<210>9<211>24<212>dna<213>人工序列<400>9agcagttactctcagcagaacacc24<210>10<211>21<212>dna<213>人工序列<400>10ccaggtggagcagtataagaa21<210>11<211>20<212>dna<213>人工序列<400>11agagcgctgtcaagaagacc20<210>12<211>27<212>dna<213>人工序列<400>12ctgtggctgattacttcaagcagattg27<210>13<211>22<212>dna<213>人工序列<400>13tagcagttctcagagcagcagc22<210>14<211>26<212>dna<213>人工序列<400>14cacttatcatgcagttgttacgtgat26<210>15<211>23<212>dna<213>人工序列<400>15cccactgagactagtcaacctca23<210>16<211>23<212>dna<213>人工序列<400>16cgagatcctgcagatgaccgtgg23<210>17<211>20<212>dna<213>人工序列<400>17ggaccaccacagccacccca20<210>18<211>20<212>dna<213>人工序列<400>18ggaccaccacagccacccca20<210>19<211>20<212>dna<213>人工序列<400>19gcagaaccagtacaacagca20<210>20<211>22<212>dna<213>人工序列<400>20acagttatccccaggtacctgg22<210>21<211>22<212>dna<213>人工序列<400>21acagttatccccaggtacctgg22<210>22<211>25<212>dna<213>人工序列<400>22aatggcgtccacggattacagtacc25<210>23<211>19<212>dna<213>人工序列<400>23gccgagatggcatccctgg19<210>24<211>21<212>dna<213>人工序列<400>24atgtccgctcgcactcgtcgc21<210>25<211>20<212>dna<213>人工序列<400>25cctggaggcgctcttcaacg20<210>26<211>22<212>dna<213>人工序列<400>26tcctacagccaagctccaagtc22<210>27<211>20<212>dna<213>人工序列<400>27cattcttccaccctggaaac20<210>28<211>20<212>dna<213>人工序列<400>28gacagcagctctgcctactg20<210>29<211>20<212>dna<213>人工序列<400>29gattccttccaacccagaca20<210>30<211>19<212>dna<213>人工序列<400>30cctccaaccmcatgaaccc19<210>31<211>20<212>dna<213>人工序列<400>31agcagaaccagtacaacagc20<210>32<211>25<212>dna<213>人工序列<400>32caaggattaaatgacagtgtgactc25<210>33<211>24<212>dna<213>人工序列<400>33ccaagcttcccatcacagtgtacc24<210>34<211>18<212>dna<213>人工序列<400>34ggctcctctccctgtacc18<210>35<211>20<212>dna<213>人工序列<400>35agcagtggaagccttactcc20<210>36<211>21<212>dna<213>人工序列<400>36cttgtcctccttgaagggctc21<210>37<211>19<212>dna<213>人工序列<400>37agggcacggaaggctaagc19<210>38<211>23<212>dna<213>人工序列<400>38ttgttggacacttgggaactgcc23<210>39<211>22<212>dna<213>人工序列<400>39ccacccatggcaaattccatgg22<210>40<211>23<212>dna<213>人工序列<400>40gtggtttctccataatgtttggt23<210>41<211>21<212>dna<213>人工序列<400>41gtacatggtgggtccagcttg21<210>42<211>22<212>dna<213>人工序列<400>42cctgaaaccttctttgcccttg22<210>43<211>21<212>dna<213>人工序列<400>43gcttcttcacggtttcgagag21<210>44<211>22<212>dna<213>人工序列<400>44gctcaatgtcggcgtttctaag22<210>45<211>20<212>dna<213>人工序列<400>45caccaaatggcaatgggctt20<210>46<211>23<212>dna<213>人工序列<400>46cgtcatcgtctttgtagtcggac23<210>47<211>23<212>dna<213>人工序列<400>47cccaggtgatgcagctggagttg23<210>48<211>25<212>dna<213>人工序列<400>48ccaggatctgatacggatctggctg25<210>49<211>19<212>dna<213>人工序列<400>49gtgggttctcgatgatgtg19<210>50<211>18<212>dna<213>人工序列<400>50gttcccggcatgtcgtag18<210>51<211>26<212>dna<213>人工序列<400>51ccctgatgtcccctgtaatttgtgag26<210>52<211>21<212>dna<213>人工序列<400>52ctggaggggctgtgggtctga21<210>53<211>18<212>dna<213>人工序列<400>53gggcagagccgtgaacac18<210>54<211>20<212>dna<213>人工序列<400>54gacacggtcttccctgtgat20<210>55<211>24<212>dna<213>人工序列<400>55gcaccagttgggctttgcaatgtg24<210>56<211>20<212>dna<213>人工序列<400>56atgtttcccccttttgggtc20<210>57<211>22<212>dna<213>人工序列<400>57gtcttgttaatcttctccaagg22<210>58<211>19<212>dna<213>人工序列<400>58gtggcttcaataaatttgg19<210>59<211>24<212>dna<213>人工序列<400>59aacactgccagggccttctgtagg24<210>60<211>23<212>dna<213>人工序列<400>60gaaggtcttgccacactcgcagc23<210>61<211>27<212>dna<213>人工序列<400>61tcatgcttaatacagattcaccattcg27<210>62<211>24<212>dna<213>人工序列<400>62atcaaaggagcggatcgagtggtc24<210>63<211>26<212>dna<213>人工序列<400>63gaactttgaccccgactgtttcctta26<210>64<211>21<212>dna<213>人工序列<400>64gagtgtggtggacaggtactg21<210>65<211>21<212>dna<213>人工序列<400>65gtaccccatcggtaccattgt21<210>66<211>20<212>dna<213>人工序列<400>66aagcccatcaacctctctca20<210>67<211>21<212>dna<213>人工序列<400>67caccctctggattgagcatcc21<210>68<211>20<212>dna<213>人工序列<400>68ataagcctggcaactgtgct20<210>69<211>22<212>dna<213>人工序列<400>69ccttcattctgcacacgaatga22<210>70<211>21<212>dna<213>人工序列<400>70ctccatctgtgcctggacttg21<210>71<211>20<212>dna<213>人工序列<400>71ctttctgtgaggagcctatg20<210>72<211>24<212>dna<213>人工序列<400>72gaggtcttgccagcaaagcagtag24<210>73<211>18<212>dna<213>人工序列<400>73caggggagtgcagaccag18<210>74<211>23<212>dna<213>人工序列<400>74gctatgagattccgagttcgaag23<210>75<211>21<212>dna<213>人工序列<400>75tttttctgggggcatcttgga21<210>76<211>20<212>dna<213>人工序列<400>76tgtggatgtgaactgcggtc20<210>77<211>20<212>dna<213>人工序列<400>77aaacctcgcctggctcccag20<210>78<211>24<212>dna<213>人工序列<400>78tctagacggcaggtcaggtccacc24<210>79<211>17<212>dna<213>人工序列<400>79gggctcctctgtagagt17<210>80<211>18<212>dna<213>人工序列<400>80tagatctggtggaattca18<210>81<211>20<212>dna<213>人工序列<400>81ggaaattgtcaccattgcag20<210>82<211>22<212>dna<213>人工序列<400>82ctcgcccaagcgcaagatgaga22<210>83<211>23<212>dna<213>人工序列<400>83aggagagaaccggagcatgagtg23<210>84<211>23<212>dna<213>人工序列<400>84agcagagttcaatctgacaggac23<210>85<211>25<212>dna<213>人工序列<400>85ctggccccgaacggccggcgccaca25<210>86<211>25<212>dna<213>人工序列<400>86cagagttcattccgacaggaccacc25<210>87<211>25<212>dna<213>人工序列<400>87agagtagctatggtcaacaaagcag25<210>88<211>25<212>dna<213>人工序列<400>88gccaagctggacaatgccaggcagt25<210>89<211>26<212>dna<213>人工序列<400>89ttcctgcagctcagcagccgccgcca26<210>90<211>26<212>dna<213>人工序列<400>90aaacaggacagcccatgattaatctg26<210>91<211>27<212>dna<213>人工序列<400>91gggagctacagccagcagcctagctat27<210>92<211>17<212>dna<213>人工序列<400>92accgggcgtgaccgcga17<210>93<211>28<212>dna<213>人工序列<400>93gttacaaccagcccagcctaggatatgg28<210>94<211>28<212>dna<213>人工序列<400>94aggagggaaaggttactttgacgcgcac28<210>95<211>28<212>dna<213>人工序列<400>95tgaccagggacagtatggaaattaccag28<210>96<211>29<212>dna<213>人工序列<400>96tgaccagggacagtatggaaattaccagc29<210>97<211>30<212>dna<213>人工序列<400>97ctatggccaagatcaatcctccatgagtag30<210>98<211>31<212>dna<213>人工序列<400>98ccaccatcttatcctcctaccagctactcct31<210>99<211>29<212>dna<213>人工序列<400>99tcttatcctcctaccagctactcctctac29<210>100<211>32<212>dna<213>人工序列<400>100aatgcatacaccgcccaacccactcaaggata32<210>101<211>31<212>dna<213>人工序列<400>101cggaccccccggaccccctggcctcggagga31<210>102<211>33<212>dna<213>人工序列<400>102agccccgcgcagcagcacgcgcacctccgccag33<210>103<211>34<212>dna<213>人工序列<400>103cagacggtgcccatgcggctgcggaagctgcccg34<210>104<211>34<212>dna<213>人工序列<400>104ctacgggcagcagagttcattccgacaggaccac34<210>105<211>34<212>dna<213>人工序列<400>105tgaagaagataactgtgtccagaggacccccagg34<210>106<211>34<212>dna<213>人工序列<400>106gcatggattttccagctatacagacagctttgtg34<210>107<211>35<212>dna<213>人工序列<400>107cccagcaccaggcatggattttccagctatacaga35<210>108<211>35<212>dna<213>人工序列<400>108cccaggtgatgagcatcctgagcaaccccagcgct35<210>109<211>32<212>dna<213>人工序列<400>109gtaactatggccaagatcaatcctccatgagt32<210>110<211>33<212>dna<213>人工序列<400>110ggttgttaagatgaacaagagaactgggcaacc33<210>111<211>34<212>dna<213>人工序列<400>111ggaaacggcagcagccgcaagcagcacaagcctc34<210>112<211>35<212>dna<213>人工序列<400>112tcccagccccgcagggggccctgaccccacctcac35<210>113<211>37<212>dna<213>人工序列<400>113tcttcgttccgcagcagcactccgacaggcagcgagt37<210>114<211>37<212>dna<213>人工序列<400>114gtgcaaacctaaagagaatgggagtgtgtcgtgtgag37<210>115<211>38<212>dna<213>人工序列<400>115cacccggccgccgcccgagcgctcgagaaagtcctctc38<210>116<211>36<212>dna<213>人工序列<400>116tccctttttgctttcttttacgaccacatgaaactt36<210>117<211>35<212>dna<213>人工序列<400>117cccatcaccatcttccaggagcgagatccctccaa35<210>118<211>10<212>dna<213>人工序列<400>118acgttggatg10当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1