癌细胞耐药的标志物、逆转癌细胞耐药的制剂组合及其应用
本发明涉及生物医药技术领域,尤其涉及癌细胞耐药的标志物、逆转癌细胞耐药的制剂组合及其应用。
背景技术:
卵巢癌细胞具有高度不稳定的基因组,在其不同的dna损伤修复通路中普遍存在遗传性或获得性基因突变。为了维持基因组的稳定性,生物进化形成了复杂的dna损伤修复反应机制以应对广泛存在的内源性或外源性基因毒性损伤。dna损伤修复反应机制由一系列相互联系的信号通路组成。对于dna单链断裂具有三种修复方式,即碱基切除修复、核酸切除修复和错配修复。而dna双链断裂则对细胞毒性较大,且难以进行修复。机体依赖同源重组修复和非同源末端连接修复这两条路径修补dna双链断裂。当某一dna损伤修复通路功能发生异常时,肿瘤细胞将转向借助其他的dna损伤修复通路以继续生存。
parp抑制剂(多腺苷二磷酸核糖聚合酶(parp)抑制剂是一类能够改善卵巢癌患者预后并应用于临床治疗的新型分子靶向药物)阻滞碱基切除修复通路,使dna单链断裂恶化为dna双链断裂。而在有同源重组修复缺陷(hrd)的卵巢癌细胞中,dna双链断裂无法得到有效修复。利用这一特点,parp抑制剂在hrd阳性的肿瘤细胞中使dna损伤积累增多,引起细胞毒性基因组不稳定,最终诱导产生合成致死效应,且在hrd阳性的肿瘤细胞中parp抑制剂的细胞毒性作用最为明显。基于此,针对hrd阳性的肿瘤患者使用parp抑制剂进行维持治疗,能有效清除手术后残存的癌细胞及术中未发现或无法摘除的微小转移灶,低毒有效地长期控制肿瘤复发和转移。parp抑制剂因而获得多项指南的许可,在铂敏感复发卵巢癌人群中,parp抑制剂作为维持治疗阶段的首选药物。
然而,对于hrd阴性的患者,其本身对parp抑制剂不敏感。且因同源重组修复功能的恢复、回复突变、药物外排或其他dna损伤修复反应机制涉及的基因发生突变等原因,在parp抑制剂的治疗过程中,部分患者难以避免的发展为获得性耐药。因此,克服此种耐药的方法值得进一步研究。
技术实现要素:
有鉴于此,本发明的目的是寻找parp抑制剂耐药的调控因子。
本发明实施例发现,c/ebpβ调控癌细胞的同源重组修复通路基因的表达,促使癌细胞出现parp抑制剂耐药,因此,检测和分析c/ebpβ的表达水平可以预测卵巢癌患者的parp抑制剂敏感性。
本发明第一方面,提供一种癌细胞耐药的标志物,所述标志物为癌细胞中的cebpb基因及其蛋白,cebpb基因及其蛋白调控癌细胞的dna损伤修复信号,所述cebpb基因表达水平与癌细胞parp抑制剂耐药性正相关。
上述技术方案中,所述dna损伤修复信号为包括作为同源重组修复通路基因的brca1、brip1、brit1和rad51,所述同源重组修复通路基因调控卵巢癌细胞的dna损伤修复效率。
上述技术方案中,所述cebpb基因表达调控所述同源重组修复通路基因的表达水平。
上述技术方案中,所述cebpb基因表达水平与卵巢癌细胞对parp抑制剂的敏感性负相关。
上述技术方案中,所述cebpb基因表达水平与卵巢癌细胞的铂敏感性负相关。
本发明第二方面提供所述的标志物作为抗癌药物筛选标记的用途。
本发明第三方面提供一种逆转癌细胞耐药的制剂组合,包括parp抑制剂和靶向c/ebpβ的治疗剂,所述治疗剂通过抑制c/ebpβ的表达、中和c/ebpβ的表达蛋白和所述治疗剂降低同源重组修复通路基因的表达水平中的至少一种方式实现靶向治疗。
上述技术方案中,所述治疗剂引起癌细胞由hrd阴性转变成hrd阳性。
上述技术方案中,所述治疗剂为直接靶向cebpb基因或者蛋白、或者靶向c/ebpβ下游信号的sirna、shrna或抗体,所述c/ebpβ下游信号优选自下述一种或多种:rictor、hifia、abcc3、g2e3、nipbl、neil3、rock1、多药耐药相关蛋白1mrp1/abccl以及brca1通路。
本发明第四方面提供所述的制剂组合在制备抗癌药物中的用途。
本发明创造性地发现,在卵巢癌细胞中,c/ebpβ介导parp抑制剂耐药,c/ebpβ是同源重组修复通路的关键调控因子,也是评估parp抑制剂反应性的标志物。通过对c/ebpβ靶向治疗可以诱导产生hrd,从而相应地恢复卵巢癌对parp抑制剂的敏感性。通过这一途径,有望实现在治疗过程中对肿瘤的parp抑制剂反应性进行动态监测并将parp抑制剂的适应群体拓宽至hrd阴性患者、或将hrd阴性患者逆转为hrd阳性患者,以便解除parp抑制剂的耐药。
附图说明
图1为本发明实施例1提供的cebpb基因与dna损伤修复信号的富集分析相关结果图;
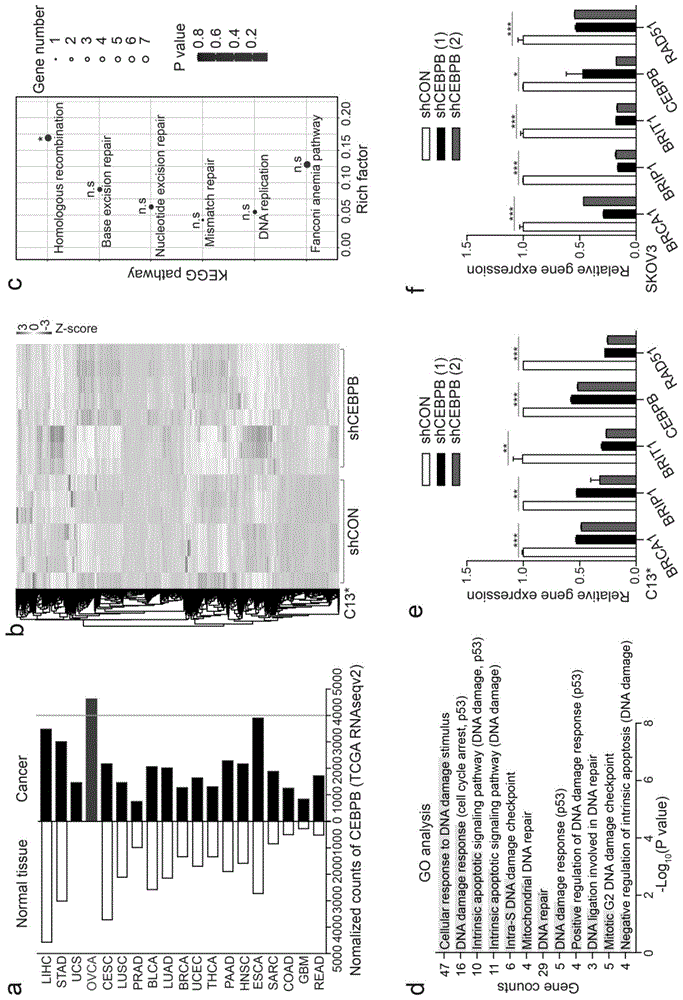
图1a为不同癌组织及癌旁组织中的cebpb基因在tcgarnaseqv2的分析结果图;图1b为cebpb沉默的c13*卵巢癌细胞系与正常细胞系的转录组测序差异表达基因热图;图1c为差异表达基因在dna损伤修复相关kegg通路富集分析的结果图;图1d为差异表达基因在dna损伤修复相关go通路分析的结果图;图1e为cebpb沉默的c13*卵巢癌细胞中相关基因的表达结果图(cebpb、brca1、brip1、brit1和rad51);图1f为cebpb沉默的卵巢癌细胞skov3中相关基因的表达结果图(cebpb、brca1、brip1、brit1和rad51)。
图2为本发明实施例2提供的cebpb表达水平与卵巢癌parp抑制剂敏感性关系的体外实验相关结果图;图2a为不同癌细胞中c/ebpβ和gapdh蛋白含量情况图;图2b为奥拉帕尼处理不同癌细胞系的ic50与相关蛋白水平的关系图;图2c为奥拉帕尼和dmso分别处理c13*、a2780、ov2008和skov3癌细胞的克隆形成结果图;图2d为在c13*细胞中通过转染慢病毒沉默c/ebpβ后,检测癌细胞中c/ebpβ和gapdh蛋白含量情况图;图2e为在c13*细胞中通过转染慢病毒沉默c/ebpβ后,检测癌细胞在奥拉帕尼处理下的生存率。
图3为本发明实施例2提供的cebpb表达水平与卵巢癌parp抑制剂敏感性关系的体内实验相关结果图;图3f为奥拉帕尼处理的接种c13*shcon和c13*shcebpb细胞的nod-scid裸鼠的动物实验结果图;图3g为c13*shcon和c13*shcebpb移植瘤模型在奥拉帕尼处理条件下的生存结果图;图3h为六例卵巢癌患者肿瘤组织的c/ebpβ表达的免疫切片图;图3i为六例卵巢癌患者肿瘤组织原代培养在奥拉帕尼处理下的生存率结果图;图3j为六例卵巢癌患者肿瘤组织原代培养对奥拉帕尼的ic50与免疫组化评分hscore定量的c/ebpβ表达水平间关系图。
图4为本发明实施例3涉及的c/ebpβ表达水平与奥拉帕尼、azd-2461或bmn673反应性(在未对cebpb进行干扰的癌细胞中)的相关结果图;图4a为a2780和ov2008中c/ebpβ和gapdh蛋白在不同浓度奥拉帕尼处理/未处理情况下的含量变化图;图4b为a2780和ov2008中c/ebpβ和gapdh蛋白在不同时间奥拉帕尼处理/未处理情况下的含量变化图;图4c为奥拉帕尼和dmso分别处理a2780和a2780(使用奥拉帕尼诱导a2780细胞构建形成的奥拉帕尼耐药细胞株)olar和skov3癌细胞的克隆形成结果图;图4d为不同浓度奥拉帕尼处理a2780和a2780olar细胞的生存率及反应性结果图;图4e为不同浓度azd-2461处理a2780和a2780olar细胞的生存率及反应性结果图;图4f为不同浓度bmn673处理a2780和a2780olar细胞的生存率及反应性结果图。
图5为本发明实施例3涉及的c/ebpβ表达水平与奥拉帕尼反应性(在对cebpb进行干扰的癌细胞中)的相关结果图;图5g为a2780细胞的及a2780olar细胞的c/ebpβ和gapdh蛋白含量图;图5h为a2780olar细胞中进行了si-cebpb干扰后的c/ebpβ和gapdh蛋白含量图;图5i为a2780olar细胞(分别进行了sinc、si-cebpb(1)和si-cebpb(2)三种方式的的rna干扰;si-nc是对照干扰,si-cebpb(1)和si-cebpb(2)分别为对cebpb两种不同序列的干扰,两个序列的应用是为了确定干扰效应是确实存在的)在奥拉帕尼处理下的生存率结果图;图5j为经奥拉帕尼处理/未处理的肿瘤组织c/ebpβ免疫切片图及免疫组化的hscore结果图。
图6为本发明实施例4提供的c/ebpβ靶向卵巢癌细胞的同源重组修复通路基因的预测和chip-qpcr检测的表达结果图;图6a为a2780转变为a2780olar产生的差异表达基因及c/ebpβ上调和下调的差异表达基因统计结果饼图;图6b为预测c/ebpβ特异性结合同源重组修复通路基因的结果图(其中p1-3为chip-qpcr引物设计位点);图6c为chip-qpcr检测c/ebpβ特异性靶向同源重组修复通路基因的结果图。
图7为本发明实施例4提供的c/ebpβ靶向卵巢癌细胞的同源重组修复通路基因表达的相关检测结果图;图7d为c13*癌细胞中的brca1、brit1、brip1和rad51启动子的荧光报告基因分析结果图;图7e为同源重组修复报告基因分析的流式细胞学检测结果,针对分别被dmso和奥拉帕尼处理的c13*癌细胞(shcon、shcebpb(1)和shcebpb(2));图7f为olaparib/dmso处理c13*癌细胞(shcon、shcebpb(1)和shcebpb(2))的彗星实验图及损伤dna含量结果图。
图8为本发明实施例5的相关结果图;图8a为癌细胞ov2008(con、cebpb)和癌细胞c13*(shcon、shcebpb(1)和shcebpb(2))中同源重组修复通路基因(brca1、brip1、brit1、rad51和gapdh(内参))表达图;图8b为癌细胞a2780olar(sinc、si-cebpb(1)和si-cebpb(2))中同源重组修复通路基因表达图;图8c为癌细胞ov2008、c13*、a2780和skov3中同源重组修复通路基因表达图;图8d为c13*癌细胞中brca1、brip1、brit1和rad51的免疫荧光分析结果图。
图9位本发明实施例5的另外相关结果图;图9e为经过奥拉帕尼处理的c13*shcon和c13*shcebpb移植瘤brca1、brip1、brit1和rad51切片图。
图10为本发明实施例6的si-rna拯救实验相关结果图;图10a为ov2008cebpb细胞中si-brca1\si-brip1\si-brit1\si-rad51的拯救实验表达图;图10b为ov2008cebpb细胞中si-brca1\si-brip1\si-brit1\si-rad51的同源重组修复报告基因分析检测结果图。
图11为本发明实施例6的si-rna拯救实验相关结果图(γh2ax分析);图11c为ov2008cebpb细胞中si-brca1\si-brip1\si-brit1
\si-rad51的γh2ax分析结果图。
图12为本发明实施例6的si-rna拯救实验相关结果图(细胞生存实验);图12d为奥拉帕尼处理ov2008cebpb细胞(sinc、si-brca1(1)、si-brca1(2)、si-brip1(1)、si-brip1(2)、si-brit1(1)、si-brit1(2)、si-rad51(1)、si-rad51(2))的细胞生存率结果图。
具体实施方式
为了使本发明的目的、技术方案及优点更加清楚明白,以下结合实施例对本发明进行进一步详细说明。应当理解,此处所描述的具体实施例仅仅用以解释本发明,并不用于限定本发明。
本发明实施例涉及的相关实验试剂、仪器及实验方法,若无特别说明,均为常规市售产品及仪器,实验方法亦为常规实验方法。
通过本发明研究结果提示ccaat增强子结合蛋白β(ccaat/enhancerbindingproteinβ,简称c/ebpβ,基因名:cebpb)是打破parp抑制剂获得性耐药的关键。c/ebpβ是亮氨酸拉链转录因子家族的成员之一,在机体生命进程的方方面面发挥重要功能,例如细胞增殖、分化、凋亡、癌基因诱导的衰老和肿瘤发生发展等。c/ebpβ还是brca1相关的dna损伤修复反应机制的重要决定因子,沉默c/ebpβ可以抑制brca1相关的dna损伤修复反应通路中多个基因的表达。
在卵巢癌细胞中,c/ebpβ介导parp抑制剂耐药,它是同源重组修复通路的关键调控因子,也是评估parp抑制剂反应性的标志物。c/ebpβ靶向治疗可以诱导产生hrd,从而相应地恢复卵巢癌对parp抑制剂的敏感性。通过这一途径,有望实现在治疗过程中对肿瘤的parp抑制剂反应性进行动态监测并将parp抑制剂的适应群体拓宽至hrd阴性人群。
既而,本发明实施例提供一种评价卵巢癌parp抑制剂耐药的标志物,该标志物为cebpb基因及其蛋白,cebpb基因及其蛋白调控卵巢癌细胞的dna损伤修复信号,cebpb基因表达水平与卵巢癌parp抑制剂耐药正相关。即,癌细胞中cebpb基因表达水平越高表明其parp抑制剂耐药性越强。因此,可通过检测和分析cebpb基因表达水平,以此来评估卵巢癌parp抑制剂耐药性。具体如通过rt-pcr检测该基因mrna表达量,以此分析卵巢癌细胞中该基因的表达水平,进而评估该癌细胞耐药情况。
具体的实施方式中,dna损伤修复信号为包括作为同源重组修复通路基因的brca1、brip1、brit1和rad51,这些同源重组修复通路基因调控卵巢癌细胞的dna损伤修复效率。其中,brca1基因位于人体细胞核的第17号染色体上,具有抑制恶性肿瘤发生的优良基因(称为“抑癌基因”),在损伤修复、细胞的正常生长方面有重要作用。brip1基因是一种抑癌基因,2006年来自英国的科学发现这一基因发生变异会增加患乳腺癌的风险。而最新的发现又表明了这一基因发生突变与卵巢癌的关联。brit1是新型细胞周期调控基因,参与细胞周期和肿瘤发生调控。rad51基因位于人体细胞核第15号染色体上,是dna损伤修复的重要组成部分。
具体的实施方式中,cebpb基因调控所述同源重组修复通路基因的表达,cebpb基因表达水平与癌细胞对parp抑制剂的敏感性负相关,cebpb基因表达水平与癌细胞的铂敏感性负相关。由此,通过分析癌细胞中cebpb基因表达情况,进而得知其同源重组修复通路相关基因表示情况,以此得知癌细胞对于parp抑制剂的敏感性及对于铂敏感性的关系,以为作为试剂抗癌药物筛选过程中提供一个药物筛选的参考。
另外,本发明实施例还提供一种逆转癌细胞耐药的制剂组合,包括parp抑制剂和靶向c/ebpβ的治疗剂,治疗剂通过抑制c/ebpβ的表达、中和c/ebpβ的表达蛋白和降低同源重组修复通路基因的表达水平中的至少一种方式实现靶向治疗。
具体的实施方式中,治疗剂引起癌细胞由hrd阴性转变成hrd阳性。由此,癌细胞hrd阴性的患者转变成为hrd阳性患者,这对于采用parp抑制剂、铂类及其他药物进行治疗具有积极意义。因此,这种制剂组合为制备抗癌药物提供了一种新型的可能。
具体的,治疗剂为直接靶向cebpb基因或者蛋白、或者靶向c/ebpβ下游信号的sirna、shrna或抗体,所述c/ebpβ下游信号优选自下述一种或多种:rictor、hifia、abcc3、g2e3、nipbl、neil3、rock1、多药耐药相关蛋白1mrp1/abccl以及brca1通路。
例如,可通过合成特异性c/ebpβ-sirna分子,并将其克隆至慢病毒表达载体pgcsil-gfp,再将重组c/ebpβ-sirna作为靶向药物感染至癌细胞中,如此能够下调癌细胞的c/ebpβ表达,进而抑制癌细胞对于parp抑制剂的耐药性。靶向c/ebpβ下游信号可采用相同方法获得对应的制剂或者药物,进而调节癌细胞耐药性。
实验方法
实验例1动物实验
从华阜康生物公司购买nod-scid裸鼠(雌性,四周龄)。将1×106个卵巢癌细胞(癌细胞ov2008(con、cebpb)和癌细胞c13*(shcon、shcebpb(1)和shcebpb(2))与细胞pbs、基质胶(matrigel,购自merck公司)混匀后,共0.2ml接种于裸鼠一侧腋下,检测肿瘤体积。当肿瘤体积大致达200mm3时开始给药。
其中,shcebpb(1)/shcebpb(2)为cebpb使用慢病毒度载体构建的沉默基因型,shcon为沉默基因对照型,参见附图。本实验例中使用从吉凯基因(中国)购买的慢病毒来沉默或过表达c/ebpβ,构建慢病毒载体时插入嘌呤霉素抗性基因,用于转染后添加嘌呤霉素进行筛选。稳定转染的细胞在表达目的基因的同时,还表达萤火虫荧光素酶,确定转染效率后细胞株方能用于后续实验。
同样地,si-cebpb(1)/si-cebpb(2)为cebpb使用sirna转染构建的基因型,si-nc为其干扰的基因对照型,参见附图;sirna干扰实验中使用的sirna购自锐博生物公司(中国),包括si-cebpb、si-brca1、si-brip1、si-brit1和si-rad51,使用lipofectaminetm3000(invitrogen,usa)辅助sirna转染。
将裸鼠随机分为四组(6只/组):
实验组:腹腔注射奥拉帕尼(olaparib,购自mce公司)(30mg/kg,溶于200μl无菌pbs),隔天给药,持续给药40天;
对照组:腹腔注射200μl含相同浓度的dmso的无菌pbs,隔天给药,持续给药40天。
到达观察终点后,给予萤火虫荧光素酶底物,随即进行肿瘤活体成像。实验完成后处死小鼠,剖检取出肿瘤用于后续实验。
另设相同的两组用于生存分析,生存终点定义为肿瘤组织体积达1500mm3或者观察期达90天。
实验例2细胞培养
使用rpmi-1640培养基(购自gibco公司)培养ov2008、c13*、a2780、nihovcar-3、ovcar-4和ovcar-8这6株卵巢癌细胞(atcc细胞库)。skov3细胞系生长在mccoy’s5a培养基(购自gibco公司)中。ov90细胞系使用的培养基是mcdb105和m199(购自sigma-aldrich公司)。caov3细胞系培养于dmem培养基(购自gibco公司)。
新鲜卵巢肿瘤组织(收集自华中科技大学同济医学院附属同济医院妇产科)在生物安全柜中分离为直径2-5mm的小块,随即使用1mg/ml的i型胶原酶分解组织(37℃恒温摇床,1-2小时)。使用无菌滤网过滤组织悬液,滤出物离心(800rpm,5分钟)后使用dmem重悬。将组织悬液均分至培养瓶,放入恒温培养箱进行原代培养。
实验例3免疫组化
将福尔马林固定和石蜡包埋过的肿瘤组织进行连续切片。石蜡切片在65-68℃加热2小时后,迅速转移至环保脱蜡液脱蜡,随即依次通过梯度酒精(100%、95%、90%、80%、75%)进行梯度补水。
组化pbs洗涤3次后,将切片置于抗原修复液中加热30min,自然冷却至室温。利用3%h2o2消耗内源性过氧化物酶,并在37℃下用5%bsa封闭60min。加入一抗4℃孵育过夜后,再与相应的hrp二抗进行反应,使用dab进行显色。随后用苏木素染核,并在显微镜下观察并进行免疫组织化学评分。
实验例4启动子荧光素酶报告基因实验
常规在六孔板中种适量待转染的细胞,待细胞贴壁且达到大约60%融合度时进行转染。每个孔大约转染2μg的萤火虫荧光素酶报告质粒(携带有目的基因(brca1、brip1、brit1和rad51)启动子,擎科生物公司),并使用海肾荧光素酶报告质粒prl-tk作为参照质粒(不携带目的基因启动子,维真生物公司)。使用转染试剂(lipofectaminetm3000,赛默飞通用型转染试剂)进行双转染,转染24小时后使用萤火虫荧光素酶报告基因检测试剂盒(dual-
实验例5westernblot检测蛋白表达
向待测细胞培养液中加入ripalysisbuffer(购自赛维尔公司)裂解,使用考马斯亮蓝溶液测定蛋白浓度。加入蛋白上样缓冲液混匀后,将蛋白样置于100℃煮沸5分钟。利用10%sds-page凝胶进行电泳,pvdf膜进行转膜,随后用含有5%bsa的tbst溶液室温封闭1小时。根据目的蛋白分子量的不同,将膜剪切为若干条,分别与一抗在4℃孵育过夜。次日条带复温后用tbst充分洗涤3次,每次10min,室温与相应二抗孵育1小时。随后tbst漂洗3次,每次10min,即可用ecl显影液显色。bio-rad曝光仪用于采集摄片。
实验例6mrna提取和rt-pcr检测
使用trizol试剂提取rna,并使用nanodrop2000(默克公司)测定rna浓度。使用rt-qpcr专用预混液(hiscriptⅱqrtsupermixforqpcr(+gdnawiper),诺唯赞公司)进行逆转录聚合酶链式反应。使用通用型高灵敏度染料法定量pcr检测试剂盒(chamquniversalsybrqpcrmastermix,诺唯赞公司)进行rt-pcr检测。
使用的引物序列如下:
靶基因babam1,上下游引物依次为如seqidno.1、2所示;
靶基因brca1,上下游引物依次为如seqidno.3、4所示;
靶基因brip1,上下游引物依次为如seqidno.5、6所示;
靶基因brit1,上下游引物依次为如seqidno.7、8所示;
靶基因cdk12,上下游引物依次为如seqidno.9、10所示;
靶基因cebpb,上下游引物依次为如seqidno.11、12所示;
靶基因dna2,上下游引物依次为如seqidno.13、14所示;
靶基因fancb,上下游引物依次为如seqidno.15、16所示;
靶基因fancm,上下游引物依次为如seqidno.17、18所示;
靶基因gapdh,上下游引物依次为如seqidno.19、20所示;
靶基因pold4,上下游引物依次为如seqidno.21、22所示;
靶基因rad50,上下游引物依次为如seqidno.23、24所示;
靶基因rad51,上下游引物依次为如seqidno.25、26所示;
靶基因rbl1,上下游引物依次为如seqidno.27、28所示;
靶基因rev1,上下游引物依次为如seqidno.29、30所示;
靶基因rfc1,上下游引物依次为如seqidno.31、32所示;
靶基因usp1,上下游引物依次为如seqidno.33、34所示;
靶基因wrn,上下游引物依次为如seqidno.35、36所示;
靶基因xrcc2,上下游引物依次为如seqidno.37、38所示。
实验例7染色质免疫共沉淀
使用chip试剂盒(chip-ithigh
靶基因brca1-1,上下游引物依次为如seqidno.39、40所示;
靶基因brca1-2,上下游引物依次为如seqidno.41、42所示;
靶基因brca1-3,上下游引物依次为如seqidno.43、44所示;
靶基因brip1-1,上下游引物依次为如seqidno.45、46所示;
靶基因brip1-2,上下游引物依次为如seqidno.47、48所示;
靶基因brip1-3,上下游引物依次为如seqidno.49、50所示;
靶基因brit1-1,上下游引物依次为如seqidno.51、52所示;
靶基因brit1-2,上下游引物依次为如seqidno.53、54所示;
靶基因brit1-3,上下游引物依次为如seqidno.55、56所示;
靶基因rad51-1,上下游引物依次为如seqidno.57、58所示;
靶基因rad51-2,上下游引物依次为如seqidno.59、60所示;
靶基因rad51-3,上下游引物依次为如seqidno.61、62所示。
实验例8细胞活力检测
将待测细胞以5×103/孔的密度接种至96孔板,次日开始使用奥拉帕尼等parp抑制剂处理细胞。48小时后使用cck8试剂盒(cellcountingkit8,博士德公司)检测细胞活力。设定酶标仪参数,读取450nm波长的吸光度。
实验例9克隆形成
计数待测细胞密度后,以2×103/孔/2ml培养基接种至六孔板,接种细胞24小时后使用olaparib/二甲亚砜(dimethylsulfoxide,dmso)处理细胞。持续用药10天后,使用结晶紫染色,扫描六孔板并计数。
实验例10免疫荧光
将待测细胞接种于爬片,次日加入olaparib/dmso,处理48小时后开始实验。加入4%的多聚甲醛后,使用5%bsa封闭60分钟。滴加一抗孵育过夜后,滴加二抗于室温结合1小时,最后用dapi染核。于暗室内使用荧光显微镜依次观察并采集图像。
实施例11同源重组修复报告基因分析
使用质粒pcbascel和pdrgfp(维真生物公司)进行同源重组修复报告基因分析。将待测细胞接种至六孔板,次日使用lipofectaminetm3000双转染。转染24小时后使用olaparib/dmso处理细胞。转染72小时后收集细胞,使用流式细胞仪检测绿色荧光蛋白表达。
实验例12彗星实验
将待测细胞悬液接种至六孔板,次日加入olaparib/dmso处理细胞,48小时后使用彗星实验试剂盒(cometassaykit,trevigen公司)进行彗星实验。染色后使用荧光显微镜观察、采集图像。使用cometscore1.5software(彗星实验分析软件,tritek公司)分析实验结果。
实验例13rna-seq与生物信息学分析
在完成对待测细胞的rna质控之后,使用2μgrna进行建库。在测序系统(illuminanovaseq6000平台,illumina公司)进行150-bp双端测序,每个样本获得≥6gb数据。使用tophat软件将测序数据映射回参考基因组hg38,并用htseq计算每个基因对应的reads数。利用edger(exact)算法界定差异表达基因。使用r语言绘制差异表达基因热图、韦恩图和饼图。使用goseqr进行差异表达基因富集geneontology(go)分析。借助kobas3.0富集差异表达基因进行kyotoencyclopediaofgenesandgenomes(kegg)分析。
实验结果
实施例1c/ebpβ调节卵巢癌的dna损伤修复信号
本实施例对同源重组修复功能完整的卵巢癌细胞系c13*进行cebpb基因沉默,再进行rna测序,结果如图1所示。结果表明,沉默cebpb基因显著影响细胞的dna损伤修复信号通路,这一群受c/ebpβ影响的基因(主要包括brca1、brip1、brit1和rad51)显著富集于与dna损伤修复相关的go通路和kegg通路中,提示c/ebpβ调节卵巢癌的dna损伤修复信号。
实施例2c/ebpβ表达水平与卵巢癌parp抑制剂敏感性负相关
本发明实施例了探究c/ebpβ与卵巢癌parp抑制剂敏感性的关系,结果如图2、3所示。
如图2a-c表明,奥拉帕尼处理的不同癌细胞系,内源性的c/ebpβ表达水平与卵巢癌细胞系对olaparib耐受显著相关。对cebpb基因沉默,能够显著降低对应的c13*在奥拉帕尼处理下的生存率(如图2d-e)。
进一步通过动物实验表明,对于其癌细胞的进行cebpb基因沉默,能够增加小鼠的生存率(如图3f-g)。这表明,c/ebpβ对卵巢癌细胞olaparib反应性的影响在动物体同样存在。
另外,还通过另存的六例卵巢癌患者肿瘤组织的c/ebpβ表达情况进行分析(如图3h-j),结果同样表明c/ebpβ表达水平与卵巢癌olaparib敏感性在卵巢癌组织原代培养中同样呈负相关。
因此,卵巢癌的c/ebpβ表达水平与parp抑制剂敏感性呈负相关。
实施例3parp抑制剂治疗诱导卵巢癌的c/ebpβ表达水平升高
本实施例探究了parp抑制剂治疗诱导卵巢癌对于其c/ebpβ表达的影响,结果如图4、5所示。
如图4所示,通过使用奥拉帕尼、azd-2461或bmn673不同的parp抑制剂对a2780和a2780olar两种卵巢癌细胞进行处理,其中a2780olar细胞系为使用奥拉帕尼诱导a2780细胞构建形成的奥拉帕尼耐药细胞株。结果表明a2780olar是稳定的parp抑制剂耐药株。a2780olar细胞c/ebpβ表达水平比a2780细胞升高,并且olaparib以剂量依赖性和时间依赖性的方式诱导c/ebpβ表达上调,说明parp抑制剂能够诱导c/ebpβ表达水平上调。
如图5所示,进一步地,对a2780olar耐药细胞系cebpb基因表达进行干扰,同样使用奥拉帕尼进行处理,其生存率在进行基因干扰后有所下降;使用olaparib治疗前后配对的卵巢癌组织样本,通过免疫组织化学染色检测患者接受治疗前后c/ebpβ的表达情况,结果发现olaparib治疗诱导卵巢癌c/ebpβ表达水平上调。
上述结果均说明,parp抑制剂治疗诱使卵巢癌c/ebpβ表达上调。
实施例4c/ebpβ靶向卵巢癌细胞同源重组修复通路基因
本实施例为探究c/ebpβ影响卵巢癌parp抑制剂反应性的确切机制,研究了c/ebpβ靶向卵巢癌细胞同源重组修复通路基因,结果如图6、7所示。
如图6,本实施例对a2780转变为a2780olar产生的差异表达基因及c/ebpβ上调或下调的差异表达基因进行统计分析,统计结果表明c/ebpβ与卵巢癌细胞对parp抑制剂从敏感到耐药的转变密切相关。进而,通过chip-qpcr分析发现了c/ebpβ与卵巢癌细胞对parp抑制剂从敏感到耐药的转变密切相关的四个基因brca1、brip1、brit1和rad51。
而这四个基因为均为同源重组修复通路基因,启动子荧光素酶报告基因分析结果(如图7d)提示c/ebpβ直接靶向这四个基因。c/ebpβ蛋白作为转录因子,能够直接结合于brca1、brip1、brit1和rad51这四个同源重组修复通路基因,并调控这些基因的表达。同源重组修复报告基因分析和彗星实验(如图7e-f)的结果证实,c/ebpβ直接靶向四个同源重组修复通路基因影响细胞的dna损伤修复。
实施例5c/ebpβ调控同源重组修复通路基因表达
本实施例通过蛋白质免疫印迹检测c/ebpβ表达量对同源重组修复通路基因表达水平的影响,如图8、9所示。结果发现c/ebpβ可以调控上述四个同源重组修复通路基因的表达,从而影响细胞的同源重组修复能力。在动物体内,c/ebpβ对同源重组修复通路基因同样发挥着调控作用。
实施例6c/ebpβ通过同源重组修复通路介导卵巢癌细胞对parp抑制剂耐受
为拯救c/ebpβ高表达细胞对parp抑制剂的不敏感性,本实施例使用si-rna开展拯救实验(参见基于block-ittm的rna干扰序列设计与pcr验证,2020,第21期),针对的靶基因及其引物如实验例6、7所示(可于genesil网站查询),选择thermofisher公司的block-ittmrnaidesigner设计工具自行设计si-rna序列并进行合成,构建载体转染癌细胞,并通过实时荧光定量pcr检测基因表达水平,验证干扰效果,结果如图10-12所示。
通过si-rna基因敲低brca1、brip1、brit1和rad51的表达可显著降低ov2008cebpb细胞的同源重组修复效率,并相应的恢复细胞对olaparib的反应性。
以上所述,仅为本发明较佳的具体实施方式,但本发明的保护范围并不局限于此,任何熟悉本技术领域的技术人员在本发明揭露的技术范围内,可轻易想到的变化或替换,都应涵盖在本发明的保护范围之内。
序列表
<110>华中科技大学
<120>癌细胞耐药的标志物、逆转癌细胞耐药的制剂组合及其应用
<160>62
<170>siposequencelisting1.0
<210>1
<211>21
<212>dna
<213>artificialsequence
<400>1
cactcgctccaatcctgaagg21
<210>2
<211>21
<212>dna
<213>artificialsequence
<400>2
ctgaagtgttgaggctcccat21
<210>3
<211>21
<212>dna
<213>artificialsequence
<400>3
gaaaccgtgccaaaagacttc21
<210>4
<211>22
<212>dna
<213>artificialsequence
<400>4
ccaaggttagagagttggacac22
<210>5
<211>21
<212>dna
<213>artificialsequence
<400>5
cttacccgtcacagcttgcta21
<210>6
<211>23
<212>dna
<213>artificialsequence
<400>6
cactaagagattgttgccatgct23
<210>7
<211>23
<212>dna
<213>artificialsequence
<400>7
atgtagtggcctatgttgaagtg23
<210>8
<211>23
<212>dna
<213>artificialsequence
<400>8
ccacaagctgtgttgtaaatgtc23
<210>9
<211>21
<212>dna
<213>artificialsequence
<400>9
ctaacagcagagagcgtcacc21
<210>10
<211>22
<212>dna
<213>artificialsequence
<400>10
aaaggtttgataactgtgccca22
<210>11
<211>18
<212>dna
<213>artificialsequence
<400>11
tccaaaccaaccgcacat18
<210>12
<211>25
<212>dna
<213>artificialsequence
<400>12
gagaggggcagagggagaagcagag25
<210>13
<211>21
<212>dna
<213>artificialsequence
<400>13
agagctgtcctgagtgaaact21
<210>14
<211>21
<212>dna
<213>artificialsequence
<400>14
gaaacacctcatggagaaccg21
<210>15
<211>21
<212>dna
<213>artificialsequence
<400>15
atgaaggatggcctaagggtc21
<210>16
<211>22
<212>dna
<213>artificialsequence
<400>16
acacactaacaactttgccagt22
<210>17
<211>21
<212>dna
<213>artificialsequence
<400>17
tcgtgacggtggttacaacac21
<210>18
<211>21
<212>dna
<213>artificialsequence
<400>18
acaagacgaattgggctcttc21
<210>19
<211>22
<212>dna
<213>artificialsequence
<400>19
acaactttggtatcgtggaagg22
<210>20
<211>19
<212>dna
<213>artificialsequence
<400>20
gccatcacgccacagtttc19
<210>21
<211>27
<212>dna
<213>artificialsequence
<400>21
tatctatacattcccagcaagcacctc27
<210>22
<211>23
<212>dna
<213>artificialsequence
<400>22
gcccagccatcagtttcttacca23
<210>23
<211>20
<212>dna
<213>artificialsequence
<400>23
tttggttggacccaatgggg20
<210>24
<211>23
<212>dna
<213>artificialsequence
<400>24
caggagggaaatctccagtacaa23
<210>25
<211>22
<212>dna
<213>artificialsequence
<400>25
caacccatttcacggttagagc22
<210>26
<211>21
<212>dna
<213>artificialsequence
<400>26
ttctttggcgcataggcaaca21
<210>27
<211>23
<212>dna
<213>artificialsequence
<400>27
atgaagaaccaccaaagttacca23
<210>28
<211>25
<212>dna
<213>artificialsequence
<400>28
gcagcatagaagtaaatgataagag25
<210>29
<211>21
<212>dna
<213>artificialsequence
<400>29
gatggaggaagcgagctgaaa21
<210>30
<211>21
<212>dna
<213>artificialsequence
<400>30
ccttctgcatagcagcatctg21
<210>31
<211>21
<212>dna
<213>artificialsequence
<400>31
ttgaacgagatgaggccaagt21
<210>32
<211>23
<212>dna
<213>artificialsequence
<400>32
actatcacgacccatgacaagat23
<210>33
<211>21
<212>dna
<213>artificialsequence
<400>33
atgcctggtgtcatacctagt21
<210>34
<211>23
<212>dna
<213>artificialsequence
<400>34
cagtcccacaaatggtaacaagt23
<210>35
<211>20
<212>dna
<213>artificialsequence
<400>35
cacagcagcggaaatgtcct20
<210>36
<211>23
<212>dna
<213>artificialsequence
<400>36
gagcaatcactagcatcgtaact23
<210>37
<211>23
<212>dna
<213>artificialsequence
<400>37
aagaaccgaatacagcaggacag23
<210>38
<211>23
<212>dna
<213>artificialsequence
<400>38
tcaagggactcagctaggcaagg23
<210>39
<211>23
<212>dna
<213>artificialsequence
<400>39
gcaggcactttatggcaaactca23
<210>40
<211>23
<212>dna
<213>artificialsequence
<400>40
caatcagaggatgggagggacag23
<210>41
<211>23
<212>dna
<213>artificialsequence
<400>41
gcaggcactttatggcaaactca23
<210>42
<211>23
<212>dna
<213>artificialsequence
<400>42
ggtacaatcagaggatgggaggg23
<210>43
<211>24
<212>dna
<213>artificialsequence
<400>43
tctaaggaacactgtggcgaagac24
<210>44
<211>23
<212>dna
<213>artificialsequence
<400>44
tcgtaagaagaggtcccaatccc23
<210>45
<211>23
<212>dna
<213>artificialsequence
<400>45
agattgtagcccgatgtcacaga23
<210>46
<211>23
<212>dna
<213>artificialsequence
<400>46
cgtagaccagcggaaaggaagta23
<210>47
<211>23
<212>dna
<213>artificialsequence
<400>47
attgtagcccgatgtcacagagc23
<210>48
<211>23
<212>dna
<213>artificialsequence
<400>48
cgtagaccagcggaaaggaagta23
<210>49
<211>23
<212>dna
<213>artificialsequence
<400>49
agatagattgtagcccgatgtca23
<210>50
<211>23
<212>dna
<213>artificialsequence
<400>50
taaattacccagctttgcagtag23
<210>51
<211>24
<212>dna
<213>artificialsequence
<400>51
cggcagttgagtttctatgctaat24
<210>52
<211>23
<212>dna
<213>artificialsequence
<400>52
agagtcacctaaacgtccgatgc23
<210>53
<211>23
<212>dna
<213>artificialsequence
<400>53
tacaggcagaaactaaggcatcg23
<210>54
<211>25
<212>dna
<213>artificialsequence
<400>54
ctcacggtcaaactctacaagacac25
<210>55
<211>23
<212>dna
<213>artificialsequence
<400>55
agcggcagttgagtttctatgct23
<210>56
<211>23
<212>dna
<213>artificialsequence
<400>56
agagtcacctaaacgtccgatgc23
<210>57
<211>23
<212>dna
<213>artificialsequence
<400>57
ccgtggttagcctcgaactccta23
<210>58
<211>24
<212>dna
<213>artificialsequence
<400>58
tttacagactgccctcttcccttt24
<210>59
<211>24
<212>dna
<213>artificialsequence
<400>59
agatactgccgaaacaaaccacaa24
<210>60
<211>23
<212>dna
<213>artificialsequence
<400>60
ccacgactcccaagaggtaatgc23
<210>61
<211>24
<212>dna
<213>artificialsequence
<400>61
atactgccgaaacaaaccacaaga24
<210>62
<211>23
<212>dna
<213>artificialsequence
<400>62
ccacgactcccaagaggtaatgc23
- 还没有人留言评论。精彩留言会获得点赞!