一种N7-甲基转移酶缺陷型冠状病毒减毒疫苗株及其制备方法与应用
本发明属于生物技术领域,涉及一种n7-甲基转移酶缺陷型冠状病毒减毒疫苗株及其制备方法与应用。
背景技术:
冠状病毒是一类分布广泛、种类多样的包膜正链rna病毒,可以感染包括人在内的多种宿主,并引发不同程度的呼吸道、胃肠道、肝脏和神经系统疾病,对人类健康和生产活动具有重大威胁。自1931年起,人类陆续发现了多种造成动物疾病的冠状病毒,包括以禽类为宿主的传染性支气管炎病毒(ibv),以猪为宿主的猪传染性胃肠炎病毒(tgev)、猪血凝性脑脊髓炎病毒(phev)、猪流行性腹泻病毒(pedv)、猪呼吸道冠状病毒(prcv)、猪德尔塔冠状病毒(pdcov)、猪急性腹泻综合征冠状病毒(sads-cov),以啮齿类动物为宿主的鼠肝炎病毒(mhv),以及牛冠状病毒(bcov)、猫传染性腹膜炎冠状病毒(fipv)、犬冠状病毒(ccov)等,它们引发的感染和疾病通常是致死性的。除了上述动物性冠状病毒外,几种常见的低致病性人冠状病毒,hcov-229e、hcov-oc43、hcov-nl63和hcov-hku1也在人群中广泛流行,但它们通常只引起类似于普通感冒的上呼吸道症状。然而,本世纪先后爆发了3种高致病性人冠状病毒,即严重急性呼吸综合征冠状病毒(sars-cov),中东呼吸系统综合征冠状病毒(mers-cov)和新型冠状病毒(2019-ncov,随后被命名为sars-cov-2)。sars-cov和sars-cov-2在进化上具有很近的亲缘关系,在发病症状上具有极高的相似性,早期均以干咳、发热、全身乏力等症候为主,晚期则发展至不同程度的呼吸衰竭和急性肺炎,患有基础病和免疫力低下的人群病死率较高。不同的是,sars-cov-2的潜伏期更长(约14天),存在无症状传染者,这使其具有超强的传播扩散能力,在短短2个月内造成了世界范围内的大流行。截止2021年5月,sars-cov-2已造成全球约1.5亿例感染,超320万人死亡。mers-cov感染引发的症候与sars相似,但感染后期可能会引起心肺功能障碍、糖尿病、肾衰竭等多种并发症,致死率高达40%,目前仍呈现局部散发的状态。
冠状病毒的频繁爆发引起了科学家们的重视,在他们的努力下,冠状病毒的进化历程、复制机制、致病机理等越来越多地被阐明。但遗憾的是,目前仍无冠状病毒的特效药物和通用型疫苗。传统的控制冠状病毒的手段主要包括物理隔离、生物安全控制等,预防部分动物性冠状病毒感染则主要通过接种减毒活疫苗的方法。然而,由于这些动物适用性减毒活疫苗主要是通过连续传代野生型株而得到,其减毒机理不明确,存在与野生型株重组且恢复毒力的风险,它们的滥用反而会加重冠状病毒的传播。在covid-19爆发之前,人用冠状病毒疫苗领域的研究仅停留在实验室阶段,没有任何批准可用的sars-cov和mers-cov疫苗。新型冠状病毒爆发后,为了控制covid-19的全球蔓延局势,包括灭活疫苗、rna疫苗、重组亚单位疫苗等在内的多种sars-cov-2疫苗被快速开发出来并进入临床试验阶段,部分疫苗已被紧急授权使用或附条件上市。不过由于缺乏冠状病毒疫苗成功的经验,再加上冠状病毒的快速突变和免疫逃逸能力,这些疫苗的安全性和有效性还需要更长期的观察和确认。因此,开发高效特异的抗冠状病毒药物和通用疫苗仍是世界范围内一项重大且艰巨的任务。
技术实现要素:
为了解决所述技术问题,本发明提供了一种n7-甲基转移酶缺陷型冠状病毒减毒疫苗株及其制备方法与应用,本发明基于“n7-甲基转移酶缺陷”策略正向设计的减毒活疫苗不仅安全、稳定,同时单剂量接种就能够诱导机体产生强有力的体液免疫和t细胞免疫应答,进而为机体提供保护作用;这种安全性好、免疫原性强、低成本的新型减毒活疫苗是一种理想的冠状病毒疫苗候选株,有望应用于通用冠状病毒疫苗的开发。
在本发明的第一方面,提供了一种n7-甲基转移酶缺陷型冠状病毒减毒疫苗株,通过将野生冠状病毒株中非结构蛋白nsp14中n7-甲基转移酶结构域内的极度保守的活性位点d和/或y定点突变成a而得到。
进一步地,所述野生冠状病毒株包括mhv、mers-cov和sars-cov-2;
对于所述mhv,非结构蛋白nsp14的第330位点由d突变为a,或/和第414位点由y突变为a;
对于所述mers-cov,非结构蛋白nsp14的第331位点由d突变为a,或/和第416位点由y突变为a;
对于所述sars-cov-2,非结构蛋白nsp14的第331位点由d突变为a,或/和第420位点由y突变为a。
在本发明的第二方面,一种n7-甲基转移酶缺陷型冠状病毒减毒疫苗,包括所述的n7-甲基转移酶缺陷型冠状病毒减毒疫苗株。
在本发明的第三方面,提供了一种n7-甲基转移酶缺陷型冠状病毒减毒疫苗株的制备方法,所述方法包括:
利用表达载体对宿主细胞进行转染,获得转染后宿主细胞;
对所述转染后宿主细胞进行培养,获得培包含编码nsp14点突变基因的重组病毒;
提取所述重组病毒dna并酶切,获得含nsp14点突变基因的全长cdna,经体外转录获得rna;
将所述rna转染宿主细胞,获得n7-甲基转移酶缺陷型冠状病毒减毒疫苗株;
其中,所述表达载体携带核酸分子,所述核酸分子编码突变体蛋白,并且所述突变体蛋白具有下列突变:非结构蛋白nsp14中n7-甲基转移酶结构域内的极度保守的活性位点d和/或y定点突变成a。
进一步地,所述突变体蛋白包括如下中的一种:
mhvnsp14突变体蛋白:野生型mhvnsp14蛋白的第330位的氨基酸由天冬氨酸d突变为丙氨酸a,或/和第414位的氨基酸由酪氨酸y突变为丙氨酸a;
mers-covnsp14突变体蛋白:野生型mers-covnsp14蛋白的第331位点由d突变为a,或/和第416位点由y突变为a;
sars-cov-2nsp14突变体蛋白:野生型sars-cov-2nsp14蛋白第420位的氨基酸由酪氨酸y突变为丙氨酸a,或/和第331位的氨基酸由天冬氨酸d突变为丙氨酸a;第420位的氨基酸由酪氨酸y突变为丙氨酸a。
在本发明的第四方面,提供了一种冠状病毒nsp14突变体蛋白,包括对野生冠状病毒株中非结构蛋白nsp14中n7-甲基转移酶结构域内的极度保守的活性位点d和/或a进行定点突变而得到。
进一步地,所述冠状病毒nsp14突变体蛋白包括如下中的一种:
mhvnsp14突变体蛋白:野生型mhvnsp14蛋白的第330位的氨基酸由天冬氨酸d突变为丙氨酸a,或/和第414位的氨基酸由酪氨酸y突变为丙氨酸a;
sars-cov-2nsp14突变体蛋白:野生型sars-cov-2nsp14蛋白第420位的氨基酸由酪氨酸y突变为丙氨酸a,或/和第331位的氨基酸由天冬氨酸d突变为丙氨酸a;第420位的氨基酸由酪氨酸y突变为丙氨酸a。
在本发明的第五方面,提供了一种编码所述突变体蛋白的核酸分子,所述核酸分子包括:
mhvnsp14突变体核酸分子:核苷酸序列如seqidno:3所示、或seqidno:4所示、或seqidno:5所示;
sars-cov-2nsp14突变体核酸分子:核苷酸序列seqidno:8所示、或seqidno:9所示、或seqidno:10所示。
在本发明的第六方面,提供了一种表达载体,包含有所述的核酸分子。
进一步地,所述表达载体包括原核表达载体和病毒载体中的一种。
在本发明的第七方面,提供了一种包含所述表达载体的工程化细胞系或重组菌。
本发明实施例中的一个或多个技术方案,至少具有如下技术效果或优点:
本发明提供的一种n7-甲基转移酶缺陷型冠状病毒减毒疫苗株及其制备方法与应用,将冠状病毒n7-甲基转移酶保守型关键酶活位点的突变引入mhv和sars-cov-2nsp14并测试了不同突变蛋白的甲基转移酶活性;考虑到sars-cov-2的生物安全风险,我们选取mhv作为模式冠状病毒,同时尝试利用反向遗传学系统拯救基于上述突变点的n7-mtase缺陷重组mhv病毒并评估其复制及稳定性,最终筛选到了能使mhvn7-mtase部分失活的点突变d330a和y414a,以及能使sars-cov-2n7-mtase部分失活的点突变d331a和y420a;并得到了重组n7-甲基转移酶缺陷型病毒mhv-nsp14-d330a和mhv-nsp14-y414a,随后在细胞和小鼠水平探索了它们的复制特征、安全性、减毒机制、致病能力、免疫原性及保护作用。结果表明基于“n7-甲基转移酶缺陷”策略正向设计的减毒活疫苗不仅安全、稳定,同时单剂量接种就能够诱导机体产生强有力的体液免疫和t细胞免疫应答,进而为机体提供保护作用。这种安全性好、免疫原性强、低成本的新型减毒活疫苗是一种理想的冠状病毒疫苗候选株,有望应用于通用冠状病毒疫苗的开发。
附图说明
为了更清楚地说明本发明实施例中的技术方案,下面将对实施例描述中所需要使用的附图作一简单地介绍,显而易见地,下面描述中的附图是本发明的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其它的附图。
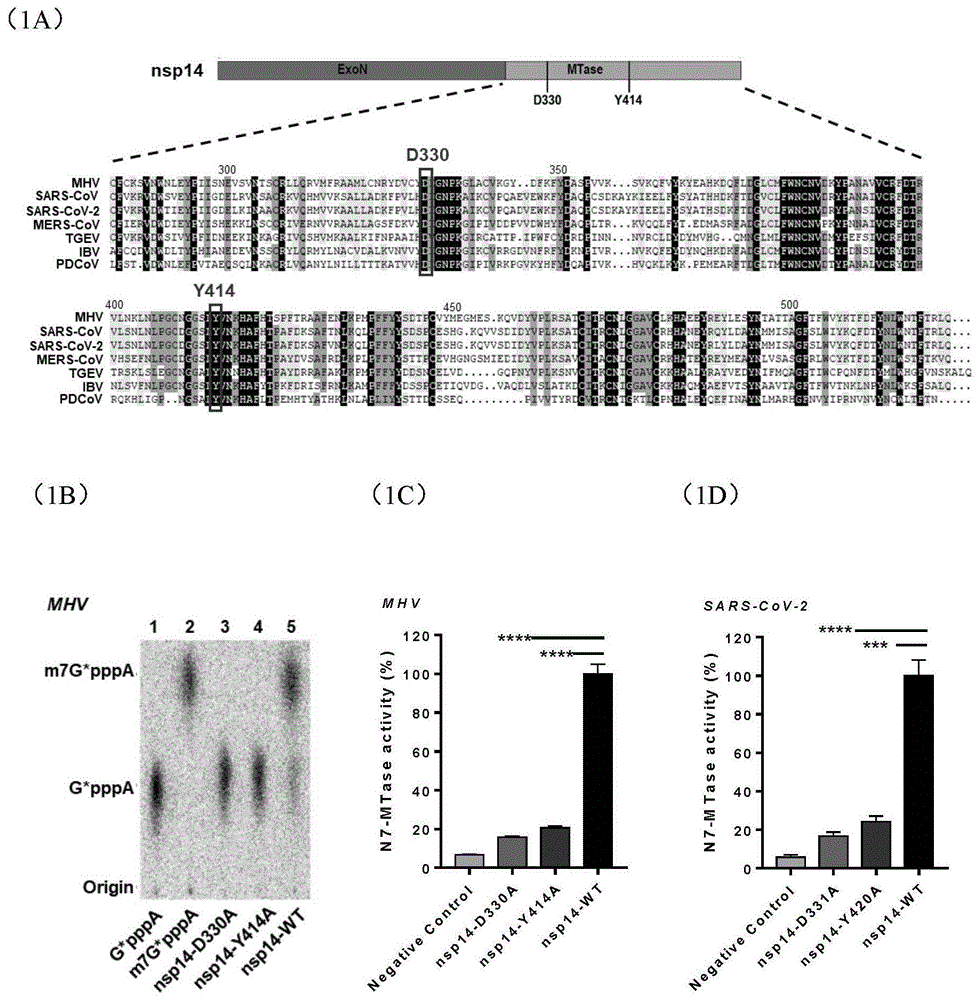
图1为实施例1的数据结果;其中,图1a为冠状病毒nsp14结构域示意图和n7-mtase氨基酸序列比对分析;图1b:基于32p同位素标记法检测mhvnsp14突变蛋白nsp14-d330a和nsp14-y414a的n7-甲基转移酶活性;图1c:基于3h同位素标记法检测mhvnsp14突变蛋白nsp14-d330a和nsp14-y414a的n7-甲基转移酶活性;图1d:基于3h同位素标记法检测sars-cov-2nsp14突变蛋白nsp14-d331a和nsp14-y420a的n7-甲基转移酶活性;
图2为实施例2的数据结果;其中,图2a:n7-mtase突变病毒mhv-nsp14-d330a(p10)和mhv-nsp14-y414a(p10)的噬斑形态;图2b:n7-mtase突变病毒mhv-nsp14-d330a(p10)和mhv-nsp14-y414a(p10)的遗传稳定性;图2c:n7-mtase突变病毒mhv-nsp14-d330a(p10)和mhv-nsp14-y414a(p10)在细胞系中的动力复制曲线;图2d:3h同位素标记法检测n7-mtase突变病毒的rna帽子甲基化状态;
图3为实施例3的数据结果;其中,图3a:n7-mtase突变病毒显著上调宿主细胞的i型干扰素表达水平;图3b:n7-mtase突变病毒在原代细胞bmdc中呈现减毒表型;图3c:包含不同帽子结构的rna诱导bmdc产生的ifn-β水平;图3d:筛选介导n7-mtase突变病毒减毒表型的宿主模式识别受体;
图4为实施例4的数据结果;其中,图4a:n7-mtase突变病毒感染c57bl/6小鼠的体重变化曲线;图4b:n7-mtase突变病毒感染c57bl/6小鼠的存活率曲线;图4c:n7-mtase突变病毒在c57bl/6小鼠肝脏内的复制情况;图4d:n7-mtase突变病毒感染c57bl/6小鼠后的肝组织病理变化;
图5为实施例5的数据结果;其中,图5a:n7-mtase突变病毒免疫c57bl/6小鼠诱导产生高效价中和抗体;图5b:n7-mtase突变病毒免疫c57bl/6小鼠诱导产生的中和抗体能够长期存在;图5c:n7-mtase突变病毒免疫c57bl/6小鼠可诱导强烈的t细胞免疫应答;
图6为实施例6的数据结果;其中,图6a:攻毒后c57bl/6小鼠的体重变化曲线;图6b:攻毒后c57bl/6小鼠的存活率曲线;图6c:攻毒后c57bl/6小鼠的肝脏病毒载量。
具体实施方式
下文将结合具体实施方式和实施例,具体阐述本发明,本发明的优点和各种效果将由此更加清楚地呈现。本领域技术人员应理解,这些具体实施方式和实施例是用于说明本发明,而非限制本发明。
在整个说明书中,除非另有特别说明,本文使用的术语应理解为如本领域中通常所使用的含义。因此,除非另有定义,本文使用的所有技术和科学术语具有与本发明所属领域技术人员的一般理解相同的含义。若存在矛盾,本说明书优先。
除非另有特别说明,本发明中用到的各种原材料、试剂、仪器和设备等,均可通过市场购买得到或者可通过现有方法制备得到。
本发明实施例提供的技术方案为解决上述技术问题,总体思路如下:
首先,本发明通过比对不同冠状病毒nsp14的编码序列,选取了一系列冠状病毒n7-甲基转移酶保守型关键酶活位点,构建了mhv和sars-cov-2的nsp14突变蛋白并测试了其甲基转移酶活性。同时尝试利用反向遗传学系统构建n7-mtase重组突变mhv病毒,最终筛选到了能使mhvn7-mtase部分失活的突变位点d330a和y414a和能使sars-cov-2n7-mtase部分失活的突变位点d331a和y420a,并成功拯救出了n7-mtase重组突变病毒mhv-nsp14-d330a和mhv-nsp14-y414a,它们遗传性状稳定且能够在细胞系中自主复制。在此基础上,测试了mhvn7-mtase突变病毒的帽子甲基化状态,发现n7-mtase缺陷导致病毒的rna帽子结构缺乏n7-甲基化修饰。
随后,本发明在原代细胞中探究了n7-mtase突变病毒的复制表型和减毒机制。发现n7-mtase突变病毒能够显著上调i型干扰素,同时突变病毒的复制能力显著下降。考虑到n7-mtase突变病毒的rna帽子结构缺失n7-甲基化修饰,我们检测了包含不同帽子结构的rna诱导原代bmdc产生i型干扰素的能力,结果发现缺乏n7-甲基化修饰的gpppg-rna诱导的ifn-β水平显著高于具有n7-甲基化修饰的m7gpppg-rna。更深层次的机制探索发现,这一现象跟宿主模式识别受体mda5和rig-i相关。由于冠状病毒丧失了n7-mtase活性,其rna的帽子结构不能被完整地甲基化,致使病毒rna被宿主mda5和rig-i所识别并激活了下游的抗病毒天然免疫,进而导致病毒被加速清除,即在感染bmdc后呈现出减毒表型。这一机制为靶向n7-甲基转移酶正向设计低毒性、高免疫原性的冠状病毒减毒活疫苗提供了理论依据。
基于此,本发明在c57bl/6小鼠中评估了n7-mtase突变病毒的致病性和安全性,发现突变病毒感染后2天即被机体完全清除,小鼠没有表现出明显疾病症状,证明n7-mtase突变病毒安全性良好。
随后,本发明在c57bl/6小鼠中评估了n7-mtase突变病毒的免疫原性,发现单剂量接种n7-mtase突变病毒能够快速诱导机体产生高滴度、长时期的中和抗体,同时能够诱导机体产生与野生型病毒相当的、甚至优于野生型病毒的辅助性t细胞免疫应答杀伤性t细胞免疫应答。
此外,本发明还评估了n7-mtase突变病毒对小鼠的保护效果,发现它们能够保护小鼠免受致死剂量的野生型病毒感染致死。
综上,基于“n7-甲基转移酶缺陷”策略正向设计安全性好、免疫原性强的冠状病毒减毒活疫苗具有极强的可行性和优越性,可应用到包括ibv、tgev、pedv、sads-cov等已存在或新发冠状病毒疫苗的设计和生产使用中。需要说明的是,本发明实施例3中对n7-mtase突变病毒的减毒机制进行了探索,验证了利用“n7-甲基转移酶缺陷”策略来制备冠状病毒减毒活疫苗为通用型策略。
具体地,基于“n7-甲基转移酶缺陷”策略正向设计的冠状病毒减毒活疫苗,其主要组成部分为n7-甲基转移酶突变株,所述突变株为在野生病毒载体的基础上对其非结构蛋白nsp14上进行突变而得;
所述野生病毒株包括mhv、mers-cov,sars-cov-2等病毒;
所述突变发生在nsp14n7-甲基转移酶结构域内极度保守的活性位点d和a,如:
对于所述mhv,非结构蛋白nsp14的第330位点由d突变为a,或/和第414位点由y突变为a;
对于mers-cov,其非结构蛋白nsp14的第331位点由d突变为a,或/和第416位点由y突变为a;
对于sars-cov-2,其非结构蛋白nsp14的第331位点由d突变为a,或/和第420位点由y突变为a;
所述n7-甲基转移酶突变株同样具有安全性好、免疫原性强的优点。
此外,为了方便研究,本发明还提供了一些载体工具:具体如实施例1中的原核表达载体pet30a-sars-cov-2nsp14-d331a和pet30a-sars-cov-2nsp14-y420a。
本发明实施例的mhvnsp14蛋白质序列如seqidno:1所示;mhvnsp14编码区核酸序列如seqidno:2所示;
本发明实施例的sars-cov-2nsp14蛋白质序列如seqidno:6所示;sars-cov-2nsp14编码区核酸序列如seqidno:7所示;
本发明实施例提供的一种编码所述突变体蛋白的核酸分子,所述核酸分子包括:
mhvnsp14突变体核酸分子:核苷酸序列如seqidno:3所示(mhvnsp14-d330a编码区核酸序列)、或seqidno:4所示(mhvnsp14-y414a编码区核酸序列)、或seqidno:5所示(mhvnsp14-d330a&y414a编码区核酸序列);
sars-cov-2nsp14突变体核酸分子:核苷酸序列seqidno:8所示(sars-cov-2nsp14-d331a编码区核酸序列)、或seqidno:9所示(sars-cov-2nsp14-y420a编码区核酸序列)、或seqidno:10所示(sars-cov-2nsp14-d331a&y420a编码区核酸序列)。
具体地,本发明还提供一种表达mhvnsp14突变体蛋白的原核表达载体及其制备方法,包括:以mhvnsp14野生型蛋白的基因片段为模板,依次采用如seqidno:11、14所示的引物对和如seqidno:12、13所示的引物对进行pcr,获得基因片段;将所述基因片段和原核表达载体pet30a分别通过bamhi和sali酶切后连接,获得原核表达载体pet30a-mhvnsp14-d330a和pet30a-mhvnsp14-y414a;
以sars-cov-2nsp14野生型蛋白的基因片段为模板,依次采用如seqidno:17、20所示的引物对和如seqidno:18、19所示的引物对进行pcr,获得基因片段;将所述基因片段和原核表达载体pet30a分别通过bamhi和xhoi酶切后连接,获得原核表达载体pet30a-sars-cov-2nsp14-d331a和pet30a-sars-cov-2nsp14-y420a。
本发明还提供一些生物材料包括表达载体、工程化细胞系或重组菌等均在本发明的保护范围之内。
此外,本发明还提供一种表达mhvnsp14突变体蛋白的重组冠状病毒的制备方法,所述制备方法包括:
以mhvnsp14野生型蛋白的基因片段为模板,通过搭桥法pcr、酶切、连接构建编码mhvnsp14突变体蛋白的t载体;
用含有被大肠杆菌鸟苷酸磷酸核糖转移酶(gpt)基因替代mhvnsp14基因片段的mhv-a59全长cdna的重组痘苗病毒感染细胞,后转染上述编码mhvnsp14突变体蛋白的t载体,经细胞内同源重组获得包含编码mhvnsp14点突变基因的重组痘苗病毒;
提取痘苗病毒dna并酶切,获得含mhvnsp14点突变基因的mhv-a59全长cdna,经体外转录得到rna;
将该rna转染宿主细胞,拯救得到编码mhvnsp14突变体蛋白的冠状病毒重组株mhv-nsp14-d330a和mhv-nsp14-y414a。
下面将结合实施例及实验数据对本申请的效果进行详细说明。如无特殊提及,以下方案中提到的分子克隆方法、蛋白表达纯化方法、细胞培养方法及各种检测方法等均为传统实验方法,可通过查询文献获得;用到的相关试剂可从对应试剂商处购买获得。
实施例1、mhvnsp14及其突变蛋白的n7-甲基转移酶活性检测
冠状病毒nsp14结构域示意图和n7-mtase氨基酸序列比对分析如图1a;所选取的冠状病毒依次为:β冠状病毒属mhv-a59(genbankaccession:ay700211),sars-cov(genbankaccession:nc_004718.3),sars-cov-2(genbankaccession:nc_045512.2),mers-cov(genbankaccession:nc_019843.3);α冠状病毒属tgev(genbankaccession:aj271965.2);γ冠状病毒属ibv(genbankaccession:nc_001451.1);δ冠状病毒属pdcov(genbankaccession:qfu80839.1)。从结果可以看出,不同种属冠状病毒的n7-mtase氨基酸序列比较保守,活性位点d330和y414极度保守。
1、构建mhvnsp14和sars-cov-2nsp14突变蛋白的原核表达载体
1)分别以实验室保存的mhv-a59nsp14野生型蛋白的基因片段(mhv-a59nsp14野生型蛋白的氨基酸序列如seqidno:1所示,核苷酸序列如seqidno:2所示)和sars-cov-2nsp14野生型蛋白的基因片段(sars-cov-2nsp14野生型蛋白的氨基酸序列如seqidno:6所示,核苷酸序列如seqidno:7所示)为模板,用搭桥法pcr扩增包含不同点突变蛋白的基因片段。引物序列如下:
表1
对于mhvnsp14:
第一轮pcr中,以mhv-a59nsp14野生型蛋白的基因片段为模板,依次采用如seqidno:11、14所示的引物对和如seqidno:12、13所示的引物对进行pcr,获得含d330a的nsp14上下游基因片段;同时采用如seqidno:11、16所示的引物对和如seqidno:12、15所示的引物对进行pcr,获得含y414a的nsp14上下游基因片段;
第二轮pcr中,以第一轮pcr的上下游产物为模板,用如seqidno:11、12所示的引物对进行pcr,分别获得含d330a和y414a的完整nsp14基因片段。
对于sars-cov-2nsp14:
第一轮pcr中,以sars-cov-2nsp14野生型蛋白的基因片段为模板,依次采用如seqidno:17、20所示的引物对和如seqidno:18、19所示的引物对进行pcr,获得含d331a的nsp14上下游基因片段;同时采用如seqidno:17、22所示的引物对和如seqidno:18、21所示的引物对进行pcr,获得含y420a的nsp14上下游基因片段;
第二轮pcr中,以第一轮pcr的上下游产物为模板,用如seqidno:17、18所示的引物对进行pcr,分别获得含d331a和y420a的完整nsp14基因片段。
2)所得含d330a或y414a突变的mhvnsp14基因片段和原核表达载体pet30a分别通过bamhi和sali酶切、t4连接酶连接,构建到原核表达载体pet30a上,获得原核表达载体pet30a-mhvnsp14-d330a和pet30a-mhvnsp14-y414a;所得含d331a或y420a突变的sars-cov-2nsp14基因片段和原核表达载体pet30a分别通过bamhi和xhoi酶切、t4连接酶连接,构建到原核表达载体pet30a上,获得原核表达载体pet30a-sars-cov-2nsp14-d331a和pet30a-sars-cov-2nsp14-y420a。
3)将上述载体分别转化感受态大肠杆菌jm109,涂布到相应抗性的lb平板。37℃倒置培养过夜(12~16h),挑取单菌落进行pcr验证。
4)菌落pcr正确的产物送测序公司测序。将测序正确的菌液进行扩增,快速质粒小提试剂盒提取质粒。所得质粒可长期冻存于-20℃。
2、原核表达纯化mhv-a59nsp14和sars-cov-2nsp14野生型及突变型蛋白
1)将上一步所得的突变蛋白原核表达质粒及野生型蛋白原核表达质粒分别转化bl21菌株感受态,涂布相应抗性平板,37℃倒置培养过夜。隔天上午挑选状态良好的菌落,接种至10mllb抗性试管中,37℃摇床220rpm震荡培养扩增。
2)至培养物吸光值(od600)在0.8~1.0之间时,将10ml菌液接种至1llb抗性培养基中,37℃摇床220rpm震荡培养。待吸光值(od600)到达0.6~0.8之间时,向菌液内加入终浓度为100~400μm的异丙基-β-d-1-硫代吡喃半乳糖苷(iptg),16℃220rpm震荡培养12h。离心收集菌体。
3)收集菌体后,用适量体积的bindingbuffer(含10mm咪唑)重悬,超声裂解破裂菌体。裂解后的菌液8000g离心30min,上清与ni填料重力柱孵育30min。随后washbuffer(含30mm咪唑)漂洗填料三次,适量elutionbuffer(含250mm咪唑)浸泡填料5min,收集流穿液。
4)sds-page凝胶电泳、考马斯亮蓝染色,检测目的蛋白的表达纯化情况。
3、mhvnsp14及其突变蛋白的n7-mtase活性检测(32p同位素标记法)
1)以mhv基因组5’端的22ntrna作为模板,设计底物并交由公司合成底物的双链dna模板,经体外转录得到未加帽修饰的单链rna。
2)利用痘病毒体外加帽和甲基化系统,以32p标记的gtp为底物,得到含有帽子核心结构但未n7甲基化的帽子前体rna(g*ppp-rna)。从反应体系中纯化得到帽子前体rna,定量后分装冻存于-80℃。
3)将待检测的nsp14野生型蛋白和突变蛋白(0.5μg)分别和帽子前体g*ppp-rna加入到甲基化反应体系中,37℃下反应1.5h。
4)核酸外切酶p1消化反应产物,随后将消化液在聚亚乙基亚胺平板上进行薄层层析。
5)台风仪扫描层析板,32p标记的帽子结构量可以被检测出来。结果展示于图1b。由图1b可知d330a和y414a突变导致mhvnsp14的n7-甲基化功能丧失。
4、mhvnsp14、sars-cov-2nsp14野生型及突变蛋白的n7-mtase活性检测(3h同位素标记法)
1)合成底物模板,方法如上。
2)利用痘病毒体外加帽和甲基化系统,得到含有帽子核心结构但未n7甲基化的帽子前体rna(gppp-rna)。从反应体系中纯化得到帽子前体rna,定量后分装冻存于-80℃。
3)将待检测的nsp14野生型蛋白和突变蛋白(0.5μg)分别和制备好的帽子前体rna(2μg)加入到甲基化反应体系(含50mmtris-hcl,6mmkcl,2mmdtt,1.25mmmgcl2,10urna酶抑制剂)中,最后向体系中加入1μci3h标记的sam底物,并在37℃下反应1.5h。
4)利用小体积deae-sephadex色谱柱分离出3h标记的产物,并通过液体闪烁检测3h掺入量。
5)将mhvnsp14的3h掺入量读值设置为100%,计算突变体及对照组的相对甲基化活性。mhvnsp14及其突变体的甲基化活性展示于图1c。由图1c可知,当nsp14-wt的n7-mtase活性为100%时,nsp14-d330a的活性仅为15%,nsp14-y414a的活性为20%,表明d330a和y414a突变可导致n7-mtase活性部分丧失。类似地,sars-cov-2nsp14及其突变体的甲基化活性展示于图1d,当nsp14-wt的n7-mtase活性为100%时,nsp14-d331a的活性仅为17%,nsp14-y420a的活性为24%,表明d331a和y420a突变可导致sars-cov-2n7-mtase活性部分丧失。
实施例2、n7-mtase突变病毒的构建与复制特征研究
1、n7-mtase突变病毒的构建
基于上述的体外n7-甲基转移酶活性结果,我们以鼠肝炎病毒mhv-59为模式病毒,利用重组痘苗病毒反向遗传学系统成功构建得到了2株n7-mtase突变病毒mhv-nsp14-d330a和mhv-nsp14-y414a,以及1株核酸外切酶突变病毒mhv-nsp14-d89a&e91a,同时构建了重组野生型病毒mhv-wt作为对照。
(1)根据实施例1中构建点突变的方法,将包含d330a、y414a、d89a&e91a突变的mhv-a59nsp14上下游基因片段克隆至pmd18-t载体。以mhvnsp14野生型蛋白的基因片段为模板,通过搭桥法pcr、酶切、连接构建,获得编码mhvnsp14突变体蛋白的pmd18-t载体。
第一轮pcr中,以包含mhvnsp14编码基因及其上下游基因片段的cdna为模板,依次采用如seqidno:23、14所示的引物对和如seqidno:13、24所示的引物对进行pcr,获得含d330a的nsp14上下游基因片段;同时采用如seqidno:23、16所示的引物对和如seqidno:15、24所示的引物对进行pcr,获得含y414a的nsp14上下游基因片段;采用如seqidno:23、26所示的引物对和如seqidno:25、24所示的引物对进行pcr,获得含d89a&e91a双突变的nsp14上下游基因片段;
第二轮pcr中,以第一轮pcr的上下游产物为模板,用如seqidno:23、24所示的引物对进行pcr,分别获得含d330a、y414a和d89a&e91a的完整nsp14基因片段。
用于构建pmd18-t载体及双突变d89a&e91a的引物补充如下:
表2
(2)用含有被大肠杆菌gpt基因替代mhvnsp14基因片段的mhv-a59株全长cdna的重组痘苗病毒感染cv-1细胞,病毒吸附1小时后转染上述编码mhvnsp14突变体蛋白的pmd18-t载体,以使得经细胞内同源重组获得包含编码mhvnsp14点突变基因的重组痘苗病毒。
转染后6小时弃掉转染液,更换新鲜dmem培养基。培养2~3天,观察到明显的细胞病变后收集细胞培养物。将细胞培养物接种至含gpt阳性筛选药物的cv-1细胞,筛选得到含gpt基因的重组痘苗病毒培养物;随后将培养物进一步接种至含gtp阴性筛选药物的d980r细胞,筛选得到经细胞内同源重组获得包含编码mhvnsp14点突变基因的重组痘苗病毒。
(3)bhk-21细胞内扩增上一步得到的重组痘苗病毒,待达到100%细胞病变后收集包含病毒的细胞沉淀,反复冻融三次。随后抽提病毒dna并定量。pcr、测序鉴定所得dna是否包含编码突变蛋白的基因片段。
(4)将上一步鉴定正确的重组痘苗病毒再次扩增,收集病毒沉淀并提取重组痘苗病毒dna。用eagi酶切dna,获得包含目的基因突变的mhv基因组全长cdna。回收、纯化酶切产物,将上述mhv基因组全长cdna进行体外转录(参考实施例1),获得mhv基因组全长rna。
(5)将上述rna转染bhk-21细胞,24小时后将转染细胞与4倍量的17-cl1细胞混合,随后继续培养并观察细胞病变。待出现明显的细胞病变后,收集细胞上清和沉淀,冻存备用。
(6)将上一步所得病毒液再次感染17-cl1细胞,并用噬斑法纯化获得的重组mhv病毒。经测序鉴定无误后冻存病毒,记为p0代病毒。
2、mhv病毒的扩增
(1)上述所有重组mhv病毒在neuro2a细胞中扩增。复苏neuro2a细胞,于含10%fbs的dmem培养基中传代。待细胞生长状态良好后,接种于15cm细胞培养皿。
(2)待细胞密度达到80~90%后,将病毒母液加入细胞,吸附1小时。随后弃去吸附液,更换新鲜培养基,37℃二氧化碳恒温箱培养16~20小时。
(3)细胞出现70%及以上的cpe后,收集细胞上清,离心去细胞碎片,所得即为扩增得到的病毒液。分装冻存于-80℃。
3、mhv病毒的滴度检测(噬斑法)
(1)上述mhv病毒在l2细胞中检测病毒滴度。复苏l2细胞,于含10%fbs的dmem培养基中传代。待细胞生长状态良好后,接种于24孔细胞培养板。
(2)细胞密度达到90%后,进行滴度测定。取出待测病毒液,分别进行10倍梯度稀释,稀释倍数覆盖101-108。取出细胞,弃去培养基,将稀释的病毒加入24孔板,吸附1小时。随后弃去吸附液,更换1%甲基纤维素半固体培养基,37℃二氧化碳恒温箱培养48小时左右。
(3)观察到有眼可见的噬斑后,取出培养板,多聚甲醛固定、结晶紫染色液染色4小时。随后冲洗细胞、噬斑计数,根据稀释倍数计算病毒滴度,计量单位为pfu/ml。
4、n7-mtase突变病毒的连续传代和稳定性检测
(1)n7-mtase突变病毒的连续传代在neuro2a细胞中进行。将neuro2a接种于6cm培养皿,待密度长至80%后,分别感染p0代突变病毒,生长16小时。同时准备同样的neuro2a细胞,用于下一代病毒的感染。
(2)收获p1代病毒,一份分装冻存,一份用于下一次传代。依次完成10次病毒传代,得到p10代突变病毒。n7-mtase突变病毒mhv-nsp14-d330a(p10)和mhv-nsp14-y414a(p10)的噬斑形态如图2a,可知n7-mtase突变病毒与野生型病毒的噬斑形态和大小无明显差别。
(3)取野生型病毒mhv-wt和p10代n7-mtase突变病毒上清,按照试剂商说明书提取病毒rna(tiangen)。
(4)将上一步所得的病毒rna进行逆转录,得到cdna作为模板,pcr扩增mhvnsp14片段。片段送公司进行一代测序。
(5)将野生型病毒的序列与p10代n7-mtase突变病毒的序列分别进行比对,确认引入的突变位点(d330a和y414a)是否稳定,以及是否有其他突变引入。包含突变位点的序列比对结果如图2b所示,可知突变病毒mhv-nsp14-d330a中,编码330位氨基酸的核苷酸由gac(d)转变为gca(a);突变病毒mhv-nsp14-y414a中,编码414位氨基酸的核苷酸由tat(y)转变为gca(a),序列单一,突变稳定存在。
5、n7-mtase突变病毒的复制曲线测定
(1)本发明分别在neuro2a细胞和l2细胞中测定了突变病毒的复制曲线。首先,分别将两种细胞接种于24孔板。
(2)待细胞密度达到80%左右后,对细胞进行计数,按感染复数(moi)为1加入病毒,吸附1小时。随后弃去吸附液,更换新鲜培养基,置于37℃恒温箱进行培养。感染后0~32小时(neuro2a细胞)或0~24小时(l2细胞),每隔4小时收取上清,用于病毒滴度检测。
(3)噬斑法测定上清样本中的病毒滴度,prism7软件绘制病毒生长曲线。生长曲线如图2c所示,可知n7-mtase突变病毒均能够在细胞系中完成自主复制,仅呈现微弱的减毒表型。
6.n7-mtase突变病毒的帽子甲基化状态检测
(1)将neuro2a接种于10cm培养皿,待密度长至80%后,分别感染野生型病毒mhv-wt和n7-mtase突变病毒,生长24小时。
(2)富集细胞中总polya-rna,操作按照试剂商说明书进行(invitrogen)。
(3)将富集得到的总polya-rna分别与nsp14蛋白或nsp10/nsp16蛋白一起加入甲基化反应体系(同实施例1),随后加入3h标记的sam底物,37℃下反应1.5h。用液闪仪检测3h掺入量(同实施例1中方法),所得结果见附图2d,可知n7-mtase突变病毒的n7-mtase是失活的,rna的帽子结构缺失n7-甲基化而非2’-o-甲基化。
实施例3、n7-mtase突变病毒的减毒机制探索
1.小鼠骨髓来源的树突状细胞(bmdcs)制备
取6~8周龄雄性c57bl/6小鼠,颈椎脱臼安乐死。无菌环境下,将小鼠后肢股骨和胫骨中的骨髓细胞分离至dmem培养基,过滤去除组织碎片,并裂解红细胞。pbs清洗细胞1遍,重悬细胞并计数。按1.5×107cell/皿的密度接种细胞于10cm皿,加入gm-csf(peprotech)细胞因子诱导7天,期间更换培养基1次。待细胞诱导成熟后,胰酶消化细胞,根据实验需求铺板。
2.bmdc的病毒感染
将成熟的bmdc铺至24孔板。待细胞贴壁后,加入mhv-wt或n7-mtase突变病毒进行感染(moi=0.1)。1小时后,弃病毒吸附液,更换新鲜培养基。感染后4、8、12、16、20小时收集细胞上清,分装为两份,一份用于检测ifn-β的量,一份用于检测病毒载量。ifn-β的检测根据试剂盒(biolegend)提供的说明书进行,病毒载量用噬斑法进行检测(同实施例2)。结果展示于图3a和图3b。由图3a可知,提示缺失n7-甲基化的帽子结构能够更早、更快地激活天然免疫信号通路,上调i型干扰素表达水平。由图3b可知,在免疫系统较为完善的原代细胞中,具有完整帽子结构的野生型病毒免疫逃逸能力较强,依然能够相对高效地进行复制;而缺失n7-甲基化帽子结构的冠状病毒免疫逃逸能力减弱,复制能力下降,呈现明显的减毒表型。
3.检测不同帽子结构rna诱导bmdc产生的ifn-β水平
(1)以mhv-a59基因组5’-端的核苷酸序列为模板,设计合成包含50kb核苷酸的dna片段。将dna片段用水溶解,用t7体外转录试剂盒(invitrogen)将其转录为单链rna(即为pppg-rna),酚氯仿抽提纯化rna备用。所得rna用痘病毒加帽和甲基化系统(neb)分别加工为gpppg-rna,m7gpppg-rna,m7gpppgm-rna。另外购买商品化polyi:c(mce)作为阳性对照刺激物。
(2)按照上述方法制备bmdc并铺板。用lipo3000(invitrogen)将上述rna和阳性对照刺激物转染细胞。转染后20小时,用trizol(life)裂解细胞,提取细胞总rna,并随机逆转录得到cdna(takara)。以cdna为模板,用q-pcr定量法检测ifn-β的mrna水平。
4.筛选介导n7-mtase突变病毒减毒表型的宿主模式识别受体
(1)按照上述方法制备bmdc并铺板。细胞内识别rna的模式识别受体主要包括rig-i、mda5等rlr受体。体外人工合成靶向rig-i和mda5的sirna并转染bmdc,转染24小时后感染mhv-wt或n7-mtase突变病毒(moi=0.1)。感染后16小时,收细胞上清,用于检测病毒载量(噬斑法)。病毒复制水平见附图3c。由图3c可知,具有不同帽子结构的rna诱导bmdc上调ifn-β水平的能力从高到低依次为:无帽子结构的pppg-rna,无n7-甲基化修饰的帽子结构的gpppg-rna,具有n7-甲基化修饰的帽子结构的m7gpppg-rna,具有n7-甲基化和2’-o-甲基化双重修饰的帽子结构的m7gpppgm-rna。polyi:c为双链核苷酸类似物,是一种i型干扰素诱导剂。
(2)筛选介导n7-mtase突变病毒减毒表型的宿主模式识别受体如图4,敲低细胞内rig-i的表达水平能够使mhv-nsp14-y414a病毒载量提高约2倍,而敲低mda5的表达水平能够使mhv-nsp14-y414a病毒载量提高约6倍,但这两种分子的表达水平变化对野生型病毒的复制并没有显著影响。综合图3c的结果,可以推测缺乏n7-甲基化帽子结构的rna作为一种病原相关分子模式,很可能被模式识别受体mda5和rig-i所识别,进而激活了宿主的抗病毒天然免疫反应,加速对病毒的清除作用,因此n7-mtase突变病毒呈现出减毒表型。这种机制为正向设计n7-mtase缺陷型冠状病毒减毒活疫苗提供了理论依据。
实施例4、n7-mtase突变病毒的致病性和安全性评估
1.mhv-wt及其突变病毒的小鼠感染实验
将56只3~4周龄的c57bl/6小鼠按下表随机分组。
表3
根据分组向小鼠肝内注射mhv-wt或n7-mtase突变病毒,剂量为2×106pfu,同时注射等量的病毒溶剂作为对照。感染后第1、2、5天,b、d、f、h每组随机取3只小鼠安乐死,分离肝脏,一半冻存,用于检测病毒载量;一半置于4%多聚甲醛中固定,用于病理切片分析。a、c、e、g组小鼠连续观察8天,记录小鼠生存状态和体重变化,绘制存活率曲线和体重变化曲线,结果如图4a和4b所示。
由如图4a可知,感染mhv野生型病毒的小鼠从第2天开始体重持续下降,直至死亡;而感染n7-mtase突变病毒的小鼠仅在感染第1天出现了短暂的体重平稳期,随后体重逐渐增加,最终恢复到与对照组小鼠相仿的水平。
由如图4b可知,感染野生型病毒的小鼠在第4~8天全部死亡,而感染n7-mtase突变病毒的小鼠和对照组小鼠全部存活。综合图4a结果可知,n7-mtase突变病毒的致病能力明显下降。
2.小鼠肝脏病毒载量检测
取肝脏,称重记录。向肝脏中加入1mlpbs,研磨匀浆,10000rpm离心5min,取上清。将匀浆液上清进行梯度稀释,随后用噬斑法检测病毒滴度。根据所得病毒滴度、稀释倍数和肝重计算肝脏病毒载量,单位为pfu/g。结果展示于图4c。由图4c可知,n7-mtase突变病毒仅能在接种后第1天被检测到,第2天时病毒已全部被清除,而野生型病毒的载量持续增加,第5天时处于峰值状态。表明n7-mtase突变病毒在免疫系统完全的机体内被快速清除,无法建立持续高效的致病性感染。
3.小鼠肝脏病理分析
将多聚甲醛固定好的肝脏送交公司,石蜡包埋、切片后进行he染色,用于判断炎症和病变程度。结果展示于图4d。由图4d可知,病毒感染后第5天,mhv-wt组小鼠的肝脏出现了严重的炎症细胞浸润、组织纤维化、局部出血等病变,而n7-mtase突变病毒的感染仅造成了极其轻微的炎症细胞浸润,再次佐证n7-mtase突变病毒的致病性严重降低。
实施例5、n7-mtase突变病毒的免疫原性评估
本部分实验中,我们选取一种已报道的基于冠状病毒nsp14核酸外切酶活性位点(d89a&e91a)而设计的减毒活疫苗,即mhv-nsp14-d89a&e91a作为对照。
1.mhv-wt及其突变病毒的小鼠免疫实验
将50只3~4周龄的c57bl/6小鼠按下表随机分组。
表4
按分组向小鼠皮下注射mhv-wt、n7-mtase突变病毒或核酸外切酶突变病毒,剂量为5×105pfu,同时注射等量的病毒溶剂作为对照。a、c、e、g、i组小鼠在免疫后第2、4、8、12、24、36、48周采集血液,用于中和抗体检测;b、d、f、h、j组小鼠在免疫后4周安乐死,取脾脏用于检测t细胞免疫反应。
2.中和抗体检测(噬斑减数法)
(1)采集小鼠血液后,室温静置1~2小时。随后2000rpm离心20min,分离血清,56℃灭活30min,分装冻存。
(2)取已知病毒滴度的野生型mhv病毒,稀释至1000pfu/ml,分装冻存。同时将l2细胞接种于24孔板,待密度达到90%后即可开始中和实验。
(3)取出血清,以1:5为起始稀释倍数,对血清进行连续2倍梯度稀释。随后将100ul血清与100ul病毒(约100pfu)等体积混合,37℃孵育1小时。
(4)孵育结束后,取l2细胞,弃掉培养基,将病毒-血清混合液转移至细胞中,吸附1小时。之后弃吸附液,更换1%甲基纤维素培养液,37℃培养48小时,直至肉眼可见的空斑形成。固定、染色,统计空斑数目。
(5)计算噬斑减少率。噬斑减少率=(1-样品孔噬斑数/对照孔噬斑数)×100%。根据reed-muech法计算prnt50,该值代表使病毒空斑数减少一半对应的血清稀释倍数,可表征中和抗体效价。中和抗体效价展示于图5a和5b。
图5a中,两种n7-mtase突变病毒快速诱导小鼠产生了高效价的中和抗体,水平与mhv-wt相当;意外的是,核酸外切酶突变病毒诱导的中和抗体水平非常低,提示基于n7-mtase设计的减毒活疫苗具有明显的优势。
由图5b可知,两种n7-mtase突变病毒诱导的中和抗体能够存在约1年时间,虽然抗体效价有所降低,但仍保持在较高的水平(69.18~131.83)。
3.t细胞免疫应答评估
(1)免疫后4周,小鼠安乐死并分离脾脏。无菌环境下轻轻碾压脾脏,将挤出的脾细胞过滤至rpmi1640培养基中,离心去除组织碎片,裂解红细胞。清洗、重悬脾细胞,计数并铺板于96孔板,密度控制在5×106cell/孔。
(2)铺好细胞后,按照moi=0.1向每孔细胞中加5×105pfumhv进行再刺激。刺激19小时后,加入高尔基体抑制剂bfa阻断蛋白转运。刺激24小时后收细胞进行检测。
(3)收脾细胞,设置空白管(×1)、单染管(×4)、实验管若干,每管分别取2×106细胞于1.5ml离心管,350g离心5min,弃上清。pbs清洗细胞1遍。加表面抗体percp/cy5.5-cd3、fitc-cd4、apc-cd8室温染色30min(空白管除外),pbs清洗细胞2遍。
(4)加fixationbuffer,室温避光固定30min。pbs清洗1遍。
(5)加permeabilizationbuffer,清洗细胞3遍。
(6)加胞内细胞因子抗体pe-ifn-γ和同型对照抗体pe-igg1,室温避光染色30min。随后清洗细胞2遍。
(7)用pbs重悬细胞,200目筛网过滤至流式管,上机检测。利用前向散射光和侧向散射光去除死细胞,设门圈出cd3+cd4+ifn-γ+t细胞群和cd3+cd8+ifn-γ+t细胞群,统计其阳性比例。所得结果展示于图5c。
由图5c可知,n7-mtase突变病毒可以同时诱导强烈的辅助性t细胞反应(cd4+)和杀伤性t细胞(cd8+)反应,其中mhv-nsp14-y414a株诱导的cd8+t细胞反应甚至强于野生型病毒;相比之下,核酸外切酶突变病毒并不能诱导显著的cd4+和cd8+t细胞应答。
实施例6、n7-mtase突变病毒的保护效果评估
将44只3~4周龄的c57bl/6小鼠按下表随机分组,并按实施例4的剂量进行免疫。免疫后30天,用野生型mhv进行肝内注射攻毒,攻毒剂量为2×107pfu。
表5
a、c、e、g组小鼠连续观察8天,记录小鼠生存状态和体重变化,绘制存活率曲线和体重变化曲线,结果如图6a和6b所示。
由图6a可知,所有接受n7-mtase突变病毒和wt病毒免疫的小鼠攻毒后,体重始终保持平稳,生长状态良好,而接受溶剂免疫的对照组小鼠攻毒后体重呈现断崖式下降,直到死亡。
由图6b可知,接受n7-mtase突变病毒和wt病毒免疫的小鼠攻毒后,所有小鼠全部存活,存活率为100%(5/5);而接受溶剂免疫的对照组小鼠攻毒后,仅有20%(1/5)小鼠存活,说明n7-mtase作为减毒活疫苗,可以为接种动物提供与野生型病毒相仿的保护效果,保护效果显著。
攻毒后第2天和第5天,b、d、f、h组随机取3只小鼠安乐死,分离肝脏,用于检测病毒载量,检测方法与实施例4相同。结果见附图6c。
由图6c可知,攻毒后第2天,100%(3/3)的mhv-wt免疫组小鼠、67%(2/3)的mhv-nsp14-y414a免疫组小鼠、33%(1/3)的mhv-nsp14-d330a免疫组小鼠肝脏中已不能检测到活病毒,而对照组小鼠的肝脏病毒载量均在较高水平;攻毒后第5天,所有病毒免疫的小鼠肝脏中均不能检测到活病毒,对照组小鼠的病毒载量依旧维持在峰值状态。说明接种n7-mtase减毒活疫苗能够及时阻断病毒的再次感染和复制。从图中可以看到,接种n7-mtase减株的小鼠在5天内完全清除了再次感染的病毒,为小鼠提供了明显的保护效果。
最后,还需要说明的是,术语“包括”、“包含”或者其任何其他变体意在涵盖非排他性的包含,从而使得包括一系列要素的过程、方法、物品或者设备不仅包括那些要素,而且还包括没有明确列出的其他要素,或者是还包括为这种过程、方法、物品或者设备所固有的要素。
尽管已描述了本发明的优选实施例,但本领域内的技术人员一旦得知了基本创造性概念,则可对这些实施例作出另外的变更和修改。所以,所附权利要求意欲解释为包括优选实施例以及落入本发明范围的所有变更和修改。
显然,本领域的技术人员可以对本发明进行各种改动和变型而不脱离本发明的精神和范围。这样,倘若本发明的这些修改和变型属于本发明权利要求及其等同技术的范围之内,则本发明也意图包含这些改动和变型在内。
序列表
<110>武汉大学
<120>一种n7-甲基转移酶缺陷型冠状病毒减毒疫苗株及其制备方法与应用
<160>26
<170>siposequencelisting1.0
<210>1
<211>521
<212>prt
<213>人工序列(artificialsequence)
<400>1
cysthrthrasnleuphelysaspcysserargsertyrvalglytyr
151015
hisproalahisalaproserpheleualavalaspasplystyrlys
202530
valglyglyaspleualavalcysleuasnvalalaaspseralaval
354045
thrtyrserargleuileserleumetglyphelysleuaspleuthr
505560
leuaspglytyrcyslysleupheilethrargaspglualailelys
65707580
argvalargalatrpvalglypheaspalagluglyalahisalaile
859095
argaspserileglythrasnpheproleuglnleuglypheserthr
100105110
glyileaspphevalvalglualathrglymetphealagluargasp
115120125
glytyrvalphelyslysalaalaalaargalaproproglyglugln
130135140
phelyshisleuileproleumetserargglyglnlystrpaspval
145150155160
valargileargilevalglnmetleuserasphisleuvalaspleu
165170175
alaaspservalvalleuvalthrtrpalaalaserphegluleuthr
180185190
cysleuargtyrphealalysvalglyarggluvalvalcysserval
195200205
cysthrlysargalathrcyspheasnserargthrglytyrtyrgly
210215220
cystrparghissertyrsercysasptyrleutyrasnproleuile
225230235240
valaspileglnglntrpglytyrthrglyserleuthrserasnhis
245250255
aspproilecysservalhislysglyalahisvalalaserserasp
260265270
alailemetthrargcysleualavalhisaspcysphecyslysser
275280285
valasntrpasnleuglutyrproileileserasngluvalserval
290295300
asnthrsercysargleuleuglnargvalmetpheargalaalamet
305310315320
leucysasnargtyraspvalcystyraspileglyasnprolysgly
325330335
leualacysvallysglytyraspphelysphetyraspalaserpro
340345350
valvallysservallysglnphevaltyrlystyrglualahislys
355360365
aspglnpheleuaspglyleucysmetphetrpasncysasnvalasp
370375380
lystyrproalaasnalavalvalcysargpheaspthrargvalleu
385390395400
asnlysleuasnleuproglycysasnglyglyserleutyrvalasn
405410415
lyshisalaphehisthrserprophethrargalaalaphegluasn
420425430
leulysprometprophephetyrtyrseraspthrprocysvaltyr
435440445
metgluglymetgluserlysglnvalasptyrvalproleuargser
450455460
alathrcysilethrargcysasnleuglyglyalavalcysleulys
465470475480
hisalagluglutyrargglutyrleuglusertyrasnthralathr
485490495
thralaglyphethrphetrpvaltyrlysthrpheaspphetyrasn
500505510
leutrpasnthrphethrargleugln
515520
<210>2
<211>1563
<212>dna
<213>人工序列(artificialsequence)
<400>2
tgtactacaaatttgtttaaggattgtagcaggagctatgtaggatatcacccagcccat60
gcaccatcctttttggcagttgatgacaaatataaggtaggcggtgatttagccgtttgc120
cttaatgttgctgattctgctgtcacttattcgcggcttatatcactcatgggattcaag180
cttgacttgacccttgatggttattgtaagctgtttataactagagatgaagctatcaaa240
cgtgttagagcctgggttggcttcgatgcagaaggtgcccatgcgatacgtgatagcatt300
gggacaaatttcccattacaattaggcttttcgactggaattgattttgttgtcgaagcc360
actggaatgtttgctgagagagatggttatgtctttaaaaaggcagccgcacgagctcct420
cctggcgaacaatttaaacaccttatcccacttatgtcaagagggcagaaatgggatgtg480
gttcgcattagaatagtacaaatgttgtcagaccacctagtggatttggcagacagtgtt540
gtacttgtgacgtgggctgccagctttgagctcacatgtttgcgatatttcgctaaagtt600
ggaagagaagttgtgtgtagtgtctgcaccaagcgtgcgacatgttttaattctagaact660
ggatactatggatgctggcgacatagttattcctgtgattacctgtacaacccactaata720
gttgacattcaacagtggggatatacaggatctttaactagcaatcatgatcctatttgc780
agcgtgcataagggtgctcatgttgcatcatctgatgctatcatgacccggtgtctagct840
gttcatgattgcttttgtaagtctgttaattggaatttagaataccccattatttcaaat900
gaggtcagtgttaatacctcctgcaggttattgcagcgcgtaatgtttagggctgcgatg960
ctatgcaataggtatgatgtgtgttatgacattggcaaccctaaaggtcttgcctgtgtc1020
aaaggatatgattttaagttttatgatgcctcccctgttgttaagtctgttaaacagttt1080
gtttataaatacgaggcacataaagatcaatttttagatggtttgtgtatgttttggaac1140
tgcaatgtggataagtatccagcgaatgcagttgtgtgtaggtttgacacgcgtgtgttg1200
aacaaattaaatctccctggctgtaatggtggcagtttgtatgttaacaaacatgcattc1260
cacaccagtccctttacccgggctgccttcgagaatttgaagcctatgcctttcttttat1320
tattcagatacgccctgtgtgtatatggaaggcatggaatctaagcaggtcgattatgtc1380
ccattgagaagcgctacatgcatcacaagatgcaatttaggtggcgctgtttgtttaaaa1440
catgctgaggagtatcgtgagtaccttgagtcttacaatacggcaaccacagcgggtttt1500
actttttgggtctataagacttttgatttttataacctttggaatacttttactaggctc1560
caa1563
<210>3
<211>1563
<212>dna
<213>人工序列(artificialsequence)
<400>3
tgtactacaaatttgtttaaggattgtagcaggagctatgtaggatatcacccagcccat60
gcaccatcctttttggcagttgatgacaaatataaggtaggcggtgatttagccgtttgc120
cttaatgttgctgattctgctgtcacttattcgcggcttatatcactcatgggattcaag180
cttgacttgacccttgatggttattgtaagctgtttataactagagatgaagctatcaaa240
cgtgttagagcctgggttggcttcgatgcagaaggtgcccatgcgatacgtgatagcatt300
gggacaaatttcccattacaattaggcttttcgactggaattgattttgttgtcgaagcc360
actggaatgtttgctgagagagatggttatgtctttaaaaaggcagccgcacgagctcct420
cctggcgaacaatttaaacaccttatcccacttatgtcaagagggcagaaatgggatgtg480
gttcgcattagaatagtacaaatgttgtcagaccacctagtggatttggcagacagtgtt540
gtacttgtgacgtgggctgccagctttgagctcacatgtttgcgatatttcgctaaagtt600
ggaagagaagttgtgtgtagtgtctgcaccaagcgtgcgacatgttttaattctagaact660
ggatactatggatgctggcgacatagttattcctgtgattacctgtacaacccactaata720
gttgacattcaacagtggggatatacaggatctttaactagcaatcatgatcctatttgc780
agcgtgcataagggtgctcatgttgcatcatctgatgctatcatgacccggtgtctagct840
gttcatgattgcttttgtaagtctgttaattggaatttagaataccccattatttcaaat900
gaggtcagtgttaatacctcctgcaggttattgcagcgcgtaatgtttagggctgcgatg960
ctatgcaataggtatgatgtgtgttatgcaattggcaaccctaaaggtcttgcctgtgtc1020
aaaggatatgattttaagttttatgatgcctcccctgttgttaagtctgttaaacagttt1080
gtttataaatacgaggcacataaagatcaatttttagatggtttgtgtatgttttggaac1140
tgcaatgtggataagtatccagcgaatgcagttgtgtgtaggtttgacacgcgtgtgttg1200
aacaaattaaatctccctggctgtaatggtggcagtttgtatgttaacaaacatgcattc1260
cacaccagtccctttacccgggctgccttcgagaatttgaagcctatgcctttcttttat1320
tattcagatacgccctgtgtgtatatggaaggcatggaatctaagcaggtcgattatgtc1380
ccattgagaagcgctacatgcatcacaagatgcaatttaggtggcgctgtttgtttaaaa1440
catgctgaggagtatcgtgagtaccttgagtcttacaatacggcaaccacagcgggtttt1500
actttttgggtctataagacttttgatttttataacctttggaatacttttactaggctc1560
caa1563
<210>4
<211>1563
<212>dna
<213>人工序列(artificialsequence)
<400>4
tgtactacaaatttgtttaaggattgtagcaggagctatgtaggatatcacccagcccat60
gcaccatcctttttggcagttgatgacaaatataaggtaggcggtgatttagccgtttgc120
cttaatgttgctgattctgctgtcacttattcgcggcttatatcactcatgggattcaag180
cttgacttgacccttgatggttattgtaagctgtttataactagagatgaagctatcaaa240
cgtgttagagcctgggttggcttcgatgcagaaggtgcccatgcgatacgtgatagcatt300
gggacaaatttcccattacaattaggcttttcgactggaattgattttgttgtcgaagcc360
actggaatgtttgctgagagagatggttatgtctttaaaaaggcagccgcacgagctcct420
cctggcgaacaatttaaacaccttatcccacttatgtcaagagggcagaaatgggatgtg480
gttcgcattagaatagtacaaatgttgtcagaccacctagtggatttggcagacagtgtt540
gtacttgtgacgtgggctgccagctttgagctcacatgtttgcgatatttcgctaaagtt600
ggaagagaagttgtgtgtagtgtctgcaccaagcgtgcgacatgttttaattctagaact660
ggatactatggatgctggcgacatagttattcctgtgattacctgtacaacccactaata720
gttgacattcaacagtggggatatacaggatctttaactagcaatcatgatcctatttgc780
agcgtgcataagggtgctcatgttgcatcatctgatgctatcatgacccggtgtctagct840
gttcatgattgcttttgtaagtctgttaattggaatttagaataccccattatttcaaat900
gaggtcagtgttaatacctcctgcaggttattgcagcgcgtaatgtttagggctgcgatg960
ctatgcaataggtatgatgtgtgttatgacattggcaaccctaaaggtcttgcctgtgtc1020
aaaggatatgattttaagttttatgatgcctcccctgttgttaagtctgttaaacagttt1080
gtttataaatacgaggcacataaagatcaatttttagatggtttgtgtatgttttggaac1140
tgcaatgtggataagtatccagcgaatgcagttgtgtgtaggtttgacacgcgtgtgttg1200
aacaaattaaatctccctggctgtaatggtggcagtttggcagttaacaaacatgcattc1260
cacaccagtccctttacccgggctgccttcgagaatttgaagcctatgcctttcttttat1320
tattcagatacgccctgtgtgtatatggaaggcatggaatctaagcaggtcgattatgtc1380
ccattgagaagcgctacatgcatcacaagatgcaatttaggtggcgctgtttgtttaaaa1440
catgctgaggagtatcgtgagtaccttgagtcttacaatacggcaaccacagcgggtttt1500
actttttgggtctataagacttttgatttttataacctttggaatacttttactaggctc1560
caa1563
<210>5
<211>1563
<212>dna
<213>人工序列(artificialsequence)
<400>5
tgtactacaaatttgtttaaggattgtagcaggagctatgtaggatatcacccagcccat60
gcaccatcctttttggcagttgatgacaaatataaggtaggcggtgatttagccgtttgc120
cttaatgttgctgattctgctgtcacttattcgcggcttatatcactcatgggattcaag180
cttgacttgacccttgatggttattgtaagctgtttataactagagatgaagctatcaaa240
cgtgttagagcctgggttggcttcgatgcagaaggtgcccatgcgatacgtgatagcatt300
gggacaaatttcccattacaattaggcttttcgactggaattgattttgttgtcgaagcc360
actggaatgtttgctgagagagatggttatgtctttaaaaaggcagccgcacgagctcct420
cctggcgaacaatttaaacaccttatcccacttatgtcaagagggcagaaatgggatgtg480
gttcgcattagaatagtacaaatgttgtcagaccacctagtggatttggcagacagtgtt540
gtacttgtgacgtgggctgccagctttgagctcacatgtttgcgatatttcgctaaagtt600
ggaagagaagttgtgtgtagtgtctgcaccaagcgtgcgacatgttttaattctagaact660
ggatactatggatgctggcgacatagttattcctgtgattacctgtacaacccactaata720
gttgacattcaacagtggggatatacaggatctttaactagcaatcatgatcctatttgc780
agcgtgcataagggtgctcatgttgcatcatctgatgctatcatgacccggtgtctagct840
gttcatgattgcttttgtaagtctgttaattggaatttagaataccccattatttcaaat900
gaggtcagtgttaatacctcctgcaggttattgcagcgcgtaatgtttagggctgcgatg960
ctatgcaataggtatgatgtgtgttatgcaattggcaaccctaaaggtcttgcctgtgtc1020
aaaggatatgattttaagttttatgatgcctcccctgttgttaagtctgttaaacagttt1080
gtttataaatacgaggcacataaagatcaatttttagatggtttgtgtatgttttggaac1140
tgcaatgtggataagtatccagcgaatgcagttgtgtgtaggtttgacacgcgtgtgttg1200
aacaaattaaatctccctggctgtaatggtggcagtttggcagttaacaaacatgcattc1260
cacaccagtccctttacccgggctgccttcgagaatttgaagcctatgcctttcttttat1320
tattcagatacgccctgtgtgtatatggaaggcatggaatctaagcaggtcgattatgtc1380
ccattgagaagcgctacatgcatcacaagatgcaatttaggtggcgctgtttgtttaaaa1440
catgctgaggagtatcgtgagtaccttgagtcttacaatacggcaaccacagcgggtttt1500
actttttgggtctataagacttttgatttttataacctttggaatacttttactaggctc1560
caa1563
<210>6
<211>527
<212>prt
<213>人工序列(artificialsequence)
<400>6
alagluasnvalthrglyleuphelysaspcysserlysvalilethr
151015
glyleuhisprothrglnalaprothrhisleuservalaspthrlys
202530
phelysthrgluglyleucysvalaspileproglyileprolysasp
354045
metthrtyrargargleuilesermetmetglyphelysmetasntyr
505560
glnvalasnglytyrproasnmetpheilethrarggluglualaile
65707580
arghisvalargalatrpileglypheaspvalgluglycyshisala
859095
thrargglualavalglythrasnleuproleuglnleuglypheser
100105110
thrglyvalasnleuvalalavalprothrglytyrvalaspthrpro
115120125
asnasnthrasppheserargvalseralalysproproproglyasp
130135140
glnphelyshisleuileproleumettyrlysglyleuprotrpasn
145150155160
valvalargilelysilevalglnmetleuseraspthrleulysasn
165170175
leuseraspargvalvalphevalleutrpalahisglyphegluleu
180185190
thrsermetlystyrphevallysileglyprogluargthrcyscys
195200205
leucysaspargargalathrcyspheserthralaseraspthrtyr
210215220
alacystrphishisserileglypheasptyrvaltyrasnprophe
225230235240
metileaspvalglnglntrpglyphethrglyasnleuglnserasn
245250255
hisaspleutyrcysglnvalhisglyasnalahisvalalasercys
260265270
aspalailemetthrargcysleualavalhisglucysphevallys
275280285
argvalasptrpthrileglutyrproileileglyaspgluleulys
290295300
ileasnalaalacysarglysvalglnhismetvalvallysalaala
305310315320
leuleualaasplyspheprovalleuhisaspileglyasnprolys
325330335
alailelyscysvalproglnalaaspvalglutrplysphetyrasp
340345350
alaglnprocysserasplysalatyrlysileglugluleuphetyr
355360365
sertyralathrhisserasplysphethraspglyvalcysleuphe
370375380
trpasncysasnvalaspargtyrproalaasnserilevalcysarg
385390395400
pheaspthrargvalleuserasnleuasnleuproglycysaspgly
405410415
glyserleutyrvalasnlyshisalaphehisthrproalapheasp
420425430
lysseralaphevalasnleulysglnleuprophephetyrtyrser
435440445
aspserprocysgluserhisglylysglnvalvalseraspileasp
450455460
tyrvalproleulysseralathrcysilethrargcysasnleugly
465470475480
glyalavalcysarghishisalaasnglutyrargleutyrleuasp
485490495
alatyrasnmetmetileseralaglypheserleutrpvaltyrlys
500505510
glnpheaspthrtyrasnleutrpasnthrphethrargleugln
515520525
<210>7
<211>1581
<212>dna
<213>人工序列(artificialsequence)
<400>7
gcggagaacgtgaccggtctgtttaaggattgcagcaaagttatcaccggcctgcacccg60
acccaggcgccgacccacctgagcgtggacaccaagttcaaaaccgaaggtctgtgcgtt120
gacatcccgggcattccgaaagatatgacctaccgtcgtctgatcagcatgatgggtttt180
aagatgaactaccaagtgaacggctatccgaacatgttcatcacccgtgaggaagcgatt240
cgtcacgtgcgtgcgtggattggttttgatgttgaaggttgccatgcgacccgtgaagcg300
gttggtaccaacctgccgctgcagctgggtttcagcaccggcgtgaacctggtggcggtt360
ccgaccggctacgttgacaccccgaacaacaccgattttagccgtgtgagcgcgaagccg420
ccgccgggtgaccagttcaaacacctgatcccgctgatgtataagggcctgccgtggaac480
gtggttcgtatcaaaattgttcaaatgctgagcgacaccctgaagaacctgagcgatcgt540
gtggtttttgtgctgtgggcgcacggtttcgagctgaccagcatgaagtactttgttaaa600
attggcccggaacgtacctgctgcctgtgcgaccgtcgtgcgacctgcttcagcaccgcg660
agcgatacctatgcgtgctggcaccacagcatcggttttgactacgtgtataacccgttc720
atgattgatgttcagcaatggggttttaccggcaacctgcagagcaaccacgacctgtac780
tgccaagtgcacggcaacgcgcacgttgcgagctgcgatgcgatcatgacccgttgcctg840
gcggtgcacgagtgcttcgtgaaacgtgttgactggaccatcgagtacccgatcattggt900
gatgaactgaagattaacgcggcgtgccgtaaagtgcagcacatggtggttaaggcggcg960
ctgctggcggacaaatttccggttctgcacgatatcggcaacccgaaggcgattaaatgc1020
gtgccgcaggcggacgttgaatggaaattttacgacgcgcaaccgtgcagcgataaggcg1080
tataaaatcgaggaactgttctacagctatgcgacccacagcgacaagtttaccgatggt1140
gtgtgcctgttctggaactgcaacgttgaccgttacccggcgaacagcattgtgtgccgt1200
ttcgatacccgtgttctgagcaacctgaacctgccgggttgcgatggtggcagcctgtat1260
gtgaacaaacacgcgttccacaccccggcgtttgataagagcgcgttcgttaacctgaaa1320
cagctgccgttcttttactatagcgacagcccgtgcgagagccacggtaaacaagtggtt1380
agcgacatcgattacgtgccgctgaagagcgcgacctgcattacccgttgcaacctgggt1440
ggcgcggtttgccgtcaccacgcgaacgaataccgtctgtatctggatgcgtataacatg1500
atgattagcgcgggttttagcctgtgggtttacaagcagttcgacacctataacctgtgg1560
aacaccttcacccgtctgcaa1581
<210>8
<211>1581
<212>dna
<213>人工序列(artificialsequence)
<400>8
gcggagaacgtgaccggtctgtttaaggattgcagcaaagttatcaccggcctgcacccg60
acccaggcgccgacccacctgagcgtggacaccaagttcaaaaccgaaggtctgtgcgtt120
gacatcccgggcattccgaaagatatgacctaccgtcgtctgatcagcatgatgggtttt180
aagatgaactaccaagtgaacggctatccgaacatgttcatcacccgtgaggaagcgatt240
cgtcacgtgcgtgcgtggattggttttgatgttgaaggttgccatgcgacccgtgaagcg300
gttggtaccaacctgccgctgcagctgggtttcagcaccggcgtgaacctggtggcggtt360
ccgaccggctacgttgacaccccgaacaacaccgattttagccgtgtgagcgcgaagccg420
ccgccgggtgaccagttcaaacacctgatcccgctgatgtataagggcctgccgtggaac480
gtggttcgtatcaaaattgttcaaatgctgagcgacaccctgaagaacctgagcgatcgt540
gtggtttttgtgctgtgggcgcacggtttcgagctgaccagcatgaagtactttgttaaa600
attggcccggaacgtacctgctgcctgtgcgaccgtcgtgcgacctgcttcagcaccgcg660
agcgatacctatgcgtgctggcaccacagcatcggttttgactacgtgtataacccgttc720
atgattgatgttcagcaatggggttttaccggcaacctgcagagcaaccacgacctgtac780
tgccaagtgcacggcaacgcgcacgttgcgagctgcgatgcgatcatgacccgttgcctg840
gcggtgcacgagtgcttcgtgaaacgtgttgactggaccatcgagtacccgatcattggt900
gatgaactgaagattaacgcggcgtgccgtaaagtgcagcacatggtggttaaggcggcg960
ctgctggcggacaaatttccggttctgcacgcaatcggcaacccgaaggcgattaaatgc1020
gtgccgcaggcggacgttgaatggaaattttacgacgcgcaaccgtgcagcgataaggcg1080
tataaaatcgaggaactgttctacagctatgcgacccacagcgacaagtttaccgatggt1140
gtgtgcctgttctggaactgcaacgttgaccgttacccggcgaacagcattgtgtgccgt1200
ttcgatacccgtgttctgagcaacctgaacctgccgggttgcgatggtggcagcctgtat1260
gtgaacaaacacgcgttccacaccccggcgtttgataagagcgcgttcgttaacctgaaa1320
cagctgccgttcttttactatagcgacagcccgtgcgagagccacggtaaacaagtggtt1380
agcgacatcgattacgtgccgctgaagagcgcgacctgcattacccgttgcaacctgggt1440
ggcgcggtttgccgtcaccacgcgaacgaataccgtctgtatctggatgcgtataacatg1500
atgattagcgcgggttttagcctgtgggtttacaagcagttcgacacctataacctgtgg1560
aacaccttcacccgtctgcaa1581
<210>9
<211>1581
<212>dna
<213>人工序列(artificialsequence)
<400>9
gcggagaacgtgaccggtctgtttaaggattgcagcaaagttatcaccggcctgcacccg60
acccaggcgccgacccacctgagcgtggacaccaagttcaaaaccgaaggtctgtgcgtt120
gacatcccgggcattccgaaagatatgacctaccgtcgtctgatcagcatgatgggtttt180
aagatgaactaccaagtgaacggctatccgaacatgttcatcacccgtgaggaagcgatt240
cgtcacgtgcgtgcgtggattggttttgatgttgaaggttgccatgcgacccgtgaagcg300
gttggtaccaacctgccgctgcagctgggtttcagcaccggcgtgaacctggtggcggtt360
ccgaccggctacgttgacaccccgaacaacaccgattttagccgtgtgagcgcgaagccg420
ccgccgggtgaccagttcaaacacctgatcccgctgatgtataagggcctgccgtggaac480
gtggttcgtatcaaaattgttcaaatgctgagcgacaccctgaagaacctgagcgatcgt540
gtggtttttgtgctgtgggcgcacggtttcgagctgaccagcatgaagtactttgttaaa600
attggcccggaacgtacctgctgcctgtgcgaccgtcgtgcgacctgcttcagcaccgcg660
agcgatacctatgcgtgctggcaccacagcatcggttttgactacgtgtataacccgttc720
atgattgatgttcagcaatggggttttaccggcaacctgcagagcaaccacgacctgtac780
tgccaagtgcacggcaacgcgcacgttgcgagctgcgatgcgatcatgacccgttgcctg840
gcggtgcacgagtgcttcgtgaaacgtgttgactggaccatcgagtacccgatcattggt900
gatgaactgaagattaacgcggcgtgccgtaaagtgcagcacatggtggttaaggcggcg960
ctgctggcggacaaatttccggttctgcacgatatcggcaacccgaaggcgattaaatgc1020
gtgccgcaggcggacgttgaatggaaattttacgacgcgcaaccgtgcagcgataaggcg1080
tataaaatcgaggaactgttctacagctatgcgacccacagcgacaagtttaccgatggt1140
gtgtgcctgttctggaactgcaacgttgaccgttacccggcgaacagcattgtgtgccgt1200
ttcgatacccgtgttctgagcaacctgaacctgccgggttgcgatggtggcagcctggca1260
gtgaacaaacacgcgttccacaccccggcgtttgataagagcgcgttcgttaacctgaaa1320
cagctgccgttcttttactatagcgacagcccgtgcgagagccacggtaaacaagtggtt1380
agcgacatcgattacgtgccgctgaagagcgcgacctgcattacccgttgcaacctgggt1440
ggcgcggtttgccgtcaccacgcgaacgaataccgtctgtatctggatgcgtataacatg1500
atgattagcgcgggttttagcctgtgggtttacaagcagttcgacacctataacctgtgg1560
aacaccttcacccgtctgcaa1581
<210>10
<211>1581
<212>dna
<213>人工序列(artificialsequence)
<400>10
gcggagaacgtgaccggtctgtttaaggattgcagcaaagttatcaccggcctgcacccg60
acccaggcgccgacccacctgagcgtggacaccaagttcaaaaccgaaggtctgtgcgtt120
gacatcccgggcattccgaaagatatgacctaccgtcgtctgatcagcatgatgggtttt180
aagatgaactaccaagtgaacggctatccgaacatgttcatcacccgtgaggaagcgatt240
cgtcacgtgcgtgcgtggattggttttgatgttgaaggttgccatgcgacccgtgaagcg300
gttggtaccaacctgccgctgcagctgggtttcagcaccggcgtgaacctggtggcggtt360
ccgaccggctacgttgacaccccgaacaacaccgattttagccgtgtgagcgcgaagccg420
ccgccgggtgaccagttcaaacacctgatcccgctgatgtataagggcctgccgtggaac480
gtggttcgtatcaaaattgttcaaatgctgagcgacaccctgaagaacctgagcgatcgt540
gtggtttttgtgctgtgggcgcacggtttcgagctgaccagcatgaagtactttgttaaa600
attggcccggaacgtacctgctgcctgtgcgaccgtcgtgcgacctgcttcagcaccgcg660
agcgatacctatgcgtgctggcaccacagcatcggttttgactacgtgtataacccgttc720
atgattgatgttcagcaatggggttttaccggcaacctgcagagcaaccacgacctgtac780
tgccaagtgcacggcaacgcgcacgttgcgagctgcgatgcgatcatgacccgttgcctg840
gcggtgcacgagtgcttcgtgaaacgtgttgactggaccatcgagtacccgatcattggt900
gatgaactgaagattaacgcggcgtgccgtaaagtgcagcacatggtggttaaggcggcg960
ctgctggcggacaaatttccggttctgcacgcaatcggcaacccgaaggcgattaaatgc1020
gtgccgcaggcggacgttgaatggaaattttacgacgcgcaaccgtgcagcgataaggcg1080
tataaaatcgaggaactgttctacagctatgcgacccacagcgacaagtttaccgatggt1140
gtgtgcctgttctggaactgcaacgttgaccgttacccggcgaacagcattgtgtgccgt1200
ttcgatacccgtgttctgagcaacctgaacctgccgggttgcgatggtggcagcctggca1260
gtgaacaaacacgcgttccacaccccggcgtttgataagagcgcgttcgttaacctgaaa1320
cagctgccgttcttttactatagcgacagcccgtgcgagagccacggtaaacaagtggtt1380
agcgacatcgattacgtgccgctgaagagcgcgacctgcattacccgttgcaacctgggt1440
ggcgcggtttgccgtcaccacgcgaacgaataccgtctgtatctggatgcgtataacatg1500
atgattagcgcgggttttagcctgtgggtttacaagcagttcgacacctataacctgtgg1560
aacaccttcacccgtctgcaa1581
<210>11
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>11
cgcggatcctgtactacaaatttgtttaag30
<210>12
<211>34
<212>dna
<213>人工序列(artificialsequence)
<400>12
gcgtgtcgacttattggagcctagtaaaagtatt34
<210>13
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>13
gatgtgtgttatgcaattggcaaccc26
<210>14
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>14
gggttgccaattgcataacacacatc26
<210>15
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>15
ggtggcagtttggcagttaacaaaca26
<210>16
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>16
tgtttgttaactgccaaactgccacc26
<210>17
<211>29
<212>dna
<213>人工序列(artificialsequence)
<400>17
cgcggatccgcggagaacgtgaccggtct29
<210>18
<211>32
<212>dna
<213>人工序列(artificialsequence)
<400>18
ccgctcgagttattgcagacgggtgaaggtgt32
<210>19
<211>27
<212>dna
<213>人工序列(artificialsequence)
<400>19
ccggttctgcacgcaatcggcaacccg27
<210>20
<211>27
<212>dna
<213>人工序列(artificialsequence)
<400>20
cgggttgccgattgcgtgcagaaccgg27
<210>21
<211>27
<212>dna
<213>人工序列(artificialsequence)
<400>21
ggtggcagcctggcagtgaacaaacac27
<210>22
<211>27
<212>dna
<213>人工序列(artificialsequence)
<400>22
gtgtttgttcactgccaggctgccacc27
<210>23
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>23
accaagctaatgtgttgtttgggtcc26
<210>24
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>24
ttacgcacagcaaaccaaaattcc24
<210>25
<211>38
<212>dna
<213>人工序列(artificialsequence)
<400>25
tgggttggcttcgcagcagcaggtgcccatgcgatacg38
<210>26
<211>38
<212>dna
<213>人工序列(artificialsequence)
<400>26
cgcatgggcacctgctgctgcgaagccaacccaggctc38
- 还没有人留言评论。精彩留言会获得点赞!