一种融合蛋白、基因及应用
本发明涉及生物制药技术领域,特别是涉及一种融合蛋白、基因及应用。
背景技术:
egfr属于酪氨酸激酶受体家族中最早被发现的一个成员,因此也称其为erbb1和her1。egfr和该受体家族的其他蛋白一样均为跨膜的糖蛋白,全长的分子量为170kda。egfr的胞外区又可以再分为四个区域,i和iii区的配体结合区域以及含半胱氨酸数量较多的ii和iv区。egfr的激活是通过和自身发生同源二聚或是和erbb家族中的其他受体比如her2,her3发生异源二聚发生的。egfr受体的过表达或突变会导致下游信号通路的失调,和一些表皮生长相关肿瘤如肺癌,结直肠癌及鳞状细胞癌等发病机制密切相关。
egfr在多种肿瘤中高表达的现象很自然的让人们考虑将其作为一个可选的药物靶点来研究,通过靶向治疗可以在某种程度上减轻传统化疗过程中非特异性杀伤所引起的副作用。目前,靶向egfr的治疗策略按作用部位可大致分为两类。一类是靶向egfr胞内酪氨酸激酶区域的小分子激酶抑制剂药物,通过和atp竞争性结合来阻断egfr以及egfr相关的下游信号通路的传导。另一种则是靶向egfr胞外区域的以单克隆抗体为代表的的大分子抗体类药物。治疗性抗体因其特异性,通常在血液循环中具备较长的半衰期,较好的稳定性,并且可以招募自身的免疫系统来作用于靶细胞,是目前临床上应用较广的一类药物。目前已经批准上市并用于临床的有4种治疗性抗体,分别是西妥昔单抗,帕尼单抗,尼妥珠单抗以及耐昔妥珠单抗。
尽管在临床治疗中抗体靶向的胞外区的氨基酸突变不像酪氨酸激酶区域那么频繁,这类突变所造成的耐药性依然大大限制了药物的适用范围。最早观察到的靶向egfr的抗体耐药位点是位于egfr胞外区的s492r突变(montagutc等,naturemedicine2012,(18),221–223)。s492r突变在经西妥昔治疗一段时间后的部分结直肠癌患者中较为常见。在对临床iii期西妥昔和帕尼治疗结直肠癌的对比治疗结果进行回顾性分析后发现,西妥昔给药组中大约有16%的患者产生了s492r突变。最近的临床研究发现,s492,g465,v441,s442,r451,i462,s464,k467,k489,i491等一系列突变位点都会导致抗体的亲和力降低,从而导致耐药现象的发生。g462r和s492r是其中比较常见的获得性耐药突变,大约会有超过10%的患者在用药后产生。相比s492r的突变只导致西妥昔的耐药而不影响帕尼单抗的亲和力,g465r突变位点由于都在西妥昔以及帕尼单抗的结合表位上,会导致西妥昔和帕尼单抗的共同耐药。
抗egfr抗体药物除了通过配体竞争性抑制以及受体内吞来抑制下游信号外,也会通过抗体的fc段来发挥免疫效应,典型的如抗体依赖的细胞介导的细胞毒性作用(adcc)和补体依赖的细胞毒性(cdc)。早期的研究表明,adcc效应是靶向egfr的抗体发挥其抗肿瘤活性的主要因素,相对应的,cdc效应则表现得不明显,因此也不太受到关注。此外,目前临床上应用较广的西妥昔单抗及帕尼单抗的cdc效应均较弱。目前的部分研究认为,要更有效的激活补体,除了要考虑抗原的种类,密度以及在细胞膜表面的流动性,还要考虑抗体结合抗原的表位,以及抗原抗体复合物的空间结构以及抗体结构的柔韧性。
技术实现要素:
本发明针对现有技术中存在的不足,提供了一种针对egfr的纳米抗体fc融合蛋白的制备方法和应用。
本发明首先提供了一种融合蛋白,包括9g8纳米抗体、7d12纳米抗体和igg1型抗体的fc,其中igg1型抗体的fc为双链结构,
9g8纳米抗体和7d12纳米抗体融合后再与igg1型抗体的fc融合,且双链结构的fc两条链各自融合一分子的9g8纳米抗体和7d12纳米抗体,
9g8纳米抗体的氨基酸序列如seqidno.1所示,
7d12纳米抗体的氨基酸序列如seqidno.3所示,
igg1型抗体的fc的氨基酸序列如seqidno.7所示。
优选的,9g8纳米抗体的c端与7d12纳米抗体的n端融合,7d12纳米抗体的c端与fc的n端融合。
更优选的,9g8纳米抗体与7d12纳米抗体之间通过柔性连接肽连接。柔性连接肽的目的在于将9g8纳米抗体与7d12纳米抗体两者之间分隔开,以避免可能存在的两者空间位阻上的相互影响。所以,一般常用的柔性连接肽都可以使用。
进一步优选的,柔性连接肽为(ggggs)n,其中n为1~3的自然数。优选n为3,此时柔性连接肽为(ggggs)3,即ggggsggggsggggs。
最优选的,所述的融合蛋白,包括序列相同的两条链,单条链的氨基酸序列如seqidno.9所示。由于fc为同源二聚体,所以融合蛋白也是同源二聚体形式。
本发明又提供了编码所述融合蛋白的基因。优选的,编码9g8纳米抗体的基因序列如seqidno.2所示,编码7d12纳米抗体的基因序列如seqidno.4所示,编码igg1型抗体的fc的基因序列如seqidno.8所示。更优选的,所述的基因,基因序列如seqidno.10所示,该基因序列为作为同源二聚体的融合蛋白中单条链对应的基因序列,由于两条链的序列相同,所以由同一个基因表达即可。
本发明还提供了所述融合蛋白在制备抗肿瘤药物中的应用。优选的,所述抗肿瘤药物针对的肿瘤类型为egfr阳性的实体瘤。
相比目前已上市的西妥昔单抗,本发明融合蛋白的cdc效应更强,且能克服一系列egfr胞外区的耐药突变,如s492r和g465r。这一类egfr胞外区耐药突变位点会使目前上市的西妥昔单抗或帕尼单抗无法有效亲和egfr,影响药效。本发明融合蛋白在结合细胞表面的egfr后,受体和抗体的复合物的内吞效率以及内吞降解效率均强于西妥昔,在细胞增殖抑制实验中表现出更好的抗肿瘤活性。
附图说明
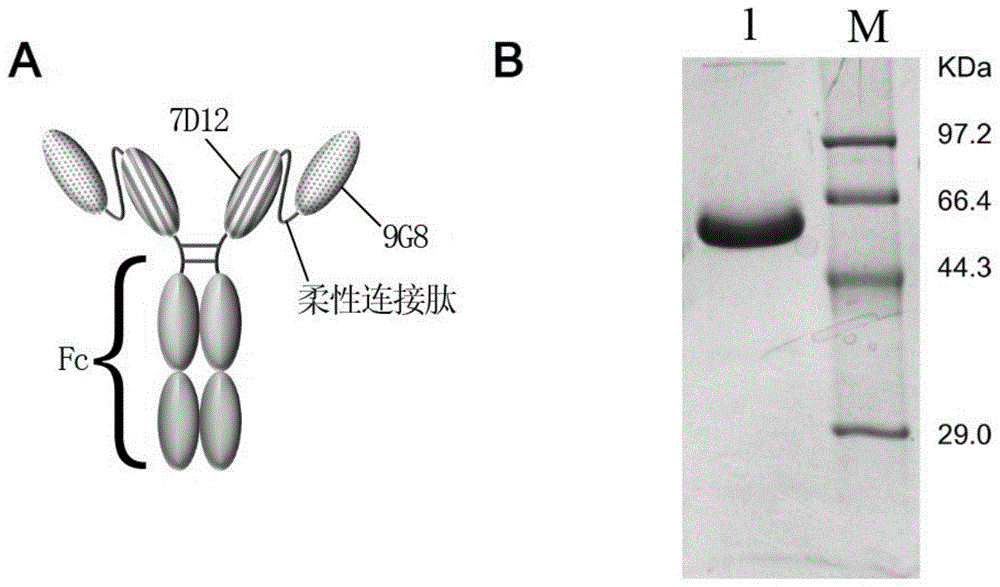
图1为融合蛋白的示意图以及sds-page分析纯化后融合蛋白的还原状态的结果,其中a:融合蛋白的示意图;b:sds-page分析纯化后融合蛋白的还原状态的结果,泳道1为融合蛋白,泳道m为标准分子量蛋白marker。
图2为融合蛋白或纳米抗体与西妥昔单抗的竞争性elisa结果。
图3为融合蛋白及西妥昔单抗对高表达egfr的a431细胞的亲和曲线。
图4为融合蛋白及西妥昔单抗对高表达s492r突变egfr的nih-3t3细胞的亲和。
图5为融合蛋白对高表达各类egfr突变的nih-3t3细胞的亲和。
图6为融合蛋白与西妥昔单抗结合a431细胞表面egfr后的内吞效率图。
图7为融合蛋白与西妥昔单抗对高表达野生型egfr以及s492regfr的293t的内吞降解。
图8为融合蛋白与西妥昔单抗对a431和sw48细胞增殖抑制效果。
图9为融合蛋白与西妥昔单抗对转染egfr的nih-3t3细胞的cdc杀伤效果图。
图10为多剂量融合蛋白给药后的balb/cnude鼠体内肿瘤生长曲线图。
图11为多剂量融合蛋白给药后的balb/cnude鼠体重变化曲线图。
图12为单剂量融合蛋白给药后的balb/cnude鼠体内肿瘤生长曲线图。
图13为单剂量融合蛋白给药后的balb/cnude鼠体重变化曲线图。
具体实施方式
实施例1
融合蛋白的制备。
以egfr为靶点的纳米抗体fc融合蛋白,7d12和9g8的纳米抗体融合而成,纳米抗体的序列来自文献(karlr.schmitz等,structure.2013;21(7):1214–1224),以下简称为97m。融合蛋白的fc段序列为实验室保存。本实施例中通过pcr法将7d12和9g8通过柔性连接肽(ggggs)3相连,c末端连接igg1型重链恒定区,最终得到dna序列。
序列如下:
9g8:氨基酸序列如seqidno.1所示,基因序列如seqidno.2所示。
7d12:氨基酸序列如seqidno.3所示,基因序列如seqidno.4所示。
柔性连接肽:氨基酸序列如seqidno.5所示,基因序列如seqidno.6所示。
igg1型重链恒定区(fc):氨基酸序列如seqidno.7所示,基因序列如seqidno.8所示。
97m:氨基酸序列如seqidno.9所示,基因序列如seqidno.10所示。
以构建好的dna序列为模板,通过pcr法将构建好的基因序列连接至pmh3表达载体中,将构建好的表达载体转染至人胚肾细胞hek293,收集培养液上清,经蛋白a亲和柱纯化后得到融合蛋白。图1a为融合蛋白的示意图,两种纳米抗体通过串联方式连接。
用sds-page分析还原状态下的融合蛋白,如图1b所示,在还原条件下,条带略高于55kd,考虑到fc段的糖基化影响,其条带和理论分子量110kd相符。
实施例2
融合蛋白与西妥昔单抗的elisa竞争性亲和分析。
通过在96孔板中包被egfr抗原来检测不同抗体之间的竞争性亲和。具体操作流程如下:首先包被稀释fc标签的egfr抗原至1μg/ml,然后按100μl/孔的添加量加入96孔板,放入37℃孵箱包被2h。弃去96孔板中的溶液,用pbst溶液清洗96孔板3次后,在96孔板中加入elisa封闭液200μl,封闭酶标板孔内未被抗原结合的部位,在37℃孵箱封闭2h。添加稀释后浓度梯度为0.014nm至30nm的西妥昔抗体,同时另外添加终浓度为10nm的7d12或9g8纳米抗体,终浓度5nm的97m作为对照组。其中9g8和7d12纳米抗体是通过将dna序列连接至原核表达载体pet21b,并转化rosetta大肠杆菌后表达纯化得到。继续孵育抗体1h后用pbst溶液清洗96孔板3次。最后用封闭液稀释hrp标记的山羊抗人kappa轻链,然后按100μl/孔的添加量加入96孔板,在37℃孵箱放置45min。再次洗板以洗去未结合的二抗后加入显色液显色。待显色反应20min后,加入终止液终止显色反应。将96孔酶标板放入酶标仪中读取od450的值,记录读数。结果如图2所示,同时添加5nm97m后的竞争性结合效果要明显高于单独添加10nm9g8或者7d12的组,证明97m自身对称二聚的结构能更有效的亲和egfr的二聚体形式。
实施例3
流式亲和力检测。
表达egfr的细胞如a431用含双抗的dmem培养基进行培养,待细胞生长至对数生长期后手机细胞,将细胞按5×105个的细胞数与一系列梯度稀释的抗体或者adc在1%bsa的pbs溶液中冰上共孵育30min。
孵育结束后,用预冷的pbs洗涤细胞两遍以除去非特异性吸附至细胞表面的蛋白。最后,在避光环境下加入1∶200稀释的fitc标记山羊抗人igg二抗,继续置于冰上避光孵育30min。孵育结束后用预冷的pbs清洗细胞,将pbs重悬后的细胞悬液上流式细胞分析仪检测细胞的平均荧光强度(mfi)。流式细胞分析仪检测融合蛋白与egfr阳性细胞的亲和,mfi信号的强弱代表结合力强弱的变化。结果如图3所示,97m的亲和曲线趋势和西妥昔接近,在100nm及以上的饱和孵育浓度下97m的荧光信号和西妥昔接近,而在12.5nm到50nm的孵育浓度下,97m的荧光信号低于西妥昔。
实施例4
对于含egfr胞外区点突变细胞的流式亲和力检测。
以野生型egfr全长基因为模板,通过pcr引入s492r,k489e,k467t,g465r以及i491m点突变,将带胞外点突变egfr全长的质粒分别转染nih-3t3细胞,挑选转染效率高的单克隆,最后得到表面高表达带各类点突变egfr的nih-3t3细胞。将消化收集的细胞悬液在4℃下以600g离心5min,并用pbs重悬以后再次离心两遍。用含1%bsa的pbs将融合蛋白或西妥昔单抗稀释10μg/ml,取200μl重悬离心管中的细胞,在冰上孵育至少30min以使抗体结合至细胞表面的抗原。孵育结束后用预冷的pbs如前述清洗细胞二次以除去非特异性吸附至细胞表面的抗体。最后在避光环境下加入一定比例稀释的fitc标记山羊抗人igg二抗(碧云天)或pe标记山羊抗人fc抗体(abcam),继续置于冰上避光孵育30min。孵育结束后用预冷的pbs清洗细胞,将pbs重悬后的细胞悬液上机。结果如图4所示,将西妥昔或97m和构建好的表面表达s492r突变型egfr的nih3t3细胞共孵育,经流式细胞检测细胞表面的荧光信号可知,该突变对西妥昔的亲和影响非常大,几乎使西妥昔完全丧失了亲和力,但是97m仍然能够保留亲和力。图5则是包含这些耐药性点突变的nih-3t3细胞的亲和,由图可知,97m可以亲和含这一系列胞外点突变的egfr细胞。
实施例5
内吞效率检测。
用胰酶小心消化培养皿中的a431细胞,收集细胞后按所需样品数分装成数量相同的两份。用含1%bsa的pbs稀释后的待测抗体重悬离心管中的细胞,在冰上孵育30min。用预冷pbs洗两次去除未结合在细胞表面的抗体后,重悬细胞。一份置于4℃,另一份置于37℃。待静置时间达到内吞检测所需时间后,将所有待测细胞转移至冰上,在避光环境下加入1:200稀释的fitc标记山羊抗人igg二抗,继续置于冰上避光孵育30min。孵育结束后用预冷的pbs清洗细胞,将pbs重悬后的细胞悬液上机检测细胞表面的平均荧光强度值。细胞表面结合抗体的内吞效率通过计算37℃下孵育待测抗体后相比4℃下孵育待测抗体的平均荧光强度的降低百分比计算得到。结果图6所示,融合蛋白无论在饱和浓度还是非饱和浓度下相比西妥昔均表现更好的egfr的内吞效率,内吞效率由20%至30%之间提升至70%至80%之间。
实施例6
内吞降解。
待培养至所需细胞数后,用胰酶小心消化培养皿中的细胞。收集消化后的细胞,并按照2.5×105个/ml的细胞密度铺于6孔细胞培养板中,放入37℃含5%co2的细胞培养箱中过夜,直至细胞贴壁,生长状态良好。第二天吸去培养板中的培养基,更换为含不同浓度抗体药物的新鲜完全培养基,其中设置对照孔,添加不含药物的完全培养基,继续放回37℃含5%co2的细胞培养箱中孵育48h。用胰酶小心消化后收集每孔中的待测细胞至离心管,用预冷的pbs洗涤细胞两遍后,轻柔吹打细胞悬液,离心管中添加300μlpbs后流式上机检测,根据最后细胞的荧光信号的数值。其中,只添加不含药物的完全培养基的细胞样品作为分母,添加含不同浓度抗体的完全培养基的细胞样品作为分子,计算egfr内吞降解的百分比。结果如图7所示,97m在转染了野生型egfr的293t细胞中,其内吞降解情况在20nm以上的浓度情况明显高于西妥昔,这表明其内吞进入细胞后的最大降解率也明显高于西妥昔。在转染了s492r突变型egfr的293t细胞中,由于西妥昔会受到s492r突变位点的影响,其亲和力较弱,因此,西妥昔所造成的egfr内吞降解也弱,只有在30nm以上的较高孵育浓度下,才会有比较微弱的内吞降解现象。
实施例7
细胞增殖抑制。
待培养至所需细胞数后,用胰酶消化离心并收集细胞。用含1%血清的培养基重悬细胞,将细胞按5000个/孔的铺板密度添加至96孔板中,每孔体积为100μl。将96孔板置于5%co2,37℃细胞培养箱中过夜。待第二天,将待测抗体也用含1%血清的培养基进行一系列的浓度梯度稀释,并且设置无细胞的空白培养基孔作为背景对照,设置含细胞但无抗体的孔作为阴性对照。按100μl的体积添加至96孔板中,继续放回37℃细胞培养箱中培养4天。取出96孔板,弃去板内的培养基,并按每孔100μl的体积添加含10%cck8试剂的培养基,放回37℃细胞培养箱中等待显色。待显色至所需时间后,将96孔酶标板放入酶标仪中读取od450的值,记录读数。结果如图8所示,随着孵育浓度的增加,融合蛋白97m对a431细胞的增殖抑制效果明显强于西妥昔,从12.5nm开始97m的抑制率已经达到20%,而西妥昔要在浓度高于50nm时才会有将近10%的抑制率。在西妥昔敏感型的sw48细胞中,97m和西妥昔的的细胞增殖抑制效果均较好,最大抑制率大于60%,虽然两者的抑制效果没有表现出明显的差异,但是97m的细胞增殖抑制效果略强于西妥昔。
实施例8
补体依赖的杀伤活性。
将带野生型egfr的质粒转染nih-3t3细胞,挑选转染效率高的单克隆,最后得到表面高表达egfr的nih-3t3细胞,命名为3t3-egfr。重悬细胞并调整细胞浓度为1×105/ml,在96孔板中以0.1ml/孔的量铺板。以50μl/孔的添加量,在样品孔中加入梯度稀释后的融合蛋白或西妥昔单抗。阴性对照孔则加同样体积的完全培养基。另外设置不加细胞的空白孔作为背景,设3个复孔。最后样品孔,阴性对照孔和空白孔均添加50μl/孔的人血清作为补体来源并混匀,保证人血清的最终比例为20%。将96孔板置于37℃含5%二氧化碳的细胞培养箱中培养1~4h,每孔添加含10%cck-8试剂的rpmi-1640培养基100μl待测。细胞培养板置于37℃含5%二氧化碳培养箱中培养等待显色,待显色至所需时间后,将96孔酶标板放入酶标仪中读取od450的值,记录读数。结果如图9所示,97m表现出较高的杀伤效果,杀伤率均高于60%,而西妥昔的杀伤效果较弱。
实施例9
多剂量给药后的体内生物学活性检测。
采用balb/cnude雌性裸小鼠构建移植瘤模型。收集处于对数生长期的a431肿瘤细胞,用预冷的pbs重悬细胞。将a431细胞悬液以4×106每只的密度接种至6-8周雌性裸鼠的右腋下。待裸鼠的肿瘤体积为100mm3至150mm3时,随机将裸鼠分为2组,每组为6只,分别为pbs空白对照组,97m6mg/kg。调整各药物浓度,按上述给药剂量各组裸鼠尾静脉注射给药100μl。实验时间自第一次给药开始计算,每四天给药一次,共给药四次,同时每两至三天测量裸鼠的体重和肿瘤的大小。肿瘤体积的具体计算公式为:v=(l×w2)/2。其中v为肿瘤体积(mm3),l为测得的肿瘤长度(mm),w为测得的肿瘤宽度(mm)。结果如图10所示,相比阴性对照pbs组,6mg/kg剂量给药的97m组能显著抑制肿瘤生长。图11为实验过程中小鼠的体重数据,可以观察到在整个实验周期里各组荷瘤小鼠的体重并没有表现出明显的下降,整体体重呈现略微增加的趋势,说明在实验周期内,经多剂量给药后没有对小鼠产生明显的副作用。
实施例10
单剂量给药后的体内生物学活性检测。
采用balb/cnude雌性裸小鼠构建移植瘤模型。收集处于对数生长期的a431肿瘤细胞,用预冷的pbs重悬细胞。将a431细胞悬液以4×106每只的密度接种至6-8周雌性裸鼠的右腋下。待裸鼠的肿瘤体积为100mm3至150mm3时,随机将裸鼠分为3组,每组为5只,分别为pbs空白对照组,97m6mg/kg,97m2mg/kg。调整各药物浓度,按上述给药剂量各组裸鼠尾静脉注射给药100μl。实验时间自第一次给药开始计算,每两至三天测量裸鼠的体重和肿瘤的大小。肿瘤体积的具体计算公式为:v=(l×w2)/2。其中v为肿瘤体积(mm3),l为测得的肿瘤长度(mm),w为测得的肿瘤宽度(mm)。结果如图12所示,在单剂量给药的情况下,6mg/kg的单剂量97m给药后,小鼠的肿瘤体积在前两周内增长趋势较缓,两周后的增长趋势和2mg/kg的低剂量组相同。单剂量给药后,高低剂量给药组的肿瘤生长趋势相较pbs组均显缓慢,证明在单剂量给药情况下,融合蛋白仍然表现出一定的抗肿瘤活性。图13则是实验过程中各组小鼠的体重数据,整个实验周期里各组荷瘤小鼠的体重整体均呈现增加的趋势,说明融合蛋白在实验周期内都没有对小鼠产生严重的毒副作用。
序列表
<110>浙江大学
<120>一种融合蛋白、基因及应用
<160>10
<170>siposequencelisting1.0
<210>1
<211>127
<212>prt
<213>人工序列(artificialsequence)
<400>1
gluvalglnleuvalgluserglyglyglyleuvalglnalaglygly
151015
serleuargleusercysalaalaserglyargthrphesersertyr
202530
alametglytrppheargglnalaproglylysgluargglupheval
354045
valalaileasntrpserserglyserthrtyrtyralaaspserval
505560
lysglyargphethrileserargaspasnalalysasnthrmettyr
65707580
leuglnmetasnserleulysprogluaspthralavaltyrtyrcys
859095
alaalaglytyrglnileasnserglyasntyrasnphelysasptyr
100105110
glutyrasptyrtrpglyglnglythrglnvalthrvalserser
115120125
<210>2
<211>381
<212>dna
<213>人工序列(artificialsequence)
<400>2
gaggtgcagctggtggagtccggcggcggactggtgcaggctggaggatccctgcggctg60
tcctgtgctgccagcggcagaaccttctccagctatgccatgggctggtttaggcaggcc120
cctggcaaggagcgggagtttgtggtggctatcaactggtcctccggcagcacctactac180
gctgactccgtgaagggccggttcaccatcagcagggacaacgccaagaacaccatgtac240
ctgcagatgaactccctgaagcctgaggataccgctgtgtactactgcgctgccggctat300
cagatcaatagcggcaactataacttcaaggactacgagtacgattattggggccagggc360
acccaggtgaccgtgtcctcc381
<210>3
<211>124
<212>prt
<213>人工序列(artificialsequence)
<400>3
glnvallysleuglugluserglyglyglyservalglnthrglygly
151015
serleuargleuthrcysalaalaserglyargthrserargsertyr
202530
glymetglytrppheargglnalaproglylysgluargglupheval
354045
serglyilesertrpargglyaspserthrglytyralaaspserval
505560
lysglyargphethrileserargaspasnalalysasnthrvalasp
65707580
leuglnmetasnserleulysprogluaspthralailetyrtyrcys
859095
alaalaalaalaglyseralatrptyrglythrleutyrglutyrasp
100105110
tyrtrpglyglnglythrglnvalthrvalserser
115120
<210>4
<211>372
<212>dna
<213>人工序列(artificialsequence)
<400>4
caggtgaagctggaggagtctggcggcggctctgtgcagaccggcggatctctgagactg60
acatgcgctgcttctggaagaacatctagatcctatggcatgggctggtttagacaggct120
cctggaaaggagagagagtttgtgtctggaatttcttggagaggagattctactggctac180
gctgattctgtgaaaggaagattcacaatttctagagataatgccaagaataccgtggac240
ctgcagatgaactctttgaagcctgaagatacagctatctactactgtgctgctgctgcc300
ggctctgcttggtatggaaccctgtatgaatatgattattggggacagggcacacaggtg360
accgtgtcttct372
<210>5
<211>15
<212>prt
<213>人工序列(artificialsequence)
<400>5
glyglyglyglyserglyglyglyglyserglyglyglyglyser
151015
<210>6
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>6
ggtggtggcggttcaggcggaggtggctctggcggtggcggatcg45
<210>7
<211>232
<212>prt
<213>人工序列(artificialsequence)
<400>7
gluprolyssercysasplysthrhisthrcysproprocysproala
151015
progluleuleuglyglyproservalpheleupheproprolyspro
202530
lysaspthrleumetileserargthrprogluvalthrcysvalval
354045
valaspvalserhisgluaspprogluvallyspheasntrptyrval
505560
aspglyvalgluvalhisasnalalysthrlysproarggluglugln
65707580
tyrasnserthrtyrargvalvalservalleuthrvalleuhisgln
859095
asptrpleuasnglylysglutyrlyscyslysvalserasnlysala
100105110
leuproalaproileglulysthrileserlysalalysglyglnpro
115120125
argargproglnvaltyrthrleuproproserargasngluleuthr
130135140
lysasnglnvalserleuthrcysleuvallysglyphetyrproser
145150155160
aspilealavalglutrpgluserasnglyglnprogluasnasntyr
165170175
lysthrthrproprovalleuaspseraspglyserphepheleutyr
180185190
serlysleuthrvalasplysserargtrpglnglnglyasnvalphe
195200205
sercysservalmethisglualaleuhisasnhistyrthrglnlys
210215220
serleuserleuserproglylys
225230
<210>8
<211>699
<212>dna
<213>人工序列(artificialsequence)
<400>8
gagcccaaatcttgtgacaaaactcacacatgcccaccgtgcccagcacctgaactcctg60
gggggaccgtcagtcttcctcttccccccaaaacccaaggacaccctcatgatctcccgg120
acccctgaggtcacatgcgtggtggtggacgtgagccacgaagaccctgaggtcaagttc180
aactggtacgtggacggcgtggaggtgcataatgccaagacaaagccgcgggaggagcag240
tacaacagcacgtaccgtgtggtcagcgtcctcaccgtcctgcaccaggactggctgaat300
ggcaaggagtacaagtgcaaggtctccaacaaagccctcccagcccccatcgagaaaacc360
atctccaaagccaaagggcagccccgagaaccacaggtgtacaccctgcccccatcccgg420
gaggagatgaccaagaaccaggtcagcctgacctgcctggtcaaaggcttctatcccagc480
gacatcgccgtggagtgggagagcaatgggcagccggagaacaactacaagaccacgcct540
cccgtgctggactccgacggctccttcttcctctacagcaagctcaccgtggacaagagc600
aggtggcagcaggggaacgtcttctcatgctccgtgatgcatgaggctctgcacaaccac660
tacacgcagaagagcctctccctgtctccgggtaaatga699
<210>9
<211>498
<212>prt
<213>人工序列(artificialsequence)
<400>9
gluvalglnleuvalgluserglyglyglyleuvalglnalaglygly
151015
serleuargleusercysalaalaserglyargthrphesersertyr
202530
alametglytrppheargglnalaproglylysgluargglupheval
354045
valalaileasntrpserserglyserthrtyrtyralaaspserval
505560
lysglyargphethrileserargaspasnalalysasnthrmettyr
65707580
leuglnmetasnserleulysprogluaspthralavaltyrtyrcys
859095
alaalaglytyrglnileasnserglyasntyrasnphelysasptyr
100105110
glutyrasptyrtrpglyglnglythrglnvalthrvalsersergly
115120125
glyglyglyserglyglyglyglyserglyglyglyglyserglnval
130135140
lysleuglugluserglyglyglyservalglnthrglyglyserleu
145150155160
argleuthrcysalaalaserglyargthrserargsertyrglymet
165170175
glytrppheargglnalaproglylysgluarggluphevalsergly
180185190
ilesertrpargglyaspserthrglytyralaaspservallysgly
195200205
argphethrileserargaspasnalalysasnthrvalaspleugln
210215220
metasnserleulysprogluaspthralailetyrtyrcysalaala
225230235240
alaalaglyseralatrptyrglythrleutyrglutyrasptyrtrp
245250255
glyglnglythrglnvalthrvalsersergluprolyssercysasp
260265270
lysthrhisthrcysproprocysproalaprogluleuleuglygly
275280285
proservalpheleupheproprolysprolysaspthrleumetile
290295300
serargthrprogluvalthrcysvalvalvalaspvalserhisglu
305310315320
aspprogluvallyspheasntrptyrvalaspglyvalgluvalhis
325330335
asnalalysthrlysproargglugluglntyrasnserthrtyrarg
340345350
valvalservalleuthrvalleuhisglnasptrpleuasnglylys
355360365
glutyrlyscyslysvalserasnlysalaleuproalaproileglu
370375380
lysthrileserlysalalysglyglnproargargproglnvaltyr
385390395400
thrleuproproserargasngluleuthrlysasnglnvalserleu
405410415
thrcysleuvallysglyphetyrproseraspilealavalglutrp
420425430
gluserasnglyglnprogluasnasntyrlysthrthrproproval
435440445
leuaspseraspglyserphepheleutyrserlysleuthrvalasp
450455460
lysserargtrpglnglnglyasnvalphesercysservalmethis
465470475480
glualaleuhisasnhistyrthrglnlysserleuserleuserpro
485490495
glylys
<210>10
<211>1497
<212>dna
<213>人工序列(artificialsequence)
<400>10
gaggtgcagctggtggagtccggcggcggactggtgcaggctggaggatccctgcggctg60
tcctgtgctgccagcggcagaaccttctccagctatgccatgggctggtttaggcaggcc120
cctggcaaggagcgggagtttgtggtggctatcaactggtcctccggcagcacctactac180
gctgactccgtgaagggccggttcaccatcagcagggacaacgccaagaacaccatgtac240
ctgcagatgaactccctgaagcctgaggataccgctgtgtactactgcgctgccggctat300
cagatcaatagcggcaactataacttcaaggactacgagtacgattattggggccagggc360
acccaggtgaccgtgtcctccggtggtggcggttcaggcggaggtggctctggcggtggc420
ggatcgcaggtgaagctggaggagtctggcggcggctctgtgcagaccggcggatctctg480
agactgacatgcgctgcttctggaagaacatctagatcctatggcatgggctggtttaga540
caggctcctggaaaggagagagagtttgtgtctggaatttcttggagaggagattctact600
ggctacgctgattctgtgaaaggaagattcacaatttctagagataatgccaagaatacc660
gtggacctgcagatgaactctttgaagcctgaagatacagctatctactactgtgctgct720
gctgccggctctgcttggtatggaaccctgtatgaatatgattattggggacagggcaca780
caggtgaccgtgtcttctgagcccaaatcttgtgacaaaactcacacatgcccaccgtgc840
ccagcacctgaactcctggggggaccgtcagtcttcctcttccccccaaaacccaaggac900
accctcatgatctcccggacccctgaggtcacatgcgtggtggtggacgtgagccacgaa960
gaccctgaggtcaagttcaactggtacgtggacggcgtggaggtgcataatgccaagaca1020
aagccgcgggaggagcagtacaacagcacgtaccgtgtggtcagcgtcctcaccgtcctg1080
caccaggactggctgaatggcaaggagtacaagtgcaaggtctccaacaaagccctccca1140
gcccccatcgagaaaaccatctccaaagccaaagggcagccccgagaaccacaggtgtac1200
accctgcccccatcccgggaggagatgaccaagaaccaggtcagcctgacctgcctggtc1260
aaaggcttctatcccagcgacatcgccgtggagtgggagagcaatgggcagccggagaac1320
aactacaagaccacgcctcccgtgctggactccgacggctccttcttcctctacagcaag1380
ctcaccgtggacaagagcaggtggcagcaggggaacgtcttctcatgctccgtgatgcat1440
gaggctctgcacaaccactacacgcagaagagcctctccctgtctccgggtaaatga1497
- 还没有人留言评论。精彩留言会获得点赞!