一种乳腺癌预后风险预测标志组合物及应用
1.本发明涉及基因技术和医学领域,特别涉及一种乳腺癌预后风险预测标志组合物及应用。
背景技术:
2.乳腺癌是女性中最高发的癌症,也是女性癌症死亡的主要原因。由于早期筛查和治疗方法的进步,乳腺癌的治疗取得了一定成效,但由于疾病的异质性,预后评估仍然面临挑战。尤其是随着乳腺癌的发病率和死亡率不断增加,且具有年轻化的趋势,现阶段乳腺癌患者的疗效及预后情况仍不乐观。因此,及时监测患者的预后尤为重要。影响乳腺癌预后的因素众多,目前尚缺乏治疗敏感和预后判断的特异性的分子生物标志物。因此,建立准确预测乳腺癌患者后的工具对于指导临床诊断治疗十分关键。
3.抑郁与乳腺癌的发生发展密切相关,但临床极易被忽视和低估。乳腺癌患者在诊治的过程中,长期处于慢性应激状态,其情绪可能由最初的对癌症的恐惧、怀疑,转变为焦虑、紧张、急躁、悲观、抑郁等心理障碍,甚至产生自杀行为,促使病情进一步恶化。抑郁可通过影响神经、内分泌系统,降低免疫功能,增加肿瘤的复发和转移和死亡风险。动物实验结果也证实慢性心理应激可以促进小鼠乳腺肿瘤的生长以及肺转移。
4.近年来,多项研究证实了基因在肿瘤预后中的预测作用,进行从分子层面对癌症进行研究更有利于个体化的治疗和预后的评估。目前乳腺癌预后预测工具在不断开发出来,例如21基因检测、乳腺癌指数等。美国国家癌症网络(nccn)指南推荐乳腺癌21基因检测适用于:ⅰ期或ⅱ期、er阳性、淋巴结阴性的新确诊乳腺癌;淋巴结阳性(1~3个)、er阳性的绝经后浸润性乳腺癌患者。可以看出目前现有的乳腺癌预后标志物仅适用于特定的乳腺癌,具有一定的局限性。因此,发现新的预后标志、构建新的预测模型仍有助于乳腺癌患者的预后判断和治疗方法的选择。
技术实现要素:
5.本发明的首要目的在于克服现有技术的缺点与不足,提供一种乳腺癌预后风险预测标志组合物。
6.本发明的另一目的在于提供所述乳腺癌预后风险预测标志组合物的应用。
7.本发明的目的通过下述技术方案实现:
8.一种乳腺癌预后风险预测标志组合物,所述的标志组合物包括mt3、sorbs1、igfals、amh、il12b、tp53aip1、pxdnl、mc5r、foxd1和lhx1基因。
9.所述的mt3、sorbs1、igfals、amh、il12b、tp53aip1、pxdnl、mc5r、foxd1和lhx1基因的ncbi登录号如下:mt3:nm_005954;sorbs1:ng_034041;igfals:ng_011778;amh:ng_012190;il12b:ng_009618;tp53aip1:ng_030401;pxdnl:nm_144651;mc5r:nm_005913;foxd1:nm_004472;lhx1:nm_005568。
10.所述的乳腺癌预后风险预测标志组合物在制备乳腺癌预后风险预测和/或诊断产
品(工具)中的应用。
11.所述的产品包括试剂、试剂盒等。
12.所述的乳腺癌预后风险预测标志组合物中的各个基因的表达水平可以通过本领域常规方法进行测定,如采用实时荧光定量法测定每个基因的mrna表达数据。
13.用于检测所述的乳腺癌预后风险预测标志组合物的表达水平的试剂在制备乳腺癌预后风险预测和/或诊断产品中的应用。
14.所述的乳腺癌预后风险预测标志组合物在构建乳腺癌患者预后死亡风险预测模型中的应用。
15.一种乳腺癌患者预后死亡风险预测模型的构建方法,包括如下步骤:
16.(1)获得基因表达水平
17.选择n个乳腺癌患者作为参照样本,从第1个参照样本开始,依次获得第1个乳腺癌患者肿瘤患者的mt3、sorbs1、igfals、amh、il12b、tp53aip1、pxdnl、mc5r、foxd1和lhx1基因的表达水平,即获得每个基因的表达值;然后将每个基因的表达值经z
‑
score标准化后,得到每个基因的标准化表达值,依次记为xi1‑1、xi2‑1……
xi
10
‑1;以此类推,分别获得第2个至第n个参照样本的基因表达水平,记为xi1‑2、xi2‑2……
xi
10
‑2,xi1‑3、xi2‑3……
xi
10
‑3,xi1‑
n
、xi2‑
n
……
xi
10
‑
n
;其中,n≥200(为正整数);
18.(2)建立预测模型
19.根据如下公式计算得到第1个乳腺癌患者的风险分数:风险分数r1=(
‑
0.160)*xi1‑1+(
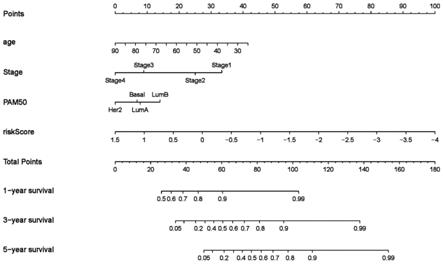
‑
0.129)*xi2‑1+(
‑
0.084)*xi3‑1+0.089*xi4‑1+(
‑
0.137)*xi5‑1+(0.164)*xi6‑1+0.137*xi7‑1+0.120*xi8‑1+0.073*xi9‑1+0.055*xi
10
‑1;以此类推,分别计算得到第2个至第n个乳腺癌患者的风险分数r2,r3……
r
n
;
20.(3)确定截断值
21.以步骤(1)中的n个乳腺癌患者5年内死亡与否为标准,以1代表死亡,0代表存活,并根据步骤(2)中计算得到的风险分数r1,r2……
r
n
分别绘制roc曲线(n个乳腺癌患者,相应绘制n条roc曲线),再根据绘制的roc曲线获得截断值;
22.(4)预后死亡风险判断
23.参考步骤(1)中的方法,先分别测定待预测乳腺癌患者(作为待测样本)的mt3、sorbs1、igfals、amh、il12b、tp53aip1、pxdnl、mc5r、foxd1和lhx1基因的表达水平,获得待预测乳腺癌患者的每个基因的表达值;将每个基因的表达值经z
‑
score标准化后,得到每个基因的标准化表达值;然后根据步骤(2)中的公式计算得到待预测乳腺癌患者的风险分数,再根据待预测乳腺癌患者的风险分数以及步骤(3)确定的截断值大小预测乳腺癌患者的预后死亡风险:风险分数高于或等于截断值的预测为高风险人群,风险分数低于截断值的预测为低风险人群。
24.步骤(1)中所述的参照样本(即n个乳腺癌患者)可通过本领域常规的方式获取,如从医院等获取足够数量(n个)的乳腺癌患者作为参照样本,或可以从现有的数据库中获取足够数量的乳腺癌患者的数据作为参照样本;优选为从tcga数据库中获取的乳腺癌患者的数据作为参照样本。
25.步骤(1)中所述的z
‑
score标准化(零
‑
均值规范化)(经过处理的数据的均值为0,标准
26.差为1)的转化公式为:标准化表达值;其中,x为实际测量值,为原始数据的均
27.值,σ为原始数据的标准差。
28.步骤(1)中所述的n的取值范围优选为:n≥500;更优选为:n≥1000;参照样本数量越多,越有利获得更精确的截断值,也就是对待预测乳腺癌患者的预后死亡风险的预测会更准确。
29.步骤(3)中所述的绘制roc曲线优选为采用medcalc软件进行绘制。
30.步骤(3)中的截断值为最佳截断值,根据约登指数最大原则,取约登指数最大时对应风险分数的数值为截断值。
31.步骤(4)中所述的基因(乳腺癌患者肿瘤样本的mt3、sorbs1、igfals、amh、il12b、tp53aip1、pxdnl、mc5r、foxd1和lhx1基因)表达水平可以通过本领域常规方法进行测定,如采用实时荧光定量法测定每个基因的mrna表达数据。
32.所述的乳腺癌患者预后死亡风险预测模型的构建方法,在步骤(4)之后还包括如下步骤:
33.(5)生存率判断
34.使用r语言中的rms包根据步骤(1)中的n个乳腺癌患者的年龄、病理学分期、分子分型和风险分数绘制生存率预测列线图(多因素cox回归分析构建列线图),并以此计算出待预测乳腺癌患者的一年、三年、五年生存率。
35.本发明相对于现有技术具有如下的优点及效果:
36.(1)本发明基于与抑郁相关的基因,筛选出了10个与乳腺癌生存率密切相关的差异表达基因mt3、sorbs1、igfals、amh、il12b、tp53aip1、pxdnl、mc5r、foxd1、lhx1,可作为在肿瘤中检测且具有较高预测准确度的乳腺癌预后的分子标记物。
37.(2)本发明基于这10个与乳腺癌生存率密切相关的差异表达基因建立了预测乳腺癌预后的风险评估模型,这10基因风险模型在预测乳腺癌患者总生存率表现出良好的性能;同时,通过验证风险模型对不同分子亚型的乳腺癌患者的预测准确性,发现此模型对于basal型和luma型的患者具有更好的预测效用,为乳腺癌癌患者预后预测提供以一种有效的预测工具。
38.(3)本发明中的roc曲线评估预测的效用结果表明,相较于常见的tnm分期和病理学分期,本发明中构建的预测模型具有更好的预测效用,并且是乳腺癌患者生存预测的独立预后指标,有利于筛选高危群体,为指导临床工作者制定个体化治疗方案提供新的思路。
39.(4)本发明通过将风险评分与其他临床指标相结合,构建了预测乳腺癌生存率的列线图。本发明通过基因表达的数据结合临床资料分析,可提高风险预测的性能。并且通过提供简单、直观和定量的预后判断,有助于临床医生预测乳腺癌患者总体生存率,指导临床医师进行治疗决策。
附图说明
40.图1是与乳腺癌预后相关的差异抑郁基因的筛选结果图;其中,a为乳腺癌差异表达基因火山图;b为乳腺癌差异表达基因热图;c为mcode模块分析蛋白互作网络中的关键基
因;d为单因素cox回归筛选与生存率相关的基因;e为lasso回归分析(表示通过lasso模型十折交叉验证的最低标准选择最佳参数(λ);横坐标为log(lambda),纵坐标为部分似然偏差)。
41.图2是乳腺癌患者高、低风险组的kaplan
‑
meier生存曲线以及1年、3年和5年生存率的时间依赖性roc曲线图;其中,a为测试集tcga以及验证集gse96058中乳腺癌患者高、低风险组的kaplan
‑
meier生存曲线(横坐标为年数,纵坐标为生存概率,红色代表高风险,蓝色代表低风险);b为测试集tcga以及验证集gse96058中1年、3年和5年生存率的时间依赖性roc曲线(横坐标为1
‑
特异性,也称为假阳性率,纵坐标为敏感度,也称为真阳性率)。
42.图3是测试集tcga以及验证集gse96058风险预测模型在不同亚型乳腺癌患者的roc曲线图(横坐标为1
‑
特异性,也称为假阳性率;纵坐标为敏感度,也称为真阳性率);其中,a为basal型;b为luma型;c为lumb型;d为her
‑
2型。
43.图4是多因素cox回归分析评估风险预测模型在乳腺癌患者的独立预后价值图(分别对年龄、tnm分期、病理学分期、风险分数进行多因素cox回归分析;年龄:hr=1.030,95%ci:1.014
‑
1.045,p<0.001;病理学分期:hr=1.618,95%ci:0.954
‑
2.742,p=0.074;tnm分期(t:hr=0.961,95%ci:0.700
‑
1.319,p=0.805;n:hr=1.270,95%ci:0.941
‑
1.716,p=0.119;m:hr=0.956,95%ci:0.409
‑
2.235,p=0.0.918)和风险模型:hr=2.671,95%ci:2.073
‑
3.411,p<0.001)。
44.图5是roc曲线分析比较风险预测模型、tnm分期、病理分期和年龄在乳腺癌患者的预测效能图。
45.图6是包含年龄、病理学分期、分子分型和风险评分的列线图。
具体实施方式
46.下面结合实施例对本发明作进一步详细的描述,但本发明的实施方式不限于此。应当指出,对于本技术领域的普通技术人员,在不脱离本发明方法的前提下,还可以做出若干改进和补充,这些改进和补充也应视为本发明的保护范围。
47.实施例1基于抑郁相关基因的乳腺癌预后风险预测模型建立及在预测乳腺癌患者预后方面的应用
48.(1)从癌症基因组图谱(tcga)数据库中收集乳腺癌患者的mrna表达数据矩阵及临床信息,该矩阵包括1096个肿瘤样本和113个正常样本,作为测试数据集(测试集);从geo数据库的gse96058数据集中收集2969名乳腺癌患者的mrna谱和临床特征,作为验证数据集(验证集)。
49.(2)筛选差异表达基因:在genecard数据库中以“depression”or“depressive”为关键词进行检索,得到抑郁相关基因集(8479个基因)。采用wilcox秩和检验筛选乳腺癌样本和正常样本中的与抑郁相关的差异表达基因,设置阈值|logfoldchange|=2,padj=0.01,见图1a和1b。
50.(3)采用string数据库获取差异基因的蛋白相互作用信息,导入cytoscape构建蛋白互作网络,并利用mcode进行模块分析(图1c),获得网络的关键基因。
51.(4)预后标志物的筛选:下载患者样本的临床信息,并整理患者的生存时间和生存状态。纳入随访时间大于或等于1个月的1027名患者(1027位患者为上述tcga数据集1096个
样本中的随访数据,且为随访时间大于或等于1个月的患者的数据),对于筛选出的关键差异表达的mrna,采用单因素cox回归模型分析与患者总生存率相关的关键基因,筛选条件为p<0.05,获得34个基因,见图1d。
52.(5)构建风险预测模型:为避免多元回归模型中的多重共线性和过度拟合,根据单因素cox回归分析结果,使用“glmnet”包进行lasso回归分析,根据参数lambda值筛选出15个mrna进行后续分析。对15个基因进行多因素cox回归分析,利用台阶法进行逐步回归(图1e)。建立了基于10个差异表达基因的cox多因素回归模型,见表1(10个基因的ncbi登录号如下:mt3:nm_005954;sorbs1:ng_034041;igfals:ng_011778;amh:ng_012190;il12b:ng_009618;tp53aip1:ng_030401;pxdnl:nm_144651;mc5r:nm_005913;foxd1:nm_004472;lhx1:nm_005568)。
53.表1基于10个差异表达基因的cox多因素回归模型
54.基因名称(id)coefhr值hr.95lhr.95hpvaluemt3
‑
0.1600.8520.7730.9400.0014sorbs1
‑
0.1290.8790.7611.0160.0802igfals
‑
0.0840.9190.8540.9900.0267amh0.0891.0931.0041.1890.0392il12b
‑
0.1370.8720.7950.9570.0039tp53aip1
‑
0.1640.8490.7640.9430.0022pxdnl0.1371.1461.0651.2340.0003mc5r0.1201.1271.0281.2360.0109foxd10.0731.0761.0041.1520.0384lhx10.0551.0560.9931.1240.0836
55.注:hr.95l表示hr值95%ci的下限,hr.95h表示hr值95%ci的上限。
56.(6)建立基于差异表达基因的预测模型:
[0057][0058]
其中,βi代表每个基因的系数,exp(xi)代表每个基因的标准化表达值;
[0059]
即:风险分数=(
‑
0.160)*mt3+(
‑
0.129)*sorbs1+(
‑
0.084)*igfals+0.089*amh+(
‑
0.137)*il12b+(0.164)*tp53aip1+0.137*pxdnl+0.120*mc5r+0.073*foxd1+0.055*lhx。
[0060]
(7)基于上述获得的差异表达基因的乳腺癌预后风险模型,计算各个患者的风险分数,使用medcalc绘制roc曲线,并找到截断值(
‑
1.281);然后根据roc工作曲线截断值将患者分为高风险组和低风险组。使用r语言的“survival包”根据分组结果绘制五年生存率,通过绘制kaplan
‑
meier(k
‑
m)曲线将两者进行比较,利用双侧对数秩检验确定高风险和低风险患者组之间的生存差异。通过比较,发现在基于差异表达水平基因的风险模型组中,测试集tcga(n=1027)以及验证集gse96058(n=2969)的乳腺癌患者高风险组和低风险组患者的五年生存率差异具有显著性意义(p<0.001),如图2a所示,低风险组的生存时间明显长于高风险组。表明该模型能够有效预测患者的预后。
[0061]
(8)模型的预测准确性分析:用r包“survivalroc”绘制受试者(测试集tcga(n=
1027)以及验证集gse96058(n=2969))工作特征roc曲线,比较受试者工作特性曲线(roc)的曲线下面积(auc),验证模型的预测准确性;发现基于抑郁差异表达基因的10
‑
基因标记风险评分模型(即上述步骤(6)建立的基于差异表达基因的预测模型)对预测患者的1、3、5年生存预后均较为稳定(1年、3年、5年生存率的roc曲线下面积为0.789、0.766、0.734),见图2b。表明该模型具有良好的特异性和稳定性。
[0062]
(9)模型的效用分析:对不同分子亚型的乳腺癌患者(不同分子亚型的乳腺癌患者数据分别来源于tcga数据集和gse96058数据集中提供的乳腺癌分子分型的患者的数据,具体为:以tcga数据库收集226例basal型(basal
‑
like型)、352例luma型(luminal a型)、158例lumb型(luminal b型)及52例her
‑
2型乳腺癌患者作为测试集,以gse96058数据集中收集的507例basal型、1501例luma型、666例lumb型及295例her
‑
2型乳腺癌患者作为验证集)使用r包“survivalroc”绘制roc曲线,验证风险模型对不同分子亚型的乳腺癌患者的预测准确性。发现此模型对于basal型和luma型的患者具有更好的预测效用,见图3。
[0063]
(10)多变量分析揭示预后预测价值:利用gse96058数据集对预测模型进行验证,km曲线说明高风险组较低风险组生存率低(p<0.001),1年、3年、5年生存率的roc曲线下面积分别为0.725、0.651、0.618,见图4。多变量分析显示,本研究的风险预测模型是乳腺癌患者生存率的独立危险因素(hr=2.671,95%ci:2.073
‑
3.411,p<0.001),优于tnm分期(t:hr=0.961,95%ci:0.700
‑
1.319,p=0.805;n:hr=1.270,95%ci:0.941
‑
1.716,p=0.119;m:hr=0.956,95%ci:0.409
‑
2.235,p=0.0.918)和病理学分期(hr=1.618,95%ci:0.954
‑
2.742,p=0.074)。与其他临床病理特征比较,风险预测模型的roc曲线的auc值(0.781)明显高于tnm分期(t:auc=0.743,n:auc=0.661,m:auc=0.549)和病理学分期(auc=0.728),见图5。
[0064]
(11)建立一个列线图:结合风险评分模型的风险评分、病理分期和年龄、分子分型4个指标,利用多因素cox回归分析构建生存率预测列线图模型。已建立的预测生存期的列线图适用于预测乳腺癌患者的1年、3年和5年生存率,如图6所示。列线图模型可根据年龄、病理学分期、分子分型和风险分数这4个自变量的回归系数设定评分标准,包括单项得分,即图中的point,表示每个变量在特定取值下所对应的单项分数,以及总得分,即total point,表示所有变量取值后对应的单项分数加起来合计的总得分,最后通过总评分与生存率之间的函数转换关系,从而计算出每个患者的一年、三年、五年生存率。列线图模型的使用方法包括:先将乳腺癌患者的风险分数、年龄、病理学分期和分子分型对应(垂直对应)的分值求和以得到其总分值,再根据总分值确定对应(垂直对应)的一年、三年、五年生存率。
[0065]
上述实施例为本发明较佳的实施方式,但本发明的实施方式并不受上述实施例的限制,其他的任何未背离本发明的精神实质与原理下所作的改变、修饰、替代、组合、简化,均应为等效的置换方式,都包含在本发明的保护范围之内。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1