用于卵巢癌和其他癌症免疫治疗的新型肽和肽组合物的制作方法
1.本发明涉及用于免疫治疗方法的肽、蛋白质、核酸和细胞。特别是,本发明涉及癌症的免疫疗法。本发明还涉及单独使用或与其他肿瘤相关肽(刺激抗肿瘤免疫反应或体外刺激t细胞和转入患者的疫苗复合物的活性药物成分)联合使用的肿瘤相关t细胞(ctl)肽表位。与主要组织兼容性复合体(mhc)分子结合的肽或与此同类的肽也可能是抗体、可溶性t细胞受体和其他结合分子的靶标。
2.本发明涉及数种新型肽序列及其变体,它们源自人肿瘤细胞的hla
‑
i类分子,可用于引发抗肿瘤免疫反应的疫苗组合物中或作为开发药物/免疫活性化合物和细胞的目标。
背景技术:
3.卵巢癌
4.卵巢癌2012年的新发病例估计为239 000例,是女性第七大最常见的癌症,占女性所有癌症的4%。相对于其他的女性生殖器官癌症,卵巢癌的死亡率往往较高,病死率在资源少的环境下更高。因此,卵巢癌是女性癌症死亡的第八大最常见的原因,有152 000人死亡。2012年,所有新发病例中近55%发生在人类发展水平高或非常高的国家;37%的新发病例和39%的死亡病例发生在欧洲和北美。北欧、东欧、北美和澳洲发病率最高,非洲和亚洲大部分地区相对较低。发病率在人类发展水平很高的某些国家一直呈下降趋势,特别是欧洲和北美。
5.最常见的卵巢癌症是卵巢癌,这也是致命性最强的妇科恶性肿瘤。基于组织病理学和分子遗传学,卵巢癌被分成五个主要类型:高级别浆液性癌(70%)、子宫内膜癌(10%)、透明细胞癌(10%)、黏液癌(3%)和低级别浆液性癌(<5%),共占卵巢癌病例的95%以上。很少见的是恶性生殖细胞瘤(无性细胞瘤、卵黄囊瘤和未成熟畸胎瘤)(占卵巢癌的3%)和潜在恶性性索间质肿瘤(1
‑
2%)(最常见的是颗粒细胞瘤)。
6.10%的病例有卵巢癌家族史;当两个或更多个一级亲属患有卵巢癌时,风险增加3倍。brca1或brca2种系突变的女性在70岁前罹患卵巢癌(主要是高级别浆液性癌)的风险为30
‑
70%(risch et al.,2006)。
7.手术切除是早期和晚期卵巢癌的主要治疗方法。手术切除之后使用铂类似物进行全身化疗,但无手术后化疗适应症的非常低等级的卵巢癌(ia期、1级)除外。对于晚期卵巢癌,一线化学疗法包括卡铂与紫杉醇联合使用,可使用贝伐单抗作为补充。对于铂类耐药的卵巢癌的标准治疗方法包括使用以下其中一种化疗药物的单一治疗:聚乙二醇化脂质体阿霉素、托泊替康、吉西他滨或紫杉醇(s3
‑
leitlinie maligne ovarialtumore,2013)。
8.免疫疗法似乎是改善卵巢癌患者治疗的一种有前景的策略,因为存在促炎性肿瘤浸润淋巴细胞,尤其是cd8阳性t细胞与良好的预后相关,肿瘤相关肽特异性t细胞可从癌组织中分离。
9.因此,大量的科学工作投入到卵巢癌不同免疫疗法的研究中。已经进行了相当多的临床前研究和临床研究,进一步的研究目前正在进行中。对于细胞因子疗法、接种疫苗、
单克隆抗体治疗、细胞过继转移和免疫调节,目前已有临床资料。
10.使用白介素
‑
2、干扰素
‑
α、干扰素
‑
γ或粒细胞
‑
巨噬细胞集落刺激因子的细胞因子治疗目的在于增强患者自身的抗肿瘤免疫反应,这些治疗已经在小型研究组中显示出有前景的结果。
11.使用来自几种肿瘤相关蛋白(her2/neu、ny
‑
eso
‑
1、p53、wilms肿瘤
‑
1)的单个或多个肽或来自自体肿瘤细胞的全肿瘤抗原的i期和ii期疫苗接种研究显示了良好的安全性和耐受性特性,但临床效果仅为低至中等。
12.特异性识别肿瘤相关蛋白的单克隆抗体被视为可增强对肿瘤细胞的免疫细胞介导的杀伤力。抗ca
‑
125抗体oregovomab和abagovomab以及抗epcam抗体catumaxomab在ii期和iii期研究中取得了有前景的结果。与此相反,抗muc1抗体hmfg1在iii期研究中未能明确地提高生存期。
13.另一种方法使用单克隆抗体来靶向和阻止肿瘤细胞上的生长因子和存活受体。虽然施用曲妥单抗(抗her2/neu抗体)和mov18与morab
‑
003(抗叶酸受体α抗体)仅对卵巢癌具有有限的临床益处,但是在标准化疗中加入贝伐单抗(抗vegf抗体)对晚期卵巢癌似乎是有利的。
14.免疫细胞的过继转移实现了临床试验中的异质结果。自体、体外扩增的肿瘤浸润性t细胞的过继转移在中试试验中被证明是一种有希望的方法。相反,转移含有叶酸受体α特异性嵌合抗原受体的t细胞在i期试验中没有诱导显著的临床反应。用肿瘤细胞裂解物或肿瘤相关蛋白体外刺激的树突细胞显示可在转移后增强抗肿瘤t细胞反应,但t细胞活化的程度与临床效果无关。在ii期研究中,自然杀伤细胞的转移造成明显的毒性。
15.内在抗肿瘤免疫性以及免疫治疗受免疫抑制肿瘤微环境影响。为了克服这个障碍,免疫调节药物(如环磷酰胺、抗cd25抗体和聚乙二醇化脂质体阿霉素)与免疫疗法组合进行了测试。增强t细胞活性的易普利姆玛(ipilimumab)、抗ctla4抗体目前有最可靠的资料。易普利姆玛被证明可在卵巢癌患者中产生显著的抗肿瘤效果(mantia
‑
smaldone et al.,2012)。
16.考虑到治疗癌症相关的严重副作用和费用,通常有必要确定可用于治疗癌症的因子,尤其是卵巢癌。通常也有必要确定代表癌症生物标志物的因子,尤其是卵巢癌,从而更好地诊断癌症、评估预后和预测治疗成功性。
17.癌症免疫治疗代表了癌症细胞特异性靶向作用的一个选项,同时最大限度地减少副作用。癌症免疫疗法利用存在的肿瘤相关抗原。
18.肿瘤相关抗原(taa)的目前分类主要包括以下几组:
19.a)癌
‑
睾丸抗原:t细胞能够识别的最先确认的taa属于这一类抗原,由于其成员表达于组织学相异的人肿瘤中、正常组织中、仅在睾丸的精母细胞/精原细胞中、偶尔在胎盘中,因此,它最初被称为癌
‑
睾丸(ct)抗原。由于睾丸细胞不表达hlai类和ii类分子,所以,在正常组织中,这些抗原不能被t细胞识别,因此在免疫学上可考虑为具有肿瘤特异性。ct抗原大家熟知的例子是mage家族成员和ny
‑
eso
‑
1。
20.b)分化抗原:肿瘤和正常组织(肿瘤源自该组织)都含有taa。大多数已知的分化抗原发现于黑色素瘤和正常黑色素细胞中。许多此类黑色素细胞谱系相关蛋白参与黑色素的生物合成,因此这些蛋白不具有肿瘤特异性,但是仍然被广泛用于癌症的免疫治疗。例子包
括,但不仅限于,黑色素瘤的酪氨酸酶和melan
‑
a/mart
‑
1或前列腺癌的psa。
21.c)过量表达的taa:在组织学相异的肿瘤中以及许多正常组织中都检测到了基因编码被广泛表达的taa,一般表达水平较低。有可能许多由正常组织加工和潜在提呈的表位低于t细胞识别的阈值水平,而它们在肿瘤细胞中的过量表达能够透过打破先前确立的耐受性而引发抗癌反应。这类taa的典型例子为her
‑
2/neu、生存素、端粒酶或wt1。
22.d)肿瘤特异性抗原:这些独特的taa产生于正常基因(如β
‑
catenin、cdk4等)的突变。这些分子变化中有一些与致瘤性转化和/或进展相关。肿瘤特异性抗原一般可在不对正常组织带来自体免疫反应风险的情况下诱导很强的免疫反应。另一方面,这些taa在多数情况下只与其上确认了有taa的确切肿瘤相关,并且通常在许多个体肿瘤之间并不都共享taa。在含有肿瘤特定(相关)同种型蛋白的情况下,如果肽源自肿瘤(相关)外显子也可能出现肽肿瘤特异性(或相关性)。
23.e)由异常翻译后修饰产生的taa:此类taa可能由肿瘤中既不具有特异性也不过量表达的蛋白产生,但其仍然具有肿瘤相关性(该相关性由主要对肿瘤具有活性的翻译后加工所致)。此类taa产生于变糖基化模式的改变,导致肿瘤产生针对muc1的新型表位或在降解过程中导致诸如蛋白拼接的事件,这可能具有也可能不具有肿瘤特异性。
24.f)肿瘤病毒蛋白:这些tta是病毒蛋白,可在致癌过程中发挥关键作用,并且由于它们是外源蛋白(非人源蛋白),所以能够激发t细胞反应。这类蛋白的例子有人乳头状瘤16型病毒蛋白、e6和e7,它们在宫颈癌中表达。
25.基于t细胞的免疫治疗靶向作用于主要组织兼容性复合体(mhc)分子提呈的来源于肿瘤相关蛋白或肿瘤特异性蛋白的肽表位。肿瘤特异性t淋巴细胞所识别的抗原,即其表位,可以是源自所有蛋白类型的分子,如酶、受体、转录因子等,它们在相应肿瘤的细胞中被表达,并且与同源未变的细胞相比,其表达通常上调。
26.mhc分子有两类:mhc i类和mhc ii类。mhc i类分子由一条α重链和β
‑2‑
微球蛋白,mhc ii类分子由一条α和一条β链组成。其三位构造形成一个结合槽,用于与肽进行非共价相互作用。
27.大部分有核细胞上都可发现mhc
‑
i类分子。他们提呈主要为内源性的蛋白、缺陷核糖体产物(drip)和较大肽裂解生成的肽。然而,源自内体结构或外源性来源的肽也经常在mhc
‑
i类分子上发现。这种i
‑
类分子非经典提呈方式在文献中被称为交叉提呈(brossart and bevan,1997;rock et al.,1990)。mhc ii类分子主要发现于专业抗原提呈细胞(apc)上,并且主要提呈,例如,在内吞作用过程中由apc占据并且随后被加工的外源性或跨膜蛋白的肽。
28.肽和mhc i类的复合体由负载相应t细胞受体(tcr)的cd8阳性t细胞进行识别,而肽和mhc ii类分子的复合体由负载相应tcr的cd4阳性辅助t细胞进行识别。因此,tcr、肽和mhc按照1:1:1的化学计量呈现,这一点已是共识。
29.cd4阳性辅助t细胞在诱导和维持cd8阳性细胞毒性t细胞的有效反应中发挥重要作用。肿瘤相关抗原(taa)衍生的cd4阳性t细胞表位的识别对开发能引发抗肿瘤免疫反应的药物产品可能非常重要(gnjatic et al.,2003)。在肿瘤部位,t辅助细胞维持着对细胞毒性t细胞(ctl)友好的细胞因子环境(mortara et al.,2006)并吸引效应细胞,如ctl、天然杀伤(nk)细胞、巨噬细胞和粒细胞(hwang et al.,2007)。
30.在没有炎症的情况下,mhc ii类分子的表达主要局限于免疫系统细胞,尤其是专业抗原提呈细胞(apc),例如,单核细胞、单核细胞源性细胞、巨噬细胞、树突状细胞。在癌症患者的肿瘤细胞中发现有mhc ii类分子的表达(dengjel et al.,2006)。
31.本发明的拉长(较长)肽可作为mhc
‑
ii类活性表位。mhc
‑
ii类表位活化的辅助t细胞在编排抗肿瘤免疫的ctl效应子功能中发挥着重要作用。触发t
h1
细胞反应的辅助t细胞表位支援cd8阳性杀伤t细胞的效应子功能,其中包括直接作用于肿瘤细胞的细胞毒性功能(该类肿瘤细胞表面显示有肿瘤相关肽/mhc复合体)。这样,肿瘤相关t辅助细胞表位单独使用或与其他肿瘤相关肽结合使用可作为刺激抗肿瘤免疫反应的疫苗化合物的活性药物成分。
32.哺乳动物(如小鼠)模型显示,即使没有cd8阳性t淋巴细胞,cd4阳性t细胞也能透过分泌干扰素
‑
γ(ifnγ)抑制血管生成而足以抑制肿瘤的表现(beatty and paterson,2001;mumberg et al.,1999)。没有cd4 t细胞作为直接抗肿瘤效应因子的证据(braumuller et al.,2013;tran et al.,2014)。
33.由于hla ii类分子的组成性表达通常仅限于免疫细胞,因此,直接从原发肿瘤中分离ii类肽之前被认为是不可能的事。然而,dengjel等人成功地在肿瘤中直接识别了多个mhc ii类表位(wo 2007/028574,ep 1 760 088b1)。
34.由于cd8依赖型和cd4依赖型这两种反应共同并协同地促进抗肿瘤作用,因此,确定和表征由cd8+ t细胞(配体:mhc i类分子+肽表位)或cd4阳性t辅助细胞(配体:mhc ii类分子)识别的肿瘤相关抗原对开发肿瘤疫苗非常重要。
35.对于mhc i类肽触发(引发)细胞免疫反应的肽,它也必须与mhc分子结合。这一过程依赖于mhc分子的等位基因以及肽氨基酸序列的特异性多态性。mhc
‑
i类
‑
结合肽的长度通常为8
‑
12个氨基酸残基,并且在其与mhc分子相应结合沟槽相互作用的序列中通常包含两个保守残基(”锚”)。这样,每个mhc的等位基因都有“结合基序”,从而确定哪些肽能与结合沟槽特异性结合。
36.在mhc
‑
i类依赖性免疫反应中,肽不仅能与肿瘤细胞表达的某些mhc
‑
i类分子结合,而且它们之后还必须能被t细胞负载的特异性t细胞受体(tcr)识别。
37.对于被t淋巴细胞识别为肿瘤特异性抗原或相关性抗原以及用于治疗的蛋白质,必须具备特殊的条件。该抗原应主要由肿瘤细胞表达,而不由正常健康组织表达,或表达数量相对较少。在一个优选的实施方案中,与正常健康组织相比,所述肽应在肿瘤细胞中过度提呈。更为适宜的情况是,该相应抗原不仅出现于一种肿瘤中,而且浓度(即每个细胞的相应肽拷贝数目)高。肿瘤特异性抗原和肿瘤相关抗原往往是源自直接参与因细胞周期控制或凋亡抑制中的其功能而发生的正常细胞向肿瘤细胞转化的蛋白。另外,这些直接导致转化事件的蛋白的下游靶标可能会被上调,因此可能与肿瘤间接相关。这些间接肿瘤相关抗原也可能是预防接种方法的靶标(singh
‑
jasuja et al.,2004)。至关重要的是,表位存在于抗原氨基酸序列中,以确保这种来自肿瘤相关抗原的肽(“免疫原性肽”)可导致体外或体内t细胞反应。
38.基本上,任何能与mhc分子结合的肽都可能充当一个t细胞表位。诱导体外或体内t细胞反应的前提是存在具有相应tcr的t细胞并且不存在对该特定表位的免疫耐受性。
39.因此,taa是基于t细胞疗法(包括但不限于肿瘤疫苗)研发的起点。识别和表征taa
的方法通常基于对患者或健康受试者t细胞的使用情况,或基于肿瘤与正常组织肽之间差别转录特性或差别表达模式的产生。然而,对肿瘤组织或人肿瘤细胞株中过量表达或选择性表达的基因的识别并不提供在免疫疗法中使用这些基因所转录抗原的准确信息。这是因为,有着相应tcr的t细胞必须要存在而且对这个特定表位的免疫耐受性必须不存在或为最低水平,因此,这些抗原的表位只有一部分适合这种应用。因此,在本发明的一非常优选的实施例中,只选择那些针对可发现功能性和/或增殖性t细胞情况的过量提呈或选择性提呈肽,这一点非常重要。这种功能性t细胞被定义为在以特异性抗原刺激后能够克隆地扩展并能够执行效应子功能(”效应子t细胞”)的t细胞。
40.在透过根据本发明的特定tcr(例如可溶性tcr)和抗体或其他结合分子(支架)靶向作用于肽
‑
mhc的情况下,潜在肽的免疫原性是次要的。在这些情况下,提呈是决定因素。
技术实现要素:
41.本发明涉及一种肽,包含选自包括seq id no:1至seq id no:640的组的一个氨基酸序列、或该序列的与seq id no:1至seq id no:640具有至少77%,优选至少88%同源(优选至少77%或至少88%相同)的一种变体序列(其中所述变体与mhc结合和/或诱导t细胞与所述肽发生交叉反应),或其药用盐(其中所述肽不是潜在全长多肽)。
42.本发明进一步涉及本发明的一种肽,包含选自包括seq id no:1至seq id no:640的组的一个序列、或与seq id no:1至seq id no:640具有至少77%、优选至少88%同源性(优选为至少77%或至少88%相同)的一种变体,其中所述肽或其变体的总长度为8至100个、优选为8至30个、最优选为8至14个氨基酸。
43.下表显示了根据本发明的肽、它们各自的seq id no、以及这些肽的可能源(潜在)基因。表1和表2中的所有肽均与hla
‑
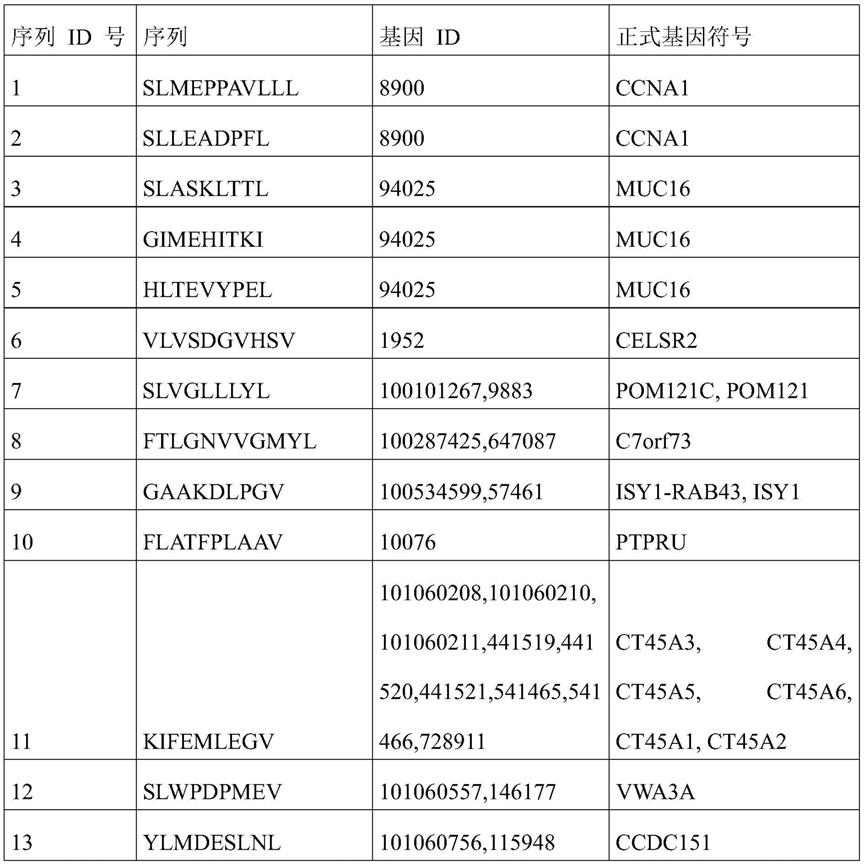
a*02结合。表2中的肽之前在大型列表中披露,作为高通量筛查结果,错误率高,或使用算法计算出,但之前与癌症毫无关联。表3中的肽是可与本发明其他肽组合使用的其他肽。表4中的肽还可用于诊断和/或治疗各种其他恶性疾病,这些疾病涉及过量表达或过度提呈各潜在多肽。
44.表1:本发明中的肽。j=磷酸丝氨酸
45.46.47.48.49.50.51.52.53.54.55.56.57.58.59.60.[0061][0062]
表2:本发明中的其他肽,之前与癌症无已知的关联。j=磷酸丝氨酸
[0063]
[0064]
[0065]
[0066]
[0067]
[0068]
[0069]
[0070]
[0071]
[0072]
[0073][0074]
表3:用于癌症疗法(例如个性化癌症疗法)的肽
[0075]
[0076][0077]
本发明还一般涉及本发明的肽用于治疗增殖性疾病,例如,非小细胞肺癌,小细胞肺癌、肾癌、脑癌、结肠或直肠癌、胃癌、肝癌、胰腺癌、前列腺癌、白血病、乳腺癌、梅克尔细胞癌、黑色素瘤、食管癌、膀胱癌、子宫癌、胆囊癌、胆管癌、胆管癌。
[0078]
肽seq id no.466(vlianlekl)及其在卵巢癌、非小细胞肺癌、小细胞肺癌、肾癌、脑癌、结肠癌或直肠癌、胃癌、肝癌、胰腺癌、前列腺癌、白血病、乳腺癌、梅克尔细胞癌、黑色素瘤、食管癌、膀胱癌、子宫癌、胆囊癌、胆管癌、优选为卵巢癌的免疫治疗用途尤其令人关注,因此为优选。
[0079]
特别优选的是本发明的肽(单独或组合),其选自包括seq id no:1、11、427、408、198、512、519和587的组群及其在卵巢癌、非小细胞肺癌、小细胞肺癌、肾癌、脑癌、结肠癌或直肠癌、胃癌、肝癌、胰腺癌、前列腺癌、白血病、乳腺癌、梅克尔细胞癌、黑色素瘤、食管癌、膀胱癌、子宫癌、胆囊癌、胆管癌、优选为卵巢癌的免疫治疗用途。
[0080]
特别优选的是本发明的肽(单独或组合),其选自包括seq id no:3、4、5、6、8、10、12、14、15、18、20、25、29、32、37、38、39、41、44、45、52、53、54、57、64、69、72、73、77、78、83、89、90、91、93、94、96、99、100、102、104、106、107、109、113、114、117、120、123、124、136、137、138、139、141、143、148、150、151、157、158、160、163、165、166、170、171、173、175、179、180、184、185、187、189、191、192、193、194、195、196、200、202、204、206、209、211、215、216、217、218、219、221、224、225、226、230、231、232、233、234、235、238、239、243、244、245、247、248、250、253、258、266、267、269、301、306、347、348、350、365、367、369、378、380、426、430、432、433、438、441、442、444、449、451、455、460、461、462、463、465、467、468、470、471、478、479、481、482、484、485、489、491、494、498、505、509、511、514、515、516、518、522、532、542、547、548、552、560、578和620的组群及其在卵巢癌、非小细胞肺癌、小细胞肺癌、肾癌、脑癌、结肠癌或直肠癌、胃癌、肝癌、胰腺癌、前列腺癌、白血病、乳腺癌、梅克尔细胞癌、黑色素瘤、食管癌、膀胱癌、子宫癌、胆囊癌、胆管癌、优选为卵巢癌的免疫治疗用途。
[0081]
特别优选的是本发明的肽(单独或组合),其选自包括seq id no:1至seq id no:640的组。更优选的是所述肽(单独或组合)选自包括seq id no:1至seq id no:259(见表1)的组,并且其用于卵巢癌、非小细胞肺癌、小细胞肺癌、肾癌、脑癌、结肠癌或直肠癌、胃癌、肝癌、胰腺癌、前列腺癌、白血病、乳腺癌、梅克尔细胞癌、黑色素瘤、食管癌、膀胱癌、子宫癌、胆囊癌、胆管癌、优选为卵巢癌的免疫治疗。
[0082]
如示下面的表4a和4b所示,其中本发明的许多肽也发现于其他肿瘤中,因此也可用于其他适应症的免疫治疗。另请参阅图1和实施例1。
[0083]
表4a:本发明的肽及其在其他增殖性疾病(特别是其他癌性疾病)中的特定用途。该表显示,对于其他肿瘤类型的选定肽,发现他们过量提呈(特定提呈)于5%以上测定的肿瘤样本,或提呈于5%以上测定的肿瘤样本且几何学平均值肿瘤与正常组织的比值大于3。过度提呈定义为与最高提呈的正常样本相比,肿瘤样本中的提呈更高。对比过度提呈组织
检测的正常组织有:脂肪组织、肾上腺、动脉、骨髓、脑、中枢神经、结肠、十二指肠、食道、胆囊、心脏、肾、肝、肺、淋巴结、单核白细胞、胰腺、外周神经、腹膜、垂体、胸膜、直肠、唾液腺、骨骼肌、皮肤、小肠、脾、胃、胸腺、甲状腺、气管、输尿管、膀胱、静脉。j=磷酸丝氨酸
[0084]
[0085]
[0086]
[0087]
[0088]
[0089]
[0090]
[0091]
[0092]
[0093]
[0094]
[0095]
[0096]
[0097]
[0098]
[0099]
[0100]
[0101]
[0102]
[0103]
[0104][0105]
nsclc=非小细胞肺癌,sclc=小细胞肺癌,rcc=肾癌,crc=结肠或直肠癌,gc=胃癌,hcc=肝癌,pc=胰腺癌,prc=前列腺癌,brca=乳腺癌,mcc=merkel细胞癌
[0106]
表4b:本发明的肽及其在其他增殖性疾病(特别是其他癌性疾病)中的特定用途(表4修订版)。该表(如表4a)显示,对于其他肿瘤类型的选定肽,发现他们过量提呈(特定提呈)于5%以上测定的肿瘤样本,或提呈于5%以上测定的肿瘤样本且几何学平均值肿瘤与正常组织的比值大于3。过度提呈定义为与最高提呈的正常样本相比,肿瘤样本中的提呈更高。经过度提呈的正常组织有:脂肪组织、肾上腺、动脉、骨髓、脑、中枢神经、结肠、十二指肠、食道、胆囊、心脏、肾、肝、肺、淋巴结、单核白细胞、胰腺、外周神经、腹膜、垂体、胸膜、直肠、唾液腺、骨骼肌、皮肤、小肠、脾、胃、胸腺、甲状腺、气管、输尿管、膀胱、静脉。
[0107]
[0108]
[0109]
[0110]
[0111]
[0112]
[0113]
[0114]
[0115]
[0116]
[0117]
[0118]
[0119]
[0120]
[0121]
[0122]
[0123]
[0124]
[0125]
[0126]
[0127]
[0128][0129]
nsclc=非小细胞肺癌,sclc=小细胞肺癌,rcc=肾癌,crc=结肠或直肠癌,gc=胃癌,hcc=肝癌,pc=胰腺癌,prc=前列腺癌,brca=乳腺癌,mcc=merkel细胞癌,oc=卵巢癌,nhl=非霍奇金淋巴瘤,aml=急性骨髓性白血病,cll=慢性淋巴细胞白血病,hnscc=头颈部鳞状细胞癌。
[0130]
因此,本发明的另一个方面涉及根据seq序列号1、11、17、27、45、57、58、61、62、65、72、74、79、84、97、98、104、105、125、126、143、150、157、161、167、176、179、183、184、195、198、201、204、213、217、222、228、234、248、263、264、268、285、287、303、313、319、323、333、335、338、343、347、348、355、356、359、373、385、394、395、403、415、421、427、428、429、430、431、434、441、443、444、446、447、450、454、456、457、458、459、463、474、477、479、480、486、489、492、493、497、501、503、506、514、517、521、526、538、539、540、541、545、554、558、568、573、576、578、579、589、595、597、599、602、607、610、613、616、627、632、635和637中任一项所述的本发明的至少一种肽与一种优选实施方案的肽联合用于治疗nsclc。
[0131]
因此,本发明的另一个方面涉及根据seq序列号1、6、17、19、20、27、28、31、34、36、38、45、47、48、51、54、55、56、57、58、59、60、61、65、66、72、75、76、79、82、85、88、91、92、98、103、108、117、123、125、126、127、135、141、142、149、152、153、166、167、169、171、176、183、184、200、205、213、214、216、228、233、234、237、240、242、248、249、251、256、263、264、277、279、283、286、288、296、300、301、312、314、315、322、323、328、331、338、341、344、345、346、366、372、373、385、388、394、399、401、404、410、418、420、421、427、428、431、433、435、437、439、441、443、444、446、449、450、451、454、461、463、469、473、474、475、479、481、492、493、500、501、514、517、521、522、523、530、531、539、541、542、545、551、552、554、555、556、558、560、561、563、565、568、574、575、581、589、590、592、595、597、602、606、610、613、616、618、622、625、626、627、628、629、632、637、638和640中任一项所述的本发明的至少一种肽与一种优选实施方案的肽联合用于治疗sclc。
[0132]
因此,本发明的另一个方面涉及根据seq序列号1、2、6、19、26、27、57、58、61、63、64、65、69、77、79、85、95、97、98、103、107、121、125、126、127、128、129、143、148、150、155、
157、166、170、174、177、200、201、204、207、213、217、222、223、229、234、235、242、243、252、258、264、267、271、275、279、285、287、294、303、306、311、313、317、319、323、328、330、332、333、336、346、347、348、354、355、356、359、360、361、375、382、385、387、393、395、405、410、415、424、430、431、441、444、447、450、459、461、465、466、472、477、480、486、491、492、497、498、499、501、505、506、508、513、514、518、528、533、539、541、542、543、554、560、561、565、568、575、576、583、588、589、591、592、594、601、610、616、619、624、629、631、633、635和640中任一项所述的本发明的至少一种肽与一种优选实施方案的肽联合用于治疗食管癌。
[0133]
因此,本发明的另一个方面涉及根据seq序列号6、48、68、106、118、127、135、143、157、174、209、247、279、292、300、313、28、332、333、340、357、358、385、389、410、425、431、450、456、464、473、474、492、501、506、514、523、528、538、539、541、558、586、589、590、592、593、606、610、619、620、628和635中任一项所述的本发明的至少一种肽与一种优选实施方案的肽联合用于治疗脑癌。
[0134]
因此,本发明的另一个方面涉及根据seq序列号6、7、17、27、56、59、61、65、76、93、103、110、131、141、143、149、169、204、212、216、226、228、229、230、242、255、264、266、268、271、273、283、284、285、286、287、288、289、303、309、331、333、335、336、340、358、362、371、372、373、375、393、395、396、401、420、422、423、427、439、446、459、466、504、521、539、554、576、580、592、595、597、610、616、635、637和640中任一项所述的本发明的至少一种肽与一种优选实施方案的肽联合用于治疗乳腺癌(brca)。
[0135]
因此,本发明的另一个方面涉及根据seq序列号6、20、139、283、373、396、418、430、441、446、472、473、474、479、501、575、578、589、627和640中任一项所述的本发明的至少一种肽与一种优选实施方案的肽联合用于治疗mcc。
[0136]
因此,本发明的另一个方面涉及根据seq序列号7、9、97、98、183、217、218、222、234、235、237、240、241、242、263、268、271、275、285、303、311、313、360、364、394、403、410、424、431、450、455、497、502、514、558、564、595、608、610和616中任一项所述的本发明的至少一种肽与一种优选实施方案的肽联合用于治疗rcc。
[0137]
因此,本发明的另一个方面涉及根据seq序列号7、24、27、64、65、84、87、95、97、125、126、127、130、137、143、183、200、272、275、291、292、311、312、332、335、346、351、357、358、364、372、398、405、407、410、421、423、427、443、459、464、499、539、540、603、609、630、631和632中任一项所述的本发明的至少一种肽与一种优选实施方案的肽联合用于治疗胰腺癌(pc)。
[0138]
因此,本发明的另一个方面涉及根据seq序列号9、31、58、183、275、335、410、421、499、514、564、616和640中任一项所述的本发明的至少一种肽与一种优选实施方案的肽联合用于治疗胃癌(gc)。
[0139]
因此,本发明的另一个方面涉及根据seq id no.9、14、19、20、28、32、36、48、54、57、58、63、64、66、87、92、94、97、98、108、125、129、139、143、144、154、157、159、163、166、167、170、174、176、178、188、197、198、201、204、207、208、209、212、213、214、217、222、229、234、237、248、256、267、269、271、273、275、286、290、294、301、306、309、313、327、328、332、338、339、340、341、346、347、349、355、359、360、367、369、370、371、372、378、383、385、387、393、394、395、396、401、406、409、410、411、415、419、420、423、427、428、429、430、432、441、
443、447、450、451、452、457、463、464、465、472、473、474、476、477、479、480、484、486、489、492、498、501、513、514、517、521、523、526、528、531、538、539、540、541、546、551、554、558、560、564、573、575、576、579、583、586、589、597、599、603、606、610、611、613、617、627、628、632、635、637、638和640中任一项所述的本发明的至少一种肽与一种优选实施方案的肽联合用于治疗肝细胞癌(hcc)。
[0140]
因此,本发明的另一个方面涉及根据seq序列号9、10、14、19、24、28、79、87、101、144、148、149、153、169、174、190、210、212、216、222、223、242、252、257、271、288、298、299、303、310、311、317、331、333、334、346、347、348、360、367、386、390、393、394、395、423、477、479、483、486、494、495、502、514、521、527、529、539、554、585、610、616、626、632和640中任一项所述的本发明的至少一种肽与一种优选实施方案的肽联合用于治疗膀胱癌。
[0141]
因此,本发明的另一个方面涉及根据seq id no.19、22、26、28、31、33、34、36、38、47、48、49、57、58、59、60、65、74、79、80、92、98、119、126、128、129、132、144、149、159、161、166、183、204、214、237、242、248、251、252、253、256、262、263、270、271、272、275、276、277、280、282、284、285、287、289、296、299、301、308、309、319、321、323、324、325、331、333、343、355、358、365、366、373、374、379、381、384、391、394、395、397、400、401、404、408、409、410、412、415、428、448、450、451,452、457、459、468、475、480、486、488、489、490、496、503、504、506、507、508、510、520、523、529、533、536、542、544、550、552、556、558、559、561、566、567、571、572、573、576、577、579、580、587、589、591、595、596、600、601、603、604、610、624、630、631、632、634和639中任一项所述的本发明的至少一种肽与一种优选实施方案的肽联合用于治疗白血病。
[0142]
因此,本发明的另一个方面涉及根据seq id no.19、22、31、34、38、48、57、58、61、62、63、64、74、77、92、97、98、101、105、107、143、144、150、155、167、176、177、183、184、199、213、217、222、230、248、251、256、264、277、282、283、287、291、309、314、316、321、323、329、331、338、339、343、344、355、365、366、373、384、388、391、394、395、401、410、412、431、443、444、450、452、457、461、463、468、472、474、487、496、499、501、514、517、521、523、525、530、540、542、544、549、550、551、552、555、560、563、564、565、566、568、572、573、575、576、578、580、584、588、589、596、599、603、611、614、616、618、621、622、624、625、626、627、631、632和633中任一项所述的本发明的至少一种肽与一种优选实施方案的肽联合用于治疗黑色素瘤。
[0143]
因此,本发明的另一个方面涉及根据seq id no.19、22、24、58、76、79、84、86、97、98、126、176、178、188、222、243、285、300、301、303、311、318、319、320、342、348、349、355、356、359、376、384、395、397、398、421、426、428、430、441、444、448、449、450、456、458、459、473、478、480、510、514、518、521、528、531、535、541、545、546、554、557、568、575、576、577、578、579、580、581、597、599、610、619、622、627、632、635和640中任一项所述的本发明的至少一种肽与一种优选实施方案的肽联合用于治疗结直肠癌(crc)。
[0144]
因此,本发明的另一个方面涉及根据seq序列号34、51、84、174、178、200、207、212、216、237、252、264、288、323、332、333、372、394、395、410、443、446、447、486、492、501、518、539、551、554、606、610、637和640中任一项所述的本发明的至少一种肽与一种优选实施方案的肽联合用于治疗前列腺癌(prc)。
[0145]
因此,本发明的另一个方面涉及根据seq序列号47、51、54、58、64、84、87、125、200、213、228、235、237、323、335、346、385、423、473、501、526、539、554、558、561、610、626和640中任一项所述的本发明的至少一种肽与一种优选实施方案的肽联合用于治疗胆囊癌。
[0146]
因此,本发明的另一个方面涉及根据seq序列号47、51、54、58、64、84、87、125、200、213、228、235、237、323、335、346、385、423、473、501、526、539、554、558、561、610、626和640中任一项所述的本发明的至少一种肽与一种优选实施方案的肽联合用于治疗胆管癌。
[0147]
因此,本发明的另一个方面涉及根据seq序列号48、126、127、129、149、153、157、207、214、228、235、237、288、323、328、332、358、360、385、395、423、427、446、497、514、539、558、565、599、619、632、637和640中任一项所述的本发明的至少一种肽与一种优选实施方案的肽联合用于治疗子宫癌。
[0148]
因此,本发明的另一个方面涉及本发明中肽的用途
‑
优选联合用于治疗选自卵巢癌、非小细胞肺癌、小细胞肺癌、肾癌、脑癌、结肠癌或直肠癌、胃癌、肝癌、胰腺癌、前列腺癌、白血病、乳腺癌、梅克尔细胞癌、黑色素瘤、食管癌、膀胱癌、子宫癌、胆囊癌、胆管癌组中的增殖性疾病。
[0149]
本发明还涉及本发明的肽,其具有与主要组织兼容性复合体(mhc)i或以拉长形式存在的例如长度变化的
‑
mhc
‑
ii类分子结合的能力。
[0150]
本发明进一步涉及本发明中的肽,其中所述肽(每种肽)系由或基本系由根据seq id no:1至seq id no:640的一个氨基酸序列组成。
[0151]
本发明进一步涉及本发明的肽,其中所述肽被修饰和/或包含非肽键。
[0152]
本发明进一步涉及本发明的肽,其中所述肽为融合蛋白的一部分,特别是与hla
‑
dr抗原相关不变链(ii)的n
‑
端氨基酸融合,或与抗体(例如,树突状细胞特定抗体)或抗体的序列融合。
[0153]
本发明进一步涉及一种核酸,其编码本发明的肽。本发明进一步涉及一种本发明的核酸,为dna、cdna、pna、rna,也可能为其组合物。
[0154]
本发明进一步涉及一种能表达和/或表达本发明核酸的表达载体。
[0155]
本发明进一步涉及本发明的一种肽、本发明的一种核酸或本发明的一种治疗疾病的药用表达载体,特别是用于治疗癌症。
[0156]
本发明进一步涉及本发明中肽或本发明中所述肽复合体(含有mhc)的特定抗体以及制造这些抗体的方法。
[0157]
本发明进一步涉及本发明的t细胞受体(tcr),特别是可溶性tcr(stcrs)和加工为自体或异体t细胞的克隆tcr,以及制造这些tcr的方法和载有所述tcr或所述tcr交叉反应的nk细胞的制造方法。
[0158]
抗体和tcr是根据本发明的肽现有免疫治疗用途的另外实施方案。
[0159]
本发明进一步涉及含本发明核酸或前述表达载体的一种宿主细胞。本发明进一步涉及本发明的宿主细胞,其为抗原提呈细胞,优选为树突细胞。
[0160]
本发明进一步涉及配制本发明一种肽的一种方法,所述方法包括培养本发明的宿主细胞和从所述宿主细胞或其培养基中分离肽。
[0161]
本发明进一步涉及本发明中的所述方法,其中抗原透过与足够量的含抗原提成细胞的抗原结合被载入表达于合适抗原提呈细胞或人工抗原呈递细胞表面的i或ii类mhc分
子。
[0162]
本发明进一步涉及本发明的方法,其中抗原提呈细胞由能表达含seq id no.1至seq id no.640、优选为含seq id no.1至seq id no.259所述肽的一个表达载体、或一个变体氨基酸序列组成。
[0163]
本发明进一步涉及以本发明方法制造的启动t细胞,其中所述t细胞有选择性地识别一种细胞,该细胞表达含一种本发明氨基酸序列的多肽。
[0164]
本发明进一步涉及一种杀伤患者靶细胞的方法,其中患者的靶细胞异常表达含本发明任何氨基酸序列的多肽,该方法包括给予患者按本发明方法制造的有效量t细胞。
[0165]
本发明进一步涉及任何所述肽、本发明的核酸、本发明的表达载体、本发明的细胞、本发明的作为药剂或制造药剂的启动t淋巴细胞、t细胞受体或抗体或其他肽
‑
和/或肽
‑
mhc结合分子的用途。所述药剂优选为具有抗癌活性。
[0166]
优选情况为,所述药剂为基于可溶性tcr或抗体的细胞治疗药物、疫苗或蛋白质。
[0167]
本发明还一般涉及本发明的用途,其中所述癌细胞为卵巢癌、非小细胞肺癌、小细胞肺癌、肾癌、脑癌、结肠癌或直肠癌、胃癌、肝癌、胰腺癌、前列腺癌、白血病、乳腺癌、梅克尔细胞癌、黑色素瘤、食管癌、膀胱癌、子宫癌、胆囊癌、胆管癌、优选为卵巢癌细胞。
[0168]
本发明进一步涉及一种基于本发明肽的生物标志物,在此成为“靶标“,其可用于诊断癌症,优选为卵巢癌。所述标志物可以肽本身过度提呈或相应基因过度表达。标志物也可以用于预测治疗成功的可能性,优选为免疫疗法,最优选为靶向作用于该生物标志物识别的相同靶目标免疫疗法。例如,抗体或可溶性tcr可用于染色肿瘤切片以检测是否存在相关肽与mhc复合。
[0169]
或者,抗体具有进一步的效应子功能,如免疫刺激域或毒素。
[0170]
本发明还涉及这些癌症治疗中新靶点的用途。
[0171]
abca1已被证明在卵巢癌和前列腺癌细胞系中超甲基化。abca1甲基化与卵巢癌患者的不良预后相关(lee et al.,2013a;chou et al.,2015)。在结肠癌中,abca1过度表达导致细胞胆固醇减少和肿瘤生长抑制。这种生长抑制可能是由于细胞凋亡所致,因为abca1过度表达增强从线粒体释放细胞色素c释放(smith and land,2012)。
[0172]
abcb8与人黑色素瘤耐药相关(chen et al.,2009b)。
[0173]
abcc1在原发性乳腺癌、肺癌、食道癌、白血病和儿童期成神经细胞瘤中上调(cole et al.,1992;burger et al.,1994;norris et al.,1996;nooter et al.,1997)。科学家们已经发现abcc1是mcf7乳腺癌细胞系的依托泊苷耐药变体中notch1信令的一个直接转录靶标(cho et al.,2011)。若干出版物已经表明,abcc1在癌症中表达增加与功能性p53损失有关(fukushima et al.,1999;sullivan et al.,2000)。
[0174]
abcc10被证明与乳腺癌的紫杉醇和吉西他滨耐药、非小细胞肺癌细胞系a549的吉西他滨、非小细胞肺癌细胞系sk
‑
lc6和nci
‑
h23的长春瑞滨耐药有关(ikeda et al.,2011;bessho et al.,2009;dorman et al.,2015)。abcc10被证明与乳腺癌发病有关(domanitskaya et al.,2014)。fhl2被证明在胰腺导管腺癌、肝细胞癌、非小细胞肺癌、慢性淋巴细胞性白血病和儿科急性髓性白血病中上调(mohelnikova
‑
duchonova et al.,2013b;borel et al.,2012;steinbach et al.,2006;wang et al.,2009c;hoellein et al.,2010)。abcc10表达被证明与结直肠癌的肿瘤分级和肺腺癌的病理分级和tnm分期相关
(hlavata et al.,2012;wang et al.,2009c)。
[0175]
abcc4水平升高存在于人nk/t细胞淋巴瘤细胞、肺癌细胞和胃癌细胞。另外,abcc4基因拷贝数变异与食管鳞状细胞癌的风险相关(sun et al.,2014b;zhao et al.,2014b;zhang et al.,2015d;zhang et al.,2015n)。此外,耐药胃癌细胞中abcc4表达的沉寂导致细胞凋亡增加和g1期细胞周期停滞。另一组表明,abcc4敲减抑制胃癌细胞的生长并阻滞细胞周期进展(chen et al.,2014d;zhang et al.,2015d)。
[0176]
在肾细胞癌、结直肠癌和黑色素瘤肿瘤发生中观察到了abcd1表达下调(heimerl et al.,2007;galamb et al.,2009;hour et al.,2009)。
[0177]
与非肿瘤组织相比,abcf1在治疗后的肿瘤中上调(hlavac et al.,2013)。此外,透过mir
‑
23a过度表达或siabcf1抑制abcf1表达过表达导致微卫星不稳定性大肠癌细胞中5
‑
氟尿嘧啶敏感度恢复(li et al.,2015c)。
[0178]
若干出版物表明abi1水平在各种癌症中升高,如卵巢癌、结直肠癌、乳腺癌和肝细胞癌(wang et al.,2007a;liu et al.,2009a;steinestel et al.,2013;steinestel et al.,2014;zhang et al.,2015f)。在上皮性卵巢癌中,abi1过度表达与晚期、高级别和ca
‑
125水平升高有关(zhang et al.,2015f)。abi1的敲减导致乳腺癌细胞系中侵袭性和迁移能力下降。同样地,abi1基因在白血病细胞中沉寂导致细胞迁移受损和肌动蛋白重塑异常(wang et al.,2007a;yu et al.,2008)。
[0179]
abl2在非小细胞肺癌、未分化甲状腺癌、黑色素瘤、结直肠癌、胰腺癌、肝细胞癌、卵巢浆液性囊腺癌、肺腺癌和肺鳞状细胞癌中过度表达(gil
‑
henn et al.,2013;greuber et al.,2013;xing et al.,2014)。在高度侵袭性乳腺癌细胞系中,abl2调节失调节egfr、her2、igfr和src激酶的增殖、存活和侵袭下游(srinivasan and plattner,2006;srinivasan et al.,2008)。
[0180]
adck3表达被证明在结直肠癌中改变(hennig et al.,2012)。
[0181]
adcy5编码腺苷酸环化酶5,这是一种膜结合腺苷酸环化酶,透过第二信使camp的合成介导g蛋白偶联受体信令(refseq,2002)。adcy5基因超甲基化和mrna表达减少发生于急性淋巴细胞性白血病、慢性淋巴细胞性白血病和肺腺癌(kuang et al.,2008;tong et al.,2010;sato et al.,2013)。
[0182]
adcy6的表达显示在喉鳞状细胞癌中差异性调节(colombo et al.,2009)。
[0183]
agl已被证明是膀胱癌的一种肿瘤抑制因子。agl在癌细胞中的损失经由甘氨酸合成酶丝氨酸羟甲2的诱导透过增加甘氨酸合成诱导体内和体外的肿瘤生长(guin et al.,2014;ritterson et al.,2015)。
[0184]
ahcy下调有助于肿瘤发生(leal et al.,2008)。ahcy能促进细胞凋亡。它能抑制食管鳞状细胞癌的黏附和迁移,提示具有食管致癌作用(li et al.,2014b)。ahcy蛋白表达在结肠癌中上调(kim et al.,2009;watanabe et al.,2008;fan et al.,2011)。ahcy可能是卵巢癌的潜在生物标志物(peters et al.,2005)。
[0185]
最近的工作确定akap6基因在胃癌中突变(li et al.,2016)。
[0186]
根据报告,aldh5a1在原位乳腺导管癌中过度表达。此外,aldh5a1抑制剂(如双硫仑和丙戊酸)能抑制乳房导管癌模型的净增殖(kaur et al.,2012)。
[0187]
已经观察到,由于alms1基因突变所致的阿尔斯特伦症候群患者也有乳头状甲状
腺癌。另一项研究确定alms1是慢性淋巴细胞性白血病的一种肿瘤新抗原(rajasagi et al.,2014;papadakis et al.,2015)。
[0188]
als2cr12被证明与皮肤基底细胞癌的易感性有关(stacey et al.,2015)。als2cr12中内含子单核苷酸多态性显示出与乳腺癌的风险有关(lin et al.,2015b)。
[0189]
在口腔鳞状细胞癌(oscc)中,alyref mrna和蛋白水平上调与细胞侵袭和转移所致的区域淋巴结转移相关(saito et al.,2013)。alyref mrna在多种肿瘤组织中过度表达,而蛋白质水平在高级别癌症中检测较低(dominguez
‑
sanchez et al.,2011)。alyref是核pi3k信令的一个靶标,它透过核akt磷酸化和磷脂酰肌醇相关性调节其子核居所、细胞增殖和mrna输出活动(okada et al.,2008)。
[0190]
ankrd26属于被证明在预后较差肿瘤患者中高度表达的基因家族(sahab et al.,2010)。ankrd26被证明与推定的肿瘤抑制因子rarres1有关(sahab et al.,2010)。
[0191]
ap1b1基因的纯合性缺失被认为在散发性脑膜瘤中无活性。这些发现提示,ap1b1基因可能在脑膜瘤发生中具有重要作用(peyrard et al.,1994;sayagues et al.,2007)。
[0192]
apobec3g与结直肠癌、肝细胞癌和淋巴瘤的肝脏转移有关(nowarski et al.,2012;chang et al.,2014b;weidle et al.,2015)。apobec3g与结肠癌伴肝转移的较差预后以及弥漫性大b细胞淋巴瘤总生存率降低相关(lan et al.,2014;jais et al.,2008)。
[0193]
apol2被证明在卵巢/腹膜癌中过度表达(davidson et al.,2011)。
[0194]
aqp5过度表达与多种癌症相关,如结直肠癌、宫颈癌、肺癌、乳腺癌和上皮卵巢癌(shan et al.,2014;yan et al.,2014a)。在非小细胞肺癌中,aqp5表达水平升高与淋巴结转移有关。此外,aqp5在iii期和iv期肿瘤中的表达水平明显高于i期和ii期肿瘤(song et al.,2015a)。先前的研究表明,aqp5可以启动直肠癌中的ras/erk/rb途径,并提高癌症发病率和进展(woo et al.,2008;kang et al.,2008b)。
[0195]
ar牵涉各种癌症的发生,如前列腺癌、去势抵抗性前列腺癌、乳腺癌、胶质母细胞瘤、结肠癌和胃癌(wang et al.,2009d;yu et al.,2015b;mehta et al.,2015;wang et al.,2015a;sukocheva et al.,2015)。除了促进前列腺癌的增殖,透过ar的雄激素信令经由诱导p21(waf1/cip1,一种细胞周期蛋白依赖性激酶抑制剂)的表达导致细胞凋亡(yeh et al.,2000)。
[0196]
一项研究表明,arfgef1在乳腺癌细胞系中下调。另一组报告了arfgef1是乳腺癌患者的一种肿瘤抑制剂(pongor et al.,2015;kim et al.,2011a)。据推测,微小rna
‑
27b介导的arfgef1上调透过启动arfgef1/akt途径促进肿瘤生长(matsuyama et al.,2016)。
[0197]
arhgap26被证明在急性髓细胞白血病中和cml进展期间下调(qian et al.,2010;aly and ghazy,2014)。arhgap26与来自原发性肺腺癌转移性脑肿瘤有关(zohrabian et al.,2007)。arhgap26与子宫肌瘤的风险、肿瘤大小和cml风险增加有关,是aml的一种良好的预后标志物(dzikiewicz
‑
krawczyk et al.,2014;aissani et al.,2015)。
[0198]
arhgef19被证明与肝细胞癌的转移有关(zhou et al.,2014a)。arhgef19被描述为平面细胞极性/非经典wnt途径的一部分,该途径与癌症有关(miller et al.,2011)。
[0199]
arid5b被证明在前列腺癌中失调(davalieva et al.,2015)。arid5b被证明与儿童急性淋巴细胞白血病的易感性、复发危险和较差治疗结果有关(xu et al.,2012;evans et al.,2014)。arid5b被证明是受sall4调节的潜在标靶,sall4是与急性骨髓性白血病相
关的一种转录因子(milanovich et al.,2015)。arid5b被证明是胃癌致癌tead4蛋白的一个靶标(lim et al.,2014)。arid5b被证明在子宫内膜样肿瘤中频繁突变(kandoth et al.,2013)。arid5b可能透过充当人乳头瘤病毒16整合位点而在宫颈癌发展中发挥作用(matovina et al.,2009)。arid5b被证明在人类唾液腺高度转移性腺样囊性癌细胞系acc
‑
m中上调,可能参与腺样囊性癌的肺转移,并可能作为诊断标志物和治疗靶标(sun et al.,2004a)。
[0200]
arl6ip1与宫颈癌细胞生长和侵袭有关(guo et al.,2010)。
[0201]
asun被证明在睾丸精原细胞瘤和卵巢癌细胞系中上调(bourdon et al.,2002)。一项研究表明,atf6b对于人类卵巢癌细胞系的溶血磷脂酸诱导的yap去磷酸化必不可少(cai and xu,2013)。
[0202]
atm是一种肿瘤抑制剂,其在广泛的人类癌症,包括肺、结直肠、乳腺和造血细胞癌症中经常发生突变(weber and ryan,2014)。atm损失与各种癌症(包括乳腺癌、结直肠癌、前列腺癌、肺癌和胰腺导管腺癌)的风险增加有关(swift et al.,1987;geoffroy
‑
perez et al.,2001;angele et al.,2004;roberts et al.,2012;grant et al.,2013;russell et al.,2015)。研究表明,il
‑
8能救援atm耗竭细胞中细胞迁移和侵袭缺陷(chen et al.,2015d)。低水平atm蛋白与乳腺癌患者的无转移生存差相关。此外,mir
‑
203和mir
‑
421过度表达可能参与这些患者的atm失调节(bueno et al.,2014;rondeau et al.,2015)。
[0203]
atp10a与b细胞前体急性淋巴细胞白血病的复发和无事件生存率下降有关(olsson et al.,2014)。
[0204]
atp2a2与皮肤癌、结肠癌和肺癌有关(korosec et al.,2006;hovnanian,2007)。
[0205]
atp2a3的表达在结肠癌、胃癌、肺癌和乳腺癌中明显降低,并且当这些细胞进行体外分化时诱导atp2a3表达(gelebart et al.,2002;arbabian et al.,2013;papp et al.,2012)。在结肠癌中,atp2a3的表达显示透过apc/β
‑
连环蛋白/tcf4致癌途径进行负调控(brouland et al.,2005)。此外,atp2a3表达被发现与淋巴管浸润负相关(gou et al.,2014)。
[0206]
研究人员指出,患有各种恶性肿瘤如黑色素瘤、非小细胞肺癌和慢性骨髓性白血病的患者在采用经照射的自体gm
‑
csf分泌肿瘤细胞接种或异体骨髓移植后会产生针对atp6ap1的高滴度igg抗体。另一份报告检测到浸润性导管和小叶癌以及乳腺癌中atp6ap1水平升高。此外,在滤泡淋巴瘤中发现atp6ap1基因突变(hodi et al.,2002;anderson et al.,2011;okosun et al.,2016)。
[0207]
atp6v1h被证明会与结肠癌细胞系hct116和sw480中的肿瘤相关基因tm9sf4产生相互作用(lozupone et al.,2015)。atp6v1h,是v
‑
atp酶v1的一部分,显示与结肠癌细胞的侵袭性为和肿瘤ph梯度相关(lozupone et al.,2015)。
[0208]
细胞内atp8a1蛋白水平升高降低了mir
‑
140
‑
3p在非小细胞肺癌细胞生长和迁移中的抑制作用(dong et al.,2015)。
[0209]
atr编码atr丝氨酸/苏氨酸激酶,其属于pi3/pi4激酶家族。这种激酶显示可磷酸化关卡激酶chk1、关卡蛋白rad17和rad9以及肿瘤抑制蛋白brca1(refseq,2002)。在口腔鳞癌细胞系中观察到atr基因的拷贝数增加、扩增或易位(parikh et al.,2014)。已经证实,子宫内膜癌截断atr突变与降低无病和总体生存下降相关(zighelboim et al.,2009)。ve
‑
822,是一种atr抑制剂,被证明可在体外对胰腺癌细胞和体内对胰腺肿瘤异种移植物有放射增敏和化学增敏作用(fokas et al.,2012;benada and macurek,2015)。
[0210]
aurka在产生于乳腺、结肠、卵巢、皮肤和其他组织的许多肿瘤中过度表达,且显示,当在许多细胞系模型中外源表达时可充当一种致癌基因(nikonova et al.,2013)。
[0211]
aurkb表达在不同的癌症类型,包括肺、结直肠和乳腺癌以及白血病中上调,因而与预后不良相关。所以,针对临床治疗研发aurkb抑制剂是一个有趣的领域(hayama et al.,2007;pohl et al.,2011;hegyi et al.,2012;goldenson and crispino,2015)。aurkb过度表达导致组蛋白h3的磷酸化和染色体的不稳定,这是癌变的关键因素(ota et al.,2002;tatsuka et al.,1998)。aurkb活性增强了致癌ras介导的细胞转化(kanda et al.,2005)。
[0212]
在前列腺和乳腺癌细胞系以及结直肠癌、甲状腺癌和子宫颈癌中检测到aurkc的过度表达和基因扩增。其他人观察到在精原细胞瘤中aurkc蛋白增加,这意味着它可能在睾丸癌进展中发挥作用。此外,aurkc已被证明具有致癌性,因为其过度表达将nih 3t3细胞转变为肿瘤(baldini et al.,2010;tsou et al.,2011;khan et al.,2011;zekri et al.,2012)。在结直肠癌中,aurkc的表达与疾病分级和肿瘤大小相关(hosseini et al.,2015)。此外,aurkc的过度表达诱导癌细胞的增殖、转化和迁移的增加(tsou et al.,2011)。
[0213]
bbs1基因的杂合子携带者似乎能增加透明细胞肾细胞癌的风险(beales et al.,2000)。此外,bbs1由黑色素瘤患者而非健康对照者的血清抗体识别(hartmann et al.,2005)。据报告,bbs1表达高的恶性胸膜间皮瘤患者与bbs1表达低的患者相比中位总生存期增加16.5个月(对比8.7个月)(vavougios et al.,2015)。
[0214]
bbx表达显示与nf
‑
kb/snail/yy1/rkip回路基因表达相关,后者与转移性前列腺癌和非霍奇金淋巴瘤相关(zaravinos et al.,2014)。
[0215]
bcl2l13显示在实体癌和血癌中过度表达,包括胶质母细胞瘤和急性淋巴细胞白血病(jensen et al.,2014;yang et al.,2010)。bcl2l13与儿童急性淋巴细胞白血病的不良临床结果相关(holleman et al.,2006)。
[0216]
定量pcr和免疫组化分析表明,bdh1在高级别前列腺癌中上调。此外,bdh1基因在转移性结膜黑色素瘤中频繁扩增(lake et al.,2011;saraon et al.,2013)。
[0217]
bhlhe41与乳腺癌转移、子宫内膜癌转移、三阴乳腺癌转移、胰腺癌、人鳞状细胞癌和肺癌有关(sato et al.,2012a;piccolo et al.,2013;liao et al.,2014a;takeda et al.,2014;falvella et al.,2008;adam et al.,2009)。bhlhe41与人口腔癌的cddp抗性相关(wu et al.,2012e)。
[0218]
bola2被描述为c
‑
myc致癌基因的一种新候选靶向基因,可能透过改变细胞周期的控制与恶性肝细胞转化有关(hunecke et al.,2012)。
[0219]
bop1与卵巢癌和结直肠癌有关(wrzeszczynski et al.,2011;killian et al.,2006)。bop1被证明是wnt连环蛋白的靶基因,其在小鼠中诱导结直肠癌细胞的emt、细胞迁移和实验性转移。因此,bop1可作为结直肠癌转移的治疗靶标(qi et al.,2015)。bop1与肝细胞癌的侵袭和转移有关(chung et al.,2011)。bop1被描述为与霉酚酸治疗后胃癌细胞系细胞周期阻滞相关的分子途径的成员,表明bop1与药物的抗癌活性具有潜在相关性(dun et al.,2013a;dun et al.,2013b)。bop1是直肠癌一种可能的标志物(lips et al.,
2008)。bop1被描述为卵巢癌的潜在致癌基因(wrzeszczynski et al.,2011)。bop1被证明在肝细胞癌中上调(chung et al.,2011)。bop1被证明是与肝细胞癌的微血管侵犯、较短的无病生存期和转移有关(chung et al.,2011)。bop1被描述为pebow复合体的亚基,这是细胞核糖体大亚基增殖和成熟必不可少的。bop1的过度表达被证明可抑制细胞增殖(rohrmoser et al.,2007)。氨基端截短形式的bop1表达导致g(1)特异性cdk2和cdk4激酶复合体、视网膜母细胞瘤和细胞周期蛋白a下调,而cdk抑制因子p21和p27上调。这导致g(1)期阻滞(pestov et al.,2001)。
[0220]
bub1b是一种肿瘤抑制蛋白。bub1b调节纺锤体装配步骤。bub1b在肿瘤中失活或下调。bub1b基因突变也与肿瘤的发展相关(aylon and oren,2011;fagin,2002;malumbres and barbacid,2007;rao et al.,2009)。bub1b透过启动癌基因而与胃癌发生有关(resende et al.,2010)。bub1b突变是结直肠癌的原因之一(karess et al.,2013;grady,2004)。
[0221]
c10orf137与鳞状细胞肺癌和结直肠癌相关(gylfe et al.,2010;zheng et al.,2013)。
[0222]
在口腔鳞状细胞癌患者唾液中发现c1r过度表达。另一方面,在喉咽癌患者中使用基于紫杉醇的治疗时观察到c1r失活(xu et al.,2013a;kawahara et al.,2016)。
[0223]
c2cd3被证明与口咽鳞状细胞癌有关(wang et al.,2013g)。
[0224]
c4orf46被证明在肾细胞癌中下调,c4orf46表达显示与透明细胞肾细胞癌的fuhrman分级呈负相关(yu et al.,2014)。
[0225]
之前报告过ca8在鳞状细胞癌、腺癌、腺细胞癌和结直肠癌中表达增加(akisawa et al.,2003)。ca8过度表达显示可诱导肺癌a549和人胚肾hek293细胞凋亡。另外,它抑制黑色素瘤、前列腺癌、肝癌和膀胱癌细胞的细胞增殖(liu et al.,2002a)。ca8的sirna
‑
介导敲减显示可显著抑制结肠癌细胞系hct116的细胞增殖和集落形成(nishikata et al.,2007)。
[0226]
caap1显示与癌症的药物抗性相关(wijdeven et al.,2015)。
[0227]
camsap1被证明与儿童急性淋巴细胞白血病的结果和喉鳞状细胞癌的预后相关(sun et al.,2014a;wang et al.,2015c)。camsap1被证明在喉鳞状细胞癌中上调(sun et al.,2014a)。
[0228]
cand1与前列腺癌和肺癌有关(zhai et al.,2014;salon et al.,2007)。
[0229]
最近的研究表明,与健康对照人群相比,canx在肺癌患者中过表达。这些结果提示,canx可用作肺癌的诊断标志物(kobayashi et al.,2015b)。canx在ht
‑
29细胞和mcf
‑
7人乳腺癌细胞中下调,相比于单层生长为集落(yeates and powis,1997)。
[0230]
cars基因的多态性与中国人群的乳腺癌发展相关。此外,cars表明与多形性胶质母细胞瘤的不同分子网具有显著较高的关联性(he et al.,2014;kim et al.,2012c)。
[0231]
ccar1与髓母细胞瘤、小细胞前列腺癌、结肠癌和非霍奇金淋巴瘤有关(bish and vogel,2014;levi et al.,2011;scott et al.,2014;ou et al.,2009)。ccar1显示在乳腺癌中下调(zhang et al.,2007)。
[0232]
ccdc110被证明与酵母双杂交系统中的高风险人乳头瘤病毒18e6癌基因相互作用,因此,是可用于癌症生物治疗的一种潜在致癌靶标(li et al.,2008b)。ccdc110被描述
为与多发性骨髓瘤相关的癌症
‑
睾丸抗原,这可能被用于接种患者(condomines et al.,2007)。ccdc110被证明是一种新型癌症
‑
睾丸抗原,其在各种类型癌症患者中引发体液免疫应答。因此,ccdc110可能是癌症免疫疗法的一个靶标(monji et al.,2004)。
[0233]
ccna1编码细胞周期蛋白a1,其属于参与cdk激酶调节的高度保守的细胞周期蛋白家族(refseq,2002)。在上皮性卵巢癌、淋巴母细胞白血病细胞系以及儿童急性淋巴细胞白血病患者中检测到ccna1水平升高。其他人观察到ccna1蛋白和mrna在前列腺癌和甲状腺未分化癌患者的肿瘤组织中过度表达(holm et al.,2006;wegiel et al.,2008;marlow et al.,2012;arsenic et al.,2015)。最近的研究表明,ccna1在表达ml1白血病细胞的高细胞周期蛋白a1中的沉寂减慢进入s期、降低增殖和抑制集落形成(ji et al.,2005)。
[0234]
ccna2的过度表达抑制肝细胞癌细胞的增殖。ccna2在子宫内膜腺癌细胞中的过度表达降低细胞生长并增加细胞凋亡。ccna2在黑色素瘤细胞中的过度表达降低肿瘤生长和转移,并且同时增加肿瘤细胞凋亡(lau,2011)。ccna2可促进癌细胞增殖、侵袭、黏附、分化、存活和转移。它在血管生成和细胞外基质的产生中起着重要作用。当在胃癌细胞中过度表达时,ccna2促进肿瘤生长并增加肿瘤血管形成。ccna2表达的沉寂降低胰腺癌细胞的肿瘤生长。ccna2可促进前列腺癌细胞的增殖(lau,2011;chen and du,2007)。ccna2过度表达诱导上皮
‑
间质转化,导致咽喉肿瘤的侵袭和转移(liu et al.,2015e)。ccna2在结直肠癌失调(chang et al.,2014)。ccna2在前列腺癌、神经胶质瘤、胰腺癌和乳腺癌中过度表达。ccna2与乳腺癌的侵袭性增加、血管化和雌激素非依赖性相关,这表明ccna2在乳腺癌进展中的主要作用(zuo et al.,2010)。
[0235]
ccnb2在结直肠腺癌中上调(park et al.,2007)。ccnb2在各种人肿瘤中过度表达。ccnb2在肿瘤细胞中的强表达与肺腺癌和浸润性乳腺癌的不良预后相关(takashima et al.,2014;albulescu,2013)。
[0236]
ccnb3
‑
bcor基因融合显示与未分化小圆形细胞肉瘤的癌症实体相关(haidar et al.,2015)。位于中轴骨骼和软组织的ccnb3
‑
bcor(尤因氏样)肉瘤被证明与相比于尤因氏肉瘤较短生存期相关(puls et al.,2014)。ccnb3被证明与cdk2相互作用,cdk2是参与细胞周期转变的蛋白质(nguyen et al.,2002)。
[0237]
在各种类型的癌症中,包括乳腺癌、结肠癌、胃癌、肺癌、子宫内膜上皮内癌、子宫浆液性癌和高级别浆液性卵巢癌中观察到了ccne1过度表达和扩增(donnellan and chetty,1999;kuhn et al.,2014;noske et al.,2015)。此外,ccne1表达增加是肺癌不良预后的有用标志物(huang et al.,2012)。一项研究表明,ccne1被mir
‑
497和mir
‑
34a下调,其协同地推迟人肺癌细胞的生长(han et al.,2015b)。
[0238]
研究表明,aml细胞长期体外增殖的急性髓细胞白血病患者显示ccnf表达改变(hatfield et al.,2014)。此外,ccnf表达与肝细胞癌患者的较差总体生存率和无复发生存率有关(fu et al.,2013a)。
[0239]
ccr4被描述为各种肿瘤的预后标志物,如肾细胞癌、头颈部鳞状细胞癌、胃癌、乳腺癌、结肠癌和霍奇金淋巴瘤(ishida et al.,2006;olkhanud et al.,2009;yang et al.,2011;tsujikawa et al.,2013;al
‑
haidari et al.,2013;liu et al.,2014d)。研究显示,有ccr4阳性肿瘤的胃癌患者与有ccr4阴性肿瘤的患者相比预后明显较差(lee et al.,2009)。
et al.,2005)。
[0259]
cep97与乳腺癌有关(rappa et al.,2014)。
[0260]
cers1在尼洛替尼抗性慢性髓性白血病细胞中下调(camgoz et al.,2013)。cers1生成的c(18)
‑
神经酰胺水平在头颈部鳞状细胞癌(hnscc)中显著下降。hnscc肿瘤组织中c(18)
‑
神经酰胺水平下降与淋巴管浸润和病理淋巴结转移的较高发生率相关(karahatay et al.,2007)。cers1生成的c(18)
‑
神经酰胺介导癌症细胞的细胞死亡(saddoughi and ogretmen,2013)。
[0261]
cers2被证明在脑膜瘤中下调(ke et al.,2014b)。cers2被证明在结直肠癌、肺鳞状细胞癌和乳腺癌中上调(moriya et al.,2012;chen et al.,2015c;schiffmann et al.,2009)。cers2与乳腺癌的转移和耐药性,前列腺的生长、侵袭和转移,膀胱癌和肝细胞癌的多样增殖、转移和侵袭有关(tang et al.,2010;zhao et al.,2013a;perez et al.,2014;xu et al.,2014a;zi et al.,2015)。cers2可能是结直肠癌、脑膜瘤和膀胱癌的潜在生物标志物(zhao et al.,2013a;ke et al.,2014b;chen et al.,2015c)。
[0262]
研究表明,cfb的表达在鼻咽癌患者的血清中减少。另一方面,与健康个体的血浆相比,cfb的表达在胰腺导管腺癌患者血浆样本中高2倍以上。其他人已经观察到cfb位点与黑色素瘤的关联性(budowle et al.,1982;seriramalu et al.,2010;lee et al.,2014a)。
[0263]
chchd7与多形性腺瘤有关(matsuyama et al.,2011)。
[0264]
chd7与皮肤t细胞淋巴瘤、cpg岛甲基化表型1结直肠癌、胃癌微卫星不稳定性和小细胞肺癌相关(kim et al.,2011b;tahara et al.,2014;litvinov et al.,2014b;pleasance et al.,2010)。chd7被证明在结肠癌中上调(scanlan et al.,2002)。chd7与胰腺癌的生存结果有关(colbert et al.,2014)。
[0265]
一项报告推测,chst1基因多态性可能解释结直肠癌患者中5
‑
氟尿嘧啶诱导的毒性。另一项研究发现,ln229胶质母细胞瘤细胞表达了升高水平的chst1(hayatsu et al.,2008;rumiato et al.,2013;arbitrio et al.,2016)。
[0266]
cklf被证明在高级别胶质瘤中上调(yang et al.,2013)。
[0267]
cldn16被证明在乳头状甲状腺癌和卵巢癌中上调(rangel et al.,2003;fluge et al.,2006)。cldn16表达被证明与乳腺癌的侵袭性、高死亡率和较差预后相关(martin et al.,2008;martin and jiang,2011)。cldn16被证明与肾癌有关(men et al.,2015)。cldn16被描述为乳腺癌的潜在生物标志物(kuo et al.,2010)。
[0268]
clspn在非小细胞肺癌(nsclc)中上调。clspn的过度表达与nsclc预后不良有关(allera
‑
moreau et al.,2012)。
[0269]
clstn3过度表达在睾丸癌以及人胚胎性癌中发现(dormeyer et al.,2008)。
[0270]
cnot1基因中的单核苷酸多态性(snp)在骨肉瘤和急性淋巴细胞白血病(all)中检测到(gutierrez
‑
camino et al.,2014;bilbao
‑
aldaiturriaga et al.,2015)。cnot1耗竭诱导mrna的稳定性和er应激介导细胞凋亡的启动(ito et al.,2011)。
[0271]
cnot4基因的单核苷酸多态性与骨肉瘤的风险相关(bilbao
‑
aldaiturriaga et al.,2015)。
[0272]
copa基因表达和rna编辑的变化被证明与肝细胞癌相关,实验研究显示了copa在
间皮瘤细胞中的抗凋亡作用(qi et al.,2014;sudo et al.,2010;wong et al.,2003)。
[0273]
shrna文库筛选确定了copb1为对2
‑
脱氧葡萄糖(癌细胞中糖酵解抑制剂)敏感性的决定因素。此外,copb1表达的沉寂让细胞对2
‑
脱氧葡萄糖毒性敏感(kobayashi et al.,2015a)。
[0274]
copb2在不同类型癌症,如乳腺癌、结肠癌、前列腺癌、胰腺癌、胶质母细胞瘤和肺腺癌中表达(erdogan et al.,2009)。其他人的研究提示,copb2参与间皮瘤的抗凋亡功能(sudo et al.,2010)。
[0275]
copg1(也称为copg)编码套体素蛋白复合体(copi)的γ亚基,其介导逆行运输(从高尔基体返回至er)和高尔基体内运输。copg1结合至arf
‑
gap(waters et al.,1991;watson et al.,2004)。copg1与患者年龄以及恶性肿瘤的较高等级以及神经胶质肉瘤的等级相关(coppola et al.,2014)。copg1被发现在肺癌和肺癌相关内皮细胞中大量表达(park et al.,2008)。
[0276]
基于功能的基因组筛选确定了copz1为不同肿瘤细胞的必需基因。copz1的敲减被证明可引起高尔基体塌陷、阻滞自噬并增殖和非分裂肿瘤细胞中诱导细胞凋亡。因此,copz1可能是一种新的治疗靶标,它提供了对肿瘤细胞的增殖非依赖性选择性杀伤的机会(shtutman et al.,2011)。
[0277]
coro2a的过度表达在乳腺癌和结肠癌中被发现(bubnov et al.,2012;rastetter et al.,2015)。研究人员发现,mapk14和prmt5信号通路在肿瘤进展中发挥关键作用(rastetter et al.,2015)。coro2a在结直肠癌细胞中下调与早期凋亡下降有关(kim et al.,2013a)。
[0278]
单核苷酸多态性以及csda基因突变与肝细胞癌有关。另一组发现,相较于相应的非肿瘤组织,csda在肝细胞癌中mrna表达水平较高。此外,相较于邻近的正常组织,在胃癌组织和细胞系中观察到csda水平升高(hayashi et al.,2002;wang et al.,2009a;yasen et al.,2012)。最近的工作表明,肝癌中csda水平升高与较差预后之间存在关联(yasen et al.,2005)。在慢性骨髓性白血病中,akt和mek/p90核蛋白体s6激酶可磷酸化csda的丝氨酸134残基(sears et al.,2010)。
[0279]
cse1l被证明在肝细胞癌、膀胱尿路上皮癌、浆液性卵巢癌、乳腺癌和转移性癌症中高表达(behrens et al.,2001;tung et al.,2009;stawerski et al.,2010;tai et al.,2012;zang et al.,2012)。研究人员证明,cse1l调节结直肠癌细胞mmp
‑
2的易位和分泌(liao et al.,2008;tsao et al.,2009)。此外,抑制mek1介导的磷酸化导致紫杉醇(紫杉醇)诱导的凋亡在乳腺癌、卵巢癌和肺癌细胞系中增强。因为cse1l也由mek1启动,透过mek1抑制改变cse1l的活性/磷酸化状态可呈现实验癌症治疗的潜在策略(behrens et al.,2003)。
[0280]
ct45基因被证明是上皮卵巢癌的潜在预后生物标志物和治疗靶标(zhang et al.,2015l)。通常仅在睾丸生殖细胞中表达的ct45a1蛋白被证明也在肺癌、乳腺癌和卵巢癌中表达(chen et al.,2009d)。ct45a1也被证明与多发性骨髓瘤的较差预后和较差结果相关(andrade et al.,2009)。ct45a1被描述为基因上调上皮
‑
间质转化(emt)和转移基因,促进emt和肿瘤的扩散。此外,ct45a1被描述为牵涉癌症干细胞样细胞的启动和维持,促进肿瘤发生和恶性进展(yang et al.,2015a)。ct45a1在乳腺癌模型中过度表达显示可导致
各种致癌和转移基因的上调,组成性活化的erk和creb信号通路并增加肿瘤发生、浸润和转移。ct45a1的沉寂被证明可降低癌细胞的迁移和侵袭。因此,ct45a1可作为一种新的原癌基因,并能是抗癌药物开发和治疗的靶标(shang et al.,2014)。
[0281]
ct45a2被证明是新双表型急性白血病儿科患者的新型剪接mll融合伴侣,因此可能与白血病的发生相关(cerveira et al.,2010)。癌/睾丸抗原家族45被证明在癌细胞系和肺癌标本中频繁表达(chen et al.,2005)。ct45基因被证明是上皮卵巢癌的潜在预后生物标志物和治疗靶标(zhang et al.,2015l)。
[0282]
癌/睾丸抗原家族45被证明在癌细胞系和肺癌标本中频繁表达(chen et al.,2005)。ct45基因被证明是上皮卵巢癌的潜在预后生物标志物和治疗靶标(zhang et al.,2015l)。
[0283]
癌/睾丸抗原家族45被证明在癌细胞系和肺癌标本中频繁表达(chen et al.,2005)。ct45基因被证明是上皮卵巢癌的潜在预后生物标志物和治疗靶标(zhang et al.,2015l)。
[0284]
癌/睾丸抗原家族45被证明在癌细胞系和肺癌标本中频繁表达(chen et al.,2005)。ct45基因被证明是上皮卵巢癌的潜在预后生物标志物和治疗靶标(zhang et al.,2015l)。
[0285]
癌/睾丸抗原家族45被证明在癌细胞系和肺癌标本中频繁表达(chen et al.,2005)。ct45基因被证明是上皮卵巢癌的潜在预后生物标志物和治疗靶标(zhang et al.,2015l)。
[0286]
与正常的黏膜相比,ctsa水平在鳞状细胞癌中水平升高。其他人检测到相较于原发性局灶裂解物,ctsa活性在恶性黑色素瘤的转移灶裂解物中水平更高。另一项报告表明,相较于眼内肿瘤患者,ctsa活性在绝对青光眼患者的玻璃体中高2倍(obuchowska et al.,1999;kozlowski et al.,2000;marques filho et al.,2006)。
[0287]
cyfip1被证明在上皮性肿瘤侵袭期间下调(silva et al.,2009)。cyfip1下调与上皮肿瘤的预后不良相关(silva et al.,2009)。
[0288]
cyfip2表达在乳腺癌新形成淋巴结中增加(gantsev et al.,2013)。与对照组织相比,cyfip2表达在人胃肿瘤样本中降低(cheng et al.,2013b)。cyfip2是慢性淋巴细胞性白血病甲基化的几种凋亡相关基因之一(halldorsdottir et al.,2012)。
[0289]
cyp2f1的表达发现于原发性卵巢癌和非癌性鼻咽组织中。但是,在乳腺肿瘤以及对照组织中不存在(downie et al.,2005;iscan et al.,2001;jiang et al.,2004)。在结直肠癌中,cyp2f1在淋巴结转移癌中的表达与其在相应原发性肿瘤是否存在强烈相关(kumarakulasingham et al.,2005)。
[0290]
cyp4x1被证明是乳腺癌中含cyp4z2p的截止框融合转录物(kim et al.,2015a)。cyp4x1被证明与乳腺癌肿瘤分级相关,可能是潜在的生物标志物以帮助对最佳辅助激素治疗作出决定(murray et al.,2010)。cyp4x1被证明是er
‑
β过度表达hek293细胞系中雌激素受体β(er
‑
β)的潜在主要靶标(zhao et al.,2009)。
[0291]
cyp7b1基因的单个多态性与前列腺癌的风险相关。此外,cyp7b1水平升高发现于高级别前列腺上皮内瘤、腺癌和乳腺癌中(jakobsson et al.,2004;olsson et al.,2007;pu et al.,2015)。
[0292]
dcbld2在胶质母细胞瘤和头颈部癌症(hncs)中上调,是egfr刺激肿瘤发生所需要的(feng et al.,2014)。此外,dcbld2在高度转移性肺癌亚系和组织样本中上调(koshikawa et al.,2002)。与此相反,在胃癌中,dcbld2的表达透过其启动子的甲基化而沉寂(kim et al.,2008b)。
[0293]
dchs2与具有高微卫星不稳定性的胃癌和结直肠癌有关(an et al.,2015)。
[0294]
ddx11属于dna解旋酶的deah家族,在晚期黑色素瘤中高度表达,是黑色素瘤细胞的生存所必需的(bhattacharya et al.,2012)。
[0295]
dx20被证明在肝细胞癌中下调(takata et al.,2013a)。ddx20与结直肠癌和膀胱癌的风险增加,乳腺癌总体存活率下降以及转移可能性增加有关(yang et al.,2008a;zhao et al.,2015b;shin et al.,2014)。ddx20可能是乳腺癌的预后生物标志物(shin et al.,2014)。
[0296]
ddx41与急性骨髓性白血病相关(antony
‑
debre and steidl,2015)。
[0297]
ddx47可能是鉴别慢性粒细胞白血病不同疾病分期的潜在标志物(oehler et al.,2009)。
[0298]
ddx6被发现在结直肠腺癌、胃癌、肝细胞癌、结节边缘区淋巴瘤、神经母细胞瘤、横纹肌肉瘤和肺癌细胞系中过度表达(akao et al.,1995;nakagawa et al.,1999;miyaji et al.,2003;lin et al.,2008a;stary et al.,2013;iio et al.,2013)。在结节边缘区淋巴瘤中,ddx6似乎以非依赖性方式干扰bcl6和bcl2的表达(stary et al.,2013)。最近的研究表明,ddx6转录后透过促进其宿主基因产物ncr143/145rna的降解下调mir
‑
143/145的表达(iio et al.,2013)。
[0299]
depdc1b被证明在口腔癌和非小细胞肺癌中上调(yang et al.,2014e;su et al.,2014)。depdc1b的表达与患者生存、非小细胞肺癌的迁移和转移以及淋巴母细胞肿瘤细胞系的放射敏感性相关(niu et al.,2010;yang et al.,2014e)。
[0300]
dffb基因高水平在膀胱癌顺铂耐药中检测到,而dffb水平在1p等位基因缺失的少突胶质细胞瘤中降低。另一组发现dffb基因在神经母细胞瘤中无突变(judson et al.,2000;mcdonald et al.,2005;kim et al.,2016)。dffb过度表达导致乙酰唑胺和磺胺苯酰培养的乳腺癌细胞生存力降低。此外,这些组中,凋亡增加,特别是使用乙酰唑胺的组。同样地,与gm
‑
csf融合的dffb被发现透过诱导细胞凋亡而促进急性髓性白血病细胞的靶向杀伤(mathew et al.,2013;bagheri et al.,2014)。
[0301]
dfna5表达被认为在肝细胞癌细胞、雌激素受体(er)阳性乳腺癌和胃癌细胞系中较低(thompson and weigel,1998;akino et al.,2007;wang et al.,2013c)。而且,黑色素瘤细胞的依托泊苷耐药性与dfna5表达降低有关(lage et al.,2001)。dfna5的敲减导致结直肠癌细胞系的细胞浸润、集落数、菌落大小和细胞生长增加(kim et al.,2008c)。
[0302]
ddx40与上皮性卵巢癌相关(zheng et al.,2004)。
[0303]
prognoscan数据库显示,dhx8在膀胱癌、血癌、脑癌、乳腺癌、结直肠癌、眼癌、头颈部癌、肺癌、卵巢癌、皮肤癌、软组织癌组织中表达(wang et al.,2014f)。研究人员观察到,dhx8同时存在于正常肾上腺皮质以及恶性肾上腺皮质癌中(szabo et al.,2009)。
[0304]
def被证明可介导肿瘤抑制基因p53的非蛋白酶体降解(tao et al.,2013)。
[0305]
研究表明,dlg5在前列腺癌以及膀胱癌中下调。另一方面,在胰腺导管腺癌中观察
到dlg5过度表达。此外,dlg5基因的单核苷酸多态性与结直肠癌的风险无关(taniuchi et al.,2005;suchy et al.,2008;tomiyama et al.,2015;zhou et al.,2015b)。内源性dlg5的敲减导致前列腺癌细胞的迁移和侵袭增加,同时抑制胰腺导管腺癌的生长(taniuchi et al.,2005;tomiyama et al.,2015)。
[0306]
dmxl2被证明在er
‑
α阳性乳腺癌中上调(faronato et al.,2015)。dmxl2是er
‑
α阳性乳腺癌的一种功能性生物标志物(faronato et al.,2015)。
[0307]
dnah3与结肠癌有关(tanaka et al.,2008a)。
[0308]
研究在口腔鳞状细胞癌患者中检测到了dnase1l1。此外,dnase1l1的表达与这些患者的无病生存率较差相关(grimm et al.,2013)。
[0309]
dock8被证明在肺鳞状细胞癌中下调(kang et al.,2010)。dock8被证明与神经母细胞瘤、小儿毛细胞型星形细胞瘤、肝细胞癌、神经胶质瘤和肺癌有关(schramm et al.,2015;saelee et al.,2009;takahashi et al.,2006;zhao et al.,2014a;idbaih et al.,2008)。dock8被证明与食管癌细胞系te
‑
11的辐射敏感性相关(ogawa et al.,2008)。
[0310]
一份报告显示,dpp3在胶质瘤细胞以及鳞状细胞肺癌中过度表达。同样地,与正常组织中的活性相比,在子宫内膜癌和卵巢恶性肿瘤中观察到更高的dpp3活性(simaga et al.,1998;simaga et al.,2003;hast et al.,2013;singh et al.,2014)。
[0311]
dppa4被证明在结肠癌中上调(zhang et al.,2015j)。dppa4与膀胱癌、前列腺癌、胚胎性癌、多能生殖细胞瘤和肉瘤相关(tung et al.,2013;amini et al.,2014)。dppa4与结肠癌的分期、浸润深度、远处转移和分化程度有关(zhang et al.,2015j)。dppa4是结肠癌无病生存和总体生存的独立预后指标(zhang et al.,2015j)。
[0312]
dtx3l被证明在黑色素瘤、宫颈鳞状细胞癌和有明显炎性浸润的弥漫性大b细胞淋巴瘤中上调(thang et al.,2015;wilting et al.,2008;juszczynski et al.,2006)。dtx3l被证明可透过fak/pi3k/akt信号通路调节黑色素瘤的侵袭和转移的调节。因此,dtx3l可作为黑色素瘤的潜在治疗靶以及潜在生物标志物(thang et al.,2015)。dtx3l被描述为一种化疗耐药因子,在ezh2获取功能的突变弥漫性大b细胞淋巴瘤中上调(johnson et al.,2015)。dtx3l被证明是转移性前列腺癌细胞中的一种新颖致癌因子,其与致癌蛋白artd8和artd9相互作用介导转移性前列腺癌的增殖、化学抗性和生存(bachmann et al.,2014)。dtx3l被证明与转移性前列腺癌细胞中的转录因子stat1和stat3以及肿瘤抑制因子irf1相关(bachmann et al.,2014)。dtx3l被描述为dna损伤应答途径的真正成员,其与肿瘤抑制因子brca1的parp1启动和招募直接相关(yan et al.,2013b)。
[0313]
整个外显子测序研究发现胰腺内导管内乳头状黏液性肿瘤患者的dync1h1基因内有体细胞突变(furukawa et al.,2011)。
[0314]
dync2h1被证明在多形性胶质母细胞瘤中上调(yokota et al.,2006)。
[0315]
与良性卵巢疾病相比,egflam启动子cgi甲基化率在上皮性卵巢癌中降低(gu et al.,2009)。egflam的启动子cgi可能卵巢癌特异性低甲基化肿瘤标志物的一种新型候选(gu et al.,2009)。
[0316]
eif2s2被证明在高度增殖性管腔乳腺肿瘤患者中扩增(gatza et al.,2014)。
[0317]
eif2s3在结直肠癌患者外周血样本和健康对照组之间差异表达的5个分子标志物之一(chang et al.,2014c)。eif2s3与n
‑
myc下游调节基因1(ndrg1)相互作用,ndrg1在前
列腺癌转移的细胞分化和抑制中发挥作用(tu et al.,2007)。
[0318]
eif3c在结肠癌中高表达(song et al.,2013)。eif3c mrna在睾丸精原细胞瘤中过度表达(rothe et al.,2000)。
[0319]
据报告,eif3f在胰腺癌和黑色素瘤中表达下调。此外,与正常组织相比,在黑色素瘤和胰腺肿瘤中均被证实eif3f损失并且基因拷贝数有统计学显著的降低(shi et al.,2006;doldan et al.,2008a;doldan et al.,2008b)。最近的工作表明,eif3f表达降低可作为受胃癌影响患者的不良结果的预后标记(cheng et al.,2015a)。高水平eif3f在黑色素瘤和胰腺癌细胞中抑制细胞增殖并诱导细胞凋亡(shi et al.,2006;doldan et al.,2008a)。
[0320]
eif4g3在弥漫性大b细胞淋巴瘤中上调。此外,透过sirna的eif4g3下调导致翻译、细胞增殖和形成集落能力的下降并诱导细胞衰老(mazan
‑
mamczarz et al.,2014)。
[0321]
emc10上调被证明与高级别胶质瘤和参与肿瘤发生信号途径的调节相关(junes
‑
gill et al.,2011;junes
‑
gill et al.,2014)。emc10被证明可抑制神经胶质瘤诱导的细胞周期进程、细胞迁移、侵袭和血管生成,因此,可能是恶性胶质母细胞瘤的一个治疗靶标(junes
‑
gill et al.,2014)。
[0322]
使用皂苷d治疗后,在肝细胞癌细胞系中发现到emg1下调(lu et al.,2015)。
[0323]
epg5与乳腺癌有关(halama et al.,2007)。
[0324]
eppk1被证明与肝内胆管细胞癌和子宫颈鳞状细胞癌相关(zou et al.,2014b;guo et al.,2015)。
[0325]
erlin2与乳腺癌和肝细胞癌有关(wang et al.,2012a)。
[0326]
ermp1被证明与乳腺癌有关(wu et al.,2012c)。
[0327]
esr1的突变和单核苷酸多态性与不同癌症类型的风险相关,包括肝癌、前列腺癌、胆囊和乳腺癌。esr1表达的上调与细胞增殖和肿瘤生长相关,但是,esr1阳性肿瘤患者由于使用选择性雌激素受体调节剂成功治疗总体生存更好(sun et al.,2015c;hayashi et al.,2003;bogush et al.,2009;miyoshi et al.,2010;xu et al.,2011;yakimchuk et al.,2013;fuqua et al.,2014)。esr1信令干扰负责细胞转化、生长和存活的不同途径,如egfr/igfr、pi3k/akt/mtor、p53、her2、nfκb和tgf
‑
β途径(frasor et al.,2015;band and laiho,2011;berger et al.,2013;skandalis et al.,2014;mehta and tripathy,2014;ciruelos gil,2014)。
[0328]
esrrg信令与他莫昔芬治疗后er+乳腺癌的无远处转移生存期下降有关(madhavan et al.,2015)。最近的工作表明,esrrg透过akt和erk1/2信号途径的启动介导雌激素对子宫内膜癌细胞增殖的影响(sun et al.,2014c)。高水平esrrg诱导er+乳腺癌细胞的增殖,不论存在或不存在雌激素。与此相反,esrrg的沉寂抑制肝癌细胞系的生长并诱导细胞凋亡(ijichi et al.,2011;yuan et al.,2015)。
[0329]
exoc8被证明与大脑中的癌症相关ras样小gtp酶rala相互作用(das et al.,2014)。exoc8与rala的相互作用被描述为前列腺癌肿瘤细胞的迁移和侵袭所需要的(hazelett and yeaman,2012)。exoc8被证明参与真皮成纤维细胞的肿瘤促进作用,它由rala执行。真皮成纤维细胞的rala信号级联涉及exoc8,并被描述为皮肤鳞状细胞癌进展的一种潜在抗癌靶标(sowalsky et al.,2011)。exoc8被描述为促进致癌ras介导肿瘤发生的
一种蛋白(issaq et al.,2010)。
[0330]
由于dna低甲基化,exosc4启动子活性在肝细胞癌增加。exosc4有效和特异地抑制癌细胞生长和细胞侵入能力(stefanska et al.,2014;drazkowska et al.,2013)。
[0331]
exosc7与宫颈癌有关(choi et al.,2007)。
[0332]
与相邻的正常组织相比,在胃肿瘤组织中,eya1表达显著降低。此外,eya1在肾母细胞瘤中显著过度表达(li et al.,2002;nikpour et al.,2014)。据报告,eya1的基因沉寂使乳腺癌细胞对pi3k/akt信号的药理学抑制显著敏感。这些发现提示,它们可一起发挥作用来调节癌细胞行为(sun et al.,2015g)。
[0333]
在几种肿瘤类型中,如上皮性卵巢肿瘤、前列腺癌、乳腺癌、泌尿道癌、胶质母细胞瘤、肺腺癌、宫颈癌、结肠癌和造血癌症中观察到eya2过度表达(bierkens et al.,2013;zhang et al.,2005a;guo et al.,2009;patrick et al.,2013;kohrt et al.,2014)。研究表明,eya2影响tgfβ途径成员的转录以及tgfbr2的磷酸化,这意味着eya2在胰腺中具有双重作用(vincent et al.,2014)。
[0334]
与间质干细胞相比,eya3在尤因氏肉瘤肿瘤样本和细胞系中高度表达。另一方面,eya3基因缺失与某些胰腺导管腺癌相关(gutierrez et al.,2011;robin et al.,2012)。最近的工作表明,eya3过度表达导致乳腺癌细胞的增殖、迁移、浸润和转化增加(pandey et al.,2010)。
[0335]
据报告,eya4在非小细胞肺癌亚型以及肺癌最早期阶段和原位腺癌、结直肠癌和肝细胞癌中被经常和伴随删除、超甲基化和低表达(selamat et al.,2011;wilson et al.,2014;hou et al.,2014;kim et al.,2015c)。在结直肠癌中,eya4是一种肿瘤抑制基因,透过诱导上调dkk1和抑制wnt信号通路起作用(kim et al.,2015c)。
[0336]
表达分析表明,肾细胞癌患者肿瘤或尿液中以及胶质母细胞瘤和黑色素瘤组织中有fabp7转录物(liang et al.,2005;seliger et al.,2005;goto et al.,2010;takaoka et al.,2011)。此外,fabp7在胶质母细胞瘤和黑色素瘤中过度表达与生存期较短相关(liang et al.,2006;slipicevic et al.,2008)。在胶质瘤细胞系中,nfi去磷酸化与fabp7表达相关(bisgrove et al.,2000)。
[0337]
fads2在肝细胞癌中上调(muir et al.,2013)。fads2活性在乳腺癌组织中增加(pender
‑
cudlip et al.,2013)。fads2表达与乳腺癌侵袭性有关(lane et al.,2003)。fads2抑制阻碍肠肿瘤发生(hansen
‑
petrik et al.,2002)。
[0338]
fam135b与食管鳞状细胞癌相关(song et al.,2014b)。
[0339]
fam86a被证明与肿瘤相关真核延伸因子2相互作用(davydova et al.,2014)。
[0340]
由于基因突变导致的fancd2下调和功能障碍在不同癌症类型中报告过,包括乳腺癌、急性淋巴性白血病和睾丸精原细胞瘤,并与癌症发展有关。另外,fancd2再次表达和上调被证明与胶质瘤和结直肠癌的肿瘤进展和转移有关(patil et al.,2014;shen et al.,2015a;shen et al.,2015b;ozawa et al.,2010;rudland et al.,2010;zhang et al.,2010a;smetsers et al.,2012)。pi3k/mtor/akt途径促进fancd2诱导atm/chk2检查点作为dna损伤应答,并且单泛素化fancd2启动肿瘤抑制因子tap63的转录(shen et al.,2013;park et al.,2013)。
[0341]
fancg mrna在新诊断的急性骨髓性白血病患者中的表达水平明显低于对照组和
急性骨髓性白血病完全缓解组。此外,fancg的种系突变可能有助于胰腺癌的进展。与此相反,fancg突变在膀胱癌细胞系中不能检测到(couch et al.,2005;neveling et al.,2007;duan et al.,2013)。人腺癌细胞系中fancg的内源破坏导致诱裂损伤、g2/m期阻滞增加以及细胞增殖减少(gallmeier et al.,2006)。
[0342]
fat2基因的突变发现于食管鳞状细胞癌以及头颈部鳞状细胞癌。此外,fat2 mrna在胃癌、胰腺癌和卵巢癌中表达(katoh and katoh,2006;lin et al.,2014;gao et al.,2014)
[0343]
fat3显示,当雄激素受体沉寂后,在紫杉醇抗卵巢癌细胞系中下调,导致在这些细胞系中对紫杉醇敏感。因此,fat3可能是与紫杉醇抗性相关的候选基因(sun et al.,2015e)。fat3被证明在食管鳞状细胞癌中突变,导致河马信号传导途径的失调(gao et al.,2014)。fat3被证明在早期t细胞前体急性淋巴细胞白血病中周期性突变(neumann et al.,2013)。fat3被描述为一种具有脑膜脑膜瘤特定签名的基因,因此,与这种亚型的良性脑膜瘤肿瘤发生有关(fevre
‑
montange et al.,2009)。fat3被描述为一种肿瘤抑制因子,对发育不良细胞的肺癌发展有抑制作用(rohrbeck and borlak,2009)。
[0344]
fbxo4被证明在肝细胞癌中下调(chu et al.,2014)。fbxo4与食管鳞状细胞癌、黑色素瘤、淋巴瘤和组织细胞肉瘤相关(vaites et al.,2011;lee et al.,2013b;lian et al.,2015)。
[0345]
fbxo5被证明在乳腺癌和肝细胞癌中上调(zhao et al.,2013c;liu et al.,2014h)。fbxo5被证明在原发性胃癌中下调(zhang et al.,2014e)。fbxo5与乳腺癌的侵袭和转移可能性,胃癌的肿瘤大小、浸润、临床分期及预后,乳腺癌的组织学分级,卵巢透明细胞癌的组织学分级和较差总体生存期,肝细胞癌的分期及预后以及食管鳞状细胞癌的较差预后相关(kogo et al.,2011;zhao et al.,2013c;min et al.,2013;liu et al.,2013d;zhang et al.,2014e;liu et al.,2014h)。fbxo5与乳腺癌、卵巢癌、肝细胞癌、前列腺癌和套细胞淋巴瘤相关(johansson et al.,2014;schraders et al.,2008)。fbxo5是乳腺癌和食管鳞状细胞癌的预后预测因子(kogo et al.,2011;liu et al.,2014h)。
[0346]
fbxw8被证明在绒毛膜癌中上调(shi et al.,2014a)。fbxw8与胰腺癌和绒毛膜癌有关(wang et al.,2014b;lin et al.,2011)。
[0347]
fgfr1op与慢性粒细胞白血病、急性髓性白血病和骨髓增殖瘤有关(hu et al.,2011;bossi et al.,2014)。fgfr1op被证明在肺癌中上调(mano et al.,2007)。fgfr1op表达与较短的肿瘤特异性生存时间相关(mano et al.,2007)。fgfr1op是肺癌的预后生物标志物(mano et al.,2007)。
[0348]
与非致瘤性细胞相比,fig4在三阴性乳腺癌中被发现过度表达(ikonomov et al.,2013)。
[0349]
flad1被证明与非小细胞肺癌相关(mitra et al.,2011)。
[0350]
根据其亚细胞定位,细丝蛋白a在癌症中起双重作用:在细胞质中,细丝蛋白a除了参与细胞迁移和黏附通路外,还在不同的生长信号通路中发挥作用。因此,其过度表达有肿瘤促进作用。相比全长细丝蛋白a,c端片段(蛋白质蛋白水解后释放)定位于细胞核,在那里,它与转录因子相互作用,从而抑制肿瘤生长和转移(savoy and ghosh,2013)。
[0351]
foxm1过度表达发现于多种侵袭性人类癌症,包括肺癌、胶质母细胞瘤、前列腺癌、
基底细胞癌、肝细胞癌、原发性乳腺癌和胰腺癌(teh et al.,2002;kalinichenko et al.,2004;kalin et al.,2006;kim et al.,2006;liu et al.,2006;wang et al.,2007b)。最近的研究表明,foxm1基因由于sonic hedgehog通路的转录调控而在胰腺癌中上调(katoh and katoh,2004)。
[0352]
若干证据提示癌症中存在gab2,例如,乳腺癌、卵巢癌以及一些转移性黑色素瘤中发现gab2水平升高。其他人的研究显示,gab2是慢性粒细胞白血病中bcr/abl介导的转化所需要的(sattler et al.,2002;daly et al.,2002;horst et al.,2009;wang et al.,2012c)。在卵巢癌中,gab2过度表达导致磷脂酰肌醇3
‑
激酶途径的活化(dunn et al.,2014)。
[0353]
gadd45gip1被证明与白血病相关lck相互作用(vahedi et al.,2015)。add45gip1被证明在急性髓性白血病中下调(ran et al.,2014)。gadd45gip1被证明在宫颈癌和卵巢癌细胞系hela和skov3中有肿瘤抑制效果(nakayama et al.,2007)。gadd45gip1被证明与前列腺癌中的肿瘤抑制因子stat3相互作用,并与cdk2相互作用作为一种细胞周期蛋白依赖性激酶抑制剂(ran et al.,2014;tan et al.,2014)。gadd45gip1被证明由nac1负调节,nac1被认为对卵巢癌和宫颈癌的预后有不良影响(nishi et al.,2012)。gadd45gip1被证明与卵巢癌中的紫杉醇抗性相关(jinawath et al.,2009)。gadd45gip1可透过其作为ar辅阻遏物在前列腺雄激素受体(ar)阳性生长的调节中发挥着重要作用(suh et al.,2008)。gadd45gip1被证明在淋巴结(+)乳腺癌中上调(abba et al.,2007)。
[0354]
gart的表达在人胶质瘤和肝细胞癌中显著上调。gart的单核苷酸多态性与中国人群的肝细胞癌风险显著相关(liu et al.,2014g;cong et al.,2014;zhang et al.,2015e)。在肝细胞癌中,gart过度表达与组织学分级、肿瘤大小、肿瘤结节数量和肝内转移呈正相关(cong et al.,2014)。gart可透过靶向作用于肿瘤相关巨噬细胞而充当肾细胞癌肿瘤进展和生存的调节因子(ohba et al.,2005)。
[0355]
gas2l3被证明在致死剂量(213)bi
‑
d9mab培育后在胃癌细胞系hsc45
‑
m2中下调。因此,gas2l3可能是肿瘤细胞选择性消除的一个新靶标(seidl et al.,2010)。
[0356]
gbgt1与卵巢癌和口腔鳞状细胞癌相关(viswanathan et al.,2003;jacob et al.,2014)。
[0357]
ggt6被证明在脉络丛乳头状瘤患者中被扩增(de leon et al.,2015)。
[0358]
研究人员发现,与正常腺体组织相比,gnb1的mrna转录水平在乳腺癌标本中更高。与对照组相比,在子宫内膜癌中,gnb1的表达显著改变(orchel et al.,2012;wazir et al.,2013)。此外,gnb1的mrna表达随着tnm分期、肿瘤分级而升高,并与患者的不良结果相关(wazir et al.,2013)。
[0359]
gon4l与肝细胞癌和唾液腺癌症相关(simons et al.,2013;kim et al.,2009b)。
[0360]
gp1ba基因的串联重复多态性的可变数目与口腔癌和乳腺癌的风险有关。相反,其他人没有检测到gp1ba基因串联重复多态性的可变数目与乳腺癌侵袭性有任何关联(oleksowicz et al.,1998;ayala et al.,2003;vairaktaris et al.,2007)。在乳腺癌中,gp1ba表达与肿瘤分期、肿瘤大小和雌激素受体阴性显著相关(oleksowicz et al.,1998)。
[0361]
gpd2丰度和活性在前列腺癌细胞中显著上调,并与癌细胞中的高活性氧簇(ros)
的产生相关(chowdhury et al.,2005;chowdhury et al.,2007)。
[0362]
在乳腺癌细胞系中,gpr64敲减导致细胞黏附和细胞迁移大大降低(peeters et al.,2015)。
[0363]
gpx5 rs451774被发现与接受铂类+吉西他滨治疗的非小细胞肺癌患者的总体存活相关(li et al.,2011c)。
[0364]
gramd1a被证明在癌细胞系中表达(song et al.,2014a)。
[0365]
grhl2被证明在结直肠癌和口腔鳞状细胞癌中上调(quan et al.,2015b;kang et al.,2009)。grhl2被证明在宫颈癌和各种乳腺癌子类中下调(cieply et al.,2012;torres
‑
reyes et al.,2014)。grhl2被证明与结直肠癌的较差预后、透明细胞肾细胞癌的较低无病生存率和乳腺癌的较差无复发生存率相关(butz et al.,2014;quan et al.,2015b;xiang et al.,2012)。grhl2被证明与乳腺癌和肝细胞癌转移相关(tanaka et al.,2008b;werner et al.,2013)。grhl2可能是结直肠癌、透明细胞肾细胞癌和肝细胞癌的预后生物标志物(butz et al.,2014;quan et al.,2015b;tanaka et al.,2008b)。
[0366]
grik3与肺腺癌(甲基化、功能修饰)、儿童中枢神经系统肿瘤、淋巴细胞性白血病和神经母细胞瘤相关(pradhan et al.,2013)。grik3在中枢神经系统的几个儿科肿瘤中差异表达(brocke et al.,2010)。
[0367]
grin2d的过度表达或体细胞突变发现于儿科中枢神经系统肿瘤、人乳腺癌以及前列腺癌细胞系中。此外,grin2d敲减不影响te671和rpmi8226癌细胞系中的癌症表型(brocke et al.,2010;pissimissis et al.,2009;luksch et al.,2011;jiao et al.,2012)。
[0368]
gsdma被描述为在胃癌细胞系中经常沉寂,与细胞凋亡相关(lin et al.,2015a)。gsdma被证明在不同癌症的3'
‑
utr中突变,导致推定的微小rna靶标位点的创建或破坏,因而,可能导致基因表达的失调(ziebarth et al.,2012)。食道癌和胃癌的gsdma表达分析表明,gsdma是一种肿瘤抑制基因(saeki et al.,2009)。
[0369]
乳腺癌患者与对照组相比,表现出更高频率的gstm1基因纯合性缺失。gstm1基因的基因多态性与伊朗人群的膀胱癌易感性,中国人群的肺癌风险,印度人群的前列腺癌、食管癌和宫颈癌有关(mittal et al.,2004;singh et al.,2008;safarinejad et al.,2013;sharma et al.,2013;possuelo et al.,2013;chen et al.,2014g)。
[0370]
研究显示,相对于正常样本,巴雷特腺癌中存在gstm5的频繁下调和启动子dna超甲基化。另一方面,在急性淋巴细胞白血病患者中没有检测到gstm5转录物(kearns et al.,2003;peng et al.,2009)。研究人员观察到,gstm5基因的单核苷酸多态性可影响i至ii期或较低期别非小细胞肺癌的总体生存(pankratz et al.,2011)。
[0371]
gstt2启动子多态性及其单倍型与在韩国人群中的结直肠癌风险相关。另一些报告称,gstt2基因的缺失可能对南非混血人群食管鳞状细胞癌的发生和发展具有保护作用。此外,在巴雷特腺癌中发现了较低频率的gstt2基因dna甲基化(peng et al.,2009;jang et al.,2007;matejcic et al.,2011)。
[0372]
gstt2b被证明与食管鳞状细胞癌相关,因为gstt2b缺失对南非混血人群中引发食管鳞状细胞癌的风险具有潜在的保护作用(matejcic et al.,2011)。
[0373]
据报告,gtf2h4中单核苷酸多态性可增加发生吸烟相关肺癌和乳头状瘤病毒诱导
的宫颈癌的风险(buch et al.,2012;mydlikova et al.,2010;wang et al.,2010)。
[0374]
研究人员观察到甲状腺癌中的gtf2ird1
‑
alk融合(stransky et al.,2014)。
[0375]
研究人员在斯皮茨肿瘤队列中确定gtf3c2为一种新型alk融合基因(yeh et al.,2015)。
[0376]
若干出版物报告了h2afy在各种人类癌症中下调,包括结直肠癌、肺癌、睾丸癌、膀胱癌、宫颈癌、乳腺癌、结肠癌、卵巢癌和子宫内膜癌(novikov et al.,2011;sporn and jung,2012)。此外,黑色素瘤细胞中h2afy的敲减导致体内增殖和迁移以及体外生长和转移显著增加(kapoor et al.,2010)。在膀胱癌中,h2afy表达耗竭与lin28b表达水平升高显著有关(park et al.,2016)。
[0377]
haus3与乳腺癌有关(shah et al.,2009)。
[0378]
hdgf高水平表达与乳腺癌和胰腺导管癌预后不良相关(uyama et al.,2006;chen et al.,2012b)。研究表明,hdgf在诱导癌细胞增殖、血管生成、侵袭和转移中起着重要作用,如口腔鳞状细胞癌、胃癌、结肠癌、肺癌和食道癌(yamamoto et al.,2007;mao et al.,2008;liao et al.,2010;meng et al.,2010;lin et al.,2012;tao et al.,2014a)。
[0379]
heatr1被证明在胶质母细胞瘤中上调(wu et al.,2014c)。
[0380]
helq被描述与rad51旁系复合体bcdx2相互作用。这种复合体的不同组件分别与卵巢癌和乳腺癌的风险增加相关(pelttari et al.,2015)。helq被证明是一个候选卵巢癌基因,因为它与rad51旁系有关(takata et al.,2013b)。helq作为聚合酶途径的一部分,被证明与口腔/咽癌有关,因为在第二外显子中错义突变(babron et al.,2014)。helq被证明在dna修复和肿瘤抑制中发挥作用(adelman et al.,2013)。使用全基因组关联分析显示,helq与中国汉族人群的食管鳞状细胞癌相关(li et al.,2013b)。
[0381]
helz2被证明是基因系列中一个生物标志物,可早期诊断上皮性卵巢癌(pils et al.,2013)。
[0382]
染色体15q13.1上的herc2/oca2区域是易患皮肤黑素瘤的几个位点之一(amos et al.,2011;xiao et al.,2014)。herc2调节不同dna修复因子的稳定性,包括chk1、p53和brca1(bekker
‑
jensen et al.,2010;cubillos
‑
rojas et al.,2014;zhu et al.,2014a;peng et al.,2015c)。
[0383]
hint1在各种癌症中转录沉寂或下调,包括肝细胞癌、一些人非小细胞肺癌细胞系和胃癌。与此相反,hint1在前列腺癌中过度表达(zhang et al.,2009;huang et al.,2011;symes et al.,2013)。已经观察到肝癌细胞系中的hint1经由tcf4抑制wnt/β
‑
连环蛋白信号传导和基因转录的活性(wang et al.,2009b)。
[0384]
hla
‑
dmb基因中的变体被证明可能与hiv相关的卡波济氏肉瘤风险关联。此外,在erg阳性和etv1阳性的前列腺癌中发现到了hla
‑
dmb基因的失调节(paulo et al.,2012;aissani et al.,2014)。此外,肿瘤上皮的hla
‑
dmb表达水平升高与晚期浆液性卵巢癌的生存改善相关(callahan et al.,2008)。
[0385]
hltf是具有解旋酶和e3泛素连接酶活性的转录调节因子swi/snf家族中的一员,被发现透过结肠、胃、子宫、膀胱和肺肿瘤的超甲基化而失活(castro et al.,2010;debauve et al.,2008;garcia
‑
baquero et al.,2014)。
[0386]
hmmr表达在不同癌症实体(乳腺癌、结肠癌、胃癌、胰腺癌和前列腺癌)中上调,并
与细胞运动性、浸润和转移有关(yamada et al.,1999;wang et al.,1998;abetamann et al.,1996;gust et al.,2009;ishigami et al.,2011;sankaran et al.,2012)。hmmr与brca1相互作用,透过促进基因组不稳定性导致肿瘤进展。此外,hmmr与src相关,这提升细胞运动性并且hmmr
‑
cd44合作刺激erk信号传导,导致促进肿瘤发生。此外,hmmr是几个肿瘤相关蛋白(包括e2f1、p53和ras)的一个靶标(blanco et al.,2015;hall et al.,1995;hall and turley,1995;maxwell et al.,2008;sohr and engeland,2008;meier et al.,2014)。
[0387]
hspa14被证明在肝细胞癌中上调(yang et al.,2015c)。hspa14与非小细胞肺癌相关(wu et al.,2011a)。
[0388]
hspa8被证明在食管鳞状细胞癌中过量表达,hspa8在食管癌细胞中的高表达在体外抵消这些细胞的氧化应激诱导的细胞凋亡。此外,hspa8在多发性骨髓瘤和结肠癌中过量表达,hspa8的bcr
‑
abl1
‑
诱导表达促进慢性髓性白血病细胞的存活(chatterjee et al.,2013;dadkhah et al.,2013;jose
‑
eneriz et al.,2008;kubota et al.,2010;wang et al.,2013b)。
[0389]
huwe1过度表达发现于不同类型肿瘤,如肺癌、乳腺癌、前列腺癌、胶质母细胞瘤和结肠癌。另一报告显示,huwe1牵涉到肝细胞癌的发病机理(yoon et al.,2005;adhikary et al.,2005;liu et al.,2012)。此外,huwe1的枯竭防止了子集人类肿瘤细胞的增殖,而huwe1水平升高与可检测的p53相关(adhikary et al.,2005;confalonieri et al.,2009)。
[0390]
ido1被发现表达于多种肿瘤,例如,结直肠癌、黑色素瘤、浆液性卵巢癌和甲状腺乳头状微癌(brandacher et al.,2006;takao et al.,2007;brody et al.,2009;ryu et al.,2014)。ido1在子宫内膜癌组织以及在儿童急性髓细胞白血病中过度表达与疾病进展和患者存活受影响正相关(ino et al.,2008;folgiero et al.,2014)。
[0391]
ifi16蛋白在某些人前列腺和乳腺癌细胞系中相对较低或检测不到(xin et al.,2003;alimirah et al.,2007)。研究人员发现,ifi16在人类乳头瘤病毒阳性头颈部鳞状细胞癌中表达,并且与较好的预后相关(azzimonti et al.,2004)。此外,使用5
‑
aza
‑
dc治疗乳腺癌细胞系导致ifi16表达上调(fujiuchi et al.,2004)。
[0392]
ifi30表达被证明与细胞启动减少相关,包括磷酸化erk1/2下降、细胞增殖和癌症患者生存期下降(rausch and hastings,2015)。ifi30被证明在原发性和转移性乳腺癌中下调(xiang et al.,2014)。乳腺癌中ifi30表达下降被证明与较差的无病生存相关,而缺少ifi30与乳腺癌不良特征(例如肿瘤大小和淋巴结状态)正相关(xiang et al.,2014)。因此,ifi30可作为乳腺癌的一个潜在肿瘤抑制因子和新的独立预后因素(xiang et al.,2014)。弥漫性大b细胞淋巴瘤中ifi30表达下降被证明与总体生存较差有关(phipps
‑
yonas et al.,2013)。ifi30的单核苷酸多态性被证明是接受雄激素剥夺疗法治疗晚期前列腺癌患者的疾病进展的一个有意义的预测因子(bao et al.,2011)。ifi30被证明是皮肤鳞状细胞癌和基底细胞癌中上调的几种基因之一(wenzel et al.,2008)。ifi30被证明与黑色素瘤和旁邻抗原提呈细胞显示的抗原性表位特征差异相关,并且因此可以在免疫防御下促进肿瘤细胞生存(haque et al.,2002)。
[0393]
ifi44l被证明与cdkn2a相关,该基因与皮肤黑色素瘤和非黑色素瘤皮肤癌以及
mir
‑
9相关,而mir
‑
9与鼻咽癌相关(gao et al.,2013;puig
‑
butille et al.,2014)。
[0394]
ifit1基因在mcf7乳腺癌细胞中下调。其他报告显示,ifit1基因在喉咽癌中失活(xu et al.,2013a;motaghed et al.,2014)。此外,mir
‑
9可以调节人类癌细胞中的ifit1基因表达(gao et al.,2013)。
[0395]
ift172与胃癌的化学抗性有关(huang et al.,2014a)。
[0396]
与相邻非癌组织相比,ighg1在人类胰腺癌组织中过度表达。与此相反,ighg1蛋白在浸润性导管癌组织中下调(kabbage et al.,2008;li et al.,2011b)。ighg1的sirna靶向沉寂能够抑制细胞活力,促进细胞凋亡(pan et al.,2013)。
[0397]
研究人员发现,ighg3在受乳腺癌影响的沙特女性中表达。同样,在前列腺癌非洲裔男性中检测到拷贝数提高以及ighg3水平升高。另一份报告显示,ighg3表达发现于鳞状非小细胞肺癌、恶性间皮瘤以及肿瘤细胞,这些细胞偶尔见于malt淋巴瘤并显示有分化成浆细胞的倾向(remmelink et al.,2005;bin amer et al.,2008;ledet et al.,2013;zhang et al.,2013c;sugimoto et al.,2014)。
[0398]
最近的工作检测到,在原发性睾丸弥漫性大b细胞淋巴瘤中涉及ighg4的重排(twa et al.,2015)。
[0399]
研究发现ighm在受横纹肌肉瘤影响的中国患者中下调。其他人检测到弥漫性大b细胞淋巴瘤中的ighm表达。另一组发现,在弥漫性大b细胞淋巴瘤中,ighm基因只在大多数igm+肿瘤中有效igh等位基因上保存。此外,上皮错构瘤样本对结合到ighm增强因子3的转录因子或转录因子eb显示任何反应性(kato et al.,2009;blenk et al.,2007;ruminy et al.,2011;liu et al.,2014a)。
[0400]
impdh2过度表达发现于骨肉瘤和人前列腺癌组织以及在白血病细胞中(nagai et al.,1991;zhou et al.,2014b)。impdh2的抑制剂,如噻唑呋林和苯甲酰胺核苷,在急性骨髓性白血病和原始细胞危象中慢性骨髓性白血病患者中显示良好的临床应答(wright et al.,1996;jayaram et al.,1999)。
[0401]
inadl在对顺铂
‑
吉西他滨联合化疗有反应的非小细胞肺癌中下调(ma et al.,2015)。
[0402]
在乳腺癌、非小细胞肺癌、肝细胞癌和喉鳞状细胞癌中观察到inppl1过度表达(prasad et al.,2008b;zhou et al.,2011;fu et al.,2013b;fu et al.,2013c)。据报告,乳腺癌细胞中的inppl1沉寂降低体外细胞增殖和体内癌症生长,并抑制肿瘤转移(prasad et al.,2008a)。
[0403]
与非癌组织相比,ipp的表达在人乳腺肿瘤样本中升高(govindaraj et al.,2012)。
[0404]
一些证据表明,iqgap1在各种肿瘤类型(包括结直肠癌、胃癌、肝细胞癌、胰腺癌、卵巢癌和食管鳞状细胞癌)中过度表达(takemoto et al.,2001;dong et al.,2006;hayashi et al.,2010;white et al.,2010;wang et al.,2013i;wang et al.,2014i)。此外,高水平的iqgap1与卵巢癌和结直肠癌的较差预后相关(dong et al.,2006;hayashi et al.,2010)。
[0405]
最近的一项研究表明,irak2 rs35060588与结直肠癌的生存存在基因关联。另一方面,在慢性淋巴细胞白血病患者的irak2中未发现突变(martinez
‑
trillos et al.,
2014;wang et al.,2014c)。研究人员观察到,irak2的过度表达与非腺癌患者的无病生存下降有关(seol et al.,2014)。
[0406]
il6在mrna和蛋白水平上调前列腺癌细胞系中的irf9(erb et al.,2013)。另一项研究表明,该上调的irf9在耐药性乳腺癌细胞总对抗微管制剂紫杉醇产生抗性(luker et al.,2001)。
[0407]
许多研究报告了isg15在多种肿瘤中过度表达,如膀胱癌、乳腺癌、口腔鳞状细胞癌、宫颈癌和前列腺癌(andersen et al.,2006;chi et al.,2009;kiessling et al.,2009;rajkumar et al.,2011;wood et al.,2012;vincent
‑
chong et al.,2012)。在乳腺癌中,isg15高表达与预后不良有关(wood et al.,2012)。
[0408]
isyna1与皮肤恶性黑色素瘤的化疗应答相关(azimi et al.,2014)。isyna1被证明在各种条件下在人肝癌细胞系hepg2中上调(guan et al.,2003)。isyna1抑制与sk
‑
n
‑
sh神经母细胞瘤细胞系的增殖下降有关(ye and greenberg,2015)。
[0409]
itgb2基因多态性与结直肠瘤和散发浸润性导管乳腺癌有关。此外,在晚期卵巢癌患者外周血嗜中性粒细胞以及白血病中观察到itgb2过度表达。与此相反,在早幼粒细胞白血病中itgb2不存在或仅依稀表达(phongpradist et al.,2010;fu et al.,2011;zhou et al.,2012b;chang et al.,2013;bednarska et al.,2016)。cibr耦合plga纳米粒靶向itgb2有望成为白血病治疗的一种选择性药物递送系统(chittasupho et al.,2010)。
[0410]
itgb4与前列腺癌、胃癌、乳腺癌、口腔鳞状细胞癌和卵巢癌相关,并且被证明在胰腺导管腺癌中上调(chen et al.,2014e;xin et al.,2014;zubor et al.,2015;masugi et al.,2015;gao et al.,2015;kawakami et al.,2015)。itgb4(也称为cd104)往往与α6亚基关联,并可能在几种浸润性癌症的生物学中发挥重要作用,如食管鳞状细胞癌、膀胱癌和卵巢癌(kwon et al.,2013;pereira et al.,2014;chen et al.,2014e)。itgb4单核苷酸多态性似乎影响肿瘤的侵袭性和存活,并可能对乳腺癌患者具有预后价值(brendle et al.,2008)。
[0411]
itgb8过度表达见于几种癌症,包括肝细胞癌、头颈部癌、一些卵巢癌和黑色素瘤细胞系以及原发性非小细胞肺癌样本和来自几种上皮癌的脑转移癌中(liu et al.,2002b;goodman et al.,2012;vogetseder et al.,2013)。此外,itgb8的沉寂造成snail和转录启动,以及肺癌细胞系中mek和akt磷酸化水平变化(xu and wu,2012)。pc
‑
3和22rv1前列腺癌细胞体外itgb8敲减导致细胞迁移和侵袭显著下降(mertens
‑
walker et al.,2015)。研究人员发现,itgb8过度表达可能是肝癌吉非替尼耐药的诱导因子。itgb8可能与tgf
‑
β途径相互作用以实现其抗吉非替尼作用(wang et al.,2015f)。
[0412]
据报告,itpr1的表达在他莫昔芬耐药性乳腺癌细胞系中被改变(elias et al.,2015)。研究人员推测,hif2α/itpr1轴在调节透明细胞肾细胞癌的细胞存活中具有作用。此外,itpr1与乳腺癌的总体生存显著相关(messai et al.,2014;gu et al.,2016)。
[0413]
itpr2基因的单核苷酸多态性与中国人群中肾细胞癌的风险相关。同样地,itpr2基因连锁不平衡中的两种常见变体rs718314和rs1049380被确定为肾细胞癌的新易感性基因座。此外,与健康人相比,在正常急性骨髓性白血病患者中观察到itpr2的过度表达(wu et al.,2012d;shi et al.,2015;zhang et al.,2016a)。在正常的急性骨髓性白血病中,itpr2表达水平升高与较短的总体生存期和无事件生存期相关(shi et al.,2015)。
[0414]
研究发现结直肠癌和肺癌腺癌中有jup表达,而在口腔鳞状细胞癌中发现高itgb4/jup比率(wang and zheng,2014;yang et al.,2012a;schuetz et al.,2012;sheng and zhu,2014;nagata et al.,2013)。
[0415]
kars过度表达发现于胃癌以及肿瘤相关炎症细胞中。此外,在结直肠患者中发现kars基因突变。其他人观察到,乳腺癌中染色体16q的整组缺失与kars表达降低相关(yen et al.,2009;hungermann et al.,2011;kim et al.,2014a)。据报告,在kars介导的转移期间kars参与细胞
‑
细胞和细胞
‑
ecm的黏附(nam et al.,2015)。
[0416]
kcnk15基因超甲基化发现于几个细胞系,包括结肠癌、白血病和膀胱癌(shu et al.,2006)。
[0417]
kdelr1在肿瘤发生中发挥作用(yi et al.,2009)。在肝癌细胞中发现kdelr1水平降低(hou et al.,2015)。kdelr1下调见于急性髓细胞白血病(caldarelli et al.,2013)。
[0418]
kdm1a过度表达促进肿瘤细胞增殖、迁移和侵袭,并与nsclc和hcc较差的预后相关(lv et al.,2012;zhao et al.,2013d)。kdm1a表达升高与前列腺癌复发和vegf
‑
a表达增加相关(kashyap et al.,2013)。用曲古霉素a(tsa)和5
‑
氮杂
‑
2'
‑
脱氧胞苷(地西他滨)的组合物抑制kdm1a可压制卵巢癌腹水细胞系skov3的成瘤(meng et al.,2013)。
[0419]
kdm1b显示由于其e3泛素连接酶活性可抑制肺癌细胞系a549的细胞生长(yang et al.,2015b)。kdm1b被证明参与推定肿瘤抑制组织因子途径抑制剂2的调节(mino et al.,2014)。kdm1b被证明在乳腺癌中上调,在高级别尿路上皮癌中扩增和上调(heidenblad et al.,2008;katz et al.,2014)。kdm1b被证明在乳腺癌的dna甲基化和基因沉寂中发挥作用。kdm1b和dna甲基转移酶的抑制被描述为乳腺癌后生治疗的新方法(katz et al.,2014)。kdm1b被证明与癌症干细胞性质的获得有关,包括透明质酸cd44v3活化头颈癌的自我更新、克隆形成和化疗耐药(bourguignon et al.,2012)。
[0420]
在临床前列腺癌中观察到kiaa0196过度表达,也在30
‑
40%的异种移植体和激素难治性肿瘤中扩增(porkka et al.,2004)。kiaa0196基因扩增与高级别雌激素受体阴性乳腺癌的较差预后相关(chin et al.,2007)。在前列腺癌中,kiaa0196在体外生长、锚定非依赖性生长或侵袭中似乎没有任何显著作用(jalava et al.,2009)。
[0421]
kiaa1324在不同癌症类型中过度表达,包括乳腺癌、肺癌、胰腺癌和卵巢癌(schlumbrecht et al.,2011;estrella et al.,2014;bauer et al.,2004)。kiaa1324在胃癌中显示了肿瘤抑制行为,其中kiaa1324被下调,这与不良预后相关(kang et al.,2015b)。
[0422]
kif11的抑制显示可停止治疗更耐药的胶质母细胞瘤肿瘤起始细胞(tic)和非tic的生长,并阻止tic人群的肿瘤发生和自我更新(venere et al.,2015)。靶向作用于kif11还显示可减少胶质瘤细胞侵袭并延长负荷原位胶质母细胞瘤小鼠的生存期(venere et al.,2015)。因此,kif11对胶质母细胞瘤的侵袭、增殖和自我更新发挥着驱动器的作用(venere et al.,2015)。有丝分裂相关基因(如kif11)的高表达被证明与肝细胞癌对经动脉化学栓塞治疗完全反应相关(gaba et al.,2015)。kif11的功能干扰被描述可导致实验肿瘤模型中肿瘤血管生成的有效抑制(exertier et al.,2013)。kif11被证明在多发性骨髓瘤和急性骨髓性白血病患者的骨髓样本中下调(cohen et al.,2014)。kif11核表达被描述为转移性去势抵抗性侵袭性前列腺癌中多西他赛反应的潜在预测生物标志物,以及前列
腺癌侵袭性的预后生物标志物(wissing et al.,2014)。kif11被证明是非小细胞肺癌及头颈部鳞状细胞癌的肿瘤细胞生存所必需的,因此可能是潜在的抗癌靶标(martens
‑
de kemp et al.,2013)。kif11上调被证明与儿童室管膜瘤复发有关(peyre et al.,2010)。
[0423]
在乳腺癌中,kif15被显示过量表达并具有免疫原性,这是因为抗kif15抗体可从乳腺癌患者中分离获得(scanlan et al.,2001)。此外,似乎肺腺癌中含有kif15(bidkhori et al.,2013)。
[0424]
在甲状腺癌、乳腺癌、头颈部鳞状细胞癌中kif1a已知频繁甲基化并显示较高水平(aviles et al.,1991;hoque et al.,2008;demokan et al.,2010;guerrero
‑
preston et al.,2014)。此外,与对照组相比,kif1a被发现于肺癌和头颈部鳞状细胞癌患者中血浆和唾液。这些结果表明,它可用作这些疾病早期检测的生物标记物(ostrow et al.,2010)。在乳腺癌中,kif1a过度表达被发现与细胞系中化疗耐药相关(de et al.,2009)。
[0425]
在胰腺导管腺癌、黑色素瘤、膀胱癌、非小细胞肺癌和胆管细胞癌中检测到kif20a过度表达(imai et al.,2011;yamashita et al.,2012;stangel et al.,2015)。最近,据报告,用kif20a衍生肽接种的胰腺导管腺癌患者与对照组相比,表现出更好的预后(asahara et al.,2013)。此外,kif20a的沉寂导致抑制胰腺癌细胞系的增殖、运动性和侵袭性(stangel et al.,2015)。
[0426]
在肺癌、腺癌和非小细胞肺癌患者中观察到kif5b基因和ret原癌基因(ret)的融合(kohno et al.,2012;cai et al.,2013b;qian et al.,2014)。根据白细胞介素
‑
3(il
‑
3)非依赖性生长确定ba/f3细胞中的kif5b
‑
ret表达导致致癌性转化(lipson et al.,2012)。
[0427]
kifc1透过焦距偏离中心微管组织中心至两个纺锤体极进行减数分裂细胞的细胞分裂而发挥至关重要的作用。在癌细胞中,kifc1被证明是独立来自大量形成的中心体(正常或多余中心体)的纺锤体正确组装、稳定极聚焦和癌细胞生存所需要的。癌症中dna损伤应答的组成性活化部分显示可介导偏离中心纺锤体的形成。来自kifc1的偏离中心纺锤体形成的依赖性使得kifc1称为癌症治疗有吸引力的靶标。一些潜在的kifc1抑制剂目前正在研究中(li et al.,2015e;kleylein
‑
sohn et al.,2012;wu et al.,2013a;watts et al.,2013;zhang et al.,2016b)。此外,kifc1显示了中心体聚类依赖性促增殖作用,这基于保护生存素免于蛋白酶体介导的降解(pannu et al.,2015)。kifc1表达被证明在乳腺癌,特别是雌激素受体阴性、孕激素受体阴性和三阴性乳腺癌和8个人乳腺癌细胞系中上调。在雌激素受体阳性乳腺癌细胞中,kifc1是由雌激素强烈诱导表达的其他19种动力蛋白之一。在乳腺癌中,kifc1过度表达及其核积累被证明与组织学分级有关,并可预测较差的无进展和总生存期。在乳腺癌细胞系中,kifc1过度表达被证明可介导对多西他赛的抗性。kifc1沉寂负面影响乳腺癌细胞生存力(zou et al.,2014a;pannu et al.,2015;de et al.,2009;li et al.,2015e)。kifc1被证明在卵巢癌中过度表达,其与肿瘤侵袭性、晚期肿瘤分级和分期相关。因此,kifc1可作为预测更差预后、较差总体存活差和转移性传播发病的潜在生物标志物(pawar et al.,2014)。kifc1被确定为三种基因之一,其在原发性nsclc肿瘤中高表达提示发生脑转移的风险较高(grinberg
‑
rashi et al.,2009)。
[0428]
klhl14与原发性中枢神经系统淋巴瘤相关(vater et al.,2015)。
[0429]
klhl15被证明可作为e3泛素连接酶连接蛋白与磷酸酶2a蛋白相互作用,后者是一
al.,2008)。
[0439]
几个研究观察到在不同类型癌症中ldlr高水平表达或无效地调节,例如,在肺腺癌细胞系、前列腺癌细胞以及人结肠癌活检物中报告有ldlr过度表达。与此相反,在急性骨髓性患者白血病细胞中报告有ldlr回馈调节降低(gueddari et al.,1993;tatidis et al.,1997;lum et al.,1999;chen and hughes
‑
fulford,2001)。
[0440]
研究发现,在结直肠癌组织以及肺癌中mrna以及lgals3bp蛋白水平上调(ozaki et al.,2004;iacovazzi et al.,2010;wu et al.,2008)。lgals3bp水平升高与弥漫性大b细胞淋巴瘤预后差呈正相关(kim et al.,2008d)。此外,在肺癌中,lgals3bp透过增加癌细胞的黏附力参与癌转移(ozaki et al.,2004)。
[0441]
lgr6与三阴性乳腺癌、胃癌和结肠癌有关(gong et al.,2012;rocken,2013;purrington et al.,2014)。lgr6被证明在胃癌中上调(steffen et al.,2012)。lgr6与胃癌的局部肿瘤生长和患者生存期相关(steffen et al.,2012)。
[0442]
llgl1表达在乳腺癌、肺癌、前列腺癌、卵巢癌、结直肠癌、黑色素瘤、子宫内膜癌和肝细胞癌中下降或缺失(schimanski et al.,2005;kuphal et al.,2006;tsuruga et al.,2007;lu et al.,2009;song et al.,2013b)。似乎llgl1透过线粒体相关途径抑制细胞增殖并促进食管癌细胞系的细胞凋亡。此外,llgl1转录下降与淋巴结转移相关,而llgl1过度表达导致细胞黏附增加和细胞迁移降低(schimanski et al.,2005;kuphal et al.,2006;tsuruga et al.,2007;song et al.,2013b)。
[0443]
在结肠癌和胃癌中lmnb1表达下降,而在前列腺癌、肝细胞癌和胰腺癌中过度表达(moss et al.,1999;lim et al.,2002;coradeghini et al.,2006;li et al.,2013a)。在肝细胞癌中,lmnb1的表达水平与肿瘤分期、肿瘤大小和结节数量呈正相关。这些结果表明,lmnb1可以用于检测早期肝细胞癌(sun et al.,2010)。
[0444]
癌/睾丸抗原家族45被证明在癌细胞系和肺癌标本中频繁表达(chen et al.,2005)。ct45基因被证明是上皮卵巢癌的潜在预后生物标志物和治疗靶标(zhang et al.,2015l)。
[0445]
lpcat2与前列腺癌有关(williams et al.,2014)。lpcat2被证明在乳腺癌、宫颈癌和结直肠癌中下调(agarwal and garg,2010)。lpcat2表达与前列腺癌的患者结果有关(williams et al.,2014)。
[0446]
透过rna干扰或透过显性负突变体抑制lrba表达导致癌细胞的生长抑制。这些发现提示,lrba表达失调促进癌细胞生长特性的改变(wang et al.,2004)。
[0447]
ltbp2被证明在肝细胞癌、胰腺导管腺癌中上调,而在食管鳞状细胞癌细胞系和肿瘤组织中,ltbp2表达下调(chan et al.,2011;turtoi et al.,2011;cho et al.,2016)。在肝细胞癌中,高水平ltbp2与较短的至肿瘤复发时间显著有关。同样地,ltbp2水平升高与er(
‑
)/pr(
‑
)乳腺癌患者的较差结果有关(naba et al.,2014;cho et al.,2016)。
[0448]
ltn1,也称为znf294,编码listerin e3泛素蛋白连接酶1,位于21q22.11染色体上(refseq,2002)。ltn1与结直肠癌的高级别微卫星不稳定性有关(reuschenbach et al.,2010)。
[0449]
lurap1被证明是一种nf
‑
κb活化剂,它可能是调节树突细胞功能以抵抗肿瘤相关因子介导功能异常的候选基因(jing et al.,2010)。
[0450]
据报告,lyst基因位于多发性骨髓瘤的拷贝数变异区域内(ivyna bong et al.,2014)。
[0451]
研究人员报告,m6pr在结肠癌细胞系以及绒毛膜细胞中有表达(braulke et al.,1992;o'gorman et al.,2002)。在乳腺癌中,m6pr低水平表达与患者预后较差相关(esseghir et al.,2006)。此外,m6pr过度表达导致在体外细胞生长率降低以及裸鼠中肿瘤生长下降(o'gorman et al.,2002)。
[0452]
macf1与结直肠癌、肾细胞癌和肺腺癌相关(bidkhori et al.,2013;arai et al.,2014;kim et al.,2015b)。macf1被证明与clb
‑
bar细胞系的神经细胞瘤有关(schleiermacher et al.,2005)。
[0453]
在许多类型人类肿瘤,包括非小细胞肺癌、肺腺癌、鳞状细胞癌、甲状腺癌、乳腺癌和卵巢癌中发现madd过度表达(subramanian et al.,2009;li et al.,2011a;wei et al.,2012;bi et al.,2013;turner et al.,2013)。研究人员证明,在a549细胞中madd水平升高抑制细胞凋亡和增加生存率,而madd的敲减促进细胞凋亡和减少细胞增殖(wei et al.,2012;bi et al.,2013)。此外,madd的功能由pten
‑
pi3k
‑
akt信号途径调节(jayarama et al.,2014)。
[0454]
magea4被描述为一种癌症睾丸抗原,发现于在一小部分典型精原细胞瘤中表达,但不在非精睾丸生殖细胞肿瘤中表达,在乳腺癌、霍奇金淋巴瘤eb病毒阴性病例、食管癌、肺癌、膀胱癌、头颈部癌、结直肠癌、口腔鳞状细胞癌和肝细胞癌中表达(ries et al.,2005;bode et al.,2014;li et al.,2005;ottaviani et al.,2006;hennard et al.,2006;chen et al.,2003)。magea4被证明在头颈部原发性黏膜黑色素瘤中频繁表达,因此可能是基于癌症睾丸抗原的免疫治疗的潜在靶标(prasad et al.,2004)。magea4被证明在来自lhk2肺腺癌细胞的癌干细胞样细胞、sw480结肠腺癌细胞和mcf7乳腺癌细胞中优先表达(yamada et al.,2013)。magea4在自发转化的正常口腔角质中过度表达表明可透过阻止细胞周期阻滞和透过抑制p53转录靶标bax和cdkn1a介导的细胞凋亡而促进生长(bhan et al.,2012)。magea4被证明在丙型肝炎病毒感染的肝硬化和晚期肝细胞癌患者中比早期肝细胞癌患者中更频繁表达,从而使magea4转录物的检测潜在有助于预测预后(hussein et al.,2012)。magea4被证明是几种癌症/睾丸抗原之一,其在肺癌中表达并可作为肺癌患者的多价免疫治疗的潜在候选抗原(kim et al.,2012b)。magea4被描述为在食管癌和肝细胞癌中上调(zhao et al.,2002;wu et al.,2011c)。称为p286
‑
1y2l9l的magea4衍生原生肽类似物被描述为适用于开发针对食管癌肽疫苗的一个新候选表位(wu et al.,2011c)。mage基因家族的几个成员,包括magea4,均显示在黑色素瘤中经常发生突变(caballero et al.,2010)。
[0455]
在各种肿瘤中,如肝细胞癌、结直肠癌和卵巢癌中检测到magea8表达(hasegawa et al.,1998;tahara et al.,1999;eng et al.,2015)。此外,magea8过度表达与高cd3肿瘤患者的较差无进展生存期相关(eng et al.,2015)。
[0456]
magec3被描述为仅在睾丸以及不同组织学来源的肿瘤中表达。因此,magec3可能是癌症免疫疗法的一个靶标(lucas et al.,2000)。
[0457]
夫拉平度诱导人肿瘤细胞增殖的抑制以及在不同人类肿瘤细胞系中magef1的下调(lu et al.,2004)。magef1在结直肠癌组织中显著过度表达(chung et al.,2010)。
[0458]
magt1被证明与淋巴瘤的易感性有关(chaigne
‑
delalande et al.,2013)。
[0459]
manba基因的多态性与瑞典人群的结直肠癌风险有关,但在中国人群中并非如此。其他人观察到manba在食管癌中水平上升(sud et al.,2004;gao et al.,2008)。
[0460]
mcm10被证明在食管鳞状细胞癌和宫颈癌中上调(das et al.,2013a;lu et al.,2014b)。mcm10表达与胶质瘤肿瘤和宫颈癌的肿瘤分级相关(das et al.,2013a;hua et al.,2014)。mcm10与早期胃癌、乳腺癌和肺癌有关(wu et al.,2012a;kang et al.,2013)。mcm10可用作食管鳞状细胞癌的一种生物标志物(lu et al.,2014b)。
[0461]
mcm2被证明是早期乳腺癌、肾细胞癌、食管癌、喉鳞状细胞癌和脑少突胶质细胞瘤增殖和预后的最敏感指标(wharton et al.,2001;going et al.,2002;rodins et al.,2002;gonzalez et al.,2003;cai et al.,2012;joshi et al.,2015)。
[0462]
研究人员在副神经节瘤中观察到mdh2表达水平较低。另一方面,其他报告显示,mdh2在胃癌以及前列腺癌细胞系和患者标本中过度表达(liu et al.,2013b;yao et al.,2015;cascon et al.,2015)。在胃癌中,mdh2水平升高与浸润深度、淋巴结转移、远处转移和tnm分期有关(yao et al.,2015)。mdh2被证明参与了阿霉素耐药性子宫癌的发展,而其他报告显示,mdh2经由jnk途径诱导前列腺癌对多西紫杉醇化学疗法的抗性(liu et al.,2013b;lo et al.,2015)。
[0463]
memo1与颊黏膜鳞状细胞癌相关(shah et al.,2013)。memo1与乳腺癌的迁移、侵袭和肺转移有关(macdonald et al.,2014)。memo1被证明在胰腺癌细胞系paca中上调(kalinina et al.,2010)。memo1是原发性乳腺癌早期远处转移的预后因素(macdonald et al.,2014)。
[0464]
在各种肿瘤中,包括乳腺癌、恶性黑色素瘤、膀胱肿瘤、卵巢癌和鳞状细胞癌中发现mfge8过度表达(jinushi et al.,2008;sugano et al.,2011;carrascosa et al.,2012;tibaldi et al.,2013;yamazaki et al.,2014)。似乎mfge8能够经由akt依赖性和twist依赖性途径来增强致瘤性和转移能力(jinushi et al.,2008)。
[0465]
mga被证明在肺腺癌中突变(2014)。mga被证明在非小细胞肺癌、小细胞肺癌和慢性淋巴细胞白血病中失活(de et al.,2013;romero et al.,2014)。
[0466]
mgrn1与骨肉瘤相关(man et al.,2004)。
[0467]
mki67ip被证明由c
‑
myc反式启动,mki67ip的沉寂导致细胞增殖抑制。因此,mki67ip可能在癌症中发挥作用(pan et al.,2015)。
[0468]
一项研究表明,mkks与同步腺瘤肿瘤中上调(kim et al.,2008a)。
[0469]
mlf1甲基化和过度表达与肺鳞状细胞癌、髓性白血病和胃癌相关。基因组谱研究确定了人食管癌中的mlf1基因(shi et al.,2012;matsumoto et al.,2000;sun et al.,2004b;chen et al.,2008)。在胃癌中,mlf1基因的甲基化与淋巴结转移数量呈正相关。但是,这对胃癌患者无任何预后价值(shi et al.,2012)。据报告,mlf1促进前列腺癌细胞增殖、集落形成并显著抑制细胞凋亡(zhang et al.,2015h)。
[0470]
mmp7常在人癌组织(包括结直肠癌、转移性肺癌和胃癌)中过度表达,与癌症进展和转移形成相关(ii et al.,2006;sun et al.,2015b;han et al.,2015a;long et al.,2014)。mmp7已显示发挥重要的肿瘤促进作用,如:细胞外基质蛋白降解、透过增加胰岛素样生长因子和肝素结合表皮生长因子活化肿瘤细胞增殖、透过裂解膜结合fas配体诱导肿瘤
相邻细胞的细胞凋亡(ii et al.,2006)。
[0471]
与正常组织相比,mrpl11被证明在鳞状细胞癌中差异表达(sugimoto et al.,2009)。mrpl11表达与无进展生存期和宫颈癌的转移性表型相关(lyng et al.,2006)。
[0472]
数项研究报告了msh2基因甲基化与多种恶性肿瘤(如,肝癌、急性淋巴细胞白血病、肾透明细胞癌和食管鳞状细胞癌)之间的联系。相反,msh2在散发性结直肠癌中启动子超甲基化是一种罕见事件(vlaykova et al.,2011;ling et al.,2012;hinrichsen et al.,2014;wang et al.,2014a;yoo et al.,2014)。最近的工作证明,顺铂可透过下调mir
‑
21来上调msh2的表达,以抑制a549细胞增殖(zhang et al.,2013e)。
[0473]
在间皮瘤中显示,msln透过增加mmp
‑
9的分泌诱导肿瘤细胞侵袭(servais et al.,2012)。若干出版物表明msln在不同类型癌症,例如间皮瘤、三阴性乳腺癌、胰腺癌、卵巢癌和肺腺癌中过度表达(chang and pastan,1996;argani et al.,2001;ho et al.,2007;tozbikian et al.,2014)。
[0474]
在许多肿瘤中,如乳腺癌、白血病、胶质母细胞瘤、非小细胞肺癌和膀胱癌中观察到mtap活性损失。此外,启动子超甲基化被认为是mtap缺陷肝细胞癌的优势灭活机制(nobori et al.,1991;smaaland et al.,1987;kamatani and carson,1980;stadler et al.,1994;nobori et al.,1993;hellerbrand et al.,2006)。mtap在mtap缺陷黏液纤维肉瘤细胞系中再表达抑制细胞迁移、侵袭、增殖、锚定非依赖性集落形成,并下调细胞周期素d1(li et al.,2014a)。
[0475]
mtbp被证明在肝细胞癌中下调(bi et al.,2015)。mtbp被证明在乳腺癌和淋巴瘤中上调(grieb et al.,2014;odvody et al.,2010)。mtbp被证明与肝细胞癌的囊/血管浸润和淋巴结转移呈负相关(bi et al.,2015)。mtbp与乳腺癌和头颈部鳞状细胞癌患者的生存相关(iwakuma and agarwal,2012;grieb et al.,2014)。mtbp可能是骨肉瘤癌症进展的一种潜在生物标志物(agarwal et al.,2013)。
[0476]
mtch1与continb和contind结肠癌细胞系中5
‑
氟尿嘧啶耐药相关(de angelis et al.,2006)。
[0477]
mthfd2被证明在伯基特淋巴瘤、弥散性大细胞淋巴瘤、乳腺癌和sapc
‑
3前列腺癌细胞系中上调(liu et al.,2014b;patrikainen et al.,2007;tedeschi et al.,2015)。mthfd2表达与乳腺癌的肿瘤大小、组织学分级、淋巴结转移及远处转移相关(liu et al.,2014b)。mthfd2与乳腺癌的生存期差以及膀胱癌的更高癌症易感性和生存期有关(nilsson et al.,2014;andrew et al.,2009)。mthfd2是乳腺癌的预后因素(liu et al.,2014b)。
[0478]
mtor信号的过度表达与各种类型癌症的临床预后差有关,如,肾癌、肺癌、乳腺癌、喉鳞状细胞癌、神经内分泌肿瘤、胆道癌、结肠癌、宫颈癌、卵巢癌、食道癌、恶性黑色素瘤和头颈部鳞状细胞癌(faried et al.,2006;hou et al.,2007;liu et al.,2007;molinolo et al.,2007;karbowniczek et al.,2008;faried et al.,2008;shao et al.,2014)。研究人员发现,透过慢病毒介导的mtor特异性shrna的mtor基因敲减导致前列腺癌细胞的活力和生长显著下降(du et al.,2014b)。
[0479]
研究人员发现,mtr基因多态性与乳腺癌、多发性骨髓瘤和头颈部鳞状细胞癌有显著的联系(zhang et al.,2005b;kim et al.,2007;cui et al.,2012;lopez
‑
cortes et al.,2013;hosseini,2013;yang et al.,2014a)。
[0480]
mtx2与急性骨髓性白血病亚组的患者预后鉴别有关(vey et al.,2004)。
[0481]
muc1在几个肿瘤,如结直肠癌、乳腺癌、肺癌和食管腺癌中上调(khodarev et al.,2009;gronnier et al.,2014;kesari et al.,2015)。在胰腺癌中,muc1透过靶向作用于特定的信号通路(如,p42
‑
44 mapk、akt、bcl
‑
2和mmp13)影响细胞增殖、迁移和侵袭。其他人观察到muc1水平在b16和b16bl6鼠黑色素瘤细胞中上升介导akt磷酸化的上调(trehoux et al.,2015;wang et al.,2015h)。muc1过度表达过度表达可减少β
‑
内连环蛋白易位到细胞核,降低t细胞因子的活性,抑制细胞周期蛋白d1和c
‑
myc表达(wang et al.,2013e)。
[0482]
muc16最初被确认在卵巢癌被过度表达。它可在卵巢癌患者的血清中检测到,并且是这种癌症类型确定的生物标志物。此外,胰腺癌和乳腺癌中报告了muc16过度表达。muc16水平升高的癌症患者表现出肿瘤复发的较高可能性(haridas et al.,2014)。
[0483]
muc20被描述为在一些上皮性肿瘤中上调的预后分子标志物(wang et al.,2015b)。muc13表达与muc20表达一起被证明是对接受新辅助化疗后手术的食管鳞状细胞癌患者的潜在预后标志物(wang et al.,2015b)。muc20被证明在结直肠癌和子宫内膜癌中上调(chen et al.,2013a;xiao et al.,2013)。muc20表达被证明与结直肠癌的复发和较差预后有关。无病生存期和总生存期在muc20上调后显著恶化(xiao et al.,2013)。muc20被证明是较差生存期的预后因素,这也与子宫内膜癌的细胞生长、迁移和侵袭有关(chen et al.,2013a)。muc20可能在癌肉瘤的肿瘤发生中发挥作用(vekony et al.,2009)。
[0484]
muc5ac在多种癌症类型(包括结直肠癌、胃癌、肺癌和胰腺癌)中失调。结直肠癌和胃癌中表达损耗或低表达与更强的侵袭性行为和较差预后相关。在肺癌中过度表达导致复发和转移的可能性增加(yonezawa et al.,1999;kocer et al.,2002;kim et al.,2014b;yu et al.,1996)。muc5ac表达由不同的途径和转录因子(包括sp1、pkc/erk/ap
‑
1、pkc/jnk/ap
‑
1、creb、nf
‑
κb和il
‑
1β/egfr/akt/gk
‑
3β/β
‑
连环蛋白调控(kato et al.,2006;raja et al.,2012;chen et al.,2014h)。
[0485]
muc5b在不同癌症实体(包括结直肠癌、肺癌和乳腺癌中)过度表达,并与肿瘤进展相关(sonora et al.,2006;valque et al.,2012;walsh et al.,2013;nagashio et al.,2015)。muc5b可在甲基化的影响下被抑制,并且由atf
‑
1、c
‑
myc、nfκb、sp1、creb、ttf
‑
11和gcr调节(perrais et al.,2001;van,i et al.,2000)。
[0486]
mvp在几种中枢神经系统肿瘤中高表达(yang et al.,2012b)。mvp在癌症以及几种化疗耐药癌细胞系中高表达(szaflarski et al.,2011;mossink et al.,2003)。mvp表达水平随着年龄的增加而增加,并促进凋亡抗性(ryu and park,2009)。
[0487]
肝细胞癌细胞中显示出脂多糖刺激后的mx2等位基因表达。此外,mx2基因单核苷酸多态性与多发原发性黑色素瘤显著相关(park et al.,2014;gibbs et al.,2015)。
[0488]
mycbp被证明在结肠癌细胞和口腔癌细胞系hep
‑
2、ssc
‑
9和tu
‑
177中上调(rey et al.,1999;jung and kim,2005)。mycbp与少突胶质细胞肿瘤的化疗敏感性有关(shaw et al.,2011)。mycbp被证明在乳腺癌细胞系mcf
‑
7有限葡萄糖和氧气可用期间与癌细胞存活相关(sedoris et al.,2010)。mycbp被证明在慢性粒细胞白血病中差异表达(pizzatti et al.,2006)。
[0489]
myo1g被证明对于乳腺癌细胞系mcf7的细胞存活和溶酶体稳定性很重要(groth
‑
pedersen et al.,2012)。
[0490]
naf1被证明与grim
‑
1(前列腺中一种潜在的共同肿瘤抑制剂)相互作用(nallar et al.,2011)。
[0491]
nampt基因多态性与发生食管鳞状细胞癌以及膀胱癌的风险有关。此外,在结直肠癌、乳腺癌、前列腺癌、胃癌、甲状腺癌、卵巢癌和胰腺癌中报告了nampt水平升高(shackelford et al.,2010;dalamaga,2012;zhang et al.,2014c;zhang et al.,2015b;sawicka
‑
gutaj et al.,2015)。此外,nampt基因的单核苷酸多态性与总体膀胱癌患者和非肌肉侵袭性膀胱癌患者的无复发生存期相关(zhang et al.,2014c)。
[0492]
naprt1被证明是与癌症相关。它也显示,使naprt1表达下降的突变可预测烟酸在nampt抑制剂肿瘤治疗中的用处(duarte
‑
pereira et al.,2014)。naprt1表达被证明由于启动子超甲基化在很多癌症类型中丢失,使两种nad补救途径之一失活。同时给予阻断第二nad补救途径的nampt抑制剂导致合成致死。因此,naprt1提供了nampt抑制剂的一种新颖预测生物标志物(shames et al.,2013)。naprt1被描述在胶质母细胞瘤、神经母细胞瘤和肉瘤中高频率丧失,可能与肿瘤细胞凋亡有关(cerna et al.,2012)。narpt1被证明在霍奇金淋巴瘤中下调(olesen et al.,2011)。
[0493]
nat8l表达在大约40%的腺癌和鳞状细胞癌病例中升高。过度表达导致nsclc患者血液中的n
‑
乙酰转移酶水平升高,提呈一个用于肺癌早期检测的潜在生物标志物(lou et al.,2016)。
[0494]
nbeal2缺乏与小鼠中癌症转移预防相关(guerrero et al.,2014)。nbeal2是一组生物标志物的一部分,用于卵巢癌期别鉴别(kashuba et al.,2012)。
[0495]
ncapd2过度表达发现于卵巢癌发展及肿瘤进展过程中的扩增和突变(emmanuel et al.,2011)。
[0496]
ncapd3是亚型1前列腺癌和前列腺癌术后生化复发的潜在生物标志物(jung et al.,2014;lapointe et al.,2008)。
[0497]
ncapg在多发性骨髓瘤、急性骨髓性白血病患者中以及来自血液的白血病细胞或骨髓瘤细胞中下调(cohen et al.,2014)。ncapg可能是结直肠癌的多重抗药性基因(li et al.,2012)。ncapg在嫌色亚型人类细胞癌中高度上调,但在传统的人类肾细胞癌中不是这样(kim et al.,2010a)。ncapg上调与黑色素瘤进展相关(ryu et al.,2007)。ncapg与葡萄膜黑色素瘤相关相关(van ginkel et al.,1998)。ncapg在不同肿瘤细胞中表现出不同表达(jager et al.,2000)。
[0498]
nckap1l过度表达与慢性淋巴细胞白血病不良结果相关。另一方面,nckap1l在患者慢性淋巴细胞白血病细胞中下调导致其对氟达拉滨(fludarabine)介导的杀伤易感性显著增加(joshi et al.,2007)。
[0499]
3p24的非同义单核苷酸多态性nek10
‑
l513s被证明与患乳腺癌的风险有关(milne et al.,2014)。brca2携带者中slc4a7/nek10的单核苷酸多态性被证明与er阳性乳腺癌有关(mulligan et al.,2011)。nek10被描述为牵涉dna损伤应答(fry et al.,2012)。nek10被描述为g2/m细胞周期阻滞的调节因子,其与mapk/erk信号通路成员erk1/2、raf
‑
1和mek1有关(moniz and stambolic,2011)。
[0500]
nfatc2被证明在人类癌症(如乳腺癌和肺癌)中表达。此外,在尤因氏肉瘤中发现nfatc2的染色体易位以及与ewsr1癌基因框内融合。此外,nfatc2基因在胰腺癌中高度扩增
(holzmann et al.,2004;yiu and toker,2006;szuhai et al.,2009;liu et al.,2013a)。在乳腺癌中,nfatc2能够透过诱导cox
‑
2来诱导侵袭性。其他报告显示,nfatc2经由lcn2/tweakr/tweak轴增加乳腺癌细胞的侵袭性(yiu and toker,2006;gaudineau et al.,2012)。
[0501]
nfe2l3损失容易使小鼠发生淋巴瘤。其他人观察到在结肠癌细胞中有高水平的nfe2l3,而nfe2l3的异常表达发现于霍奇金淋巴瘤中。此外,nfe2l3在er阳性肿瘤中显示出超甲基化(kuppers et al.,2003;chevillard et al.,2011;palma et al.,2012;rauscher et al.,2015)。
[0502]
在含有神经内分泌元素的肺肿瘤中以及小细胞肺癌中发现了nhp2l1水平升高(jensen et al.,1994;harken et al.,1999)。
[0503]
nlrc3被证明在结直肠癌中下调,下调与癌症进展相关(liu et al.,2015d)。nlrc3被描述为炎症反应的潜在负调节因子,其与具有不同炎性成分(如,胱天蛋白酶1和5)相互作用(gultekin et al.,2015)。
[0504]
noa1过度表达被证明透过增加线粒体蛋白酪氨酸硝化和细胞色素c释放在人乳腺腺癌细胞系mcf
‑
7中诱导凋亡(parihar et al.,2008a)。noa1被证明可调节人类神经母细胞瘤细胞的细胞凋亡(parihar et al.,2008b)。
[0505]
nod2与结直肠癌、胃癌风险、malt淋巴瘤、乳腺癌、肺癌、喉癌和前列腺癌相关(kang et al.,2012;liu et al.,2014c;castano
‑
rodriguez et al.,2014;ahangari et al.,2014)。nod2与尿路上皮膀胱癌的淋巴结转移有关(guirado et al.,2012)。nod2基因多态性可能与睾丸、肝脏、胆囊、胆道、胰腺、小肠、肾和皮肤癌、非甲状腺内分泌肿瘤、淋巴瘤和白血病的风险改变有关(kutikhin,2011)。
[0506]
nploc4被证明与复合体介导的替代nf
‑
kb途径中的p97和ufd1相关,该途径牵涉癌症(zhang et al.,2015o)。
[0507]
nr4a2在几种癌症(如,膀胱癌、结直肠癌和胃癌)中高度表达。与此相反,相对于正常乳腺组织,在乳腺癌中观察到nr4a2表达下调(holla et al.,2006;inamoto et al.,2008;llopis et al.,2013;han et al.,2013)。在鼻咽癌中,nr4a2高细胞质表达与肿瘤大小、淋巴结转移及临床分期显著相关。此外,与细胞质nr4a2表达较低的患者相比,细胞质nr4a2表达较高的患者表现出显著较低的生存率(wang et al.,2013f)。
[0508]
靶向作用于mark的sirna抑制宫颈癌细胞系生长,并导致nup188的下调(huang et al.,2008;yuan et al.,2010)。nup188似乎是乳腺癌中肿瘤抑制基因brca1的一个靶标(bennett et al.,2008)。透过k
‑
纤维形成和募集numa到纺锤体极的有丝分裂中染色体排列需要nup188(itoh et al.,2013)。
[0509]
nup205被tmem209稳定。这种相互作用是肺癌细胞增殖的关键驱动因子(fujitomo et al.,2012)。
[0510]
nup62与经培养的高级别卵巢癌细胞的药物抗性相关(kinoshita et al.,2012)。
[0511]
opa1过度表达在嗜酸甲状腺肿瘤以及肺腺癌中检测到(fang et al.,2012;ferreira
‑
da
‑
silva et al.,2015)。其他报告显示,肝细胞癌细胞可透过opa1 sirna敲减对索拉非尼诱导的凋亡变得敏感。此外,opa1表达的沉寂导致顺铂抗性降低,增加细胞色素c的释放和启动胱天蛋白酶依赖性凋亡途径(fang et al.,2012;zhao et al.,2013b)。
[0512]
在转移性透明细胞肾细胞癌标本中观察到orc2水平升高(tan et al.,2008)。研究人员证明,表达orc2的plk1非磷酸化变体的胰腺癌细胞对吉西他滨治疗更敏感(song et al.,2013a)。
[0513]
osbpl10被证明是在乳腺癌中突变的致癌基因(pongor et al.,2015)。osbpl10被证明是原发性中枢神经系统淋巴瘤相关的异常体细胞超突变的靶标(vater et al.,2015)。
[0514]
pak6被证明在结肠癌组织和细胞系以及肝细胞癌中上调(chen et al.,2014b;tian et al.,2015)。pak6被证明在透明细胞肾细胞癌下调(liu et al.,2014e)。pak6被证明可促进结肠癌的化学抵抗性和进展,以及细胞系lncap中前列腺细胞的活动性和侵袭性(liu et al.,2013c;chen et al.,2015b)。pak6与前列腺癌有关(zapatero et al.,2014)。pak6与透明细胞肾细胞癌的不良总生存期和无复发生存期、肝细胞癌的不良预后以及头颈癌细胞系的药物(吉非替尼)抗性有关(chen et al.,2014b;liu et al.,2014e;hickinson et al.,2009)。pak6是ii、iii期结肠癌5
‑
fu辅助化疗、结直肠的总体和无病生存率、以及透明细胞肾细胞癌肾切除后的无复发生存率的预后标志物(liu et al.,2014e;chen et al.,2015b)。pak6可能是区分宫颈腺癌和宫颈鳞状细胞癌的有用标记物(lee et al.,2010)。
[0515]
pard6b被证明是p53的新候选靶向基因(garritano et al.,2013)。pard6b被证明在乳腺癌细胞系中上调(cunliffe et al.,2012)。pard6b被证明在人类上皮结肠直肠腺癌细胞系caco
‑
2的形态发生中发挥作用(durgan et al.,2011)。pard6b被证明受乳腺癌细胞系mcf
‑
7中的癌基因类固醇受体共启动因子
‑
3调节(labhart et al.,2005)。
[0516]
parp10被证明与细胞凋亡、nf
‑
kb信令和dna损伤修复相关,可能在癌症生物学发挥功能(kaufmann et al.,2015)。pard6b被证明是nf
‑
kb信令的一个调节因子(verheugd et al.,2013)。parp10被证明与原癌基因c
‑
myc相互作用(yu et al.,2005)。
[0517]
parp14是介导转移性前列腺癌细胞的增殖、化学抗性和存活的一个因子(bachmann et al.,2014)。parp14在骨髓瘤浆细胞高表达,与疾病进展和不良生存期相关。parp14与jnk2依赖型生存期极度相关。parp14被发现可透过结合和抑制jnk1促进骨髓瘤细胞的存活(barbarulo et al.,2013)。
[0518]
研究人员发现在原发性胶质母细胞瘤组织中mrna和parp3蛋白水平升高。另一组发现在乳腺癌以及非小细胞肺癌中parp3下调(frias et al.,2008;bieche et al.,2013;quan et al.,2015a)。parp3基因沉寂导致胶质母细胞瘤异种移植物小鼠模型中体内细胞增殖和肿瘤生长抑制下降。在肺癌细胞系中,mir
‑
630透过下调几个凋亡调节剂(如,parp3)减少细胞凋亡(cao et al.,2014;quan et al.,2015a)。
[0519]
parpbp被证明在胰腺癌中上调(o'connor et al.,2013)。parpbp被证明在hela细胞系中与宫颈癌潜在相关(van et al.,2012)。
[0520]
pawr被证明在许多癌症中下调,包括乳腺癌、淋巴瘤和肾细胞癌(cook et al.,1999;boehrer et al.,2001;nagai et al.,2010)。此外,pawr表达下降与乳腺癌患者预后不良相关(nagai et al.,2010;alvarez et al.,2013)。透过akt的pawr磷酸化导致其在细胞质中结合和隐蔽,从而防止前列腺癌细胞凋亡(goswami et al.,2005)。
[0521]
pbxip1被证明在结直肠癌、口腔鳞状细胞癌、高级别胶质瘤、室管膜瘤和肝癌中上
调(xu et al.,2013b;van vuurden et al.,2014;okada et al.,2015;feng et al.,2015b)。pbxip1与乳腺癌和肝细胞癌有关(okada et al.,2015;bugide et al.,2015;wang et al.,2008)。pbxip1促进结直肠癌细胞迁移和侵袭(feng et al.,2015b)。pbxip1与结直肠的不良临床预后和平滑肌肉瘤的总生存期相关(silveira et al.,2013;feng et al.,2015b)。
[0522]
pcbp4被证明在肺癌中下调(pio et al.,2004)。
[0523]
pdia3可用作生物标志物和用于肿瘤诊断(shishkin et al.,2013)。pdia3在神经胶质瘤中分化表达(deighton et al.,2010)。pdia3涉及人体病理,包括癌症和阿尔茨海默氏病(coe and michalak,2010)。pdia3是在mhc i类分子上加载病毒和自我肽的tap辅助因子(coe and michalak,2010;abele and tampe,2011)。
[0524]
phb启动参与细胞生长和恶性转化的raf/mek/erk通路(rajalingam and rudel,2005)。phb是鼻咽癌的一种潜在生物标志物,可预测对放射疗法的治疗反应(chen et al.,2015e)。phb在耐药癌细胞、药物作用和疾病状态组织的蛋白质组分析中被确定(guo et al.,2013)。phb在许多癌症实体中过度表达(zhou and qin,2013)。丙型肝炎病毒的核心蛋白是肝细胞癌的主要危险因素,其透过削弱抑制素诱导过度产生氧化应激(theiss and sitaraman,2011;schrier and falk,2011;koike,2014)。phb在神经胶质瘤中分化表达(deighton et al.,2010)。
[0525]
phf20l1被证明在细胞系zr
‑
75
‑
30中与乳腺癌相关(schulte et al.,2012)。phf20l1与卵巢癌有关(wrzeszczynski et al.,2011)。
[0526]
phkg2在甲状腺乳头状癌中经常甲基化(kikuchi et al.,2013)。phkg2在子宫内膜癌中失调,并可充当一种分子生物标志物(colas et al.,2011)。
[0527]
phrf1与急性早幼粒细胞白血病有关(prunier et al.,2015)。phrf1被证明在乳腺癌中删除或沉寂(ettahar et al.,2013)。
[0528]
与正常肝组织相比,在肝细胞癌中观察到pi4ka水平升高。另外,在胰腺癌细胞系中检测到pi4ka基因(ishikawa et al.,2003;ilboudo et al.,2014)。pi4kamrna浓度较高的肝细胞癌患者肿瘤复发以及疾病特定生存期较短风险较高(ilboudo et al.,2014)。最近,pi4ka被发现参与髓母细胞瘤细胞系的细胞增殖和顺铂耐药。其他人的研究显示,pi4ka在胰腺癌的侵袭和转移中起着至关重要的作用(ishikawa et al.,2003;guerreiro et al.,2011)。
[0529]
研究人员证明了应用piga突变导致gpi锚定蛋白表达丧失作为发现癌症突变基因(mut)表型的新技术(chen et al.,2001)。最近的研究表明,piga引起大鼠c6胶质瘤细胞凋亡。此外,在piga处理细胞中观察到细胞色素c的胞质积累、胱天蛋白酶
‑
3活化和dna片段化。其他报告显示,存在piga突变的白血病细胞与其被自然杀伤细胞体外杀伤的对照配对物相比较不敏感(nagakura et al.,2002;chelli et al.,2005)。
[0530]
在受结直肠癌影响的患者中检测到了pigk基因的单核苷酸多态性。另一份报告观察到pigk mrna水平在膀胱癌、肝细胞癌和结肠癌中下调(nagpal et al.,2008;dasgupta et al.,2012)。
[0531]
pja1被证明在胃癌中上调(mishra et al.,2005a)。
[0532]
相对于未分化甲状腺癌,pja2过度表达发现于乳头状甲状腺癌和胶质母细胞瘤样
本的裂解物(cantara et al.,2012;lignitto et al.,2013)。此外,pja2
‑
fer酪氨酸激酶mrna嵌合体也被发现与非小细胞肺癌的术后预后差相关(kawakami et al.,2013)。最近的研究证明,pja2是控制camp依赖性pka活性和促存活信令的关键元素(hedrick et al.,2013)。
[0533]
pkhd1l1被证明在t
‑
细胞大颗粒淋巴细胞白血病中被表达为融合转录物(izykowska et al.,2014)。
[0534]
在胃癌中,pla2g6的水平升高与肿瘤大小、肿瘤分化程度和tnm分期相关,它是胃癌患者生存期的一种独立预测因子(wang et al.,2013h)。在多种人类癌症中,包括胆管癌、胃癌、结直肠癌、肺癌、胰腺癌、膀胱癌和巴雷特腺癌中检测到pla2g6过度表达(wu et al.,2002;lagorce
‑
pages et al.,2004;cai et al.,2013a;wang et al.,2013h)。溴烯醇内酯——pla2g6抑制剂引起卵巢癌细胞凋亡以及诱导s
‑
和g2/m期细胞周期阻滞的增加(song et al.,2007)。
[0535]
若干出版物显示,plaur在各种肿瘤中上调,例如,膀胱尿路上皮瘤、结直肠癌和乳腺癌(bianchi et al.,1994;illemann et al.,2014;dohn et al.,2015)。plaur过度表达与结直肠癌和胃癌患者的总体生存相关(yang et al.,2000;seetoo et al.,2003;alpizar
‑
alpizar et al.,2012)。
[0536]
plch1与肺鳞状细胞癌有关(zhang et al.,2013d)。
[0537]
plekha8被证明与结直肠癌有关(eldai et al.,2013)。plekha8被证明与原发性乳腺癌培养细胞对5
‑
氟尿嘧啶的反应性相关(tsao et al.,2010)。
[0538]
最近的研究确定了plxnc1的体细胞错义突变以及胰腺导管腺癌和黑色素瘤的拷贝数量损失。另一组显示了plxnc1在转移性黑色素瘤中相对于原发性黑色素瘤的显著损失。其他的报告显示,plxnc1在急性髓性白血病中下调(stirewalt et al.,2008;lazova et al.,2009;balakrishnan et al.,2009)。似乎plxnc1显著抑制黑色素瘤的迁移和增殖(chen et al.,2013c)。
[0539]
一项基因关联分析显示poln在肺癌中边缘性显著(kazma et al.,2012)。poln被证明与黑色素瘤家族(有和没有cdkn2a突变)中黑色素瘤风险增加相关(liang et al.,2012b)。poln被证明参与dna修复,与同源重组和交联修复有关(moldovan et al.,2010)。poln显示被神经母细胞瘤的易位断点破坏,因此,可能在神经母细胞瘤的发展中发挥作用(schleiermacher et al.,2005)。
[0540]
rna聚合酶i(聚合酶i)的活性在人类癌症中通常失调。polr1a充当pol i大催化亚基蛋白,因此可代表癌症的治疗靶点(colis et al.,2014)。此外,药物诱发的polr1a破坏被证明与nci60癌细胞系的癌细胞杀伤相关(peltonen et al.,2014)。polr1a的干扰显示可抑制rrna合成并阻碍含灭活p53的细胞的细胞周期进展。因此,polr1a可能是妨碍p53缺陷癌细胞增殖的一种新颖选择性靶标(donati et al.,2011)。
[0541]
polr1b被证明受原癌基因c
‑
myc的调节(poortinga et al.,2011)。polr1b被证明与治疗有关急性骨髓性白血病的发病机制相关(cahan and graubert,2010)。
[0542]
最近的研究确定了pom121为白血病和儿童急性淋巴细胞白血病中的一种pax5融合蛋白(nebral et al.,2009;fortschegger et al.,2014)。
[0543]
在mcf7dap3kd和mda
‑
mb
‑
231dap1kd乳腺癌细胞系中发现了低水平的ppip5k1
(wazir et al.,2015a;wazir et al.,2015b)。高水平ppip5k1被证明可促进促凋亡基因trail的诱导,而抗凋亡基因,如bcl2、birc3和prkce受到抑制。此外,ppip5k1能够诱导胱天蛋白酶的活化。最近的研究表明,ppip5k1透过lkb1灭活诱导癌细胞迁移、侵袭和肿瘤转移(rao et al.,2015;kumar et al.,2015)。
[0544]
ppp2r1a基因的突变可归因于各种癌症,例如,乳腺癌、前列腺癌和子宫浆液性癌。其他人观察到,ppp2r1a突变在卵巢癌、子宫内膜癌中不常见,在透明细胞和癌肉瘤亚型中不存在(calin et al.,2000;shih et al.,2011;cheng et al.,2011;nagendra et al.,2012;rahman et al.,2013)。研究人员证明,
[0545]
prame显示在多发性骨髓瘤、透明细胞肾细胞癌、乳腺癌、急性骨髓性白血病、黑色素瘤、慢性粒细胞白血病、头颈部鳞状细胞癌和骨肉瘤细胞系中上调(dannenmann et al.,2013;yao et al.,2014a;zou et al.,2012;szczepanski and whiteside,2013;zhang et al.,2013b;beard et al.,2013;abdelmalak et al.,2014;qin et al.,2014)。prame与黏液样和圆形细胞脂肪肉瘤相关(hemminger et al.,2014)。prame与接受r
‑
chop治疗的弥漫性大b细胞淋巴瘤的较短无进展生存期和化疗反应、头颈部鳞状细胞癌不良预后的指标、尿路上皮癌的化疗反应不佳以及骨肉瘤的不良预后和肺转移相关(tan et al.,2012;dyrskjot et al.,2012;szczepanski et al.,2013;mitsuhashi et al.,2014)。prame与急性淋巴细胞性白血病的较低复发、较低死亡率和总生存率相关(abdelmalak et al.,2014)。prame可能是接受r
‑
chop疗法治疗的弥漫性大b细胞淋巴瘤的预后标志物(mitsuhashi et al.,2014)。
[0546]
若干出版物表明,乳头状肾细胞癌中发现的易位导致prcc基因融合至tfe3转录因子(sidhar et al.,1996;weterman et al.,1996;weterman et al.,2001)。
[0547]
一些研究人员观察到prkar1a表达在未分化甲状腺癌中相对于正常甲状腺组织和分化型甲状腺肿瘤的显著增加。与此相反,在牙源性肿瘤子集中报告了prkar1a表达下调。另一组显示,prkar1a可能参与牙源性黏液瘤以及散发性肾上腺皮质腺瘤的发病机制(bertherat et al.,2003;perdigao et al.,2005;ferrero et al.,2015;sousa et al.,2015)。
[0548]
prkdc是子宫内膜异位症相关卵巢癌和乳腺癌中常见的突变基因(er et al.,2016;wheler et al.,2015)。在结直肠癌中,与正常组织相比,prkdc在癌组织中上调。prkdc高表达的患者表现出较差的总生存期(sun et al.,2016b)。
[0549]
在颚骨的角化牙源性肿瘤中检测到prkx过度表达(kong et al.,2015)。据报告,prkx下调导致肾癌和黑色素瘤细胞系对舒尼替尼(sunitinib)敏感。同样地,在三种folr1 sirna治疗的紫杉醇耐药性鼻咽癌细胞中检测到prkx水平降低(bender and ullrich,2012;song et al.,2015b)。
[0550]
研究已经检测到prky在前列腺癌组织中表达,而在性腺母细胞瘤中检测不到prky表达(dasari et al.,2001;lau and zhang,2000;su et al.,2006)。
[0551]
prpf8与急性髓细胞白血病的不良预后以及mcl1依赖型神经母细胞瘤的耐药性有关(laetsch et al.,2014;kurtovic
‑
kozaric et al.,2015)。
[0552]
prrc1被证明与继发性急性淋巴细胞性白血病中的mll融合(douet
‑
guilbert et al.,2014)。
[0553]
据报告,psap在许多雄激素非依赖性前列腺癌细胞系、乳腺癌细胞系和食道鳞状细胞癌中被扩增和过度表达(koochekpour et al.,2005b;pawar et al.,2011;wu et al.,2012f)。此外,psap的高mrna水平与接受一线三苯氧胺疗法治疗复发性疾病的乳腺癌患者的较短无进展生存期显著相关(meijer et al.,2009)。最近的研究表明,psap诱导前列腺癌细胞系的细胞增殖、迁移和侵袭(lee et al.,2004;koochekpour et al.,2005a)。
[0554]
psma4基因的单核苷酸多态性与中国汉族人群肺癌风险相关。其他报告显示,psma4基因的单核苷酸多态性不是非小细胞肺癌易感性的主要促成因素。此外,与正常肺组织相比,在肺肿瘤中观察到了psma4过度表达(liu et al.,2008a;liu et al.,2009b;yongjun zhang et al.,2013;wang et al.,2015e)。
[0555]
在转基因小鼠肿瘤以及人肝细胞癌中报告了psmc2上调(cui et al.,2006)。psmc2推测可能在急性早幼粒细胞白血病细胞系的细胞凋亡和部分分化中起着重要作用(wang et al.,2003)。
[0556]
psmc3被确定为人胃癌相关抗原。此外,psmc3能够与肝细胞癌患者的血清反应(zeng et al.,2007;uemura et al.,2003)。
[0557]
与相应的邻近正常前列腺组织相比,psmc4在前列腺癌细胞中显著和相干上调(hellwinkel et al.,2011)。
[0558]
在结肠癌、骨髓瘤和肝细胞癌中检测到psmd4水平升高(arlt et al.,2009;midorikawa et al.,2002;shaughnessy,jr.et al.,2011)。
[0559]
psmd8在周围肺细胞中表达增加可能对哪些临界细胞群体参与侵入性癌症发生提供潜在的信息(zhou et al.,1996)。
[0560]
ptplad2被证明在食管鳞状细胞癌中下调,这是与不良预后相关(zhu et al.,2014b)。ptplad2被证明与stat3相互作用并在上调后抑制肿瘤增殖。因此,ptplad2是一种潜在的肿瘤抑制因子和预后指标以及食管鳞状细胞癌治疗的可能靶标(zhu et al.,2014b)。ptplad2被描述为包含于胶质母细胞瘤纯合缺失位点内的一个新候选肿瘤抑制基因(nord et al.,2009)。
[0561]
在人乳腺癌细胞系的子集中观察到了ptpn2蛋白水平下调。此外,ptpn2在所有人类t细胞急性淋巴细胞白血病中删除。另外,在霍奇金淋巴瘤细胞系sup
‑
hd1中发现到ptpn2基因的双等位基因失活(kleppe et al.,2010;kleppe et al.,2011a;kleppe et al.,2011b;shields et al.,2013)。最近的工作表明,ptpn2基因损失和较低的mrna水平与乳腺癌预后不良相关(karlsson et al.,2015)。似乎ptpn2透过抑制jak/stat信号通路充当一种典型的癌症抑制因子(kleppe et al.,2011b)。
[0562]
在胃癌组织中以及胶质瘤中发现了ptpru表达水平升高。其他的报告显示,ptpru充当结肠癌的一种肿瘤抑制因子(yan et al.,2006;zhu et al.,2014c;liu et al.,2014i)。此外,ptpru敲减抑制胃癌的生长和活动性,而在胶质瘤中,它抑制增殖、存活、侵袭、迁移和黏附。在乳腺癌中,ptpru阻止肿瘤生长和转移形成(zhu et al.,2014c;liu et al.,2014i;liu et al.,2015f)。
[0563]
pwp1被证明在胰腺癌中上调(honore et al.,2002)。
[0564]
在多重耐药性癌细胞系中观察到了pygl过度表达。此外,pygl基因多态性与儿童急性淋巴细胞白血病复发的较高风险相关(heim and lage,2005;yang et al.,2012c)。
[0565]
rad54l2与胃肠道间质瘤较短的总体生存期相关(schoppmann et al.,2013)。
[0566]
ralgapb枯竭被证明可导致染色体错位,降低有丝分裂细胞周期蛋白b1,而过度表达干扰细胞分裂。ralgapb失调可引起基因组不稳定性,导致癌变(personnic et al.,2014)。ral gtp酶启动蛋白的抑制被证明可造成mtorc1依赖性胰腺肿瘤细胞侵袭,提示ral和mtor信号网络之间的串扰。mtor信令与癌症相关(martin et al.,2014)。
[0567]
若干出版物观察到了rarres3表达在基底细胞癌和鳞状细胞癌中减弱(disepio et al.,1998;duvic et al.,2000;duvic et al.,2003)。此外,rarres3被证明可抑制宫颈癌细胞的ras信令途径(tsai et al.,2006)。在皮肤癌中,rarres3显示可诱导中心体周围细胞器聚集,而这又导致细胞周期蛋白d1、细胞周期蛋白e和细胞周期蛋白a水平降低和p21水平升高。此外,在睾丸癌细胞中,rarres3显著抑制细胞迁移和侵袭(scharadin et al.,2011;wu et al.,2012b)。
[0568]
rasal2是一种ras
‑
gtp酶活化蛋白,在雌激素受体阳性乳腺癌、卵巢癌和肺癌中具有肿瘤抑制功能(huang et al.,2014d;li and li,2014)。与此相反,rasal2在三阴乳腺癌中具有致癌性,并促进间质浸润和转移(feng et al.,2014b)。
[0569]
rasgef1b被描述为一种ras启动启动子,其由细胞周期相关转录因子e2f1调节(korotayev et al.,2008)。
[0570]
rbm47与乳腺癌的进展和转移相关(vanharanta et al.,2014)。
[0571]
最近的工作显示,rcc1在低分化胃细胞系和胃癌组织中下调。其他人报告显示,在pten null t细胞白血病系中回应于pten表达的rcc1水平升高(huang et al.,2005b;lin et al.,2015c)。在胃癌中,rcc1表达缺失与肿瘤的分化和浸润深度有关(lin et al.,2015c)。
[0572]
rec8编码rec8减数分裂重组蛋白,它是染色体蛋白伙伴结构维持kleisin家族的成员(refseq,2002)。最近的一项研究显示,rec8基因在皮肤t细胞淋巴瘤以及在源自患者的细胞系中不均匀表达。其他人的研究表明,rec8在黑色素瘤中超甲基化。此外,rec8在内多倍体肿瘤细胞上组成型表达(litvinov et al.,2014a;furuta et al.,2006;erenpreisa et al.,2009)。rec8的超甲基化与甲状腺癌患者的较差临床病理结果(包括晚期肿瘤、疾病期别和患者死亡率)相关(liu et al.,2015a)。
[0573]
在松果体区乳头状肿瘤和原发性睾丸弥漫性大b细胞淋巴瘤中检测到基因组重排或rfx3的过度表达。其他报告显示,rfx3在胃癌细胞中表达水平低(twa et al.,2015;fevre
‑
montange et al.,2006;seidl et al.,2010)。
[0574]
rfx5基因突变发现于微卫星不稳定性结直肠癌病变中。这些发现表明,rfx5基因突变代表着肿瘤细胞中hla ii类抗原表达损失的新机制。最近的工作表明,rfx5与胃肠癌相关(satoh et al.,2004;michel et al.,2010;surmann et al.,2015)。
[0575]
rhpn2被证明与结直肠癌有关(he et al.,2015)。rhpn2多态性可能结直肠癌手术切除患者的预后生物标志物(he et al.,2015)。rhpn2被证明与结直肠癌的生存结果、无病生存和总体生存预后更差以及胶质母细胞瘤患者生存期下降有关(danussi et al.,2013;kang et al.,2015a)。rhpn2被证明在人类垂体非功能性腺瘤形成中发挥作用(zhan and desiderio,2006)。
[0576]
rint1被描述为胶质母细胞瘤的一种癌基因,作为乳腺癌和lynch症候群相关癌症
中所见的一种适度渗透剂癌症易感基因(ngeow and eng,2014;quayle et al.,2012)。
[0577]
ripk3被证明在结直肠癌、乳腺癌和浆液性卵巢癌中下调(mccabe et al.,2014;koo et al.,2015a;feng et al.,2015a)。ripk3表达与宫颈癌基于polylc
‑
免疫治疗方法的临床结果以及在5
‑
氨基乙酰丙酸介导的光动力疗法后骨肉瘤细胞系u2os更好的生存有关(coupienne et al.,2011;schmidt et al.,2015)。ripk3与非霍奇金淋巴瘤和肺癌有关(yang et al.,2005;fukasawa et al.,2006;cerhan et al.,2007)。ripk3是结直肠癌总体生存率和无病生存率的独立预后因素(feng et al.,2015a)。ripk3是预测凋亡抗性和晚期食管癌的顺铂敏感性的潜在标志物(xu et al.,2014b)。
[0578]
ripk4被证明在皮肤鳞状细胞癌中下调(poligone et al.,2015)。ripk4与舌鳞癌细胞系tca
‑
8113的迁移和侵袭,弥漫性大b细胞淋巴瘤的生存期以及宫颈鳞状细胞癌总体生存、无病生存、进展和不良预后相关(wang et al.,2014h;liu et al.,2015b;kim et al.,2008e)。ripk4与家族性胰腺癌相关(lucito et al.,2007)。ripk4可能是宫颈鳞状细胞癌的一个潜在的诊断性和独立的预后标志物,是舌癌预后和治疗的生物标志物(wang et al.,2014h;liu et al.,2015b)。
[0579]
rnf167的ring结构域点突变在人肿瘤样本中确定,其阻止了泛素连接酶的活性和功能(van dijk et al.,2014)。rnf167与ubch6一起充当推定肿瘤抑制因子tssc5的一种泛素连接酶,tssc5是一种在某些肿瘤中发生突变的基因。rnf167与ubch6一起可确定靶向作用于tssc5的一种新泛素
‑
蛋白酶体途径(yamada and gorbsky,2006)。
[0580]
rnf20被证明在睾丸精原细胞瘤和转移性前列腺癌中下调(jaaskelainen et al.,2012;chernikova et al.,2012)。
[0581]
rnf213与慢性骨髓性白血病有关(zhou et al.,2013b)。rnf213与间变性淋巴瘤激酶阳性间变性大细胞淋巴瘤的预后较差相关(moritake et al.,2011)。
[0582]
rnf31被证明在乳腺癌中以及骨肉瘤lm8细胞系的肺转移中上调(tomonaga et al.,2012;zhu et al.,2015)。rnf31与弥漫性大b细胞淋巴瘤活化的b细胞样亚型有关(yang et al.,2014f;grumati and dikic,2014)。rnf31与卵巢癌顺铂耐药性相关(mackay et al.,2014)。
[0583]
相比于正常睾丸,睾丸生殖细胞癌精原细胞瘤中报告了rnf40下调。其他人也观察到了结直肠癌中的rnf40低水平(chernikova et al.,2012;tarcic et al.,2016)。此外,rnf40损失强烈阻碍了前列腺癌细胞的生长(jaaskelainen et al.,2012)。
[0584]
最近,在黑色素瘤中确定了rqcd1基因突变。此外,在乳腺癌标本以及乳腺癌细胞系中发现了rqcd1过度表达(ajiro et al.,2009;wong et al.,2015)。在乳腺癌细胞系中,rqcd1蛋白被证明与gigyf1和gigyf2蛋白相互作用,这些蛋白参与了akt启动的调节。此外,rqcd1敲减导致akt磷酸化水平降低,这由表皮生长因子的刺激引起(ajiro et al.,2009;ajiro et al.,2010)。
[0585]
最近的工作证明,rtn2过度表达在人乳腺癌细胞系中诱导抗雌激素抗性(near et al.,2007;makkinje et al.,2009)。
[0586]
在口腔鳞状细胞癌患者的唾液中检测到了rtn3过度表达。同样地,在各个期别的上皮性卵巢癌患者的血清中检测到了rtn3水平升高,特别是第iii期最高。其他人在白血病和泌尿生殖癌中观察到了rtn3高水平(mitchell et al.,1988;dunzendorfer et al.,
1980;chen et al.,2009c;jessie et al.,2013)。另外,循环rtn3显示与上皮性卵巢癌患者的肿瘤期别和生存期相关(zhao et al.,2007)。
[0587]
samd9被证明在乳腺癌、结肠癌、非小细胞肺癌和纤维瘤病中下调(ma et al.,2014;li et al.,2007)。samd9与非小细胞肺癌细胞系h1299的侵袭、迁移和增殖,食管鳞状细胞癌和髓系白血病的淋巴浸润和转移相关(nagamachi et al.,2013;tang et al.,2014b;ma et al.,2014)。
[0588]
samsn1被证明在多形性胶质母细胞瘤中上调(yan et al.,2013c)。samsn1被证明在肝细胞癌、多发性骨髓瘤和大细胞肺癌细胞系calu
‑
6中下调(noll et al.,2014;sueoka et al.,2015;yamada et al.,2008)。samsn1与溃疡性结肠炎相关癌症和急性骨髓性白血病相关(watanabe et al.,2011;claudio et al.,2001)。samsn1与肝细胞癌的较短总生存期和无复发生存期以及多形性胶质母细胞瘤的总生存期较差相关(yan et al.,2013c;sueoka et al.,2015)。samsn1是肝细胞癌进展的独立预后因素和多发性骨髓瘤的潜在预后标志物(ni et al.,2012;sueoka et al.,2015)。
[0589]
scara3被证明在卵巢/原发性腹膜癌中上调(bock et al.,2012)。scara3是多发性骨髓瘤进展和治疗反应的预测因子(brown et al.,2013)。
[0590]
在乳腺癌细胞系以及在神经母细胞瘤中检测到scnn1a的甲基化。最近的一项研究提议scnn1a可能牵涉睾丸生殖细胞肿瘤的病因,因为视黄酸抑制胚胎癌细胞的致瘤性(giuliano et al.,2005;roll et al.,2008;caren et al.,2011)。研究人员已使用了cox比例风险模型,显示scnn1a可以预测腺癌患者预后(endoh et al.,2004)。
[0591]
sec61a1与前列腺癌有关(bull et al.,2001)。
[0592]
sec61g被证明在胃癌中上调(tsukamoto et al.,2008)。sec61g与胶质瘤有关(neidert et al.,2013)。
[0593]
sesn3被描述为mtor复合体1的一个独特的细胞抑制剂(vakana et al.,2013)。sesn3被描述在活性氧簇解毒背景下,可由肿瘤抑制因子foxo3诱导(hagenbuchner and ausserlechner,2013)。sesn3抑制被描述为在细胞增殖后活性氧簇调节背景下透过致癌ras诱导(zamkova et al.,2013)。sesn3被证明在pc12细胞系神经生长因子介导的分化后由肿瘤抑制基因p53调节(brynczka et al.,2007)。sesn3 5'cpg岛甲基化被证明是一种新的子宫内膜癌特异性标志物(zighelboim et al.,2007)。
[0594]
研究人员已经确定setdb1是斑马鱼黑色素瘤模型以及人肺癌中新的癌基因。此外,在非小细胞肺癌、前列腺癌和胶质瘤中发现setdb1过度表达(ceol et al.,2011;rodriguez
‑
paredes et al.,2014;spyropoulou et al.,2014;sun et al.,2014d;sun et al.,2015f)。似乎setdb1能够积极刺激wnt
‑
β
‑
连环蛋白途径的活性(sun et al.,2015f)。此外,透过sirna的setdb1敲减抑制了前列腺癌细胞的生长、侵袭、迁移,降低了集落形成和诱导了细胞周期停滞(sun et al.,2014d)。
[0595]
sgpp2被证明在鞘氨醇
‑1‑
磷酸酯富集胶质母细胞瘤中下调(abuhusain et al.,2013)。sgpp2被证明是一种nf
‑
κb依赖性基因,因此可能是促炎症信号传导的一种潜在的新基因(mechtcheriakova et al.,2007)。
[0596]
sh3glb2被证明在前列腺癌中上调(fasso et al.,2008)。
[0597]
shisa5与头颈部鳞状细胞癌有关(ghosh et al.,2008)。
[0598]
在脾边缘细胞淋巴瘤以及aids相关卡波济氏肉瘤中观察到siglec1表达增加。其他人发现,siglec1基因的突变与胰腺导管腺癌的发展有关(zhou et al.,2012a;cornelissen et al.,2003;marmey et al.,2006)。siglec1表达水平升高与结直肠癌和恶性黑色素瘤患者的更好预后相关(ohnishi et al.,2013;saito et al.,2015)。
[0599]
sin3a被证明与肺腺癌细胞系a549中的侵袭相关(das et al.,2013b)。sin3a与乳腺癌有关(ellison
‑
zelski and alarid,2010)。sin3a被证明在非小细胞肺癌中下调(suzuki et al.,2008)。
[0600]
在人乳腺癌细胞系、肺腺癌细胞系、黑色素瘤和骨肉瘤观察到skil过度表达。其他报告显示,skil在原发性食管鳞状细胞癌中扩增(imoto et al.,2001;zhang et al.,2003;zhu et al.,2007)。在乳腺癌中,skil表达下降与雌激素受体阳性患者较长的远期无病生存率相关(zhang et al.,2003)。
[0601]
一项研究显示,与pc
‑
3和du145细胞相比较,在前列腺癌细胞系lncap上发现slc15a2 mrna水平最高。其他的报告显示,slc15a2基因的基因组变体可能与肝细胞癌患者对索拉非尼(sorafenib)反应相关(tai et al.,2013;lee et al.,2015)。
[0602]
slc15a3与结直肠癌有关(zhou et al.,2013a)。slc15a3被证明与前列腺癌细胞系lncap、du
‑
145、pc
‑
3和mda2b的前列腺癌相关(ibragimova et al.,2010)。
[0603]
与非肿瘤甲状腺组织相比,在甲状腺髓样癌中报告了slc16a2下调(hudson et al.,2013)。
[0604]
报告显示,slc25a14表达与erα/erβ比率低的绝经后人类乳腺肿瘤显著负相关。其他人在erα/erβ比率低的乳腺癌细胞系中观察到slc25a14水平升高。此外,在结肠癌细胞中发现高水平slc25a14,这与线粒体功能障碍相关(santandreu et al.,2009;nadal
‑
serrano et al.,2012;sastre
‑
serra et al.,2013)。
[0605]
slc28a3被证明在胰腺导管腺癌下调(mohelnikova
‑
duchonova et al.,2013a)。slc28a3与接受紫杉醇和吉西他滨化疗的转移性乳腺癌临床结果、吉西他滨治疗的非小细胞肺癌总体生存、基于吉西他滨化放疗的胰腺癌的总体生存相关(li et al.,2012c;lee et al.,2014b;marechal et al.,2009)。slc28a3与慢性淋巴细胞性白血病的氟达拉滨抗性以及t细胞白血病的耐药性相关(karim et al.,2011;fernandez
‑
calotti et al.,2012)。
[0606]
slc29a3与接受吉西他滨化疗的非小细胞肺癌患者的总体存活以及接受核苷类似物治疗的胰腺癌患者的总体生存相关(mohelnikova
‑
duchonova et al.,2013a;chen et al.,2014f)。slc29a3接受吉西他滨治疗晚期非小细胞肺癌患者一个潜在的预后生物标志物(chen et al.,2014f)。
[0607]
slc34a2的表达在非小细胞肺癌手术样本和正常组织之间显著不同。此外,在肺腺癌细胞系中发现slc34a2表达水平低。其他人已经证明,slc34a2可能是mx35的靶标,mx35是一种开发用于治疗卵巢癌的抗体(yin et al.,2008;yang et al.,2014c;wang et al.,2015k)。另外,肺腺癌细胞系中slc34a2上调能在体外显著抑制细胞生存力和侵袭(wang et al.,2015k)。另一方面,slc34a2表达的减少使乳腺癌干细胞透过slc34a2
‑
bmi1
‑
abcc5信令对阿霉素敏感(ge et al.,2015)。
[0608]
slc35b3与结直肠癌有关(kamiyama et al.,2011)。slc35b3被证明与卵巢癌中的
化疗耐药性相关(cheng et al.,2010)。
[0609]
slc35e1被证明与直肠癌对新辅助放化疗的反应相关(rimkus et al.,2008)。
[0610]
根据报告,slc35e2在神经母细胞瘤中下调(thorell et al.,2009)。
[0611]
slc35e2b转录物显示在不良神经母细胞瘤中表达显著降低(thorell et al.,2009)。
[0612]
在结肠癌和肝细胞癌中观察到slc4a2过度表达。另一方面,slc4a2表达在胃癌中下调(wu et al.,2006;yang et al.,2008b;song et al.,2012)。在结肠癌中,slc4a2水平升高与不良预后相关(song et al.,2012)。此外,slc4a2表达的抑制降低了细胞活力,在亚g1期阻滞细胞周期,并且在低分化肝癌细胞中诱导细胞凋亡(hwang et al.,2009)。
[0613]
slc7a8与平滑肌瘤相关(xia et al.,2010;luo et al.,2009)。slc7a8被证明与卵巢癌细胞系w1变体的药物抗性相关(januchowski et al.,2013)。slc7a8被证明在雌激素受体α阳性乳腺癌细胞系t
‑
47d中上调(thakkar et al.,2010)。
[0614]
若干出版物报告了在前列腺癌、结直肠癌和宫颈上皮内瘤中smarcc1 mrna和蛋白表达增加。与此相反,在卵巢癌细胞系中没有检测到smarcc1蛋白表达(shadeo et al.,2008;heeboll et al.,2008;andersen et al.,2009;delbove et al.,2011)。此外,smarcc1过度表达与结直肠癌预后差和复发有关(andersen et al.,2009)。研究人员证明,smarcc1在精氨酸残基r1064甲基化影响mcf7乳腺癌细胞的集落形成能力。此外,似乎这种修饰完全依赖于carm1(wang et al.,2014e)。
[0615]
smchd1与造血癌有关(leong et al.,2013)。
[0616]
smg1被证明在胰腺癌中上调(wang et al.,2015d)。smg1被证明在肝细胞癌中下调(han et al.,2014)。smg1被证明与胰腺癌细胞系和肺癌细胞系h1299的吉西他滨与顺铂化学敏感性以及肝癌细胞系的索拉非尼抗性有关(xia et al.,2011;nam et al.,2014;wang et al.,2015d)。smg1与急性骨髓性白血病相关(du et al.,2014a)。smg1与肝细胞癌的总体生存较差相关(han et al.,2014)。
[0617]
smpd4被证明与细胞应激反应、dna损伤和p53活化有关,表达被证明在几种原发肿瘤中失调(corcoran et al.,2008)。
[0618]
snd1被证明在非小细胞肺癌、乳腺癌、结肠癌、肝细胞癌、胶质瘤和前列腺癌中上调(cappellari et al.,2014;emdad et al.,2015;yu et al.,2015a;zagryazhskaya et al.,2015)。snd1与非小细胞肺癌的化学抗性相关(zagryazhskaya et al.,2015)。snd1与前列腺癌、原发性皮肤恶性黑色素瘤和皮肤恶性黑色素瘤转移有关(sowalsky et al.,2015;sand et al.,2012)。snd1与肝细胞癌的迁移和侵袭有关(santhekadur et al.,2014)。snd1与结肠癌较短总生存期和不良预后相关(wang et al.,2012b)。snd1是一种很有前途的前列腺癌生物标志物(kuruma et al.,2009)。
[0619]
snrpe在肝细胞癌以及高级别前列腺癌中过度表达(jia et al.,2011;anchi et al.,2012;xu et al.,2015c)。此外,snrpe水平升高与肺癌患者的预后较差相关(valles et al.,2012)。snrpe的sirna
‑
介导耗尽导致乳腺癌、肺癌和黑色素瘤癌细胞系的细胞活力下降(quidville et al.,2013)。
[0620]
研究发现,与健康对照组相比,在滤泡性淋巴瘤、弥漫性大b细胞淋巴瘤和外周t细胞淋巴瘤患者中血清sorl1水平高。另一份报告也指出,sorl1水平在急性白血病患者升高,
al.,2013;park et al.,2015;bandres et al.,2004)。stk17a是肿瘤抑制基因p53的直接靶标,是活性氧簇(ros)的调节剂(kerley
‑
hamilton et al.,2005;mao et al.,2011)。
[0631]
在软组织肉瘤中发现stk3超甲基化,而在头颈部鳞状细胞癌中不那么频繁。其他报告显示,stk3损失导致肝细胞癌的发展(seidel et al.,2007;zhou et al.,2009;steinmann et al.,2009)。
[0632]
stk35被证明可调节cdkn2a和抑制内皮细胞中的g1期到s期过渡,从而在内皮细胞的细胞周期和迁移的连接中发挥作用(goyal et al.,2011)。
[0633]
stk38与b细胞淋巴瘤相关(bisikirska et al.,2013)。stk38被证明与宫颈癌细胞系hela的辐射敏感性相关(enomoto et al.,2013)。stk38显示在胃癌中下调(cui et al.,2005)。
[0634]
stk38l被证明在人体皮肤肿瘤中下调(hummerich et al.,2006)。stk38l与胶质瘤有关(deng et al.,2005)。
[0635]
strada是肿瘤抑制因子lkb1的调节伴侣(sun et al.,2015a)。strada被证明在前列腺癌细胞系lncap的细胞增殖和活力中发挥作用,因此可能是前列腺癌的一种新的药物靶标(dahlman et al.,2012)。strada被证明参与髓母细胞瘤细胞系的细胞增殖和顺铂耐药(guerreiro et al.,2011)。strada被证明在神经管细胞瘤中上调(guerreiro et al.,2011)。strada被证明是一种乳腺癌抗原(scanlan et al.,2001)。
[0636]
在乳腺癌以及小细胞肺癌中发现stx1a过度表达。最近的工作确定stx1a为转移性骨肉瘤的治疗靶标(graff et al.,2001;diao et al.,2014;fernandez
‑
nogueira et al.,2016)。有研究表明,stx1a的表达与乳腺癌亚型的较短总生存期和无远处转移生存率显著相关(fernandez
‑
nogueira et al.,2016)。stx1a的抑制降低了胶质母细胞瘤细胞的增殖和迁移能力(ulloa et al.,2015)。
[0637]
styxl1与尤因氏肉瘤家族肿瘤有关(siligan et al.,2005)。
[0638]
svil主要透过启动子甲基化在前列腺癌组织中显著下调(vanaja et al.,2006)。svil透过p53水平的控制调节细胞的存活。svil表达是生存信号传导和细胞运动途径之间的串扰所必需的(fang and luna,2013)。
[0639]
研究人员在高级别浆液性卵巢癌中发现到扩增、拷贝数增加和taf2的mrna过度表达(ribeiro et al.,2014)。
[0640]
在高级别浆液性卵巢癌中发现到扩增、拷贝数增加或taf4b的mrna上调(ribeiro et al.,2014)。此外,taf4b能够与ap
‑
1一起调节参与上皮至间质转变的靶基因整合素6,因此,改变癌症相关的迁移性质(kalogeropoulou et al.,2010)。
[0641]
tanc2显示在乳腺癌中上调(mahmood et al.,2014)。
[0642]
tap1基因的单核苷酸多态性以及损失似乎牵涉于某些癌症类型,如黑色素瘤、宫颈癌、结直肠癌和头颈部鳞状细胞癌。另一方面,在肺癌和卵巢浆液性癌中观察到tap1上调(yang et al.,2003;meissner et al.,2005;vermeulen et al.,2007;yamauchi et al.,2014;zhang et al.,2015g;nymoen et al.,2015)。此外,tap1的表达与前列腺癌患者的肿瘤分级、临床分期、总生存期和无进展生存期相关(tahara et al.,2015)。在肺癌中,tap1损失抑制细胞增殖,并以p53依赖性方式引起细胞周期阻滞(zhang et al.,2015g)。
[0643]
一些研究没有发现tap2基因多态性与肾细胞癌和宫颈癌之间的相关性。与此相
反,其他研究观察到了tap2基因多态性与慢性淋巴白血病易感性之间的相关性。此外,tap2的表达在乳腺癌、胃癌、小细胞肺癌及头颈部鳞状细胞癌中下降(restifo et al.,1993;vitale et al.,1998;kang et al.,2000;hodson et al.,2003;kordi tamandani et al.,2009;bandoh et al.,2010;ozbas
‑
gerceker et al.,2013)。
[0644]
tcerg1被证明可充当dach1的转录共调节因子,这是一种显示与各种类型癌症相关的转录因子(zhou et al.,2010)。
[0645]
telo2在不同癌症类型(包括白血病、乳腺癌和鼻咽癌)中失调(he et al.,2007;sang et al.,2015;kawagoe et al.,2004)。telo2过度表达降低细胞周期长度,让所述细胞对细胞凋亡超敏感,并增加端粒长度。telo2表达的抑制可逆地停滞细胞周期(jiang et al.,2003)。活化的telo2对于pikk家族蛋白质(如,mtor、atm、atr和smg
‑
1)的稳定性必不可少。telo2在调节翻译、细胞生长和dna损伤信令中具有重要作用(kaizuka et al.,2010;horejsi et al.,2010)。
[0646]
tet3被证明在肝细胞癌、结直肠癌及胃癌中下调(rawluszko
‑
wieczorek et al.,2015;sajadian et al.,2015;du et al.,2015a)。tet3被证明在弥漫性固有脑桥胶质瘤中上调(ahsan et al.,2014)。tet3被证明与肿瘤缺氧、肿瘤恶性和乳腺癌的较差预后相关(wu et al.,2015)。tet3被证明与tnfα
‑
p38
‑
mapk信令有关(wu et al.,2015)。tet3被描述为5
‑
羟甲基化(与恶性肿瘤相关的表观遗传修饰)的调节因子。在平滑肌瘤中,5
‑
羟甲基化内容的表观遗传失衡被描述为tet3上调所致,其可能导致发现平滑肌瘤治疗的新靶标(navarro et al.,2014)。tet3被证明在结肠癌中重复突变,并可提供潜在的治疗干预机会(seshagiri et al.,2012)。tet3被描述为组蛋白修饰和骨髓增生异常症候群和急性髓细胞性白血病wnt通路的的潜在调节因子(gelsi
‑
boyer et al.,2009)。
[0647]
在乳腺癌以及生殖细胞肿瘤中发现tfap2c过度表达(turner et al.,1998;hoei
‑
hansen et al.,2004)。根据报告,tfap2c诱导p21表达、阻滞细胞周期和抑制乳腺癌细胞的肿瘤生长(li et al.,2006)。
[0648]
人类乳头状癌组织中观察到tfdp2下调,而其他报告显示,相比于正常肝脏组织,tfdp2在肝细胞癌中过度表达。此外,tfdp2变体与卵巢癌相关联(liu et al.,2003;lapouge et al.,2005;cunningham et al.,2009)。
[0649]
th1l可能在人乳腺癌细胞增殖和侵袭的调节中起着重要作用,并且可能是人乳腺癌治疗的潜在靶点(zou et al.,2010)。
[0650]
一些研究人员报告,timeless蛋白和mrna在结直肠癌、宫颈癌、肺癌和前列腺癌中过度表达。另一方面,另一项研究报告了timeless在肝细胞癌中下调。此外,timeless基因的单核苷酸多态性与前列腺癌风险无关,但与乳腺癌风险相关(lin et al.,2008b;fu et al.,2012;mazzoccoli et al.,2011;yoshida et al.,2013;mao et al.,2013b;markt et al.,2015;elgohary et al.,2015)。在肺癌中,timeless水平升高与不良的总体生存相关(yoshida et al.,2013)。
[0651]
在各种癌症(包括肺肿瘤、滑膜肉瘤、恶性间皮瘤、白血病和淋巴瘤)中发现tle1过度表达和表观遗传失活(allen et al.,2006;fraga et al.,2008;matsuyama et al.,2010;seo et al.,2011;rekhi et al.,2012)。此外,tle1抑制滑膜肉瘤细胞中阿霉素诱导的细胞凋亡。在肺癌细胞系中,tle1能够透过抑制肿瘤抑制基因e
‑
cadherin而增强上皮间
质转化(seo et al.,2011;yao et al.,2014b)。此外,曲古霉素a被发现显著抑制tle1转基因小鼠中的肺癌发生(liu et al.,2015c)。
[0652]
与良性和非典型脑膜瘤相比,在一些恶性脑膜瘤中观察到tle3过度表达。其他报告显示,tle3剪接同种型水平在前列腺肿瘤以及前列腺肿瘤细胞系中升高(cuevas et al.,2005;nakaya et al.,2007)。研究表明,tle3 mrna水平可预测接受他莫昔芬的乳腺癌患者的无进展生存期。相比之下,其他人报告了tle3表达不代表是乳腺癌紫杉烷益处的一个可行的生物标志物。另一项报告表明,tle3表达预示对含卵巢癌化疗方案的紫杉烷反应良好(van et al.,2009;samimi et al.,2012;bartlett et al.,2015)。
[0653]
最近的工作确定了tle4基因在急性骨髓性白血病中的错义突变。其他研究显示,tle4在结直肠癌以及腺瘤中过度表达(greif et al.,2011;ruebel et al.,2006;wang et al.,2016a)。在结肠癌中,tle4水平升高与结直肠癌的dukes晚期分期、淋巴结转移和预后不良相关(wang et al.,2016a)。似乎mir
‑
93过度表达负调节tle4的mrna和蛋白表达(yu et al.,2011)。
[0654]
以前的研究在多种肿瘤(包括前列腺癌、口腔鳞状细胞癌、卵巢浆液性癌和鼻咽癌)中发现tln1过度表达(sakamoto et al.,2010;lai et al.,2011;tang et al.,2013;xu et al.,2015d)。tln1过度表达与口腔鳞状细胞癌患者的总体生存期下降有关(lai et al.,2011)。似乎tln1s425磷酸化在β1整合素启动、细胞黏附、迁移、侵袭和前列腺癌细胞转移中起着至关重要的作用。此外,tln1水平升高与肝细胞癌细胞系的侵袭性和迁移降低以及恶性下降相关(fang et al.,2014;jin et al.,2015)。
[0655]
tlr7被证明在胰腺癌、口腔鳞状细胞癌和肝细胞癌中上调(mohamed et al.,2015;ni et al.,2015;grimmig et al.,2015)。tlr7与胰腺癌的肿瘤细胞增殖和化学抗性相关(grimmig et al.,2015)。tlr7过度表达与肺癌的不良临床结果和化疗耐药以及口腔鳞状细胞癌的较差预后相关(ni et al.,2015;dajon et al.,2015)。tlr7与膀胱癌有关(cheng et al.,2014)。
[0656]
tmem14c与乳腺癌存活相关(burleigh et al.,2015)。tmem14c与乳腺癌细胞系zr
‑
75
‑
1的他莫昔芬抗性相关(zarubin et al.,2005)。
[0657]
tmem189
‑
ube2v1同种型2(uev1b)被证明与泛素和hrs相关,并且蛋白过度表达阻止了hrs与癌症相关蛋白egfr共定位的能力(duex et al.,2010)。
[0658]
tmprss13编码ii型跨膜丝氨酸蛋白酶家族的一员,已知在发展、稳态、感染和肿瘤发生中发挥作用(refseq,2002)。tmprss13被证明可充当肝细胞生长因子(hgf)
‑
转化酶,转化亲hgf为生物活性hgf。hgf被证明与癌基因c
‑
met相互作用,并与多种癌症相关(hashimoto et al.,2010)。
[0659]
tnfaip2编码tnfα诱导蛋白2,它已表明是急性早幼粒细胞性白血病的一种视黄酸靶基因(refseq,2002)。tnfaip2 rs8126多态性与头颈部鳞状细胞癌、胃癌和食管鳞状细胞癌的易感性显著相关。此外,与邻近正常组织相比,tnfaip2 mrna和蛋白被发现在鼻咽癌肿瘤细胞中升高。其他人观察到tnfaip2在胶质瘤样本过度表达(chen et al.,2011;liu et al.,2011;xu et al.,2013c;zhang et al.,2014b;cheng et al.,2015b)。此外,tnfaip2过度表达与鼻咽癌患者的较短无远处转移生存期相关(chen et al.,2011)。
[0660]
在卵巢癌和恶性间皮瘤中观察到了tnxb上调,而在周围神经鞘瘤中,tnxb显著下
al.,2015)。trip13过度表达被描述为癌症细胞的标志,显示染色体不稳定性(wang et al.,2014d)。透过trip13过度表达引发的有丝分裂关卡提前沉寂表明可促进癌症发展(wang et al.,2014d)。trip13被证明参与乳腺癌肿瘤细胞运动性的调节(maurizio et al.,2016)。trip13在头颈部鳞状细胞癌的高表达被证明可侵袭性和治疗抵抗性肿瘤,增强dna损伤的修复,并促进容易出错的非同源末端连接(banerjee et al.,2014)。trip13被描述为前列腺癌进展的推定标记物,可以与手术前psa水平和gleason评分结合来预测前列腺癌复发(larkin et al.,2012)。trip13被描述为证明在早期非小细胞肺癌中有高基因拷贝数变化的几个基因之一(kang et al.,2008a)。
[0668]
最近的研究提示,trps1出现于多种人类癌症,如乳腺癌、结肠癌、骨肉瘤、白血病、子宫内膜癌和前列腺癌(常牵连(chang et al.,2004;asou et al.,2007;chen et al.,2010;liang et al.,2012a;hong et al.,2013;li et al.,2015f)。此外,trps1表达与乳腺癌患者的生存期改善显著相关(chen et al.,2010)。此外,trps1过度表达透过影响乳腺癌的血管内皮生长因子表达而诱导血管生成(hu et al.,2014)。
[0669]
在结直肠癌和黑色素瘤中发现trrap基因突变,而在甲状腺和卵巢癌中无trrap基因突变(wei et al.,2011;murugan et al.,2013;mouradov et al.,2014;zou et al.,2015)。此外,trrap敲减导致培养的脑肿瘤起始细胞自我更新减少,并使细胞对替莫唑胺(temozolomide)诱导的细胞凋亡敏感(wurdak et al.,2010)。
[0670]
tsc2基因的单核苷酸多态性与结肠癌显著相关。此外,在肝细胞癌和急性髓细胞白血病患者中观察到了tsc2下调。在一个病例中,tsc2基因的突变似乎是形成胰腺神经内分泌肿瘤的原因。其他人指出磷酸化tsc2水平在非小细胞肺癌中升高(xu et al.,2009;yoshizawa et al.,2010;slattery et al.,2010;bombardieri et al.,2013;huynh et al.,2015)。最近的工作证明,erc
‑
18细胞中tsc2的表达增加oka和磷脂酰肌醇
‑
3'激酶抑制剂ly294002诱导的细胞凋亡易感性(kolb et al.,2005)。
[0671]
tsen15是mirna
‑
449a的一个靶标,其功能是作为神经母细胞瘤的肿瘤抑制因子。tsen15在介导mirna
‑
449a分化诱导功能中起重要作用(zhao et al.,2015)。tsen15与人胎儿股骨来源的细胞的细胞分化潜能相关(mirmalek
‑
sani et al.,2009)。
[0672]
tsga13被证明与除了胶质母细胞瘤和肺癌的相邻正常组织相比,在大多数类型的人癌组织中下调。因此,tsga13与肿瘤恶性之间可能存在关系(zhao et al.,2015a)。
[0673]
辐射转化和致瘤性乳腺癌细胞系染色体重排列的tuba1a和一些其他基因的去调节表达可能反映乳腺癌发生的早期分子事件(unger et al.,2010)。采用晚期浆液性卵巢癌的比较蛋白质组学分析发现,tuba1a为化学耐药的一个潜在预测因子(kim et al.,2011c)。
[0674]
tuba1b的差异表达与某些其他基因表达结合与套细胞淋巴瘤的预后相关,预测ii期结直肠癌患者的复发并且区别随后转移和不转移的葡萄膜黑素瘤(blenk et al.,2008;agesen et al.,2012;linge et al.,2012)。tuba1b表达在肝细胞癌组织和增殖性肝细胞癌细胞中上调。tuba1b表达增加与较差的整体生存率和肝细胞癌患者对紫杉醇的耐药相关(lu et al.,2013)。在卵巢癌细胞中,tuba1b表达下降与奥沙利铂耐药有关(tummala et al.,2009)。
[0675]
tuba1c的表达被证明在骨肉瘤和hcv相关肝细胞癌中上调,可能是骨肉瘤肿瘤发
生或分化良好的hcv相关肝细胞癌的潜在生物标志物(li et al.,2010;kuramitsu et al.,2011)。
[0676]
食管鳞状细胞癌(escc)的比较蛋白质组学分析显示tuba4a表达增加(qi et al.,2005)。
[0677]
在小鼠肝脏中,tuba8用一种非基因毒性致癌物苯巴比妥处理后诱导。在肝细胞癌细胞株中,tuba8过度表达显示可影响细胞生长、增殖和迁移(kamino et al.,2011)。
[0678]
若干出版物在人乳腺癌细胞系以及在前列腺癌和鳞状颈癌观察到tyk2过度表达。与此相反,小鼠缺乏tyk2与埃布尔森诱导的b淋巴白血病和淋巴瘤发展有关。此外,tyk2基因的单核苷酸多态性与直肠癌相关(stoiber et al.,2004;ide et al.,2008;song et al.,2008;zhu et al.,2009;slattery et al.,2013)。在前列腺癌细胞系中,sirna对tyk2的压制抑制了这些细胞的迁移能力(ide et al.,2008)。
[0679]
最近的工作确定了肝细胞癌中ube2h基因的拷贝数量增加。其他人观察到了乳腺癌中ube2h水平增加,而在结肠癌中并非如此(chen and madura,2005;keng et al.,2009)。
[0680]
在鼻咽癌中观察到了ube2l6下调,而在食管鳞状细胞癌中,ube2l6过度表达(dadkhah et al.,2013;zhou et al.,2015a)。此外,ube2l6水平低与鼻咽癌患者的较差结果相关(zhou et al.,2015a)。此外,ube2l6已被证明可在乳腺癌细胞系中干扰f
‑
肌动蛋白结构和黏着斑的形成,并促进细胞迁移。此外,ube2l6表达恢复抑制鼻咽癌细胞的增殖和集落形成,而同时诱导细胞凋亡(desai et al.,2012;zhou et al.,2015a)。研究人员推测,ube2l6可以作为急性早幼粒细胞白血病患者中硼替佐米治疗反应的生物标志物(takenokuchi et al.,2015)。
[0681]
乳腺癌样本以及在培养的肿瘤细胞系中检测到ube2v1表达水平升高。此外,ube2v1基因已被确定为与前列腺癌的发展有关(stubbs et al.,1999;xiao et al.,1998;tanner et al.,1995)。研究人员的研究表明,ube2v1诱导乳腺癌的细胞迁移和侵袭。类似地,高水平ube2v1在异种移植小鼠模型中促进肿瘤生长和转移。nsc697923是一种ube2v1抑制剂,能够抑制弥漫性大b细胞淋巴瘤细胞的增殖和存活(pulvino et al.,2012;wu et al.,2014b)。
[0682]
一些研究人员观察到,与邻近正常组织相比,ube3c在透明细胞肾细胞癌组织中过度表达。其他人也发现ube3c水平在肝细胞癌中升高。此外,根据报告,在骨髓瘤的侧群细胞中ube3c基因上调(jiang et al.,2014a;tagawa,2014;wen et al.,2015)。此外,肝细胞癌组织中ube3c过度表达与术后肝细胞癌患者的生存期下降和早期肿瘤复发相关(jiang et al.,2014a)。
[0683]
研究人员已经确定了ube4b基因在神经母细胞瘤患者中突变。全基因组关联分析显示,ube4b基因可能参与乙型肝炎病毒相关肝细胞癌。其他人报告了ube4b在乳腺癌和脑肿瘤中过度表达(krona et al.,2003;zhang et al.,2010b;wu et al.,2011b;zhang et al.,2014f)。此外,ube4b下调与神经母细胞瘤患者不良预后相关(zage et al.,2013)。
[0684]
ubr4被证明与乳腺浸润性微乳头状癌有关(gruel et al.,2014)。
[0685]
unc45a被证明在乳腺癌和卵巢癌中上调(guo et al.,2011;bazzaro et al.,2007)。unc45a与乳腺癌转移有关(guo et al.,2011)。unc45a与神经母细胞瘤的药物抗性
al.,2013b;seliger,2014;liu et al.,2014f;chen et al.,2014i;fan et al.,2014;dong and ma,2015;zhang et al.,2015a)。vtcn1与肾透明细胞癌相关(xu et al.,2014c)。vtcn1表达水平被证明与卵巢癌的患者生存期呈负相关(smith et al.,2014)。vtcn1可能是尿路上皮细胞癌和胃癌的一个潜在预后指标(shi et al.,2014b;fan et al.,2014)。
[0696]
vwa1与透明细胞卵巢癌有关(cicek et al.,2013)。
[0697]
vwa2被证明与结直肠癌有关(hoff et al.,2015)。vwa2被证明在ii、iii和iv期结肠癌、结肠腺瘤和结肠癌细胞系中被高度诱导。因此,vwa2是开发早期结肠癌血清诊断标志物的新候选基因(xin et al.,2005)。
[0698]
vwa3a被证明与卵巢癌生存相关(madden et al.,2014)。
[0699]
vwde在乳腺癌患者中突变,并显示出致癌性质(pongor et al.,2015)。
[0700]
wdfy3被证明在结直肠癌中下调(piepoli et al.,2012)。
[0701]
最近的研究观察到whsc1蛋白水平在几种类型的人类癌症中上升,如,胃肠道癌(食管癌、胃癌、结肠癌、肛管癌)、小细胞肺癌、前列腺癌和膀胱肿瘤、女性生殖道肿瘤和皮肤肿瘤。其他人已经报导,染色体易位造成的whsc1过度表达在异种移植物模型中显著影响多发性骨髓瘤细胞的致瘤性(lauring et al.,2008;hudlebusch et al.,2011;yang et al.,2012d)。前列腺癌细胞系中whsc1敲减导致软琼脂中细胞增殖、集落形成下降以及细胞迁移和侵袭下降。同样地,在头颈部细胞的鳞状细胞癌中,whsc1敲减导致显著生长抑制,诱导细胞凋亡和延迟细胞周期进程。此外,whsc1表达显示出可在多发性骨髓瘤中诱导细胞黏附、克隆源性生长和致瘤性(kassambara et al.,2009;ezponda et al.,2013;saloura et al.,2015)。
[0702]
wrn基因的单核苷酸多态性德国和澳大利亚人群的乳腺癌风险相关。其他人发现wrn基因的单核苷酸多态性与结直肠癌、前列腺癌和食管癌之间存在相关性。此外,在宫颈癌标本观察到了wrn异常甲基化(wirtenberger et al.,2006;wang et al.,2011;li et al.,2012d;masuda et al.,2012;sun et al.,2015d;zins et al.,2015)。另外,wrn基因的sirna 介导沉寂在体外抑制癌细胞生长(arai et al.,2011)。
[0703]
越来越多的证据表明,wt1基因在不同形式的肿瘤(包括急性骨髓性白血病、急性淋巴性白血病、肝细胞癌和头颈部鳞状细胞癌)中高表达(miwa et al.,1992;perugorria et al.,2009;li et al.,2015d)。此外,wt1过度表达是原发性高级别浆液性卵巢癌的总体生存率和无进展生存率的显著积极的预后因素。同样地,总生存率和无病生存率在wt1基因突变的急性粒细胞性白血病患者中显著降低。其他人也报告了wt1变体rs2234593与急性髓细胞白血病的复发和总生存之间存在相关性(niavarani et al.,2015;taube et al.,2016;toogeh et al.,2016)。
[0704]
一些研究人员在肝细胞癌、浆液性卵巢癌和乳腺癌中观察到了xdh低表达。但是,其他人报告了在血吸虫膀胱癌和非血吸虫膀胱癌,脑肿瘤,小细胞及非小细胞肺癌中xdh活性显著增加(kokoglu et al.,1990;stirpe et al.,2002;linder et al.,2005;kaynar et al.,2005;metwally et al.,2011;linder et al.,2012)。另外,根据报告,xdh下调与浆液性卵巢癌和乳腺癌患者的预后较差有关(linder et al.,2005;linder et al.,2012)。
[0705]
xpo4表达被肝细胞癌中的启动子甲基化下调,与肿瘤大小、病理组织学分级和患者生存预后显著较差相关(liang et al.,2011;zhang et al.,2014a)。pi3k的催化亚基突变形成高度活化的akt/mtor途径并下调肿瘤抑制基因pten、xpo4和dlc1(kudo et al.,2011)。
[0706]
ybx1被证明在不同类型癌症(包括结直肠癌、胃癌、多发性骨髓瘤和乳腺癌)中上调(bargou et al.,1997;chatterjee et al.,2008;wu et al.,2012g;yan et al.,2014b)。在乳腺癌中,ybx1过度表达与淋巴结状态和高病理分级均不相关,但与er阴性、her2阳性相关,这对5年总生存率产生不良影响(wang et al.,2015g)。研究人员证明,ybx1可透过调节上皮
‑
间质转变而促进结直肠癌细胞的增殖、凋亡、侵袭和迁移(yan et al.,2014c)。
[0707]
tut酶zcchc6被证明由肿瘤发生相关rna结合蛋白lin28募集,以阻止let
‑
7生物合成。透过tut酶抑制剂恢复癌症中let
‑
7表达可能在未来的药物发明中利用(lin and gregory,2015)。
[0708]
znf583被描述为结直肠癌的潜在生物标志物(mori et al.,2011)。
[0709]
znf700被证明是检测结直肠癌自身抗体的捕获抗原。在锌指蛋白的系列检查中,znf特异性自身抗体检测可发现到结直肠癌(o'reilly et al.,2015)。
[0710]
znfx1可作为一种新型的前列腺癌抗原(dunphy and mcneel,2005)。
[0711]
zranb2被证明在iii级卵巢浆液性乳头状癌中过度表达(schaner et al.,2003;mangs and morris,2008)。
[0712]
zwint被证明与cox
‑
2抑制后的前列腺癌细胞周期进程阻滞有关(bieniek et al.,2014)。淋巴结中慢性淋巴细胞白血病细胞的zwint表达被证明与临床结果相关(gilling et al.,2012)。zwint被描述为雄激素受体的靶基因,显示在去势抗性前列腺癌中上调(urbanucci et al.,2012)。zwint在肺腺癌预后模型中被描述为具有特别预测价值的基因(endoh et al.,2004)。
[0713]
zyg11a在非小细胞肺癌中作为致癌基因,并影响ccne1的表达(wang et al.,2016b)。
[0714]
zzef1被描述可能与癌症相关,位于髓母细胞瘤相关染色体区域(cvekl,jr.et al.,2004)。
[0715]
发明的详细说明
[0716]
是否能刺激免疫反应取决于是否存在被宿主免疫系统视为异物的抗原。发现肿瘤相关抗原的存在增加了运用宿主免疫系统干预肿瘤生长的可能性。目前,针对癌症免疫治疗,正在探索利用免疫系统的体液和细胞进行免疫的各种机制。
[0717]
细胞免疫反应的特定元素能特异性地识别和破坏肿瘤细胞。从肿瘤浸润细胞群或外周血中分离出的t
‑
细胞表明,这些细胞在癌症的天然免疫防御中发挥了重要作用。特别是cd8阳性t细胞在这种反应中发挥重要作用,tcd8
+
能识别通常8至10个源自蛋白或位于细胞质的缺损核糖体产物(drip)的氨基酸残基的主要组织兼容性复合体(mhc)所载的肽中所含的i类分子。人mhc分子也称为人白细胞
‑
抗原(hla)。
[0718]
除非另有说明,否则本文使用的所有术语定义如下。
[0719]
术语”t细胞反应”是指由一种肽在体外或体内诱导的效应子功能的特异性扩散和
启动。对于mhc i类限制性细胞毒性t细胞,效应子功能可能为溶解肽脉冲的、肽前体脉冲的或天然肽提呈的靶细胞、分泌细胞因子,优选为肽诱导的干扰素
‑
γ,tnf
‑
α或il
‑
2,分泌效应分子,优选为肽诱导的颗粒酶或穿孔素,或脱颗粒。
[0720]
本文所用”肽”这一术语,系指一系列氨基酸残基,通常透过相邻氨基酸的α
‑
氨基和羰基之间的肽键来连接。这些肽的长度优选为9个氨基酸,但至短可为8个氨基酸长度,至长可为10、11、12、13或14个氨基酸或更长,如果为mhc
‑
ii类肽时(本发明肽的拉长变体),至长可为14、15、16、17、18、19或20个氨基酸长度或更长。
[0721]
因此,”肽”这一术语应包括一系列氨基酸残基的盐,通常透过相邻氨基酸的α
‑
氨基和羰基之间的肽键来连接。优选的情况是,盐为肽的药用盐,例如:氯化物或乙酸(三氟乙酸)盐。必须注意的是,本发明肽的盐与其体内状态的肽基本上不同,因为该不是体内的盐。
[0722]
术语“肽“应也包括“寡肽“。本文使用的术语”寡肽”是指一系列氨基酸残基,通常透过相邻氨基酸的α
‑
氨基和羰基之间的肽键来连接。寡肽的长度对于本发明来说并不十分关键,只要在寡肽中保持正确的表位即可。通常,寡肽长度约小于30个氨基酸残基,约长于15个氨基酸。
[0723]“多肽”这一术语是指一系列氨基酸残基,通常透过相邻氨基酸的α
‑
氨基和羰基之间的肽键来连接。多肽的长度对于本发明来说并不十分关键,只要保持正确的表位即可。与术语肽或寡肽相对,“多肽”这一术语是指包含多于约30个氨基酸残基的分子。
[0724]
一种肽、寡肽、蛋白质或编码该分子的核苷酸如果能诱导免疫反应,则具有”免疫原性”(因此是本发明中的一种”免疫原”)。在本发明的情况下,免疫原性的更具体定义是诱导t细胞反应的能力。因此,”免疫原”是一种能够诱导免疫反应的分子,并且在本发明的情况下,是一种能诱导t细胞反应的分子。在另一方面,所述免疫原可以是肽,肽与mhc的复合体、和/或用于提高特异性抗体或tcr抗性的蛋白。
[0725]
i类t细胞”表位”要求的是一种结合至mhc i类受体上的短肽,从而形成一种三元复合体(mhc i类α链、β
‑2‑
微球蛋白和肽),其可以透过t细胞负载匹配t细胞受体与具有适当亲和力的mhc/肽复合物结合来识别。结合至mhc i类分子的肽的典型长度为8
‑
14个氨基酸,最典型为9个氨基酸长度。
[0726]
在人类中,有三种编码mhc i类分子的不同基因位点(人mhc分子也是指定的人白细胞抗原(hla)):hla
‑
a、hla
‑
b和hla
‑
c。hla
‑
a*01、hla
‑
a*02和hla
‑
b*07是可从这些基因位点表达的不同mhc i类等位基因的实例。
[0727]
表5:hla
‑
a*02和hla
‑
a*24和最常见hla
‑
dr血清类型的表达频率f。频率根据mori等人(mori et al.,1997)使用的hardy
‑
weinberg公式f=1
–
(1
‑
gf)2改编,从美国人群范围内的单体型频率中推导出。由于连锁不平衡,某些hla
‑
dr等位基因内的a*02或a*24组合与其预期单一频率相比,可能是浓缩的或频率较低。有关详细信息,请参阅chanock等人的文献(chanock et al.,2004)。
[0728]
[0729]
[0730][0731]
本发明的肽,优选当如本文描述纳入本发明的疫苗时与a*02。疫苗还可能包括泛结合mhc ii类肽。因此,本发明的疫苗可用于治疗a*02阳性患者中的癌症,但不因为这些肽的广泛结核性而必须选择ii类mhc同种异型。
[0732]
如果本发明的a*02肽与结合至另一等位基因例如a*24的肽组合,与单独的mhc i类等位基因相比,可治疗更高比例的患者群体。虽然在大多数人群中,低于50%的患者可由单独的等位基因来解决问题,但是本发明中一种含hla
‑
a*24和hla
‑
a*02表位的疫苗可以治疗任何相关人群中至少60%的患者。具体来说,各区域中,以下比例的患者这些等位基因中的至少一个有肯定效果:美国61%、西欧62%、中国75%、韩国77%、日本86%(根据www.allelefrequencies.net计算)。
[0733]
在一项优选的实施方案中,术语”核苷酸序列”系指脱氧核苷酸的杂聚物。
[0734]
编码特定肽、寡肽或多肽的核苷酸序列可为天然核苷酸序列,也可为合成核苷酸序列。一般来说,编码肽、多肽以及本发明蛋白的dna片段由cdna片段和短寡核苷酸衔接物,或一系列寡核苷酸组成,以提供一种合成基因,该基因能够在包含源自微生物或病毒操纵子的调节元素的重组转录单元中被表达。
[0735]
如本文所用的术语”肽的核苷酸编码”系指对肽进行核苷酸序列编码,其中该肽包括与将由用于产生tcr的树突细胞或另一细胞系统所表达该序列的生物系统兼容的人工(人造)启动和停止密码子。
[0736]
本文提到的核酸序列既包括单链核酸也包括双链核酸。因此,除非本文另有所指,否则,例如对于dna,具体的序列是该序列的单链dna、该序列与其互补序列的双工(双链dna)以及该序列的互补序列。
[0737]“编码区”这一术语是指在基因的天然基因组环境中天然或正常编码该基因的表达产物的那部分基因,即,体内编码该基因的天然表达产物的区域。
[0738]
编码区可来自非突变(”正常”)基因、突变基因或异常基因,甚至还可以来自dna序列,完全可在实验室中使用本领域熟知的dna合成方法合成。
[0739]“表达产物”这一术语是指多肽或蛋白,它是基因和遗传码退化并因而编码同样的氨基酸所造成的任何核酸序列编码同等物的翻译产物。
[0740]“片断”这一术语,当指的是一种编码序列时,表示包含非完整编码区的dna的一部分,其表达产物与完整编码区表达产物基本上具有相同的生物学功能或活性。
[0741]“dna片段”这一术语是指一种dna聚合物,以单独的片段形式或一种较大dna结构的组分形式存在,它们从至少分离过一次的dna中以基本纯净的形式获得,即不含污染性内
源性材料,并且获得的数量或浓度能够使用标准生化方法,例如使用克隆载体,进行识别、操纵和回收该片段及其组分核苷酸序列。此类片段以开放阅读框架(未被内部未翻译序列打断)或内含子(通常提呈于真核基因中)的形式存在。未翻译dna序列可能存在于开放阅读框架的下游,在那里其不会干预编码区的操纵或表达。
[0742]“引物”这一术语表示一种短核酸序列,其可与一个dna链配对,并在dna聚合酶开始合成脱氧核糖核酸链之处提供一个游离的3'
‑
oh末端。
[0743]“启动子”这一术语表示参与rna聚合酶的结合从而启动转录的dna区域。
[0744]
术语”分离”表示一种物质从其原来的环境(例如,如果是天然发生的则是天然环境)中被移走。例如,活体动物中的天然核苷酸或多肽不是分离的,但是,从天然系统中一些或所有共存物质中分离出来的核苷酸或多肽是分离的。此类多核苷酸可能是载体的一部分和/或此类多核苷酸和多肽可能是一种组合物的一部分,并且由于该载体或组合物不是其天然环境的一部分,因此它仍然是分离的。
[0745]
本发明中披露的多核苷酸和重组或免疫原性多肽也可能以”纯化”的形式存在。术语”纯化”并非要求绝对的纯度;它只是一个相对的定义,可以包括高度纯化或部分纯化的制剂,相关领域技术人员能理解这些术语。例如,各个从已用传统方法纯化为具有电泳同构型的cdna库中分离出的各种克隆物。明确考虑到将起始材料或天然物质纯化至少一个数量级,优选为两或三个数量级,更优选为四或五个数量级。此外,明确涵盖所述多肽的纯度优选为99.999%,或至少为99.99%或99.9%;甚而适宜为以重量计99%或更高。
[0746]
根据本发明公开的核酸和多肽表达产物,以及包含此类核酸和/或多肽的表达载体可能以”浓缩的形式”存在。本文使用的术语”浓缩”是指材料的浓度至少是其自然浓度的大约2、5、10、100或1000倍,有优势的是,按重量计为0.01%,优选为至少0.1%。也明确考虑到,按重量计约为0.5%、1%、5%、10%和20%的浓缩制剂。序列、构型、载体、克隆物以及包含本发明的其他材料可有优势地以浓缩或分离的形式存在。”活性片段”这一术语是指产生免疫反应的片段(即具有免疫原性活性),通常是一种肽、多肽或核酸序列的片段,不论是单独或可选地与合适的佐剂一起或在载体中给予一种动物,比如哺乳动物,例如兔子或小鼠,也包括人;这种免疫反应采用的形式是在接受动物(如:人)体内刺激t细胞反应。或者,”活性片段”也可用于诱导体外t细胞反应。
[0747]
本文使用的”部分”(portion)、”节段”(segment)、”片段”(fragment)这几个术语,当与多肽相关地使用时是指残基的连续序列,比如氨基酸残基,其序列形成一个较大序列的子集。例如,如果一个多肽以任一种肽链内切肽酶(如胰蛋白酶或糜蛋白酶)进行处理,则该处理获得的寡肽会代表起始多肽的部分、节段或片段。当与多核苷酸相关地使用时,这些术语系指用任何核酸内切酶处理所述多核苷酸产生的产物。
[0748]
根据本发明,术语”等同度百分比”或”等同百分比”,如果指的是序列,则表示在待对比序列(”被对比序列”)与所述序列或权利要求的序列(”参考序列”)对准之后将被对比序列与所述序列或权利要求的序列进行比较。然后根据下列公式计算等同度百分比:
[0749]
等同度百分比=100[1
‑
(c/r)]
[0750]
其中c是参考序列与被对比序列之间对准长度上参考序列与被对比序列之间的差异数量,其中
[0751]
(i)参考序列中每个碱基或氨基酸序列在被对比序列中没有对应的对准碱基或氨
基酸;
[0752]
(ii)参考序列中每个空隙,以及
[0753]
(iii)参考序列中每个对准碱基或氨基酸与被比对比序列中对准碱基或氨基酸不同,即构成一个差异以及
[0754]
(iiii)必须在对准序列的第1位置开始对准;
[0755]
并且r是参考序列与被对比序列对准长度上在参考序列中产生任何空隙也计算为一个碱基或氨基酸的参考序列中的碱基或氨基酸数目。
[0756]
如果”被对比序列”和”参考序列”之间存在的一个对准按上述计算的等同度百分比大致等于或大于指定的最低等同度百分比,则被对比序列与参考序列具有指定的最低等同度百分比,虽然可能存在按本文上述计算的等同度百分比低于指定等同度百分比的对准。
[0757]
因此,如上所述,本发明提出了一种肽,其包括选自seq id no:1至seq id no:640群组的一个序列、或与seq id no:1至seq id no:640具有88%同源性的其变体、或诱导与该肽发生t细胞交叉反应的一个变体。本发明所述的肽具有与主要组织兼容性复合体(mhc)i或所述肽拉长版本的ii类分子结合的能力。
[0758]
在本发明中,”同源性”一词系指两个氨基酸序列之间的同一度(参见上文的等同度百分比,如肽或多肽序列。前文所述的”同源”是透过将理想条件下调整的两个序列与待比较序列进行比对后确定的。此类序列同源性可透过使用clustalw等算法创建一个排列而进行计算。也可用使用一般序列分析软件,更具体地说,是vector nti、genetyx或由公共数据库提供的其他工具。
[0759]
本领域技术人员能评估特定肽变体诱导的t细胞是否可与该肽本身发生交叉反应(appay et al.,2006;colombetti et al.,2006;fong et al.,2001;zaremba et al.,1997)。
[0760]
发明人用给定氨基酸序列的“变体“表示,一个或两个氨基酸残基等的侧链透过被另一个天然氨基酸残基的侧链或其他侧链取代而发生改变,这样,这种肽仍然能够以含有给定氨基酸序列(由seq id no:1至seq id no:640组成)的肽大致同样的方式与hla分子结合。例如,一种肽可能被修饰以便至少维持(如没有提高)其能与hla
‑
a*02或
‑
dr等合适mhc分子的结合槽相互作用和结合,以及至少维持(如没有提高)其与启动t细胞的tcr结合的能力。
[0761]
随后,这些t细胞可与细胞和杀伤细胞发生交叉反应,这些细胞表达多肽(其中包含本发明中定义的同源肽的天然氨基酸序列)。正如科学文献和数据库(rammensee et al.,1999;godkin et al.,1997)中所述,hla
‑
a结合肽的某些位点通常为锚定残基,可形成一种与hla结合槽的结合模序相称的核心序列,其定义由构成结合槽的多肽链的极性、电物理、疏水性和空间特性确定。因此,本领域技术人员能够透过保持已知的锚残基来修饰seq id no:1至seq id no:640提出的氨基酸序列,并且能确定这些变体是否保持与mhc i或ii类分子结合的能力。本发明的变体保持与启动t细胞的tcr结合的能力,随后,这些t细胞可与表达一种包含本发明定义的同源肽的天然氨基酸序列的多肽的细胞发生交叉反应并杀死该等细胞。
[0762]
如果无另有说明,那么本文公开的原始(未修饰)肽可以透过在肽链内的不同(可
能为选择性)位点上取代一个或多个残基而被修饰。优选情况是,这些取代位于氨基酸链的末端。此取代可能是保守性的,例如,其中一个氨基酸被具有类似结构和特点的另一个氨基酸所取代,比如其中一个疏水性氨基酸被另一个疏水性氨基酸取代。更保守的取代是具有相同或类似的大小和化学性质的氨基酸间的取代,例如,亮氨酸被异亮氨酸取代。在天然同源蛋白质家族序列变异的研究中,某些氨基酸的取代往往比其他氨基酸更具有耐受性,这些氨基酸往往表现出与原氨基酸的大小、电荷、极性和疏水性之间的相似性相关,这是确定”保守取代”的基础。
[0763]
在本文中,保守取代定义为在以下五种基团之一的内部进行交换:基团1—小脂肪族、非极性或略具极性的残基(ala,ser,thr,pro,gly);基团2—极性、带负电荷的残基及其酰胺(asp,asn,glu,gln);基团3—极性、带正电荷的残基(his,arg,lys);基团4—大脂肪族非极性残基(met,leu,ile,val,cys)以及基团5—大芳香残基(phe,tyr,trp)。
[0764]
较不保守的取代可能涉及一个氨基酸被另一个具有类似特点但在大小上有所不同的氨基酸所取代,如:丙氨酸被异亮氨酸残基取代。高度不保守的取代可能涉及一个酸性氨基酸被另一个具有极性或甚至具有碱性性质的氨基酸所取代。然而,这种”激进”取代不能认为是无效的而不予考虑,因为化学作用是不完全可预测的,激进的取代可能会带来其简单化学原理中无法预见的偶然效果。
[0765]
当然,这种取代可能涉及普通l
‑
氨基酸之外的其他结构。因此,d
‑
氨基酸可能被本发明的抗原肽中常见的l
‑
氨基酸取代,也仍在本公开的范围之内。此外,非标准氨基酸(即,除了常见的天然蛋白原氨基酸)也可以用于取代之目的,以生产根据本发明的免疫原和免疫原性多肽。
[0766]
如果在一个以上位置上的取代发现导致肽的抗原活性基本上等于或大于以下定义值,则对这些取代的组合进行测试,以确定组合的取代是否产生对肽抗原性的迭加或协同效应。肽内被同时取代的位置最多不能超过4个。
[0767]
基本上由本文所指氨基酸序列组成的一种肽可能有一个或两个非锚定氨基酸(见下面锚基序相关内容)被交换,而不存在这种情况,即相比于未修饰的肽,与人类主要组织兼容性复合体(mhc)
–
i或ii类分子的能力基本上被改变或受到不利影响。在另一实施方案中,在基本上由本文所述氨基酸序列组成的肽中,一个或两个氨基酸可与其保守交换伙伴交换(见下文),而不存在这种情况,即相比于未修饰的肽,与人类主要组织兼容性复合体(mhc)
–
i或ii类分子的能力基本上被改变或受到不利影响。
[0768]
这些基本不与t细胞受体互动的氨基酸残基可透过取代其他几乎不影响t细胞反应并不妨碍与相关mhc结合的氨基酸而得到修饰。因此,除了特定限制性条件外,本发明的肽可能为任何包括给定氨基酸序列或部分或其变体的肽(发明人所用的这个术语包括寡肽或多肽)。
[0769]
表6:根据seq id no:20、40和217的肽的变体和基序
[0770]
[0771]
[0772][0773]
较长(拉长)的肽也可能适合。mhc i类表位(通常长度为8至11个氨基酸)可能由肽从较长的肽或包含实际表位的蛋白中加工而产生。两侧有实际表位的残基优选为在加工过程中几乎不影响暴露实际表位所需蛋白裂解的残基。
[0774]
本发明的肽可被拉长多达四个氨基酸,即1、2、3或4个氨基酸,可按照4∶0与0∶4之间的任何组合添加至任意一端。本发明的拉长组合可见表8。
[0775]
表7:本发明肽的拉长组合
[0776]
c
‑
端n
‑
端4030或120或1或210或1或2或300或1或2或3或4n
‑
端c
‑
端4030或120或1或210或1或2或300或1或2或3或4
[0777]
拉伸/延长的氨基酸可以是所述蛋白或任何其他氨基酸的原序列肽。拉长可用于增强所述肽的稳定性或溶解性。
[0778]
因此,本发明所述的表位可能与天然肿瘤相关表位或肿瘤特异性表位相同,也可能包括来自参考肽的不超过四个残基的不同肽,只要它们有基本相同的抗原活性即可。
[0779]
在一项替代实施方案中,肽的一边或双边被拉长4个以上的氨基酸,优选最多30个氨基酸的总长度。这可形成mhc
‑
ii类结合肽。结合至mhc ii类肽可透过本领域中已知的方法进行测试。
[0780]
因此,本发明提出了mhc i类表位的肽和变体,其中所述肽或抗体的总长度为8至100个、优选为8至30个、最优选为8至14个氨基酸长度(即10、11、12、13、14个氨基酸,如果为拉长ii类结合肽时,长度也可为15、16、17、18、19、20、21或22个氨基酸)。
[0781]
当然,本发明的肽或变体能与人主要组织兼容性复合体(mhc)i或ii类分子结合。肽或变体与mhc复合物的结合可用本领域内的已知方法进行测试。
[0782]
优选情况是,当本发明的肽特异性t细胞相比于取代肽受到检测时,如果取代肽在相对于背景肽溶解度增加达到最大值的一半,则该肽浓度不超过约1mm,优选为不超过约1μm,更优选为不超过约1nm,再优选为不超过约100pm,最优选为不超过约10pm。也优选为,取代肽被一个以上的t细胞识别,最少为2个,更优选为3个。
[0783]
在本发明的一个特别优选实施方案中,肽系由或基本系由根据seq id no:1至seq id no:640所选的氨基酸序列组成。
[0784]
基本由“...组成“系指本发明的肽,除了根据seq id no:1至seq id no:640中的任一序列或其变体组成外,还含有位于其他n和/或c端延伸处的氨基酸,而它们不一定能形成作为mhc分子表位的肽。
[0785]
但这些延伸区域对有效将本发明中的肽引进细胞具有重要作用。在本发明的一实施例中,该肽为融合蛋白的一部分,含来自ncbi、genbank登录号x00497的hla
‑
dr抗原相关不变链(p33,以下称为”ii”)的80个n
‑
端氨基酸等。在其他的融合中,本发明的肽可以被融合到本文所述的抗体、或其功能性部分,特别是融合入抗体的序列,以便所述抗体进行特异性靶向作用,或者,例如进入本文所述的树突状细胞特异性抗体。
[0786]
此外,该肽或变体可进一步修饰以提高稳定性和/或与mhc分子结合,从而引发更强的免疫反应。肽序列的该类优化方法是本领域内所熟知的,包括,例如,反式肽键和非肽键的引入。
[0787]
在反式肽键氨基酸中,肽(
‑
co
‑
nh
‑
)并未连接其残基,但是其肽键是反向的。这种逆向反向模拟肽(retro
‑
inverso peptidomimetics)可透过本领域已知的方法制备,例如:meziere等人在(meziere et al.,1997)中所述的方法,以引用的方式并入本文。这种方法涉及制备包含骨架(而并非侧链)改变的模拟肽。meziere等人(meziere et al.,1997)的研究显示,这些模拟肽有利于mhc的结合和辅助性t细胞的反应。以nh
‑
co键替代co
‑
nh肽键的逆向反向肽大大地提高了抗水解性能。
[0788]
非肽键为
‑
ch2‑
nh、
‑
ch2s
‑
、
‑
ch2ch2‑
、
‑
ch=ch
‑
、
‑
coch2‑
、
‑
ch(oh)ch2‑
和
‑
ch2so
‑
等。美国4897445号专利提出了多肽链中非肽键(
‑
ch2‑
nh)的非固相合成法,该方法涉及按标准程序合成的多肽以及透过氨基醛和一种含nacnbh3的氨基酸相互作用而合成的非肽键。
[0789]
含上述序列的肽可与其氨基和/或羧基末端的其他化学基团进行合成,从而提高肽的稳定性、生物利用度、和/或亲和力等。例如,苄氧羰基、丹酰基等疏水基团或叔丁氧羰基团可加入肽的氨基末端。同样,乙酰基或9
‑
芴甲氧羰基可能位于肽的氨基末端。此外,疏水基团、叔丁氧羰基团或氨基团都可能被加入肽的羧基末端。
[0790]
另外,本发明中的所有肽都可能经合成而改变其空间构型。例如,可能使用这些肽的一个或多个氨基酸残基的右旋体,通常不是其左旋体。更进一步地,本发明中肽的至少一个氨基酸残基可被熟知的一个非天然氨基酸残基取代。诸如此类的改变可能有助于增加本发明肽的稳定性、生物利用度和/或结合作用。
[0791]
同样,本发明中的肽或变体可在合成肽之前或之后透过特异氨基酸的反应而进行化学修饰。此类修饰的实施例为本领域所熟知,例如,在r.lundblad所著的《chemical reagents for protein modification》(3rd ed.crc press,2004)(lundblad,2004)中有概述,以参考文献的方式并入本文。虽然氨基酸的化学修饰方法无限制,但其包括(但不限于)透过以下方法修饰:酰基化、脒基化、赖氨酸吡哆基化、还原烷基化、以2,4,6
‑
三硝基苯磺酸(tnbs)三硝基苯基化氨基团、透过将半胱氨酸过甲酸氧化为磺基丙氨酸而对羧基团和巯基进行氨基修饰、形成易变衍生物、与其他巯基化合物形成混合二硫化合物、与马来酰亚胺反应,与碘乙酸或碘乙酰胺羧甲基化、在碱性ph值下与氰酸盐甲氨酰化。在这方面,技术
al.,2004)以及本文引用的参考文献)
[0798]
三氟乙酸用真空中蒸发、随后用承载粗肽的二乙基乙醚滴定进行去除。用简单萃取程序(水相冻干后,该程序制得不含清道夫混合物的肽)清除任何存在的清道夫混合物。肽合成试剂一般可从calbiochem
‑
novabiochem(英国诺丁汉)获得。
[0799]
纯化可透过以下技术的任何一种或组合方法进行,如:再结晶法、体积排阻色谱法、离子交换色谱法、疏水作用色谱法以及(通常)反相高效液相色谱法(如使用乙腈/水梯度分离)。
[0800]
可以使用薄层色谱法、电泳特别是毛细管电泳、固相萃取(cspe)、反相高效液相色谱法、酸解后的氨基酸分析、快原子轰击(fab)质谱分析以及maldi和esi
‑
q
‑
tof质谱分析进行肽分析。
[0801]
为了选择过度提呈的肽,计算了提呈图,其显示样本中位提呈量以及复制变化。该特点使相关肿瘤实体的样本与正常组织样本的基线值并列。可透过计算调节线性混合效应模型(pinheiro et al.,2015)的p值将以上每个特点并入过度提呈分数中,从而透过假发现率(benjamini and hochberg,1995)调整多项检验。
[0802]
对于透过质谱法对hla配体的识别和相对定量,对来自冲击冷冻组织样本的hla分子进行纯化并对hla相关肽进行分离。分离的肽分开,并透过在线纳米
‑
电喷雾
‑
电离(nanoesi)液相色谱
‑
谱(lc
‑
ms)实验进行鉴定。由此产生的肽序列的验证方法是,将卵巢癌样本(n=20个a*02阳性样本)中记录的自然肿瘤相关肽(tumap)的片段模式与相同序列相应合成参考肽的片段模式进行比较。由于这些肽被直接鉴定为原发肿瘤hla分子的配体,因此这些结果为来自20名卵巢癌患者的原发性癌组织上确定肽的自然加工和提呈提供了直接证据。
[0803]
发现管道v2.1(例如,参见us 2013
‑
0096016,并在此透过引用将其整体并入本文)考虑到识别和选择相关过量提呈的候选肽疫苗,这基于与几种不同的非癌组织和器官相比癌症或其他受感染组织的hla限制肽水平直接相对定量结果。这透过以下方法实现:使用专有数据分析管道处理的lc
‑
ms采集数据、结合序列识别算法、谱聚类、计算离子、保留时间调整、充电状态卷积以及正态化而开发无标记差异化定量方法。
[0804]
为每种肽和样本确立了提呈水平,包括误差估计值。肿瘤组织大量提呈的肽以及肿瘤与非肿瘤组织和器官中过量提呈的肽已经得到确定。
[0805]
对来自卵巢癌组织样本的hla肽复合物进行纯化,并且对hla相关肽使用lc
‑
ms进行分离和分析(见实施例)。本技术中包含的所有tumap使用原发性卵巢癌样本的方法进行鉴定,确认其在原发性卵巢癌上的提呈。
[0806]
在多个卵巢癌和正常组织上确定的tumap用无标记lc
‑
ms数据的离子计数方法进行量化。该方法假定肽的lc
‑
ms信号区域与样本中其丰度相关。各种lc
‑
ms实验中肽的所有量化信号在集中趋势基础上进行正常化,根据每个样品进行平均,并合并入柱状图(被称为提呈图)。提呈图整合了不同分析方法,如:蛋白数据库检索、谱聚类、充电状态卷积(除电)和保留时间校准和正态化。
[0807]
此外,发现管道v2.x可对癌症或其他感染组织上的mhc
‑
肽(优选为hla限制性肽)进行直接的绝对定量。简言之,总细胞计数根据被分析的组织样本的总dna含量来计算。组织样本中tumap的总肽量用nanolc
‑
ms/ms测定为天然tumap的比率以及
tumap同位素标记版本的已知量,称为内部标准。tumap分离效率确定方法:把肽:所有选定tumap的mhc在tumap分离程序尽早的时间点加入组织裂解液,并在肽分离成后完成后透过nanolc
‑
ms/ms检测。总细胞计数和总肽量根据每份组织样本三次测量值来计算。所述肽特异性隔离效率计算为三次测量10次加标实验的平均值(见实施例6和表12)。
[0808]
除了过量提呈肽之外,也分析了潜在基因的mrna表达。mrna数据透过rna测序分析正常组织和癌组织获得(见实施例2)。正常组织数据的额外来源是从3000个正常组织样本中公开获得的rna表达数据的数据库(lonsdale,2013)。获得自蛋白的肽在癌组织中显示高表达编码mrna,但是在重要健康(正常)组织中非常低或不存在,这些肽作为优选肽纳入本发明。
[0809]
本发明提出了有利于治疗癌肿/肿瘤,优选为治疗过量提呈或只提呈本发明肽的卵巢癌。这些肽由质谱分析法直接显示出,而由hla分子自然提呈于原发性人卵巢癌样本中。
[0810]
与正常组织相比,癌症中高度过量表达肽来源的许多源基因/蛋白质(也指定为“全长蛋白“或“潜在蛋白“)
‑
本发明相关的“正常组织“是健康卵巢或其他正常组织,这表明肿瘤与这些源基因的高度关联性(见实施例2)。此外,这些肽本身也在肿瘤组织中过度提呈(本发明相关的“肿瘤组织“是指来自卵巢癌患者的样本),但不在正常组织中过度提呈(见实施例1)。
[0811]
hla结合肽能够被免疫系统识别,特别是t淋巴细胞。t细胞可破坏提呈被识别hla/肽复合体的细胞(如:提呈衍生肽的卵巢癌细胞)。
[0812]
本发明的所有肽已被证明具有刺激t细胞反应的能力,并过量提呈,因而可用于制备本发明的抗体和/或tcr,例如可溶性tcr(参见实施例3和实施例4)。此外,肽与相应的mhc组合时,也可用于制备本发明的抗体和/或tcr,特别是stcr。各个方法均为技术人员所熟知,并在各个文献中可找到。因此,本发明的肽可用于在患者中产生免疫反应,从而能够毁灭肿瘤细胞。患者的免疫反应能够透过直接给予患者所述肽或前体物质(如,加长肽、蛋白或编码这些肽的核酸),较理想是与加强免疫原性的制剂相结合,而进行诱导。源自该治疗性疫苗的免疫反应预期能够高度特异性地对抗肿瘤细胞,因为本发明的目标肽在正常组织上提呈的复制数目较少,防止患者发生对抗正常细胞的不良自体免疫反应的风险。
[0813]
本说明书还涉及包含一个α链和一个β链(“α/βtcr”)的t细胞受体(tcr)。还提供了由mhc分子提呈时可与tcr和抗体结合的havcr1
‑
001肽。本说明书还涉及核酸、载体和用于表达tcr的宿主细胞和本说明书的肽;以及使用它们的方法。
[0814]
术语“t细胞受体”(缩写tcr)是指一种异二聚体分子,其包含一个α多肽链(α链)和一个β多肽链(β链),其中所述异二聚体受体能够结合由hla分子提呈的肽抗原。该术语还包括所谓的γ/δtcr。
[0815]
在一个实施方案中,本说明书提供了如本文中所描述的产生tcr的方法,该方法包括在适于促进tcr表达的条件下培养能够表达tcr的宿主细胞。
[0816]
另一个方面,本说明书涉及一种根据本说明书的方法,其中所述抗原透过与足够量的含抗原提成细胞的抗原结合被载入表达于合适抗原提呈细胞或人工抗原呈递细胞表面的i或ii类mhc分子,或该抗原透过四聚化被加载i或ii类mhc四聚体/i或ii类mhc复合单体。
[0817]
α/βtcr的α和β链和γ/δtcr的γ和δ链通常被视为各自有两个”结构域”,即可变和恒定结构域。可变结构域由可变区(v)和连接区(j)的组合。可变结构域还可能包括一个前导区(l)。β和δ链还可能包括一个多样区(d)。α和β恒定结构域还可能包括锚定α和β链至细胞膜的c末端跨膜(tm)结构域。
[0818]
相对于γ/δ的tcr,如本文所用的术语“tcrγ可变域”是指无前导区(l)的tcrγv(trgv)区与tcrγ(trgj)区的组合,术语tcrγ恒定结构域是指细胞外trgc区域,或c
‑
末端截短trgc序列。同样地,”tcrδ可变域”是指无前导区(l)的tcrδv(trdv)区与tcrδd/j(trdd/trdj)区的组合,术语“tcrδ恒定结构域”是指细胞外trdc区域,或c
‑
末端截短trdc序列。
[0819]
本说明书的tcr优选结合至havcr1
‑
001肽hla分子复合体,其具有约100μm或更小、约50μm或更小、约25μm或更小或约10μm或更小的结合亲和力(kd)。更为优选的情况是具有约1μm或更小、约100nm或更小、约50nm或更小或约25nm或更小结合亲和力的高亲和力tcr。本发明tcr优选结合亲和力范围的非限制性示例包括约1nm至约10nm;约10nm至约20nm;约20nm至约30nm;约30nm至约40nm;约40nm至约50nm;约50nm至约60nm;约60nm至约70nm;约70nm至约80nm;约80nm至约90nm;以及约90nm至约100nm。
[0820]
与本说明书tcr相关,本文使用的”特异性结合”及其语法变体用于表示对100μm或更小的havcr1
‑
001肽
‑
hla分子复合体有结合亲和力(kd)的tcr。
[0821]
本说明书的α/β异二聚体tcr可能具有其恒定结构域之间的引入二硫键。这种类型的优选tcr包括那些具有一个trac恒定域序列和trbc1或trbc2恒定域序列的tcr,除非trac的苏氨酸48和trbc1或trbc2的丝氨酸57被半胱氨酸残基取代,所述半胱氨酸形成trac恒定域序列和tcr的trbc1或trbc2恒定区序列之间的二硫键。
[0822]
不论具有或不具有上述的引入链间键,本说明书的α/β杂二聚体tcr可能具有一个trac恒定域序列和一个trbc1或trbc2恒定结构域序列,并且trac恒定结构域序列和tcr的trbc1或trbc2恒定结构域序列可能透过trac外显子2的cys4和trbc1或trbc2外显子2的cys4之间的天然二硫键相连。
[0823]
本说明书的tcr可能包括选自由放射性核素、荧光团和生物素组成组中的可检测标记。本说明书的tcr可能共轭至治疗活性剂,如放射性核素、化学治疗剂或毒素。
[0824]
在一个实施方案中,具有在α链中至少一个突变和/或具有在β链中至少一个突变的tcr与未突变的tcr相比,已经修改了糖基化。
[0825]
在一个实施方案中,在tcrα链和/或tcrβ链中包括至少一个突变的tcr对havcr1
‑
001肽hla分子复合体有结合亲和力和/或结合半衰期,其是包含未突变tcrα链和/或未突变tcrβ链的tcr的结合亲和力的至少两倍。肿瘤特异性tcr亲和力增强及其开发依赖于存在最佳tcr亲和力的窗口。这样窗口的存在是根据观察结果:hla
‑
a2限制性病原体特异性tcr与hla
‑
a2限制性肿瘤相关自身抗原特异性tcr相比,kd值通常大约低10倍。现已知,尽管肿瘤抗原可能具有免疫原性,但是因为肿瘤来自个体自身的细胞,因此仅突变蛋白质或翻译加工改变的蛋白将被免疫系统视为外来物质。上调或过度表达(所谓的自体抗原)的抗原不一定诱导针对肿瘤的功能免疫应答:表达对这些抗原具有高度反应性的tcr的t细胞会在一种称为中枢耐受的程序中在胸腺内被不利选择,也就是说只有对自身抗原具有低亲和力tcr的细胞才仍然存在。因此,本说明书的tcr或变体对havcr1
‑
001的亲和力可透过本领域熟知的方法来增强。
[0826]
本说明书还涉及一种识别和分离本发明tcr的一种方法,所述方法包括:用a2/havcr1
‑
001肽单体从hla
‑
a*02阴性健康供体孵育pbmc,用四聚体
‑
藻红蛋白(pe)孵育pbmc并透过荧光启动细胞分选(facs)
–
calibur方法分析分离高亲和力t细胞。
[0827]
本说明书还涉及一种识别和分离本发明tcr的一种方法,所述方法包括:获得含整个人体tcrαβ基因位点(1.1and 0.7mb)的转基因小鼠(其t细胞表达多样化人类tcr,用于补偿小鼠tcr缺乏),用havcr1
‑
001对小鼠进行免疫处理,用四聚体
‑
藻红蛋白(pe)孵育从转基因小鼠中获得的pbmc,并透过荧光启动细胞分选(facs)
–
calibur方法分析分离高亲和力t细胞。
[0828]
一方面,为了获得表达本说明书tcr的t细胞,编码本说明书tcr
‑
α和/或tcr
‑
β链的核酸被克隆入表达载体,诸如γ反转录病毒或慢病毒。重组病毒产生,然后测试功能,如抗原专一性和功能性亲合力。然后,最终产品的等分试样被用于转导靶t细胞群体(一般纯化自患者的pbmc),在输入患者前展开。另一方面,为了获得表达本说明书tcr的t细胞,tcr rna透过本领域中已知的技术(例如,体外转录系统)合成。然后,体外合成的tcr rna透过电穿孔来重新表达肿瘤特异性tcr
‑
α和/或tcr
‑
β链被引入获得自健康供体的初级cd8+ t细胞。
[0829]
为了增加表达,编码本说明书tcr的核酸在操作上可连接到强启动子,例如逆转录病毒长末端重复序列(ltr)、巨细胞病毒(cmv)、鼠干细胞病毒(mscv)u3、磷酸甘油酸激酶(pgk)、β肌动蛋白、泛素蛋白和猿猴病毒40(sv40)/cd43复合启动子、延伸因子(ef)
‑
1a和脾脏病灶形成病毒(sffv)启动子。在一优选实施方案中,启动子与被表达的核酸异源。除了强启动子外,本说明书的tcr表达盒可能含有附加的元素,可提高转基因表达,包括中枢多聚嘌呤区(cppt),其促进了慢病毒构建体的核易位(follenzi et al.,2000),和土拨鼠肝炎病毒转录后调控元素(wpre),其透过提高rna稳定性增加转基因表达水平(zufferey et al.,1999)。
[0830]
本发明tcr的α和β链可由位于分开的载体核酸进行编码,或者可透过位于同一载体的多核苷酸编码。
[0831]
实现高水平的tcr表面表达需要引入tcr的tcr
‑
α和tcr
‑
β链高水平转录。为了实现它,本说明书的tcr
‑
α和tcr
‑
β链可在单一的载体中被克隆入双顺反子构建体,其已被证明能够克服这一障碍。使用tcr
‑
α和tcr
‑
β链在之间的病毒核糖体间进入位点(ires)导致两链的协同表达,因为tcr
‑
α和tcr
‑
β链均由在翻译过程中分成两个蛋白质的单一转录物产生,从而确保了产生tcr
‑
α和tcr
‑
β链的相等摩尔比。(schmitt et al.2009)。
[0832]
编码本说明书tcr的核酸可以是被优化以从宿主细胞增加表达的密码子。遗传密码冗余让一些氨基酸被一个以上的密码子编码,但某些密码子没有其他密码子”优化”,因为匹配trna以及其他因子的相对可用性(gustafsson et al.,2004)。修改tcr
‑
α和tcr
‑
β基因序列使得每个氨基酸被用于哺乳动物基因表达的最佳密码子编码,以及消除mrna不稳定性基序或隐蔽剪接位点,已显示可显著提高tcr
‑
α和tcr
‑
β基因表达(scholten et al.,2006)。
[0833]
此外,引入的和内源性tcr链之间的错配可能会导致获得特异性,其构成自身免疫的显著风险。例如,混合tcr二聚体的形成可能会减少可用以形成正确配对tcr复合体的cd3分子数目,因此,可以显著降低表达所引入tcr的细胞的功能性亲合力(kuball et al.,
2007)。
[0834]
为了减少错配,本说明书引入的tcr链的c
‑
末端结构域可以进行修改以促进链间亲和力,同时降低引入链与内源tcr配对的能力。这些策略可能包括用鼠配对物取代人类tcr
‑
α和tcr
‑
βc端结构域(鼠化c端结构域);透过引入第二个半胱氨酸残基到引入tcr的tcr
‑
α和tcr
‑
β链产生c末端结构域的第二个链间二硫键(半胱氨酸修饰);交换tcr
‑
α和tcr
‑
β链c端结构域的相互作用残基(”杵臼结构”);直接融合tcr
‑
α和tcr
‑
β链可变结构域至cd3ζ(cd3ζ融合)(schmitt et al.2009)。
[0835]
在一实施方案中,宿主细胞被改变结构以表达本说明书的tcr。在一优选实施方案中,宿主细胞为人t细胞或t细胞祖细胞。在一些实施方案中,t细胞或t细胞祖细胞从癌症患者中获得。在另一些实施方案中,t细胞或t细胞祖细胞从健康供体中获得。本说明书的宿主细胞相对于待治疗的患者可以为同种异体或自体的。在一实施方案中,宿主是被转化以表达α/βtcr的γ/δt细胞。
[0836]“药物组合物“是指适合在医疗机构用于人体的组合物。优选地,药物组合物为无菌状态,并根据gmp指南生产。
[0837]
药物组合物包括游离形式或以一种药用盐形式存在的肽(也参见上文)。此处使用的”药用盐”系指所公开的肽的一种衍生物,其中该肽由制酸或药剂的碱盐进行改性。例如,用与适合的酸反应的游离碱(通常其中的中性药物有一个中性
–
nh2基团)制备酸式盐。适合制备酸盐的酸包括有机酸,如:乙酸、丙酸、羟基酸、丙酮酸、草酸、苹果酸、丙二酸、丁二酸、马来酸、富马酸、酒石酸、柠檬酸、苯甲酸酸、肉桂酸、扁桃酸、甲磺酸、甲磺酸、苯磺酸、水杨酸等等、以及无机酸,如:盐酸、氢溴酸、硫酸、硝酸和磷酸等。相反,可在一种肽上提呈的酸性基团的碱盐制剂使用药用碱基进行制备,如氢氧化钠、氢氧化钾、氢氧化铵、氢氧化钙、三甲胺等等。
[0838]
在特别优选的实施方案中,药物组合物包括乙酸(醋酸盐),三氟乙酸盐或盐酸(氯化物)形式的肽。
[0839]
本发明中所述的药剂优选为一种免疫治疗药剂,例如,一种疫苗。该疫苗可直接给到患者的受影响器官,也可i.d.、i.m.、s.c.、i.p.和i.v.注射方式全身给药,或体外应用到来自患者或其细胞株的细胞(随后再将这些细胞注入到患者中),或体外用于从来自患者的免疫细胞的一个细胞亚群(然后再将细胞重新给予患者)。如果核酸体外注入细胞,可能有益于细胞转染,以共同表达免疫刺激细胞因子(如白细胞介素
‑
2)。肽可完全单独给药,也可与免疫刺激佐剂相结合(见下文)、或与免疫刺激细胞因子联合使用、或以适当的输送系统给药(例如脂质体)。该肽也可共轭形成一种合适的载体(如钥孔虫戚血蓝蛋白(klh)或甘露)到合适的载体(参阅wo 95/18145及(longenecker et al.,1993))。肽也可能被标记,可能是融合蛋白,或可能是杂交分子。在本发明中给出序列的肽预计能刺激cd4或cd8 t细胞。然而,在有cd4 t
‑
辅助细胞的帮助时,cd8 t细胞刺激更加有效。因此,对于刺激cd8 t细胞的mhc
‑
i类表位,一种杂合分子的融合伙伴或片段提供了刺激cd4阳性t细胞的适当表位。cd4
‑
和cd8刺激表位为本领域所熟知、并包括本发明中确定的表位。
[0840]
一方面,疫苗包括至少含有seq id no:1至seq id no:640中提出的一种肽以及至少另外一种肽,优选为2至50个、更优选为2至25个、再优选为2至20个、最优选为2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17或18个肽。肽可能从一个或多个特定taa中衍生,并且可
能与mhc i类分子结合。
[0841]
另一方面,本发明提出了一种编码本发明中肽或肽变体的核酸(如多聚核苷酸)。多聚核苷酸可能为,例如,dna、cdna、pna、rna或其组合物,它们可为单链和/或双链、或多聚核苷酸的原生或稳定形式(如:具有硫代磷酸骨架的多聚核苷酸),并且只要它编码肽,就可能包含也可能不包含内含子。当然,多聚核苷酸只能编码加入天然肽键并含有天然氨基酸残基的肽。另一个方面,本发明提出了一种可根据本发明表达多肽的表达载体。
[0842]
对于连接多核苷酸,已经开发出多种方法,尤其是针对dna,可透过向载体补充可连接性末端等方法进行连接。例如,可向dna片段加入补充性均聚物轨道,之后dna片段被插入到载体dna。然后,透过补充性均聚物尾巴的氢键结合,将载体和dna片段结合,从而形成重组dna分子。
[0843]
含有一个或多个酶切位点的合成接头为dna片段与载体连接提供了另一种方法。含各种限制性核酸内切酶的合成接头可透过多种管道购得,其中包括从国际生物技术公司(international biotechnologies inc,new haven,cn,美国)购得。
[0844]
编码本发明多肽的dna理想修饰方法是使用saiki等人(saiki et al.,1988)所采用的聚合酶链反应方法。此方法可用于将dna引入合适的载体(例如,透过设计合适的酶切位点),也可用于本领域已知的其他有用方法修饰dna。如果使用病毒载体,痘病毒载体或腺病毒载体为优选。
[0845]
之后,dna(或在逆转录病毒载体情况下,rna)可能表达于合适的宿主,从而制成含本发明肽或变体的多肽。因此,可根据已知技术使用编码本发明肽或变体的dna,用本文所述方法适当修饰后,构建表达载体,然后表达载体用于转化合适宿主细胞,从而表达和产生本发明中的多肽。此类技术包括那些公开于,例如,美国专利4,440,859、4,530,901、4,582,800、4,677,063、4,678,751、4,704,362、4,710,463、4,757,006、4,766,075和4,810,648。
[0846]
编码含本发明化合物多肽的dna(或在逆转录病毒载体情况下,rna)可能被加入到其他多种dna序列,从而引入到合适的宿主中。同伴dna将取决于宿主的性质、dna引入宿主的方式、以及是否需要保持为游离体还是要相互结合。
[0847]
一般来说,dna可以适当的方向和正确的表达阅读框架附着到一种表达载体(如质粒)中。如有必要,该dna可能与所需宿主所识别的相应转录和翻译调节控制核苷酸序列连接,尽管表达载体中一般存在此类控制功能。然后,该载体透过标准方法被引入宿主。一般来说,并不是所有的宿主都会被载体转化。因此,有必要选择转化过的宿主细胞。选择方法包括用任何必要的控制元素向表达载体插入一个dna序列,该序列对转化细胞中的可选择性属性(如抗生素耐药性)进行编码。
[0848]
另外,有这种选择属性的基因可在另外一个载体上,该载体用来协同转化所需的宿主细胞。
[0849]
然后,本发明中的重组dna所转化的宿主细胞在本文中所述本领域技术人员熟悉的合适条件下培养足够长的时间,从而表达之后可回收的肽。
[0850]
有许多已知的表达系统,包括细菌(如大肠杆菌和枯草芽孢杆菌)、酵母(如酵母菌)、丝状真菌(如曲霉菌)、植物细胞、动物细胞及昆虫细胞。该系统可优选为哺乳动物细胞,如来自atcc细胞生物学库(cell biology collection)中的cho细胞。
[0851]
典型的哺乳动物细胞组成型表达载体质粒包括cmv或含一个合适的多聚a尾巴的
sv40启动子以及抗性标志物(如新霉素)。一个实例为从pharmacia公司(piscataway,新泽西,美国)获得的psvl。一种可诱导型哺乳动物表达载体的例子是pmsg,也可以从pharmacia公司获得。有用的酵母质粒载体是prs403
‑
406和prs413
‑
416,一般可从stratagene cloning systems公司(la jolla,ca92037,美国)获得。质粒prs403、prs404、prs405和prs406是酵母整合型质粒(yip),并插入了酵母可选择性标记物his3、trp1、leu2和ura3。prs413
‑
416质粒为酵母着丝粒质粒(ycp)。基于cmv启动子的载体(如,来自于sigma
‑
aldrich公司)提供了瞬时或稳定的表达、胞浆表达或分泌,以及flag、3xflag、c
‑
myc或matn不同组合物中的n
‑
端或c
‑
端标记。这些融合蛋白可用于检测、纯化及分析重组蛋白。双标融合为检测提供了灵活性。
[0852]
强劲的人巨细胞病毒(cmv)启动子调控区使得cos细胞中的组成蛋白表达水平高达1mg/l。对于较弱的细胞株,蛋白水平一般低于0.1mg/l。sv40复制原点的出现将导致dna在sv40复制容纳性cos细胞中高水平复制。例如,cmv载体可包含细菌细胞中的pmb1(pbr322的衍生物)复制原点、细菌中进行氨苄青霉素抗性选育的钙
‑
内酰胺酶基因、hgh polya和f1的原点。含前胰岛素原引导(ppt)序列的载体可使用抗flag抗体、树脂和板引导flag融合蛋白分泌到进行纯化的培养基中。其他与各种宿主细胞一起应用的载体和表达系统是本领域熟知众所周知的。
[0853]
在另一个实施方案中,对本发明的两个或更多的肽或肽变体进行编码,因此,以一个连续顺序(类似于”一串珠子”的构建体)表达。在达到目标,所述肽或肽变体可能透过连接符氨基酸的延伸处(例如llllll)连接或融合一起,也可能他们之间没有任何附加的肽而被连接。这些构建体也可用于癌症治疗,可诱导涉及mhc i和mhc ii类分子的免疫应答。
[0854]
本发明还涉及一种宿主细胞,其以本发明的多核苷酸载体构建转化而来。宿主细胞可为原核细胞,也可为真核细胞。在有些情况下,细菌细胞为优选原核宿主细胞,典型为大肠杆菌株,例如,大肠杆菌菌株dh5(从bethesda research laboratories公司(bethesda,md,美国)获得)和rr1(从美国菌种保藏中心(atcc,rockville,md,美国),atcc编号31343获得)。首选的真核宿主细胞包括酵母、昆虫和哺乳动物细胞,优选为脊椎动物细胞,如:小鼠、大鼠、猴子或人成纤维细胞和结肠癌细胞株中的细胞。酵母宿主细胞包括yph499、yph500和yph501,一般可从stratagene cloning systems公司(la jolla,ca92037,美国)获得。首选哺乳动物宿主细胞包括中国仓鼠卵巢(cho)细胞为atcc中的ccl61细胞、nih瑞士小鼠胚胎细胞nih/3t3为atcc中的crl 1658细胞、猴肾源性cos
‑
1细胞为atcc中的crl 1650细胞以及人胚胎肾细胞的293号细胞。首选昆虫细胞为sf9细胞,可用杆状病毒表达载体转染。有关针对表达选择合适宿主细胞的概要,可从教科书(paulina balb
á
s and argelia lorence《methods in molecular biology recombinant gene expression,reviews and protocols》part one,second edition,isbn 978
‑1‑
58829
‑
262
‑
9)和技术人员知道的其他文献中查到。
[0855]
含本发明dna结构的适当宿主细胞的转化可使用大家熟知的方法完成,通常取决于使用载体的类型。关于原核宿主细胞的转化,请参见,例如,cohen等人的文献(cohen et al.,1972)和(green and sambrook,2012)。酵母细胞的转化在sherman等人的文章(sherman et al.,1986)中进行了描述。beggs(beggs,1978)中所述的方法也很有用。对于脊椎动物细胞,转染这些细胞的试剂等,例如,磷酸钙和deae
‑
葡聚糖或脂质体配方,可从
stratagene cloning systems公司或life technologies公司(gaithersburg,md 20877,美国)获得。电穿孔也可用于转化和/或转染细胞,是本领域用于转化酵母细胞、细菌细胞、昆虫细胞和脊椎动物细胞大家熟知的方法。
[0856]
被成功转化的细胞(即含本发明dna结构的细胞)可用大家熟知的方法(如pcr)进行识别。另外,上清液存在的蛋白可使用抗体进行检测。
[0857]
应了解,本发明中的某些宿主细胞用于制备本发明中的肽,例如细菌细胞、酵母细胞和昆虫细胞。但是,其他宿主细胞可能对某些治疗方法有用。例如,抗原提呈细胞(如树突状细胞)可用于表达本发明中的肽,使他们可以加加载相应的mhc分子中。因此,本发明提出了含本发明中核酸或表达载体的一种宿主细胞。
[0858]
在一个优选实施方案中,宿主细胞为抗原提呈细胞,尤其是树突状细胞或抗原提呈细胞。2010年4月29日,美国食品和药物管理局(fda)批准载有含前列腺酸性磷酸酶(pap)的重组融合蛋白可用于治疗无症状或症状轻微的转移性hrpc(rini et al.,2006;small et al.,2006)。
[0859]
另一方面,本发明提出了一种配制一种肽及其变体的方法,该方法包括培养宿主细胞和从宿主细胞或其培养基中分离肽。
[0860]
在另一个实施方案中,本发明中的肽、核酸或表达载体用于药物中。例如,肽或其变体可制备为静脉(i.v.)注射剂、皮下(s.c.)注射剂、皮内(i.d.)注射剂、腹膜内(i.p.)注射剂、肌肉(i.m.)注射剂。肽注射的优选方法包括s.c.、i.d.、i.p.、i.m.和i.v.注射。dna注射的优选方法为i.d.、i.m.、s.c.、i.p.和i.v.注射。例如,给予50μg至1.5mg,优选为125μg至500μg的肽或dna,这取决于具体的肽或dna。上述剂量范围在以前的试验中成功使用(walter et al.,2012)。
[0861]
用于主动免疫接种的多聚核苷酸可为基本纯化形式,也可包被于载体或输送系统。核酸可能为dna、cdna、pna、rna,也可能为其组合物。这种核酸的设计和引入方法为本领域所熟知。例如,文献中有其概述(teufel et al.,2005)。多核苷酸疫苗很容易制备,但这些载体诱导免疫反应的作用模式尚未完全了解。合适的载体和输送系统包括病毒dna和/或rna,如基于腺病毒、牛痘病毒、逆转录病毒、疱疹病毒、腺相关病毒或含一种以上病毒元素的混合病毒的系统。非病毒输送系统包括阳离子脂质体和阳离子聚合物,是dna输送所属领域内熟知的系统。也可使用物理输送系统,如透过”基因枪”。肽或核酸编码的肽可以是一种融合蛋白,例如,含刺激t细胞进行上述cdr的表位。
[0862]
本发明的药剂也可能包括一种或多种佐剂。佐剂是那些非特异性地增强或加强免疫反应的物质(例如,透过cd8
‑
阳性t细胞和辅助t(t
h
)细胞介导的对一种抗原的免疫应答,因此被视为对本发明的药剂有用。适合的佐剂包括(但不仅限于)1018iss、铝盐、as15、bcg、cp
‑
870,893、cpg7909、cyaa、dslim、鞭毛蛋白或鞭毛蛋白衍生的tlr5配体、flt3配体、gm
‑
csf、ic30、ic31、咪喹莫特resiquimod、imufact imp321、白细胞介素il
‑
2、il
‑
13、il
‑
21、干扰素α或β,或其聚乙二醇衍生物、is patch、iss、iscomatrix、iscoms、lipovac、malp2、mf59、单磷酰脂a、montanide ims 1312、montanide isa 206、montanide isa 50v、montanide isa
‑
51、水包油和油包水乳状液、ok
‑
432、om
‑
174、om
‑
197
‑
mp
‑
ec、ontak、ospa、载体系统、基于聚丙交酯复合乙交酯
[plg]和右旋糖苷微粒、重组人乳铁传递蛋白srl172、病毒颗粒和其他病毒样颗粒、yf
‑
17d、vegf trap、r848、β
‑
葡聚糖、pam3cys、源自皂角苷、分支杆菌提取物和细菌细胞壁合成模拟物的aquila公司的qs21刺激子,以及其他专有佐剂,如:ribi's detox、quil或superfos。优选佐剂如:弗氏佐剂或gm
‑
csf。前人对一些树突状细胞特异性免疫佐剂(如mf59)及其制备方法进行了描述(allison and krummel,1995)。也可能使用细胞因子。一些细胞因子直接影响树突状细胞向淋巴组织迁移(如,tnf
‑
),加速树突状细胞成熟为t淋巴细胞的有效抗原提呈细胞(如,gm
‑
csf、il
‑
1和il
‑
4)(美国5849589号专利,特别以其完整引用形式并入本文),并充当免疫佐剂(如il
‑
12、il
‑
15、il
‑
23、il
‑
7、ifn
‑
α、ifn
‑
β)(gabrilovich et al.,1996)。
[0863]
据报告,cpg免疫刺激寡核苷酸可提高佐剂在疫苗中的作用。如果没有理论的约束,cpg寡核苷酸可透过toll样受体(tlr)(主要为tlr9)启动先天(非适应性)免疫系统从而起作用。cpg引发的tlr9活化作用提高了对各种抗原的抗原特异性体液和细胞反应,这些抗原包括肽或蛋白抗原、活病毒或被杀死的病毒、树突状细胞疫苗、自体细胞疫苗以及预防性和治疗性疫苗中的多糖结合物。更重要的是,它会增强树突状细胞的成熟和分化,导致t
h1
细胞的活化增强以及细胞毒性t淋巴细胞(ctl)生成加强,甚至cd4 t细胞说明的缺失。甚至有疫苗佐剂的存在也能维持tlr9活化作用诱发的t
h1
偏移,这些佐剂如:正常促进t
h2
偏移的明矾或弗氏不完全佐剂(ifa)。cpg寡核苷酸与以下其他佐剂或配方一起制备或联合给药时,表现出更强的佐剂活性,如微粒、纳米粒子、脂肪乳或类似制剂,当抗原相对较弱时,这些对诱发强反应尤为必要。他们还能加速免疫反应,使抗原剂量减少约两个数量级,在有些实验中,对不含cpg的全剂量疫苗也能产生类似的抗体反应(krieg,2006)。美国6406705b1号专利对cpg寡核苷酸、非核酸佐剂和抗原结合使用促使抗原特异性免疫反应进行了描述。一种cpg tlr9拮抗剂为mologen公司(德国柏林)的dslim(双干环免疫调节剂),这是本发明药物组合物的优选成分。也可使用其他如tlr结合分子,如:rna结合tlr7、tlr8和/或tlr9。
[0864]
其他有用的佐剂例子包括(但不限于)化学修饰性cpg(如cpr、idera)、dsrna模拟物,如,poly(i:c)及其衍生物(如:ampligen、hiltonol、多聚
‑
(iclc)、多聚(ic
‑
r)、多聚(i:c12u))、非cpg细菌性dna或rna以及免疫活性小分子和抗体,如:环磷酰胺、舒尼替单抗、贝伐单抗、西乐葆、ncx
‑
4016、西地那非、他达拉非、伐地那非、索拉非尼、替莫唑胺、temsirolimus、xl
‑
999、cp
‑
547632、帕唑帕尼、vegf trap、zd2171、azd2171、抗
‑
ctla4、免疫系统的其他抗体靶向性主要结构(如:抗
‑
cd40、抗
‑
tgfβ、抗
‑
tnfα受体)和sc58175,这些药物都可能有治疗作用和/或充当佐剂。技术人员无需过度进行不当实验就很容易确定本发明中有用的佐剂和添加剂的数量和浓度。
[0865]
首选佐剂是抗
‑
cd40、咪喹莫特、瑞喹莫德、gm
‑
csf、环磷酰胺、舒尼替尼、贝伐单抗、干扰素α、cpg寡核苷酸及衍生物、多聚(i:c)及衍生物、rna、西地那非和plg或病毒颗粒的微粒制剂。
[0866]
本发明药物组合物的一个优选实施方案中,佐剂从含集落刺激因子制剂中选择,如粒细胞巨噬细胞集落刺激因子(gm
‑
csf,沙格司亭)、环磷酰胺、咪喹莫特、resiquimod和干扰素
‑
α。
[0867]
本发明药物组合物的一个优选实施方案中,佐剂从含集落刺激因子制剂中选择,如粒细胞巨噬细胞集落刺激因子(gm
‑
csf,沙格司亭)、环磷酰胺、咪喹莫特和
resimiquimod。在本发明药物组合物的一个优选实施方案中,佐剂为环磷酰胺、咪喹莫特或resiquimod。更优选的佐剂是montanide ims 1312、montanide isa 206、montanide isa 50v、montanide isa
‑
51、聚
‑
iclc和抗cd40 mab或其组合物。
[0868]
此组合药物为非肠道注射使用,如皮下、皮内、肌肉注射,也可口服。为此,肽和其他选择性分子在药用载体中分解或悬浮,优选为水载体。此外,组合物可包含辅料,如:缓冲剂、结合剂、冲击剂、稀释剂、香料、润滑剂等。这些肽也可与免疫刺激物质合用,如:细胞因子。可用于此类组合物的更多辅料可在从a.kibbe所著的handbook of pharmaceutical excipients(kibbe,2000)等书中获知。此组合药物可用于阻止、预防和/或治疗腺瘤或癌性疾病。例如,ep2112253中有示例制剂。
[0869]
重要的是要认识到,透过本发明的疫苗引发的免疫应答在不同的细胞阶段和开发的不同阶段攻击癌症。而且不同的癌症相关信号通路被攻击。这相对于其他疫苗的优势,这些疫苗只针对一个或几个靶标,这可能会导致肿瘤很容易适应于攻击(肿瘤逃逸)。此外,并非所有的个体肿瘤都表达相同模式的抗原。因此,几个肿瘤相关肽的组合确保了每个肿瘤都承担至少一些靶标。该组合物以这样的方式设计,预期每个肿瘤可表达几种抗原并覆盖肿瘤生长和维持所需要的几种独立的途径。因此,疫苗可易于”现成的”用于较大患者群体。这意味着,预选择接受疫苗治疗的患者可限制为hla分型,无需抗原表达的任何额外的生物标志物评估,但仍然确保多个靶标同时被诱导的免疫应答攻击,这对于疗效很重要(banchereau et al.,2001;walter et al.,2012)。
[0870]
本文所用的”支架”一词是指与(如抗原)决定因子特异性结合的分子。在一项实施方案中,支架是能够引导其所连接的实体(例如,(第二)抗原结合部分)至目标靶点,例如,至特定类型的肿瘤细胞或承载抗原决定簇的肿瘤基质(如根据目前申请中肽和mhc的复合体)。在另一项实施例中,支架能够透过其靶抗原(例如t细胞受体复合体抗原)启动信号通路。支架包括但不限于抗体及其片段,抗体的抗原结合区,其包含抗体重链可变区和抗体轻链可变区,结合的蛋白包括至少一个锚蛋白重复序列基元和单域抗原结合(sdab)分子、适体、(可溶)tcr和(经修饰的)细胞,例如同种异体或自体t细胞。为了评估某个分子是否是结合至靶点的支架,可进行结合测定。
[0871]“特定”结合系指,与其他天然肽
‑
mhc复合体相比,该支架与感兴趣的肽
‑
mhc复合体更好地结合,结合程度为,拥有能够杀死承载特定靶点细胞的活性分子的支架不能够杀死无特定靶点但提呈一个或多个其他肽
‑
mhc复合体的另一细胞。如果交叉反应性肽
‑
mhc的肽并不是天然的,即,并非来自人hla
‑
多肽组,则结合至其他肽
‑
mhc复合体是无关紧要的。评估靶细胞杀伤的测试在本领域中是公知的。它们应该含有未改变的肽
‑
mhc提呈的靶细胞(原发细胞或细胞系)或载有肽的细胞进行,以便达到天然肽
‑
mhc的水平。
[0872]
各支架可包括一个标记,其透过确定是否存在或不存在卷标所提供的信号可检测到结合支架。例如,该支架可用荧光染料或任何其他适用的细胞标记分子进行标记。此类标记分子是本领域中公知的。例如,透过荧光染料进行的荧光标记可透过荧光或激光扫描显微术或流式细胞术提供结合适体的可视化。
[0873]
各支架可与第二个活性分子(例如il
‑
21、抗cd3、抗cd28)共轭。
[0874]
关于多肽支架的进一步信息,可参阅,例如,在wo 2014/071978a1背景技术部分,并作为参考文献引用。
[0875]
本发明还涉及适体。适体(例如,参见wo 2014/191359及其中引用的文献)是短的单链核酸分子,其可以折迭为所定义的三维结构并识别特定的靶标结构。它们似乎是开发靶向治疗的合适替代方法。适体已显示可选择性与具有高亲和力和特异性的复合体靶标相结合。
[0876]
识别细胞表面分子的适体在过去十年内已经确定,并为开发诊断和治疗方法提供了手段。由于适体已显示几乎无毒性和免疫原性,因此,它们是生物医学应用中有前景的候选物质。事实上适体,例如前列腺特异性膜抗原识别适体,已被成功地用于靶向治疗并在体内模型的异种移植物中显示出功能。此外,认识到特定肿瘤细胞系的适体也已确定。
[0877]
可选择dna适体来揭示各种癌细胞的广谱标识属性,特别是那些来自于实体瘤的细胞,而非致瘤和主要健康细胞不被识别。如果所识别的适体不仅识别肿瘤特异性子类型,而且与一系列肿瘤相互作用,这使适体适用于作为所谓的广谱诊断和治疗手段。
[0878]
此外,用流式细胞仪对细胞结合行为的研究显示,适体在纳摩尔范围内显示出很好的亲和力。
[0879]
适体用于诊断和治疗目的。此外,也可能显示,一些适体被肿瘤细胞吸取,因而可作为抗癌剂靶向递送的分子赋形剂,例如sirna进入肿瘤细胞。
[0880]
可选择适体针对复合体的靶标,如细胞和组织以及包含、优选包括根据任何seq id no 1至seq id no 640的一个序列、根据当前发明的肽复合体与mhc分子,使用细胞selex(透过指数富集的配体系统进化)技术。
[0881]
本发明中的肽可用于生成和开发出针对mhc/肽复合物的特定抗体。这些抗体可用于治疗,将毒素或放射性物质靶向病变组织。这些抗体的另一用途是为了成像之目的(如pet)将放射性核素靶向病变组织。这可有助于检测小转移灶或确定病变组织的大小和准确位置。
[0882]
因此,本发明的另一方面是提出产生特异性结合至与hla限制性抗原络合的i或ii类人主要组织兼容性复合体(mhc)的一种重组抗体的方法,该方法包括:用可溶形式的与hla限制性抗原络合的(mhc)i或ii类分子对包含表达所述主要组织兼容性说复合体(mhc)i或ii类的基因工程非人哺乳动物进行免疫;将mrna分子与产生所述非人哺乳动物细胞的抗体分离;产生一个噬菌体显示库,显示由所述mrna分子编码的蛋白分子;以及将至少一个噬菌体与所述噬菌体显示库分离,所述的至少一个噬菌体显示所述抗体特异性地结合至与hla限制性抗原络合的所述人主要组织兼容性说复合体(mhc)i或ii类。
[0883]
本发明的另一方面提出一种抗体,其特异性结合至与一种hla限制性抗原络合的i或ii类人主要组织兼容性说复合体(mhc),其中该抗体优选为多克隆抗体、单克隆抗体、双特异性抗体和/或嵌合抗体。
[0884]
产生这种抗体和单链i类主要组织兼容性复合物的相应方法,以及产生这些抗体的其他工具在wo 03/068201、wo 2004/084798、wo 01/72768、wo 03/070752以及出版物(cohen et al.,2003a;cohen et al.,2003b;denkberg et al.,2003)中进行了披露,为了本发明之目的,所有参考文献透过引用被完整地并入本文。
[0885]
优选地,该抗体与复合体的结合亲和力低于20纳摩尔,优选为低于10纳摩尔,这在本发明情况下也被视为具有”特异性”。
[0886]
本发明涉及一种肽,包含选自seq id no:1至seq id no:640组成的组的一个序列
或该序列的与seq id no:1至seq id no:640具有88%同源性(优选为相同)的一种变体,或诱导与所述变异肽发生t细胞交叉反应的一种变体,其中,所述肽不是基本的全长多肽。
[0887]
本发明进一步涉及一种肽,包含选自seq id no:1至seq id no:640组成的组的一个序列、或与seq id no:1至seq id no:640具有至少88%同源性(优选为相同)的一种变体,其中所述肽或变体的总长度为8至100个、优选为8至30个、最优选为8至14个氨基酸。
[0888]
本发明进一步涉及本发明的肽,其具有与主要组织兼容性复合体(mhc)i或ii类分子结合的能力。
[0889]
本发明进一步涉及本发明中的肽,其中肽系由或基本系由根据seq id no:1至seq id no:640的一个氨基酸序列组成。
[0890]
本发明进一步涉及本发明的肽,其中该肽(在化学上)被修饰和/或包含非肽键。
[0891]
本发明进一步涉及本发明的肽,其中该肽为融合蛋白的一部分,特别包括hla
‑
dr抗原相关不变链(ii)的n
‑
端氨基酸,或其中该肽与一种抗体(例如,树突状细胞特定抗体)融合。
[0892]
本发明进一步涉及一种核酸,其编码本发明所述肽,前提是该肽并非完整(完全)的人蛋白。
[0893]
本发明进一步涉及一种本发明的核酸,为dna、cdna、pna、rna,也可能为其组合物。
[0894]
本发明进一步涉及一种能表达本发明核酸的表达载体。
[0895]
本发明进一步涉及本发明的一种肽、本发明的一种核酸或本发明的一种药用表达载体,特别是用于治疗卵巢癌。
[0896]
本发明进一步涉及含本发明核酸或本发明表达载体的一种宿主细胞。
[0897]
本发明进一步涉及本发明的宿主细胞,其为抗原提呈细胞,优选为树突细胞。
[0898]
本发明进一步涉及配制本发明一种肽的一种方法,所述方法包括培养本发明的宿主细胞和从所述宿主细胞或其培养基中分离肽。
[0899]
本发明进一步涉及本发明中的方法,其中抗原透过与足够量的含抗原提成细胞的抗原结合被载入表达于合适抗原提呈细胞表面的i或ii类mhc分子。
[0900]
本发明进一步涉及本发明的方法,其中该抗原提呈细胞包括一个表达载体,该载体有能力表达含seq id no:1至seq id no:640的肽或所述变体氨基酸序列。
[0901]
本发明进一步涉及以本发明方法制造的启动t细胞,其中所述t细胞有选择性地识别一种细胞,该细胞异常表达含一种本发明氨基酸序列的多肽。
[0902]
本发明进一步涉及一种杀伤患者靶细胞的方法,其中患者的靶细胞异常表达含本发明任何氨基酸序列的多肽,该方法包括给予患者本发明的有效量t细胞。
[0903]
本发明进一步涉及任何所述肽、本发明的一种核酸、本发明的一种表达载体、本发明的一种细胞、本发明一种作为药剂或制造药剂的启动细胞毒性t淋巴细胞的用途。本发明进一步涉及一种本发明的用途,其中药剂可有效抗癌。
[0904]
本发明进一步涉及一种本发明的用途,其中该药剂为一种疫苗。本发明进一步涉及一种本发明的用途,其中药剂可有效抗癌。
[0905]
本发明还一般涉及本发明的用途,其中所述癌细胞为卵巢癌细胞或其他实体或血液肿瘤细胞,如:卵巢癌、非小细胞肺癌、小细胞肺癌、肾癌、脑癌、结肠癌或直肠癌、胃癌、肝癌、胰腺癌、前列腺癌、白血病、乳腺癌、梅克尔细胞癌、黑色素瘤、食管癌、膀胱癌、子宫癌、
胆囊癌、胆管癌。
[0906]
本发明进一步涉及一种基于本发明肽的特定标志物蛋白和生物标志物,在此成为“靶标”,其可用于诊断和/或判断卵巢癌的预后。本发明还涉及这些供癌症治疗使用的新靶点。
[0907]
本文中术语”抗体”为广义上的定义,既包括多克隆也包括单克隆抗体。除了完整或”全部”的免疫球蛋白分子,”抗体”这一术语还包括这些免疫球蛋白分子和人源化免疫球蛋白分子的片段(如,cdr、fv、fab和fc片段)或聚合物,只要它们表现出本发明的任何期望属性(例如,卵巢癌标志物(多)肽的特异性结合、将毒素传递给癌症标志物基因表达水平增加时的卵巢癌细胞和/或抑制卵巢癌标志物多肽的活性)。
[0908]
只要有可能,本发明的抗体可从商业来源购买。本发明的抗体也可能使用已知的方法制得。技术人员会了解全长卵巢癌标志物多肽或其片段可用于制备本发明的抗体。用于产生本发明抗体的多肽可部分或全部地由天然源经纯化而得,也可利用重组dna技术生产。
[0909]
例如,本发明的编码肽的cdna,例如,该肽为根据seq id no:1至seq id no:640多肽的肽,或其中一个变体或片段,可在原核细胞中(如:细菌)或真核细胞(如:酵母、昆虫或哺乳动物细胞)中表达,之后,可纯化重组蛋白,并用于产生一种特异性结合用于产生本发明抗体的卵巢癌标志物多肽的单克隆或多克隆抗体制剂。
[0910]
本领域的技术人员会认识到,两种或两种以上不同集合的单克隆抗体或多克隆抗体能最大限度地增加获得一种含预期用途所需的特异性和亲和力(例如,elisa法、免疫组织化学、体内成像、免疫毒素疗法)的抗体的可能性。根据抗体的用途,用已知的方法对其期望活性进行测试(例如,elisa法、免疫组织化学、免疫治疗等;要获取产生和测试抗体的进一步指导,请参阅,例如,greenfield,2014(greenfield,2014))。例如,该抗体可用elisa法或免疫印迹法、免疫组织化学染色福尔马林固定的癌组织或冰冻的组织切片进行检测。在初次体外表征后,用于治疗或体内诊断用途的抗体根据已知的临床测试方法进行检测。
[0911]
此处使用的术语”单克隆抗体”系指从大量同质抗体中获得的一种抗体,即,由相同的抗体组成的抗体群,但可能少量提呈的自然突变除外。此处所述的单克隆抗体具体包括”嵌合”抗体,其中一部分重链和/或轻链与从特定物种中获得的抗体或属于特定抗体类型和分类型抗体的相应序列相同(同质),同时,剩余链与从其他物种中获得的抗体或属于特定抗体类型和子类型抗体的相应序列以及这些抗体的片段相同(同质),只要他们表现出预期的拮抗活性(美国4816567号专利,其在此以其整体并入)。
[0912]
本发明的单克隆抗体可能使用杂交瘤方法制得。在杂交瘤方法中,老鼠或其他适当的宿主动物,通常用免疫制剂以引发产生或能产生将特异性结合至免疫制剂的抗体。或者,淋巴细胞可在体外进行免疫。
[0913]
单克隆抗体也可由dna重组方法制得,如:美国4816567号专利所述。编码本发明单克隆抗体的dna可很容易地使用传统程序进行分离和测序(例如:透过使用能与编码鼠抗体重链和轻链的基因特异性结合的寡核苷酸探针)。
[0914]
体外方法也适用于制备单价抗体。抗体消化以产生抗体的片段,尤其是fab片段,可以透过使用本领域已知的常规技术完成。例如,可以透过使用木瓜蛋白酶完成消化。木瓜蛋白酶消化的实施例在wo 94/29348和美国4342566号专利中有描述。抗体的木瓜蛋白酶消
化通常产生两种相同的抗原结合性片段,称为fab片段(每个片段都有一个抗原结合点)和残余fc片段。胃蛋白酶处理产生一个f(ab')2片段和一个pfc'片段。
[0915]
抗体片段,不论其是否附着于其他序列,均可包括特定区域或特定氨基酸残基的插入、删除、替换、或其他选择性修饰,但前提是,片段的活性与非修饰的抗体或抗体片段相比没有显著的改变或损害。这些修饰可提供一些额外的属性,如:删除/添加可与二硫键结合的氨基酸,以增加其生物寿命、改变其分泌特性等。在任何情况下,抗体片段必须拥有生物活性的特性,如:结合活性、调节结合域的结合力等。抗体的功能性或活性区域可透过蛋白特定区域的基因突变、随后表达和测试所表达的多肽进行确定。这些方法为本行业技术人员所熟知,可包括编码抗体片段的核酸的特定位点基因突变。
[0916]
本发明的抗体可进一步包括人源化抗体或人抗体。非人(如:鼠)抗体的人源化形式为嵌合抗体免疫球蛋白、免疫球蛋白链或其片段(如:fv、fab、fab'或抗体的其他抗原结合序列),其中包含从非人免疫球蛋白中获得的最小序列。人源化抗体包括人免疫球蛋白(受体抗体),其中来自受体互补决定区(cdr)的残基被来自非人物种(供体抗体)(如具有与其特异性、亲和力和能力的小鼠、大鼠或兔子)cdr的残基取代。在某些情况下,人类免疫球蛋白的fv框架(fr)残基被相应的非人残基取代。人源化抗体可能还包括既非受体抗体、也非输入cdr或框架序列中发现的残基。一般来说,人源化抗体将包括几乎所有的至少一个、通常为二个可变域,其中,全部或几乎全部的cdr区域均对应于非人免疫球蛋白的区域并且全部或几乎全部的fr区域均为人免疫球蛋白相同序列的区域。理想情况是,人源化抗体还将包括至少免疫球蛋白恒定区(fc)的一部分,通常是人免疫球蛋白的恒定区的一部分。
[0917]
人源化非人抗体的方法为本行业所熟知。一般来说,人源化抗体具有一个或多个从非人源头引入的氨基酸残基。这些非人氨基酸残基往往被称为”输入”残基,通常从”输入”可变域中获得。人源化基本上可以透过将啮齿动物cdr或cdr序列取代为相应的人抗体序列而完成。因此,这种”人源化”抗体为嵌合抗体(美国4816567号专利),其中大大少于完整的人可变域被来自于非人物种的相应序列取代。在实践中,人源化抗体通常为人抗体,其中有些cdr残基以及可能的一些fr残基被来自啮齿动物抗体中的类似位点的残基取代。
[0918]
可使用免疫后在内源性免疫球蛋白产生缺失时能产生完整人抗体的转基因动物(如:小鼠)。例如,它被描述为,嵌合和种系突变小鼠中的抗体重链连接区域基因的纯合性缺失导致内源性抗体生成的完全抑制。在此种系变种小鼠中人种系免疫球蛋白基因数组的转移在抗原挑战后将导致人抗体的生成。人抗体也可在噬菌体展示库中产生。
[0919]
本发明的抗体优选为透过药用载体的形式给予受试者。通常,在制剂中使用适量的药用盐,以使制剂等渗。药用载体的例子包括生理盐水、林格氏液和葡萄糖溶液。溶液的ph值优选为约5至8,更优选为约7至7.5。此外,载体还包括缓释制剂,如:含有抗体的固体疏水性聚合物半透性基质,其中基质为有形物品形式,如:薄膜、脂质体或微粒。本行业的技术人员熟知,某些载体可能为更优选,取决于例如,抗体的给药途径和浓度。
[0920]
该抗体可透过注射(如:静脉内、腹腔内、皮下、肌肉内)或透过输注等其他方法给予受试者、患者或细胞,确保其以有效的形式传输到血液中。这些抗体也可以透过瘤内或瘤周途径给予,从而发挥局部和全身的治疗作用。局部或静脉注射为优选。
[0921]
抗体给药的有效剂量和时间表可根据经验确定,并且作出此类决定属本行业的技术范围内。本行业的技术人员会明白,必须给予的抗体剂量根据以下因素会有所不同,例
drive,rockville,maryland 20852,美国)目录号crl1992;果蝇细胞株schneider 2号株从属atcc目录crl 19863;小鼠rma
‑
s细胞株ljunggren等人描述过(ljunggren and karre,1985)。
[0929]
优选情况是,宿主细胞在转染前基本上不表达mhc i类分子。刺激因子细胞还优选为表达对t细胞共刺激信号起到重要作用的分子,如,b7.1、b7.2、icam
‑
1和lfa3中的任一种分子。大量mhc i类分子和共刺激分子的核酸序列可从genbank和embl数据库中公开获得。
[0930]
当mhc i类表位用作一种抗原时,t细胞为cd8阳性t细胞。
[0931]
如果抗原提呈细胞受到转染而表达这种表位,则优选的细胞包括一个表达载体,该载体有能力表达含seq id no:1至seq id no:640的肽或变体氨基酸序列。
[0932]
可使用其他一些方法来体外生成t细胞。例如,自体肿瘤浸润性淋巴细胞可用于生成ctl。plebanski等人在(plebanski et al.,1995)使用自体外周血淋巴细胞(plb)制得t细胞。另外,也可能用肽或多肽脉冲处理树突状细胞或透过与重组病毒感染而制成自体t细胞。此外,b细胞可用于制备自体t细胞。此外,用肽或多肽脉冲处理或用重组病毒感染的巨噬细胞可用于配制自体t细胞。s.walter等人在(walter et al.,2003)中描述了透过使用人工抗原提呈细胞(aapc)体外启动t细胞,这也是生成作用于所选肽的t细胞的一种合适方法。在本发明中,根据生物素:链霉素生物化学方法透过将预制的mhc:肽复合物耦合到聚苯乙烯颗粒(微球)而生成aapc。该系统实现了对aapc上的mhc密度进行精确调节,这使得可以在血液样本中选择地引发高或低亲合力的高效抗原特异性t细胞反应。除了mhc:肽复合物外,aapc还应携运含共刺激活性的其他蛋白,如耦合至表面的抗
‑
cd28抗体。此外,此类基于aapc的系统往往需要加入适当的可溶性因子,例如,诸如白细胞介素12的细胞因子。
[0933]
也可用同种异体细胞制得t细胞,在wo 97/26328中详细描述了一种方法,以参考文献方式并入本文。例如,除了果蝇细胞和t2细胞,也可用其他细胞来提呈肽,如cho细胞、杆状病毒感染的昆虫细胞、细菌、酵母、牛痘感染的靶细胞。此外,也可使用植物病毒(例如,参阅porta等人在(porta et al.,1994)中描述了将豇豆花叶病毒开发为一种提呈外来肽的高产系统。
[0934]
被启动的t细胞直接针对本发明中的肽,有助于治疗。因此,本发明的另一方面提出了用本发明前述方法制得的启动t细胞。
[0935]
按上述方法制成的启动t细胞将会有选择性地识别异常表达含seq id no:1至seq id no 640氨基酸序列的多肽。
[0936]
优选情况是,t细胞透过与其含hla/肽复合物的tcr相互作用(如,结合)而识别该细胞。t细胞是杀伤患者靶细胞方法中有用的细胞,其靶细胞异常表达含本发明中氨基酸序列的多肽。此类患者给予有效量的启动t细胞。给予患者的t细胞可能源自该患者,并按上述方法启动(即,它们为自体t细胞)。或者,t细胞不是源自该患者,而是来自另一个人。当然,优选情况是该供体为健康人。发明人使用”健康个人”系指一个人一般状况良好,优选为免疫系统合格,更优选为无任何可很容易测试或检测到的疾病。
[0937]
根据本发明,cd8
‑
阳性t细胞的体内靶细胞可为肿瘤细胞(有时表达mhc
‑
ii类抗原)和/或肿瘤周围的基质细胞(肿瘤细胞)(有时也表达mhc
‑
ii类抗原;(dengjel et al.,2006))。
[0938]
本发明所述的t细胞可用作治疗性组合物中的活性成分。因此,本发明也提出了一
种杀伤患者靶细胞的方法,其中患者的靶细胞异常表达含本发明中氨基酸序列的多肽,该方法包括给予患者上述有效量的t细胞。
[0939]
发明人所用的”异常表达”的意思还包括,与正常表达水平相比,多肽过量表达,或该基因在正常(健康)组织中未表达。”过量表达”系指多肽水平至少为正常组织中的1.2倍;优选为至少为正常组织中的2倍,更优选为至少5或10倍。
[0940]
t细胞可用本领域已知的方法制得(如,上述方法)。
[0941]
t细胞继转移方案为本领域所熟知的方案。综述可发现于:gattioni et al.和morgan et al.(gattinoni et al.,2006;morgan et al.,2006)。
[0942]
本发明的另一个方面包括使用与mhc复合的肽,以生成t细胞受体,其核酸被克隆并被引入至宿主细胞,优选为t细胞。然后,该透过基因工程改变的t细胞可转给患者用于癌症治疗。
[0943]
本发明的任一分子(即肽、核酸、抗体、表达载体、细胞,启动t细胞、t细胞受体或编码核酸)都有益于治疗疾病,其特点在于细胞逃避免疫反应的打击。因此,本发明的任一分子都可用作药剂或用于制造药剂。这种分子可单独使用也可与本发明中的其他分子或已知分子联合使用。
[0944]
本发明进一步提出了一种药剂,其用于治疗癌症,特别是卵巢癌和其他恶性肿瘤。
[0945]
本发明还涉及一种套件,其包括:
[0946]
(a)一个容器,包含上述溶液或冻干粉形式的药物组合物;
[0947]
(b)可选的第二个容器,其含有冻干粉剂型的稀释剂或重组溶液;和
[0948]
(c)可选的(i)溶液使用或(ii)重组和/或冻干制剂使用的说明。
[0949]
该套件还步包括一个或多个(iii)缓冲剂,(iv)稀释剂,(v)过滤液,(vi)针,或(v)注射器。容器最好是瓶子、小瓶、注射器或试管,可以为多用途容器。药物组合物最好是冻干的。
[0950]
本发明中的套件优选包含一种置于合适容器中的冻干制剂以及重组和/或使用说明。适当的容器包括,例如瓶子、西林瓶(如双室瓶)、注射器(如双室注射器)和试管。该容器可能由多种材料制成,如玻璃或塑料。试剂盒和/或容器最好有容器或关于容器的说明书,指明重组和/或使用的方向。例如,标签可能表明冻干剂型将重组为上述肽浓度。该标签可进一步表明制剂用于皮下注射。
[0951]
存放制剂的容器可使用多用途西林瓶,使得可重复给予(例如,2
‑
6次)重组剂型。该套件可进一步包括装有合适稀释剂(如碳酸氢钠溶液)的第二个容器。
[0952]
稀释液和冻干制剂混合后,重组制剂中的肽终浓度优选为至少0.15mg/ml/肽(=75μg),不超过3mg/ml/肽(=1500μg)。该套件还可包括商业和用户角度来说可取的其他材料,包括其他缓冲剂、稀释剂,过滤液、针头、注射器和带有使用说明书的包装插页。
[0953]
本发明中的套件可能有一个单独的容器,其中包含本发明所述的药物组合物制剂,该制剂可有其他成分(例如,其他化合物或及其药物组合物),也可无其他成分,或者每种成分都有其不同容器。
[0954]
优选情况是,本发明的套件包括与本发明的一种制剂,包装后与第二种化合物(如佐剂(例如gm
‑
csf)、化疗药物、天然产品、激素或拮抗剂、抗血管生成剂或抑制剂、凋亡诱导剂或螯合剂)或其药物组合物联合使用。该套件的成分可进行预络合或每种成分在给予患
者之前可放置于单独的不同容器。该套件的成分可以是一种或多种溶液,优选为水溶液,更优选为无菌水溶液。该套件的成分也可为固体形式,加入合适的溶剂后转换为液体,最好放置于另一个不同的容器中。
[0955]
治疗套件的容器可能为西林瓶、试管、烧瓶、瓶子、注射器、或任何其他盛装固体或液体的工具。通常,当成分不只一种时,套件将包含第二个西林瓶或其他容器,使之可以单独定量。该套件还可能包含另一个装载药用液体的容器。优选情况是,治疗套件将包含一个设备(如,一个或多个针头、注射器、滴眼器、吸液管等),使得可注射本发明的药物(本套件的组合物)。
[0956]
本发明的药物配方适合以任何可接受的途径进行肽给药,如口服(肠道)、鼻内、眼内、皮下、皮内、肌内,静脉或经皮给药。优选为皮下给药,最优选为皮内给药,也可透过输液泵给药。
[0957]
由于本发明的肽从卵巢癌中分离而得,因此,本发明的药剂优选用于治疗卵巢癌。
[0958]
本发明进一步涉及为个体患者制备个体化药物的一种方法,其中包括:制造含选自预筛选tumap存储库至少一种肽的药物组合物,其中药物组合物中所用的至少一种肽选择为适合于个体患者。在一项实施方案中,药物组合物为一种疫苗。该方法也可以改动以产生下游应用的t细胞克隆物,如:tcr隔离物或可溶性抗体和其他治疗选择。
[0959]“个体化药物“系指专门针对个体患者的治疗,将仅用于该等个体患者,包括个体化活性癌症疫苗以及使用自体组织的过继细胞疗法。
[0960]
如本文所述,“存储库“应指已经接受免疫原性预筛查和/或在特定肿瘤类型中过量提呈的一组或一系列肽。“存储库“一词并不暗示,疫苗中包括的特定肽已预先制造并储存于物理设备中,虽然预期有这种可能性。明确预期所述肽可以用于新制造每种个体化疫苗,也可能被预先制造和储存。存储库(例如,数据库形式)由肿瘤相关肽组成,其在各种hla
‑
ahla
‑
b和hla
‑
c等位基因卵巢癌患者的肿瘤组织中高度过度表达。其可能含有包括mhc i类和mhc ii类肽或拉长的mhc i类肽。除了从几种卵巢癌组织中采集的肿瘤相关肽外,存储库还可能包含hla
‑
a*02和hla
‑
a*24标记肽。这些肽可对tumap诱导的t细胞免疫进行量化比较,从而可得出疫苗抗肿瘤反应能力的重要结论。其次,在没有观察到来自患者“自身”抗原tumap的任何疫苗诱导的t细胞反应时,它们可作为来自“非自身“抗原的重要阳性对照肽。第三,它还可对患者的免疫功能状态得出结论。
[0961]
存储库的tumap透过使用一种功能基因组学方法进行鉴定,该方法结合了基因表达分析、质谱法和t细胞免疫学。该方法确保了只选择真实存在于高百分比肿瘤但在正常组织中不表达或仅很少量表达的tumap用于进一步分析。对于初始肽的选择,患者卵巢癌样本和健康供体的血液以循序渐进的方法进行分析:
[0962]
1.恶性材料的hla配体用质谱法确定
[0963]
2.使用全基因组信使核糖核酸(mrna)表达分析法用于确定恶性肿瘤组织(卵巢癌)与一系列正常器官和组织相比过度表达的基因。
[0964]
3.确定的hla配体与基因表达数据进行比较。肿瘤组织上过度提呈或选择性提呈的肽,优选为第2步中检测到的选择性表达或过量表达基因所编码的考虑为多肽疫苗的合适候选tumap。
[0965]
4.文献检索以确定更多证据以支持确认为tump的肽的相关性
[0966]
5.过度表达在mrna水平的相关性由肿瘤组织第3步选定的tumap重新检测而确定,并且在健康组织上缺乏(或不经常)检测。
[0967]
6.为了评估透过选定的肽诱导体内t细胞反应是否可行,使用健康供体以及卵巢癌患者的人t细胞进行体外免疫原性测定。
[0968]
一方面,在将所述肽加入存储库之前,对其进行筛查以了解免疫原性。举例来说(但不限于此),纳入存储库的肽的免疫原性的确定方法包括体外t细胞启动,具体为:用装载肽/mhc复合物和抗cd28抗体的人工抗原提呈细胞反复刺激来自健康供体的cd8+ t细胞。
[0969]
这种方法优选用于罕见癌症以及有罕见表达谱的患者。与含目前开发为固定组分的多肽鸡尾酒相反的是,存储库可将肿瘤中抗原的实际表达于疫苗进行更高程度的匹配。在多目标方法中,每名患者将使用几种“现成“肽的选定单一肽或组合。理论上来说,基于从50抗原肽库中选择例如5种不同抗原肽的一种方法可提供大约170万种可能的药物产品(dp)组分。
[0970]
在一方面,选择所述肽用于疫苗,其基于个体患者的适合性,并使用本发明此处或后文所述的方法。
[0971]
hla表型、转录和肽组学资料从患者的肿瘤材料和血液样本中收集,以确定最合适每名患者且含有“存储库“和患者独特(即突变)tumap的肽。将选择的那些肽选择性地或过度表达于患者肿瘤中,并且可能的情况下,如果用患者个体pbmc进行检测,则表现出很强的体外免疫原性。
[0972]
优选的情况是,疫苗所包括的肽的一种确定方法包括:(a)识别由来自个体患者肿瘤样本提呈的肿瘤相关肽(tumap);(b)将(a)中鉴定的肽与上述肽的存储库(数据库)进行比对;且(c)从与患者中确定的肿瘤相关肽相关的存储库(数据库)中选择至少一种肽。例如,肿瘤样本提呈的tumap的鉴定方法有:(a1)将来自肿瘤样本的表达数据与所述肿瘤样本组织类型相对应的正常组织样本的表达数据相比对,以识别肿瘤组织中过量表达或异常表达的蛋白;以及(a2)将表达数据与结合到肿瘤样本中i类mhc和/或ii类分子的mhc配体序列想关联,以确定来源于肿瘤过量表达或异常表达的蛋白质的mhc配体。优选情况是,mhc配体的序列的确定方法是:洗脱来自肿瘤样本分离的mhc分子结合肽,并测序洗脱配体。优选情况是,肿瘤样本和正常组织从同一患者获得。
[0973]
除了使用存储库(数据库)模型选择肽以外,或作为一种替代方法,tumap可能在新患者中进行鉴定,然后列入疫苗中。作为一种实施例,患者中的候选tumap可透过以下方法进行鉴定:(a1)将来自肿瘤样本的表达数据与所述肿瘤样本组织类型相对应的正常组织样本的表达数据相比对,以识别肿瘤组织中过量表达或异常表达的蛋白;以及(a2)将表达数据与结合到肿瘤样本中i类mhc和/或ii类分子的mhc配体序列想关联,以确定来源于肿瘤过量表达或异常表达的蛋白质的mhc配体。作为另一实施例,蛋白的鉴定方法为可包含突变,其对于肿瘤样本相对于个体患者的相应正常组织是独特的,并且tumap可透过特异性靶向作用于变异来鉴定。例如,肿瘤以及相应正常组织的基因组可透过全基因组测序方法进行测序:为了发现基因蛋白质编码区域的非同义突变,从肿瘤组织中萃取基因组dna和rna,从外周血单核细胞(pbmc)中提取正常非突变基因组种系dna。运用的ngs方法只限于蛋白编码区的重测序(外显子组重测序)。为了这一目的,使用供货商提供的靶序列富集试剂盒来捕获来自人样本的外显子dna,随后使用hiseq2000(illumina公司)进行测序。此外,对肿瘤的
mrna进行测序,以直接定量基因表达,并确认突变基因在患者肿瘤中表达。得到的数以百万计的序列读数透过软件算法处理。输出列表中包含突变和基因表达。肿瘤特异性体突变透过与pbmc衍生的种系变化比较来确定,并进行优化。然后,为了存储库可能测试新确定的肽了解如上所述的免疫原性,并且选择具有合适免疫原性的候选tumap用于疫苗。
[0974]
在一个示范实施方案中,疫苗中所含肽透过以下方法确定:(a)用上述方法识别由来自个体患者肿瘤样本提呈的肿瘤相关肽(tumap);(b)将(a)中鉴定的肽与进行肿瘤(与相应的正常组织相比)免疫原性和过量提呈预筛查肽的存储库进行比对;(c)从与患者中确定的肿瘤相关肽相关的存储库中选择至少一种肽;及(d)可选地在(a)中选择至少一种新确定的肽,确认其免疫原性。
[0975]
在一个示范实施方案中,疫苗中所含肽透过以下方法确定:(a)识别由来自个体患者肿瘤样本提呈的肿瘤相关肽(tumap);以及(b)在(a)中选择至少一种新确定的肽,并确认其免疫原性。
[0976]
一旦选定了用于个体化肽疫苗的肽时,则产生疫苗。该疫苗优选为一种液体制剂,包括溶解于20
‑
40%dmso之间,优选为约30
‑
35%dmso,例如,约33%dmso中的个体肽。
[0977]
列入产品的每种肽都溶于dmso中。单个肽溶液浓度的选择取决于要列入产品中的肽的数量。单肽
‑
dmso溶液均等混合,以实现一种溶液中包含所有的肽,且浓度为每肽~2.5mg/ml。然后该混合溶液按照1:3比例用注射用水进行稀释,以达到在33%dmso中每肽0.826mg/ml的浓度。稀释的溶液透过0.22μm无菌筛检程序进行过滤。从而获得最终本体溶液。
[0978]
最终本体溶液填充到小瓶中,在使用前储存于
‑
20℃下。一个小瓶包含700μl溶液,其中每种肽含有0.578mg。其中的500μl(每种肽约400μg)将用于皮内注射。
[0979]
本发明的肽除了用于治疗癌症,也可用于诊断。由于肽由卵巢癌样本产生,并且已确定这些肽在正常组织中不存在或水平较低,因此这些肽可用于诊断癌症是否存在。
[0980]
血液样本中组织活检物含权利要求的肽,可有助于病理师诊断癌症。用抗体、质谱或其他本领域内已知的方法检测某些肽可使病理师判断该组织样本为恶性的还是炎症或一般病变,也可用作卵巢癌的生物标志物。肽基团的提呈使得能对病变组织进行分类或进一步分成子类。
[0981]
对病变标本中肽的检测使得能对免疫系统治疗方法的利益进行判断,特别是如果t
‑
淋巴细胞已知或预计与作用机制有关。mhc表达的缺失是一种机制,充分说明了哪些受感染的恶性细胞逃避了免疫监视。因此,肽的提呈表明,分析过的细胞并没有利用这种机制。
[0982]
本发明的肽可用于分析淋巴细胞对肽的反应(如t细胞反应),或抗体对肽或mhc分子络合的肽发生的反应。这些淋巴细胞反应可以作为预后指标,决定是否采取进一步的治疗。这些反应也可以用作免疫疗法中的替代反应指标,旨在以不同方式诱导淋巴细胞反应,如接种蛋白疫苗、核酸、自体材料、淋巴细胞过继转移。基因治疗中,淋巴细胞对肽发生的反应可以在副作用的评估中考虑。淋巴细胞反应监测也可能成为移植疗法随访检查中的一种有价值的工具,如,用于检测移植物抗宿主和宿主抗移植物疾病。
附图说明
[0983]
下列描述优选方案的实施例将对本发明进行说明,并参照随附图表(但是不仅限
于此)。考虑到本发明的目的,文中引用的所有参考文献透过引用的方式并入在本文中。
[0984]
图1a 至1ae显示了正常组织(白色柱)和卵巢癌(黑色柱)中各种肽的过量提呈。图1a)基因符号:clsr2,肽:vlvsdgvhsv(seq id no.:6);从左至右的组织:1脂肪组织,3肾上腺,6动脉,5骨髓,7大脑,3乳房,1中枢神经,13结肠,1十二指肠,8食管,2胆囊,5心脏,16肾脏,2淋巴结,21肝,46肺,1淋巴结转移,4白细胞样本,7胰腺,4周围神经,1腹膜,3垂体腺,2胎盘,3胸膜,3前列腺,6直肌,7唾液腺,3骨骼肌肉,5皮肤,2小肠,4脾脏,7胃,4睾丸,3胸腺,4甲状腺,7气管,3输尿管,6膀胱,2子宫,2静脉,3卵巢,20oc。该肽还在1/6乳腺癌,1/2梅克尔细胞癌,3/17食管癌,3/91肺癌,10/29脑癌,1/22肾癌和1/15小细胞肺癌中检测出(未列出)。图1b)基因符号:ccna1,肽:slmeppavlll(seq id no.:1);从左至右的组织:1脂肪组织,3肾上腺,6动脉,5骨髓,7大脑,3乳房,1中枢神经,13结肠,1十二指肠,8食管,2胆囊,5心脏,16肾脏,2淋巴结,21肝,46肺,1淋巴结转移,4白细胞样本,7胰腺,4周围神经,1腹膜,3垂体腺,2胎盘,3胸膜,3前列腺,6直肌,7唾液腺,3骨骼肌肉,5皮肤,2小肠,4脾脏,7胃,4睾丸,3胸腺,4甲状腺,7气管,3输尿管,6膀胱,2子宫,2静脉,3卵巢,20oc。该肽还在1/2aml,1/28结直肠癌,2/17食管癌,7/91肺癌,1/29脑癌,1/22肾癌和2/15小细胞肺癌中检测出(未列出)。图1c)基因符号:vtcn1,肽:allplspyl(seq id no.:427);从左至右的组织:1脂肪组织,3肾上腺,6动脉,5骨髓,7大脑,3乳房,1中枢神经,13结肠,1十二指肠,8食管,2胆囊,5心脏,16肾脏,2淋巴结,21肝,46肺,1淋巴结转移,4白细胞样本,7胰腺,4周围神经,1腹膜,3垂体腺,2胎盘,3胸膜,3前列腺,6直肌,7唾液腺,3骨骼肌肉,5皮肤,2小肠,4脾脏,7胃,4睾丸,3胸腺,4甲状腺,7气管,3输尿管,6膀胱,2子宫,2静脉,3卵巢,20oc。该肽还在4/43前列腺癌,3/6乳腺癌,4/16肝癌,1/17食管癌,4/19胰腺癌,19/91肺癌,1/15小细胞肺癌,1/4膀胱癌和3/4子宫癌中检测出(未列出)。图1d)基因符号:ap1b1,肽:fldtlkdli seq id no.:514);从左至右的组织:6细胞系(1淋巴细胞,1肾脏,1胰腺,2pbmc,k562
‑
a2),4正常组织(2骨髓,2脾脏),49癌组织(1乳腺癌,3结肠癌,2食管癌,1胆囊癌,2白血病,3肝癌,21肺癌,7卵巢癌,23直肠癌,1皮肤癌,4胃癌,1睾丸癌,1膀胱癌)。正常组织系列和癌症细胞系和测试的异种移植物与图1a
‑
c相同,包括1脂肪组织,3肾上腺,6动脉,5骨髓,7大脑,3乳房,1中枢神经,13结肠,1十二指肠,8食管,2胆囊,5心脏,16肾脏,2淋巴结,21肝,46肺,1淋巴结转移,4白细胞样本,7胰腺,4周围神经,1腹膜,3垂体腺,2胎盘,3胸膜,3前列腺,6直肌,7唾液腺,3骨骼肌肉,5皮肤,2小肠,4脾脏,7胃,4睾丸,3胸腺,4甲状腺,7气管,3输尿管,6膀胱,2子宫,2静脉,3卵巢,20oc。该肽还在2/12慢性淋巴细胞白血病,5/28结肠癌,2/16肝癌,1/2黑色素瘤,2/17食管癌,17/91肺癌,4/46胃癌,4/15小细胞肺癌和1/4膀胱癌中检测到。图1d和表4之间的肿瘤类型相关的差异可能是由于表4采用更严格的选择标准所致(详情请参照表4)。图1d显示了可检测提呈肽y的所有样本,无论过度提呈参数和技术样本质量检查如何。图1e)基因符号:celsr2,肽:vlvsdgvhsv(seq id no.:6)。从左到右的组织:6脂肪组织,8肾上腺,24血细胞,15血管,10骨髓,13大脑,7乳房,9食管,2眼,3胆囊,16心脏,17肾脏,25大肠,24肝,49肺,7淋巴结,12神经,3卵巢,13胰腺,6甲状旁腺,1腹膜,6脑垂体,7胎盘,1胸膜,4前列腺,7唾液腺,9骨骼肌,11皮肤,9小肠,12脾,8胃,5睾丸,3胸腺,5甲状腺,16气管,7输尿管,8膀胱,6子宫,20卵巢癌样本。该肽还发现于15/34脑癌,3/18乳腺癌,3/18食管癌,4/12头颈部癌,1/23肾癌,6/107肺癌,5/18皮肤癌,5/15膀胱癌,3/16子宫癌。图1f)基因符号:suco,肽:lllditpei(seq id no.:143)。从左到右的组织:6脂肪组织,8肾上腺,24血细胞,
15血管,10骨髓,13大脑,7乳房,9食管,2眼,3胆囊,16心脏,17肾脏,25大肠,24肝,49肺,7淋巴结,12神经,3卵巢,13胰腺,6甲状旁腺,1腹膜,6脑垂体,7胎盘,1胸膜,4前列腺,7唾液腺,9骨骼肌,11皮肤,9小肠,12脾,8胃,5睾丸,3胸腺,5甲状腺,16气管,7输尿管,8膀胱,6子宫,20卵巢癌样本。该肽还发现于2/34脑癌,4/18乳腺癌,2/18食管癌,1/12头颈部癌,2/21肝癌,6/107肺癌,2/18皮肤癌,1/45胃癌,2/15膀胱癌。图1g)基因符号:plaur,肽:rlweegeelel(seq id no.:150)。从左到右的组织:6脂肪组织,8肾上腺,24血细胞,15血管,10骨髓,13大脑,7乳房,9食管,2眼,3胆囊,16心脏,17肾脏,25大肠,24肝,49肺,7淋巴结,12神经,3卵巢,13胰腺,6甲状旁腺,1腹膜,6脑垂体,7胎盘,1胸膜,4前列腺,7唾液腺,9骨骼肌,11皮肤,9小肠,12脾,8胃,5睾丸,3胸腺,5甲状腺,16气管,7输尿管,8膀胱,6子宫,20卵巢癌样本。该肽还发现于4/17胆囊和胆管癌症,1/18乳腺癌,1/29结肠癌,2/18食管癌,1/12头颈部癌,10/107肺癌,2/18皮肤癌,1/16子宫癌。图1h)基因符号:heatr2,肽:slndevpev(seq id no.:157)。从左到右的组织:6脂肪组织,8肾上腺,24血细胞,15血管,10骨髓,13大脑,7乳房,9食管,2眼,3胆囊,16心脏,17肾脏,25大肠,24肝,49肺,7淋巴结,12神经,3卵巢,13胰腺,6甲状旁腺,1腹膜,6脑垂体,7胎盘,1胸膜,4前列腺,7唾液腺,9骨骼肌,11皮肤,9小肠,12脾,8胃,5睾丸,3胸腺,5甲状腺,16气管,7输尿管,8膀胱,6子宫,20卵巢癌样本。该肽还发现于1/17胆管癌,5/34脑癌,1/18乳腺癌,1/29结肠癌,2/18食管癌,1/12头颈部癌,2/23肾癌,1/21肝癌,4/107肺癌,2/20淋巴结癌,1/18皮肤癌,1/15膀胱癌,1/16子宫癌。图1i)基因符号:vtcn1,肽:allplspyl(seq id no.:427)。从左到右的组织:6脂肪组织,8肾上腺,24血细胞,15血管,10骨髓,13大脑,7乳房,9食管,2眼,3胆囊,16心脏,17肾脏,25大肠,24肝,49肺,7淋巴结,12神经,3卵巢,13胰腺,6甲状旁腺,1腹膜,6脑垂体,7胎盘,1胸膜,4前列腺,7唾液腺,9骨骼肌,11皮肤,9小肠,12脾,8胃,5睾丸,3胸腺,5甲状腺,16气管,7输尿管,8膀胱,6子宫,20卵巢癌样本。该肽还发现于7/17胆囊和胆管癌,9/18乳腺癌,2/18食管癌,1/12头颈部癌,7/21肝癌,22/107肺癌,4/19胰脏癌,4/87前列腺癌,2/15膀胱癌,11/16子宫癌。图1j)基因符号:ddx11、ddx12p、loc642846,肽:gllrdealaev(seq id no.:444)。从左到右的组织:6脂肪组织,8肾上腺,24血细胞,15血管,10骨髓,13大脑,7乳房,9食管,2眼,3胆囊,16心脏,17肾脏,25大肠,24肝,49肺,7淋巴结,12神经,3卵巢,13胰腺,6甲状旁腺,1腹膜,6脑垂体,7胎盘,1胸膜,4前列腺,7唾液腺,9骨骼肌,11皮肤,9小肠,12脾,8胃,5睾丸,3胸腺,5甲状腺,16气管,7输尿管,8膀胱,6子宫,20卵巢癌样本。该肽还发现于2/18乳腺癌,3/29结肠或直肠癌,1/18食管癌,1/12头颈部癌,1/23肾癌,2/17白细胞白血病癌症,9/107肺癌,6/20淋巴结癌,1/18骨髓细胞癌,2/18皮肤癌,2/15膀胱癌,1/16子宫癌。图1k)基因符号:kdm1b,肽:klaegldiql(seq id no.:449)。从左到右的组织:6脂肪组织,8肾上腺,24血细胞,15血管,10骨髓,13大脑,7乳房,9食管,2眼,3胆囊,16心脏,17肾脏,25大肠,24肝,49肺,7淋巴结,12神经,3卵巢,13胰腺,6甲状旁腺,1腹膜,6脑垂体,7胎盘,1胸膜,4前列腺,7唾液腺,9骨骼肌,11皮肤,9小肠,12脾,8胃,5睾丸,3胸腺,5甲状腺,16气管,7输尿管,8膀胱,6子宫,20卵巢癌样本。该肽还发现于3/29结肠或直肠癌,6/107肺癌,1/20淋巴结癌。图1l)基因符号:ccna1,肽:slmeppavlll(seq id no.:1)。从左到右的组织:1癌细胞系,1正常组织(1淋巴结),45癌组织(3骨髓癌,1脑癌,1乳腺癌,2食管癌,1头颈部癌,1肾癌,3白细胞白血病癌症,12肺癌,1骨髓细胞癌,11卵巢癌,2膀胱癌,7子宫癌)。测试的正常组织系列与图1e
‑
k中相同。图1m)基因符号:ct45a5、loc101060208、ct45a3、ct45a1、loc101060211、
ct45a6、ct45a4、loc101060210、ct45a2,肽:kifemlegv(seq id no.:11)。从左到右的组织:3正常组织(1脑,1肺,1输尿管),21癌组织(1胆管癌,1食管癌,1肝癌,10肺癌,1淋巴结癌,5卵巢癌,2子宫癌)。测试的正常组织系列与图1e
‑
k中相同。图1n)基因符号:fgfr1op,肽:klddltqdltv(seq id no.:32)。从左到右的组织:1细胞系,1正常组织(1肝),29癌组织(2胆管癌,1食管癌,2头颈部癌,4肝癌,4肺癌,3淋巴结癌,8卵巢癌,1前列腺癌,1直肠癌,2膀胱癌,1子宫癌)。测试的正常组织系列与图1e
‑
k中相同。图1o)基因符号:tsen15,肽:flleddihvs(seq id no.:38)。从左到右的组织:1原代培养物,1正常组织(1气管),28癌组织(2乳腺癌,1头颈部癌,4白细胞白血病癌症,5肺癌,6淋巴结癌,1骨髓细胞癌,2卵巢癌,1直肠癌,3皮肤癌,2膀胱癌,1子宫癌)。测试的正常组织系列与图1e
‑
k中相同。图1p)基因符号:znf527、znf829、znf383、znf850、znf583,肽:slleqgkepwmv(seq id no.:54)。从左到右的组织:1细胞系,18癌组织(2脑癌,1乳腺癌,1胆囊癌,1白细胞白血病癌症,2肝癌,7肺癌,1淋巴结癌,2卵巢癌,1膀胱癌)。测试的正常组织系列与图1e
‑
k中相同。图1q)基因符号:camsap1,肽:tlaelqppvql(seq id no.:57)。从左到右的组织:4细胞系和原代培养物,32癌组织(1胆管癌,1脑癌,2食管癌,3头颈部癌,2白细胞白血病癌症,1肝癌,9肺癌,4淋巴结癌,5卵巢癌,2皮肤癌,1膀胱癌,1子宫癌)。测试的正常组织系列与图1e
‑
k中相同。图1r)基因符号:stk38l,肽:ilveadgawvv(seq id no.:77)。从左到右的组织:4细胞系,19癌组织(1脑癌,2乳腺癌,1结肠癌,1白细胞白血病癌症,4肺癌,3淋巴结癌,3卵巢癌,1前列腺癌,1皮肤癌,1膀胱癌,1子宫癌)。测试的正常组织系列与图1e
‑
k中相同。图1s)基因符号:piga,肽:alnpeivsv(seq id no.:148)。从左到右的组织:3细胞系,20癌组织(1食管癌,2头颈部癌,1白细胞白血病癌症,5肺癌,3淋巴结癌,2卵巢癌,2皮肤癌,4膀胱癌)。测试的正常组织系列与图1e
‑
k中相同。图1t)基因符号:nploc4,肽:ylnhleppv(seq id no.:166)。从左到右的组织:2细胞系,20癌组织(3脑癌,1乳腺癌,1食管癌,3白细胞白血病癌症,2肝癌,4肺癌,2淋巴结癌,1骨髓细胞癌,3卵巢癌)。测试的正常组织系列与图1e
‑
k中相同。图1u)基因符号:rnf213,肽:ylmdingkmwl(seq id no.:184)。从左到右的组织:1细胞系,19癌组织(1乳腺癌,1胆囊癌,1白细胞白血病癌症,6肺癌,2淋巴结癌,5卵巢癌,2皮肤癌,1子宫癌)。测试的正常组织系列与图1e
‑
k中相同。图1v)基因符号:skil,肽:ktinkvptv(seq id no.:198)。从左到右的组织:2细胞系和原代培养物,1正常组织(1气管),36癌组织(3脑癌,2乳腺癌,2结肠癌,1头颈部癌,1肝癌,14肺癌,1淋巴结癌,8卵巢癌,1直肠癌,2膀胱癌,1子宫癌)。测试的正常组织系列与图1e
‑
k中相同。图1w)基因符号:sec24c,肽:flfpnqyvdv(seq id no.:248)。从左到右的组织:3细胞系和原代培养物,1正常组织(1脾),24癌组织(1胆管癌,2乳腺癌,2白细胞白血病癌症,1肝癌,9肺癌,2淋巴结癌,3卵巢癌,1前列腺癌,2皮肤癌,1子宫癌)。测试的正常组织系列与图1e
‑
k中相同。图1x)基因符号:pdik1l、stk35,肽:allenpkmel(seq id no.:441)。从左到右的组织:5细胞系和原代培养物,1正常组织(1肾上腺),26癌组织(1乳腺癌,1结肠癌,1食管癌,1头颈部癌,2肝癌,10肺癌,5卵巢癌,1前列腺癌,1直肠癌,2膀胱癌,1子宫癌)。测试的正常组织系列与图1e
‑
k中相同。图1y)基因符号:emc10,肽:slveshlsdqltl(seq id no.:463)。从左到右的组织:1原代培养物,32癌组织(1胆管癌,2脑癌,2乳腺癌,2头颈部癌,3白细胞白血病癌症,1肝癌,8肺癌,3淋巴结癌,5卵巢癌,2皮肤癌,2膀胱癌,1子宫癌)。测试的正常组织系列与图1e
‑
k中相同。图1z)基因符号:zyg11a,肽:vlianlekl(seq id no.:466)。从左到右的组织:5细胞系,17癌组织(3乳腺癌,2食管癌,1肝
癌,2肺癌,5淋巴结癌,3卵巢癌,1膀胱癌)。测试的正常组织系列与图1e
‑
k中相同。图1aa)基因符号:cep192,肽:slfgnsgilenv(seq id no.:479)。从左到右的组织:7细胞系,1正常组织(1脾),33癌组织(1乳腺癌,1结肠癌,1食管癌,1头颈部癌,1白细胞白血病癌症,3肝癌,10肺癌,1淋巴结癌,1骨髓细胞癌,7卵巢癌,2皮肤癌,3膀胱癌,1子宫癌)。测试的正常组织系列与图1e
‑
k中相同。图1ab)基因符号:ccna1,肽:slseivpcl(seq id no.:512)。从左到右的组织:9癌组织(1头颈部癌,2肺癌,1骨髓细胞癌,3卵巢癌,2子宫癌)。测试的正常组织系列与图1e
‑
k中相同。图1ac)基因符号:gnb1,肽:alwdietgqqttt(seq id no.:560),从左至右的组织:5细胞系和原代培养物,26癌组织(1脑癌,1食管癌,1食管和胃癌,1胆囊癌,2头颈部癌,1白细胞白血病癌症,1肝癌,5肺癌,6淋巴结癌,3卵巢癌,1前列腺癌,1皮肤癌,1膀胱癌,1子宫癌)。测试的正常组织系列与图1e
‑
k中相同。图1ad)基因符号:klhl14,肽:vmndrlyai(seq id no.:587),从左至右的组织:5正常组织(1胰腺,3脾脏,1甲状腺),38癌组织(14白细胞白血病癌症,10淋巴结癌,9卵巢癌,1前列腺癌,4子宫癌)。测试的正常组织系列与图1e
‑
k中相同。图1ae)基因符号:urb1,肽:kllnkiyea(seq id no.:620),从左至右的组织:3细胞系和原代培养物,2正常组织(1肺,1子宫),27癌组织(5脑癌,2乳腺癌,2食管癌,5肺癌,1淋巴结癌,1骨髓细胞癌,5卵巢癌,3前列腺癌,1直肠癌,1膀胱癌,1子宫癌)。测试的正常组织系列与图1e
‑
k中相同。
[0985]
图2a至2d显示了本发明的源基因的代表性表达特征,这些基因在一系列正常组织(白色柱)的卵巢癌中以及20个卵巢癌症样本(黑色柱)中高度过度表达或专门表达。从左到右的组织:7动脉,1脑,1心脏,2肝,2肺,2静脉,1脂肪组织,1肾上腺,4骨髓,1结肠,2食管,2胆囊,1肾,6淋巴结,1胰腺,1脑垂体,1直肠,1骨骼肌,1皮肤,1小肠,1脾,1胃,1胸腺,1甲状腺,5气管,1膀胱,1乳腺,3卵巢,3胎盘,1前列腺,1睾丸,1子宫。图2a)ct45a1、ct45a3、ct45a5、ct45a6、ct45a2、rp11
‑
342l5.1;图2b)cldn16;图2c)esr1;图2d)ido1。
[0986]
图3a至f显示了示例性的免疫原性资料:肽特定多聚体染色后流式细胞仪结果。cd8+ t细胞制备的方法为:使用抗cd28 mab和hla
‑
a*02涂层的人工apc分别与sseqid no 662肽(a,左图)、seqid no 663肽(b,左图)、seqid no 11肽(c,左图)、seqid no 198肽(d,左图)、seqid no 587肽(e,左图)和seqid no 427肽(f,左图)合成。经过3个周期的刺激后,用a*02/seqid no 662(a)、a*02/seqid no 663(b)、a*02/seqid no 11(c)、a*02/seqid no 198(d)、a*02/seqid no 587(e)或a*02/seqid no 427(f)的2d多聚体染色法对肽反应性细胞进行检测。右图(a、b、c、d、e和f)显示用不相关a*02/肽复合体刺激的细胞对照染色。活单细胞在cd8+淋巴细胞上得到门控。boolean门控帮助排除用不同肽特定的多聚体检测的假阳性事件。提示了特异性多聚体+细胞和cd8+淋巴细胞的频率。
具体实施方式
[0987]
实施例1
[0988]
细胞表面提呈的肿瘤相关肽的识别和定量
[0989]
组织样本
[0990]
患者的肿瘤组织获得自asterand(detroit,usaand royston,herts,uk)、val d201hebron大学医院(barcelona)、proteogenex inc.,(culver city,ca,usa)、stanford癌症中心(stanford,ca,usa)、蒂宾根大学医院。正常(健康)组织获得自asterand
(detroit,usa和royston,herts,uk)、bio
‑
options inc.,ca,usa、bioserve,beltsville,md,usa、capital bioscience inc.,rockville,md,usa、geneticist inc.,glendale,ca,usa、日内瓦大学医院、海德堡大学医院、慕尼黑大学医院、proteogenex inc.,culver city,ca,usa、德国蒂宾根大学医院、京都府立医科大学(kpum)。所有患者在手术或尸检前都获得了书面知情同意。切除后组织立即进行冷休克处理,在分离tumap前储存于
‑
70℃或以下。
[0991]
从组织样本中分离hla肽
[0992]
根据方案(falk et al.,1991;seeger et al.,1999)略加修改,使用hla
‑
a*02特异性抗体bb7.2、hla
‑
a、hla
‑
b、hlac特异性抗体w6/32、cnbr活化的琼脂糖凝胶、酸处理和超滤方法以免疫沉淀法从实体组织中获得了冷冻组织样本的hla肽库。
[0993]
质谱分析
[0994]
获得的hla肽库根据其疏水性用反相色谱(nanoacquity uplc system,waters)分离,洗脱肽用装有电喷雾源的ltq
‑
velos融合杂交质谱(thermoelectron)进行了分析。肽库被直接加载填充有1.7μm c18反相材料(waters)的分析用熔炼石英微毛细管柱(75μm内径x 250mm),应用流速为400nl每分钟。随后,使用来自流速为300nl每分钟、浓度为10%至33%溶剂b中的两步180分钟二元梯度法对肽进行分离。梯度由溶剂a(含0.1%甲酸的水)和溶剂b(含0.1%甲酸的乙腈)。金镀膜玻璃毛细管(picotip,new objective)用于引入到纳升电喷雾源。使用前5(top5)策略在数据依赖模式下操作ltq
‑
orbitrap质谱仪。简言之,首先以高精确质量完全扫描在orbitrap开始一个扫描周期(r=30 000),之后用先前选定离子的动态排除技术在orbitrap中对5种含量最为丰富的前体离子进行ms/ms扫描(r=7500)。串联质谱以sequest和另一种手动控制器进行解读。生成的自然肽破碎模式与合成序列相同参考肽的破碎模式进行比较后,确保了被识别的肽序列。
[0995]
无标记相对lc
‑
ms定量透过离子计数(即透过lc
‑
ms功能提取和分析)来进行(mueller et al.,2007)。该方法假定肽的lc
‑
ms信号区域与样本中其丰度相关。提取的特征透过充电状态去卷积和保留时间校准进行进一步处理(mueller et al.,2008;sturm et al.,2008)。最后,所有的lc
‑
ms特征与序列鉴定结果交叉引用,以将不同样本和组织的定量数据与肽呈递特征结合。定量数据根据集中数据以两层方式进行正态化处理,以说明技术和生物学复制变异。因此,每个被识别的肽均可与定量资料相关,从而可得出样本和组织之间的相对定量。此外,对候选肽获得的所有定量数据进行手动检查,以确保数据的一致性,并验证自动化分析的准确度。对于每种肽,计算了提呈图,其显示样本平均提呈量以及复制变化。这些特征使卵巢癌样本与正常组织样本的基线值并列。示范性过度提呈肽的提呈谱示于图1中。示范性肽的提呈分数见表8。
[0996]
表8:提呈分数。该表列出了与一系列正常组织相比在肿瘤上非常高度过量提呈(+++)、与一系列正常组织相比在肿瘤上高度过量提呈(++)或与一系列正常组织相比在肿瘤上过量提呈(+)的肽。系列正常组织包括:脂肪组织、肾上腺、动脉、静脉、骨髓、脑、中枢和周围神经、结肠、直肠、小肠(包括十二指肠)、食道、胆囊、心脏、肾、肝、肺、淋巴结、单核白细胞、胰腺、腹膜、垂体、胸膜、唾液腺、骨骼肌、皮肤、脾、胃、胸腺、甲状腺、气管、输尿管、膀胱。
[0997]
[0998]
[0999]
[1000]
[1001]
[1002]
[1003]
[1004]
[1005]
[1006]
[1007]
[1008]
[1009]
[1010]
[1011]
[1012]
[1013]
[1014]
[1015][1016]
实施例2
[1017]
编码本发明肽的基因的表达谱
[1018]
与正常细胞相比在肿瘤细胞上一种肽过度提呈或特定提呈足够其在免疫治疗中有效使用,一些肽为肿瘤特异性的,尽管存在其源蛋白也存在于正常组织中。但是,mrna表达谱增加了免疫治疗目标肽选择中其他级别的安全性。特别是对于具有高安全性风险的治疗选择,诸如亲和力成熟的tcr,理想的目标肽将来源于对该肿瘤独一无二且不出现于正常组织中的蛋白。
[1019]
rna来源与制备
[1020]
手术切除组织标本按如上所述(参见实施例1)在获得每名患者的书面知情同意后
提供。手术后立即速冻肿瘤组织标本,之后在液态氮中用杵臼匀浆。使用tri试剂(ambion公司,darmstadt,德国)之后用rneasy(qiagen公司,hilden,德国)清理从这些样本中制备总rna;这两种方法都根据制造商的方案进行。
[1021]
用于rnaseq实验来自健康人体组织的总rna获得自:asterand,detroit,usaand royston,herts,uk、proteogenex inc.culver city,ca,usa、geneticist inc.,glendale,ca,usa、istituto nazionale tumori"pascale",molecular biology and viral oncology unit(irccs),naples,italy、德国海德堡大学医院、biocat gmbh,heidelberg,germany。
[1022]
所有rna样本的质量和数量都在agilent 2100bioanalyzer分析仪(agilent公司,waldbronn,德国)上使用rna6000pico labchip kit试剂盒(agilent公司)进行评估。
[1023]
rnaseq实验
[1024]
透过新一代测序技术(rnaseq)由cegat(t
ü
bingen,germany)对肿瘤和正常组织的rna样本进行基因表达分析。简言之,根据供货商的方案(illumina inc.,san diego,ca,usa),其中包括rna碎片化、cdna转化和测序适配器的加入,利用illumina hiseq v4试剂盒准备测序文库。从多个样本获得的文库根据制造商的说明等摩尔混合并在illumina hiseq 2500序列发生器上测序,产生50bp的单端读数。处理的读数使用star软件映像至人类基因组(grch38)。根据ensembl序列数据库的说明(ensembl77),表达数据在转录水平设置为rpkm(每百万映射读数每千碱基读数,由cufflinks软件生成)并在外显子水平上设置(总读数,由bedtools软件生成)。外显子读数被归为外显子长度和校准尺寸,以获得rpkm值。
[1025]
本发明的代表性源基因在卵巢癌中高度过量表达的表达谱如图2a至2d所示。进一步代表性基因的表达分数见表9。
[1026]
表9:表达分数。该表列出了与一系列正常组织相比在肿瘤上非常高度过量表达(+++)、与一系列正常组织相比在肿瘤上高度过量表达(++)或与一系列正常组织相比在肿瘤上过量表达(+)的基因的肽。本得分基线根据以下正常组织的测量值计算:脂肪组织、肾上腺、动脉、骨髓、脑、结肠、食道、胆囊、心脏、肾、肝、肺、淋巴结、胰腺、垂体、直肠、骨骼肌、皮肤、小肠、脾、胃、胸腺、甲状腺、气管、膀胱、静脉。
[1027]
[1028]
[1029]
[1030][1031]
实施例3
[1032]
mhc
‑
i类提呈肽的体外免疫原性
[1033]
为了获得关于本发明tumap的免疫原性信息,发明人使用体外t细胞扩增分析方法进行了研究,其中该分析方法基于使用装载肽/mhc复合物和抗cd28抗体的人工抗原提呈细胞(aapc)进行反复刺激。用这种方法,发明人可显示出本发明目前为止22种hla
‑
a*0201限制tumap具有免疫原性,这表明这些肽为对抗人cd8+前体t细胞的t细胞表位(表11)。
[1034]
cd8+ t细胞体外启动
[1035]
为了用载有肽
‑
mhc复合物(pmhc)和抗cd28抗体的人工抗原提呈细胞进行体外刺
激,发明人首先从university clinics mannheim,germany中获取健康供体cd8微珠(miltenyi biotec,bergisch
‑
gladbach,germany)透过积极选择白细胞清除术后新鲜hla
‑
a*02产物而分离出cd8+ t细胞。
[1036]
pbmc和分离出的cd8+淋巴细胞使用前在t细胞培养基(tcm)中培养,培养基包括rpmi
‑
glutamax(invitrogen公司,karlsruhe,德国)并补充10%热灭活人ab血清(pan
‑
biotech公司,aidenbach,德国)、100u/ml青霉素/100μg/ml链霉素(cambrex公司,cologne,德国),1mm丙酮酸钠(cc pro公司,oberdorla,德国)和20μg/ml庆大霉素(cambrex公司)。在此步骤,2.5ng/ml的il
‑
7(promocell公司,heidelberg,德国)和10u/ml的il
‑
2(novartis pharma公司,n
ü
rnberg,德国)也加入tcm。
[1037]
对于pmhc/抗
‑
cd28涂层珠的生成、t细胞的刺激和读出,使用每刺激条件四个不同pmhc分子以及每个读出条件8个不同的pmhc分子在高度限定的体外系统中进行。
[1038]
纯化的共刺激小鼠igg2a抗人cd28抗体9.3(jung et al.,1987)使用制造商(perbio公司,波恩,德国)推荐的n
‑
羟基琥珀酰亚胺生物素进行化学生物素化处理。所用珠为5.6μm的链霉抗生物素蛋白包裹的多聚苯乙烯颗粒(bangs labooratories,伊利诺伊州,美国)。
[1039]
用于阳性和阴性对照刺激物的pmhc分别为a*0201/mla
‑
001(从melan
‑
a/mart
‑
1中修饰制得的肽elagigiltv(seq id no.664))和a*0201/ddx5
‑
001(从ddx5中获得的yllpaivhi(seq id no.665))。
[1040]
800.000珠/200μl包裹于含有4x 12.5ng不同生物素
‑
pmhc的96孔板、进行洗涤,随后加入体积为200μl的600ng生物素抗
‑
cd28。在37℃下,在含5ng/ml il
‑
12(promocell)的200μl tcm中共培养1x10
6 cd8+ t细胞与2x105的清洗涂层珠3天,从而启动刺激。之后,一半培养基与补充80u/ml il
‑
2的新鲜tcm进行交换,并且培养在37℃下持续4天。这种刺激性周期总共进行3次。对于使用每条件8种不同pmhc分子的pmhc多聚体读出,二维组合编码方法如前述使用(andersen et al.,2012),稍作修饰,涵盖耦合至5种不同的荧光染料。最后,用live/dead near ir染料(invitrogen公司,karlsruhe,德国)、cd8
‑
fitc抗体克隆sk1(bd公司,heidelberg,德国)和荧光pmhc多聚体而执行多聚体分析。对于分析,使用了配有合适激光仪和筛检程序的bd lsrii sorp细胞仪。肽特异性细胞以占总cd8+细胞的百分比形式进行计算。多聚体分析结果使用flowjo软件(tree star公司,oregon,美国)进行评估。特定多聚体+cd8+淋巴细胞的体外填装用与阴性对照刺激组比较而进行检测。如果健康供体中的至少一个可评价的体外刺激孔在体外刺激后发现含有特异性cd8+ t细胞株(即该孔包含至少1%特定多聚体+cd8+ t细胞,并且特定多聚体+的百分比至少为阴性对照刺激中位数的10倍),则检测给定抗原的免疫原性。
[1041]
卵巢癌肽体外免疫原性
[1042]
对于受到测试的hla
‑
i类肽,可透过肽特异性t细胞株的生成证明其体外免疫原性。tumap特异性多聚体对本发明的2种肽染色后流式细胞仪检测的典型结果如图3所示,同时也含有相应的阴性对照信息。本发明6种肽的结果汇总于表10a和b。
[1043]
表10a:本发明中hlai类肽的体外免疫原性。申请人对本发明的肽所做的体外免疫原性实验的示例性结果。<20%=+;20%
‑
49%=++;50%
‑
69%=+++;>=70%=++++
[1044]
seq id no:序列孔供体
283alyigdgyvihla++++648llwgnaifl+++++652tlwyrapev+++++++659ilfpdiiara++++662kiqeiltqv++++663kiqemqhfl++++
[1045]
表10b:本发明其他hla
‑
i类肽的体外免疫原性。
[1046]
申请人对本发明的hla
‑
a*02限制肽所做的体外免疫原性实验的示例性结果。提示了体外免疫原性实验的结果。阳性孔和供体(其他可评价)的百分比概括为<20%=+;20%
‑
49%=++;50%
‑
69%=+++;>=70%=++++
[1047]
[1048][1049]
实施例4
[1050]
肽的合成
[1051]
所有的肽透过使用fmoc策略以标准、广为接受的固相肽合成法合成。每个肽的身份和纯度已使用质谱和rp
‑
hplc分析法确定。用冻干法(三氟乙酸盐)获得白色至类白色的肽,纯度为>50%。所有的tumap优选作为三氟乙酸盐或乙酸盐进行给药,其他药用盐形式也可以。
[1052]
实施例5
[1053]
mhc结合测定
[1054]
本发明基于t细胞疗法的候选肽进一步测试其mhc结合能力(亲和性)。单个肽
‑
mhc复合体透过uv
‑
配体交换产生,其中,紫外线敏感肽经紫外线照射后裂解,与分析的相关肽交换。只有能够有效地结合并稳定肽接受mhc分子的候选肽才能阻止mhc复合物的解离。为了确定交换反应的产率,将基于稳定mhc复合物轻链(β2m)的检测结果进行elisa测定。检测总体上按照rodenko等人在(rodenko et al.,2006)中描述的方法进行。
[1055]
96孔maxisorp板(nunc)在室温下在pbs中以2ug/ml链霉包被过夜,用4倍洗涤并在37℃下在含封闭缓冲液的2%bsa中封闭1小时。折迭的hla
‑
a*02:01/mla
‑
001单体作为标准品,涵盖15
‑
500ng/ml的范围。紫外线交换反应的肽
‑
mhc单体在封闭缓冲液中稀释100倍。样本在37℃下孵育1小时,洗涤四次,在37℃下以2ug/ml hrp缀合抗
‑
β2m温育1小时,再次洗涤,并以nh2so4封堵的tmb溶液进行检测。在450nm处测量吸收。在生成和产生抗体或其片段时和/或t细胞受体或其片段时,通常优选显示为高交换产率(优选为高于50%,最优选为高于75%)的候选肽,这是因为它们对mhc分子表现出足够的亲合力,并能防止mhc复合物的解离。
[1056]
表11:mhc
‑
i类结合分数。hla
‑
i类限制肽与hla
‑
a*02:01的结合根据肽交换产量分类:≥10%=+;≥20%=++;≥50=+++;≥75%=++++
[1057]
[1058]
[1059]
[1060]
[1061]
[1062]
[1063]
[1064]
[1065]
[1066]
[1067]
[1068]
[1069]
[1070]
[1071]
[1072]
[1073]
[1074]
[1075]
[1076]
[1077]
[1078]
[1079][1080][1081]
实施例6
[1082]
细胞表面提呈的肿瘤相关肽的绝对定量
[1083]
黏合剂例如抗体和/或tcr的产生是一个费力的过程,其可以仅针对一些选定靶标进行。在肿瘤相关和特异性肽的情况下,选择标准包括但不限于排除提呈于细胞表面上肽的提呈和浓度。除了如实施例1中所述肽的分离和相对定量,发明人也分析了每个细胞的绝对肽拷贝数,如专利申请pct/ep2015/79873所述。实体肿瘤样本中每个细胞的tumap拷贝数定量需要分离tumap的绝对定量、tumap分离效率和分析的组织样本细胞计数。实验步骤如下所述。
[1084]
nanolc
‑
ms/ms肽定量
[1085]
对于透过质谱法对肽的准确定量,使用内标法生成每种肽的校准曲线。内标是每种肽的双同位素标记的变体,即,tumap合成中纳入2个同位素标记的氨基酸。它与肿瘤相关肽仅在质量上不同,但在其他的物理化学性质方面无差异(anderson et al.,2012)。内标被掺入到每个ms样本,所有ms信号均标准化为内标ms信号,以平衡ms实验之间潜在的技术差异。
[1086]
校准曲线用至少三种不同的矩阵绘制,即,来自于类似于常规ms样本的天然样本的hla肽洗脱液,并且每个制备品以重复ms运行进行测量。对于评价,ms信号被标准化为内标信号,校准曲线透过logistic回归计算。
[1087]
对于来自组织样本的肿瘤相关肽的定量,各样本也掺有内标;ms信号标准化为内标并使用该肽校正曲线进行定量。
[1088]
肽/mhc分离的效率
[1089]
对于任何蛋白质纯化过程,来自组织样本蛋白的分离与相关蛋白的一定损失相关联。为了确定tumap分离的效率,针对选定为绝对定量的所有tumap产生了肽/mhc复合体。为了能够天然肽mhc/复合体与加样物,使用了单同位素标记版本的tumap,即tumap合成期间纳入1个同位素标记的氨基酸。这些复合物被掺入新制备的组织裂解物中,例如,在tumap分离过程中最早可能时间点,然后在之后的亲和纯化中像天然肽mhc/复合物被获取。因此,测量单标记tumap的恢复可得到个体tumap分离效率相关的结论。
[1090]
分离效率使用少量样本进行分析,且这些组织样本可比较。与此相反,个体肽之间的分离效率不同。这表明,分离效率虽然只在有限数量的样本中进行测定,但可外推至任何其他组织制备品中。但是,由于分离效率不能从一种肽外推至其他肽,因此,有必要单独分析每个tumap。
[1091]
固体、冷冻组织中细胞计数的测定
[1092]
为了确定经过绝对肽定量的组织样本的细胞数,发明人采用了dna 含量分析。此方法适用于不同来源的广泛样本,最重要的是,冷冻样本(alcoser et al.,2011;forsey and chaudhuri,2009;silva et al.,2013)。在肽分离方案期间,组织样本被加工为均匀的裂解物,从中取一小等份裂解物。样本等分为三份,从中分离dna被分离(qiaamp dna mini kit,qiagen,hilden,德国)。每次dna 分离的总dna含量至少重复两次使用基于荧光的dna定量测定法(qubit dsdna hs assay kit,life technologies,darmstadt,德国)进行定量。
[1093]
为了计算细胞数,生成了来自单个健康血细胞等分试样的dna标准曲线,使用一系列指定的细胞数。标准曲线用于计算每次dna分离总dna含量的总细胞含量。用于肽分离的
组织样本的平均总细胞计数,在考虑裂解物等份的已知体积和总裂解物体积的情况下进行推算。
[1094]
每细胞的肽拷贝数
[1095]
使用前述实验的数据,发明人以总肽量除以样本总细胞计数计算得出每个细胞的tumap拷贝数,随后除以分离效率。选定肽的细胞拷贝数如表12所示。
[1096]
表12:绝对拷贝数。该表列出了nsclc肿瘤样本中绝对肽定量的结果。针对每种肽,每个细胞的中位拷贝数表示:<100=+;>=100=++;>=1,000+++;>=10,000=++++.提示样本数量,其中提供评估的高质量ms资料。
[1097][1098][1099]
参考文献列表
[1100]
nature 511(2014):543
‑
550
[1101]
abba,m.c.et al.,mol.cancer res 5(2007):881
‑
890
[1102]
abdelmalak,c.a.et al.,clin lab 60(2014):55
‑
61
[1103]
abele,r.et al.,essays biochem.50(2011):249
‑
264
[1104]
abetamann,v.et al.,clin cancer res 2(1996):1607
‑
1618
[1105]
abuhusain,h.j.et al.,j biol chem 288(2013):37355
‑
37364
[1106]
adam,a.p.et al.,cancer res 69(2009):5664
‑
5672
[1107]
addou
‑
klouche,l.et al.,mol.cancer 9(2010):213
[1108]
adelaide,j.et al.,cancer res 67(2007):11565
‑
11575
[1109]
adelman,c.a.et al.,nature 502(2013):381
‑
384
[1110]
adhikary,s.et al.,cell 123(2005):409
‑
421
[1111]
agarwal,a.k.et al.,j lipid res 51(2010):2143
‑
2152
[1112]
agarwal,n.et al.,oncogene 32(2013):462
‑
470
[1113]
agesen,t.h.et al.,gut 61(2012):1560
‑
1567
[1114]
ahangari,f.et al.,med.oncol 31(2014):173
[1115]
ahsan,s.et al.,acta neuropathol.commun.2(2014):59
[1116]
aissani,b.et al.,genes immun.15(2014):424
‑
429
[1117]
aissani,b.et al.,fertil.steril.103(2015):528
‑
534
[1118]
ajiro,m.et al.,int.j oncol 35(2009):673
‑
681
[1119]
ajiro,m.et al.,int.j oncol 37(2010):1085
‑
1093
[1120]
akao,y.et al.,cancer res 55(1995):3444
‑
3449
[1121]
akino,k.et al.,cancer sci.98(2007):88
‑
95
[1122]
akisawa,y.et al.,virchows arch.442(2003):66
‑
70
[1123]
al
‑
haidari,a.a.et al.,int.j colorectal dis.28(2013):1479
‑
1487
[1124]
albulescu,r.,biomark.med.7(2013):203
[1125]
alimirah,f.et al.,mol.cancer res 5(2007):251
‑
259
[1126]
allen,t.et al.,cancer res 66(2006):1294
‑
1301
[1127]
allera
‑
moreau,c.et al.,oncogenesis.1(2012):e30
[1128]
allison,j.p.et al.,science 270(1995):932
‑
933
[1129]
alpizar
‑
alpizar,w.et al.,int.j cancer 131(2012):e329
‑
e336
[1130]
alvarez,j.v.et al.,cancer cell 24(2013):30
‑
44
[1131]
aly,r.m.et al.,blood cells mol.dis.53(2014):185
‑
188
[1132]
amini,s.et al.,anat.cell biol 47(2014):1
‑
11
[1133]
amos,c.i.et al.,hum.mol.genet.20(2011):5012
‑
5023
[1134]
an,c.h.et al.,pathol.oncol res 21(2015):181
‑
185
[1135]
anchi,t.et al.,oncol lett.3(2012):264
‑
268
[1136]
andersen,c.l.et al.,br.j cancer 100(2009):511
‑
523
[1137]
andersen,j.b.et al.,br.j cancer 94(2006):1465
‑
1471
[1138]
andersen,j.n.et al.,sci.transl.med.2(2010):43ra55
[1139]
andersen,r.s.et al.,nat.protoc.7(2012):891
‑
902
[1140]
anderson,k.s.et al.,j proteome.res 10(2011):85
‑
96
[1141]
andrade,v.c.et al.,exp.hematol.37(2009):446
‑
449
[1142]
andrew,a.s.et al.,hum.genet.125(2009):527
‑
539
[1143]
angele,s.et al.,br.j cancer 91(2004):783
‑
787
[1144]
ansari,d.et al.,j cancer res clin oncol 141(2015):369
‑
380
[1145]
antony
‑
debre,i.et al.,cancer cell 27(2015):609
‑
611
[1146]
appay,v.et al.,eur.j immunol.36(2006):1805
‑
1814
[1147]
arai,a.et al.,cancer res 71(2011):4598
‑
4607
[1148]
arai,e.et al.,int.j cancer 135(2014):1330
‑
1342
[1149]
arbabian,a.et al.,febs j 280(2013):5408
‑
5418
[1150]
arbitrio,m.et al.,cancer chemother.pharmacol.77(2016):205
‑
209
[1151]
argani,p.et al.,clin cancer res 7(2001):3862
‑
3868
[1152]
arlt,a.et al.,oncogene 28(2009):3983
‑
3996
[1153]
arsenic,r.et al.,bmc.cancer 15(2015):784
[1154]
asahara,s.et al.,j transl.med.11(2013):291
[1155]
asmarinah,a.et al.,int.j oncol 45(2014):1489
‑
1496
[1156]
asou,n.et al.,blood 109(2007):4023
‑
4027
[1157]
aviles,velastegui j.et al.,minerva chir 46(1991):533
‑
537
[1158]
ayala,f.et al.,breast cancer res treat.80(2003):145
‑
154
[1159]
aylon,y.et al.,mol.oncol 5(2011):315
‑
323
[1160]
azimi,a.et al.,br.j cancer 110(2014):2489
‑
2495
[1161]
azzimonti,b.et al.,histopathology 45(2004):560
‑
572
[1162]
babron,m.c.et al.,carcinogenesis 35(2014):1523
‑
1527
[1163]
bachmann,s.b.et al.,mol cancer 13(2014):125
[1164]
bacsi,k.et al.,bmc.cancer 8(2008):317
[1165]
bagheri,f.et al.,mol.biol rep.41(2014):7387
‑
7394
[1166]
balakrishnan,a.et al.,hum.mutat.30(2009):1167
‑
1174
[1167]
baldini,e.et al.,andrologia 42(2010):260
‑
267
[1168]
balgkouranidou,i.et al.,clin chem lab med.51(2013):1505
‑
1510
[1169]
ball,a.r.,jr.et al.,mol.cell biol 22(2002):5769
‑
5781
[1170]
banat,g.a.et al.,plos.one.10(2015):e0139073
[1171]
banchereau,j.et al.,cell 106(2001):271
‑
274
[1172]
band,a.m.et al.,j mammary.gland.biol neoplasia.16(2011):109
‑
115
[1173]
bandoh,n.et al.,oncol rep.23(2010):933
‑
939
[1174]
bandres,e.et al.,oncol rep.12(2004):287
‑
292
[1175]
banerjee,r.et al.,nat commun.5(2014):4527
[1176]
bao,b.y.et al.,clin cancer res.17(2011):928
‑
936
[1177]
bar
‑
peled,l.et al.,science 340(2013):1100
‑
1106
[1178]
barbarulo,a.et al.,oncogene 32(2013):4231
‑
4242
[1179]
bargou,r.c.et al.,nat med.3(1997):447
‑
450
[1180]
bartlett,j.m.et al.,br.j cancer 113(2015):722
‑
728
[1181]
bauer,m.et al.,oncol rep.11(2004):677
‑
680
[1182]
bazzaro,m.et al.,am.j pathol.171(2007):1640
‑
1649
[1183]
beales,p.l.et al.,nephrol.dial.transplant.15(2000):1977
‑
1985
[1184]
beard,r.e.et al.,clin cancer res 19(2013):4941
‑
4950
[1185]
beatty,g.et al.,j immunol 166(2001):2276
‑
2282
[1186]
bednarska,k.et al.,immunobiology 221(2016):323
‑
332
[1187]
beggs,j.d.,nature 275(1978):104
‑
109
[1188]
behrens,p.et al.,anticancer res 21(2001):2413
‑
2417
[1189]
behrens,p.et al.,apoptosis.8(2003):39
‑
44
[1190]
bekker
‑
jensen,s.et al.,nat cell biol 12(2010):80
‑
86
[1191]
benada,j.et al.,biomolecules.5(2015):1912
‑
1937
[1192]
bender,c.et al.,int.j cancer 131(2012):e45
‑
e55
[1193]
benjamini,y.et al.,journal of the royal statistical society.series b(methodological),vol.57(1995):289
‑
300
[1194]
bennett,c.b.et al.,plos.one.3(2008):e1448
[1195]
berger,c.et al.,curr.mol.med.13(2013):1229
‑
1240
[1196]
bertherat,j.et al.,cancer res 63(2003):5308
‑
5319
[1197]
bessho,y.et al.,oncol rep.21(2009):263
‑
268
[1198]
bhan,s.et al.,oncol rep.28(2012):1498
‑
1502
[1199]
bhattacharya,c.et al.,mol cancer 11(2012):82
[1200]
bi,q.et al.,clin exp.metastasis 32(2015):301
‑
311
[1201]
bi,w.et al.,oncol rep.29(2013):1533
‑
1539
[1202]
bianchi,e.et al.,cancer res 54(1994):861
‑
866
[1203]
bidkhori,g.et al.,plos.one.8(2013):e67552
[1204]
bieche,i.et al.,int.j cancer 133(2013):2791
‑
2800
[1205]
bieniek,j.et al.,prostate 74(2014):999
‑
1011
[1206]
bierkens,m.et al.,genes chromosomes.cancer 52(2013):56
‑
68
[1207]
bilbao
‑
aldaiturriaga,n.et al.,pediatr.blood cancer 62(2015):766
‑
769
[1208]
bin amer,s.m.et al.,saudi.med.j 29(2008):507
‑
513
[1209]
bisgrove,d.a.et al.,j biol chem 275(2000):30668
‑
30676
[1210]
bish,r.et al.,mol.cells 37(2014):357
‑
364
[1211]
bisikirska,b.c.et al.,oncogene 32(2013):5283
‑
5291
[1212]
blanco,i.et al.,plos.one.10(2015):e0120020
[1213]
blenk,s.et al.,cancer inform.3(2007):399
‑
420
[1214]
blenk,s.et al.,bmc.cancer 8(2008):106
[1215]
bloch,d.b.et al.,j biol chem 271(1996):29198
‑
29204
[1216]
bock,a.j.et al.,hum.pathol.43(2012):669
‑
674
[1217]
bode,p.k.et al.,mod.pathol.27(2014):899
‑
905
[1218]
boehrer,s.et al.,hematol.j 2(2001):103
‑
107
[1219]
boehringer,j.et al.,biochem.j 448(2012):55
‑
65
[1220]
bogush,t.a.et al.,antibiot.khimioter.54(2009):41
‑
49
[1221]
boland,a.et al.,nat struct.mol.biol 20(2013):1289
‑
1297
[1222]
bombardieri,r.et al.,endocr.pract.19(2013):e124
‑
e128
[1223]
borel,f.et al.,hepatology 55(2012):821
‑
832
[1224]
bossi,d.et al.,mol.oncol 8(2014):221
‑
231
[1225]
boulter,j.m.et al.,protein eng 16(2003):707
‑
711
[1226]
bourdon,v.et al.,cancer res 62(2002):6218
‑
6223
[1227]
bourguignon,l.y.et al.,j biol chem 287(2012):32800
‑
32824
[1228]
brandacher,g.et al.,clin cancer res 12(2006):1144
‑
1151
[1229]
brandenberger,r.et al.,nat biotechnol.22(2004):707
‑
716
[1230]
braulke,t.et al.,arch.biochem.biophys.298(1992):176
‑
181
[1231]
braumuller,h.et al.,nature(2013)
[1232]
brendle,a.et al.,carcinogenesis 29(2008):1394
‑
1399
[1233]
brocke,k.s.et al.,cancer biol ther.9(2010):455
‑
468
[1234]
broderick,p.et al.,bmc.cancer 6(2006):243
[1235]
brody,j.r.et al.,cell cycle 8(2009):1930
‑
1934
[1236]
brossart,p.et al.,blood 90(1997):1594
‑
1599
[1237]
brouland,j.p.et al.,am.j pathol.167(2005):233
‑
242
[1238]
brown,c.o.et al.,leuk.res 37(2013):963
‑
969
[1239]
bruckdorfer,t.et al.,curr.pharm.biotechnol.5(2004):29
‑
43
[1240]
brule,h.et al.,biochemistry 43(2004):9243
‑
9255
[1241]
brynczka,c.et al.,bmc.genomics 8(2007):139
[1242]
bubnov,v.et al.,exp.oncol 34(2012):370
‑
372
[1243]
buch,s.c.et al.,mol carcinog.51 suppl 1(2012):e11
‑
e20
[1244]
budowle,b.et al.,cancer genet.cytogenet.5(1982):247
‑
251
[1245]
bueno,r.c.et al.,ann.oncol 25(2014):69
‑
75
[1246]
bugide,s.et al.,oncogene 34(2015):4601
‑
4612
[1247]
bujo,h.,rinsho byori 60(2012):469
‑
476
[1248]
bull,j.h.et al.,br.j cancer 84(2001):1512
‑
1519
[1249]
burger,h.et al.,leukemia 8(1994):990
‑
997
[1250]
burkhart,r.a.et al.,mol.cancer res 11(2013):901
‑
911
[1251]
burleigh,a.et al.,breast cancer res 17(2015):4
[1252]
burton,j.d.et al.,clin cancer res 10(2004):6606
‑
6611
[1253]
butz,h.et al.,clin chem 60(2014):1314
‑
1326
[1254]
caballero,o.l.et al.,plos.one.5(2010)
[1255]
caceres
‑
gorriti,k.y.et al.,plos.one.9(2014):e91000
[1256]
cahan,p.et al.,bmc.genomics 11(2010):638
[1257]
cai,h.et al.,plos.one.8(2013a):e57081
[1258]
cai,h.et al.,cell commun.signal.11(2013):31
[1259]
cai,k.et al.,lin.chung er.bi yan.hou tou.jing.wai ke.za zhi.26(2012):425
‑
428
[1260]
cai,w.et al.,cancer 119(2013b):1486
‑
1494
[1261]
caldarelli,a.et al.,leukemia 27(2013):2301
‑
2310
[1262]
calin,g.a.et al.,oncogene 19(2000):1191
‑
1195
[1263]
callahan,m.j.et al.,clin cancer res 14(2008):7667
‑
7673
[1264]
camgoz,a.et al.,leuk.lymphoma 54(2013):1279
‑
1287
[1265]
campone,m.et al.,breast cancer res treat.109(2008):491
‑
501
[1266]
cantara,s.et al.,j clin endocrinol.metab 97(2012):4253
‑
4259
[1267]
cao,j.x.et al.,cell death.dis.5(2014):e1426
[1268]
cao,l.et al.,biochem.biophys.res commun.333(2005):1050
‑
1059
[1269]
cappellari,m.et al.,oncogene 33(2014):3794
‑
3802
[1270]
card,k.f.et al.,cancer immunol immunother.53(2004):345
‑
357
[1271]
caren,h.et al.,bmc.cancer 11(2011):66
[1272]
carrascosa,c.et al.,oncogene 31(2012):1521
‑
1532
[1273]
carton,j.m.et al.,j histochem.cytochem.51(2003):715
‑
726
[1274]
cascon,a.et al.,j natl.cancer inst.107(2015)
[1314]
chen,l.et al.,cancer res 65(2005):5599
‑
5606
[1315]
chen,l.c.et al.,mod.pathol.24(2011):175
‑
184
[1316]
chen,q.et al.,plos.one.9(2014e):e88386
[1317]
chen,r.et al.,cancer res 61(2001):654
‑
658
[1318]
chen,w.t.et al.,elife.4(2015d)
[1319]
chen,x.et al.,pathol.res pract.208(2012b):437
‑
443
[1320]
chen,x.et al.,med.oncol 31(2014f):865
[1321]
chen,x.p.et al.,asian pac.j cancer prev.15(2014g):7741
‑
7746
[1322]
chen,y.et al.,j cell biochem.100(2007):1337
‑
1345
[1323]
chen,y.et al.,am.j physiol lung cell mol.physiol 306(2014h):l797
‑
l807
[1324]
chen,y.et al.,int.j cancer 91(2001):41
‑
45
[1325]
chen,y.et al.,j hematol.oncol 2(2009c):37
[1326]
chen,y.et al.,oncogene 32(2013c):4941
‑
4949
[1327]
chen,y.et al.,onco.targets.ther.7(2014i):1465
‑
1472
[1328]
chen,y.t.et al.,int.j cancer 124(2009d):2893
‑
2898
[1329]
chen,y.t.et al.,proc.natl.acad.sci.u.s.a 102(2005):7940
‑
7945
[1330]
chen,z.t.et al.,int.j mol.sci.16(2015e):15497
‑
15530
[1331]
cheng,a.n.et al.,cancer lett.337(2013a):218
‑
225
[1332]
cheng,a.s.et al.,gastroenterology 144(2013b):122
‑
133
[1333]
cheng,l.et al.,gynecol.oncol 117(2010):159
‑
169
[1334]
cheng,s.et al.,int.j clin exp.pathol.7(2014):8118
‑
8126
[1335]
cheng,y.et al.,cancer genet.204(2011):375
‑
381
[1336]
cheng,y.et al.,clin transl.sci.8(2015a):320
‑
325
[1337]
cheng,z.et al.,j exp.clin cancer res 34(2015b):27
[1338]
chernikova,s.b.et al.,cancer res 72(2012):2111
‑
2119
[1339]
chevillard,g.et al.,blood 117(2011):2005
‑
2008
[1340]
chi,l.m.et al.,mol.cell proteomics.8(2009):1453
‑
1474
[1341]
chin,s.f.et al.,genome biol 8(2007):r215
[1342]
chittasupho,c.et al.,mol.pharm.7(2010):146
‑
155
[1343]
cho,h.j.et al.,dna cell biol 35(2016):71
‑
80
[1344]
cho,s.et al.,proc.natl.acad.sci.u.s.a 108(2011):20778
‑
20783
[1345]
choi,y.l.et al.,j thorac.oncol 9(2014):563
‑
566
[1346]
choi,y.w.et al.,int.j gynecol.cancer 17(2007):687
‑
696
[1347]
choschzick,m.et al.,hum.pathol.41(2010):358
‑
365
[1348]
chou,j.l.et al.,clin epigenetics.7(2015):1
[1349]
chowdhury,s.k.et al.,biochem.biophys.res commun.333(2005):1139
‑
1145
[1350]
chowdhury,s.k.et al.,free radic.res 41(2007):1116
‑
1124
[1351]
chu,x.et al.,biochem.biophys.res commun.447(2014):158
‑
164
[1352]
chuang,j.y.et al.,oncogene 31(2012):4946
‑
4959
[1353]
chung,f.y.et al.,j surg.oncol 102(2010):148
‑
153
[1354]
chung,k.y.et al.,hepatology 54(2011):307
‑
318
[1355]
cicek,m.s.et al.,hum.mol.genet.22(2013):3038
‑
3047
[1356]
cieply,b.et al.,cancer res 72(2012):2440
‑
2453
[1357]
ciruelos gil,e.m.,cancer treat.rev 40(2014):862
‑
871
[1358]
clarke,l.e.et al.,j cutan.pathol.36(2009):433
‑
438
[1359]
claudio,j.o.et al.,oncogene 20(2001):5373
‑
5377
[1360]
coe,h.et al.,int.j biochem.cell biol 42(2010):796
‑
799
[1361]
cohen,c.j.et al.,j mol recognit.16(2003a):324
‑
332
[1362]
cohen,c.j.et al.,j immunol 170(2003b):4349
‑
4361
[1363]
cohen,s.n.et al.,proc.natl.acad.sci.u.s.a 69(1972):2110
‑
2114
[1364]
cohen,y.et al.,hematology.19(2014):286
‑
292
[1365]
colak,d.et al.,plos.one.8(2013):e63204
[1366]
colas,e.et al.,int.j cancer 129(2011):2435
‑
2444
[1367]
colbert,l.e.et al.,cancer res 74(2014):2677
‑
2687
[1368]
cole,s.p.et al.,science 258(1992):1650
‑
1654
[1369]
coligan,j.e.et al.,current protocols in protein science(1995)
[1370]
colis,l.et al.,j med.chem 57(2014):4950
‑
4961
[1371]
colombetti,s.et al.,j immunol.176(2006):2730
‑
2738
[1372]
colombo,j.et al.,oncol rep.21(2009):649
‑
663
[1373]
condomines,m.et al.,j immunol.178(2007):3307
‑
3315
[1374]
confalonieri,s.et al.,oncogene 28(2009):2959
‑
2968
[1375]
cong,x.et al.,hum.pathol.45(2014):1370
‑
1378
[1376]
cook,j.et al.,oncogene 18(1999):1205
‑
1208
[1377]
coppola,d.et al.,j geriatr.oncol 5(2014):389
‑
399
[1378]
coradeghini,r.et al.,oncol rep.15(2006):609
‑
613
[1379]
corcoran,c.a.et al.,mol.cancer res 6(2008):795
‑
807
[1380]
cornelissen,m.et al.,bmc.cancer 3(2003):7
[1381]
couch,f.j.et al.,cancer res 65(2005):383
‑
386
[1382]
coupienne,i.et al.,lasers surg.med.43(2011):557
‑
564
[1383]
creancier,l.et al.,cancer lett.365(2015):107
‑
111
[1384]
cubillos
‑
rojas,m.et al.,j biol chem 289(2014):14782
‑
14795
[1385]
cuevas,i.c.et al.,cancer res 65(2005):5070
‑
5075
[1386]
cuevas,r.et al.,cancer res 73(2013):1400
‑
1410
[1387]
cui,d.x.et al.,world j gastroenterol.11(2005):1273
‑
1282
[1388]
cui,f.et al.,proteomics.6(2006):498
‑
504
[1389]
cui,l.h.et al.,med.oncol 29(2012):1837
‑
1842
[1390]
cui,x.et al.,oncogene 26(2007):4253
‑
4260
[1391]
cunliffe,h.e.et al.,am.j cancer res 2(2012):478
‑
491
[1392]
cunningham,j.d.et al.,am.j surg.173(1997):521
‑
522
[1393]
cunningham,j.m.et al.,br.j cancer 101(2009):1461
‑
1468
[1394]
curry,j.m.et al.,laryngoscope(2015)
[1395]
cvekl,a.,jr.et al.,eur.j cancer 40(2004):2525
‑
2532
[1396]
dadkhah,e.et al.,arch.iran med.16(2013):463
‑
470
[1397]
dahlman,k.b.et al.,plos.one.7(2012):e34414
[1398]
dajon,m.et al.,oncoimmunology 4(2015):e991615
[1399]
dalamaga,m.,med.hypotheses 79(2012):617
‑
621
[1400]
daly,r.j.et al.,oncogene 21(2002):5175
‑
5181
[1401]
dannenmann,s.r.et al.,cancer immunol.res.1(2013):288
‑
295
[1402]
danussi,c.et al.,cancer res 73(2013):5140
‑
5150
[1403]
das,a.et al.,j cell sci.127(2014):686
‑
699
[1404]
das,m.et al.,plos.one.8(2013a):e69607
[1405]
das,t.k.et al.,oncogene 32(2013b):3184
‑
3197
[1406]
dasari,v.k.et al.,j urol.165(2001):1335
‑
1341
[1407]
dasgupta,s.et al.,int.j oncol 41(2012):1405
‑
1410
[1408]
datta,m.w.et al.,appl.immunohistochem.mol.morphol.8(2000):210
‑
215
[1409]
davalieva,k.et al.,prostate 75(2015):1586
‑
1600
[1410]
david
‑
watine,b.,plos.one.6(2011):e22423
[1411]
davidson,b.et al.,j cell mol.med.15(2011):535
‑
544
[1412]
davydova,e.et al.,j biol chem 289(2014):30499
‑
30510
[1413]
de angelis,p.m.et al.,mol.cancer 5(2006):20
[1414]
de leon,f.c.et al.,childs nerv.syst.31(2015):141
‑
146
[1415]
de,paoli l.et al.,leuk.lymphoma 54(2013):1087
‑
1090
[1416]
de,s.et al.,cancer res 69(2009):8035
‑
8042
[1417]
debauve,g.et al.,cell mol life sci.65(2008):591
‑
604
[1418]
deighton,r.f.et al.,brain pathol.20(2010):691
‑
703
[1419]
delbove,j.et al.,epigenetics.6(2011):1444
‑
1453
[1420]
demelash,a.et al.,mol.biol cell 23(2012):2856
‑
2866
[1421]
demokan,s.et al.,int.j cancer 127(2010):2351
‑
2359
[1422]
deng,s.et al.,breast cancer res treat.104(2007):21
‑
30
[1423]
deng,y.c.et al.,ai.zheng.24(2005):680
‑
684
[1424]
dengjel,j.et al.,clin cancer res 12(2006):4163
‑
4170
[1425]
denkberg,g.et al.,j immunol 171(2003):2197
‑
2207
[1426]
desai,s.d.et al.,exp.biol med.(maywood.)237(2012):38
‑
49
[1427]
diao,c.y.et al.,asian pac.j cancer prev.15(2014):1817
‑
1822
[1428]
diefenbacher,m.e.et al.,j clin invest 124(2014):3407
‑
3418
[1429]
diggle,c.p.et al.,plos.genet.10(2014):e1004577
[1430]
disepio,d.et al.,proc.natl.acad.sci.u.s.a 95(1998):14811
‑
14815
[1431]
dobashi,y.et al.,int.j cancer 110(2004):532
‑
541
[1432]
dohn,l.h.et al.,urol.oncol 33(2015):165
‑
24
[1433]
doldan,a.et al.,mol.carcinog 47(2008a):235
‑
244
[1434]
doldan,a.et al.,mol.carcinog 47(2008b):806
‑
813
[1435]
domanitskaya,n.et al.,br.j cancer 111(2014):696
‑
707
[1436]
dominguez
‑
sanchez,m.s.et al.,bmc.cancer 11(2011):77
[1437]
donati,g.et al.,j cell sci.124(2011):3017
‑
3028
[1438]
dong,p.et al.,cancer lett.243(2006):120
‑
127
[1439]
dong,q.et al.,biomed.res int.2015(2015):156432
[1440]
dong,w.et al.,tumour.biol(2015)
[1441]
donnellan,r.et al.,faseb j 13(1999):773
‑
780
[1442]
dorman,s.n.et al.,mol.oncol(2015)
[1443]
dormeyer,w.et al.,j proteome.res 7(2008):2936
‑
2951
[1444]
douet
‑
guilbert,n.et al.,leuk.res 38(2014):1316
‑
1319
[1445]
downie,d.et al.,clin cancer res.11(2005):7369
‑
7375
[1446]
drazkowska,k.et al.,nucleic acids res 41(2013):3845
‑
3858
[1447]
du,c.et al.,gastric.cancer 18(2015a):516
‑
525
[1448]
du,l.et al.,tumori 101(2015b):384
‑
389
[1449]
du,y.et al.,int.j mol.sci.15(2014a):17065
‑
17076
[1450]
du,y.f.et al.,int.j clin exp.pathol.7(2014b):923
‑
931
[1451]
duan,x.l.et al.,zhongguo shi yan.xue.ye.xue.za zhi.21(2013):7
‑
11
[1452]
duarte
‑
pereira,s.et al.,sci.rep.4(2014):6311
[1453]
duex,j.e.et al.,exp.cell res 316(2010):2136
‑
2151
[1454]
dun,b.et al.,am.j transl.res 6(2013a):28
‑
42
[1455]
dun,b.et al.,int.j clin exp.pathol.6(2013b):2880
‑
2886
[1456]
dunn,g.p.et al.,proc.natl.acad.sci.u.s.a111(2014):1102
‑
1107
[1457]
dunphy,e.j.et al.,j immunother.28(2005):268
‑
275
[1458]
dunzendorfer,u.et al.,eur.urol.6(1980):232
‑
236
[1459]
durgan,j.et al.,j biol chem 286(2011):12461
‑
12474
[1460]
dusseau,c.et al.,int.j oncol 18(2001):393
‑
399
[1461]
duursma,a.et al.,mol.cell biol 25(2005):6937
‑
6947
[1462]
duvic,m.et al.,clin cancer res 6(2000):3249
‑
3259
[1463]
duvic,m.et al.,j invest dermatol.121(2003):902
‑
909
[1464]
dyrskjot,l.et al.,br.j cancer 107(2012):116
‑
122
[1465]
dzikiewicz
‑
krawczyk,a.et al.,j hematol.oncol 7(2014):43
[1466]
eggers,j.p.et al.,clin cancer res 17(2011):6140
‑
6150
[1467]
eldai,h.et al.,plos.one.8(2013):e76251
[1468]
elgohary,n.et al.,int.j oncol 46(2015):597
‑
606
[1469]
elias,d.et al.,oncogene 34(2015):1919
‑
1927
[1470]
ellison
‑
zelski,s.j.et al.,mol.cancer 9(2010):263
[1471]
emdad,l.et al.,neuro.oncol 17(2015):419
‑
429
[1472]
emmanuel,c.et al.,plos.one.6(2011):e17617
[1473]
endoh,h.et al.,j clin oncol 22(2004):811
‑
819
[1474]
enesa,k.et al.,adv.exp.med.biol.809(2014):33
‑
48
[1475]
eng,k.h.et al.,genes cancer 6(2015):399
‑
407
[1476]
enomoto,a.et al.,eur.j cancer 49(2013):3547
‑
3558
[1477]
epping,m.t.et al.,mol.cancer res 7(2009):1861
‑
1870
[1478]
er,t.k.et al.,j mol.med.(berl)(2016)
[1479]
erb,h.h.et al.,endocr.relat cancer 20(2013):677
‑
689
[1480]
erdogan,e.et al.,clin cancer res 15(2009):1527
‑
1533
[1481]
erenpreisa,j.et al.,exp.cell res 315(2009):2593
‑
2603
[1482]
escobar
‑
hoyos,l.f.et al.,mod.pathol.27(2014):621
‑
630
[1483]
esseghir,s.et al.,j pathol.210(2006):420
‑
430
[1484]
estrella,j.s.et al.,pancreas 43(2014):996
‑
1002
[1485]
ettahar,a.et al.,cell rep.4(2013):530
‑
541
[1486]
evans,t.j.et al.,plos.one.9(2014):e110255
[1487]
exertier,p.et al.,oncotarget.4(2013):2302
‑
2316
[1488]
ezponda,t.et al.,oncogene 32(2013):2882
‑
2890
[1489]
fackler,m.et al.,febs j 281(2014):2123
‑
2135
[1490]
fagin,j.a.,mol.endocrinol.16(2002):903
‑
911
[1491]
fairfield,k.m.et al.,int.j cancer 110(2004):271
‑
277
[1492]
falk,k.et al.,nature 351(1991):290
‑
296
[1493]
falvella,f.s.et al.,oncogene 27(2008):3761
‑
3764
[1494]
fan,j.et al.,clin cancer res 17(2011):2908
‑
2918
[1495]
fan,m.et al.,int.j clin exp.pathol.7(2014):6768
‑
6775
[1496]
fang,h.y.et al.,hum.pathol.43(2012):105
‑
114
[1497]
fang,k.p.et al.,asian pac.j cancer prev.15(2014):2655
‑
2661
[1498]
fang,z.et al.,j biol chem 288(2013):7918
‑
7929
[1499]
faried,l.s.et al.,mol.carcinog 47(2008):446
‑
457
[1500]
faried,l.s.et al.,oncol rep.16(2006):57
‑
63
[1501]
faronato,m.et al.,oncotarget.(2015)
[1502]
fasso,m.et al.,proc.natl.acad.sci.u.s.a 105(2008):3509
‑
3514
[1503]
feldmann,g.et al.,cancer res 70(2010):4460
‑
4469
[1504]
feng,h.et al.,j clin invest 124(2014a):3741
‑
3756
[1505]
feng,m.et al.,j clin invest 124(2014b):5291
‑
5304
[1506]
feng,x.et al.,neoplasma 62(2015a):592
‑
601
[1507]
feng,y.et al.,sci.rep.5(2015b):9429
[1508]
fernandez
‑
calotti,p.x.et al.,haematologica 97(2012):943
‑
951
[1509]
fernandez
‑
nogueira,p.et al.,oncotarget.7(2016):5313
‑
5326
[1510]
ferreira
‑
da
‑
silva,a.et al.,plos.one.10(2015):e0122308
[1511]
ferrero,s.et al.,histol.histopathol.30(2015):473
‑
478
[1512]
fevre
‑
montange,m.et al.,int.j oncol 35(2009):1395
‑
1407
[1513]
fevre
‑
montange,m.et al.,j neuropathol.exp.neurol.65(2006):675
‑
684
[1514]
fitzgerald,j.et al.,febs lett.517(2002):61
‑
66
[1515]
fluge,o.et al.,thyroid 16(2006):161
‑
175
[1516]
fokas,e.et al.,cell death.dis.3(2012):e441
[1517]
folgiero,v.et al.,oncotarget.5(2014):2052
‑
2064
[1518]
fong,l.et al.,proc.natl.acad.sci.u.s.a 98(2001):8809
‑
8814
[1519]
fortschegger,k.et al.,mol.cancer res 12(2014):595
‑
606
[1520]
fraga,m.f.et al.,cancer res 68(2008):4116
‑
4122
[1521]
frasor,j.et al.,mol.cell endocrinol.418 pt 3(2015):235
‑
239
[1522]
frias,c.et al.,lung cancer 60(2008):416
‑
425
[1523]
fry,a.m.et al.,j cell sci.125(2012):4423
‑
4433
[1524]
fu,a.et al.,mol.carcinog 51(2012):923
‑
929
[1525]
fu,d.y.et al.,tumour.biol(2015)
[1526]
fu,j.et al.,cancer sci.104(2013a):508
‑
515
[1527]
fu,m.et al.,int.j clin exp.pathol.6(2013b):2185
‑
2191
[1528]
fu,m.et al.,int.j clin exp.pathol.6(2013c):2515
‑
2522
[1529]
fu,z.et al.,breast cancer res treat.127(2011):265
‑
271
[1530]
fujimura,k.et al.,clin chim.acta 430(2014):48
‑
54
[1531]
fujitomo,t.et al.,cancer res 72(2012):4110
‑
4118
[1532]
fujiuchi,n.et al.,j biol chem 279(2004):20339
‑
20344
[1533]
fukasawa,m.et al.,j hum.genet.51(2006):368
‑
374
[1534]
fukushima,y.et al.,eur.j cancer 35(1999):935
‑
938
[1535]
fuqua,s.a.et al.,breast cancer res treat.144(2014):11
‑
19
[1536]
furukawa,t.et al.,sci.rep.1(2011):161
[1537]
furuta,j.et al.,cancer res 66(2006):6080
‑
6086
[1538]
gaba,r.c.et al.,j vasc.interv.radiol.26(2015):723
‑
732
[1539]
gabrilovich,d.i.et al.,nat med.2(1996):1096
‑
1103
[1540]
galamb,o.et al.,cell oncol 31(2009):19
‑
29
[1541]
gallmeier,e.et al.,gastroenterology 130(2006):2145
‑
2154
[1542]
gantsev,s.k.et al.,biomed.pharmacother.67(2013):363
‑
366
[1543]
gao,f.et al.,biochem.biophys.res commun.431(2013):610
‑
616
[1544]
gao,j.et al.,acta oncol 47(2008):372
‑
378
[1545]
gao,w.et al.,bmc.cancer 15(2015):367
[1546]
gao,y.b.et al.,nat genet.46(2014):1097
‑
1102
[1547]
gao,z.et al.,biochem.biophys.res commun.407(2011):271
‑
276
[1548]
garcia
‑
baquero,r.et al.,tumour.biol.35(2014):5777
‑
5786
[1549]
garritano,s.et al.,oncogenesis.2(2013):e54
[1550]
gattinoni,l.et al.,nat rev.immunol 6(2006):383
‑
393
[1551]
gatza,m.l.et al.,nat genet.46(2014):1051
‑
1059
[1552]
gaudineau,b.et al.,j cell sci.125(2012):4475
‑
4486
[1553]
ge,g.et al.,tumour.biol(2015)
[1554]
geiger,t.r.et al.,plos.one.9(2014):e111813
[1555]
gelebart,p.et al.,j biol chem 277(2002):26310
‑
26320
[1556]
gelsi
‑
boyer,v.et al.,br.j haematol.145(2009):788
‑
800
[1557]
gentile,m.et al.,oncogene 20(2001):7753
‑
7760
[1558]
geoffroy
‑
perez,b.et al.,int.j cancer 93(2001):288
‑
293
[1559]
georgiou,g.k.et al.,world j surg.oncol 11(2013):213
[1560]
ghosh,s.et al.,int.j cancer 123(2008):2594
‑
2604
[1561]
gibbs,d.c.et al.,cancer epidemiol.biomarkers prev.24(2015):992
‑
997
[1562]
gil
‑
henn,h.et al.,oncogene 32(2013):2622
‑
2630
[1563]
gilling,c.e.et al.,br.j haematol.158(2012):216
‑
231
[1564]
giuliano,c.j.et al.,biochim.biophys.acta 1731(2005):48
‑
56
[1565]
glaser,r.et al.,plos.one.6(2011):e25160
[1566]
gnjatic,s.et al.,proc natl.acad.sci.u.s.a100(2003):8862
‑
8867
[1567]
godkin,a.et al.,int.immunol 9(1997):905
‑
911
[1568]
going,j.j.et al.,gut 50(2002):373
‑
377
[1569]
gold,d.v.et al.,int.j clin exp.pathol.4(2010):1
‑
12
[1570]
goldenson,b.et al.,oncogene 34(2015):537
‑
545
[1571]
gong,x.et al.,plos.one.7(2012):e37137
[1572]
gonzalez,m.a.et al.,j clin oncol 21(2003):4306
‑
4313
[1573]
goodman,s.l.et al.,biol open.1(2012):329
‑
340
[1574]
goswami,a.et al.,mol.cell 20(2005):33
‑
44
[1575]
goto,y.et al.,j invest dermatol.130(2010):221
‑
229
[1576]
gou,w.f.et al.,oncol rep.31(2014):232
‑
240
[1577]
govindaraj,v.et al.,horm.mol.biol clin investig.9(2012):173
‑
178
[1578]
goyal,p.et al.,plos.one.6(2011):e16249
[1579]
grady,w.m.,cancer metastasis rev 23(2004):11
‑
27
[1580]
graff,l.et al.,cancer res 61(2001):2138
‑
2144
[1581]
grant,r.c.et al.,hum.genomics 7(2013):11
[1582]
green,m.r.et al.,molecular cloning,a laboratory manual 4th(2012)
[1583]
greenfield,e.a.,antibodies:a laboratory manual 2nd(2014)
[1584]
greif,p.a.et al.,leukemia 25(2011):821
‑
827
[1585]
greuber,e.k.et al.,nat rev cancer 13(2013):559
‑
571
[1586]
grieb,b.c.et al.,mol.cancer res 12(2014):1216
‑
1224
[1587]
grimm,m.et al.,bmc.cancer 13(2013):569
[1588]
grimmig,t.et al.,int.j oncol 47(2015):857
‑
866
[1589]
grinberg
‑
rashi,h.et al.,clin cancer res 15(2009):1755
‑
1761
[1590]
gronnier,c.et al.,biochim.biophys.acta 1843(2014):2432
‑
2437
[1591]
groth
‑
pedersen,l.et al.,plos.one.7(2012):e45381
[1592]
gruel,n.et al.,breast cancer res 16(2014):r46
[1593]
grumati,p.et al.,cancer discov 4(2014):394
‑
396
[1594]
gu,x.h.et al.,zhonghua fu chan ke.za zhi.44(2009):754
‑
759
[1595]
gu,y.et al.,mol.carcinog 55(2016):292
‑
299
[1596]
guan,g.et al.,arch.biochem.biophys.417(2003):251
‑
259
[1597]
guan,x.et al.,carcinogenesis 34(2013):812
‑
817
[1598]
gueddari,n.et al.,biochimie 75(1993):811
‑
819
[1599]
guerreiro,a.s.et al.,mol.cancer res 9(2011):925
‑
935
[1600]
guerrero,j.a.et al.,blood 124(2014):3624
‑
3635
[1601]
guerrero
‑
preston,r.et al.,oncol rep.32(2014):505
‑
512
[1602]
guin,s.et al.,j natl.cancer inst.106(2014)
[1603]
guirado,m.et al.,hum.immunol.73(2012):668
‑
672
[1604]
gultekin,y.et al.,j innate.immun.7(2015):25
‑
36
[1605]
guo,f.et al.,mol.biol rep.37(2010):3819
‑
3825
[1606]
guo,g.et al.,tumour.biol 35(2014):4017
‑
4022
[1607]
guo,j.t.et al.,zhonghua zhong.liu za zhi.31(2009):528
‑
531
[1608]
guo,s.et al.,drug des devel.ther.7(2013):1259
‑
1271
[1609]
guo,w.et al.,j mol.biol 412(2011):365
‑
378
[1610]
guo,x.et al.,tumour.biol 36(2015):1711
‑
1720
[1611]
gust,k.m.et al.,neoplasia.11(2009):956
‑
963
[1612]
gutierrez,m.l.et al.,plos.one.6(2011):e22315
[1613]
gutierrez
‑
camino,a.et al.,pediatr.res 75(2014):767
‑
773
[1614]
guyonnet,duperat,v et al.,biochem.j 305(pt 1)(1995):211
‑
219
[1615]
gylfe,a.e.et al.,int.j cancer 127(2010):2974
‑
2980
[1616]
hagenbuchner,j.et al.,front physiol 4(2013):147
[1617]
haidar,a.et al.,am.j case.rep.16(2015):87
‑
94
[1618]
halama,n.et al.,int.j oncol 31(2007):205
‑
210
[1619]
hall,c.l.et al.,j neurooncol.26(1995):221
‑
229
[1620]
hall,c.l.et al.,cell 82(1995):19
‑
26
[1621]
halldorsdottir,a.m.et al.,am.j hematol.87(2012):361
‑
367
[1622]
hammam,o.et al.,j egypt.soc.parasitol.44(2014):733
‑
740
[1623]
han,j.c.et al.,world j surg.oncol 13(2015a):5
[1624]
han,l.l.et al.,oncol rep.31(2014):2569
‑
2578
[1625]
han,y.et al.,cancer 119(2013):3436
‑
3445
[1626]
han,z.et al.,oncotarget.6(2015b):13149
‑
13163
[1627]
hansen
‑
petrik,m.b.et al.,cancer lett.175(2002):157
‑
163
[1628]
hao,j.et al.,oncol lett.9(2015):2525
‑
2533
[1629]
haque,m.a.et al.,j exp.med.195(2002):1267
‑
1277
[1630]
haridas,d.et al.,faseb j 28(2014):4183
‑
4199
[1631]
harken,jensen c.et al.,tumour.biol 20(1999):256
‑
262
[1632]
hartmann,t.b.et al.,int.j cancer 114(2005):88
‑
93
[1633]
hasegawa,h.et al.,arch.pathol.lab med.122(1998):551
‑
554
[1634]
hashimoto,t.et al.,febs j 277(2010):4888
‑
4900
[1635]
hast,b.e.et al.,cancer res 73(2013):2199
‑
2210
[1636]
hatfield,k.j.et al.,expert.opin.ther.targets.18(2014):1237
‑
1251
[1637]
hayama,s.et al.,cancer res 67(2007):4113
‑
4122
[1638]
hayashi,h.et al.,int.j cancer 126(2010):2563
‑
2574
[1639]
hayashi,j.et al.,int.j oncol 21(2002):847
‑
850
[1640]
hayashi,s.i.et al.,endocr.relat cancer 10(2003):193
‑
202
[1641]
hayatsu,n.et al.,biochem.biophys.res commun.368(2008):217
‑
222
[1642]
hazelett,c.c.et al.,plos.one.7(2012):e39602
[1643]
he,d.et al.,biomed.pharmacother.74(2015):164
‑
168
[1644]
he,h.et al.,j clin endocrinol.metab 98(2013):e973
‑
e980
[1645]
he,j.et al.,cancer biol ther.6(2007):76
‑
82
[1646]
he,y.et al.,mol carcinog.(2014)
[1647]
hedrick,e.d.et al.,j mol.signal.8(2013):10
[1648]
heeboll,s.et al.,histol.histopathol.23(2008):1069
‑
1076
[1649]
hegyi,k.et al.,pathobiology 79(2012):314
‑
322
[1650]
heidenblad,m.et al.,bmc.med.genomics 1(2008):3
[1651]
heim,s.et al.,in vivo 19(2005):583
‑
590
[1652]
heimerl,s.et al.,melanoma res 17(2007):265
‑
273
[1653]
hellerbrand,c.et al.,carcinogenesis 27(2006):64
‑
72
[1654]
hellwinkel,o.j.et al.,prostate cancer prostatic.dis.14(2011):38
‑
45
[1655]
hemminger,j.a.et al.,mod.pathol.27(2014):1238
‑
1245
[1656]
hennard,c.et al.,j pathol.209(2006):430
‑
435
[1657]
hennig,e.e.et al.,j mol.med.(berl)90(2012):447
‑
456
[1658]
hickinson,d.m.et al.,clin transl.sci.2(2009):183
‑
192
[1659]
hider,j.l.et al.,bmc.evol.biol.13(2013):150
[1660]
hinrichsen,i.et al.,plos.one.9(2014):e84453
[1661]
hirota,y.et al.,nucleic acids res 28(2000):917
‑
924
[1662]
hlavac,v.et al.,pharmacogenomics.14(2013):515
‑
529
[1663]
hlavata,i.et al.,mutagenesis 27(2012):187
‑
196
[1664]
ho,m.et al.,clin cancer res 13(2007):1571
‑
1575
[1665]
hodi,f.s.et al.,proc.natl.acad.sci.u.s.a 99(2002):6919
‑
6924
[1666]
hodson,i.et al.,int.j oncol 23(2003):991
‑
999
[1667]
hoei
‑
hansen,c.e.et al.,clin cancer res 10(2004):8521
‑
8530
[1668]
hoellein,a.et al.,j cancer res clin oncol 136(2010):403
‑
410
[1669]
hoff,a.m.et al.,oncotarget.(2015)
[1670]
holla,v.r.et al.,j biol chem 281(2006):2676
‑
2682
[1671]
holleman,a.et al.,blood 107(2006):769
‑
776
[1672]
holm,c.et al.,leuk.res 30(2006):254
‑
261
[1673]
holzmann,k.et al.,cancer res 64(2004):4428
‑
4433
[1674]
hong,j.et al.,biomed.res int.2013(2013):454085
[1675]
honore,b.et al.,oncogene 21(2002):1123
‑
1129
[1676]
hoque,m.o.et al.,cancer res 68(2008):2661
‑
2670
[1677]
horani,a.et al.,am j hum.genet.91(2012):685
‑
693
[1678]
horejsi,z.et al.,mol.cell 39(2010):839
‑
850
[1679]
horst,b.et al.,am.j pathol.174(2009):1524
‑
1533
[1680]
hosseini,m.,pol.j pathol.64(2013):191
‑
195
[1681]
hosseini,s.et al.,clin lab 61(2015):475
‑
480
[1682]
hou,g.et al.,cancer lett.253(2007):236
‑
248
[1683]
hou,j.et al.,mol.oncol 9(2015):1312
‑
1323
[1684]
hou,x.et al.,ann.surg.oncol 21(2014):3891
‑
3899
[1685]
hou,y.et al.,med.oncol 29(2012):3498
‑
3503
[1686]
hour,t.c.et al.,int.j biol markers 24(2009):171
‑
178
[1687]
hovnanian,a.,subcell.biochem.45(2007):337
‑
363
[1688]
hsu,p.k.et al.,j gastroenterol.49(2014):1231
‑
1240
[1689]
hsu,w.h.et al.,plos.one.10(2015):e0121298
[1690]
hu,h.et al.,oncol lett.10(2015):268
‑
272
[1691]
hu,j.et al.,exp.biol med.(maywood.)239(2014):423
‑
429
[1692]
hu,s.et al.,pediatr.hematol.oncol 28(2011):140
‑
146
[1693]
hua,c.et al.,bmc.cancer 14(2014):526
[1694]
huang,c.et al.,cell biol int.32(2008):1081
‑
1090
[1695]
huang,h.et al.,clin cancer res 11(2005a):4357
‑
4364
[1696]
huang,h.et al.,beijing da.xue.xue.bao.46(2014a):183
‑
189
[1697]
huang,h.et al.,int.j oncol 38(2011):1557
‑
1564
[1698]
huang,l.n.et al.,clin chim.acta 413(2012):663
‑
668
[1699]
huang,x.et al.,apmis 122(2014b):1070
‑
1079
[1700]
huang,y.et al.,int.j mol.sci.15(2014c):18148
‑
18161
[1701]
huang,y.et al.,oncogene 24(2005b):3819
‑
3829
[1702]
huang,y.et al.,oncotarget.5(2014d):6734
‑
6745
[1703]
hudlebusch,h.r.et al.,clin cancer res 17(2011):2919
‑
2933
[1704]
hudson,j.et al.,exp.mol.pathol.95(2013):62
‑
67
[1705]
hui,l.et al.,oncol rep.34(2015):2627
‑
2635
[1706]
hummerich,l.et al.,oncogene 25(2006):111
‑
121
[1707]
hunecke,d.et al.,j pathol.228(2012):520
‑
533
[1708]
hungermann,d.et al.,j pathol.224(2011):517
‑
528
[1709]
hunter,s.m.et al.,oncotarget.6(2015):37663
‑
37677
[1710]
hussein,y.m.et al.,med.oncol 29(2012):3055
‑
3062
[1711]
huynh,h.et al.,mol.cancer ther.14(2015):1224
‑
1235
[1712]
hwang,c.f.et al.,plos.one.8(2013):e84218
[1713]
hwang,j.m.et al.,mol.cell biochem.327(2009):135
‑
144
[1714]
hwang,m.l.et al.,j immunol.179(2007):5829
‑
5838
[1715]
iacovazzi,p.a.et al.,immunopharmacol.immunotoxicol.32(2010):160
‑
164
[1716]
iakovlev,v.et al.,cancer epidemiol.biomarkers prev.21(2012):1135
‑
1142
[1717]
ibragimova,i.et al.,cancer prev.res(phila)3(2010):1084
‑
1092
[1718]
ida
‑
yonemochi,h.et al.,mod.pathol.25(2012):784
‑
794
[1719]
idbaih,a.et al.,j neurooncol.90(2008):133
‑
140
[1720]
ide,h.et al.,biochem.biophys.res commun.369(2008):292
‑
296
[1721]
ii,m.et al.,exp.biol.med.(maywood.)231(2006):20
‑
27
[1722]
iio,a.et al.,biochim.biophys.acta 1829(2013):1102
‑
1110
[1723]
ijichi,n.et al.,j steroid biochem.mol.biol 123(2011):1
‑7[1724]
ikeda,r.et al.,int.j oncol 38(2011):513
‑
519
[1725]
ikonomov,o.c.et al.,biochem.biophys.res commun.440(2013):342
‑
347
[1726]
ilboudo,a.et al.,bmc.cancer 14(2014):7
[1727]
illemann,m.et al.,cancer med.3(2014):855
‑
864
[1728]
imai,k.et al.,br.j cancer 104(2011):300
‑
307
[1729]
imoto,i.et al.,biochem.biophys.res commun.286(2001):559
‑
565
[1730]
inamoto,t.et al.,mol.cancer ther.7(2008):3825
‑
3833
[1731]
ino,k.et al.,clin cancer res 14(2008):2310
‑
2317
[1732]
inoda,s.et al.,am.j pathol.178(2011a):1805
‑
1813
[1733]
inoda,s.et al.,j immunother.32(2009):474
‑
485
[1734]
inoda,s.et al.,exp.mol.pathol.90(2011b):55
‑
60
[1735]
ioachim,h.l.et al.,am.j surg.pathol.20(1996):64
‑
71
[1736]
iscan,m.et al.,breast cancer res treat.70(2001):47
‑
54
[1737]
ishida,t.et al.,leukemia 20(2006):2162
‑
2168
[1738]
ishigami,s.et al.,cancer lett.168(2001):87
‑
91
[1739]
ishigami,s.et al.,bmc.cancer 11(2011):106
[1740]
ishikawa,s.et al.,j exp.clin cancer res 22(2003):299
‑
306
[1741]
issaq,s.h.et al.,mol.cancer res 8(2010):223
‑
231
[1742]
ito,k.et al.,protein cell 2(2011):755
‑
763
[1743]
ito,m.et al.,jpn.j clin oncol 36(2006):116
‑
120
[1744]
ito,y.et al.,oncology 59(2000):68
‑
74
[1745]
itoh,g.et al.,cancer sci.104(2013):871
‑
879
[1746]
ivyna bong,p.n.et al.,mol.cytogenet.7(2014):24
[1747]
iwakuma,t.et al.,cancer metastasis rev 31(2012):633
‑
640
[1748]
iwanaga,k.et al.,cancer lett.202(2003):71
‑
79
[1749]
izykowska,k.et al.,eur.j haematol.93(2014):143
‑
149
[1750]
jaaskelainen,t.et al.,mol.cell endocrinol.350(2012):87
‑
98
[1751]
jackson,r.s.et al.,cell cycle 6(2007):95
‑
103
[1752]
jacob,f.et al.,bmc.mol.biol 15(2014):24
[1753]
jacques,c.et al.,j clin endocrinol.metab 90(2005):2314
‑
2320
[1754]
jager,d.et al.,cancer res 60(2000):3584
‑
3591
[1755]
jaggi,m.et al.,prostate 66(2006):193
‑
199
[1756]
jais,j.p.et al.,leukemia 22(2008):1917
‑
1924
[1757]
jakobsson,j.et al.,pharmacogenomics.j 4(2004):245
‑
250
[1758]
jalava,s.e.et al.,int.j cancer 124(2009):95
‑
102
[1759]
jang,s.g.et al.,bmc.cancer 7(2007):16
[1760]
januchowski,r.et al.,biomed.pharmacother.67(2013):240
‑
245
[1761]
janus,j.r.et al.,laryngoscope 121(2011):2598
‑
2603
[1762]
jayaram,h.n.et al.,curr.med.chem 6(1999):561
‑
574
[1763]
jayarama,s.et al.,j cell biochem.115(2014):261
‑
270
[1764]
jeffery,j.et al.,faseb j 29(2015a):1999
‑
2009
[1765]
jeffery,j.et al.,oncogene(2015b)
[1766]
jensen,c.h.et al.,eur.j biochem.225(1994):83
‑
92
[1767]
jensen,s.a.et al.,proc.natl.acad.sci.u.s.a 111(2014):5682
‑
5687
[1768]
jessie,k.et al.,electrophoresis 34(2013):2495
‑
2502
[1769]
ji,p.et al.,oncogene 24(2005):2739
‑
2744
[1770]
jia,d.et al.,hepatology 54(2011):1227
‑
1236
[1771]
jiang,j.h.et al.,ai.zheng.23(2004):672
‑
677
[1772]
jiang,j.h.et al.,hepatology 59(2014a):2216
‑
2227
[1773]
jiang,n.et al.,j biol chem 278(2003):21678
‑
21684
[1774]
jiang,p.et al.,mol.med.rep.9(2014b):2347
‑
2351
[1775]
jiang,q.et al.,histopathology 64(2014c):722
‑
730
[1776]
jiao,x.et al.,genes chromosomes.cancer 51(2012):480
‑
489
[1777]
jin,j.k.et al.,oncogene 34(2015):1811
‑
1821
[1778]
jinawath,n.et al.,oncogene 28(2009):1941
‑
1948
[1779]
jing,z.et al.,j immunol.185(2010):6719
‑
6727
[1780]
jinushi,m.et al.,cancer res 68(2008):8889
‑
8898
[1781]
johansson,p.et al.,j biol chem 289(2014):18514
‑
18525
[1782]
johnson,d.p.et al.,oncotarget.6(2015):4863
‑
4887
[1783]
joosse,s.a.et al.,clin cancer res 18(2012):993
‑
1003
[1784]
jose
‑
eneriz,e.s.et al.,br.j haematol.142(2008):571
‑
582
[1785]
joshi,a.d.et al.,clin cancer res 13(2007):5295
‑
5304
[1786]
joshi,s.et al.,bmc.cancer 15(2015):546
[1787]
judson,h.et al.,hum.genet.106(2000):406
‑
413
[1788]
junes
‑
gill,k.s.et al.,j neurooncol.102(2011):197
‑
211
[1789]
junes
‑
gill,k.s.et al.,bmc.cancer 14(2014):920
[1790]
jung,g.et al.,proc natl acad sci u s a 84(1987):4611
‑
4615
[1791]
jung,h.c.et al.,life sci.77(2005):1249
‑
1262
[1792]
jung,w.y.et al.,appl.immunohistochem.mol.morphol.22(2014):652
‑
657
[1793]
juszczynski,p.et al.,mol.cell biol 26(2006):5348
‑
5359
[1794]
kabbage,m.et al.,j biomed.biotechnol.2008(2008):564127
[1795]
kaizuka,t.et al.,j biol chem 285(2010):20109
‑
20116
[1796]
kalin,t.v.et al.,cancer res 66(2006):1712
‑
1720
[1797]
kalinichenko,v.v.et al.,genes dev.18(2004):830
‑
850
[1798]
kalinina,t.et al.,bmc.cancer 10(2010):295
[1799]
kalogeropoulou,m.et al.,mol.cancer res 8(2010):554
‑
568
[1800]
kamatani,n.et al.,cancer res 40(1980):4178
‑
4182
[1801]
kamino,h.et al.,cancer genet.204(2011):382
‑
391
[1802]
kamiyama,s.et al.,glycobiology 21(2011):235
‑
246
[1803]
kanda,a.et al.,oncogene 24(2005):7266
‑
7272
[1804]
kandoth,c.et al.,nature 497(2013):67
‑
73
[1805]
kang,b.w.et al.,plos.one.10(2015a):e0119649
[1806]
kang,g.et al.,plos.one.8(2013):e82770
[1807]
kang,j.k.et al.,int.j oncol 16(2000):1159
‑
1163
[1808]
kang,j.m.et al.,cancer res 75(2015b):3087
‑
3097
[1809]
kang,j.u.et al.,int.j oncol 37(2010):327
‑
335
[1810]
kang,j.u.et al.,cancer genet.cytogenet.182(2008a):1
‑
11
[1811]
kang,m.j.et al.,prostate 72(2012):1351
‑
1358
[1812]
kang,s.k.et al.,am.j pathol.173(2008b):518
‑
525
[1813]
kang,x.et al.,oncogene 28(2009):565
‑
574
[1814]
kapoor,a.et al.,nature 468(2010):1105
‑
1109
[1815]
karahatay,s.et al.,cancer lett.256(2007):101
‑
111
[1816]
karbowniczek,m.et al.,j invest dermatol.128(2008):980
‑
987
[1817]
karess,r.e.et al.,int.rev cell mol.biol 306(2013):223
‑
273
[1818]
karim,h.et al.,biochem.biophys.res commun.411(2011):156
‑
161
[1819]
karlsson,e.et al.,breast cancer res treat.153(2015):31
‑
40
[1820]
karytinos,a.et al.,j biol chem 284(2009):17775
‑
17782
[1821]
kashuba,v.et al.,int.j mol.sci.13(2012):13352
‑
13377
[1822]
kashyap,v.et al.,mol oncol 7(2013):555
‑
566
[1823]
kassambara,a.et al.,biochem.biophys.res commun.379(2009):840
‑
845
[1824]
kato,i.et al.,pathol.int.59(2009):38
‑
43
[1825]
kato,s.et al.,int.j oncol 29(2006):33
‑
40
[1826]
katoh,m.et al.,int.j oncol 25(2004):1495
‑
1500
[1827]
katoh,y.et al.,int.j mol.med.18(2006):523
‑
528
[1828]
katz,t.a.et al.,breast cancer res treat.146(2014):99
‑
108
[1829]
kaufmann,m.et al.,curr.top.microbiol.immunol.384(2015):167
‑
188
[1830]
kaur,h.et al.,plos.one.7(2012):e50249
[1831]
kawagoe,h.et al.,cancer res 64(2004):6091
‑
6100
[1832]
kawahara,r.et al.,proteomics.16(2016):159
‑
173
[1833]
kawakami,k.et al.,int.j oncol(2015)
[1834]
kawakami,m.et al.,cancer sci.104(2013):1447
‑
1454
[1835]
kaynar,h.et al.,cancer lett.227(2005):133
‑
139
[1836]
kazma,r.et al.,carcinogenesis 33(2012):1059
‑
1064
[1837]
ke,j.y.et al.,j zhejiang.univ sci.b 15(2014a):1032
‑
1038
[1838]
ke,r.h.et al.,j neurooncol.118(2014b):369
‑
376
[1839]
kearns,p.r.et al.,br.j haematol.120(2003):80
‑
88
[1840]
keng,v.w.et al.,nat biotechnol.27(2009):264
‑
274
[1841]
kerley
‑
hamilton,j.s.et al.,oncogene 24(2005):6090
‑
6100
[1842]
kesari,m.v.et al.,indian j gastroenterol.34(2015):63
‑
67
[1843]
khan,j.et al.,plos.one.6(2011):e26512
[1844]
khodarev,n.n.et al.,cancer res 69(2009):2833
‑
2837
[1845]
kibbe,a.h.,handbook of pharmaceutical excipients rd(2000)
[1846]
kiessling,a.et al.,oncogene 28(2009):2606
‑
2620
[1847]
kikuchi,y.et al.,front genet.4(2013):271
[1848]
killian,a.et al.,genes chromosomes.cancer 45(2006):874
‑
881
[1849]
kim,b.h.et al.,ann.surg.oncol 21(2014a):2020
‑
2027
[1850]
kim,d.h.,yonsei med.j 48(2007):694
‑
700
[1851]
kim,d.s.et al.,j proteome.res 9(2010):3710
‑
3719
[1852]
kim,h.e.et al.,plos.one.7(2012a):e43223
[1853]
kim,h.j.et al.,j proteome.res 8(2009a):1368
‑
1379
[1854]
kim,h.n.et al.,am.j hematol.82(2007):798
‑
801
[1855]
kim,i.m.et al.,cancer res 66(2006):2153
‑
2161
[1856]
kim,j.et al.,genes chromosomes.cancer 54(2015a):681
‑
691
[1857]
kim,j.c.et al.,int.j radiat.oncol biol phys.86(2013a):350
‑
357
[1858]
kim,j.c.et al.,world j gastroenterol.14(2008a):6662
‑
6672
[1859]
kim,j.h.et al.,cancer 85(1999):546
‑
553
[1860]
kim,j.h.et al.,bmb.rep.44(2011a):523
‑
528
[1861]
kim,j.w.et al.,int.j oncol 35(2009b):129
‑
137
[1862]
kim,k.et al.,mol.cell 52(2013b):459
‑
467
[1863]
kim,m.et al.,mol cancer res 6(2008b):222
‑
230
[1864]
kim,m.s.et al.,oncogene 27(2008c):3624
‑
3634
[1865]
kim,m.s.et al.,histopathology 58(2011b):660
‑
668
[1866]
kim,r.et al.,plos.one.10(2015b):e0126670
[1867]
kim,s.h.et al.,investig.clin urol.57(2016):63
‑
72
[1868]
kim,s.j.et al.,acta haematol.120(2008d):211
‑
216
[1869]
kim,s.j.et al.,mol.carcinog 54(2015c):1748
‑
1757
[1870]
kim,s.m.et al.,int.j cancer 134(2014b):114
‑
124
[1871]
kim,s.w.et al.,omics.15(2011c):281
‑
292
[1872]
kim,s.w.et al.,blood 111(2008e):1644
‑
1653
[1873]
kim,y.d.et al.,int.j mol.med.29(2012b):656
‑
662
[1874]
kim,y.w.et al.,plos.one.7(2012c):e40960
[1875]
kindt,n.et al.,j cancer res clin oncol 140(2014):937
‑
947
[1876]
kinoshita,y.et al.,am.j pathol.180(2012):375
‑
389
[1877]
kitange,g.j.et al.,j neurooncol.100(2010):177
‑
186
[1878]
klatka,j.et al.,eur.arch.otorhinolaryngol.270(2013):2683
‑
2693
[1879]
kleppe,m.et al.,nat genet.42(2010):530
‑
535
[1880]
kleppe,m.et al.,blood 117(2011a):7090
‑
7098
[1881]
kleppe,m.et al.,haematologica 96(2011b):1723
‑
1727
[1882]
kleylein
‑
sohn,j.et al.,j cell sci.125(2012):5391
‑
5402
[1883]
knapp,p.et al.,prostaglandins other lipid mediat.92(2010):62
‑
66
[1884]
ko,h.w.et al.,dev.cell 18(2010):237
‑
247
[1885]
kobayashi,h.et al.,biochem.biophys.res commun.467(2015a):121
‑
127
[1886]
kobayashi,m.et al.,lung cancer 90(2015b):342
‑
345
[1887]
kobayashi,y.et al.,placenta 34(2013):110
‑
118
[1888]
kocer,b.et al.,pathol.int.52(2002):470
‑
477
[1889]
kogo,r.et al.,int.j oncol 39(2011):155
‑
159
[1890]
kohno,t.et al.,nat med.18(2012):375
‑
377
[1891]
kohrt,d.et al.,cell cycle 13(2014):62
‑
71
[1892]
koike,k.,recent results cancer res 193(2014):97
‑
111
[1893]
kokoglu,e.et al.,cancer lett.50(1990):179
‑
181
[1894]
kolb,t.m.et al.,toxicol.sci.88(2005):331
‑
339
[1895]
kollmann,k.et al.,cancer cell 24(2013):167
‑
181
[1896]
kong,l.et al.,shanghai kou qiang.yi.xue.24(2015):89
‑
93
[1897]
koo,g.b.et al.,cell res 25(2015a):707
‑
725
[1898]
koo,s.et al.,anticancer res 35(2015b):3209
‑
3215
[1899]
koochekpour,s.et al.,asian j androl 7(2005a):147
‑
158
[1900]
koochekpour,s.et al.,genes chromosomes.cancer 44(2005b):351
‑
364
[1901]
kordi tamandani,d.m.et al.,j assist.reprod.genet.26(2009):173
‑
178
[1902]
korosec,b.et al.,cancer genet.cytogenet.171(2006):105
‑
111
[1903]
korotayev,k.et al.,cell signal.20(2008):1221
‑
1226
[1904]
kortum,k.m.et al.,ann.hematol.94(2015):1205
‑
1211
[1905]
koshikawa,k.et al.,oncogene 21(2002):2822
‑
2828
[1906]
kozlowski,l.et al.,arch.dermatol.res 292(2000):68
‑
71
[1907]
kraemer,n.et al.,cell mol life sci.68(2011):1719
‑
1736
[1908]
kramer,m.et al.,biomed.res int.2015(2015):208017
[1909]
krieg,a.m.,nat rev.drug discov.5(2006):471
‑
484
[1910]
krona,c.et al.,oncogene 22(2003):2343
‑
2351
[1911]
kuang,s.q.et al.,leukemia 22(2008):1529
‑
1538
[1912]
kuasne,h.et al.,clin epigenetics.7(2015):46
[1913]
kubota,h.et al.,cell stress.chaperones.15(2010):1003
‑
1011
[1914]
kudo,y.et al.,j hepatol.55(2011):1400
‑
1408
[1915]
kuhn,e.et al.,mod.pathol.27(2014):1014
‑
1019
[1916]
kulkarni,a.a.et al.,clin cancer res 15(2009):2417
‑
2425
[1917]
kumar,s.et al.,cell death.dis.6(2015):e1758
[1918]
kumarakulasingham,m.et al.,clin cancer res 11(2005):3758
‑
3765
[1919]
kuo,i.y.et al.,int.j cancer 135(2014):563
‑
573
[1920]
kuo,s.j.et al.,oncol rep.24(2010):759
‑
766
[1921]
kuphal,s.et al.,oncogene 25(2006):103
‑
110
[1922]
kuppers,r.et al.,j clin invest 111(2003):529
‑
537
[1923]
kuramitsu,y.et al.,anticancer res 31(2011):3331
‑
3336
[1924]
kurtovic
‑
kozaric,a.et al.,leukemia 29(2015):126
‑
136
[1925]
kuruma,h.et al.,am.j pathol.174(2009):2044
‑
2050
[1926]
kutikhin,a.g.,hum.immunol.72(2011):955
‑
968
[1927]
kuznetsova,e.b.et al.,mol.biol.(mosk)41(2007):624
‑
633
[1928]
kwon,j.et al.,int j oncol 43(2013):1523
‑
1530
[1929]
la,vecchia c.,eur.j cancer prev.10(2001):125
‑
129
[1930]
labhart,p.et al.,proc.natl.acad.sci.u.s.a 102(2005):1339
‑
1344
[1931]
laetsch,t.w.et al.,cell death.dis.5(2014):e1072
[1932]
lage,h.et al.,febs lett.494(2001):54
‑
59
[1933]
lagorce
‑
pages,c.et al.,virchows arch.444(2004):426
‑
435
[1934]
lai,j.m.et al.,methods mol.biol 623(2010):231
‑
242
[1935]
lai,m.t.et al.,j pathol.224(2011):367
‑
376
[1936]
lake,s.l.et al.,invest ophthalmol.vis.sci.52(2011):5598
‑
5604
[1937]
lan,h.et al.,int.j clin exp.med.7(2014):665
‑
672
[1938]
lan,q.et al.,eur.j haematol.85(2010):492
‑
495
[1939]
lane,j.et al.,int.j mol.med.12(2003):253
‑
257
[1940]
langemeijer,s.m.et al.,cell cycle 8(2009):4044
‑
4048
[1941]
lapointe,j.et al.,am.j surg.pathol.32(2008):205
‑
209
[1942]
lapouge,g.et al.,cell mol.life sci.62(2005):53
‑
64
[1943]
lara,p.c.et al.,radiat.oncol 6(2011):148
[1944]
larkin,s.e.et al.,br.j cancer 106(2012):157
‑
165
[1945]
laske,k.et al.,cancer immunol.res 1(2013):190
‑
200
[1946]
lau,l.f.,cell mol.life sci.68(2011):3149
‑
3163
[1947]
lau,y.f.et al.,mol.carcinog 27(2000):308
‑
321
[1948]
lauring,j.et al.,blood 111(2008):856
‑
864
[1949]
lazova,r.et al.,am.j dermatopathol.31(2009):177
‑
181
[1950]
leal,j.f.et al.,carcinogenesis 29(2008):2089
‑
2095
[1951]
ledet,e.m.et al.,prostate 73(2013):614
‑
623
[1952]
lee,b.h.et al.,cancer res 73(2013a):1211
‑
1218
[1953]
lee,e.j.et al.,oncol res 18(2010):401
‑
408
[1954]
lee,e.k.et al.,mol.cell biol 33(2013b):4422
‑
4433
[1955]
lee,j.h.et al.,ann.surg.249(2009):933
‑
941
[1956]
lee,m.j.et al.,j proteome.res 13(2014a):4878
‑
4888
[1957]
lee,s.y.et al.,eur.j cancer 50(2014b):698
‑
705
[1958]
lee,t.j.et al.,mol.cancer 3(2004):31
[1959]
lee,y.s.et al.,oncotarget.6(2015):16449
‑
16460
[1960]
leong,h.s.et al.,cancer res 73(2013):1591
‑
1599
[1961]
leong,s.r.et al.,mol.pharm.12(2015):1717
‑
1729
[1962]
levi,e.et al.,cancer chemother.pharmacol.67(2011):1401
‑
1413
[1963]
levy,p.et al.,clin cancer res 13(2007):398
‑
407
[1964]
li,b.h.et al.,biochem.biophys.res commun.369(2008a):554
‑
560
[1965]
li,b.s.et al.,oncogene 34(2015a):2556
‑
2565
[1966]
li,c.f.et al.,oncotarget.5(2014a):11428
‑
11441
[1967]
li,c.f.et al.,bmc.genomics 8(2007):92
[1968]
li,c.m.et al.,am.j pathol.160(2002):2181
‑
2190
[1969]
li,h.et al.,neoplasia.8(2006):568
‑
577
[1970]
li,j.et al.,zhonghua bing.li xue.za zhi.43(2014b):546
‑
550
[1971]
li,j.f.et al.,zhonghua wei chang wai ke.za zhi.15(2012a):388
‑
391
[1972]
li,j.y.et al.,chin med.j(engl.)125(2012b):3526
‑
3531
[1973]
li,l.et al.,clin cancer res 19(2013a):4651
‑
4661
[1974]
li,l.et al.,pharmacogenet.genomics 22(2012c):105
‑
116
[1975]
li,l.c.et al.,am.j obstet.gynecol.205(2011a):362
‑
25
[1976]
li,m.et al.,clin cancer res 11(2005):1809
‑
1814
[1977]
li,n.et al.,biochem.biophys.res commun.455(2014):358
‑
362
[1978]
li,q.et al.,mol.biol rep.41(2014c):2409
‑
2417
[1979]
li,s.et al.,j huazhong.univ sci.technolog.med.sci.28(2008b):93
‑
96
[1980]
li,s.et al.,proc.natl.acad.sci.u.s.a 111(2014d):6970
‑
6975
[1981]
li,t.et al.,j thorac.oncol 7(2012d):448
‑
452
[1982]
li,w.et al.,cancer cell int.15(2015b):17
[1983]
li,w.et al.,med.oncol 31(2014e):208
[1984]
li,w.q.et al.,carcinogenesis 34(2013b):1536
‑
1542
[1985]
li,x.et al.,curr.protein pept.sci.16(2015c):301
‑
309
[1986]
li,x.et al.,pancreas 40(2011b):753
‑
761
[1987]
li,x.et al.,bmc.cancer 15(2015d):342
[1988]
li,x.et al.,cancer res(2016)
[1989]
li,y.et al.,cancer genet.cytogenet.198(2010):97
‑
106
[1990]
li,y.et al.,cancer biol ther.16(2015e):1316
‑
1322
[1991]
li,y.et al.,clin cancer res 17(2011c):3830
‑
3840
[1992]
li,y.et al.,lung cancer 80(2013c):91
‑
98
[1993]
li,z.et al.,diagn.pathol.10(2015f):167
[1994]
li,z.et al.,nan.fang yi.ke.da.xue.xue.bao.33(2013d):1483
‑
1488
[1995]
lian,z.et al.,cancer biol ther.16(2015):750
‑
755
[1996]
liang,b.et al.,zhonghua yi.xue.za zhi.95(2015a):408
‑
411
[1997]
liang,b.et al.,dig.dis.sci.60(2015b):2360
‑
2372
[1998]
liang,h.et al.,genome res 22(2012a):2120
‑
2129
[1999]
liang,q.et al.,sci.rep.3(2013):2932
[2000]
liang,x.s.et al.,int.j cancer 130(2012b):2062
‑
2066
[2001]
liang,x.t.et al.,j gastroenterol.hepatol.26(2011):544
‑
549
[2002]
liang,y.et al.,bmc.cancer 6(2006):97
[2003]
liang,y.et al.,proc.natl.acad.sci.u.s.a 102(2005):5814
‑
5819
[2004]
liao,c.f.et al.,j exp.clin cancer res 27(2008):15
[2005]
liao,f.et al.,med.oncol 27(2010):1219
‑
1226
[2006]
liao,y.et al.,bmc.cancer 14(2014a):487
[2007]
liao,y.et al.,plos.one.9(2014b):e99907
[2008]
liddy,n.et al.,nat med.18(2012):980
‑
987
[2009]
lignitto,l.et al.,nat commun.4(2013):1822
[2010]
lim,b.et al.,carcinogenesis 35(2014):1020
‑
1027
[2011]
lim,s.o.et al.,biochem.biophys.res commun.291(2002):1031
‑
1037
[2012]
lin,d.c.et al.,nat genet.46(2014):467
‑
473
[2013]
lin,f.et al.,cancer biol ther.7(2008a):1669
‑
1676
[2014]
lin,h.s.et al.,arch.otolaryngol.head neck surg.130(2004):311
‑
316
[2015]
lin,p.et al.,mol.biol rep.38(2011):1741
‑
1747
[2016]
lin,p.h.et al.,j biomed.sci.22(2015a):44
[2017]
lin,s.et al.,rna.biol 12(2015):792
‑
800
[2018]
lin,w.y.et al.,hum.mol.genet.24(2015b):285
‑
298
[2019]
lin,y.l.et al.,int.j clin exp.pathol.8(2015c):14257
‑
14269
[2020]
lin,y.m.et al.,mol.carcinog 47(2008b):925
‑
933
[2021]
lin,y.w.et al.,oral oncol 48(2012):629
‑
635
[2022]
lindberg,d.et al.,neuroendocrinology 86(2007):112
‑
118
[2023]
linder,n.et al.,gynecol.oncol 124(2012):311
‑
318
[2024]
linder,n.et al.,clin cancer res 11(2005):4372
‑
4381
[2025]
ling,z.q.et al.,eur.j surg.oncol 38(2012):326
‑
332
[2026]
linge,a.et al.,invest ophthalmol.vis.sci.53(2012):4634
‑
4643
[2027]
lips,e.h.et al.,bmc.cancer 8(2008):314
[2028]
lipson,d.et al.,nat med.18(2012):382
‑
384
[2029]
litvinov,i.v.et al.,clin.cancer res.20(2014a):3799
‑
3808
[2030]
litvinov,i.v.et al.,oncoimmunology 3(2014b):e970025
[2031]
liu,b.et al.,biochem.biophys.res commun.293(2002a):1396
‑
1404
[2032]
liu,c.et al.,int.j clin exp.pathol.7(2014a):690
‑
698
[2033]
liu,d.et al.,oncotarget.6(2015a):39211
‑
39224
[2034]
liu,d.q.et al.,sci.rep.5(2015b):11955
[2035]
liu,f.et al.,tumour.biol 35(2014b):8685
‑
8690
[2036]
liu,j.et al.,plos.one.9(2014c):e89340
[2037]
liu,j.et al.,biochem.biophys.res commun.463(2015c):1230
‑
1236
[2038]
liu,j.f.et al.,cancer cell int.13(2013a):41
[2039]
liu,l.x.et al.,world j gastroenterol.8(2002b):631
‑
637
[2040]
liu,l.x.et al.,oncol rep.10(2003):1771
‑
1775
[2041]
liu,l.z.et al.,cancer res 67(2007):6325
‑
6332
[2042]
liu,m.et al.,cancer res 66(2006):3593
‑
3602
[2043]
liu,p.et al.,j natl.cancer inst.100(2008a):1326
‑
1330
[2044]
liu,q.et al.,prostate 73(2013b):1028
‑
1037
[2045]
liu,q.et al.,med.oncol 31(2014d):882
[2046]
liu,r.et al.,proc.natl.acad.sci.u.s.a 105(2008b):7570
‑
7575
[2047]
liu,r.et al.,oncotarget.6(2015d):33456
‑
33469
[2048]
liu,s.y.et al.,zhonghua wai ke.za zhi.47(2009a):1732
‑
1735
[2049]
liu,w.et al.,ann.surg.oncol 21 suppl 4(2014e):s575
‑
s583
[2050]
liu,w.et al.,j biol.chem.279(2004):10167
‑
10175
[2051]
liu,w.et al.,mol.clin oncol 2(2014f):219
‑
225
[2052]
liu,x.et al.,plos.one.8(2013c):e77367
[2053]
liu,x.et al.,pathol.res pract.210(2014g):256
‑
263
[2054]
liu,x.et al.,zhonghua yi.xue.za zhi.94(2014h):2008
‑
2012
[2055]
liu,x.et al.,med.oncol 30(2013d):735
[2056]
liu,y.et al.,cancer res 69(2009b):7844
‑
7850
[2057]
liu,y.et al.,asian pac.j cancer prev.16(2015e):2659
‑
2664
[2058]
liu,y.et al.,int.j clin exp.pathol.7(2014i):5750
‑
5761
[2059]
liu,y.x.et al.,oncol lett.4(2012):847
‑
851
[2060]
liu,z.et al.,oncol rep.33(2015f):1908
‑
1914
[2061]
liu,z.et al.,bmc.cancer 14(2014j):274
[2062]
liu,z.et al.,carcinogenesis 32(2011):1668
‑
1674
[2063]
ljunggren,h.g.et al.,j exp.med.162(1985):1745
‑
1759
[2064]
llopis,s.et al.,bmc.cancer 13(2013):139
[2065]
lo,y.w.et al.,j cell mol.med.19(2015):744
‑
759
[2066]
long,z.w.et al.,tumour.biol.35(2014):11415
‑
11426
[2067]
longenecker,b.m.et al.,ann n.y.acad.sci.690(1993):276
‑
291
[2068]
lonsdale,j.,nat.genet.45(2013):580
‑
585
[2069]
lopez
‑
cortes,a.et al.,am.j med.sci.346(2013):447
‑
454
[2070]
lou,t.f.et al.,cancer prev.res(phila)9(2016):43
‑
52
[2071]
lozupone,f.et al.,oncogene(2015)
[2072]
lu,c.et al.,mol.cell biochem.312(2008):71
‑
80
[2073]
lu,c.et al.,dig.dis.sci.58(2013):2713
‑
2720
[2074]
lu,j.et al.,oncol rep.32(2014a):2571
‑
2579
[2075]
lu,j.j.et al.,chin j nat med.13(2015):673
‑
679
[2076]
lu,p.et al.,plos.one.9(2014b):e88918
[2077]
lu,x.et al.,mol.cancer ther.3(2004):861
‑
872
[2078]
lu,x.et al.,clin cancer res 15(2009):3287
‑
3296
[2079]
lucas,s.et al.,int.j cancer 87(2000):55
‑
60
[2080]
lucito,r.et al.,cancer biol ther.6(2007):1592
‑
1599
[2081]
ludwig,a.et al.,anticancer res 22(2002):3213
‑
3221
[2082]
lukas,t.j.et al.,proc.natl.acad.sci.u.s.a 78(1981):2791
‑
2795
[2083]
luker,k.e.et al.,cancer res 61(2001):6540
‑
6547
[2084]
luksch,h.et al.,anticancer res 31(2011):3181
‑
3192
[2085]
lum,d.f.et al.,int.j cancer 83(1999):162
‑
166
[2086]
lundblad,r.l.,chemical reagents for protein modification 3rd(2004)
[2087]
luo,x.et al.,j clin endocrinol.metab 94(2009):4533
‑
4539
[2088]
lv,t.et al.,plos.one.7(2012):e35065
[2089]
lyng,h.et al.,bmc.genomics 7(2006):268
[2090]
ma,g.f.et al.,eur.rev.med.pharmacol.sci.19(2015):578
‑
585
[2091]
ma,q.et al.,biochem.biophys.res commun.454(2014):157
‑
161
[2092]
macdonald,g.et al.,sci.signal.7(2014):ra56
[2093]
macdonald,t.j.et al.,methods mol.biol 377(2007):203
‑
222
[2094]
mackay,c.et al.,cancer res 74(2014):2246
‑
2257
[2095]
madden,s.f.et al.,mol.cancer 13(2014):241
[2096]
madhavan,s.et al.,j exp.clin cancer res 34(2015):45
[2097]
magold,a.i.et al.,plos.one.4(2009):e6952
[2098]
mahmood,s.f.et al.,carcinogenesis 35(2014):670
‑
682
[2099]
makkinje,a.et al.,cell signal.21(2009):1423
‑
1435
[2100]
malta
‑
vacas,j.et al.,clin chem lab med.47(2009):427
‑
431
[2101]
malumbres,m.et al.,curr.opin.genet.dev.17(2007):60
‑
65
[2102]
man,t.k.et al.,bmc.cancer 4(2004):45
[2103]
mang,j.et al.,transl.oncol 8(2015):487
‑
496
[2104]
mangs,a.h.et al.,int.j biochem.cell biol 40(2008):2353
‑
2357
[2105]
mano,y.et al.,cancer sci.98(2007):1902
‑
1913
[2106]
mantia
‑
smaldone,g.m.et al.,hum.vaccin.immunother.8(2012):1179
‑
1191
[2107]
mao,j.et al.,cancer sci.99(2008):2120
‑
2127
[2108]
mao,p.et al.,j biol chem 286(2011):19381
‑
19391
[2109]
mao,p.et al.,plos.one.8(2013a):e81803
[2110]
mao,y.et al.,bmc.cancer 13(2013b):498
[2111]
marechal,r.et al.,clin cancer res 15(2009):2913
‑
2919
[2112]
marian,c.et al.,eur.j clin nutr.65(2011):683
‑
689
[2113]
marini,f.et al.,j biol chem 277(2002):8716
‑
8723
[2114]
markt,s.c.et al.,cancer causes control 26(2015):25
‑
33
[2115]
marlow,l.a.et al.,j cell sci.125(2012):4253
‑
4263
[2116]
marmey,b.et al.,hum.pathol.37(2006):68
‑
77
[2117]
marques filho,m.f.et al.,braz.j otorhinolaryngol.72(2006):25
‑
30
[2118]
martens
‑
de kemp,s.r.et al.,clin cancer res 19(2013):1994
‑
2003
[2119]
martin,l.et al.,oncogene 31(2012):4076
‑
4084
[2120]
martin,t.a.et al.,j cell biochem.105(2008):41
‑
52
[2121]
martin,t.a.et al.,methods mol.biol 762(2011):383
‑
407
[2122]
martin,t.d.et al.,mol.cell 53(2014):209
‑
220
[2123]
martinez
‑
trillos,a.et al.,blood 123(2014):3790
‑
3796
[2124]
masuda,k.et al.,oncol rep.28(2012):1146
‑
1152
[2125]
masuda,t.a.et al.,clin cancer res 9(2003):5693
‑
5698
[2126]
masugi,y.et al.,lab invest 95(2015):308
‑
319
[2127]
matejcic,m.et al.,plos.one.6(2011):e29366
[2128]
mathew,m.et al.,apoptosis.18(2013):882
‑
895
[2129]
matovina,m.et al.,gynecol.oncol 113(2009):120
‑
127
[2130]
matsumoto,k.et al.,genes cells 6(2001):1101
‑
1111
[2131]
matsumoto,n.et al.,leukemia 14(2000):1757
‑
1765
[2132]
matsuyama,a.et al.,virchows arch.459(2011):539
‑
545
[2133]
matsuyama,a.et al.,virchows arch.457(2010):577
‑
583
[2134]
matsuyama,r.et al.,cancer sci.107(2016):28
‑
35
[2135]
maurizio,e.et al.,mol.cell proteomics.15(2016):109
‑
123
[2136]
maxwell,c.a.et al.,j cell sci.121(2008):925
‑
932
[2137]
mayne,m.et al.,eur.j immunol.34(2004):1217
‑
1227
[2138]
mazan
‑
mamczarz,k.et al.,plos.genet.10(2014):e1004105
[2139]
mazzoccoli,g.et al.,chronobiol.int.28(2011):841
‑
851
[2140]
mccabe,k.e.et al.,cell death.dis.5(2014):e1496
[2141]
mcclung,j.k.et al.,exp.gerontol.30(1995):99
‑
124
[2142]
mcdonald,j.m.et al.,mol.cancer 4(2005):35
[2143]
mechtcheriakova,d.et al.,cell signal.19(2007):748
‑
760
[2144]
mehta,a.et al.,breast 23(2014):2
‑9[2145]
mehta,j.et al.,plos.one.10(2015):e0120622
[2146]
meier,c.et al.,j pathol.234(2014):351
‑
364
[2147]
meijer,d.et al.,breast cancer res treat.113(2009):253
‑
260
[2148]
meissner,m.et al.,clin cancer res 11(2005):2552
‑
2560
[2149]
men,w.et al.,cancer genomics proteomics.12(2015):1
‑8[2150]
meng,f.et al.,int j oncol 43(2013):495
‑
502
[2151]
meng,j.et al.,acta biochim.biophys.sin.(shanghai)42(2010):52
‑
57
[2152]
mertens
‑
walker,i.et al.,bmc.cancer 15(2015):164
[2153]
messai,y.et al.,cancer res 74(2014):6820
‑
6832
[2154]
metwally,n.s.et al.,cancer cell int.11(2011):8
[2155]
meziere,c.et al.,j immunol 159(1997):3230
‑
3237
[2156]
michel,s.et al.,int.j cancer 127(2010):889
‑
898
[2157]
midorikawa,y.et al.,jpn.j cancer res 93(2002):636
‑
643
[2158]
mikami,t.et al.,oral oncol 47(2011):497
‑
503
[2159]
mikami,t.et al.,virchows arch.466(2015):559
‑
569
[2160]
milanovich,s.et al.,exp.hematol.43(2015):53
‑
64
[2161]
miller,r.k.et al.,j am.soc.nephrol.22(2011):1654
‑
1664
[2162]
milne,r.l.et al.,hum.mol.genet.23(2014):6096
‑
6111
[2163]
min,k.w.et al.,int.j gynecol.pathol.32(2013):3
‑
14
[2164]
mino,k.et al.,biosci.biotechnol.biochem.78(2014):1010
‑
1017
[2165]
mirmalek
‑
sani,s.h.et al.,j cell mol.med.13(2009):3541
‑
3555
[2166]
mishra,l.et al.,cancer biol ther.4(2005a):694
‑
699
[2167]
mishra,s.et al.,febs j 277(2010):3937
‑
3946
[2168]
mishra,s.et al.,trends mol.med.11(2005b):192
‑
197
[2169]
mitchell,r.j.et al.,hum.hered.38(1988):144
‑
150
[2170]
mitra,r.et al.,clin cancer res 17(2011):2934
‑
2946
[2171]
mitsuhashi,k.et al.,int.j hematol.100(2014):88
‑
95
[2172]
mittal,r.d.et al.,indian j cancer 41(2004):115
‑
119
[2173]
miwa,h.et al.,leukemia 6(1992):405
‑
409
[2174]
miwa,t.et al.,cancer med.4(2015):1091
‑
1100
[2175]
miyaji,k.et al.,j viral hepat.10(2003):241
‑
248
[2176]
miyoshi,y.et al.,med.mol.morphol.43(2010):193
‑
196
[2177]
mo,l.et al.,anticancer res 30(2010):3413
‑
3420
[2178]
mohamed,f.e.et al.,liver int.35(2015):1063
‑
1076
[2179]
mohelnikova
‑
duchonova,b.et al.,cancer chemother.pharmacol.72(2013a):669
‑
682
[2180]
mohelnikova
‑
duchonova,b.et al.,pancreas 42(2013b):707
‑
716
[2181]
moldovan,g.l.et al.,mol.cell biol 30(2010):1088
‑
1096
[2182]
molinolo,a.a.et al.,clin cancer res 13(2007):4964
‑
4973
[2183]
moniz,l.s.et al.,mol.cell biol 31(2011):30
‑
42
[2184]
monji,m.et al.,clin cancer res 10(2004):6047
‑
6057
[2185]
morgan,r.a.et al.,science 314(2006):126
‑
129
[2186]
mori,m.et al.,transplantation 64(1997):1017
‑
1027
[2187]
mori,y.et al.,endocr.relat cancer 18(2011):465
‑
478
[2188]
moritake,h.et al.,am.j hematol.86(2011):75
‑
78
[2189]
moriya,y.et al.,j hum.genet.57(2012):38
‑
45
[2190]
mortara,l.et al.,clin cancer res.12(2006):3435
‑
3443
[2191]
moss,s.f.et al.,gut 45(1999):723
‑
729
[2192]
mossink,m.h.et al.,oncogene 22(2003):7458
‑
7467
[2193]
mostafa,w.z.et al.,j cutan.pathol.37(2010):68
‑
74
[2194]
motaghed,m.et al.,int.j mol.med.33(2014):8
‑
16
[2195]
mouradov,d.et al.,cancer res 74(2014):3238
‑
3247
[2196]
mueller,l.n.et al.,j proteome.res 7(2008):51
‑
61
[2197]
mueller,l.n.et al.,proteomics.7(2007):3470
‑
3480
[2198]
muir,k.et al.,cancer res 73(2013):4722
‑
4731
[2199]
mulligan,a.m.et al.,breast cancer res 13(2011):r110
[2200]
mumberg,d.et al.,proc.natl.acad.sci.u.s.a 96(1999):8633
‑
8638
[2201]
munoz,i.m.et al.,mol.cell 35(2009):116
‑
127
[2202]
murphy,n.c.et al.,int.j cancer 126(2010):1445
‑
1453
[2203]
murray,g.i.et al.,histopathology 57(2010):202
‑
211
[2204]
murrin,l.c.et al.,j neuroimmune.pharmacol.2(2007):290
‑
295
[2205]
murugan,a.k.et al.,oncol lett.6(2013):437
‑
441
[2206]
muto,y.et al.,cell cycle 7(2008):2738
‑
2748
[2207]
mydlikova,z.et al.,neoplasma 57(2010):287
‑
290
[2208]
naba,a.et al.,elife.3(2014):e01308
[2209]
nadal
‑
serrano,m.et al.,j cell biochem.113(2012):3178
‑
3185
[2210]
nagai,m.et al.,cancer res 51(1991):3886
‑
3890
[2211]
nagai,m.a.et al.,int.j oncol 37(2010):41
‑
49
[2212]
nagakura,s.et al.,blood 100(2002):1031
‑
1037
[2213]
nagamachi,a.et al.,cancer cell 24(2013):305
‑
317
[2214]
nagashio,r.et al.,sci.rep.5(2015):8649
[2215]
nagata,m.et al.,bmc.cancer 13(2013):410
[2216]
nagendra,d.c.et al.,mol.carcinog 51(2012):826
‑
831
[2217]
nagi,c.et al.,breast cancer res treat.94(2005):225
‑
235
[2218]
nagpal,j.k.et al.,mod.pathol.21(2008):979
‑
991
[2219]
nakagawa,y.et al.,br.j cancer 80(1999):914
‑
917
[2220]
nakaya,h.i.et al.,biochem.biophys.res commun.364(2007):918
‑
923
[2221]
nakayama,k.et al.,cancer res 67(2007):8058
‑
8064
[2222]
nallar,s.c.et al.,plos.one.6(2011):e24082
[2223]
nam,s.h.et al.,oncotarget.6(2015):21655
‑
21674
[2224]
nam,s.w.et al.,int.j oncol 45(2014):1450
‑
1456
[2225]
nam
‑
cha,s.h.et al.,mod.pathol.22(2009):1006
‑
1015
[2226]
nantajit,d.et al.,plos.one.5(2010):e12341
[2227]
narita,t.et al.,mol cell biol.23(2003):1863
‑
1873
[2228]
navarro,a.et al.,j clin endocrinol.metab 99(2014):e2437
‑
e2445
[2229]
naylor,d.j.et al.,j biol chem 273(1998):21169
‑
21177
[2230]
near,r.i.et al.,j cell physiol 212(2007):655
‑
665
[2231]
nebral,k.et al.,leukemia 23(2009):134
‑
143
[2232]
neidert,m.c.et al.,j neurooncol.111(2013):285
‑
294
[2233]
nelson,c.r.et al.,j cell biol 211(2015):503
‑
516
[2234]
nelson,m.a.et al.,cancer genet.cytogenet.108(1999):91
‑
99
[2235]
neumann,m.et al.,blood 121(2013):4749
‑
4752
[2236]
neveling,k.et al.,cytogenet.genome res 118(2007):166
‑
176
[2237]
ng,y.et al.,j biol chem 279(2004):34156
‑
34164
[2238]
ngeow,j.et al.,cancer discov.4(2014):762
‑
763
[2239]
nguyen,t.b.et al.,j biol chem 277(2002):41960
‑
41969
[2240]
ni,i.b.et al.,hematol.rep.4(2012):e19
[2241]
ni,y.h.et al.,histopathology(2015)
[2242]
ni,z.et al.,j urol.167(2002):1859
‑
1862
[2243]
niavarani,a.et al.,ann.hematol.(2015)
[2244]
niimi,r.et al.,bmc.cancer 13(2013):309
[2245]
nikonova,a.s.et al.,cell mol.life sci.70(2013):661
‑
687
[2246]
nikpour,p.et al.,med.oncol 31(2014):955
[2247]
nilsson,r.et al.,nat commun.5(2014):3128
[2248]
nishi,t.et al.,pathol.int.62(2012):802
‑
810
[2249]
nishikata,m.et al.,mol.carcinog 46(2007):208
‑
214
[2250]
niu,n.et al.,genome res 20(2010):1482
‑
1492
[2251]
niu,n.et al.,bmc.cancer 12(2012):422
[2252]
nobori,t.et al.,cancer res 51(1991):3193
‑
3197
[2253]
nobori,t.et al.,cancer res 53(1993):1098
‑
1101
[2254]
noll,j.e.et al.,neoplasia.16(2014):572
‑
585
[2255]
nooter,k.et al.,br.j cancer 76(1997):486
‑
493
[2256]
nord,h.et al.,neuro.oncol 11(2009):803
‑
818
[2257]
norris,m.d.et al.,n.engl.j med.334(1996):231
‑
238
[2258]
noske,a.et al.,exp.mol.pathol.98(2015):47
‑
54
[2259]
novikov,l.et al.,mol.cell biol 31(2011):4244
‑
4255
[2260]
nowarski,r.et al.,blood 120(2012):366
‑
375
[2261]
nymoen,d.a.et al.,gynecol.oncol 139(2015):30
‑
39
[2262]
o'connor,k.w.et al.,cancer res 73(2013):2529
‑
2539
[2263]
o'gorman,d.b.et al.,endocrinology 143(2002):4287
‑
4294
[2264]
o'malley,s.et al.,int.j cancer 125(2009):1805
‑
1813
[2265]
o'reilly,j.a.et al.,plos.one.10(2015):e0123469
[2266]
oberg,e.a.et al.,j biol chem 287(2012):43378
‑
43389
[2267]
obuchowska,i.et al.,klin.oczna 101(1999):167
‑
168
[2268]
odvody,j.et al.,oncogene 29(2010):3287
‑
3296
[2269]
oehler,v.g.et al.,blood 114(2009):3292
‑
3298
[2270]
ogawa,c.et al.,j biol chem 278(2003):1268
‑
1272
[2271]
ogawa,r.et al.,dis.esophagus.21(2008):288
‑
297
[2272]
oguri,t.et al.,mol.cancer ther.7(2008):1150
‑
1155
[2273]
ohba,k.et al.,j urol.174(2005):461
‑
465
[2274]
ohnishi,k.et al.,cancer sci.104(2013):1237
‑
1244
[2275]
ohshima,k.et al.,mol biol.evol.27(2010):2522
‑
2533
[2276]
oishi,y.et al.,tumour.biol 33(2012):383
‑
393
[2277]
okada,m.et al.,proc.natl.acad.sci.u.s.a105(2008):8649
‑
8654
[2278]
okada,s.et al.,j oral pathol.med.44(2015):115
‑
125
[2279]
okosun,j.et al.,nat genet.48(2016):183
‑
188
[2280]
olayioye,m.a.et al.,j biol chem 280(2005):27436
‑
27442
[2281]
oleksowicz,l.et al.,cancer j sci.am.4(1998):247
‑
253
[2282]
olesen,u.h.et al.,apmis 119(2011):296
‑
303
[2283]
olkhanud,p.b.et al.,cancer res 69(2009):5996
‑
6004
[2284]
olsson,l.et al.,leukemia 28(2014):302
‑
310
[2285]
olsson,m.et al.,prostate 67(2007):1439
‑
1446
[2286]
ooe,a.et al.,breast cancer res treat.101(2007):305
‑
315
[2287]
orchel,j.et al.,int.j gynecol.cancer 22(2012):937
‑
944
[2288]
ostrow,k.l.et al.,clin cancer res 16(2010):3463
‑
3472
[2289]
ota,t.et al.,cancer res 62(2002):5168
‑
5177
[2290]
ottaviani,s.et al.,cancer immunol.immunother.55(2006):867
‑
872
[2291]
ou,c.y.et al.,j biol chem 284(2009):20629
‑
20637
[2292]
ozaki,y.et al.,oncol rep.12(2004):1071
‑
1077
[2293]
ozawa,h.et al.,ann.surg.oncol 17(2010):2341
‑
2348
[2294]
ozbas
‑
gerceker,f.et al.,asian pac.j cancer prev.14(2013):5213
‑
5217
[2295]
ozgur,s.et al.,rna.biol 10(2013):528
‑
539
[2296]
palma,m.et al.,bmc.clin pathol.12(2012):2
[2297]
pan,b.et al.,mol.biol rep.40(2013):27
‑
33
[2298]
pan,w.a.et al.,rna.biol 12(2015):255
‑
267
[2299]
pandey,r.n.et al.,oncogene 29(2010):3715
‑
3722
[2300]
pankratz,v.s.et al.,j thorac.oncol 6(2011):1488
‑
1495
[2301]
pannu,v.et al.,oncotarget.6(2015):6076
‑
6091
[2302]
papadakis,m.et al.,fam.cancer 14(2015):599
‑
602
[2303]
papp,b.et al.,biomolecules.2(2012):165
‑
186
[2304]
parihar,a.et al.,life sci.82(2008a):1077
‑
1082
[2305]
parihar,m.s.et al.,biochim.biophys.acta 1780(2008b):921
‑
926
[2306]
parikh,r.a.et al.,genes chromosomes.cancer 53(2014):25
‑
37
[2307]
park,e.et al.,mol.cell 50(2013):908
‑
918
[2308]
park,h.j.et al.,j proteome.res 7(2008):1138
‑
1150
[2309]
park,s.h.et al.,clin cancer res.13(2007):858
‑
867
[2310]
park,s.j.et al.,oncogene 35(2016):1292
‑
1301
[2311]
park,y.et al.,oncogene 34(2015):5037
‑
5045
[2312]
park,y.m.et al.,gene 551(2014):236
‑
242
[2313]
patil,a.a.et al.,oncotarget.5(2014):6414
‑
6424
[2314]
patrick,a.n.et al.,nat struct.mol.biol 20(2013):447
‑
453
[2315]
patrikainen,l.et al.,eur.j clin invest 37(2007):126
‑
133
[2316]
paulo,p.et al.,neoplasia.14(2012):600
‑
611
[2317]
pavelec,d.m.et al.,genetics 183(2009):1283
‑
1295
[2318]
pawar,h.et al.,cancer biol ther.12(2011):510
‑
522
[2319]
pawar,s.et al.,j ovarian.res 7(2014):53
[2320]
peddaboina,c.et al.,bmc.cancer 12(2012):541
[2321]
peeters,m.c.et al.,cell signal.27(2015):2579
‑
2588
[2322]
peltonen,k.et al.,cancer cell 25(2014):77
‑
90
[2323]
pelttari,l.m.et al.,fam.cancer(2015)
[2324]
pender
‑
cudlip,m.c.et al.,cancer sci.104(2013):760
‑
764
[2325]
peng,d.f.et al.,gut 58(2009):5
‑
15
[2326]
peng,h.x.et al.,biomed.res int.2015(2015a):326981
[2327]
peng,j.et al.,sichuan.da.xue.xue.bao.yi.xue.ban.46(2015b):413
‑
416
[2328]
peng,y.et al.,cancer res 75(2015c):378
‑
386
[2329]
perdigao,p.f.et al.,genes chromosomes.cancer 44(2005):204
‑
211
[2330]
pereira,j.s.et al.,endocrine.49(2015):204
‑
214
[2331]
pereira,p.m.et al.,org.biomol.chem.12(2014):1804
‑
1811
[2332]
perez,a.et al.,cancers(basel)6(2014):179
‑
192
[2333]
perez
‑
tomas,r.,curr.med.chem 13(2006):1859
‑
1876
[2334]
perrais,m.et al.,j biol chem 276(2001):15386
‑
15396
[2335]
perrotti,d.et al.,lancet oncol 14(2013):e229
‑
e238
[2336]
personnic,n.et al.,febs j 281(2014):2977
‑
2989
[2337]
perugorria,m.j.et al.,cancer res 69(2009):1358
‑
1367
[2338]
pestov,d.g.et al.,mol.cell biol 21(2001):4246
‑
4255
[2339]
peters,d.g.et al.,cancer epidemiol.biomarkers prev.14(2005):1717
‑
1723
[2340]
peyrard,m.et al.,hum.mol.genet.3(1994):1393
‑
1399
[2341]
peyre,m.et al.,plos.one.5(2010):e12932
[2342]
phipps
‑
yonas,h.et al.,front immunol.4(2013):425
[2343]
phongpradist,r.et al.,curr.pharm.des 16(2010):2321
‑
2330
[2344]
piccolo,s.et al.,cancer res 73(2013):4978
‑
4981
[2345]
piepoli,a.et al.,exp.biol med.(maywood.)237(2012):1123
‑
1128
[2346]
pils,d.et al.,bmc.cancer 13(2013):178
[2347]
pinheiro,j.et al.,nlme:linear and nonlinear mixed effects models(http://cran.r
‑
project.org/packe=nlme) (2015)
[2348]
pio,r.et al.,cancer res 64(2004):4171
‑
4179
[2349]
pissimissis,n.et al.,anticancer res 29(2009):371
‑
377
[2350]
pizzatti,l.et al.,biochim.biophys.acta 1764(2006):929
‑
942
[2351]
placke,t.et al.,blood 124(2014):13
‑
23
[2352]
pleasance,e.d.et al.,nature 463(2010):184
‑
190
[2353]
plebanski,m.et al.,eur.j immunol 25(1995):1783
‑
1787
[2354]
pohl,a.et al.,pharmacogenomics.j 11(2011):93
‑
99
[2355]
poligone,b.et al.,j invest dermatol.135(2015):869
‑
876
[2356]
polisetty,r.v.et al.,j proteomics.74(2011):1918
‑
1925
[2357]
pongor,l.et al.,genome med.7(2015):104
[2358]
poomsawat,s.et al.,j oral pathol.med.39(2010):793
‑
799
[2359]
poortinga,g.et al.,nucleic acids res 39(2011):3267
‑
3281
[2360]
popov,n.et al.,nat cell biol 9(2007):765
‑
774
[2361]
porkka,k.p.et al.,genes chromosomes.cancer 39(2004):1
‑
10
[2362]
porta,c.et al.,virology 202(1994):949
‑
955
[2363]
possuelo,l.g.et al.,rev bras.ginecol.obstet.35(2013):569
‑
574
[2364]
pozo,k.et al.,cancer cell 24(2013):499
‑
511
[2365]
pradhan,m.p.et al.,bmc.syst.biol 7(2013):141
[2366]
prasad,m.l.et al.,head neck 26(2004):1053
‑
1057
[2367]
prasad,n.k.et al.,carcinogenesis 29(2008a):25
‑
34
[2368]
prasad,n.k.et al.,tumour.biol 29(2008b):330
‑
341
[2369]
prunier,c.et al.,cell rep.(2015)
[2370]
pu,h.et al.,world j surg.oncol 13(2015):323
[2371]
puig
‑
butille,j.a.et al.,oncotarget.5(2014):1439
‑
1451
[2372]
puls,f.et al.,am.j surg.pathol.38(2014):1307
‑
1318
[2373]
pulukuri,s.m.et al.,mol.cancer res 7(2009):1285
‑
1293
[2374]
pulvino,m.et al.,blood 120(2012):1668
‑
1677
[2375]
purrington,k.s.et al.,carcinogenesis 35(2014):1012
‑
1019
[2376]
qi,j.et al.,gut(2015)
[2377]
qi,l.et al.,cancer res 74(2014):1301
‑
1306
[2378]
qi,y.et al.,proteomics.5(2005):2960
‑
2971
[2379]
qian,y.et al.,mol.cancer 13(2014):176
[2380]
qian,z.et al.,j exp.clin cancer res 29(2010):111
[2381]
qin,y.et al.,chin med.j(engl.)127(2014):1666
‑
1671
[2382]
quan,j.j.et al.,tumour.biol 36(2015a):8617
‑
8624
[2383]
quan,y.et al.,j cancer 6(2015b):342
‑
350
[2384]
quayle,s.n.et al.,neuro oncol 14(2012):1325
‑
1331
[2385]
quek,h.h.et al.,dna cell biol.16(1997):275
‑
280
[2386]
quidville,v.et al.,cancer res 73(2013):2247
‑
2258
[2387]
rahman,m.et al.,anticancer res 33(2013):113
‑
118
[2388]
raja,s.b.et al.,j cell sci.125(2012):703
‑
713
[2389]
rajalingam,k.et al.,cell cycle 4(2005):1503
‑
1505
[2390]
rajasagi,m.et al.,blood 124(2014):453
‑
462
[2391]
rajkumar,t.et al.,bmc.cancer 11(2011):80
[2392]
ramachandran,c.,curr.pharm.biotechnol.8(2007):99
‑
104
[2393]
rammensee,h.g.et al.,immunogenetics 50(1999):213
‑
219
[2394]
ran,q.et al.,plos.one.9(2014):e85328
[2395]
rangel,l.b.et al.,oncogene 22(2003):7225
‑
7232
[2396]
rao,c.v.et al.,carcinogenesis 30(2009):1469
‑
1474
[2397]
rao,f.et al.,proc.natl.acad.sci.u.s.a 112(2015):1773
‑
1778
[2398]
rappa,g.et al.,mol.cancer res 12(2014):1840
‑
1850
[2399]
rasinpera,h.et al.,gut 54(2005):643
‑
647
[2400]
rastetter,r.h.et al.,bmc.cancer 15(2015):638
[2401]
rausch,m.p.et al.,mol.immunol.68(2015):124
‑
128
[2402]
rauscher,g.h.et al.,bmc.cancer 15(2015):816
[2403]
rawluszko
‑
wieczorek,a.a.et al.,j cancer res clin oncol 141(2015):
1379
‑
1392
[2404]
refseq,the ncbi handbook[internet],chapter 18,(2002),http://www.ncbi.nlm.nih.gov/books/nbk21091/
[2405]
rekhi,b.et al.,indian j med.res 136(2012):766
‑
775
[2406]
remmelink,m.et al.,int.j oncol 26(2005):247
‑
258
[2407]
ren,g.et al.,omics.18(2014):615
‑
624
[2408]
ren,s.et al.,cell res 22(2012):806
‑
821
[2409]
resende,c.et al.,helicobacter.15 suppl 1(2010):34
‑
39
[2410]
restifo,n.p.et al.,j exp.med.177(1993):265
‑
272
[2411]
reuschenbach,m.et al.,fam.cancer 9(2010):173
‑
179
[2412]
rey,o.et al.,oncogene 18(1999):827
‑
831
[2413]
ribeiro,j.r.et al.,front oncol 4(2014):45
[2414]
rieger
‑
christ,k.m.et al.,hum.pathol.32(2001):18
‑
23
[2415]
ries,j.et al.,int.j oncol 26(2005):817
‑
824
[2416]
rimkus,c.et al.,clin gastroenterol.hepatol.6(2008):53
‑
61
[2417]
rini,b.i.et al.,cancer 107(2006):67
‑
74
[2418]
risch,h.a.et al.,j natl.cancer inst.98(2006):1694
‑
1706
[2419]
ritterson,lew c.et al.,nat rev urol.12(2015):383
‑
391
[2420]
roberts,n.j.et al.,cancer discov 2(2012):41
‑
46
[2421]
robin,t.p.et al.,mol.cancer res 10(2012):1098
‑
1108
[2422]
robles,l.d.et al.,j biol chem 277(2002):25431
‑
25438
[2423]
rock,k.l.et al.,science 249(1990):918
‑
921
[2424]
rocken,c.,pathologe 34(2013):403
‑
412
[2425]
rodins,k.et al.,clin cancer res 8(2002):1075
‑
1081
[2426]
rodriguez
‑
paredes,m.et al.,oncogene 33(2014):2807
‑
2813
[2427]
rohrbeck,a.et al.,plos.one.4(2009):e7315
[2428]
rohrmoser,m.et al.,mol.cell biol 27(2007):3682
‑
3694
[2429]
roll,j.d.et al.,mol.cancer 7(2008):15
[2430]
romero,o.a.et al.,cancer discov 4(2014):292
‑
303
[2431]
rondeau,s.et al.,br.j cancer 112(2015):1059
‑
1066
[2432]
rosado,i.v.et al.,rna.10(2004):1073
‑
1083
[2433]
ross,h.m.et al.,mod.pathol.24(2011):390
‑
395
[2434]
rothe,m.et al.,am.j pathol.157(2000):1597
‑
1604
[2435]
rudland,p.s.et al.,am.j pathol.176(2010):2935
‑
2947
[2436]
ruebel,k.h.et al.,endocrine.29(2006):435
‑
444
[2437]
rumiato,e.et al.,cancer chemother.pharmacol.72(2013):483
‑
488
[2438]
ruminy,p.et al.,leukemia 25(2011):681
‑
688
[2439]
russell,r.et al.,nat commun.6(2015):7677
[2440]
ryu,b.et al.,plos.one.2(2007):e594
[2441]
ryu,h.s.et al.,thyroid 24(2014):1232
‑
1240
[2442]
ryu,s.j.et al.,expert.opin.ther.targets.13(2009):479
‑
484
[2443]
s3
‑
leitlinie maligne ovarialtumore,032
‑
035ol,(2013)
[2444]
saddoughi,s.a.et al.,adv.cancer res.117(2013):37
‑
58
[2445]
saeki,n.et al.,genes chromosomes.cancer 48(2009):261
‑
271
[2446]
saelee,p.et al.,asian pac.j cancer prev.10(2009):501
‑
506
[2447]
safadi,r.a.et al.,oral surg.oral med.oral pathol.oral radiol.121(2016):402
‑
411
[2448]
safarinejad,m.r.et al.,urol.oncol 31(2013):1193
‑
1203
[2449]
sahab,z.j.et al.,j cancer 1(2010):14
‑
22
[2450]
saiki,r.k.et al.,science 239(1988):487
‑
491
[2451]
saito,y.et al.,j cancer res clin oncol 139(2013):585
‑
594
[2452]
saito,y.et al.,cancer immunol.res 3(2015):1356
‑
1363
[2453]
sajadian,s.o.et al.,clin epigenetics.7(2015):98
[2454]
sakai,s.et al.,clin chim.acta 413(2012):1542
‑
1548
[2455]
sakamoto,s.et al.,cancer res 70(2010):1885
‑
1895
[2456]
sakurikar,n.et al.,j biol chem 287(2012):39193
‑
39204
[2457]
salon,c.et al.,j pathol.213(2007):303
‑
310
[2458]
saloura,v.et al.,mol.cancer res 13(2015):293
‑
304
[2459]
samimi,g.et al.,cancer epidemiol.biomarkers prev.21(2012):273
‑
279
[2460]
sand,m.et al.,cell tissue res 350(2012):119
‑
126
[2461]
sang,y.et al.,oncotarget.(2015)
[2462]
sankar,s.et al.,mol.cell biol 33(2013):4448
‑
4460
[2463]
sankaran,d.et al.,j biol chem 287(2012):5483
‑
5491
[2464]
santandreu,f.m.et al.,cell physiol biochem.24(2009):379
‑
390
[2465]
santhekadur,p.k.et al.,febs open.bio 4(2014):353
‑
361
[2466]
saraon,p.et al.,mol.cell proteomics.12(2013):1589
‑
1601
[2467]
sarbia,m.et al.,am.j clin pathol.128(2007):255
‑
259
[2468]
sarto,c.et al.,electrophoresis 18(1997):599
‑
604
[2469]
sastre
‑
serra,j.et al.,free radic.biol med.61(2013):11
‑
17
[2470]
sato,f.et al.,int.j mol.med.30(2012a):495
‑
501
[2471]
sato,t.et al.,plos.one.8(2013):e59444
[2472]
sato,t.et al.,j cell sci.125(2012b):1544
‑
1555
[2473]
satoh,a.et al.,oncogene 23(2004):8876
‑
8886
[2474]
sattler,m.et al.,cancer cell 1(2002):479
‑
492
[2475]
savoy,r.m.et al.,endocr.relat cancer 20(2013):r341
‑
r356
[2476]
sawicka
‑
gutaj,n.et al.,tumour.biol 36(2015):7859
‑
7863
[2477]
sayagues,j.m.et al.,med.clin(barc.)128(2007):226
‑
232
[2478]
scagliotti,g.v.et al.,ann.oncol 10 suppl 5(1999):s83
‑
s86
[2479]
scanlan,m.j.et al.,cancer immun.1(2001):4
[2480]
scanlan,m.j.et al.,cancer res 62(2002):4041
‑
4047
[2481]
schaner,m.e.et al.,mol.biol cell 14(2003):4376
‑
4386
[2482]
scharadin,t.m.et al.,plos.one.6(2011):e23230
[2483]
scheffer,g.l.et al.,curr.opin.oncol 12(2000):550
‑
556
[2484]
schiffmann,s.et al.,carcinogenesis 30(2009):745
‑
752
[2485]
schimanski,c.c.et al.,oncogene 24(2005):3100
‑
3109
[2486]
schleiermacher,g.et al.,oncogene 24(2005):3377
‑
3384
[2487]
schlumbrecht,m.p.et al.,mod.pathol.24(2011):453
‑
462
[2488]
schmidt,s.v.et al.,oncotarget.6(2015):8635
‑
8647
[2489]
schoppmann,s.f.et al.,clin cancer res 19(2013):5329
‑
5339
[2490]
schraders,m.et al.,br.j haematol.143(2008):210
‑
221
[2491]
schramm,a.et al.,nat genet.47(2015):872
‑
877
[2492]
schrier,s.a.et al.,curr.opin.ophthalmol.22(2011):325
‑
331
[2493]
schuetz,j.m.et al.,cancer epidemiol.biomarkers prev.21(2012):2272
‑
2274
[2494]
schulte,i.et al.,bmc.genomics 13(2012):719
[2495]
scott,a.f.et al.,genes(basel)5(2014):366
‑
384
[2496]
scotto,l.et al.,genes chromosomes.cancer 47(2008):755
‑
765
[2497]
sears,d.et al.,cell death.dis.1(2010):e93
[2498]
sedoris,k.c.et al.,bmc.cancer 10(2010):157
[2499]
seeger,f.h.et al.,immunogenetics 49(1999):571
‑
576
[2500]
seeling,j.m.et al.,science 283(1999):2089
‑
2091
[2501]
seetoo,d.q.et al.,j surg.oncol 82(2003):184
‑
193
[2502]
seidel,c.et al.,mol.carcinog 46(2007):865
‑
871
[2503]
seidl,c.et al.,invest new drugs 28(2010):49
‑
60
[2504]
sekine,i.et al.,jpn.j clin oncol 37(2007):329
‑
336
[2505]
selamat,s.a.et al.,plos.one.6(2011):e21443
[2506]
seliger,b.,methods mol.biol 1102(2014):367
‑
380
[2507]
seliger,b.et al.,proteomics.5(2005):2631
‑
2640
[2508]
seo,s.w.et al.,j orthop.res 29(2011):1131
‑
1136
[2509]
seol,h.s.et al.,cancer lett.353(2014):232
‑
241
[2510]
seriramalu,r.et al.,electrophoresis 31(2010):2388
‑
2395
[2511]
servais,e.l.et al.,clin cancer res 18(2012):2478
‑
2489
[2512]
seshagiri,s.et al.,nature 488(2012):660
‑
664
[2513]
shackelford,r.e.et al.,int.j clin exp.pathol.3(2010):522
‑
527
[2514]
shadeo,a.et al.,bmc.genomics 9(2008):64
[2515]
shah,s.p.et al.,nature 461(2009):809
‑
813
[2516]
shah,t.m.et al.,oral oncol 49(2013):604
‑
610
[2517]
shames,d.s.et al.,clin cancer res 19(2013):6912
‑
6923
[2518]
shan,t.et al.,oncol rep.32(2014):1564
‑
1570
[2519]
shang,b.et al.,cell death.dis.5(2014):e1285
[2520]
shao,j.et al.,plos.one.9(2014):e97085
[2521]
sharma,a.et al.,tumour.biol 34(2013):3249
‑
3257
[2522]
shaughnessy,j.d.,jr.et al.,blood 118(2011):3512
‑
3524
[2523]
shaw,e.j.et al.,cell oncol(dordr.)34(2011):355
‑
367
[2524]
shen,c.et al.,cancer res 73(2013):3393
‑
3401
[2525]
shen,y.et al.,oncotarget.6(2015a):20396
‑
20403
[2526]
shen,y.et al.,cancer cell microenviron.2(2015b)
[2527]
sheng,s.h.et al.,clin transl.oncol 16(2014):153
‑
157
[2528]
sherman,f.et al.,laboratory course manual for methods in yeast genetics(1986)
[2529]
shi,d.et al.,biochem.biophys.res commun.450(2014a):1241
‑
1246
[2530]
shi,h.et al.,world j surg.oncol 12(2014b):188
[2531]
shi,j.et al.,oncogene 25(2006):4923
‑
4936
[2532]
shi,j.et al.,am.j cancer res 2(2012):116
‑
129
[2533]
shi,j.l.et al.,oncotarget.6(2015):5299
‑
5309
[2534]
shields,b.j.et al.,mol.cell biol 33(2013):557
‑
570
[2535]
shih,iem et al.,am.j pathol.178(2011):1442
‑
1447
[2536]
shin,e.m.et al.,j clin invest 124(2014):3807
‑
3824
[2537]
shiraishi,t.et al.,j transl.med.9(2011):153
[2538]
shishkin,s.s.et al.,biochemistry(mosc.)78(2013):1415
‑
1430
[2539]
shruthi,d.k.et al.,j oral maxillofac.pathol.18(2014):365
‑
371
[2540]
shtutman,m.et al.,proc.natl.acad.sci.u.s.a 108(2011):12449
‑
12454
[2541]
shu,g.s.et al.,cancer biomark.11(2012):107
‑
114
[2542]
shu,j.et al.,cancer res.66(2006):5077
‑
5084
[2543]
sidhar,s.k.et al.,hum.mol.genet.5(1996):1333
‑
1338
[2544]
siligan,c.et al.,oncogene 24(2005):2512
‑
2524
[2545]
silva,j.m.et al.,cell 137(2009):1047
‑
1061
[2546]
silveira,s.m.et al.,plos.one.8(2013):e67643
[2547]
simaga,s.et al.,eur.j cancer 34(1998):399
‑
405
[2548]
simaga,s.et al.,gynecol.oncol 91(2003):194
‑
200
[2549]
simonova,o.a.et al.,mol.biol(mosk)49(2015):667
‑
677
[2550]
simons,a.l.et al.,lab invest 93(2013):711
‑
719
[2551]
singh,g.,pharmaceuticals.(basel)7(2014):192
‑
206
[2552]
singh,h.et al.,am.j obstet.gynecol.198(2008):303
‑
306
[2553]
singh,p.k.et al.,immunobiology 220(2015):103
‑
108
[2554]
singh,r.et al.,febs j 281(2014):1629
‑
1641
[2555]
singh
‑
jasuja,h.et al.,cancer immunol.immunother.53(2004):187
‑
195
[2556]
sinha,s.et al.,mol.oncol 5(2011):454
‑
464
[2557]
skandalis,s.s.et al.,matrix biol 35(2014):182
‑
193
[2558]
slattery,m.l.et al.,carcinogenesis 31(2010):1604
‑
1611
[2559]
slattery,m.l.et al.,mol.carcinog 52(2013):155
‑
166
[2560]
slipicevic,a.et al.,bmc.cancer 8(2008):276
[2561]
smaaland,r.et al.,breast cancer res treat.9(1987):53
‑
59
[2562]
small,e.j.et al.,j clin oncol.24(2006):3089
‑
3094
[2563]
smetsers,s.et al.,fam.cancer 11(2012):661
‑
665
[2564]
smith,b.et al.,cell rep.2(2012):580
‑
590
[2565]
smith,j.b.et al.,gynecol.oncol 134(2014):181
‑
189
[2566]
sohr,s.et al.,cell cycle 7(2008):3448
‑
3460
[2567]
soman,n.r.et al.,proc.natl.acad.sci.u.s.a 88(1991):4892
‑
4896
[2568]
soman,n.r.et al.,proc.natl.acad.sci.u.s.a87(1990):738
‑
742
[2569]
song,b.et al.,mol.cancer ther.12(2013a):58
‑
68
[2570]
song,j.et al.,world j gastroenterol.19(2013b):4127
‑
4136
[2571]
song,l.j.et al.,j mol.med.(berl)90(2012):707
‑
718
[2572]
song,n.et al.,j zhejiang.univ sci.b 14(2013c):451
‑
459
[2573]
song,t.et al.,oncol lett.9(2015a):2799
‑
2804
[2574]
song,x.et al.,monoclon.antib.immunodiagn.immunother.33(2014a):246
‑
253
[2575]
song,x.c.et al.,mol.cell proteomics.7(2008):163
‑
169
[2576]
song,y.et al.,nature 509(2014b):91
‑
95
[2577]
song,y.et al.,int.j clin exp.pathol.8(2015b):11314
‑
11322
[2578]
song,y.et al.,biochem.j 406(2007):427
‑
436
[2579]
sonora,c.et al.,j histochem.cytochem.54(2006):289
‑
299
[2580]
soupene,e.et al.,j lipid res.49(2008):1103
‑
1112
[2581]
sousa,s.f.et al.,endocr.relat cancer 22(2015):399
‑
408
[2582]
sowalsky,a.g.et al.,cancer res 71(2011):758
‑
767
[2583]
sowalsky,a.g.et al.,mol.cancer res.13(2015):98
‑
106
[2584]
sporn,j.c.et al.,am.j pathol.180(2012):2516
‑
2526
[2585]
spyropoulou,a.et al.,neuromolecular.med.16(2014):70
‑
82
[2586]
srinivasan,d.et al.,cancer res 66(2006):5648
‑
5655
[2587]
srinivasan,d.et al.,oncogene 27(2008):1095
‑
1105
[2588]
st
‑
denis,n.et al.,mol.cell proteomics.14(2015):946
‑
960
[2589]
stacey,s.n.et al.,nat commun.6(2015):6825
[2590]
stadler,w.m.et al.,cancer res 54(1994):2060
‑
2063
[2591]
stangel,d.et al.,j surg.res 197(2015):91
‑
100
[2592]
stary,s.et al.,genes chromosomes.cancer 52(2013):33
‑
43
[2593]
stawerski,p.et al.,pol.j pathol.61(2010):219
‑
223
[2594]
stefanska,b.et al.,clin cancer res 20(2014):3118
‑
3132
[2595]
steffen,j.s.et al.,virchows arch.461(2012):355
‑
365
[2596]
steinbach,d.et al.,clin cancer res 12(2006):4357
‑
4363
[2597]
steinestel,k.et al.,mol.cancer 13(2014):145
[2598]
steinestel,k.et al.,pathologe 34 suppl 2(2013):189
‑
194
[2599]
steinmann,k.et al.,oncol rep.22(2009):1519
‑
1526
[2600]
stirewalt,d.l.et al.,genes chromosomes.cancer 47(2008):8
‑
20
[2601]
stirpe,f.et al.,am.j gastroenterol.97(2002):2079
‑
2085
[2602]
stoiber,d.et al.,j clin invest 114(2004):1650
‑
1658
[2603]
stone,b.et al.,gene 267(2001):173
‑
182
[2604]
stransky,n.et al.,nat commun.5(2014):4846
[2605]
strock,c.j.et al.,cancer res 66(2006):7509
‑
7515
[2606]
strojnik,t.et al.,anticancer res 26(2006):2887
‑
2900
[2607]
strojnik,t.et al.,anticancer res 29(2009):3269
‑
3279
[2608]
stubbs,a.p.et al.,am.j pathol.154(1999):1335
‑
1343
[2609]
sturm,m.et al.,bmc.bioinformatics.9(2008):163
[2610]
su,m.t.et al.,gynecol.oncol 103(2006):357
‑
360
[2611]
su,y.f.et al.,j biomed.sci.21(2014):67
[2612]
subramanian,m.et al.,j clin endocrinol.metab 94(2009):1467
‑
1471
[2613]
suchy,j.et al.,bmc.cancer 8(2008):112
[2614]
sud,n.et al.,int.j cancer 112(2004):905
‑
907
[2615]
sudo,h.et al.,genomics 95(2010):210
‑
216
[2616]
sueoka,s.et al.,ann.surg.oncol(2015)
[2617]
sugano,g.et al.,oncogene 30(2011):642
‑
653
[2618]
sugimoto,k.j.et al.,int.j clin exp.pathol.7(2014):8980
‑
8987
[2619]
sugimoto,t.et al.,genes chromosomes.cancer 48(2009):132
‑
142
[2620]
suh,j.h.et al.,mol.endocrinol.22(2008):33
‑
46
[2621]
sukocheva,o.a.et al.,world j gastroenterol.21(2015):6146
‑
6156
[2622]
sullivan,g.f.et al.,j clin invest 105(2000):1261
‑
1267
[2623]
sun,a.et al.,prostate(2015a)
[2624]
sun,d.w.et al.,cancer epidemiol.(2015b)
[2625]
sun,h.et al.,j buon.20(2015c):296
‑
308
[2626]
sun,j.y.et al.,zhonghua kou qiang.yi.xue.za zhi.39(2004a):114
‑
117
[2627]
sun,k.et al.,tumour.biol 36(2015d):1549
‑
1559
[2628]
sun,n.k.et al.,oncotarget.6(2015e):27065
‑
27082
[2629]
sun,q.y.et al.,j pathol.235(2015f):559
‑
570
[2630]
sun,s.et al.,gene 584(2016):90
‑
96
[2631]
sun,s.et al.,j proteome.res 9(2010):70
‑
78
[2632]
sun,w.et al.,cancer lett.212(2004b):83
‑
93
[2633]
sun,x.et al.,int.j oncol 44(2014a):1678
‑
1684
[2634]
sun,y.et al.,oncogene 34(2015g):2527
‑
2537
[2635]
sun,y.et al.,carcinogenesis 35(2014b):1941
‑
1950
[2636]
sun,y.et al.,eur.j cancer prev.23(2014c):418
‑
424
[2637]
sun,y.et al.,asian j androl 16(2014d):319
‑
324
[2638]
sung,w.w.et al.,bmc.cancer 14(2014):951
[2639]
surmann,e.m.et al.,cancer immunol.immunother.64(2015):357
‑
366
[2640]
suzuki,h.et al.,lung cancer 59(2008):24
‑
31
[2641]
svendsen,j.m.et al.,cell 138(2009):63
‑
77
[2642]
svojgr,k.et al.,immunol.lett.122(2009):185
‑
192
[2643]
svojgr,k.et al.,exp.hematol.40(2012):379
‑
385
[2644]
swanson,k.d.et al.,genes chromosomes.cancer 47(2008):253
‑
259
[2645]
swift,m.et al.,n.engl.j med.316(1987):1289
‑
1294
[2646]
symes,a.j.et al.,plos.one.8(2013):e84295
[2647]
szabo,p.m.et al.,virchows arch.455(2009):133
‑
142
[2648]
szaflarski,w.et al.,postepy biochem.57(2011):266
‑
273
[2649]
szczepanski,m.j.et al.,oral oncol 49(2013):144
‑
151
[2650]
szczepanski,m.j.et al.,biomark.med.7(2013):575
‑
578
[2651]
szuhai,k.et al.,clin cancer res 15(2009):2259
‑
2268
[2652]
tagawa,h.,nihon rinsho 72(2014):1052
‑
1057
[2653]
tahara,h.et al.,prostate cancer prostatic.dis.18(2015):56
‑
62
[2654]
tahara,k.et al.,cancer 85(1999):1234
‑
1240
[2655]
tahara,t.et al.,gastroenterology 146(2014):530
‑
538
[2656]
tai,c.j.et al.,int.j biol markers 27(2012):e280
‑
e284
[2657]
tai,w.et al.,mol.pharm.10(2013):477
‑
487
[2658]
takahashi,k.et al.,int.j oncol 28(2006):321
‑
328
[2659]
takahashi,y.et al.,ann.oncol 26(2015):935
‑
942
[2660]
takao,m.et al.,oncol rep.17(2007):1333
‑
1339
[2661]
takaoka,n.et al.,bmc.mol.biol 12(2011):31
[2662]
takashima,s.et al.,tumour.biol.35(2014):4257
‑
4265
[2663]
takata,a.et al.,hepatology 57(2013a):162
‑
170
[2664]
takata,k.et al.,nat commun.4(2013b):2338
[2665]
takayanagi,s.et al.,j exp.ther.oncol 4(2004):239
‑
246
[2666]
takeda,s.et al.,j toxicol.sci.39(2014):711
‑
716
[2667]
takemoto,h.et al.,int.j cancer 91(2001):783
‑
788
[2668]
takenokuchi,m.et al.,anticancer res 35(2015):3307
‑
3316
[2669]
takeyama,k.et al.,j biol chem 278(2003):21930
‑
21937
[2670]
talieri,m.et al.,thromb.haemost.91(2004):180
‑
186
[2671]
tamir,a.et al.,j ovarian.res 7(2014):109
[2672]
tan,j.a.et al.,mol.cell endocrinol.382(2014):302
‑
313
[2673]
tan,p.et al.,biochem.biophys.res commun.419(2012):801
‑
808
[2674]
tan,x.et al.,int.j cancer 123(2008):1080
‑
1088
[2675]
tanahashi,n.et al.,biochem.biophys.res commun.243(1998):229
‑
232
[2676]
tanaka,m.et al.,cancer sci.99(2008a):979
‑
985
[2677]
tanaka,y.et al.,j hepatol.49(2008b):746
‑
757
[2678]
tang,c.y.et al.,clin chem lab med.52(2014a):1843
‑
1850
[2679]
tang,h.et al.,int.j mol.med.32(2013):381
‑
388
[2680]
tang,n.et al.,sheng li xue.bao.62(2010):196
‑
202
[2681]
tang,s.et al.,xi.bao.yu fen.zi.mian.yi.xue.za zhi.30(2014b):411
‑
413
[2682]
taniuchi,k.et al.,cancer res 65(2005):105
‑
112
[2683]
tanner,m.m.et al.,clin cancer res 1(1995):1455
‑
1461
[2684]
tano,k.et al.,febs lett.584(2010):4575
‑
4580
[2685]
tao,f.et al.,world j gastroenterol.20(2014a):9564
‑
9569
[2686]
tao,j.et al.,tumour.biol 35(2014b):4389
‑
4399
[2687]
tao,t.et al.,cell res 23(2013):620
‑
634
[2688]
taouji,s.et al.,j biol chem 288(2013):17190
‑
17201
[2689]
tarcic,o.et al.,cell rep.14(2016):1462
‑
1476
[2690]
tatidis,l.et al.,j lipid res 38(1997):2436
‑
2445
[2691]
tatsuka,m.et al.,cancer res 58(1998):4811
‑
4816
[2692]
taube,e.t.et al.,gynecol.oncol 140(2016):494
‑
502
[2693]
tedeschi,p.m.et al.,mol.cancer res(2015)
[2694]
teh,m.t.et al.,cancer res 62(2002):4773
‑
4780
[2695]
terada,t.,int.j clin exp.pathol.5(2012):596
‑
600
[2696]
teufel,r.et al.,cell mol life sci.62(2005):1755
‑
1762
[2697]
thakkar,a.d.et al.,biomark.cancer 2(2010):1
‑
15
[2698]
thang,n.d.et al.,oncotarget.6(2015):14290
‑
14299
[2699]
theiss,a.l.et al.,biochim.biophys.acta 1813(2011):1137
‑
1143
[2700]
thomas,a.et al.,cancer med.2(2013):836
‑
848
[2701]
thome,c.h.et al.,mol.cell proteomics.11(2012):1898
‑
1912
[2702]
thompson,d.a.et al.,eur.j biochem.252(1998):169
‑
177
[2703]
thorell,k.et al.,bmc.med.genomics 2(2009):53
[2704]
tian,x.et al.,oncol rep.34(2015):707
‑
714
[2705]
tibaldi,l.et al.,plos.one.8(2013):e72708
[2706]
timofeeva,o.a.et al.,int.j oncol 35(2009):751
‑
760
[2707]
tomiyama,l.et al.,oncogene 34(2015):1141
‑
1149
[2708]
tomonaga,m.et al.,int.j oncol 40(2012):409
‑
417
[2709]
tong,w.g.et al.,epigenetics.5(2010):499
‑
508
[2710]
toogeh,g.et al.,clin lymphoma myeloma.leuk.16(2016):e21
‑
e26
[2711]
torres
‑
reyes,l.a.et al.,int.j clin exp.pathol.7(2014):7409
‑
7418
[2712]
tozbikian,g.et al.,plos.one.9(2014):e114900
[2713]
tran,e.et al.,science 344(2014):641
‑
645
[2714]
travis,r.c.et al.,int.j cancer 132(2013):1901
‑
1910
[2715]
trehoux,s.et al.,biochem.biophys.res commun.456(2015):757
‑
762
[2716]
trifonov,v.et al.,bmc.syst.biol 7(2013):25
[2717]
tripodi,d.et al.,bmc.med.genomics 2(2009):65
[2718]
trotta,c.r.et al.,nature 441(2006):375
‑
377
[2719]
tsai,f.m.et al.,cell signal.18(2006):349
‑
358
[2720]
tsao,d.a.et al.,dnacell biol 29(2010):285
‑
293
[2721]
tsao,t.y.et al.,mol.cell biochem.327(2009):163
‑
170
[2722]
tsou,j.h.et al.,j pathol.225(2011):243
‑
254
[2723]
tsujikawa,t.et al.,int.j cancer 132(2013):2755
‑
2766
[2724]
tsukamoto,y.et al.,j pathol.216(2008):471
‑
482
[2725]
tsuruga,t.et al.,oncol res 16(2007):431
‑
435
[2726]
tu,l.c.et al.,mol.cell proteomics.6(2007):575
‑
588
[2727]
tucci,m.et al.,curr.top.med.chem 9(2009):218
‑
224
[2728]
tummala,r.et al.,cancer chemother.pharmacol.64(2009):1187
‑
1194
[2729]
tung,m.c.et al.,cancer epidemiol.biomarkers prev.18(2009):1570
‑
1577
[2730]
tung,p.y.et al.,stem cells 31(2013):2330
‑
2342
[2731]
turner,a.et al.,plos.one.8(2013):e56817
[2732]
turner,b.c.et al.,cancer res 58(1998):5466
‑
5472
[2733]
turtoi,a.et al.,j proteome.res 10(2011):4302
‑
4313
[2734]
twa,d.d.et al.,j pathol.236(2015):136
‑
141
[2735]
uchikado,y.et al.,int.j oncol 29(2006):1337
‑
1347
[2736]
uchiyama,k.et al.,j cell biol.159(2002):855
‑
866
[2737]
uemura,m.et al.,cancer 97(2003):2474
‑
2479
[2738]
ulloa,f.et al.,plos.one.10(2015):e0119707
[2739]
unger,k.et al.,endocr.relat cancer 17(2010):87
‑
98
[2740]
urbanucci,a.et al.,oncogene 31(2012):2153
‑
2163
[2741]
uyama,h.et al.,clin cancer res 12(2006):6043
‑
6048
[2742]
vahedi,s.et al.,oncol rep.34(2015):43
‑
50
[2743]
vainio,p.et al.,plos.one.7(2012):e39801
[2744]
vairaktaris,e.et al.,anticancer res 27(2007):4121
‑
4125
[2745]
vaites,l.p.et al.,mol.cell biol 31(2011):4513
‑
4523
[2746]
vakana,e.et al.,plos.one.8(2013):e78780
[2747]
valles,i.et al.,plos.one.7(2012):e42086
[2748]
valque,h.et al.,plos.one.7(2012):e46699
[2749]
van de rijn,m.et al.,am.j pathol.161(2002):1991
‑
1996
[2750]
van den heuvel
‑
eibrink mm et al.,int.j clin pharmacol.ther.38(2000):94
‑
110
[2751]
van der zwan,y.g.et al.,eur.urol.67(2015):692
‑
701
[2752]
van dijk,j.r.et al.,biochem.j 459(2014):27
‑
36
[2753]
van ginkel,p.r.et al.,biochim.biophys.acta 1448(1998):290
‑
297
[2754]
van vuurden,d.g.et al.,neuro.oncol 16(2014):946
‑
959
[2755]
van,agthoven t.et al.,j clin oncol 27(2009):542
‑
549
[2756]
van,dam s.et al.,bmc.genomics 13(2012):535
[2757]
van,seuningen,i et al.,biochem.j 348 pt 3(2000):675
‑
686
[2758]
vanaja,d.k.et al.,clin cancer res 12(2006):1128
‑
1136
[2759]
vanderstraeten,a.et al.,cancer immunol.immunother.63(2014):545
‑
557
[2760]
vanharanta,s.et al.,elife.3(2014)
[2761]
vanneste,d.et al.,curr.biol.19(2009):1712
‑
1717
[2762]
vasca,v.et al.,oncol lett.8(2014):2501
‑
2504
[2763]
vater,i.et al.,leukemia 29(2015):677
‑
685
[2764]
vavougios,g.d.et al.,am.j physiol lung cell mol.physiol 309(2015):l677
‑
l686
[2765]
veigaard,c.et al.,cancer genet.204(2011):516
‑
521
[2766]
vekony,h.et al.,oral oncol 45(2009):259
‑
265
[2767]
venere,m.et al.,sci.transl.med.7(2015):304ra143
[2768]
verheugd,p.et al.,nat commun.4(2013):1683
[2769]
vermeulen,c.f.et al.,gynecol.oncol 105(2007):593
‑
599
[2770]
vey,n.et al.,oncogene 23(2004):9381
‑
9391
[2771]
vincent,a.et al.,oncotarget.5(2014):2575
‑
2587
[2772]
vincent
‑
chong,v.k.et al.,oral dis.18(2012):469
‑
476
[2773]
viswanathan,m.et al.,clin cancer res 9(2003):1057
‑
1062
[2774]
vitale,m.et al.,cancer res 58(1998):737
‑
742
[2775]
vlaykova,t.et al.,j buon.16(2011):265
‑
273
[2776]
vogetseder,a.et al.,int.j cancer 133(2013):2362
‑
2371
[2777]
volkmer,j.p.et al.,proc.natl.acad.sci.u.s.a109(2012):2078
‑
2083
[2778]
vrabel,d.et al.,klin.onkol.27(2014):340
‑
346
[2779]
walker,f.et al.,biol chem 395(2014):1075
‑
1086
[2780]
walsh,m.d.et al.,mod.pathol.26(2013):1642
‑
1656
[2781]
walter,s.et al.,j immunol 171(2003):4974
‑
4978
[2782]
walter,s.et al.,nat med.18(2012):1254
‑
1261
[2783]
wang,b.s.et al.,clin sci.(lond)124(2013a):203
‑
214
[2784]
wang,b.s.et al.,cell stress.chaperones.18(2013b):359
‑
366
[2785]
wang,c.et al.,mol.cancer res 5(2007a):1031
‑
1039
[2786]
wang,c.et al.,nucleic acids res 43(2015a):4893
‑
4908
[2787]
wang,c.et al.,clin cancer res 4(1998):567
‑
576
[2788]
wang,c.j.et al.,mol.biol rep.40(2013c):6525
‑
6531
[2789]
wang,c.x.et al.,asian pac.j cancer prev.15(2014a):355
‑
362
[2790]
wang,d.et al.,j biol.chem.277(2002):36216
‑
36222
[2791]
wang,e.et al.,proc.natl.acad.sci.u.s.a 110(2013d):3901
‑
3906
[2792]
wang,f.et al.,oncol rep.30(2013e):260
‑
268
[2793]
wang,g.et al.,biochem.j 446(2012a):415
‑
425
[2794]
wang,g.r.et al.,acta pharmacol.sin.30(2009a):1436
‑
1442
[2795]
wang,h.et al.,j biol.chem 289(2014b):4009
‑
4017
[2796]
wang,h.et al.,j biol chem 289(2014c):23123
‑
23131
[2797]
wang,h.et al.,chin med.j(engl.)116(2003):1074
‑
1077
[2798]
wang,h.et al.,j cancer res ther.11 suppl 1(2015b):c74
‑
c79
[2799]
wang,j.et al.,oncotarget.6(2015c):16527
‑
16542
[2800]
wang,j.et al.,asian pac.j cancer prev.14(2013f):2805
‑
2809
[2801]
wang,j.w.et al.,oncogene 23(2004):4089
‑
4097
[2802]
wang,k.et al.,j biol chem 289(2014d):23928
‑
23937
[2803]
wang,l.et al.,acta med.okayama 65(2011):315
‑
323
[2804]
wang,l.et al.,int.j cancer 124(2009b):1526
‑
1534
[2805]
wang,l.et al.,cancer cell 25(2014e):21
‑
36
[2806]
wang,m.et al.,int.j mol.med.33(2014f):1019
‑
1026
[2807]
wang,n.et al.,mol.biol rep.39(2012b):10497
‑
10504
[2808]
wang,p.et al.,zhongguo fei.ai.za zhi.12(2009c):875
‑
878
[2809]
wang,q.et al.,cell 138(2009d):245
‑
256
[2810]
wang,q.et al.,mol.med.rep.12(2015d):475
‑
481
[2811]
wang,s.s.et al.,plos.one.5(2010):e8667
[2812]
wang,s.y.et al.,oncotarget.7(2016a):2878
‑
2888
[2813]
wang,t.et al.,clin transl.oncol 17(2015e):564
‑
569
[2814]
wang,v.w.et al.,head neck 35(2013g):831
‑
835
[2815]
wang,w.w.et al.,int.j clin exp.med.8(2015f):3063
‑
3071
[2816]
wang,w.x.et al.,sichuan.da.xue.xue.bao.yi.xue.ban.40(2009e):857
‑
860
[2817]
wang,x.et al.,int.j clin exp.med.8(2015g):1780
‑
1791
[2818]
wang,x.et al.,hum.pathol.44(2013h):2020
‑
2027
[2819]
wang,x.et al.,am.j cancer res 5(2015h):2590
‑
2604
[2820]
wang,x.et al.,j biol chem 290(2015i):3925
‑
3935
[2821]
wang,x.et al.,oncotarget.7(2016b):8029
‑
8042
[2822]
wang,x.et al.,hum.immunol.75(2014g):1203
‑
1209
[2823]
wang,x.et al.,biochim.biophys.acta 1783(2008):1220
‑
1228
[2824]
wang,x.et al.,int.j biol markers 29(2014h):e150
‑
e159
[2825]
wang,x.x.et al.,hepatobiliary.pancreat.dis.int.12(2013i):540
‑
545
[2826]
wang,x.x.et al.,plos.one.9(2014i):e96501
[2827]
wang,y.et al.,j thorac.dis.7(2015j):672
‑
679
[2828]
wang,y.et al.,oncogene 31(2012c):2512
‑
2520
[2829]
wang,y.et al.,j biomed.sci.22(2015k):52
[2830]
wang,y.et al.,pathol.oncol res.20(2014):611
‑
618
[2831]
wang,y.f.et al.,tumour.biol 34(2013j):1685
‑
1689
[2832]
wang,z.et al.,cancer res 67(2007b):8293
‑
8300
[2833]
warfel,n.a.et al.,cell cycle 12(2013):3689
‑
3701
[2834]
waseem,a.et al.,oral oncol 46(2010):536
‑
542
[2835]
watanabe,m.et al.,proteomics.clin appl.2(2008):925
‑
935
[2836]
watanabe,t.et al.,clin colorectal cancer 10(2011):134
‑
141
[2837]
waters,m.g.et al.,nature 349(1991):248
‑
251
[2838]
watson,p.j.et al.,traffic.5(2004):79
‑
88
[2839]
watts,c.a.et al.,chem biol 20(2013):1399
‑
1410
[2840]
wazir,u.et al.,cancer genomics proteomics.10(2013):69
‑
73
[2841]
wazir,u.et al.,oncol rep.33(2015a):1450
‑
1458
[2842]
wazir,u.et al.,oncol rep.33(2015b):2575
‑
2582
[2843]
weber,a.m.et al.,pharmacol.ther(2014)
[2844]
weeks,l.d.et al.,mol.cancer ther.12(2013):2248
‑
2260
[2845]
wegiel,b.et al.,j natl.cancer inst.100(2008):1022
‑
1036
[2846]
wei,p.et al.,j transl.med.11(2013):313
[2847]
wei,x.et al.,nat genet.43(2011):442
‑
446
[2848]
wei,y.p.et al.,xi.bao.yu fen.zi.mian.yi.xue.za zhi.28(2012):354
‑
357
[2849]
weidle,u.h.et al.,clin exp.metastasis 32(2015):623
‑
635
[2850]
weigert,o.et al.,cancer discov 2(2012):47
‑
55
[2851]
wen,j.l.et al.,plos.one.10(2015):e0115622
[2852]
wenzel,j.et al.,int.j cancer 123(2008):2605
‑
2615
[2853]
werner,s.et al.,j biol chem 288(2013):22993
‑
23008
[2854]
weterman,m.a.et al.,cytogenet.cell genet.92(2001):326
‑
332
[2855]
weterman,m.a.et al.,proc.natl.acad.sci.u.s.a93(1996):15294
‑
15298
[2856]
wharton,s.b.et al.,neuropathol.appl.neurobiol.27(2001):305
‑
313
[2857]
wheler,j.j.et al.,bmc.cancer 15(2015):442
[2858]
whitaker
‑
menezes,d.et al.,cell cycle 10(2011):4047
‑
4064
[2859]
white,c.d.et al.,bmc.gastroenterol.10(2010):125
[2860]
wijdeven,r.h.et al.,cancer res 75(2015):4176
‑
4187
[2861]
wikman,h.et al.,genes chromosomes.cancer 42(2005):193
‑
199
[2862]
willcox,b.e.et al.,protein sci.8(1999):2418
‑
2423
[2863]
williams,k.a.et al.,plos.genet.10(2014):e1004809
[2864]
wilson,i.m.et al.,oncogene 33(2014):4464
‑
4473
[2865]
wilting,s.m.et al.,genes chromosomes.cancer 47(2008):890
‑
905
[2866]
wirtenberger,m.et al.,carcinogenesis 27(2006):1655
‑
1660
[2867]
wissing,m.d.et al.,oncotarget.5(2014):7357
‑
7367
[2868]
wong,n.et al.,j hepatol.38(2003):298
‑
306
[2869]
wong,s.q.et al.,oncotarget.6(2015):1115
‑
1127
[2870]
woo,j.et al.,biochem.biophys.res commun.367(2008):291
‑
298
[2871]
wood,l.m.et al.,cancer immunol.immunother.61(2012):689
‑
700
[2872]
wright,d.g.et al.,anticancer res 16(1996):3349
‑
3351
[2873]
wrzeszczynski,k.o.et al.,plos.one.6(2011):e28503
[2874]
wu,c.et al.,bmc.bioinformatics.13(2012a):182
[2875]
wu,c.c.et al.,proteomics.clin appl.2(2008):1586
‑
1595
[2876]
wu,c.c.et al.,biochim.biophys.acta 1823(2012b):2227
‑
2236
[2877]
wu,c.y.et al.,j biomed.sci.18(2011a):1
[2878]
wu,h.et al.,nat med.17(2011b):347
‑
355
[2879]
wu,h.c.et al.,nat commun.5(2014a):3214
[2880]
wu,j.et al.,oncogene 31(2012c):333
‑
341
[2881]
wu,j.et al.,acs chem biol 8(2013a):2201
‑
2208
[2882]
wu,m.z.et al.,cancer res 75(2015):3912
‑
3924
[2883]
wu,t.et al.,hepatology 36(2002):363
‑
373
[2884]
wu,t.t.et al.,chin j physiol 49(2006):192
‑
198
[2885]
wu,w.et al.,cancer res 67(2007):951
‑
958
[2886]
wu,x.et al.,hum.mol.genet.21(2012d):456
‑
462
[2887]
wu,y.et al.,biomed.res 33(2012e):75
‑
82
[2888]
wu,y.et al.,cancer sci.103(2012f):1820
‑
1825
[2889]
wu,y.et al.,cell rep.5(2013b):224
‑
236
[2890]
wu,y.et al.,j surg.oncol 105(2012g):724
‑
730
[2891]
wu,z.et al.,neoplasia.11(2009):66
‑
76
[2892]
wu,z.et al.,breast cancer res 16(2014b):r75
[2893]
wu,z.b.et al.,j immunol.res 2014(2014c):131494
[2894]
wu,z.y.et al.,scand.j immunol.74(2011c):561
‑
567
[2895]
wurdak,h.et al.,cell stem cell 6(2010):37
‑
47
[2896]
xia,luo et al.,reprod.sci.17(2010):791
‑
797
[2897]
xia,q.s.et al.,zhonghua yi.xue.za zhi.91(2011):554
‑
559
[2898]
xiang,x.et al.,plos.one.7(2012):e50781
[2899]
xiang,y.j.et al.,plos.one.9(2014):e109449
[2900]
xiao,f.et al.,hum.genet.133(2014):559
‑
574
[2901]
xiao,j.et al.,j biol.chem.276(2001):6105
‑
6111
[2902]
xiao,w.et al.,nucleic acids res 26(1998):3908
‑
3914
[2903]
xiao,x.et al.,j transl.med.11(2013):151
[2904]
xin,b.et al.,oncogene 24(2005):724
‑
731
[2905]
xin,h.et al.,oncogene 22(2003):4831
‑
4840
[2906]
xin,z.et al.,virchows arch.465(2014):35
‑
47
[2907]
xing,q.t.et al.,onco.targets.ther.7(2014):881
‑
885
[2908]
xu,c.et al.,biomarkers 20(2015a):271
‑
274
[2909]
xu,c.z.et al.,int.j clin exp.pathol.6(2013a):2745
‑
2756
[2910]
xu,h.et al.,j clin oncol 30(2012):751
‑
757
[2911]
xu,j.et al.,oncol rep.34(2015b):1424
‑
1430
[2912]
xu,l.et al.,zhongguo fei.ai.za zhi.14(2011):727
‑
732
[2913]
xu,w.et al.,med.oncol 32(2015c):96
[2914]
xu,x.et al.,iubmb.life 65(2013b):873
‑
882
[2915]
xu,x.et al.,zhonghua bing.li xue.za zhi.43(2014a):177
‑
183
[2916]
xu,y.et al.,plos.one.9(2014b):e100127
[2917]
xu,y.et al.,plos.one.8(2013c):e64973
[2918]
xu,y.et al.,oncol lett.7(2014c):1474
‑
1478
[2919]
xu,y.f.et al.,bmc.cancer 15(2015d):332
[2920]
xu,z.et al.,leuk.res 33(2009):891
‑
897
[2921]
xu,z.et al.,anat.rec.(hoboken.)295(2012):1446
‑
1454
[2922]
xue,l.y.et al.,zhonghua zhong.liu za zhi.32(2010):838
‑
844
[2923]
yakimchuk,k.et al.,mol.cell endocrinol.375(2013):121
‑
129
[2924]
yamada,h.et al.,genes chromosomes.cancer 47(2008):810
‑
818
[2925]
yamada,h.y.et al.,oncogene 25(2006):1330
‑
1339
[2926]
yamada,r.et al.,tissue antigens 81(2013):428
‑
434
[2927]
yamada,y.et al.,jpn.j cancer res 90(1999):987
‑
992
[2928]
yamamoto,s.et al.,ann.surg.oncol 14(2007):2141
‑
2149
[2929]
yamashita,j.et al.,acta derm.venereol.92(2012):593
‑
597
[2930]
yamauchi,t.et al.,environ.health prev.med.19(2014):265
‑
270
[2931]
yamazaki,m.et al.,lab invest 94(2014):1260
‑
1272
[2932]
yamazoe,s.et al.,j exp.clin cancer res 29(2010):53
[2933]
yan,c.et al.,j ovarian.res 7(2014a):78
[2934]
yan,h.x.et al.,j biol chem 281(2006):15423
‑
15433
[2935]
yan,l.et al.,tumour.biol 34(2013a):4089
‑
4100
[2936]
yan,q.et al.,mol.cell biol 33(2013b):845
‑
857
[2937]
yan,x.et al.,int.j clin exp.pathol.7(2014b):8715
‑
8723
[2938]
yan,x.b.et al.,mol.med.rep.10(2014c):2720
‑
2728
[2939]
yan,y.et al.,plos.one.8(2013c):e81905
[2940]
yang,h.et al.,cancer res 68(2008a):2530
‑
2537
[2941]
yang,h.y.et al.,j proteomics.75(2012a):3639
‑
3653
[2942]
yang,j.et al.,neurosurg.clin n.am.23(2012b):451
‑
458
[2943]
yang,j.et al.,cell biochem.biophys.70(2014a):1943
‑
1949
[2944]
yang,j.j.et al.,blood 120(2012c):4197
‑
4204
[2945]
yang,j.l.et al.,int.j cancer 89(2000):431
‑
439
[2946]
yang,p.et al.,mol.cell biol 32(2012d):3121
‑
3131
[2947]
yang,p.et al.,curr.pharm.des 21(2015a):1292
‑
1300
[2948]
yang,p.et al.,zhonghua yi.xue.za zhi.93(2013):5
‑7[2949]
yang,t.et al.,tumour.biol 35(2014b):11199
‑
11207
[2950]
yang,t.et al.,j biol chem 278(2003):15291
‑
15296
[2951]
yang,w.et al.,mol.med.rep.10(2014c):1205
‑
1214
[2952]
yang,x.et al.,pathol.oncol res 20(2014d):641
‑
648
[2953]
yang,y.et al.,biochem.biophys.res commun.332(2005):181
‑
187
[2954]
yang,y.et al.,biochem.biophys.res commun.450(2014e):899
‑
905
[2955]
yang,y.et al.,cancer discov 4(2014f):480
‑
493
[2956]
yang,y.et al.,exp.oncol 30(2008b):81
‑
87
[2957]
yang,y.et al.,mol.cell 58(2015b):47
‑
59
[2958]
yang,y.l.et al.,leuk.res 34(2010):18
‑
23
[2959]
yang,y.m.et al.,cancer sci.102(2011):1264
‑
1271
[2960]
yang,z.et al.,int.j med.sci.12(2015c):256
‑
263
[2961]
yao,j.et al.,cancer immunol.res.2(2014a):371
‑
379
[2962]
yao,x.et al.,biochem.biophys.res commun.455(2014b):277
‑
284
[2963]
yao,y.s.et al.,clin transl.sci.8(2015):137
‑
142
[2964]
yasen,m.et al.,clin cancer res 11(2005):7354
‑
7361
[2965]
yasen,m.et al.,int.j oncol 40(2012):789
‑
797
[2966]
ye,c.et al.,j neurochem.133(2015):273
‑
283
[2967]
ye,z.et al.,int.j clin exp.med.8(2015):3707
‑
3715
[2968]
yeates,l.c.et al.,biochem.biophys.res commun.238(1997):66
‑
70
[2969]
yeh,i.et al.,am.j surg.pathol.39(2015):581
‑
591
[2970]
yeh,s.et al.,proc.natl.acad.sci.u.s.a 97(2000):11256
‑
11261
[2971]
yen,l.c.et al.,clin cancer res 15(2009):4508
‑
4513
[2972]
yi,c.h.et al.,cancer lett.284(2009):149
‑
156
[2973]
yildiz,m.et al.,blood 125(2015):668
‑
679
[2974]
yin,b.w.et al.,cancer immun.8(2008):3
[2975]
yin,j.et al.,med.oncol 31(2014):272
[2976]
yiu,g.k.et al.,j biol chem 281(2006):12210
‑
12217
[2977]
yokota,t.et al.,acta neuropathol.111(2006):29
‑
38
[2978]
yonezawa,s.et al.,pathol.int.49(1999):45
‑
54
[2979]
yongjun zhang,m.m.et al.,j cancer res ther.9(2013):660
‑
663
[2980]
yoo,k.h.et al.,oncol lett.8(2014):2135
‑
2139
[2981]
yoon,d.h.et al.,eur.j haematol.88(2012):292
‑
305
[2982]
yoon,s.y.et al.,biochem.biophys.res commun.326(2005):7
‑
17
[2983]
yoshida,a.et al.,am.j surg.pathol.38(2014):552
‑
559
[2984]
yoshida,k.et al.,cancer sci.104(2013):171
‑
177
[2985]
yoshida,y.et al.,genes dev.17(2003):1201
‑
1206
[2986]
yoshizawa,a.et al.,clin cancer res 16(2010):240
‑
248
[2987]
young,a.n.et al.,am.j pathol.158(2001):1639
‑
1651
[2988]
yu,c.j.et al.,int.j cancer 69(1996):457
‑
465
[2989]
yu,j.et al.,cancer 88(2000):1801
‑
1806
[2990]
yu,l.et al.,cancer res 75(2015a):1275
‑
1286
[2991]
yu,m.et al.,oncogene 24(2005):1982
‑
1993
[2992]
yu,w.et al.,carcinogenesis 29(2008):1717
‑
1724
[2993]
yu,x.et al.,tumour.biol 36(2015b):967
‑
972
[2994]
yu,x.f.et al.,world j gastroenterol.17(2011):4711
‑
4717
[2995]
yu,z.et al.,mol.med.rep.10(2014):1583
‑
1589
[2996]
yuan,b.et al.,cancer sci.106(2015):819
‑
824
[2997]
yuan,j.y.et al.,oncol lett.1(2010):649
‑
655
[2998]
yuan,y.et al.,am.j surg.pathol.33(2009):1673
‑
1682
[2999]
zage,p.e.et al.,cancer 119(2013):915
‑
923
[3000]
zagryazhskaya,a.et al.,oncotarget.6(2015):12156
‑
12173
[3001]
zamkova,m.et al.,cell cycle 12(2013):826
‑
836
[3002]
zang,h.et al.,zhonghua shi yan.he.lin.chuang.bing.du xue.za zhi.26(2012):285
‑
287
[3003]
zapatero,a.et al.,urol.oncol 32(2014):1327
‑
1332
[3004]
zaravinos,a.et al.,tumour.biol 35(2014):4987
‑
5005
[3005]
zaremba,s.et al.,cancer res.57(1997):4570
‑
4577
[3006]
zarubin,t.et al.,cell res 15(2005):439
‑
446
[3007]
zekri,a.et al.,oncol res 20(2012):241
‑
250
[3008]
zekri,a.r.et al.,asian pac.j cancer prev.16(2015):3543
‑
3549
[3009]
zeng,x.et al.,ai.zheng.26(2007):1080
‑
1084
[3010]
zhai,w.et al.,eur.rev med.pharmacol.sci.18(2014):1354
‑
1360
[3011]
zhan,x.et al.,anal.biochem.354(2006):279
‑
289
[3012]
zhang,b.et al.,j huazhong.univ sci.technolog.med.sci.30(2010a):322
‑
325
[3013]
zhang,c.et al.,j surg.res 197(2015a):301
‑
306
[3014]
zhang,c.et al.,bmc.gastroenterol.15(2015b):49
[3015]
zhang,c.y.et al.,asian j androl 17(2015c):106
‑
110
[3016]
zhang,f.et al.,j viral hepat.21(2014a):241
‑
250
[3017]
zhang,f.et al.,cancer res 63(2003):5005
‑
5010
[3018]
zhang,g.et al.,oncol rep.33(2015d):1147
‑
1154
[3019]
zhang,h.et al.,tumour.biol 36(2015e):997
‑
1002
[3020]
zhang,h.et al.,nat genet.42(2010b):755
‑
758
[3021]
zhang,h.h.et al.,int.j clin exp.pathol.6(2013a):1734
‑
1746
[3022]
zhang,j.et al.,hum.pathol.46(2015f):1331
‑
1340
[3023]
zhang,j.et al.,tumour.biol 36(2015g):2163
‑
2168
[3024]
zhang,j.et al.,plos.one.9(2014b):e109318
[3025]
zhang,k.et al.,tumour.biol 35(2014c):4031
‑
4040
[3026]
zhang,l.et al.,med.oncol 32(2015h):454
[3027]
zhang,l.et al.,mol.cancer ther.6(2007):1661
‑
1672
[3028]
zhang,l.et al.,j cell mol.med.19(2015i):799
‑
805
[3029]
zhang,l.et al.,cancer res 65(2005a):925
‑
932
[3030]
zhang,m.et al.,j exp.clin cancer res 34(2015j):60
[3031]
zhang,m.et al.,cancer lett.243(2006):38
‑
46
[3032]
zhang,n.et al.,oncotarget.(2016a)
[3033]
zhang,p.et al.,genome 57(2014d):253
‑
257
[3034]
zhang,s.q.et al.,mol.med.rep.12(2015k):1177
‑
1182
[3035]
zhang,w.et al.,epigenetics.10(2015l):736
‑
748
[3036]
zhang,w.et al.,acta haematol.130(2013b):297
‑
304
[3037]
zhang,w.et al.,tumour.biol(2015m)
[3038]
zhang,w.et al.,j biol chem 286(2011):35899
‑
35905
[3039]
zhang,w.et al.,biochem.j(2016b)
[3040]
zhang,x.et al.,oncotarget.5(2014e):6178
‑
6190
[3041]
zhang,x.et al.,plos.one.8(2013c):e72458
[3042]
zhang,x.et al.,leuk.res 39(2015n):1448
‑
1454
[3043]
zhang,y.et al.,clin lung cancer 14(2013d):45
‑
49
[3044]
zhang,y.et al.,plos.one.9(2014f):e90154
[3045]
zhang,y.j.et al.,cancer lett.275(2009):277
‑
284
[3046]
zhang,y.x.et al.,biomed.pharmacother.67(2013e):97
‑
102
[3047]
zhang,z.et al.,gynecol.oncol 135(2014g):69
‑
73
[3048]
zhang,z.et al.,cancer epidemiol.biomarkers prev.14(2005b):1188
‑
1193
[3049]
zhang,z.et al.,j biol chem 290(2015o):19558
‑
19568
[3050]
zhao,c.et al.,neoplasia.9(2007):1
‑7[3051]
zhao,c.et al.,endocrine.36(2009):224
‑
232
[3052]
zhao,h.et al.,cancer gene ther.21(2014a):448
‑
455
[3053]
zhao,h.et al.,cell tissue res(2015a)
[3054]
zhao,h.et al.,zhonghua gan zang.bing.za zhi.10(2002):100
‑
102
[3055]
zhao,q.et al.,exp.ther.med.5(2013a):942
‑
946
[3056]
zhao,x.et al.,cancer res 65(2005):2125
‑
2129
[3057]
zhao,x.et al.,onco.targets.ther.7(2014b):343
‑
351
[3058]
zhao,x.et al.,lab invest 93(2013b):8
‑
19
[3059]
zhao,y.et al.,onco.targets.ther.8(2015b):421
‑
425
[3060]
zhao,y.et al.,hum.pathol.44(2013c):365
‑
373
[3061]
zhao,z.et al.,eur.j surg.oncol 40(2014c):1361
‑
1368
[3062]
zhao,z.et al.,rna.biol 12(2015c):538
‑
554
[3063]
zhao,z.k.et al.,tumour.biol.34(2013d):173
‑
180
[3064]
zheng,c.x.et al.,int.j oncol 43(2013):755
‑
764
[3065]
zheng,m.et al.,ai.zheng.23(2004):771
‑
776
[3066]
zhou,b.et al.,cancer biol.ther 13(2012a):871
‑
879
[3067]
zhou,d.et al.,cancer cell 16(2009):425
‑
438
[3068]
zhou,d.et al.,plos.one.8(2013a):e53310
[3069]
zhou,j.et al.,lung cancer 14(1996):85
‑
97
[3070]
zhou,j.et al.,j biol chem 285(2010):40342
‑
40350
[3071]
zhou,j.et al.,j surg.res 188(2014a):129
‑
136
[3072]
zhou,j.b.et al.,mol.med.rep.7(2013b):591
‑
597
[3073]
zhou,j.r.et al.,zhonghua er.bi yan.hou tou.jing.wai ke.za zhi.42(2007):934
‑
938
[3074]
zhou,l.et al.,clin transl.oncol 16(2014b):906
‑
913
[3075]
zhou,t.b.et al.,j recept.signal.transduct.res 33(2013):28
‑
36
[3076]
zhou,x.et al.,arch.med.res 42(2011):589
‑
595
[3077]
zhou,x.et al.,oncotarget.6(2015a):41077
‑
41091
[3078]
zhou,y.et al.,am.j clin pathol.138(2012b):744
‑
750
[3079]
zhou,z.et al.,exp.cell res 331(2015b):399
‑
407
[3080]
zhu,f.et al.,zhongguo shi yan.xue.ye.xue.za zhi.21(2013a):396
‑
398
[3081]
zhu,j.et al.,asian pac.j cancer prev.14(2013b):3011
‑
3015
[3082]
zhu,j.et al.,oncogene(2015)
[3083]
zhu,m.et al.,nucleic acids res 42(2014a):13074
‑
13081
[3084]
zhu,q.et al.,mol.cell biol 27(2007):324
‑
339
[3085]
zhu,s.et al.,febs lett.588(2014b):981
‑
989
[3086]
zhu,x.et al.,gynecol.oncol 112(2009):248
‑
256
[3087]
zhu,z.et al.,carcinogenesis 35(2014c):1901
‑
1910
[3088]
zi,y.et al.,int.j clin exp.pathol.8(2015):1312
‑
1320
[3089]
ziebarth,j.d.et al.,plos.one.7(2012):e47137
[3090]
zighelboim,i.et al.,clin cancer res 13(2007):2882
‑
2889
[3091]
zighelboim,i.et al.,j clin oncol 27(2009):3091
‑
3096
[3092]
zins,k.et al.,int.j mol.sci.16(2015):29643
‑
29653
[3093]
zohrabian,v.m.et al.,oncol rep.18(2007):321
‑
328
[3094]
zou,c.et al.,cancer 118(2012):1845
‑
1855
[3095]
zou,j.x.et al.,mol.cancer res 12(2014a):539
‑
549
[3096]
zou,s.et al.,nat commun.5(2014b):5696
[3097]
zou,t.t.et al.,oncogene 21(2002):4855
‑
4862
[3098]
zou,w.et al.,cancer sci.101(2010):2156
‑
2162
[3099]
zou,y.et al.,biomed.rep.3(2015):33
‑
37
[3100]
zubor,p.et al.,mol.biol.rep.42(2015):977
‑
988
[3101]
zuo,g.w.et al.,histol.histopathol.25(2010):795
‑
806
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1