基于分子对接的PAP的高特异性的偶联酶YND突变体、方法和应用
基于分子对接的pap的高特异性的偶联酶ynd突变体、方法和应用
技术领域
1.本发明属于生物技术领域,尤其是一种基于分子对接的pap(3’,5
’‑
二磷酸腺苷)的高特异性的偶联酶ynd突变体、方法和应用。
背景技术:
2.磺酰基转移酶(sulfotransferase,st)是一类负责催化磺酰基(so3)基团从通用供体分子3
′‑
磷酸腺苷-5
′‑
磷酸硫酸盐(paps)转移到碳水化合物、蛋白质和其他多种低分子量代谢产物中的酶。磺酰基转移酶存在于大多数生物体和所有人体组织中,介导不同种类受体的硫酸化以实现多种生物功能。目前发现在生物系统中,磺酰基转移酶发挥着各种重要作用,包括解毒、信号传导和调节受体结合等。在生物硫酸化过程中,无机硫酸盐首先被激活为高能辅助因子,然后将硫酸盐基团转移到最终的受体上。
3.对于st的活性测定方法有同位素标记法、分光光度法、荧光分析法以及质谱分析法,以上这些方法由于对操作人员的毒害作用、并非所有底物或产物都有荧光吸收和荧光特性、以及仪器昂贵等原因,不作为st的活性测定的首选方法。目前最方便的分析方法是偶联酶反应。即在磺酰基转移酶类研究过程中一般以3'-磷酸腺苷-5'-磷酰硫酸(3'-phosphoadenosine-5'-phosphosulfate,paps)作为磺酰基供体,在磺酰基转移酶催化作用下paps失去磺基后成为3’,5
’‑
二磷酸腺苷(3'-phosphate 5'-phosphate adenosine,pap),可以通过定量产生的pap来判断磺基转移酶的活性以及酶活力,而pap经过酶催化会分解成5'-腺嘌呤核苷酸(adeno sine 5'-monophosphate,amp)和磷酸根离子,通过产生的磷酸根离子可以确定pap的产量,进而确定磺酰基转移酶的活性,整个流程如下所示:
[0004][0005]
目前市面上测定磺酰基转移酶活性的酶活试剂盒存在价格贵,反应体系缓冲液单一等缺点,成为磺酰基转移酶及对应底物的大量筛选的阻力,另外试剂盒的试剂所用缓冲体系为tr is-hcl,在某些反应中tris可能成为磺酰基受体的竞争性底物,为磺酰基转移酶底物特异性筛选带来困难。
[0006]
目前,现有技术中存在如下缺陷:
[0007]
1、对于st的活性测定方法有同位素标记法、分光光度法、荧光分析法以及质谱分析法,以上这些方法由于对操作人员的毒害作用、并非所有底物或产物都有荧光吸收和荧光特性、以及仪器昂贵等原因,不作为st的活性测定的首选方法。目前最方便的分析方法是偶联酶反应。
[0008]
2、目前市面上测定磺酰基转移酶活性的酶活试剂盒存在价格贵。
[0009]
3、反应体系缓冲液单一等缺点,成为磺酰基转移酶及对应底物的大量筛选的阻力,另外试剂盒的试剂所用缓冲体系为tris-hcl,在某些反应中tris可能成为磺酰基受体的竞争性底物,为磺酰基转移酶底物特异性筛选带来困难。
[0010]
4、现有酶的优化方法具有一定的盲目性,即根据大致结合位点进行突变,造成筛选实验任务量大且不一定准确。本发明应用分子对接技术,精确到酶与底物的结合位点的氨基酸,对结合的位点进行精确突变,使实验任务量大大减小,目的性和方向性更明确。
[0011]
通过检索,尚未发现与本发明专利申请相关的专利公开文献。
技术实现要素:
[0012]
本发明目的在于克服现有技术中的不足之处,提供一种基于分子对接的pap(3’,
bisphosphate nucleotida se,ynd)(www.pdb.org;pdb:1ka1)为出发点设计并改造了一种pap特异性强的偶联酶。本发明改造的酶由大肠杆菌表达系统诱导表达,储存于储存液中,-80℃条件下,能维持活性3个月,用相应的缓冲体系稀释使用,方便且可更换反应体系,解决了体系缓冲物质对st活性检测局限性的问题。
[0031]
2、本发明方法对已知晶体结构的磷酸腺苷酶ynd首先通过分子对接设计,推算出可能的正向突变位点,以此为依据进行酶分子的改造,大大降低了筛选工作量,极大降低了st活性检测成本。
[0032]
3、本发明方法以分子对接为基础进行突变设计,并佐以实验验证来提高ynd对pap的底物特异性,纯化后的蛋白可以根据实验要求更改反应体系。本发明以hepes为缓冲体系,对其活性测定,条件优化,以优化后反应条件检验突变酶在磺酰基转移酶活性测定体系中的应用效果。
附图说明
[0033]
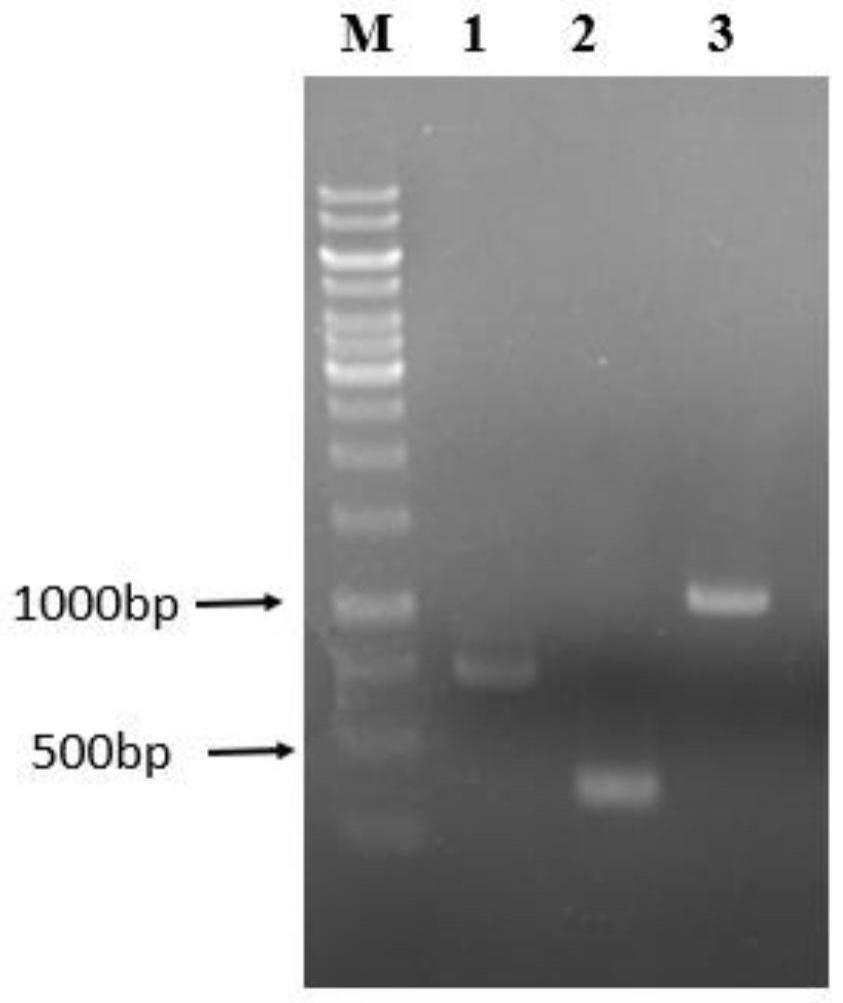
图1为本发明中重叠pcr验证图;其中,1:pcr左臂;714bp,2:pcr右臂;410bp,3:重叠pcr产物;1079bp,m:marker;
[0034]
图2为本发明中酶切验证图;其中,1:pet28a(+)线性化质粒;2:ynd酶切片段;3:236d酶切片段;4:236y酶切片段;5:236w酶切片段;m:marker;
[0035]
图3为本发明中sds-page蛋白胶图;其中,1:ynd纯化蛋白;2:236d纯化蛋白;3:236y纯化蛋白;4:236w纯化蛋白;m:蛋白marker;
[0036]
图4为本发明中236d突变蛋白在磺酰基转移酶中的应用图(采用孔雀石绿-钼酸铵染色法进行显色);其中,实验组:10ulpaps+25ul st+5ul 236d+10ul 1-萘酚,对照组1:10ul paps+25ul st+5ul反应液+10ul 1-萘酚,对照组2:10ulpaps+25ul反应液+5ul 236d+10ul 1-萘酚,对照组3:10ulpaps+30ul反应液+10ul 1-萘酚。
具体实施方式
[0037]
下面详细叙述本发明的实施例,需要说明的是,本实施例是叙述性的,不是限定性的,不能以此限定本发明的保护范围。
[0038]
本发明中所使用的原料,如无特殊说明,均为常规的市售产品;本发明中所使用的方法,如无特殊说明,均为本领域的常规方法。
[0039]
一种基于分子对接的3’,5
’‑
二磷酸腺苷的高特异性的偶联酶ynd突变体,所述ynd突变体的基因序列为seq id no.2、seq id no.3或seq id no.4,所述ynd突变体的氨基酸序列为seq id no.6、seq id no.7或seq id no.8。
[0040]
较优地,分子对接软件vina1被用于设计对pap底物具有较高特异性的ynd,ynd的三维结构使用pdb id为1k9y2的蛋白质结构;ynd的突变体使用discovery studio visualizer构建;底物pap和paps均使用chemdraw绘制;在对接研究中,格点间距被设为同时其格点中心(x,y,z)为(9.623,16.925,-23.414),格点参数设为25对接所得构象数为20。
[0041]
较优地,所述ynd突变体由大肠杆菌表达系统诱导表达。
[0042]
较优地,所述ynd突变体储存于储存液中,-80℃条件下,能维持活性3个月,用相应
的缓冲体系稀释使用。
[0043]
如上所述的基于分子对接的3’,5
’‑
二磷酸腺苷的高特异性的偶联酶ynd突变体在磺酰基转移酶测定方面中的应用。
[0044]
如上所述的基于分子对接的3’,5
’‑
二磷酸腺苷的高特异性的偶联酶ynd突变体的制备方法,步骤如下:
[0045]
首先对ncbi获得的ynd基因进行设计,在基因两端插入xhoi、ncoi酶切位点,检查并利用密码子的兼并性去掉位于基因上存在的xhoi、ncoi酶切位点,修改后的氨基酸序列为seq id no.5,基因序列为seq id no.1;
[0046]
通过人工合成基因的方法合成修饰后的ynd基因,将获得的ynd基因与经过xhoi、ncoi限制性核酸内切酶酶切后的pet28a(+)线性载体,利用solution i连接酶进行连接,构建表达载体用于表达ynd;
[0047]
利用重叠pcr的方法获得突变dna,根据ynd的序列与分子对接获得的需要突变的位点,设计定点突变pcr引物,以突变位点为中心,选取含突变后碱基的同源序列15-25bp,并向上游与下游选取17-30bp碱基作为引物,设计引物序列为seq id no.7至seq id no.16;
[0048]
将构建后的突变质粒化转e coli jm109感受态细胞进行扩增并对其进行测序验证;
[0049]
将测序成功的突变质粒化转e coli bl21,挑取化转后的单菌落于5mllb培养基试管中过夜培养,将获得的菌液接种到1l三角瓶液体培养基中进行液体培养,37℃培养至od
600
到0.6,加入iptg置于16℃摇床中进行诱导表达;20h后收集菌体;
[0050]
取裂解液悬浮菌体,冰浴超声破碎菌体,12000rpm,30min,收集上清;
[0051]
通过镍柱亲和层析的方法进行目的蛋白的纯化,用储存液对镍柱亲和层析获得的蛋白进行超滤,置换其缓冲体系,即得ynd突变体,-80℃保存;
[0052]
其中,所述储存液为50mm hepes缓冲液,ph7.5。
[0053]
较优地,所述ynd为酵母3’,5
’‑
二磷酸腺苷酶,www.pdb.org;pdb:1ka1。
[0054]
具体地,相关制备如下:
[0055]
分子对接软件vina1被用于设计对pap底物具有较高特异性的ynd。ynd的三维结构使用pdb id为1k9y2的蛋白质结构。ynd的突变体使用discovery studio visualizer构建。此外,底物pap和paps均使用chemdraw绘制。在对接研究中,格点间距被设为同时其格点中心(x,y,z)为(9.623,16.925,-23.414)。此外,格点参数设为格点参数设为对接所得构象数为20。
[0056]
通过观察底物pap与ynd结合的三维结构来确定饱和突变的氨基酸。在这里,对残基d263、e238、g236、h241、k267、q265、s264、v237以及y268进行全饱和突变。随后,将底物pap和paps分别与ynd的野生型和突变型的目标结合区域进行对接研究。根据分子对接的最终得分进行筛选。最终,选择筛选出对底物pap具有较高特异性的ynd突变体g236y、g236d、g236w(后续以突变后的位点加突变后的氨基酸命名突变蛋白,如236y、236d、236w。)。
[0057]
更为具体地:
[0058]
首先对ncbi获得的ynd(酵母3’,5
’‑
二磷酸腺苷酶(yeast 3',5'-bisphosphate nucleotidase,ynd)(www.pdb.org;pdb:1ka1))基因进行设计,在基因两端插入xhoi、ncoi
酶切位点,检查并利用密码子的兼并性去掉位于基因上存在的xhoi、ncoi酶切位点,修改后的碱基序列如碱基序列1所示。
[0059]
通过人工合成基因的方法合成修饰后的ynd基因,将获得的ynd基因与经过xhoi、ncoi限制性核酸内切酶酶切后的pet28a(+)线性载体,利用solution i连接酶进行连接,构建表达载体用于表达ynd。酶切线性质粒以及目的基因如酶切验证胶图2所示。
[0060]
利用重叠pcr的方法获得突变dna,根据ynd的序列与分子对接获得的需要突变的位点,设计定点突变pcr引物,以突变位点为中心,选取含突变后碱基的同源序列大约15-25bp,并向上游与下游选取17-30bp碱基作为引物,设计引物序列表1所示:
[0061]
表1单点突变对应引物序列
[0062][0063]
将构建后的突变质粒化转e coli jm109感受态细胞进行扩增并对其进行测序验证。
[0064]
将测序成功的突变质粒化转e coli bl21,挑取化转后的单菌落与5ml lb试管中过夜培养,将获得的菌液接种到1l三角瓶中进行液体培养,37℃培养至od
600
到0.6左右,加入iptg置于16℃摇床中进行诱导表达。20h后收集菌体。
[0065]
取裂解液30ml,悬浮菌体,冰浴超声破碎菌体。12000rpm,30min,收集上清。
[0066]
通过镍柱亲和层析的方法进行目的蛋白的纯化,用储存液(50mm hepes缓冲液ph7.5)对镍柱亲和层析获得的蛋白进行超滤,置换其缓冲体系,-80℃保存备用。超滤浓缩后的目的蛋白及突变蛋白如图3sds-page蛋白电泳胶所示。
[0067]
相关检测如下:
[0068]
1、本发明通过分子对接及分子模拟的方法,以结合能为判断依据,结果显示对236位点的氨基酸进行突变可以改变ynd的特异性,以重叠pcr的技术手段对ynd基因进行单点突变获得突变基因。结果如图1所示。
[0069]
2、本发明对获得效果较好的突变蛋白进行反应条件优化,优化从反应温度、反应体系ph值、以及添加剂如甘油添加量、酶活保护剂bsa的添加与否。结果如表2至表6所示。
[0070]
表2不同突变蛋白对paps,pap两种底物酶活比值的比较
[0071][0072]
从结果来看,原始蛋白ynd对两种底物的酶活比值接近1.即同一条件下对两种底物的特异性差别不大,对底物pap活性更大一点,即原始蛋白对pap特异性稍微强一点。突变后的蛋白对pap的特异性从大到小的排列顺序为236d,236w,236y,所有突变均为正向突变,
因为最终目的是筛选pap特异性强的突变蛋白,后续以最优突变236d为基础进行条件优化。
[0073]
表3反应温度对突变蛋白236d底物特异性的影响
[0074][0075]
表4反应ph对突变蛋白236d底物特异性的影响
[0076][0077]
表5甘油添加量对突变蛋白236d底物特异性的影响
[0078][0079]
表6 bsa的添加对突变蛋白236d底物特异性的影响
[0080][0081]
本发明将对磺酰基转移酶活性检测以及特定底物的筛选提供便利。
[0082]
以236d为例,对点突变以及活性测定进行描述,
[0083]
首先以人工合成的基因ynd为模板,用引物f-ynd
,236rd;236fd,r-ynd
分别pcr获得突变后的左臂和右臂,以上述获得的左臂和右臂为模板,f-ynd
,r-ynd
为引物进行重叠pcr获得完整的突变基因。用限制性核酸内切酶xhoi、ncoi对此基因进行酶切,去除保护碱基以及切出粘性末端,同时用相同的限制性核酸内切酶对pet28a(+)质粒进行线性化。线性化的质粒与突变基因通过连接酶solution i进行16℃过夜连接。将构建后的突变质粒化转e coli jm109感受态细胞进行扩增并对其进行测序验证。
[0084]
将测序成功的突变质粒化转e coli bl21,挑取化转后的单菌落与5ml lb试管中过夜培养,将获得的菌液接种到1l三角瓶中进行液体培养,37℃培养至od
600
到0.6左右,加入iptg置于16℃摇床中进行诱导表达。20h后收集菌体。
[0085]
取裂解液30ml,悬浮菌体,冰浴超声破碎菌体。12000rpm,30min,收集上清。
[0086]
通过镍柱亲和层析的方法进行目的蛋白的纯化,将纯化后的蛋白经过超滤置换为储存液进行储存备用。
[0087]
对最终获得的蛋白进行稀释,以0.01ug/ul作为使用浓度进行后续反应。
[0088]
向96孔板中依次加入1mmpaps/1mmpap 10ul,0.01ug/ul 236d蛋白5ul,反应液((50mm hepes缓冲液50mm kcl 2mm mgcl
2 ph7.5))35ul,轻拍混匀后进行反应。所用底物
及蛋白的稀释均用过程中的反应液进行。初筛反应条件为37℃水浴,ph7.5,反应20分钟。反应结束后,每孔加入30ul孔雀石绿显色剂a,100ul过膜水,30ul孔雀石绿显色剂b,混匀后室温静置20分钟进行显色。在620nm下进行比色。比色后不同底物的吸光度值作为筛选的依据,这里以paps/pap的比值作为特异性判断的依据。比值越接近1说明对两种底物的活性越接近,即底物特异性越小。比值越接近0说明对底物paps活性越低,而对底物pap活性越高,即特异性越强。
[0089]
初筛结果说明。所有突变皆为正向突变即酶对底物pap的特异性得到提高。本发明对突变效果最好的236d蛋白进行进一步的反应条件的优化。
[0090]
以ph7.550mm hepes缓冲液50mm kcl 2mm mgcl2作为反应液,反应时间20分钟,进行反应温度的优化,从表3可以看出,结果证明温度降低,酶对底物的特异性提高,综合后续此酶在磺酰基转移酶酶活测定中的应用,取20℃作为最佳反应温度。
[0091]
以20℃,不同ph的50mm hepes缓冲液50mm kcl 2mm mgcl2反应液,反应时间20分钟进行反应体系ph的优化。结果证明ph7.5时酶对底物的特异性最高,因此以ph7.550mm hepes缓冲液50mm kcl 2mm mgcl2作为反应液进行后续优化。如表4所示。
[0092]
在反应液中加入不同体积的甘油,以最终甘油含量(体积)为1/1000,1/100,2/100,5/100的浓度作为反应液,以20℃、ph7.5反应20分钟作为反应条件进行条件优化,实验结果证明不含甘油时酶对底物的特异性最高。如表5所示。
[0093]
向ph7.550mm hepes 50mm kcl 2mm mgcl2中加入牛血清白蛋白(bsa)使其终浓度为1mg/ml,作为反应液,20℃水浴,反应时间20分钟,表6结果显示bsa的加入提高了酶对底物的特异性。
[0094]
是以最终以ph7.550mm hepes 50mm kcl 2mm mgcl
2 bsa 1mg/ml作为反应体系,20℃水浴作为反应条件,反应20分钟作为最优的反应条件。后续以此作为基础进行突变酶236d在磺酰基转移酶活性测定及底物验证中的应用效果。
[0095]
并对获得的突变酶以实验室已有且对应底物明确的磺酰基转移酶作为检验,验证突变酶236d对磺酰基转移酶酶活检测的应用是可行的。
[0096]
检验体系如下:
[0097]
实验组以10ul 1mmpaps+25ul 100ng/ul st+5ul 0.01ug/ul 236d+10ul 2mm 1-萘酚其他对照组的缺失成分以上述最优反应液进行代替,同时反应中的成分稀释均以此反应液作为稀释溶液。反应条件为ph7.550mm hepes 50mm kcl 2mm mgcl
2 bsa 1mg/ml作为反应体系,20℃水浴,反应20分钟。
[0098]
由实验结果图4可看出实验组与对照组2,可见明显颜色差异,即此突变的236d 3’,5
’‑
二磷酸腺苷酶的底物特异性足以用以磺酰基转移酶的活性检测及底物鉴定。对照组1,3用以确定236d的活性,以及paps的纯度。
[0099]
本发明涉及基因序列:
[0100]
ynd(cp036482)
[0101]
ccatggcattggaaagagaattattggttgcaactcaagctgtacgaaaggcgtctttattgactaagagaattcaatctgaagtgatttctcacaaggactccactactattaccaagaatgataattctccagtaaccacaggtgattatgctgcacaaacgatcatcataaatgctatcaagagcaattttcctgatgataaggtagttggtgaagaatcctcatcaggattgagcgacgcattcgtctcaggaattttaaacgaaataaaagccaatgacgaagtttataacaaga
attataaaaaggatgattttctgtttacaaacgatcagtttccgctaaaatctttggaggacgtcaggcaaatcatcgatttcggcaattacgaaggtggtagaaaaggaagattttggtgtttggatcctattgacggaaccaaggggtttttaagaggtgaacagtttgcagtatgtctggccttaattgtggacggtgttgttcagcttggttgtattggatgccccaacttagttttaagttcttatggggcccaagatttgaaaggccatgagtcatttggttatatctttcgtgctgttagaggtttaggtgccttctattctccatcttcagatgcagagtcatggaccaaaatccacgttagacacttaaaagacactaaagacatgattactttagagggagttgaaaagggacactcctctcatgatgaacaaactgctatcaaaaacaaactaaatatatccaaatctttgcacttggattctcaagccaagtactgtttgttagcattgggcttagcagacgtatatttacgtctgcctatcaaactttcttaccaagaaaagatctgggaccatgctgcaggcaacgttattgtccatgaagctggaggtatccatacagatgcaatggaagatgttcctctagacttcggtaacggtagaacgctagctacgaagggagttatagcgtcaagtggcccacgcgagttacatgacttggtggtgtctacatcatgcgatgtcattcagtcaagaaacgccctcgag
[0102]
236d
[0103]
ccatggcattggaaagagaattattggttgcaactcaagctgtacgaaaggcgtctttattgactaagagaattcaatctgaagtgatttctcacaaggactccactactattaccaagaatgataattctccagtaaccacaggtgattatgctgcacaaacgatcatcataaatgctatcaagagcaattttcctgatgataaggtagttggtgaagaatcctcatcaggattgagcgacgcattcgtctcaggaattttaaacgaaataaaagccaatgacgaagtttataacaagaattataaaaaggatgattttctgtttacaaacgatcagtttccgctaaaatctttggaggacgtcaggcaaatcatcgatttcggcaattacgaaggtggtagaaaaggaagattttggtgtttggatcctattgacggaaccaaggggtttttaagaggtgaacagtttgcagtatgtctggccttaattgtggacggtgttgttcagcttggttgtattggatgccccaacttagttttaagttcttatggggcccaagatttgaaaggccatgagtcatttggttatatctttcgtgctgttagaggtttaggtgccttctattctccatcttcagatgcagagtcatggaccaaaatccacgttagacacttaaaagacactaaagacatgattactttagaggacgttgaaaagggacactcctctcatgatgaacaaactgctatcaaaaacaaactaaatatatccaaatctttgcacttggattctcaagccaagtactgtttgttagcattgggcttagcagacgtatatttacgtctgcctatcaaactttcttaccaagaaaagatctgggaccatgctgcaggcaacgttattgtccatgaagctggaggtatccatacagatgcaatggaagatgttcctctagacttcggtaacggtagaacgctagctacgaagggagttatagcgtcaagtggcccacgcgagttacatgacttggtggtgtctacatcatgcgatgtcattcagtcaagaaacgccctcgag
[0104]
236w
[0105]
ccatggcattggaaagagaattattggttgcaactcaagctgtacgaaaggcgtctttattgactaagagaattcaatctgaagtgatttctcacaaggactccactactattaccaagaatgataattctccagtaaccacaggtgattatgctgcacaaacgatcatcataaatgctatcaagagcaattttcctgatgataaggtagttggtgaagaatcctcatcaggattgagcgacgcattcgtctcaggaattttaaacgaaataaaagccaatgacgaagtttataacaagaattataaaaaggatgattttctgtttacaaacgatcagtttccgctaaaatctttggaggacgtcaggcaaatcatcgatttcggcaattacgaaggtggtagaaaaggaagattttggtgtttggatcctattgacggaaccaaggggtttttaagaggtgaacagtttgcagtatgtctggccttaattgtggacggtgttgttcagcttggttgtattggatgccccaacttagttttaagttcttatggggcccaagatttgaaaggccatgagtcatttggttatatctttcgtgctgttagaggtttaggtgccttctattctccatcttcagatgcagagtcatggaccaaaatccacgttagacacttaaaagacactaaagacatgattactttagagccagttgaaaagggacactcctctcatgatgaacaaactgctatcaaaaacaaactaaatatatccaaatctttgcacttggattctcaagccaagtactgtttgttagcattgggcttagcagacgtatat
ttacgtctgcctatcaaactttcttaccaagaaaagatctgggaccatgctgcaggcaacgttattgtccatgaagctggaggtatccatacagatgcaatggaagatgttcctctagacttcggtaacggtagaacgctagctacgaagggagttatagcgtcaagtggcccacgcgagttacatgacttggtggtgtctacatcatgcgatgtcattcagtcaagaaacgccctcgag
[0106]
236y
[0107]
ccatggcattggaaagagaattattggttgcaactcaagctgtacgaaaggcgtctttattgactaagagaattcaatctgaagtgatttctcacaaggactccactactattaccaagaatgataattctccagtaaccacaggtgattatgctgcacaaacgatcatcataaatgctatcaagagcaattttcctgatgataaggtagttggtgaagaatcctcatcaggattgagcgacgcattcgtctcaggaattttaaacgaaataaaagccaatgacgaagtttataacaagaattataaaaaggatgattttctgtttacaaacgatcagtttccgctaaaatctttggaggacgtcaggcaaatcatcgatttcggcaattacgaaggtggtagaaaaggaagattttggtgtttggatcctattgacggaaccaaggggtttttaagaggtgaacagtttgcagtatgtctggccttaattgtggacggtgttgttcagcttggttgtattggatgccccaacttagttttaagttcttatggggcccaagatttgaaaggccatgagtcatttggttatatctttcgtgctgttagaggtttaggtgccttctattctccatcttcagatgcagagtcatggaccaaaatccacgttagacacttaaaagacactaaagacatgattactttagagtacgttgaaaagggacactcctctcatgatgaacaaactgctatcaaaaacaaactaaatatatccaaatctttgcacttggattctcaagccaagtactgtttgttagcattgggcttagcagacgtatatttacgtctgcctatcaaactttcttaccaagaaaagatctgggaccatgctgcaggcaacgttattgtccatgaagctggaggtatccatacagatgcaatggaagatgttcctctagacttcggtaacggtagaacgctagctacgaagggagttatagcgtcaagtggcccacgcgagttacatgacttggtggtgtctacatcatgcgatgtcattcagtcaagaaacgccctcgag
[0108]
本发明涉及氨基酸序列
[0109]
ynd(np_014577.1)
[0110]
malerellvatqavrkaslltkriqsevishkdsttitkndnspvttgdyaaqtiiinaiksnfpddkvvgeesssglsdafvsgilneikandevynknykkddflftndqfplksledvrqiidfgnyeggrkgrfwcldpidgtkgflrgeqfavclalivdgvvqlgcigcpnlvlssygaqdlkghesfgyifravrglgafyspssdaeswtkihvrhlkdtkdmitlegvekghsshdeqtaiknklniskslhldsqakycllalgladvylrlpiklsyqekiwdhaagnvivheaggihtdamedvpldfgngrtlatkgviassgprelhdlvvstscdviqsrnale
[0111]
236d
[0112]
malerellvatqavrkaslltkriqsevishkdsttitkndnspvttgdyaaqtiiinaiksnfpddkvvgeesssglsdafvsgilneikandevynknykkddflftndqfplksledvrqiidfgnyeggrkgrfwcldpidgtkgflrgeqfavclalivdgvvqlgcigcpnlvlssygaqdlkghesfgyifravrglgafyspssdaeswtkihvrhlkdtkdmitledvekghsshdeqtaiknklniskslhldsqakycllalgladvylrlpiklsyqekiwdhaagnvivheaggihtdamedvpldfgngrtlatkgviassgprelhdlvvstscdviqsrnale
[0113]
236w
[0114]
malerellvatqavrkaslltkriqsevishkdsttitkndnspvttgdyaaqtiiinaiksnfpddkvvgeesssglsdafvsgilneikandevynknykkddflftndqfplksledvrqiidfgnyeggrkgrfwcldpidgtkgflrgeqfavclalivdgvvqlgcigcpnlvlssygaqdlkghesfgyifravrglgafyspssdaeswtkihvrhlkdtkdmitlewvekghsshdeqtaiknklniskslhldsqakycllalgladvylrlpiklsyqekiwdhaagnvivheaggihtdamedvpldfgngrtlatkgviassgprelhdlvvstscdviqsrnale
[0115]
236y
[0116]
malerellvatqavrkaslltkriqsevishkdsttitkndnspvttgdyaaqtiiinaiksnfpddkvvgeesssglsdafvsgilneikandevynknykkddflftndqfplksledvrqiidfgnyeggrkgrfwcldpidgtkgflrgeqfavclalivdgvvqlgcigcpnlvlssygaqdlkghesfgyifravrglgafyspssdaeswtkihvrhlkdtkdmitleyvekghsshdeqtaiknklniskslhldsqakycllalgladvylrlpiklsyqekiwdhaagnvivheaggihtdamedvpldfgngrtlatkgviassgprelhdlvvstscdviqsrnale
[0117]
尽管为说明目的公开了本发明的实施例,但是本领域的技术人员可以理解:在不脱离本发明及所附权利要求的精神和范围内,各种替换、变化和修改都是可能的,因此,本发明的范围不局限于实施例所公开的内容。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1