一种R-3-羟基丁酸乙酯微生物发酵制备方法与流程
一种r-3-羟基丁酸乙酯微生物发酵制备方法
技术领域
1.本发明涉及生物工程领域,特别涉及一种r-3-羟基丁酸乙酯微生物发酵制备方法。
背景技术:
2.3-羟基丁酸乙酯是合成许多手性药物的非常重要的手性医药中间体,3-羟基丁酸乙酯分子中含有多功能基团,其手性单一对映异构体(r)-3-羟基丁酸乙酯和(s)-3-羟基丁酸乙酯均是非常有前景的重要的手性砌块。如(s)-3-羟基丁酸乙酯是薰衣草醇、核球壳菌、格哈菌素、卡包霉素和灰绿霉素前提等天然产物的手性源,(r)-3-羟基丁酸乙酯是合成亚胺培南、美罗培南、帕尼培南、法罗培南、厄他培南、多尼培南、比阿培南等培南类抗生素和l-肉碱重要手性中间体。
3.现有技术中,手性拆分法反应条件温和,效率高,但应用于工业化制备时成本较高;化学不对称合成法反应快速,但手性催化剂制备困难、价格昂贵;微生物不对称还原合成3-羟基丁酸乙酯所得产物多为s型,r构型产物不易得到,且生物转化过程中底物浓度偏低也是生物催化不对称还原合成3-羟基丁酸乙酯所需要解决的问题。现有技术中公开了一种手性医药中间体r-3-羟基丁酸乙酯微生物发酵制备方法,先发酵生成3-羟基丁酸,然后再使用化学催化剂进行酯化反应,酯化反应是需要强酸/高温剧烈条件进行,只能在发酵结束后进行酯化,耗能,成本高,周期长,不易于提取精制,制备的3-羟基丁酸乙酯也容易发生水解,且发酵过程需要灭菌,极大的浪费能源。
技术实现要素:
4.针对现有技术中的缺陷,本发明提出了一种r-3-羟基丁酸乙酯微生物发酵制备方法。构建敲除聚3-羟基丁酸酯聚合酶基因和过表达β-酮硫解酶及乙酰乙酰辅酶a还原酶基因的工程菌。利用该工程菌进行发酵,在发酵过程中进行原位萃取。
5.本发明提供一种r-3-羟基丁酸乙酯微生物发酵制备方法,包括如下步骤:
6.s1:敲除halomonas lutescens mdf-9中的聚3-羟基丁酸酯聚合酶基因(phac),其核苷酸序列为seq id no.1,敲除后的聚3-羟基丁酸酯聚合酶基因失去聚合生成聚3-羟基丁酸的的功能,仍保留脱去辅酶a的功能,得到敲除菌株;
7.步骤s1中敲除phac酶基因序列中表达为使得(r)-3-羟基丁酰辅酶a脱去辅酶a的3-羟基丁酸聚合为聚3-羟基丁酸的片段,仍保留催化(r)-3-羟基丁酰辅酶a脱去辅酶a的基因片段。phac酶是聚3-羟基丁酸酯生物合成过程中的关键酶,能够用羟基脂肪酸的辅酶a硫酯作为底物,催化3-羟基丁酸脱去辅酶a聚合为聚3-羟基丁酸酯聚合物。而敲除聚3-羟基丁酸酯聚合酶基因片段,能使得在工程菌体内代谢产物为(r)-3-羟基丁酸单体,不再发生聚合反应生成聚合物。
8.s2:采用基因工程技术构建β-酮硫解酶(核苷酸序列为seq id no.2)和乙酰乙酰辅酶a还原酶(核苷酸序列为seq id no.3)的表达载体并导入s1得到的敲除菌株中,得到工
程菌;
9.将目的基因cds序列克隆到细胞内的质粒上,使两者基因可以在人为控制的条件下实现大量转录和翻译,产生足够的β-酮硫解酶和乙酰乙酰辅酶a还原酶,使得胞内能实现(r)-3-羟基丁酸的生成两分子乙酰辅酶a,经β-酮硫解酶生成(r)-3-羟基乙酰乙酰辅酶a,再经乙酰乙酰辅酶a还原酶生成(r)-3-羟基丁酰辅酶a。
10.将制得的工程菌株在以葡萄糖、果糖等六碳糖和乙醇为底物,细胞中引入外源及内源脂肪酶编码基因的表达并进行发酵,在脂肪酶的催化下,3-羟基丁酸乙酰辅酶a与乙醇发生酯化反应,脂肪酶由胞体自带和外源加入两部分组成。
11.s3:将s2得到的工程菌进行发酵,在发酵过程中同时使用萃取剂进行原位萃取,使发酵得到的3-羟基丁酸乙酯直接进入萃取相;有利于其后续的分离提取;
12.s4:提取产物。
13.进一步的,所述halomonas lutescens mdf-9的保藏编号为gdmcc no.61850。
14.进一步的,所述步骤s4采用间歇共沸精馏法分离萃取剂和3-羟基丁酸乙酯,得到最终产物3-羟基丁酸乙酯。
15.进一步的,所述萃取剂为中长链烷烃。
16.进一步的,所述萃取剂为正十二烷、正十四烷、乙酸乙酯中的任意一种。
17.进一步的,所述步骤s3具体包括如下步骤:
18.(1)平板种子培养:菌种活化;
19.(2)摇瓶种子培养;
20.(3)溶氧和ph电极校正;
21.(4)发酵参数的设定;
22.(5)接种;
23.(6)发酵过程控制。
24.进一步的,所述步骤(4)中发酵罐温度控制在35-40℃。
25.进一步的,所述步骤(4)中调节ph为7.5-9.5。
26.进一步的,所述步骤(6)中控制溶氧为35-80%。
27.进一步的,所述步骤(6)中控制发酵罐温度37
±
1℃,ph 8.5
±
1。
28.综上,与现有技术相比,本发明达到了以下技术效果:
29.(1)本发明的制备方法发酵和酯化同步进行,中间不经过生成3-羟基丁酸步骤。
30.(2)本发明的制备方法3-羟基丁酸乙酯生产和萃取同步进行,可有效避免3-羟基丁酸乙酯的水解。
31.(3)本发明的制备方法使用嗜盐单胞菌halomonas lutescens mdf-9为底盘生物,发酵过程不需要灭菌,操作更加方便,可实现连续接种或补充底物进行不间断发酵,更加节省能源。
32.(4)本发明的制备方法条件温和,能利用易获取的廉价底物快速繁殖、节能、环境友好,具有成本低,产量高,副产物少,易转化,周期短,易于提取精制等优点。
附图说明
33.为了更清楚地说明本发明实施例的技术方案,下面将对实施例中所需要使用的附
图作简单地介绍,应当理解,以下附图仅示出了本发明的某些实施例,因此不应被看作是对范围的限定,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他相关的附图。
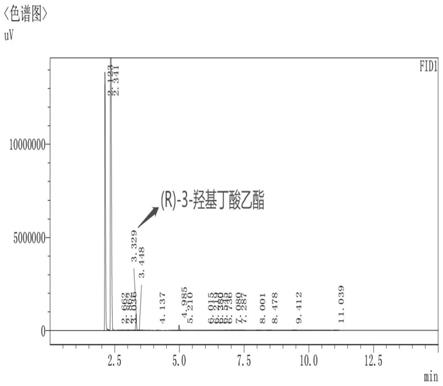
34.图1为本发明实施例3的产物气相色谱图。
具体实施方式
35.为了使本技术领域的人员更好地理解本发明方案,下面将结合本发明实施例中的附图,对本发明实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例仅仅是本发明一部分的实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动的前提下所获得的所有其他实施例,都应当属于本发明保护的范围。
36.本发明通过通过敲除halomonas lutescens mdf-9(保藏编号为gdmcc no.61850)中的聚β-羟基丁酸酯聚合酶基因,将halomonas lutescens mdf-9的β-酮硫解酶和乙酰乙酰辅酶a还原酶进行过表达构建基因工程菌。本发明的制备方法中发酵和酯化同步进行,中间不经过生成3-羟基丁酸步骤,这个效果通过引入外源脂肪酶的表达合成和胞内自有的脂肪酶在发酵过程中催化3-羟基丁酸乙酰辅酶a(3hb-coa)和摄入乙醇的酯化反应合成,脂肪酶的适用条件与halomonas lutescens mdf-9菌株发酵条件相似,在适宜ph条件下可达到60%酶活性以上。本发明的制备方法中3-羟基丁酸乙酯生产和萃取同步进行,可有效避免3-羟基丁酸乙酯的水解。本发明使用嗜盐单胞菌mdf为底盘生物,mdf系列菌株本身是改造过的工程菌,耐高盐/高碱,但不是所有嗜盐单胞菌属可以开放发酵,耐盐不一定耐碱。因为发酵过程为高盐高碱,培养基的高ph高浓度盐会使得大部分杂菌失活,所以利用mdf不需要灭菌。发酵过程由于不需要灭菌,所以操作更加方便,可实现连续接种或补充底物进行不间断发酵,比使用埃希氏菌属、假单包菌属、气单包菌属的菌株更加节省能源。
37.制备方法具体包括如下步骤:
38.s1:敲除halomonas lutescens mdf-9gdmcc no.61850中的聚3-羟基丁酸酯聚合酶基因,敲除后的聚3-羟基丁酸酯聚合酶基因失去聚合生成聚3-羟基丁酸的的功能,仍保留脱去辅酶a的功能,得到敲除菌株;
39.s2:采用基因工程技术构建β-酮硫解酶和乙酰乙酰辅酶a还原酶的表达载体并导入s1得到的敲除菌株中,得到工程菌;
40.s3:将s2得到的工程菌进行发酵,在发酵过程中同时使用萃取剂进行原位萃取,使发酵得到的3-羟基丁酸乙酯直接进入萃取相;所述萃取剂为中长链烷烃,为正十二烷、正十四烷、乙酸乙酯中的任意一种。
41.发酵过程如下:
42.(1)平板种子培养:菌种活化;
43.(2)摇瓶种子培养;
44.(3)溶氧和ph电极校正;
45.(4)发酵参数的设定;发酵罐温度控制在35-40℃,调节ph为7.5-9.5。
46.(5)接种;
47.(6)发酵过程控制。控制溶氧为35-80%,控制发酵罐温度37
±
1℃,ph 8.5
±
1。
73.(3)目的基因的扩增
74.扩增体系和反应程序如下:
75.扩增体系如下:
[0076][0077][0078]
pcr反应完成后,配制相应浓度的琼脂糖凝胶,进行电泳以便观察dna的条带大小,将凝胶置于紫外灯下,迅速的切下目的dna片段的凝胶,尽可能的将多余的凝胶切掉。
[0079]
(4)载体的限制性酶切反应
[0080]
按照如下体系进行双酶切反应,加完所有试剂后放置于37℃孵育器中3-4h,后将产物进行电泳,以便观察其酶切是否成功。
[0081][0082]
pcr反应完成后,配制相应浓度的琼脂糖凝胶,进行电泳以便观察dna的条带大小,将凝胶置于紫外灯下,迅速的切下目的dna片段的凝胶,尽可能的将多余的凝胶切掉。
[0083]
(5)扩增产物与载体酶切产物回收
[0084]
步骤1:将扩增产物和酶切产物用琼脂糖凝胶dna回收试剂盒(hipure gel pure dna mini kit)进行回收;
[0085]
步骤2:将在紫外灯下切的目的dna片段的凝胶装入2ml收集管中,加入500μl buffer gdp。如果凝胶浓度较大,可以适当的增加buffer gdp的体积。将凝胶放入烘箱10-15min,期间颠倒混匀,以至完全溶解;
[0086]
步骤3:将hipure dna mini colum装到2ml离心管中,将溶胶液转移至柱子中,如果溶胶液超过700μl则分两次转入。12000rpm离心1min
[0087]
步骤4:弃掉滤液,将柱子重新装入2ml收集管中。加入300μl buffer gdp,并且静置1min;
[0088]
步骤5:弃掉滤液,将柱子重新装入2ml收集管中。加入600μl buffer dw2(已提前加入无水乙醇)到柱子中。12000rpm离心1min;
[0089]
步骤6:重复上述步骤一次;
[0090]
步骤7:弃掉滤液,将柱子重新装入2ml收集管中。空甩12000rpm离心2min;
[0091]
步骤8:将柱子重新装入新的1.5ml离心管中。放入烘箱中5min,打开柱子的盖子以彻底去除无水乙醇(无水乙醇残留会影响后续反应);
[0092]
步骤9:往柱子膜中央加入15-30μl eb,室温(rt)放置2min。12000rpm离心1min。
[0093]
可重复上柱一次,洗脱下来的dna保存于-20℃。
[0094]
(6)同源重组反应体系
[0095]
采用一步克隆酶(clon expressii one step cloning kit),按照如下反应:
[0096]
加入微量管中混匀,短暂离心使反应液收集至管底。放置37℃孵育器30min。拿出微量管立即放至冰上或降至4℃
[0097][0098]
x(载体使用量)=(0.01
×
载体碱基数)/载体回收产物浓度;
[0099]
y(载体使用量)=(0.02
×
目的基因碱基数)/目的基因回收产物浓度。
[0100]
(7)mdf-9转化
[0101]
步骤1:将提前制备好的克隆感受态细胞从-80℃拿出,在冰上解冻,5min后待菌块融化;
[0102]
步骤2:往感受态细胞中加入10μl连接产物,轻轻的弹管壁将反应液混匀(请勿振荡混匀),后将其冰上静止10-30min。注:连接产物转化体积最多不应超过所用感受态细胞体积1/10;
[0103]
步骤3:42℃水浴热激45-90s后,立即置于冰上冷却2-3min,晃动会降低转化效率;
[0104]
步骤4:向离心管中加入700μl lb培养基(不含抗生素),混匀后放入37℃摇床中200rpm复苏60min;
[0105]
步骤5:5000rpm离心5min收菌,弃掉600μl上清留取100μl轻轻吹打重悬菌块并涂布到含相应抗生素的lb培养基上;
[0106]
步骤6:将培养基倒置到37℃培养箱中培养12-15h。
[0107]
(8)质粒小提
[0108]
采用magen生物公司质粒小提试剂盒(hipure plasmid micro kit)
[0109]
步骤1:将阳性的单克隆菌落接种于5-10ml含有相应抗生素的lb培养基中,置于37℃;摇床中12-16h。保存菌液于-80℃,以便后续接菌繁殖;
[0110]
步骤2:取2ml菌液装入提前准备好的2ml离心管中,12000rpm离心30-60s,收集2-3次;
[0111]
步骤3:倒弃上清,在吸水纸上轻轻拍打吸尽残液。加入250μl预冷的buffer p1/rnase a混合液,在振荡仪上高速涡旋彻底重悬细菌;
[0112]
步骤4:往2ml管中加入250μl buffer p2,上下轻轻颠倒混匀8-10次,溶液变得粘稠而透亮表明细菌已经充分裂解。注:如果涡旋会导致基因组dna污染。这一步如果样品较多,操作要迅速;
[0113]
步骤5:往上述重悬液中加入350μl buffer p3,立即颠倒混匀8-10次让溶液中和,防止沉淀团聚而影响中和效果;
[0114]
步骤6:12000rpm离心10min;
[0115]
步骤7:将hipure dna mini colum ii装入2ml collection tube中,将上清转至柱子中。12000rpm离心30-60s;
[0116]
步骤8:弃掉滤液,加入500μl buffer pw1至柱子中。12000rpm离心30-60s;
[0117]
步骤9:弃掉滤液,加入600μl已经用无水乙醇稀释过的buffer pw2至柱子中。12000rpm离心30-60s;
[0118]
步骤10:重复上一步骤;
[0119]
步骤11:将柱子装入提前准备好的1.5ml离心管中,往柱子膜中央加入15-30μl eb,室温放(rt)放置2min。12000rpm离心1min洗脱dna;
[0120]
步骤12:弃掉柱子,质粒用于后续反应保存与-20℃。
[0121]
酶切、测序鉴定,结果表明本实施例在实施例1的敲除菌株中成功转入了β-酮硫解酶和乙酰乙酰辅酶a还原酶基因,得到本发明用于发酵的工程菌。
[0122]
实施例3利用实施例2得到的工程菌进行发酵
[0123]
本实施例中使用的培养基:
[0124]
lb平板培养基:酵母浸粉,0.5%;胰蛋白胨,1%;氯化钠6%,琼脂粉1.8g/100ml,ph 8.0。
[0125]
lb摇瓶培养基:酵母浸粉,0.1%;氯化钠6%,ph 8.0,30ml/250ml。
[0126]
组分i:硫酸镁:0.2g/l;尿素:0.6g/l;(配50倍浓缩的母液:10g/l硫酸镁,30g/l尿素);
[0127]
组分ii:磷酸二氢钾(5.2g/l),配50倍母液260g/l;
[0128]
葡萄糖溶液(30g/l):配葡萄糖母液500g/l;
[0129]
组分iii(10ml/l):柠檬酸铁铵5g/l,无水氯化钙1.5g/l,12mol/l盐酸41.7ml/l);
[0130]
组分iv(1ml/l):(七水硫酸锌100mg/l,四水硫酸锰30mg/l,硼酸300mg/l,六水氯化钴200mg/l,五水硫酸铜10mg/l,氯化镍六水合物20mg/l,钼酸钠30mg/l。
[0131]
发酵培养基:
[0132]
玉米浆干粉36g(单独溶解后加入);
[0133]
mgso4(硫酸镁)0.6g;
[0134]
urea(尿素)6g;
[0135]
kh2po4(磷酸氢二钾)15.6g;
[0136]
c6h
12
o6(葡萄糖)60g(20g/l);
[0137]
nacl(氯化钠)150g。
[0138]
补料培养基:
[0139][0140][0141]
4%的naoh调节培养基ph,消泡剂5%量配置后添加。
[0142]
ph控制在7.5-9.0。
[0143]
(一)平板种子培养:菌种活化
[0144]
实验室4℃冰箱取菌种,用酒精棉将双手消毒,待双手酒精干透后,点酒精灯。平皿底部写上接种菌名、日期和时间。用接种环挑取单菌落划线接种于平板上,培养24小时。重复上述操作,接种平板二级,培养24小时。
[0145]
(二)摇瓶种子培养
[0146]
一级菌液:取二级平板,挑取单菌种接种于lb摇瓶培养基中,将培养液置于摇床37℃、220rpm培养12小时。
[0147]
二级菌液:吸取一级菌液300微升(1%接种量),接种于二级摇瓶培养基中将培养液置于摇床37℃、220rpm培养12小时。
[0148]
(三)溶氧和ph电极校正
[0149]
发酵罐用水冲洗干净,对do电极进行标零,ph电极进行两点校准(标准缓冲液放置常温),校准后正确安装在发酵罐上,空消。(ph电极和溶氧电极不用空消)。
[0150]
(四)发酵参数的设定
[0151]
准备好组分iii和组分iv(提前溶解),发酵罐温度控制在35-40℃。利用碱液调节ph为8.2,加入消泡剂0.3ml,打开空气进罐阀调节初始空气流量2l/min进罐,初始转速调至400rpm,待od示数稳定后标定100%。
[0152]
(五)接种
[0153]
挑选颜色均匀,少沉淀物的种子液300ml,将种子液接种进入发酵罐,发酵罐中使用发酵培养基,摇瓶残液并预留10ml菌液测定od,残糖,接种后倒入组分iii(30ml)和组分iv(3ml)。
[0154]
(六)发酵过程控制
[0155]
控制发酵罐温度37
±
1℃,ph 8.5
±
1,转速和空气流量交替调节控制溶氧35-80%,初始转速为400rpm,通气量2l/min,转速每次调节50rpm,最高转速800rpm,空气流量每次调节0.5l/min,最高调节3l/min,前四个小时,每两个小时取样,测od,残糖。
[0156]
(1)正常情况下,溶氧会逐渐下降,转气控制溶氧35%以上,每半个小时对罐内液面情况进行确认,液面太高,需要消泡(少量多次,避免过量)。
[0157]
(2)每两小时取样,先放出几秒,再取菌液2ml,检测菌液离线ph,稀释,测残糖和和od值。
[0158]
流加控制残糖5-15g/l。
[0159]
(七)离心纯化提取
[0160]
(1)使用中长链烷烃(本实施例采用正十二烷)做为萃取剂进行原位萃取,使发酵
得到3-羟基丁酸乙酯直接进入中长链烷烃相。
[0161]
(2)采用间歇共沸精馏法分离中长链烷烃和3-羟基丁酸乙酯,得到最终产物3-羟基丁酸乙酯。
[0162]
(3)产物浓度的测定
[0163]
气相色谱条件为:柱温80℃保留3min,升温速率8℃/min,升温至120℃,进样器温度220℃,检测器温度250℃,载气为氮气,进样量为1μl,以正十二烷为内标物。
[0164]
(4)产物光学纯度的测定
[0165]
样品衍生化处理:取样品1ml放入80℃水浴中蒸干溶剂加入100μl三氟乙酸酐和1ml正己烷后置于30℃水浴衍生化15min。然后将反应液置于80℃水浴中挥发干瓶内液体,加入适量乙酸乙酯。
[0166]
光学纯度的气相测定:采用以β-环糊精衍生物为填充物的手性柱。气相色谱条件为:柱温60℃保留时间7min升温速率2℃/min升温至180℃。进样器温度220℃,检测器温度250℃,进样量为1μl。气相图谱如图1所示。e.e.的计算公式为:e.e.%=(sr-ss)/(sr+ss)
×
100%。由图1结果可知,本实施例中发酵得到的产物确为r-3-羟基丁酸乙酯。
[0167]
发酵结束后,测量在ph 8.2条件下5l发酵罐培养中获得的细胞干重为40g/l,其中3-羟基丁酸乙酯含量为80%,3-羟基丁酸乙酯产量为32g/l,r型3-羟基丁酸乙酯手性纯度达到99%。
[0168]
实施例4利用实施例2得到的工程菌进行发酵
[0169]
操作步骤与实施例3相同,利用碱液调节ph为7.5,使用正十四烷做为萃取剂进行原位萃取,使发酵得到3-羟基丁酸乙酯直接进入正十四烷烃相。
[0170]
发酵结束后,测量在ph 7.5条件下5l发酵罐培养中获得的细胞干重为40g/l,其中3-羟基丁酸乙酯含量为60%,3-羟基丁酸乙酯产量为24g/l,r型3-羟基丁酸乙酯手性纯度达到99.8%。
[0171]
综合以上实施例,本发明公开了一种r-3-羟基丁酸乙酯微生物发酵制备方法。包括如下步骤:s1:敲除halomonas lutescens mdf-9中的聚3-羟基丁酸酯聚合酶基因,得到敲除菌株;s2:采用基因工程技术构建β-酮硫解酶和乙酰乙酰辅酶a还原酶的表达载体并导入s1得到的敲除菌株中,得到工程菌;s3:将s2得到的工程菌进行发酵,在发酵过程中同时使用萃取剂进行原位萃取;s4:提取产物。本发明的制备方法发酵和酯化同步进行,中间不经过生成3-羟基丁酸步骤。3-羟基丁酸乙酯生产和萃取同步进行,可有效避免3-羟基丁酸乙酯的水解。发酵过程不需要灭菌,操作方便,能利用易获取的廉价底物快速繁殖、节能、环境友好,具有成本低,产量高,副产物少,易转化,周期短,易于提取精制等优点。
[0172]
以上所述仅为本发明的较佳实施例,并不用以限制本发明,凡在本发明的精神和原则之内,所作的任何修改、等同替换、改进等,均应包含在本发明的保护范围之内。
[0173]
序列表
[0174]
seq id no.1:
[0175]
atgcagaacaacacgcattcattcgcaaccttctggtcgggacaggttcctttcatcgcctcctttgccgtgcaacaactgcgcctgtgggtgagcaccactccgtggttttccgggcatgaccaagacacctggttcgacgtgcctcgcgagaccctggacagccttcaagcggactaccaggttgaatgggctcaactcggccagaaactgctgaccggccagcccttcagctttgatgatcgacgcttcaccagcggcgactggagcacccccctgttcggctccctcgccgcgttt
tacctgctcaactccagcttcttgctgaaactgctcgacaagctgcgaatcgacgaaaagaaagcacaccaacgcctgcgctatctggtggagcaatccatggccgccagtgcaccgagcaatttcctggccagcaaccctgatgcgttgcagactgcggtcgagacccaaggtgccagcctgatcacagggatgcagcatatggccagcgacatgaacgaaggcaaaatgcgccagtgcgatagcggcgccttcaaggtgggtgtcgacctggccaatacccccggcgaagtcgtcttcgagaatcgcatcttccaactcatccagtactacccgcaaagcgaaacccagtaccagcagccggtgctcgtcgtacctccatcgatcaacaagtactacatccttgatctgcgccccgacaactcgatggtccgccacctgctggaaaagggacatcccgtattcctggtctcctggcgcaactttgaccaggaacacgccggtaccacctgggacgactggatcgagcacggggtgataaacgccgtgagcgtgacccgcgagatcagtggccaacagcgcctcaattgcgtgggtttctgcattggcggcaccctgttgagtacggcgctggccgtattggctgcacgtggcgaccgcgagatcgccagcctgagcctgttcgccacgttcctcgattacctggataccggtcccatcgacatcttcgtcgatgaagaactggtgcgttaccgcgagcgcaccattggcggtgtcaacggccctatcggattgttccgtggcgaagacatgggcaataccttttccctgctgcgccccaacgaactggtgtggaactacaacgtcgacaaatacctcaaggggcaaaagccgattccactggacctgctgttctggaacaacgacagcaccaaccttccagggccgatgtactgctggtacctgcgccacacctacctgcagaacgacttgaagtcaggcgagctggagtgctgcgggaccaagctggattttcgtgcaatcgacgcgcccgcctacatcctcgccacccatgacgatcacatcgtgccgtggaagagcgcctacgccagcacccaggtgctttccgggcccaagcgtttcgtactgggtgcttccgggcacatcgccggcgtcgtcaatccgccggccaagcagaagcgtcattactggaccaacaatggcgggtcactaaaaaccccgagacctggttcaagaatgccgagcagcaacaaggtagctggtggaatgactggttcaactggctggcggggcactcaggcgagcagcaacccgcggttgcgcacatcggcaatgaccaatacccgcctctggagccggcgccaggacgttatgaagcaatga
[0176]
seq id no.2:
[0177]
atgactcagcgcattgcgtatgtgaccggcggcatgggtggtatcggaaccgccatttgccagcggctggccaaggatggctttcgtgtggtggccggttgcggccccaactcgccgcgccgcgaaaagtggctggagcagcagaaggccctgggcttcgatttcattgcctcggaaggcaatgtggctgactgggactcgaccaagaccgcattcgacaaggtcaagtccgaggtcggcgaggttgatgtgctgatcaacaacgccggtatcacccgcgacgtggtgttccgcaagatgacccgcgccgactgggatgcggtgatcgacaccaacctgacctcgctgttcaacgtcaccaagcaggtgatcgacggcatggccgaccgtggctggggccgcatcgtcaacatctcgtcggtgaacgggcagaagggccagttcggccagaccaactactccaccgccaaggccggcctgcatggcttcaccatggcactggcgcaggaagtggcgaccaagggcgtgaccgtcaacacggtctctccgggctatatcgccaccgacatggtcaaggcgatccgccaggacgtgctcgacaagatcgtcgcgacgatcccggtcaagcgcctgggcctgccggaagagatcgcctcgatctgcgcctggttgtcgtcggaggagtccggtttctcgaccggcgccgacttctcgctcaacggcggcctgcatatgggctga
[0178]
seq id no.3:
[0179]
atgactgacgttgtcatcgtatccgccgcccgcaccgcggtcggcaagtttggcggctcgctggccaagatcccggcaccggaactgggtgccgtggtcatcaaggccgcgctggagcgcgccggcgtcaagccggagcaggtgagcgaagtcatcatgggccaggtgctgaccgccggttcgggccagaaccccgcacgccaggccgcgatcaaggccggcctgccggcgatggtgccggccatgaccatcaacaaggtgtgcggctcgggcctgaaggccgtgatgctggccgccaacgcgatcatggcgggcgacgccgagatcgtggtggccggcggccaggaaaacatgagcgccgccccgcacgtgctgccgggctcgcgcgatggtttccgcatgggcgatgccaagctggtcgacaccatgatcgtcgacggcctgtgggacgtgtacaaccagtaccacatgggcatcaccgccgagaacgtggccaaggaatacggcatcacacgcgaggcgcaggatgagttcgccgtcggctcgcagaacaaggccgaagccgcgcagaaggccggcaagtttgacgaagagatcgtcccggtgct
gatcccgcagcgcaagggcgacccggtggccttcaagaccgacgagttcgtgcgccagggcgccacgctggacagcatgtccggcctcaagcccgccttcgacaaggccggcacggtgaccgcggccaacgcctcgggcctgaacgacggcgccgccgcggtggtggtgatgtcggcggccaaggccaaggaactgggcctgaccccgctggccacgatcaagagctatgccaacgccggtgtcgatcccaaggtgatgggcatgggcccggtgccggcctccaagcgcgccctgtcgcgcgccgagtggaccccgcaagacctggacctgatggagatcaacgaggcctttgccgcgcaggcgctggcggtgcaccagcagatgggctgggacacctccaaggtcaatgtgaacggcggcgccatcgccatcggccacccgatcggcgcgtcgggctgccgtatcctggtgacgctgctgcacgagatgaagcgccgtgacgcgaagaagggcctggcctcgctgtgcatcggcggcggcatgggcgtggcgctggcagtcgagcgcaaataa
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1