用于快速RNA-腺苷酸化和RNA测序的组合物和方法
用于快速rna-腺苷酸化和rna测序的组合物和方法
1.相关申请的交叉引用
2.本技术要求于2020年2月6日提交的美国临时专利申请第62/971,214号的优先权,所述美国临时专利申请的整个公开内容通过引用并入。
技术领域
3.本公开涉及用于rna测序的改善的组合物和方法。
背景技术:
4.用于查询细胞dna、rna和蛋白质含量的全基因组分析的出现已彻底改变了介导细胞稳态的基因表达的研究。rna-seq使用下一代测序(ngs)以揭示生物样品中rna分子的存在和数量。尽管rna-seq是用于量化基因表达的有力方法,但其不能够可靠地鉴定先前未知的转录物、剪接同种型和天然存在的rna片段。已设计了专门的rna-seq方法以鉴定转录物(1)的5末端,如cage(基因表达的帽分析)、strt(单细胞标签逆转录)、nanocage(基因表达的纳米帽分析)、tss-seq(寡聚加帽)和gro帽(全局核连缀帽(global nuclear run-on cap))。然而,这些方法检测rna分子上5'末端帽的存在。因此,这些方法不适用于鉴定rna片段,而所述rna片段的鉴定对于rna代谢的研究至关重要。作为一个实例,核糖体谱(ribo-seq)捕获通过核酸酶消化和其后的深度测序产生的整套受核糖体保护的mrna片段(rpf)(2)。其以亚密码子分辨率提供了在整个转录组中核糖体的位置和密度的简要说明。由于常用的rna-seq方法不能够应用于核糖体谱,因此ribo-seq的广泛应用由于方案的复杂性和费用而放缓。另外,由于样品制备的细节可能会引入偏差和伪影,因此对ribo-seq结果的解释已经产生了担忧(3)。在文库制备期间,例如,环化或接头连接的效率可能受到rpf的5'末端核苷酸身份的影响。因此,在技术上扩大或耗竭的rpf可以改变核糖体足迹的整体模式。另外,使用翻译抑制剂放线菌酮(chx)进行的预处理已显示出可使密码子密度有偏差,并且诱导不需要的细胞应答(4)。尽管省略chx预处理已经成为惯例,但消除由各种方案引入的伪影仍然是具有挑战性的。
5.最近引入了无连接ribo-seq方法(5)。然而,所述方法依赖于模板转换技术,所述技术以更高的效率严重偏向于在5'末端具有g核苷酸的rna分子(6)。因此,此无连接方法由于不可接受的5'末端偏差将扭曲后续数据分析而不适用于ribo-seq。出于类似理由,illumina超低rna测序试剂盒(clontech)不能够用于ribo-seq,因为其使用smart(在rna转录物的5'末端处的转换机制)以生成mrna分子的全长cdna拷贝。尽管smart方法能够由单细胞量的rna制备用于测序的cdna,但是其费时、昂贵并且限于mrna测序。对于rna片段,smart方法不能够准确地捕获具有随机5'核苷酸的rna分子。在先前ribo-seq的方法中显而易见的rna测序挑战也与对任何rna多核苷酸进行测序有关。因此,对于用于对rna多核苷酸进行测序的改善的组合物和方法有着持续存在且未被满足的需要。本公开与此需要相关。
技术实现要素:
6.本公开提供了用于rna测序的组合物和方法。所述方法在本文中称为简单rna-腺苷酸化测序(“ezra-seq”)。在图1a中提供了可用方法与ezra-seq在对受核糖体保护的片段进行测序的方面的比较。在图1b中提供了所述方法的概述。本公开提供在单管中加工rna样品和生成cdna,如图1c中大体上描绘的。
7.在实施方式中,所述方法包括使用酶的混合物修饰rna以产生用于测序的cdna,并且进一步提供包括可以用于所述方法中的酶的区段的融合蛋白。除了其它酶以外,酶的所述混合物还含有环化酶和聚合酶。所述环化酶和聚合酶可以以融合蛋白的形式提供。
8.在一实施方式中,本公开提供了一种用于确定rna多核苷酸的核苷酸序列的方法。所述方法通常包括:a)提供从所述rna多核苷酸获得的多个rna和/或rna片段;b)通过酶促将所述多个rna片段的5'末端磷酸化以提供含有单磷酸化5'末端的多个rna片段;c)通过酶促将所述多个rna片段的3'末端去磷酸化以提供含有游离3'羟基的多个rna片段;d)通过酶促将所述多个rna片段的磷酸化5'末端腺苷酸化以提供多个5'单腺苷酸化的rna片段;e)通过酶促将含有所述游离3'羟基的所述多个rna片段的3'末端多腺苷酸化以提供含有多腺苷酸化3'末端的多个rna片段;f)将寡核苷酸衔接子连接到所述多个5'单腺苷酸化的rna片段的所述5'末端,以提供在所述5'末端处含有所述寡核苷酸衔接子的多个rna片段,所述寡核苷酸衔接子任选地包括dna/rna杂合体3'末端,所述dna/rna杂合体3'末端任选地包括rcrc-oh或rgrg-oh作为所述dna/rna杂合体的rna组分;g)由f)的所述多个rna片段生成cdna;以及h)扩增所述cdna。本公开进一步提供:i)确定所述cdna的核苷酸序列,从而确定所述rna多核苷酸的所述核苷酸序列。
9.在某些方法中,至少所述步骤b)-h)在单个反应容器中进行。在某些方法中,所述反应容器包括底物,如链霉亲和素。这可以例如与用于通过逆转录生成cdna的引物一起使用,所述引物包括与所述底物结合的结合配偶体(binding partner),例如生物素部分。本公开提供了改善的5'末端测序。
10.在实施方式中,所述方法部分地使用连接酶进行以通过酶促将rna片段的5'末端磷酸化以提供含有单磷酸化5'末端的多个rna片段。所述连接酶还通过酶促将所述rna片段的3'末端去磷酸化以提供含有所述游离3'羟基的多个rna片段。在某些方法中,通过所述连接酶修饰的所述rna多核苷酸使用上述环化酶和聚合酶进一步修饰,所述环化酶和聚合酶可以以单独蛋白的形式提供或以单一融合蛋白的组分的形式提供。使用此方法促进了通过酶促将所述多个rna片段的所述磷酸化5'末端腺苷酸化,以及将所述多个rna片段的所述3'末端多腺苷酸化。在非限制性实施方式中,聚合酶包括poly(a)聚合酶,所述poly(a)聚合酶获自或衍生自大肠杆菌(e.coli)poly(a)聚合酶(大肠杆菌pap1)或酿酒酵母(saccharomyces cerevisiae)poly(a)聚合酶(酿酒酵母pap1)。在非限制性实施方式中,所述环化酶催化rna 2',3'-环磷酸末端的合成并且催化所述多个rna片段的5'-磷酸末端的腺苷酸化(adenylylation)。代表性环化酶包括rtca。在实施方式中,所述方法还包括将寡核苷酸衔接子连接到所述多个5'单腺苷酸化的rna片段的所述5'末端,所述连接可以使用t4 rna连接酶进行。
11.因此,在代表性实施方式中,所述方法提供了一种用于cdna文库构建的重新设计的方案(图1b)。示出了用于对受核糖体保护的片段(rpf)进行测序的方法,但所述方法可以
经调适以与任何其它类型的rna一起使用。具体地,代替使用3'末端接头连接和环化,本公开提供了能够对同一rna片段施加3'末端poly(a)加尾和5'末端腺苷酸化的酶促系统。特别设计的5'寡核苷酸允许将衔接子高效地连接到所述腺苷酸化的rpf。通过将生物素化的寡核苷酸用于逆转录,整个程序可以在单管内完成,其中非特异性产物极少。与标准ribo-seq相比,ezra-seq明显降低了起始材料的量(约1ng rna),将整个文库加工时间从4天缩短至约4小时,并且增加了rpf的分辨率,其中平均ifr》90%。从相同原始样品中,ezra-seq以完美的阅读框使rpf的量接近翻倍(图1a,底部小图)。优异的分辨率在包括实体组织在内的不同细胞类型中具有高度可重复性和可实现性。在实施方式中,对根据所述方法加工的所述多个rna片段的所述5'末端的至少80%进行测序。在某些和非限制性实施方式中,如在对受核糖体保护的rna片段进行测序的情况下,本公开提供了针对测序的rna多核苷酸确定具有至少90%的框内比率(ifr)的序列。如以下进一步解释的,用于ribo-seq的所述方法可适用于确定任何类型的rna多核苷酸的序列。
12.对于用于进行所述方法和用于包括在试剂盒和制品中,本公开还提供了一种用于rna测序的组合物,所述组合物包含两种不同蛋白质的混合物或融合蛋白,所述两种不同蛋白质或所述融合蛋白包括poly(a)聚合酶和rna 3'-磷酸环化酶。还提供了包含此类融合蛋白或蛋白质的混合物的组合物。本公开包括分离的融合蛋白,所述分离的融合蛋白包括poly(a)聚合酶和rna3'-磷酸环化酶。
13.一方面,本公开提供了一种试剂盒,所述试剂盒包括两种所述不同蛋白质的混合物或融合蛋白,其中所述试剂盒还可以含有rna连接酶或rna激酶中的至少一种。所述试剂盒还可以包括至少一个容器,所述至少一个容器至少含有所述两种不同蛋白质的所述混合物或所述融合蛋白。可以使用任何容器,如小瓶、广口瓶、可密封管等。所述试剂盒可以进一步包括用于cdna合成的至少一种寡核苷酸引物。通常,所述寡核苷酸引物含有poly-t区段。所述引物可以被标记,使得所述引物可以与结合配偶体结合。在一个实施方式中,所述标记包括生物素。所述试剂盒还可以包括珠,所述珠包括被配置成与所述标记结合的部分。在一实施方式中,所述部分是链霉亲和素。可以使用任何合适的珠,并且所述珠是可商购获得的。在实施方式中,珠包括磁珠。所述试剂盒还可以包括适用于rna测序的缓冲液。在实施方式中,所述缓冲液具有大约7.0的ph和/或大于1mm并且任选地为大约2mm的atp浓度。
14.本公开还提供了制品,所述制品包括至少一个密封容器,所述至少一个密封容器可以含有与所述试剂盒相同或类似的组分。所述制品还可以含有印刷材料和标牌,所述印刷材料和标牌提供所述组分用于rna测序,并且可以包括用于使用所述试剂盒组分的说明。
附图说明
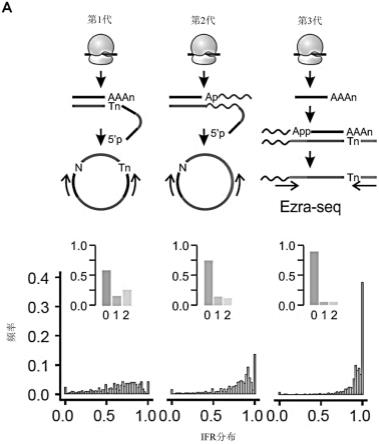
15.图1.ezra-seq和常规ribo-seq方法的示意性表示(a)。下面列出了在ifr分辨率方面的结果的直接比较。ifr:核糖体足迹的框内比率。(b)应用于核糖体谱的ezra-seq的工作流程。rpf:受核糖体保护的rna片段。(c)使用ezra酶的单管反应的整体过程。
16.图2.rtca通过atp依赖性途径催化rna2',3'-环磷酸末端的合成。在用t4 pnk预处理后,rtca催化rna5'-单磷酸末端的连接酶样腺苷酸化(adenylylation)。
17.图3.针对ezra系统(rtca+pap1)测试了不同缓冲液,在(a)中示出。p指示单个步骤中分别的阳性对照。(b)针对ezra系统(rtca +pap1)在不同atp浓度的情况下测试了新的缓
冲液。p指示单个步骤中分别的阳性对照。(c)ezra系统的优化缓冲液。
18.图4.如(a)中所示的,重组ezra酶包括pap1和rtca,使得能够对具有5'单磷酸和3'oh的rna分子进行5'末端腺苷酸化和3'末端多腺苷酸化。(b)酵母pap1与大肠杆菌pap1之间的多腺苷酸化效率。
19.图5.(a)中示出了生物素rt-引物的全序列(seq id no:5)。(b)用于连接的5'衔接子的全序列(seq id no:2)。(c)具有各种3'核糖核苷酸的5'衔接子与由t4 rnl2催化的apprna之间的连接效率。
20.图6.(a)中示出了使用ezra-seq技术结合基于蔗糖梯度的核糖体分级进行的mef细胞的ribo-seq。(b)在未使用基于蔗糖梯度的核糖体分级的情况下使用ezra-seq技术进行的mef细胞的ribo-seq。(c)使用ezra-seq技术的线粒体ribo-seq和rna-seq。(d)使用ezra-seq技术的染色质相关rna-seq。
21.图7.3'和5'腺苷酸化的图形描绘。
22.图8.珠结合的图形描绘。
23.图9.连接的图形描绘。
24.图10.cdna合成的图形描绘。
25.图11.pcr扩增的图形描绘。
具体实施方式
26.除非有相反的规定,否则本说明书通篇中给出的每个最大数值限制包括每个较低的数值限制,如同此类较低的数值限制在本文中明确写出一样。在整个说明书中给出的每个最小数值限制将包括每个较高的数值限制,如同此类较高的数值限制在本文中明确写出一样。贯穿本说明书给出的每一数值范围将包括落在此类较宽数值范围内的每一较窄数值范围,如同此类较窄数值范围在本文中明确写出一样。
27.本公开包括本文所描述的每个氨基酸序列,以及编码所述氨基酸序列的每个多核苷酸序列,包括但不限于cdna序列和rna序列。还包括互补序列和反向互补序列。本公开涵盖包括此类核苷酸序列的表达载体。
28.包括了包括与本公开的氨基酸序列至少80%相同的氨基酸序列的多肽。在实施方式中,蛋白质包括相对于内源性蛋白的突变。“内源性”蛋白是通常由未经修饰的基因编码的蛋白质。同样地,内源性基因或其它多核苷酸包括未如通过重组、基因编辑或其它方法修饰的dna序列。突变可以包括氨基酸插入、缺失和改变。
29.在实施方式中,本公开提供了用于rna腺苷酸化和测序的组合物和方法。所述方法有时被称为ezra-seq,其代表简单rna-腺苷酸化测序。术语“简单”应在本公开的上下文中看待,这提供了用于以先前无法获得的效率和分辨率进行rna测序的新型组合物和方法,但并非旨在表示本公开的简单化性质。
30.通常,本公开提供了用于rna相关测序的组合物和方法,其中rna片段用3'末端poly(a)加尾和5'末端腺苷酸化进行修饰,然后直接扩增。与在不使用酶的情况下进行的化学修饰相反,所述修饰是通过酶促(例如,酶)完成的。
31.在实施方式中,所提供的本公开的方法可以使用或不使用融合蛋白,如通过使用不同酶的混合物。因此,一方面,本公开提供了适用于所述rna修饰方法的一种或多种融合
蛋白,所述rna修饰方法包括但不必限于rna的5'和3'腺苷酸化。融合蛋白包括单一连续多肽,其中在融合蛋白内具有不同蛋白的区段。在实施方式中,本公开的融合蛋白被称为“ezra”酶,其代表简单rna-腺苷酸化酶。因此,在实施方式中,所述组合物、方法和试剂盒中使用的所有酶可以是单独蛋白,或者所述酶中的一些酶可以存在于至少一种融合蛋白中。在实施方式中,所述酶中的至少两种酶可以存在于融合蛋白中。在实施方式中,融合蛋白包括作为环化酶的区段和作为聚合酶的区段。在实施方式中,环化酶包括催化rna 2',3'-环磷酸末端的合成并且还催化rna链的5'-磷酸末端的腺苷酸化(adenylylation)的rna 3'-磷酸环化酶。在实施方式中,所述蛋白获自或衍生自:原核生物,例如细菌;或真核生物,例如酵母。在实施方式中,可以用作不同的蛋白或融合蛋白的组分的聚合酶包括poly(a)聚合酶。poly(a)聚合酶可以分离或衍生自原核生物或真核生物来源。“衍生自”是指内源性产生的蛋白可以被修饰,如以包括纯化标签,或氨基酸序列中的一个或多个变化,条件是所述蛋白质保留其酶促功能。在实施方式中,所述蛋白质包括任何合适的纯化标签,包括但不必限于通常含有2至10个组氨酸的多组氨酸标签、strep标签、小泛素样修饰物(sumo)、麦芽糖结合蛋白(mbp)标签、n端谷胱甘肽s-转移酶(gst)等。
32.在一实施方式中,poly(a)聚合酶是大肠杆菌poly(a)聚合酶或酿酒酵母poly(a)聚合酶。此类酶的代表性和非限制性实施方式提供为大肠杆菌poly(a)聚合酶(pap1)或酿酒酵母poly(a)聚合酶(pap1)。环化酶的代表性和非限制性实例是大肠杆菌rtca。在实施方式中,可以使用本文所述的酶的功能区段。功能区段包括所述酶的执行其预期功能所必需且足以执行其预期功能的区段,所述酶的功能在本文中进一步描述并且在某些图中展示。
33.结合本公开,已知rna片段的高通量测序(hts)通常需要制备文库,其中rna置于已知5'端序列与3'端序列之间。在本公开之前,用于文库构建的可用方法利用rna或dna衔接子来连接到靶rna分子的5'末端和3'末端。衔接子提供首先用于逆转录(rt)引物并且然后用于聚合酶链反应(pcr)和hts测序的引物退火位点。然而,以这种方式连接衔接子不仅耗时,而且过程效率低下,需要数微克投入物。另外,所得的cdna文库被交叉连接和自连接衔接子副产物污染,并且在预扩增之前和之后需要另外的纯化步骤。另外,具有5'凹陷末端的小rna是酶促衔接子连接的不良底物(8)。
34.许多以前可用的小rna测序方案在cdna文库制备期间使用具有5'预腺苷酸化的合成的dna寡核苷酸衔接子。衔接子5'末端的预腺苷酸化促进衔接子与rna分子的3'末端的连接,而无需添加atp,从而避免了atp依赖性副反应。然而,dna衔接子的预腺苷酸化可能既昂贵又困难。以前用于dna衔接子的化学腺苷酸化的可用方法效率低下,并且需要另外的纯化步骤。最近引入了使用商业rna连接酶的替代性的酶促方法,但此酶作为化学计量的腺苷酸化试剂而不是催化剂效果最好(9)。因此,在实施方式中,本公开包括rna腺苷酸化不是使用预腺苷酸化的寡核苷酸而进行的附带条件。相反,腺苷酸化直接在rna多核苷酸上通过酶促进行,包括但不限于rna多核苷酸的片段。
35.本公开演示了不仅催化rna 2',3'-环磷酸末端的合成,而且还催化rna链的5'-磷酸末端的腺苷酸化(adenylylation)的rna 3'-磷酸环化酶(rtca)的使用(图2)。腺苷酸化(adenylylation)产生图2所示的“app”结构,其示出了具有两个磷酸的单个a。此腺苷酸化(adenylylation)也可以称为腺苷酸化(adenylation),这在本领域中经常如此。本公开包括但不限于图2所示的rna的所有酶促修饰。
36.当用将5'末端磷酸化但将3'末端去磷酸化的合适的激酶预处理rna片段时,一旦5'末端被rtca腺苷酸化,rna片段就成为活性“接头”。合适的激酶的代表性和非限制性实例在本文中展示为t4多核苷酸激酶。
37.对于要测序的rna片段,文库制备需要5'和3'衔接子。如本文所使用的rna片段可以是任何合适的大小,所述大小的非限制性实施方式包括最小长度为大约20个核苷酸的rna多核苷酸。在实施方式中,长度为20-100nt,但较短或较长的多核苷酸不排除在本公开的范围之外。因此,在实施方式中,rna片段可以包括未必如通过机械片段化而被片段化的rna多核苷酸。虽然本公开适用于对如完整mrna等完整rna或如某些mirna和trna等小rna进行测序,但在实施方式中,本公开涉及对已片段化的rna多核苷酸进行测序。可以使用任何合适的技术实现rna片段的生成,这通常涉及完整rna多核苷酸的机械破坏。合适的方法包括但不限于超声处理、声波剪切、流体动力剪切,但可以使用替代性方法,如加热和二价金属阳离子暴露。
38.标准方案使用单独的步骤,并需要对连接产物进行另外的纯化。与常用的3'接头连接相比,3'末端处的多腺苷酸化是高效且无偏差的。在atp存在的情况下,来自大肠杆菌或酿酒酵母的poly(a)聚合酶(pap1)将poly(a)尾添加到rna片段。由于5'腺苷酸化(adenylylation)和3'多腺苷酸化都需要atp分子并且在相同的温度(37℃)下发生,因此本公开提供了将这两种反应组合成单个反应(例如,ezra)。系统可以通过将加工时间从2小时缩短到30分钟来大大简化整个程序。重要的是,所述方法通过省略纯化步骤,如乙醇沉淀预防了产物损失。因此,在一实施方式中,本公开的方法可以不含乙醇沉淀或通过其它溶剂进行的沉淀。
39.然而,在本公开中揭示了用于pap1和rtca酶的工作缓冲液不相容。具体地,用于pap1的最佳缓冲液的ph为7.9,而rtca在ph 6.0下效果最佳。在对ph值进行滴定后,发现ph 7.0对pap1和rtca都有效(图3a)。另外,通过将atp浓度从1mm增加到2mm,获得了更高的效率(图3b)。利用这些新型缓冲液条件(图3c),本公开提供了能够对同一rna样品进行5'腺苷酸化(adenylylation)和3'多腺苷酸化的ezra系统。所述所有缓冲液都包括在本公开的范围内。
40.为了增加在rna片段的两个末端处rna腺苷酸化的效率,对由rtca和pap1构成的几种融合蛋白进行工程化。具有xten接头的最活跃的酶(pap1-rtca)被重新命名为ezra酶,所述ezra酶代表简单rna腺苷化酶(图4)。因此,rna5'-腺苷酸化和3'-多腺苷酸化可以在同一管中实现而无需纯化。因此,在实施方式中,环化酶和聚合酶可以通过任何合适的接头在融合蛋白内彼此分离,所述接头的非限制性实施方式是所述xten接头。然而,如在本领域中已知的,合适的接头可以包括不同的长度和不同的氨基酸序列,并且任何合适的接头都可以用于产生环化酶和聚合酶的融合蛋白。在实施方式中,接头可以包括1至20个氨基酸,包括端点,并且包括其间的所有整数和整数范围。在实施方式中,使用柔性接头。在实施方式中,接头可以包括甘氨酸和丝氨酸。
41.进一步地,虽然本公开的非限制性演示示出了聚合酶位于n端而后是位于c端的环化酶,但可以使用其它构型,如使聚合酶相对于环化酶位于c端。
42.在实施方式中,所述组合物、方法和试剂盒可以包括两种不同蛋白质,或包括两种不同蛋白质的氨基酸序列的融合蛋白。在一实施方式中,不同蛋白质是rna 3'-磷酸环化酶
(rtca)和poly(a)聚合酶。在非限制性实施方式中,poly(a)聚合酶是大肠杆菌poly(a)聚合酶(pap1)或酿酒酵母poly(a)聚合酶(pap1)。以下描述了合适的环化酶和聚合酶的代表性和非限制性序列。
43.图4a中提供了包括pap1和rtca的代表性ezra融合蛋白的图示。大肠杆菌poly(a)聚合酶(pap1)和酿酒酵母poly(a)聚合酶(pap1)的活性相似(图4b)。下面列出了ezra融合蛋白序列,其中his标签以斜体示出,pap1序列以粗体示出,接头为下标形式,并且rtca序列放大。seq id no:1用于含有大肠杆菌poly(a)聚合酶(pap1)的ezra融合蛋白,而seq id no:2用于酿酒酵母poly(a)聚合酶(pap1)。
[0044][0045][0046]
参考seq id no:1,氨基酸1-11对应于poly-his亲和标签,氨基酸12-466对应于大肠杆菌pap1,氨基酸467-484对应于xten接头,并且氨基酸485-822对应于rtca。
[0047]
在实施方式中,本公开的方法是使用连续多肽进行的,所述连续多肽包括与seq id no:1的包括氨基酸12-466的区段至少80%相同的氨基酸序列,以及与seq id no:1的包括氨基酸485至822的区段至少80%类似的氨基酸序列。
[0048][0049]
参考seq id no:2,氨基酸1-11对应于亲和标签,氨基酸12-578对应于酿酒酵母pap1,氨基酸579-596对应于xten接头,并且氨基酸597-934对应于rtca。
[0050]
在实施方式中,本公开的方法是使用连续多肽进行的,所述连续多肽包括与seq id no:2的包括氨基酸12-578的区段至少80%相同的氨基酸序列,以及与seq id no:2的包括氨基酸597至934的区段至少80%类似的氨基酸序列。
[0051]
以下针对编码序列使用与以上氨基酸序列中相同的样式示出了编码ezra融合蛋白的代表性和非限制性dna序列,seq id no:3用于大肠杆菌poly(a)聚合酶(pap1),并且seq id no:4用于酿酒酵母poly(a)聚合酶(pap1):
[0052]
[0053][0054]
如以上所讨论的,本公开的各方面使用受核糖体保护的片段(rfp)进行展示,但相同的方法可以适于其它rna片段,除了如果使用rfp,通常不需要去除核糖体rna。在此方面,在图1a中示意性地提供了用于对rfp进行测序的ezra-seq与常规“ribo-seq”方法的比较,所述rfp作为可以根据本公开测序的rna类型的非限制性实例。在图1b中示意性地提供了本公开的方法的非限制性描绘。如图1b所描绘的,本公开提供了对多个rna多核苷酸进行测序。所述方法一般包括:1)使多个rna多核苷酸与如下进一步描述的一种或多种酶和寡核苷酸接触,使得rna多核苷酸经受5'-腺苷酸化和3'-多腺苷酸化,以及2)将rna多核苷酸扩增成cdna,这促进了对rna多核苷酸的测序。
[0055]
使用本文所述的组合物和方法进行测序的rna的类型没有特别限制。在实施方式中,rna由原核生物、真核生物或病毒产生。在实施方式中,根据本公开进行测序的rna多核苷酸包括但不限于信使rna(mrna),如上所述。在实施方式中,可以对mrna进行片段化,以便对mrna的尚未具有poly-a尾的区段进行测序。如果需要,则也可以对任何被测序的rna进行片段化。可以被测序的rna还包括转移rna(trna)、核糖体rna(rrna)、转移信使rna(tmrna)、小核rna(snrna)、任何类型的反义rna、核酶、微rna(mirna)、小干扰rna(sirna)、短发夹rna(shrna)、rna病毒基因组、任何crispr rna,包括但不限于向导rna和反式激活crrna、双链rna(dsrna)和任何其它类型的rna,无论rna是否含有开放阅读框或具有已知或未知的功能。rna可以位于细胞的细胞核或细胞质中,或者rna可以从细胞中分泌,如在含有rna的被分泌的外泌体内。在实施方式中,根据本公开进行测序的rna多核苷酸包括一种或多种n6甲基腺苷。在实施方式中,对新生的、主动转录的rna进行测序。在实施方式中,本文所述的组合物和方法适于与任何现有或后来开发的rna测序方法一起使用。现有方法的非限制性实例包括rip-seq(rna免疫沉淀)、clip-seq(交联免疫沉淀)、chip-seq(染色质免疫沉淀)以及rna修饰的全基因组检测(例如,如上所述的m6a-seq)。
[0056]
在与对rpf进行测序有关的实施方式中,对rna的受核糖体保护以免受核酸酶消化的区段进行测序。因此,在实施方式中,对mrna的受核糖体保护的片段进行测序。在一个实施方式中,对来自样品的整套受核糖体保护的mrna rpf进行测序。在实施方式中,所述组合物和所述方法因此适用于例如核糖体谱(在本文中有时称为“ribo-seq”)。在实施方式中,本公开提供了一种rna测序方法,使得以亚密码子分辨率提供了在整个转录组中核糖体的位置和/或密度。在实施方式中,本公开使得相对于框外足迹,核糖体足迹的框内比率较高,其中核糖体“足迹”意指rna多核苷酸的受核糖体保护以免受酶促降解的区段。“框内”意味着rna中密码子的顺序在0框中是完整的,所述0框是从所测序的rna中的第一核苷酸开始。在实施方式中,如本文所述的ribo-seq是在没有基于蔗糖梯度的核糖体分离的情况下进行的。在实施方式中,可以使用全细胞裂解物进行ribo-seq以提供rna片段。
[0057]
在实施方式中,作为使用3'末端接头连接和环化的替代方法,当前提供的公开提供了在同一rna片段上进行3'末端poly(a)加尾和5'末端腺苷酸化。在实施方式中,5'-腺苷酸化的rna被称为apprna。在实施方式中,通过使用本公开的组合物和方法,rna 5'-腺苷酸化和3'-多腺苷酸化可以在单个反应容器中实现,而无需纯化步骤,如纯化rna多核苷酸或dna多核苷酸,包括但不限于寡核苷酸、引物等。
[0058]
本公开包括本文所描述的所有试剂以及试剂的组合。本公开包括如本文所述的组分的所有浓度,其代表性和非限制性实例包括缓冲液、ph值、核苷酸、rna和酶浓度、体积以及本文所述的任何其它定量值。本公开包括所有时间段、温度和值区间。在非限制性实例中,本公开的方法是在溶液中进行的,所述溶液的ph为大约6.0至7.9,包括端点,并且包括其间的到第一个小数点的所有数字。在实施方式中,本公开的方法是ph大约为或精确地为7.0的溶液中进行的。在实施方式中,本公开的溶液中的atp浓度大于1mm。在实施方式中,本公开的溶液中的atp浓度大约为或精确地为2mm。在实施方式中,本公开提供了包含大约50mm tris-hcl、250mm nacl、10mm mgcl2、1mm dtt和2mm atp的缓冲液。在实施方式中,多个rna多核苷酸的测序在不超过大约8小时的时间段内进行。在实施方式中,cdna文库在不超过大约2小时的时间段内产生。在实施方式中,cdna文库在大约30分钟的时间内产生。
oh或rgrg-oh)。在实施方式中,使用在3'末端处具有rsrs的5'衔接子。在实施方式中,本公开中使用的寡核苷酸在其3'末端处不具有rara。在实施方式中,本公开的组合物和方法中使用的寡核苷酸不包括如用于在常规cdna文库制备期间使用的5'预腺苷酸化。在实施方式中,5'衔接子使用rna连接酶连接到锚定的rna多核苷酸,所述rna连接酶的一个非限制性实例包括截短的t4 rna连接酶2(t4 rnl2tr)。
[0067]
在图5c中通过如上所述制备的连接的寡核苷酸和rna衔接子的电泳分离的照片,示出了使用如上所述的某些代表性寡核苷酸和apprna底物的连接效率的非限制性证明。在图5b中示出了用作衔接子的寡核苷酸的代表性和非限制性实例。在实施方式中,图5b所示的寡核苷酸提供平均核苷酸长度。因此,可以使用具有较短或较长长度的寡核苷酸。
[0068]
在特异性和非限制性实施方式中,使用5',5'-腺苷焦磷酰基帽结构,将rna片段转化为“接头”池,其可以通过截短的t4 rna连接酶2(t4 rnl2tr)连接到具有3'-oh的定制rna衔接子。设计了dna/rna杂合寡核苷酸,其在3'末端处以各种核糖核苷酸(rara、rcrc、rgrg和ruru)结束。发现截短的t4 rna连接酶2(t4 rnl2tr)有效地将rcrc-oh和rgrg-oh连接到5'-apprna(图5a)。rara的连接不良很重要,因为这避免了多腺苷酸化的apprna的自连接。考虑到illumina nextseq平台,并且不打算受到任何特定理论的约束,以rsrs结束的5'衔接子被认为最适用于后续扩增和测序。
[0069]
在实施方式中,所述组合物和方法包括直接发生在珠上的cdna合成(图1b)。在去除未连接的衔接子和t4 rnl2tr后,通过m-mulv逆转录酶实现cdna合成。
[0070]
pcr反应的最后一个步骤通过使用与illumina序列元件和条形码序列互补的普通引物进行。条形编码系统允许将不同的原始样品汇集到一个管中,从而大大降低了测序成本。在数据分析期间,提供的条形码信息允许快速分离原始样品。此策略最小化了测序期间引入的技术偏差。在清洁最终产物具有正确大小(约180bp)的情况下,样品准备好进行测序。
[0071]
鉴于前述,将认识到本文所述的组合物和方法的一种应用是在ribo-seq领域。在这方面,ribo-seq的标志是rpf的3-nt周期性,这归功于通过延长核糖体进行的相对精确的5'末端保护。因此,映射到阅读框0的读段的百分比或框内比率(ifr)通常用于反映ribo-seq的分辨率。文库构建的优化已将rpf的ifr从约50%提高到约75%(图1a,中间小图与左侧小图)(7)。然而,在本公开之前,大量的读段仍然处于框外,从而对理解核糖体动力学,尤其是翻译期间的阅读框保真度施加了极大的障碍。本公开解决了这些缺陷并且包括另外的改善。具体地,当前提供的ezra-seq方法大大减少了起始材料的量(约1ng rna),将整个文库加工时间从4天缩短到约4小时,并且提高了rpf的分辨率,其中平均ifr》90%。如根据典型的ribo-seq结果所预期的,ezra-seq在起始密码子处揭示出突出的峰,从而表示启动核糖体的暂停(图6a)。当考虑5'和3'末端两者时,其揭示了rpf的大小为29-nt。
[0072]
利用ezra-seq的超分辨率,可以在没有基于蔗糖梯度的核糖体分离的情况下实现核糖体谱,所述基于蔗糖梯度的核糖体分离已成为其广泛应用的瓶颈。为了测试这一点,收集了全细胞裂解物,将其用rna酶i消化,并按大小选择25-35nt rna物种。值得注意的是,获得了与使用蔗糖梯度核糖体分离的结果相似的结果(图6b)。
[0073]
线粒体有其自身的基因组和翻译装置。线粒体翻译不像细菌和真核生物细胞质翻译被很好地表征(11)。从相同的原始样品中,ezra-seq可以以极高的灵敏度捕获线粒体翻
译。用毒胡萝卜素(tg,0.1μm)或铼化合物(trip,25μm)处理小鼠胚胎成纤维细胞(mef)2小时,然后进行ezra-seq。与媒剂对照相比,trip处理显著降低了线粒体翻译(图6c)。
[0074]
如以上所讨论的,ezra-seq的应用不限于ribo-seq。ezra-seq可以很容易地转化为rna-seq,并行地用作ribo-seq对照。应用ezra-seq来监测细胞rna水平,并在tg或trip处理后发现了相当的线粒体转录物(图6c,中间小图)。
[0075]
鉴于ezra-seq的优越灵敏度,还应用ezra-seq来量化细胞核中染色质相关rna种类。对于许多转录物,ezra-seq发现了来自内含子和外显子两者的读段,这指示了未剪接的新生的rna种类(图6d)。有趣的是,氨基酸饥饿2小时导致转录减弱,如通过gapdh所例示的(图6d)。
[0076]
本公开还提供了制品,包括但不必限于试剂盒。在实施方式中,所述制品含有提供在一个或多个密封容器中的一种或多种酶和/或引物和/或缓冲液,所述密封容器的非限制性实例包括可密封的玻璃器皿或塑料小瓶。所述制品可以包括任何合适的包装材料,如用于容纳容器的盒子或包封或管。包装可以包括如在包装或容器本身上或在标牌上或在纸质插入物上的印刷材料。印刷材料可以提供对在本文中描述的用于确定任何rna的序列的测定中使用酶、引物和缓冲液的组合中的任一种的描述。所述制品/试剂盒中的任何试剂都可以以供用户重构的形式提供。例如,缓冲液、引物、酶等可以以用于用所述试剂制备溶液的干燥/功率/冻干形式提供。在实施方式中,基于确定rna序列的结果可以固定在有形的表达介质中,如保存在便携式存储器装置上或硬盘驱动器上的数字文件。例如,此信息可以存储在数字数据库中,以用于各种目的。
[0077]
以下是对说明本公开的实施方式的材料和方法的说明性和非限制性描述。其包括根据所述描述而显而易见的用于进行ribo-seq的某些特定步骤和组合物,但通过省略关于去除rrna的步骤和试剂,以也适应于对任何其它多个rna片段(例如,不是rpf的rna片段)进行测序。所述方法可以使用单独的或作为融合蛋白的组分的rna3'磷酸环化酶和酵母poly(a)聚合酶来进行。
[0078]
材料:
[0079]
试剂和缓冲液:
[0080]
1.rrna耗竭:
[0081]
1-1. 20
×
ssc:3m nacl和0.3m柠檬酸钠。
[0082]
1-2. 100%乙醇和70%乙醇。
[0083]
1-3. 3m乙酸钠(ph 5.2)。
[0084]
1-4.糖原(英杰公司(invitrogen),am9510)。
[0085]
2.对于腺苷酸化:
[0086]
2-1.t4多核苷酸激酶。
[0087]
2-2.rna 3'磷酸环化酶
[0088]
2-3.酵母poly(a)聚合酶
[0089]
2-4.atp溶液(10mm)(赛默飞世尔科技(thermo fisher scientific),pv3227)。
[0090]
2-5. 10
×
腺苷酸化缓冲液:700mm tris-hcl(ph 7.5)、100mm mgcl2和50mm dtt。
[0091]
2-6.superase_in rna酶抑制剂(英杰公司,am2696)。
[0092]
3.对于珠结合:
[0093]
3-1.链霉亲和素磁珠(新英格兰生物实验室(new england biolabs),s1420s)
[0094]
4.对于寡核苷酸连接:
[0095]
4-1.t4 rna连接酶2,截短的,k227q(新英格兰生物实验室,m0351l),具有peg8000 50%(w/v)和10
×
t4 rna连接酶缓冲液。
[0096]
5.对于cdna合成:
[0097]
5-1. 5
×
第一链缓冲液:250mm tris-hcl(ph 8.3)、375mm kcl和15mm mgcl2。
[0098]
5-2. 0.1m dtt。
[0099]
5-3.m-mulv逆转录酶mut5(自制)。
[0100]
5-4.rnaseout(英杰公司,10777-019)。
[0101]
6.对于pcr反应:
[0102]
6-1.phusion hf缓冲液(赛默飞世尔科技,f518l)
[0103]
6-2.dntp混合物(10mm)(赛默飞世尔科技,18427013)
[0104]
6-3.phusion dna聚合酶(自制)。
[0105]
7.对于大小选择:(仅用于ribo-seq)
[0106]
7-1.novex高密度tbe样品缓冲液(5x)(英杰公司,lc6678)。
[0107]
7-2.novex 5
×
tbe运行缓冲液(英杰公司,lc6675)。
[0108]
7-3.dna凝胶提取缓冲液:10mm tris(ph 8.0)、300mm nacl和1mm edta。
[0109]
7-4.sybr金核酸凝胶染色剂(英杰公司,s-11494)。
[0110]
设备:
[0111]
1.对于文库构建:
[0112]
1-1.dna lobind管1.5ml(艾本德(eppendorf),022431021)。
[0113]
1-2.加热块。
[0114]
1-3.冷冻式微型离心机。
[0115]
1-4.磁分离架。
[0116]
1-5.带盖的pcr管。
[0117]
2.对于凝胶运行和大小选择:
[0118]
2-1.电泳电源。
[0119]
2-2.迷你电池聚丙烯酰胺凝胶盒(赛默飞世尔科技,ei0001)。
[0120]
2-3.novex tbe凝胶,8%(英杰公司,ec6215box)。
[0121]
2-4.蓝光照明器。
[0122]
2-5.剃刀。
[0123]
2-6.spin-x离心管过滤器,0.22μm孔隙ca膜(西格玛(sigma),cls8160)。
[0124]
寡核苷酸和引物:
[0125]
1.rrna耗竭(de-rrna)寡核苷酸:(5'
→
3')
[0126]
/生物素-teg/agcgagcgaccaaaggaaccataactgattt(seq id no:7)
[0127]
/生物素-teg/cgaggttatctagagtcacca(seq id no:8)
[0128]
/生物素-teg/tcctagctgcggtatccaggcg(seq id no:9)
[0129]
/生物素-teg/agatagtcaagttcgaccgtcttctcagc(seq id no:10)
[0130]
/生物素-teg/gggcctcgatcagaaggacttgggccccccacga(seq id no:11)
[0131]
/生物素-teg/gcgagacgggccggtggtgcgccctcggcgg(seq id no:12)
[0132]
/生物素-teg/cgcttggcgccagaagcgagagcc(seq id no:13)
[0133]
/生物素-teg/agcgacgctcagacaggcgtagccccgggag(seq id no:14)
[0134]
/生物素-teg/ccggctatccgaggccaaccg(seq id no:15)
[0135]
/生物素-teg/tgatctgataaatgcacgcatccccc(seq id no:16)
[0136]
/生物素-teg/gtgtcgagggctgactttcaatagatcgcagcg(seq id no:17)
[0137]
/生物素-teg/agtagtggtatttcaccggcg(seq id no:18)
[0138]
/生物素-teg/tagaattaccacagttatc(seq id no:19)
[0139]
2.连接寡核苷酸混合物:(5'
→
3')
[0140]
acactctttccctacacgacgctcttccgatctrsrs(seq id no:6)
[0141]
rs:ribo-g或c
[0142]
3.生物素rt-引物:(5'
→
3')
[0143]
/5biosg/gtgactggagttgacgtgtgctcttccgatct
(25)
vn(seq id no:5)
[0144]
4.pcr引物:(5'
→
3')
[0145]
f:
[0146]
aatgatacggcgaccaccgagatctacactctttccctacacgacgctcttccgatct(seq id no:20)
[0147]
r:caagcagaagacggcatacgagat nnnnnn
[0148]
gtgactggagttcagacgtgtgctcttccgatct(seq id no:21)
[0149]
*nnnnnn:条形码(索引)
[0150]
程序:
[0151]
起始材料:在10μl无核酸酶的h2o中的rna片段
[0152]
1.rrna耗竭(任选的,定时:80分钟):
[0153]
1-1.制备rrna耗竭主混合物,如表1:
[0154]
表a
[0155][0156][0157]
1-2.向rna样品中添加10μl rrna耗竭主混合物(总计20μl)
[0158]
1-3.在80℃下温育30秒,然后缓慢冷却(约3℃/分钟)至37℃。
[0159]
1-4.在温育期间如下预洗涤链霉亲和素磁珠:
[0160]
1-4-1.将悬浮的链霉亲和素磁珠(20μl/样品)添加到新的1.5ml管中。
[0161]
1-4-2.将管放置到磁力架中1-2分钟。取出并丢弃上清液。
[0162]
1-4-3.向管中添加200μl 2
×
ssc,轻轻涡旋混合。用磁力架收集珠,然后取出并丢弃上清液。
[0163]
1-4-4.将链霉亲和素磁珠(20μl/样品)悬浮在2
×
ssc中。
[0164]
1-5.添加20μl链霉亲和素珠(预洗涤),通过若干次移液充分混合,在室温下保持10分钟。
[0165]
1-6.将管放置在磁体上2分钟,然后将上清液(总计40μl)转移到新的1.5ml管中,将珠丢弃。
[0166]
1-7.使rna样品沉淀:
[0167]
1-7-1.添加160μl无核酸酶的水、4μl糖原和40μl 3m乙酸钠,并且然后添加500μl 100%乙醇。
[0168]
1-7-2.在-20℃下沉淀至少30分钟。
[0169]
1-7-3.通过在20,000g、4℃下离心15分钟使rna沉淀。
[0170]
1-7-4.用500μl 70%乙醇洗涤rna沉淀,然后在20,000g、4℃下离心5分钟,用移液器从管中吸取所有液体并风干5分钟。
[0171]
1-8.将rna沉淀溶解在10μl无核酸酶的h2o中。
[0172]
2. 3'和5'-腺苷酸化(时间:30分钟)(描绘于图7),其中“插入物”是要测序的rna区段,并且对于ribo-seq,插入物是受核糖体保护的片段(rpf)。
[0173]
2-1.制备腺苷酸化主混合物,如表b所示:
[0174]
表b
[0175][0176]
2-2.向样品管中添加8.0μl腺苷酸化主混合物(总计20μl)。
[0177]
2-3.混合并在37℃下温育30分钟。
[0178]
3.珠结合(定时:30分钟);(描绘于图8)
[0179]
3-1.制备珠结合主混合物,如表c所述:
[0180]
表c
[0181][0182]
3-2.向样品管中添加20μl结合主混合物(总计40μl)。
[0183]
3-3.混合并在65℃下温育3分钟,然后冷却(约3℃/分钟)至室温。
[0184]
3-4.在温育期间如在步骤1-4处所述预洗涤链霉亲和素磁珠(10μl/样品)。
[0185]
3-5.向每个管中添加10μl预洗涤的链霉亲和素珠,通过若干次移液混合。
[0186]
3-6.在室温下温育10分钟。
[0187]
3-7.将管放置在磁体上1-2分钟以收集珠并去除上清液。
[0188]
3-8.将珠重新悬浮在200μl无核酸酶的h2o中。
[0189]
3-9.将管放置在磁体上1-2分钟并去除上清液。
[0190]
4.连接[定时:125分钟](描绘于图9,其中“读段1”和“读段2”是适于用于例如illumina测序的引物序列)
[0191]
4-1.通过若干次移液,用9μl无核酸酶的h2o和1μl 10μm连接寡核苷酸使珠重新悬浮。
[0192]
4-2.制备连接主混合物,如表d所述:
[0193]
表d
[0194][0195]
4-3.将10μl连接主混合物添加到样品管中(20μl总体积)。
[0196]
4-4.混合并在25℃(或室温)下温育120分钟,以800rpm连续振荡。
[0197]
4-5.将管放置在磁体上1-2分钟以收集珠并去除上清液。
[0198]
4-6.将珠重新悬浮在200μl无核酸酶的h2o中。
[0199]
4-7.将管放置在磁体上1-2分钟并去除所有上清液。
[0200]
5.cdna合成[定时:35分钟](描绘于图10)
[0201]
5-1.通过若干次移液,用13μl无核酸酶的h2o使珠重新悬浮。
[0202]
5-2.在70℃下温育2分钟,然后让样品冷却至室温。
[0203]
5-3.制备cdna合成主混合物,如表e所述:
[0204]
表e
[0205][0206]
5-4.将7μl cdna合成主混合物添加到样品管中(总共20μl)。
[0207]
5-5.充分混合并在50℃下温育30分钟,以800rpm连续振荡,然后在85℃下温育10分钟以终止。
[0208]
6.pcr扩增[定时:30分钟](描绘于图11,其中p5和p7是用于簇形成的illumina序列,并且i7索引为条形码序列)。
[0209]
6-1.制备pcr主混合物,如表f所述:
[0210]
表f
[0211][0212]
6-2.将步骤5中的2μl cdna和18μl pcr主混合物(总计20μl)添加到pcr管中。
[0213]
对于要汇集的每个样品,使用具有不同条形码的不同pcr引物。
[0214]
6-3.如下表g所述进行pcr反应:
[0215]
表g:
[0216][0217]
7.大小选择(定时:50分钟)(仅适用于ribo-seq):
[0218]
7-1.向每个pcr管中添加5μl novex
tm
高密度tbe样品缓冲液(5x)。
[0219]
7-2.将样品加载到8%novex
tm tbe凝胶上,并在180v下运行电泳45分钟。
[0220]
7-3.将凝胶在1
×
sybr金中染色3-5分钟。
[0221]
7-4.可视化凝胶并切除目标pcr产物。
[0222]
7-5.在4℃下在旋转过夜的情况下在400μl dna凝胶提取缓冲液中回收pcr产物。
[0223]
8.qc和测序(仅适用于ribo-seq):
[0224]
8-1.如步骤1-7处所述使dna沉淀。
[0225]
8-2.将rna沉淀溶解在15μl无核酸酶的h2o中。
[0226]
8-3.发送以进行qc和测序。
[0227]
以下参考文献列表并不表明所述参考文献中的任何参考文献对可专利性是必要的。
[0228]
1.x.adiconis等人,《自然方法(nat methods)》15,505(2018)。
[0229]
2.n.t.ingolia,s.ghaemmaghami,j.r.newman,j.s.weissman,《科学(science)》324,218(2009)。
[0230]
3.m.v.gerashchenko,v.n.gladyshev,《核酸研究(nucleic acids res)》42,e134(2014)。
[0231]
4.d.a.santos,l.shi,b.p.tu,j.s.weissman,《核酸研究》47,4974(2019)。
[0232]
5.n.hornstein等人,《基因组生物学(genome biol)》17,149(2016)。
[0233]
6.a.turchinovich等人,《rna生物学(rna biol)》11,817(2014)。
[0234]
7.n.t.ingolia,g.a.brar,s.rouskin,a.m.mcgeachy,j.s.weissman,《自然实验室指南(nat protoc)》7,1534(2012)。
[0235]
8.l.lama,j.cobo,d.buenaventura,k.ryan,《生物方法杂志(j biol methods)》6,(2019)。
[0236]
9.y.wang,s.k.silverman,《rna》12,1142(2006)。
[0237]
10.a.k.chakravarty,s.shuman,《生物化学杂志(j biol chem)》286,4117(2011)。
[0238]
11.b.e.christian,l.l.spremulli,《生物化学与生物物理学学报(biochim biophys acta)》1819,1035(2012)。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1