一种水稻谷氨酰胺氨基转移酶基因OsGAT2的基因工程应用
一种水稻谷氨酰胺氨基转移酶基因osgat2的基因工程应用
技术领域
1.本发明属于基因工程技术和植物学领域,涉及一种水稻谷氨酰胺氨基转运酶基因osgat2 及其在低氮肥施用时水稻提高产量及氮素利用效率中的应用。
背景技术:
2.氮素是植物中最重要的大量矿质元素之一,是植物体内构成叶绿素、核酸、酶等重要有机化合物的必需元素。禾本科植物缺氮时,会导致植株分蘖减少,茎秆细长,穗粒数少,籽粒不饱满,并且易出现早衰现象从而导致产量下降(s k de datta,1986)。在实际农业生产中,施加氮肥是使农作物高产优产的保障。但氮肥的过量施加,也造成了许多环境和生产上的问题。过度施用氮肥不仅会加速土壤酸化,破坏环境生态稳定。同时也会降低植株对氮素的利用效率,影响生育期发育,导致贪青晚熟,使植株的营养生长过长,生殖生长延后,扰乱了正常的生长时期,从而导致产量和氮素生理利用效率降低。因此,分析植株氮素吸收的途径和机制,研究相关基因的功能,对于实际生产中产量的提高和氮肥的施用具有重要作用。
3.氨基转移酶(ec 2.6.1.x)是一类普遍存在于动植物及微生物中的重要且多样的蛋白质,这类酶的生理功能是催化氨基供体转移到氨基受体化合物:一般来说,氨基酸作为氨基供体, 2-氧代酸作为氨基受体,反应产物则分别是相应的2-氧代酸和氨基酸(givan,1980)。这类酶在新陈代谢中起着核心作用,它们将氨基转入氨基酸,氨基糖,嘌呤和嘧啶核苷酸,辅酶和抗生素中。目前已知氨基转移酶家族成员至少有16个,所催化的反应在植物的氨基酸合成和代谢、次生代谢、碳氮同化、光呼吸、乙醛酸解毒以及抗胁迫等多种代谢途径中起着不可或缺的作用(liepman a h et al.,2004)。
4.水稻中氮素在水稻体内的运输形式主要为氨基酸和少量的硝态氮(no
3-)。其中,天冬酰胺(asparagine,asn)和谷氨酰胺(glutamate,gln)是有效的氮储存和转运化合物,也是氮素从根向地上运输的最重要的形式。在水稻的木质部和韧皮部汁液中,谷氨酰胺是运输氮的最丰富的氨基酸(p.j.lea et al.,2007)。水稻中谷氨酰胺氨基转移酶家族(gats)成员较多,且功能大多不同。其中报道较多的是天冬酰胺合成酶(asparagine synthetase)和谷氨酸合成酶(glutamate synthetase,gogat)。天冬酰胺合成酶(asparagine synthetase)的水稻编码基因是osasn1和osasn2(ohashi m et al.,2015),osasn1过表达可以促进水稻幼苗对氮的吸收和同化,增强幼苗对缺氮的耐受性,缺氮条件下产量增加(lee s et al.,2020)。谷氨酸合成酶(glutamate synthetase,gogat)在水稻中的编码基因中,osnadh-gogat2的突变将导致小穗数显著减少,产量和生物量显著降低(tamura w et al.,2011)。
技术实现要素:
5.本发明的目的是提供水稻谷氨酰胺氨基转移酶基因osgat2的工程应用,主要是在低氮肥条件下增加分蘖,提高产量及提高氮素养分利用效率的功能。
6.水稻谷氨酰胺氨基转移酶基因osgat2在提高水稻产量与氮素利用效率的应用;所述的水稻谷氨酰胺氨基转移酶基因osgat2基因组序列在msu数据库中的登录号为os06g48620,编码的蛋白序列在msu数据库中的登录号为os06g48620。
7.作为本发明的一种优选,所述的水稻谷氨酰胺氨基转移酶基因osgat2在提高水稻株高、分蘖数和/或氮素利用效率的应用。
8.作为本发明的进一步优选,敲除或突变水稻中的水稻谷氨酰胺氨基转移酶基因osgat2,能够提高水稻株高、分蘖数和/或氮素利用效率。
9.有益效果:
10.本发明所述的水稻谷氨酰胺氨基转移酶基因osgat2,在水稻根茎结合处表达。以日本晴水稻为背景,通过基因工程crispr-cas9技术对该基因进行敲除后,株高显著增高,氮素利用效率提高。在低氮肥施用土地中,突变体分蘖数和单株产量显著增加。对水稻增产和提高氮素生理利用效率具有重要意义。
附图说明
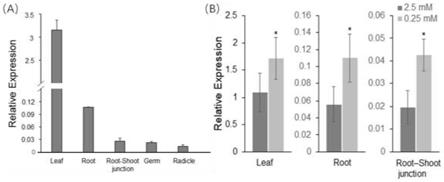
11.图1:osgat2基因在水稻中的表达量。
12.a:osgat2不同时期不同部位的表达情况;b:缺氮处理下的osgat2表达情况。数值代表平均值
±
sd(n=3),*表示p<0.05
13.图2:osgat2基因敲除突变体的突变位点鉴定。
14.基因结构中浅色方框代表外显子,深色方框代表5'-utr和3'-utr,黑色的线代表内含子。基序列中下划线表示spacer序列,删除线代表碱基缺失。
15.图3:缺氮条件下osgat2突变体成熟期表型。
16.a:osgat2突变体拍照;b:osgat2突变体株高;c:有效分蘖数;数值代表平均值
±
sd(n=5), *表示p<0.05,**表示p<0.01
17.图4:缺氮条件下osgat2突变体产量。
18.a:osgat2突变体生物量(dw);b:单株产量;数值代表平均值
±
sd(n=5),*表示p<0.05, **表示p<0.01
19.图5:osgat2突变体氮素利用效率。
20.a:osgat2突变体谷草比;b:谷/草氮浓度;c:农学生理利用效率;数值代表平均值
±
sd(n=5), **表示p<0.01,***表示p<0.001
21.上述附图中的gat2-1、gat2-2、gat2-3分别代表3个利用crispr/cas9技术构建osgat2突变体材料。
具体实施方式
22.实施例1:osgat2的表达研究
23.1)总rna的提取及cdna的合成
24.选用水稻品种日本晴。挑选饱满健康的成熟种子去壳后,用70%的酒精溶液浸泡30s进行初步消毒,再用含1%活性氯的次氯酸钠溶液消毒1h。在超净台内,用灭菌水清洗种子5-6 次后,将种子移到1/2ms培养基中培养,当水稻幼苗长至两叶一心后,选取长势一致的幼苗移到国际水稻所标准营养液中培养,同时进行不同氮浓度处理,分别用nh
4+
浓度为
0.25mm、 2.5mm的营养液处理。根据需要在不同时期不同部位取样,用液氮研磨成粉状。采用trizol (life technologies,carisbad,ca,usa)法提取叶片总rna,cdna用primescript tmrt试剂盒(takara,shiga,japan)反转录合成。
25.2)定量检测osgat2的表达
26.采用实时pcr法。使用qpcr绿色主混合试剂盒(vazyme,中国南京)。
27.osgat2基因特异性引物序列为:
28.上游引物f1:5
’‑
acaccagaggaagatgaatg-3’(seq id no.1)
29.下游引物r1:5
’‑
ggttccacgaatggtactta-3’(seq id no.2)
30.所使用的对照参考osactin基因特异性引物序列为:
31.osactin-f:5
’‑
caacacccctgctatgtacg-3’(seq id no.3)
32.osactin-r:5
’‑
catcaccagagtccaacacaa-3’(seq id no.4)
33.扩增反应在thermo lifetech abi quantstudiotm6 flex系统(life technologies,carisbad, ca,usa)进行。步骤如下:95℃持续30s;95℃持续5s;60℃持续30s,40个周期; 95℃持续15s;60℃持续60s;95℃持续15s。反应完成后,进行溶解曲线以检测引物特异性。pcr定量结果显示,osgat2在水稻萌发3天的胚芽、胚根及水培9天的叶、根及根茎结合处均有表达,在叶片中的相对表达量较高(图1a)。在缺氮条件下,各部位中osgat2 的表达均显著上调(图1b)。
34.实施例2:osgat2突变体植株的创制及鉴定
35.1)osgat2突变体植株的创制
36.利用crispr/cas9技术构建osgat2突变体材料。首先根据所述基因序列,利用 crispr-cas9系统设计含有的ngg或ccn结构靶序列。设计引物:
37.spacer 1-f:5
’‑
gccgatcgataatcttggggcca-3’(seq id no.5)
38.spacer 1-r:5
’‑
aaactggccccaagattatcgat-3’(seq id no.6)
39.spacer 2-f:5
’‑
ggcagattttgttttcgcaatttc-3’(seq id no.7)
40.spacer 2-r:5
’‑
aaacgaaattgcgaaaacaaaatc-3’(seq id no.8)
41.将这2对spacer通过pcr退火形成寡聚核苷酸双链,将双链连入入门载体 pylgrna-osu3/lacz和pylgrna-osu6a中,该载体能够通过驱动u3和u6启动子表达 snrna。将重组质粒在lr clonase ii enzyme的作用下进行gateway lr反应,转入含有cas9 的最终表达质粒pylcrispr/cas9-mh中。测序正确的表达载体通过电转法转入农杆菌 eha105中,通过水稻转基因技术获得osgat2 crispr/cas9突变体水稻材料。
42.2)osgat2突变体植株的鉴定
43.采集转基因t0代苗的叶片,提取水稻基因组dna。根据osgat2突变位点设计鉴定引物:
44.osgat2-f:5
’‑
ttgatcacaacaatggggat-3’(seq id no.9)
45.osgat2-r:5
’‑
gcctgtatttcaactgcaaa-3’(seq id no.10)
46.以基因组dna为模板,pcr扩增osgat2突变位点,产物测序后与野生型序列比对,挑选出3个有效突变的株系,命名为gat2-1,gat2-2,gat2-3。gat2-2在第1个外显子上缺失 9个碱基,gat2-3在第1个外显子上缺失12个碱基,gat2-6在第1个外显子上缺失9个碱基 (图2)。以osgat2突变体株系基因组dna为模板,扩增cas9载体序列,所有突变体株系基因组均
不存在cas9载体序列。cas9载体序列引物:
47.cas9-f:5
’‑
acaagggcagggatttcg-3’(seqidno.11)
48.cas9-r:5
’‑
actggtggatgagggtggc-3’(seqidno.12)
49.实施例3:生物量、单株产量、谷草比、植株氮浓度及氮素利用效率评价
50.将osgat2突变体株系与其原背景日本晴野生型材料以每穴一株,种植于南京农业大学白马科研教学基地,设置正常氮素供应处理(250kg/ha)的同时,设置田间缺氮处理(75kg/ha)。观察各个突变体株系和野生型整个生育期的生长情况,在成熟期统计株高、有效分蘖数等农艺性状。在成熟期收集各个材料的整株地上部样品,迅速放至105℃烘箱,杀青30min,置于70℃烘箱中烘干至衡重,分别称重各突变体株系和野生型的单株产量。测定植株氮含量及氮素利用效率采用凯氏定氮法,经过烘干、消煮后,用流动分析仪(型号为aa3,德国bran+luebbe公司)测定总氮。所计算的氮素利用效率为氮素生理利用效率(植株产量/植株吸收总氮量)。分析结果显示,在缺氮条件下,osgat2突变体的株高增加了约7cm(图3b),有效分蘖数增加了约2~3个(图3c),生物量相对于野生型提高了约34.5%(图4a),单株产量提高了约36.1%(图4b)。正常氮条件下,osgat2突变体谷草比相对于野生型增加了约43.4%(图5a),同时植株谷、草中氮含量均显著下降(图5b),草中平均氮含量约降低了18%,谷中平均氮含量约降低了13.6%。与野生型相比,osgat2突变体水稻对氮素的生理利用效率显著提升了约29.2%(图5c)。
51.参考文献
52.[1]dedattask.improvingnitrogenfertilizerefficiencyinlowlandriceintropicalasia[j].fertilizerresearch,1986,9(1-2):171-186.
[0053]
[2]givancv.aminotransferasesinhigherplants-sciencedirect[j].aminoacidsandderivatives,1980:329-357.
[0054]
[3]leapj,miflinbj.alternativeroutefornitrogenassimilationinhigherplants[j].nature,1974,251:614-616,420(6913):316-320.
[0055]
[4]lees,parkj,leej,etal.osasn1overexpressioninriceincreasesgrainproteincontentandyieldundernitrogen-limitingconditions[j].plantcellphysiol,2020,61(7):1309-1320.
[0056]
[5]liepmanah,olsenlj.taylor&francisonline::genomicanalysisofaminotransferasesinarabidopsisthaliana-criticalreviewsinplantsciences-volume23,issue1[j].criticalreviewsinplantsciences,2004.
[0057]
[6]ohashim,ishiyamak,kojimas,etal.asparaginesynthetase1,butnotasparaginesynthetase2,isresponsibleforthebiosynthesisofasparaginefollowingthesupplyofammoniumtoriceroots[j].plantcellphysiol,2015,56(4):769-778.
[0058]
[7]tamuraw,kojimas,toyokawaa,etal.disruptionofanovelnadh-glutamatesynthase2genecausedmarkedreductioninspikeletnumberofrice[j].frontiersinplantscience,2011,2:57.
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1