用于预测胰腺癌预后的评估基因集、试剂盒、应用及系统的制作方法
1.本发明属于肿瘤分子生物学技术领域,具体涉及一种用于预测胰腺癌预后的评估基因集,本发明同时还提供了一种用于预测胰腺癌患者预后的试剂盒、检测3种线粒体自噬相关基因表达水平的试剂盒在制备胰腺癌患者总体生存率诊断或辅助诊断产品中的应用和用于预测胰腺癌患者预后的系统。
背景技术:
2.胰腺癌(pc)是一种恶性的消化道恶性肿瘤,占癌症相关死亡的4.7%,据报道,到2025年可能成为癌症死亡的第三大原因(sunget al.,2021);高异质性、早期诊断困难、疗效有限是pc预后不良的原因,现有研究表明,基于基因检测的靶向治疗可能为克服pc治疗缺陷提供了有效的治疗选择,然而,由于pc患者复杂的分子分型和肿瘤异质性,大多数基因表达变异的特征仍然很差,抑制了它们的临床转化。因此,迫切需要一种全新的生物标记物用于预后分层和治疗选择,以改善pc患者的预后。
3.线粒体自噬是一种线粒体质量控制机制,主要负责肿瘤细胞内的代谢重塑和调节肿瘤细胞之间的相互作用。线粒体自噬通过缺氧和代谢应激的功能激活,通过去除多余的线粒体和减少氧气消耗来提高肿瘤细胞的生存。线粒体自噬缺失会对胰腺癌干细胞的干细胞性产生负面影响,损害其致瘤能力。
4.越来越多的证据表明,肿瘤的发展和进展在很大程度上取决于其所处的复杂微环境,包括肿瘤细胞及其周围的免疫细胞、成纤维细胞和内皮细胞。现有研究认为,在传统tnm分期系统的基础上,可以将免疫评分作为癌症分期系统的一部分,进一步提高对总体预后的评估。现有针对结肠癌患者的研究中建立了基于免疫评分的癌症分类,结果表明适应性免疫细胞浸润的预后价值优于当前的经典肿瘤浸润(tnm期)。因此,免疫评分系统的引入将进一步有助于为pc分层和预后预测提供新的见解。
技术实现要素:
5.本发明基于tcga数据库、icgc数据库和geo数据库中获取胰腺癌病例的基因表达谱及相应的临床信息,将tcga队列中的患者分为两组并筛选了两组之间的差异表达基因,根据基因表达值得到预测胰腺癌预后的评估基因集。
6.本发明的第一目的是提供一种用于预测胰腺癌患者预后的评估基因集,其中,评估基因集为线粒体自噬相关基因集,其包括3种线粒体自噬相关基因,3种线粒体自噬相关基因为prkn、src、vdac1。
7.本发明的第二目的是提供一种用于预测胰腺癌患者预后的试剂盒,其包括检测前述评估基因集中3中线粒体自噬相关基因表达水平的试剂。
8.本发明的第三目的是提供一种检测3种线粒体自噬相关基因表达水平的试剂盒在制备胰腺癌患者总体生存率诊断或辅助诊断产品中的应用,其中,试剂盒为前述的试剂盒。
9.本发明的第四目的是提供一种用于预测胰腺癌患者预后的系统,其包括以下模
块:
10.1)数据收集模块:收集患者的样本,测定其基因表达值,其中基因为权利要求1所述的评估基因集中的3种线粒体自噬相关基因;
11.2)模型计算模块:计算胰腺癌患者的3种线粒体自噬相关基因的总表达值,即风险评分,风险评分的计算公式如下:
12.风险评分=(-0.929609894*prkn的表达水平)+(0.006652335*src的表达水平)+(0.001093884*vdac1的表达水平);
13.3)输出预测模块:根据胰腺癌患者的风险评分数据来预测患者的预后情况,患者的风险评分越低,则预后情况越好。
14.作为本发明的进一步实施方案,将患者的风险评分与设定值进行比较,如果高于设定值,则预后风险为高风险,如果低于设定值,则预后风险为低风险。
15.作为本发明的进一步实施方案,设定值为1.070577。
16.本发明具备以下有益效果:
17.本发明提供了一种用于胰腺癌预后预测的评估基因集及相应的试剂盒,能更可靠地应用到临床实践中,相较于现有技术,可以更准确地预测胰腺癌患者预后,为医疗决策提供科学依据。
18.下面结合附图对本发明作进一步的详细说明。
附图说明
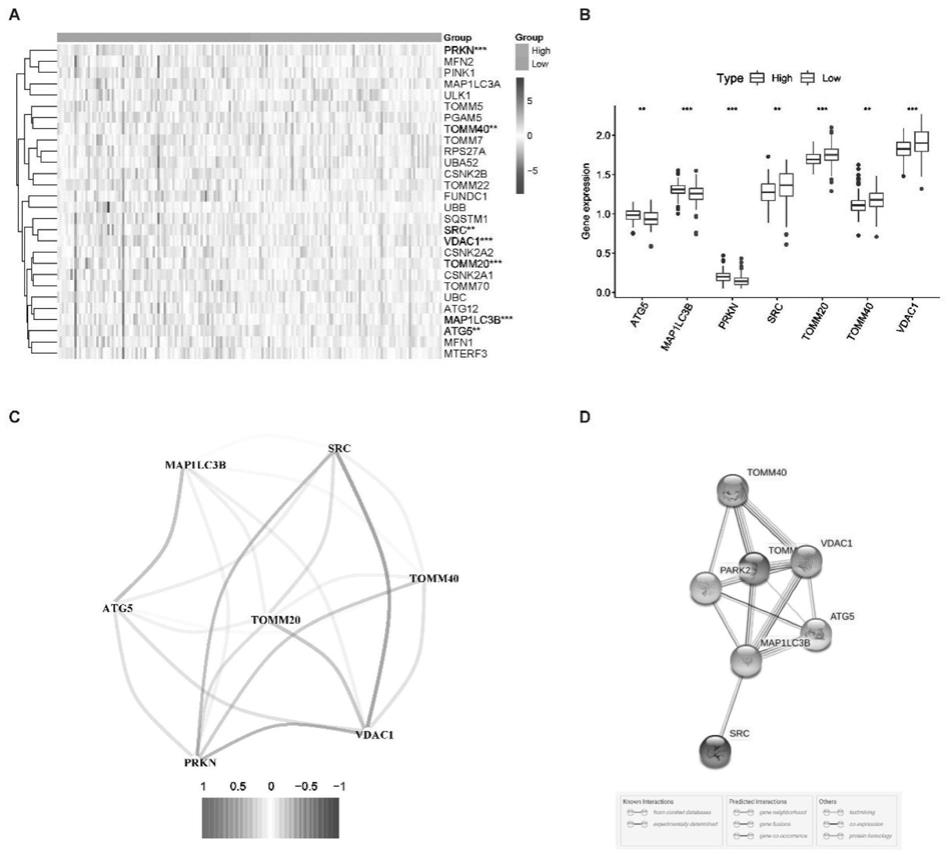
19.图1是tcga队列中两个免疫评分亚群之间的线粒体自噬相关差异表达基因(degs);
20.其中1a是tcga队列中高免疫评分组和低免疫评分组胰腺癌患者中29个线粒体相关基因表达谱的热图,*表示p《0.05;**代表p《0.01;***代表p《0.001;1b是degs表达的箱线图;1c是degs之间的相关性网络,红色代表正相关,蓝色代表负相关;1d是degs之间的蛋白-蛋白相互作用(ppi)网络;
21.图2基于线粒体自噬相关基因的肿瘤分类;
22.其中2a是累积分布函数曲线;2b是共识聚类的delta面积曲线;2c是共识聚类矩阵(k=3);2d是三个亚组的kaplan-meier生存分析;2e是三种亚型线粒体自噬相关基因表达的热图;
23.图3是tcga队列中线粒体自噬相关基因特征的建立;
24.其中3a是单变量cox回归分析后预后相关基因的森林图;3b是最小绝对收缩和选择算子(lasso)变量筛选过程;3c是基于最小lambda值的候选基因交叉验证;3d是使用多变量cox回归分析的独立预后相关基因的森林图;3e是tcga队列中pc样本的风险评分分布;3f是根据风险评分,每个pc样本的生存时间和状态;3g是3基因特征的主成分分析(pca);3h是两个危险亚组之间的生存分析;3i是3基因特征的roc曲线;
25.图4是icgc、gse28735、gse62452队列中线粒体自噬相关基因特征的验证;
26.其中4a、4f、4j是pc样本的风险评分分布;4b、4g、4k是基于风险评分的pc样本生存时间和状态、4c、4h、4l是两个危险亚组之间的生存分析;4e、4i、4m是3基因特征的roc曲线;
27.图5是3个特征基因分析;
28.其中5a是tcga队列中3个特征基因表达水平及风险评分与临床特征相关性的heatmap;5b、5c、5d是tcga队列中3个特征基因的生存分析;5e、5f、5g是胰腺正常组织和胰腺癌组织中3个特征基因的免疫组化染色情况;5h是3个特征基因和6个免疫细胞之间的关系,*表示p《0.05;**代表p《0.01;***代表p《0.001;5i是基于timer数据库发现,prkn与肿瘤纯度负相关,并与6种免疫浸润显著正相关;
29.图6是独立预后因素的鉴定和列线图的构建;
30.其中6a是tcga队列风险评分及临床特征的单因素cox回归分析和多因素cox回归分析;6b是tcga队列的列线图预测pc患者1、3、5年生存率;6c、6d、6e是列线图的校准图;6f、6g、6h是3个独立预后因素与列线图的roc曲线分析;
31.图7是tcga队列中3基因特征的功能分析;
32.其中7a是高、低风险组degs的go富集分析,bp:生物过程,mf:分子功能,cc:细胞成分;7b是高危组与低危组degs的kegg富集分析;7c是在两个风险亚组之间的16种免疫细胞富集分数的差异;7d是两个风险亚组之间13种免疫通路富集分数的差异,*表示p《0.05;**代表p《0.01;***代表p《0.001;
33.图8是tcga队列中3基因签名的免疫状况和免疫治疗预测;
34.其中8a-8f是风险评分与6种免疫浸润细胞(b细胞、cd4+t细胞、cd8+t细胞、中性粒细胞、巨噬细胞、树突状细胞)的相关性;8g是22种免疫细胞在pc样本中的比例;8h是22种免疫细胞在高危和低风险亚组之间的相对比例;8i是两个危险亚群之间22种免疫细胞组成的差异,*表示p《0.05;**代表p《0.01;***代表p《0.001;8j是2个危险亚群中8个免疫检查点基因的表达水平;
35.图9是3基因特征的突变情况;
36.其中9a是低危患者突变状态瀑布图;9b是高危患者突变状态瀑布图;
37.图10是3基因特征的化疗反应预测;
38.其中10a-10i分别是阿西替尼、喜树碱、托泊苷、尼罗替尼、帕佐帕尼、舒尼替尼、替西莫司、厄洛替尼、紫杉醇的ic50值。
具体实施方式
39.为了能够更清楚地理解本发明的上述目的、特征和优点,下面结合附图和具体实施方式对本发明进行进一步的详细描述。需要说明的是,在不冲突的情况下,本技术的实施例及实施例中的特征可以相互组合。
40.在下面的描述中阐述了很多具体细节以便于充分理解本发明,但是,本发明还可以采用其他不同于在此描述的其他方式来实施,因此,本发明的保护范围并不限于下面公开的具体实施例的限制。
41.实施例1
42.数据收集
43.pc的转录组测序数据[fragments per kilobase of transcript per million mapped reads(fpkm)标准化]、突变数据和相应的临床信息均来自tcga数据库(https://tcga-data.nci.nih.gov/tcga/),以此作为训练集。
[0044]
验证队列从icgc数据库(https://dcc.icgc.org)和geo数据库(https://
www.ncbi.nlm.nih.gov/geo)下载;共收集了176份tcga-胰腺癌(tcga-paad)样本、79份icgc-胰腺癌-澳大利亚(icgc-paca-au)样本、41份gse28735(zhang et al.,2013a)样本和62份gse62452(yang et al.,2016)样本进行分析。
[0045]
tcga的转录本测序数据、icgc的读计数数据以及gse28735、gse62452的序列矩阵数据,采用r包“limma”进行“log2(data+1)”处理(ritchie et al.,2015)。此外,pc患者的免疫评分来自estimate数据库(https://bioinformatics.mdanderson.org/estimate/disease.html)。从pathway unification数据库(https://pathcards.genecards.org/)获得29个线粒体自噬相关基因(mrgs),分别为:atg12、atg5、csnk2a1、csnk2a2、csnk2b、fundc1、map1lc3a、map1lc3b、mfn1、mfn2、mterf3、pgam5、pink1、prkn、rps27a、sqstm1、src、tomm20、tomm22、tomm40、tomm5、tomm6、tomm7、tomm70、uba52、ubb、ubc、ulk1、vdac1。
[0046]
差异表达基因的筛选
[0047]
根据中位免疫评分,将来自tcga队列的pc样本分为高免疫评分组和低免疫评分组。接下来,从tcga队列的表达谱数据中提取29个mrgs表达谱,并使用r包“pheatmap”生成热图。
[0048]
在两个免疫评分组间筛选显著差异的基因(degs),错误发现率阈值(fdr)小于0.05;使用r包“reshape2”和“ggpubr”绘制degs表达的箱线图;用r包“igraph”生成degs的相关性网络,用交互作用基因检索工具(string)数据库(http://string-db.org)构建degs的蛋白质-蛋白质相互作用网络(protein-protein interaction,ppi)。
[0049]
具体展开的,根据中位免疫评分,将pc样本分为两个免疫评分亚组。7个mrgs在高、低免疫评分组间有差异表达(均为fdr《0.05,图1a-1b),其中atg5、map1lc3b、prkn的基因表达水平下调,其他4个基因(src、tomm20、tomm40、vdac1)的表达水平在高免疫评分组上调。
[0050]
degs之间的相关性网络如图1c所示,不同颜色代表不同的相关系数。接着发明人还进一步构建了ppi网络来识别这些线粒体自噬相关degs之间的相互作用,ppi网络分析结果表明tomm20是一个枢纽基因(图1d)。
[0051]
基于mrgs的肿瘤分类
[0052]
为了进一步探讨29个mrgs与pc患者分层之间的关系,使用r包“consensusclusterplus”进行一致性分析;采用r软件包“survival”和“survminer”对不同亚型进行kaplan-meier生存分析,应用r包“pheatmap”绘制热图。
[0053]
具体展开的,通过一致性聚类分析来评估mrgs对pc样本的影响。
[0054]
如图2a-b所示,根据累积分布函数(cdf)曲线和cdf曲线下面积的相对变化结果,最佳聚类数被确定为k=3,即最大的聚类数在保持聚类内最大一致性的情况下,引起cdf曲线下面积增量变化最小。图2c的结果显示,pc样本可以根据线粒体自噬相关基因分为三组;tcga队列pc患者被分为三组(n=124、48、6),三种亚型之间的生存期差异有统计学意义(p=0.023,图2d),其中三个聚类之间的基因表达谱如图2e所示。
[0055]
线粒体自噬相关基因特征的构建与验证
[0056]
使用单变量cox回归分析筛选生存相关基因,然后应用多因素cox回归分析筛选与独立的生存相关基因。
[0057]
为了进一步缩小候选基因的范围,发明人还采用最小绝对收缩选择算子(least absolute contraction and selection operator,lasso)算法来防止模型过拟合;采用
lasso回归系数计算风险评分。
[0058]
具体展开的,经单因素cox回归分析,从tcga队列中确定3个mrgs为预后相关基因。src和vdac1是危险基因(hr》1),而prkn是一个保护性基因(hr《1)(图3);lasso回归进一步缩小了候选基因的范围,最终以λ的最佳值0.02895928为基础建立了3个基因的预后特征(图3b-3c)。
[0059]
多因素cox回归进一步筛选了预后相关基因,确定了prkn是一个独立的预后基因(p=0.0192,图3d)。
[0060]
风险评分的的计算公式如下:
[0061]
风险评分=(-0.929609894*prkn的表达水平)+(0.006652335*src的表达水平)+(0.001093884*vdac1的表达水平);
[0062]
根据中位风险评分,将pc样本分为高、低风险组,kaplan-meier分析两危险组生存差异,和采用r中“stats”软件包的“prcomp”函数进行主成分分析(pca),和采用r软件包“timeroc”进行roc曲线下面积(auc)分析,以评估预后模型的性能。
[0063]
具体展开的,根据中位风险评分,pc样本被分为高风险组(n=88)和低风险组(n=88),风险评分越高的患者死亡风险越大,生存时间越短(图3e-f)。pca显示出高、低风险组之间的明显区别(图3g)。生存曲线显示高危患者的生存期远低于低危患者(p=0.006267,图3h)。tcga队列的3年和5年生存auc值分别为0.785和0.782(图3i)。
[0064]
在验证队列(icgc-paca-au、gse28735和gse62452)中,使用相同的公式计算风险评分,并使用上述方法验证风险特征的性能。
[0065]
其中外部验证集显示上述3种基因特征具有较高的预测精度,具体展开的,在三个验证队列中,风险评分分布和他们的生存状态与tcga队列的结果一致(图4b、4f、4g、4j、4k),高危患者生存时间比低风险病人更低(p=0.02327,icgc-paca-au,图4c;gse28735,p=0.04975,图4h;gse62452,p=0.0062,图4l)。
[0066]
同时,三个验证队列的auc值显示了对3年生存率的良好预测能力和稳定性(icgc-paca-au,auc=0.818,图4e;gse28735,auc=0.821,图4i;gse62452,auc=0.822,图4m),表明本实施例中3种基因特征能够可靠地预测pc患者的生存情况。
[0067]
进一步的,本实施例还进行有如下过程:
[0068]
3种特征基因与生存和免疫活性相关
[0069]
为了评估3种特征基因的预后预测能力和免疫相关性,发明人评估了它们在tcga队列和人类免疫组化组织中的表达水平,并对这些基因进行了生存和免疫相关性分析。
[0070]
具体展开的,图5a显示了tcga队列中3种特征基因的表达以及风险评分与临床特征之间的相关性(年龄、性别、酒精、分级、分期、tnm和状态)。prkn在高危组的表达水平低于低危组,而src和vdac1的表达水平则相反,3种特征基因的生存分析也显示了它们与预后的紧密联系(图5b-d)。
[0071]
此外,在hpa数据库的肿瘤样本和正常样本中验证了特征基因的表达;如图5e-5g所示,prkn在pc组织中低表达,src和vdac1在pc组织中高表达,与基因表达分析结果一致。接下来进一步分析了这3种特征基因与免疫活性的关系,发现prkn与6种免疫细胞(b细胞、巨噬细胞、髓系树突状细胞、中性粒细胞、cd4+t细胞、cd8+t细胞)密切相关(p《0.05,图5h);从timer数据库中提取的结果显示,prkn与6个免疫浸润细胞呈正相关(图5i)。
[0072]
预后模型与临床特征的相关性
[0073]
为了评估重要的临床预后因素和我们预后模型的临床适用性,发明人进行了单因素和多因素分析以确定独立的预后因素,并建立了列线图。
[0074]
从tcga队列中提取pc患者的年龄、性别、酒精、分级、分期、t、n等临床信息,经单因素和多因素cox回归分析,年龄、n分期和风险评分被确定为独立的预后因素(p=0.0341、0.0103和0.0375,图6a);根据独立预后因素构建列线图预测pc患者1年、3年、5年生存率(图6b);校准图显示预测结果和实际结果之间有很好的一致性(图6c-6e)。列线图预测1年、3年、5年生存率的auc分别为0.647、0.870、1.00(图6f-6h)。
[0075]
3基因特征的功能富集
[0076]
为了进一步探索这3个基因特征的功能富集,发明人对这3中基因特征进行了go富集、kegg分析和免疫富集评分。在tcga队列中,两组共筛选出2128个degs。go富集结果显示,这些degs主要富集于膜电位调控和信号释放(图7a)。kegg分析显示,degs在神经活性配体-受体相互作用和细胞因子-细胞因子受体相互作用的相关通路显著富集(图7b)。对预后模型的进一步功能分析表明,3基因特征与免疫活性显著相关。
[0077]
高危组多数免疫细胞浸润水平明显低于低危组(14/16,87.5%,所有p《0.05,图7c);与低危组相比,高危组的ccr、checkpoint、溶细胞活性、hla、促炎、t细胞共抑制、t细胞共刺激、ii型ifn反应等免疫通路富集评分显著降低(所有p《0.05,图7d)。
[0078]
3基因特征与免疫微环境和免疫检查点相关
[0079]
为进一步探讨3基因标记与免疫微环境的差异,采用spearman相关性分析风险评分与免疫评分。如图8a-8f所示,风险评分与6个免疫浸润细胞(b细胞、cd4+t细胞、cd8+t细胞、中性粒细胞、巨噬细胞、骨髓树突状细胞)呈负相关(均为p《0.05),来自tcga队列的pc患者中22个免疫细胞的比例见图8g,m0巨噬细胞、m2巨噬细胞、静息记忆cd4+t细胞所占比例最高。
[0080]
22个肿瘤免疫细胞在高危和低危患者之间的分布如图8h所示,具体而言,大多数免疫细胞浸润细胞(18/22,81.8%)在两个危险亚组之间有显著差异(所有p《0.05),表明3基因特征与免疫微环境有很强的相关性(图8i)。
[0081]
此外,发明人还研究了预后模型与免疫检查点基因表达值之间的相关性,这可以作为预测免疫应答的一个指标;如图8j所示,在tcga队列的高危患者中,除siglec15基因外,其余7个免疫检查点基因均下调(所有p《0.05)。
[0082]
3基因特征的突变情况
[0083]
发明人比较了tcga队列中高风险和低风险患者的突变情况,如图9a-9b所示,高危患者的突变率高于低危患者(97.67%vs.75.9%);在基因突变频率方面,kras、肿瘤蛋白p53(tp53)和smad家族成员4(smad4)是高危患者比低危患者改变最多的基因(分别为90%比61%、73%比53%和30%比17%)。此外,错义突变是pc患者最常见的突变类型。
[0084]
3基因特征可以预测化疗药物敏感性
[0085]
化疗药物一直是pc治疗的主要手段,不良预后与化疗耐药性有关;在此,发明人进一步预测了两个危险亚组对常用化疗药物的化疗反应。
[0086]
如图10a-10g所示,高危患者对八种化疗药物(阿西替尼、喜树碱、依泊苷、尼罗替尼、帕佐帕尼、舒尼替尼、替西莫司)的ic50值均高于低危组患者,提示低危患者可从这些化
疗药物中获益。此外,发明人还发现高危患者对厄洛替尼和紫杉醇更敏感(p=0.038,p=0.00011,图10h-10i)。
[0087]
虽然本发明以较佳实施例揭露如上,但并非用以限定本发明实施的范围。任何本领域的普通技术人员,在不脱离本发明的发明范围内,当可作些许的改进,即凡是依照本发明所做的同等改进,应为本发明的范围所涵盖。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1