检测SCN4A和SCN7AmRNA表达量的产品在制备肝癌预后预测产品中的应用
mrna在肿瘤组织中低表达,其中scn4a和scn7a的结果与我们分析组织芯片的结果一致。timer数据库的泛肿瘤分析显示,scn7a在肿瘤中的表达稳定降低。scn4a和scn7a均与肿瘤分级、淋巴结转移状态、组织学亚型、患者种族、个体肿瘤分期和tp53突变状态有不同程度的相关性。kaplan-meier plotter预后分析结果显示,scn4a mrna高表达与较长的总生存期(os)、疾病特异性生存期(dss)和无进展生存期(pfs)相关,scn7a mrna高表达与较长的os相关;在亚洲人,更高的scn4a与更长的os和dss相关,scn7a高表达与更长的os、无复发生存(rfs)、dss和pfs相关。cbioportal的数据分析显示,scn7a突变与rfs和pfs相关。进一步使用免疫组化检测scn4a和scn7a蛋白在肝细胞癌中的表达并结合生存资料做了生存分析。单变量生存分析显示,scn7a蛋白高表达与更长的os和rfs显著相关。多因素分析显示scn7a为os和rfs独立的预后因素。综上所述,scn4a和scn7a在hcc中表达存在显著而稳定的变化。其中scn7a在肝癌中的表达有更好的预后价值。
12.因此本发明要求保护以下内容:
13.检测scn4a mrna和/或scn7a mrna表达量的产品在制备肝癌预后预测产品中的应用。
14.其中,scn4agene id 6329,scn7a gene id:6332。
15.优选地,所述预后为肝癌的总生存期(os)、无复发生存期(rfs)、疾病特异性生存期(dss)和无进展生存期(pfs)。
16.更优选地,scn4a mrna高表达的肝癌患者的总生存期(os)、疾病特异性生存期(dss)和无进展生存期(pfs)中的一项或几项较scn4a mrna低表达的肝癌患者长。
17.优选地,scn7a mrna高表达的肝癌患者的总生存期(os)较scn7a mrna低表达的肝癌患者长。
18.优选地,肝癌患者为亚洲裔,scn4a mrna高表达的肝癌患者的总生存期(os)和疾病特异性生存期(dss)的一项或几项较scn4a mrna低表达的肝癌患者长。
19.优选地,肝癌患者为亚洲裔,scn7a mrna高表达的肝癌患者的总生存期(os)、无复发生存期(rfs)、疾病特异性生存期(dss)和无进展生存期(pfs)的一项或几项较scn7a mrna低表达的肝癌患者长。
20.作为一个具体的实施例,所述检测scn4a mrna和/或scn7a mrna表达量的产品为芯片。
21.以及,检测scn7a突变的产品在制备肝癌预后预测产品中的应用。
22.优选地,所述预后为无复发生存期(rfs)和无进展生存期(pfs)中的一项或几项,scn7a突变肝癌患者的无复发生存期(rfs)和无进展生存期(pfs)中的一项或几项较scn7a无突变肝癌患者的长。。
23.以及,检测scn7a蛋白表达量的产品在制备肝癌预后预测产品中的应用。
24.优选地,所述预后为肝癌的总生存期(os)和无复发生存期(rfs)中的一项或几项,scn7a蛋白表达越高总生存期(os)和无复发生存期(rfs)中的一项或几项越长,表达越高生存率越好。
25.作为一个具体的实施例,所述检测scn7a 蛋白表达量的产品为scn7a 蛋白免疫组化检测试剂。
26.优选地,所述肝癌为肝细胞癌。
27.与现有技术相比,本发明具有以下有益效果:
28.发明人发现钠通道编码基因家族(scn family)可以作为肝细胞癌的预后分子标志物,尤其是scn4a和scn7a在肝癌的预后预测中有很好的效果;其中的scn7a在亚洲hcc患者中有更好的预后价值。并且scn7a在mrna和蛋白层面表达水平越高,肝癌患者预后越好,而且scn7a的dna突变也可以区分肝癌患者的不同预后。
附图说明
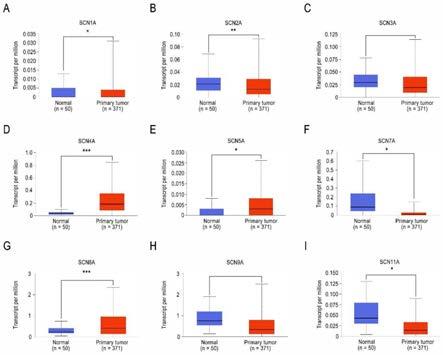
29.图1为scn家族成员在hcc中的mrna表达;(a)scn1a mrna在肿瘤组织和正常组织中的表达;(b)scn2a mrna在肿瘤组织和正常组织中的表达;(c)scn3a mrna在肿瘤组织和正常组织中的表达;(d)scn4a mrna在肿瘤组织和正常组织中的表达;(e)scn5a mrna在肿瘤组织和正常组织中的表达;(f)scn7a mrna在肿瘤组织和正常组织中的表达;(g)scn8a mrna在肿瘤组织和正常组织中的表达;(h)scn9a mrna在肿瘤组织和正常组织中的表达;(i)scn11a mrna在肿瘤组织和正常组织中的表达;*p《0.05,**p《0.01,***p《0.001。
30.图2为肝细胞癌组织与相配对的癌旁组织的组织芯片分析;(a)分级聚类热图显示了在hcc组织和邻近非肿瘤组织中表达显著变化的基因;(b)火山图中显示了癌组织和邻近非肿瘤组织中的所有基因。
31.图3为scn4a和scn7a的泛肿瘤分析以及scn4a和scn7a与肝癌患者临床病理因素的关系;(a)scn4a的泛肿瘤分析;(b)scn7a的泛肿瘤分析。(c)scn4a和scn7a与肿瘤分级的关系;(d)scn4a/7a与淋巴结转移的关系;(e)scn4a/7a与肿瘤组织类型的关系。(f)the relationship scn4a/7a与病人种族的关系;(g)scn4a/7a与肿瘤等级的关系。(h)scn4a/7a与tp53突变的关系。*p《0.05,**p《0.01,***p《0.001。
32.图4为kaplan
–
meier plotter分析hcc患者scn4a/7a mrna的预后意义;(a)scn4a mrna高表达与较长的os(p《0.001)、dss(p=0.007)和pfs(p=0.012)相关;(b)scn4a mrna高表达与较长的os(p=0.018)相关。
33.图5为kaplan
–
meier plotter分析亚洲hcc患者scn4a和scn7a mrna的预后意义;(a)scn4a mrna高表达与更长的os相关(p《0.001)和dss(p=0.013);(b)scn7a mrna高表达与较长的os(p=0.004)、rfs(p《0.001)、dss(p=0.001)和pfs(p《0.001)。
34.图6为cbioportal对scn4a和scn7a突变在hcc中的生存分析;(a)scn4a突变与os(p》0.05),rfs(p》0.05),dss(p》0.05)和pfs(p》0.05)无关;(b)scn7a突变与rfs(p=0.034)和pfs(p《0.001)相关。
35.图7为免疫组化检测scn4a和scn7a蛋白在hcc中的表达及scn4a和scn7a蛋白表达与患者生存的预后分析;(a)scn4a在hcc组织中的表达;(b)scn4a蛋白表达与rfs(p》0.05)无关;(c)scn4a蛋白表达与os(p》0.05)无关;(d)scn7a在hcc组织中的表达;(e)scn7a蛋白表达高与较长的rfs相关(p=0.003);(f)scn7a蛋白表达高与较长的os相关(p=0.003);(g)hcc rfs和os的多因素生存分析。
具体实施方式
36.下面结合说明书附图及具体实施例对本发明作出进一步地详细阐述,所述实施例只用于解释本发明,并非用于限定本发明的范围。下述实施例中所使用的试验方法如无特
殊说明,均为常规方法;所使用的材料、试剂等,如无特殊说明,为可从商业途径得到的试剂和材料。
37.1、伦理陈述和组织标本
38.以下实施例(伦理号:gzr2017-130)经中山大学肿瘤防治中心临床研究伦理委员会批准,按照《赫尔辛基宣言》的原则进行。大部分数据均来自在线数据库。
39.在中山大学肿瘤中心肝胆肿瘤科接受肝切除术的患者中获得6对hcc及相应的癌旁组织样本和306例肿瘤组织样本,并获得每位患者的知情同意。患者未接受术前治疗,且无其他恶性肿瘤病史。排除有肝外转移和肝细胞癌侵犯胆道系统的患者。术后病理结果均为肝细胞癌。复发方式有肝内和肝外两种。
40.2、ualcan
41.ualcan是一个基于the cancer genome atlas(tcga)数据库的癌症数据分析网站。它帮助医学研究人员分析肿瘤和正常样本之间的基因转录差异,然后进行更彻底的分析,如发现目标基因时,对生物标记物进行识别和生存分析。此外,还可以通过相关链接查询其他数据库中的相关信息。
42.3、timer
43.timer(http://timer.cistrome.org/)是一个包含多种癌症类型资源的分析网站。
44.4、kaplan-meier plotter
45.kaplan
–
meier plotter是最大的肿瘤数据集之一,包含了54000个与多种癌症的生存相关的基因。
46.5、数据分析
47.所有数据使用graphpad software 6(graphpad,la jolla,ca,usa)和statistical package for the social sciences(spss,version 22.0)进行分析。采用kaplan-meier法评估scn4a或scn7a表达与生存预后的关系。通过卡方检验验证临床病理因素与scn4a或scn7a表达的相关性,单因素和多因素分析采用cox比例风险回归模型评估预后因素。p值小于0.05为有统计学意义。
48.实施例1不同scn家族成员在肝癌患者中的表达差异
49.一、不同scn成员在正常组织和原发肿瘤中的mrna表达情况
50.1、实验方法
51.使用ualcan数据库分析了不同scn成员在正常组织和原发肿瘤中的mrna表达情况。
52.2、实验结果
53.分析了scn家族9个成员的表达水平,结果如图1所示,结果显示,scn4a/5a/8a在肿瘤组织中高表达,而scn1a/2a/7a/11a在肿瘤组织中低表达。
54.二、芯片分析检测了肝癌患者的肿瘤组织和邻近的非肿瘤组织中scn家族成员的表达
55.1、实验方法
56.用芯片分析检测了6对hcc患者的肿瘤组织和邻近的非肿瘤组织中scn家族成员的表达。使用根据agilent公司(agilent technologies inc.,ca,usa)提供的单色微阵列基
因表达分析操作说明(agilent one-color microarrays-based gene expression analysis protocol,ca,usa)进行6对hcc患者的肿瘤组织和相邻的非肿瘤组织的纯化和杂交处理。阵列图像使用agilent feature extraction软件(版本11.0.1.1)进行分析。使用genesspring gx v11.5.1软件包(agilent technologies inc.,ca,usa)进行分位数归一化和后续数据处理。
57.2、实验结果
58.如图2所示,只有scn4a(p=0.049)和scn7a(p=0.011)的表达发生了显著变化;此外,scn4a和scn7a在肿瘤组织和非肿瘤组织中的表达趋势与ualcan数据库中的结果一致,即scn4a在肿瘤中表达较高,而scn7a在肿瘤中表达较低。
59.实施例2 scn4a和scn7a的泛肿瘤分析
60.一、实验方法
61.在timer数据库中对scn4a和scn7a进行了泛肿瘤分析。检测38种肿瘤组织中scn4a和scn7a的mrna表达,并与正常组织进行比较。
62.二、实验结果
63.如图3a和b所示,scn4a在几种癌症中表达较高,如肝癌;然而,它在某些类型的肿瘤组织中表达则较低。而scn7a的表达在许多类型的肿瘤表达都比较稳定的下调,其中包括了肝癌。
64.实施例3 scn4a和scn7a与hcc患者临床病理因素的关系
65.一、实验方法
66.通过ualcan数据库,分析了scn4a/7a与肿瘤分级、淋巴结转移状态、组织学亚型、患者种族、肿瘤个体分期、tp53突变状态等病理因素之间的关系。
67.二、实验结果
68.如图3c所示,scn4a的mrna表达在各个级别均显著升高,而scn7a的表达随着肿瘤级别的增加逐渐降低,且在1级和3级发生显著变化。scn4a和scn7a mrna表达水平也与肿瘤分期显著相关;同样,scn4a在各期均高表达,而scn7a在3期和4期表达下降(图3g)。scn4a在有淋巴结转移的组织中比无淋巴结转移的组织中表达更高。在组织学亚型方面,与正常组织相比,scn4a和scn7a的表达仅在肝胆癌中有显著差异(图3e)。从补充图1f显示的结果中,发现只有高加索或亚洲患者的肿瘤样本scn4a表达较高,而非裔或亚洲患者的肿瘤样本scn7a表达较低。scn4a的表达水平在tp53突变组和tp53未突变组均较高,而scn7a的表达仅在tp53突变组肿瘤组织中显著降低(图3h)。
69.实施例4 scn4a和scn7a在肝癌中的预后意义
70.一、实验方法
71.使用kaplan
–
meier plotter分析了scn4a mrna和scn7a mrna的表达与肝癌的总生存期(os)、无复发生存期(rfs)、疾病特异性生存期(dss)和无进展生存期(pfs)之间的关系。
72.并通过cbioportal数据库检测了scn4a和scn7a基因突变与肝癌患者os、rfs、dss或pfs之间的关系。
73.二、实验结果
74.scn4a mrna的高表达与较长的os、dss和pfs相关(图4a),而高scn7a mrna的表达
与更长的os相关(图4b)。
75.而在亚洲患者中,scn4a mrna的高表达与较长的os和dss(图5a),scn7a mrna的高表达与更长的os、rfs、dss和pfs相关(图5b)。
76.scn4a突变与os,rfs,dss和pfs无关(图6a),但scn7a突变与rfs和pfs相关(图6b)。
77.实施例5 scn4a和scn7a蛋白在肝细胞癌(hcc)组织中的表达及预后价值
78.一、实验方法
79.对306例肿瘤组织样本进行免疫组化分析,具体方法:
80.肝细胞癌组织用酒精梯度再水化,并被h2o2阻断过氧化物酶。然后,在柠檬酸缓冲液(ph 6.0)中用微波加热进行抗原修复。接下来,将组织与一抗在4℃孵育一夜。与二抗孵育后,dab显色法显色。
81.将载玻片分为四组(chen,d.t.,j.h.pan,y.h.chen,w.xing,y.yan,y.f.yuan,et al.,the mu-opioid receptor is a molecular marker for poor prognosis in hepatocellular carcinoma and represents a potential therapeutic target.br j anaesth,2019.122(6):p.e157-e167 doi:10.1016/j.bja.2018.09.030.):染色强度分级标准为:0=未染色、1=弱染色、2=中等染色、3=强染色。
82.肿瘤组织阳性百分率分级标准为:0(0-10%)、1(10%-25%)、2(26%-50%)、3(51%-75%)、4(75%-100%)。
83.将2个评分相乘,分为四组免疫组化:缺失染色(-)(评分0~3)、弱染色(+)(评分4~6)、中等染色(++)(评分7~9)、强染色(+++)(评分10~12)。
84.二、实验结果
85.根据免疫组化结果,将四种不同染色强度分组(图7a和7d)。306例患者,平均年龄50.7岁(范围20~83),其中男性270例,女性36例,中位随访期59.1个月(范围1~129)。如图7b和7c所示,scn4a蛋白表达与os或rfs无关;然而,scn7a蛋白表达与os(p=0.001)和rfs(p=0.003)显著相关(图7e和7f),且表达越高生存率越高,这与在线数据库的结果一致。
86.患者的临床病理因素以及scn4a/7a蛋白表达与预后的单因素生存分析如表1所示。
87.表1单因素生存分析:
88.89.[0090][0091]
没有child-pugh c级患者;根据美国癌症联合委员会(ajcc)第七版评估肿瘤-淋巴结转移(tnm)分期;缩写:drd1多巴胺受体d1;hcc肝细胞癌;afpγ-谷氨酰转移酶;tnm,肿瘤-淋巴结-转移。
[0092]
scn7a表达与hcc患者临床病理因素相关性分析如表2所示:
[0093]
表2 scn7a表达与hcc患者临床病理因素相关性分析
[0094]
[0095]
[0096][0097]
具有统计意义的值以粗体显示;没有child-pugh c级患者。
[0098]
患者的临床病理因素以及scn4a/7a蛋白表达与预后的多因素生存分析如表3所示:scn7a(p=0.034)、卫星结节(p=0.032)和alt(p=0.001)是rfs的独立预后因素。此外,scn7a(p=0.025)、白蛋白(p=0.020)和tnm分类(p=0.029)也是os的独立预后因素(图7g和表3)。
[0099]
表3多因素生存分析:
[0100][0101]
此表仅显示了显著影响rfs或os的因素。
[0102]
最后所应当说明的是,以上实施例仅用以说明本发明的技术方案而非对本发明保护范围的限制,对于本领域的普通技术人员来说,在上述说明及思路的基础上还可以做出其它不同形式的变化或变动,这里无需也无法对所有的实施方式予以穷举。凡在本发明的精神和原则之内所作的任何修改、等同替换和改进等,均应包含在本发明权利要求的保护范围之内。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1