一种筛选肿瘤特异TCR的方法
一种筛选肿瘤特异tcr的方法
技术领域
1.本发明涉及肿瘤免疫技术领域,具体涉及一种筛选肿瘤特异tcr的方法。
背景技术:
2.t细胞识别对应的抗原是通过其细胞表面的t细胞受体(tcr),tcr是t细胞表面的一种受体分子,它特异性识别抗原提呈细胞上的抗原肽-mhc复合物,进而激发t细胞免疫应答。由于大部分t细胞表面的tcr无法识别肿瘤细胞,因而t细胞无法有效杀伤肿瘤细胞,导致肿瘤细胞的快速扩增。如果找到特异识别肿瘤细胞的t细胞,克隆其对应的tcr,进而通过基因编辑(比如慢病毒)将该tcr导入t细胞,就会生成t 细胞受体基因修饰t 细胞 (tcr-t),肿瘤抗原特异性的tcr转入普通t细胞后,能赋予该t细胞识别肿瘤抗原的能力,该tcr-t具备特异识别和杀伤肿瘤细胞的能力,经体外活化增殖后再输入患者体内,可以发挥抗肿瘤功效,并已在多种癌症中显示出有效的抗肿瘤活性,因此可知肿瘤特异性 tcr的筛选是tcr-t治疗成功的关键,是肿瘤免疫治疗中研究的热点。
3.t细胞被肿瘤抗原激活后会表达t细胞标志物,因此前期研究通过肿瘤抗原刺激t细胞,进而筛选表达t细胞标志物的t细胞并完成对应tcr的克隆,由此获得肿瘤特异的tcr,其中标志物主要为ifn-γ(对应基因名称为ifng)或41bb(对应基因名称为tnfrsf9)。但是不同类型t细胞(比如幼稚t细胞,效应t细胞和记忆t细胞)以及抗原刺激后的不同时间,都会影响t细胞标志物的表达,即不同标志物的表达水平差异很大,因此使用单一标志物筛选肿瘤特异t细胞可能会漏掉部分未表达某一标志物的肿瘤特异t细胞,进而造成假阴性。为使筛选结果更准确,有必要使用更多的t细胞标志物综合评分来筛选肿瘤特异t细胞,进而筛选肿瘤特异的tcr。但是在选用多个t细胞标志物筛选肿瘤特异t细胞的过程中,发明人发现并非选择的t细胞标志物越多越好,也并非均选用本领域内公知的与激发t细胞免疫应答关联度最强的标志物的表达量数值进行评分所对应的筛选效果就最好,事实上发明人经过持续的研究发现,特定的t细胞标志物组合的表达量平均值用于筛选肿瘤特异tcr,效果更准确,过程更高效。
技术实现要素:
4.本发明的目的是提供一种高效、准确筛选肿瘤tcr的方法。
5.发明人在研究过程中,选择了众多业内普遍认同的t细胞标志物,将tils与对应肿瘤细胞共孵育后得到的激活的tils进行单细胞转录组测序,可以获得每个t细胞标志物(基因)的表达水平,即每个基因对应的mrna含量,进而利用软件的程序(seurat addmodulescore)(参考文献和使用说明分别见:cell. 2021 nov 11;184(23):5838. cell 177, 1888-1902 e1821 (2019). 和https://rdrr.io/cran/seurat/man/addmodulescore.html),根据软件默认参数计算每个tils细胞的激活得分,大概的计算过程为:将每个t细胞标志物正态化表达数值输入该软件就可得出tils细胞激活得分,该得分为标志物正态化表达数值减去背景值后的平均表达值,因此每个tils细胞可以计算出一个
tils细胞激活得分,然后将具有相同tcr的所有tils细胞的tils细胞激活得分再取平均值,即为该种tcr激活得分的数值。
6.通过不断研究和探索,本发明确定采用10个t细胞标志物表达量的综合评分来筛选肿瘤特异tcr具有最高的灵敏度和特异度,即假阳性和假阴性率最低,这10个标志物对应的基因分别为:ifng, il2, tnf, il2ra, cd69, tnfrsf9, gzmb, gzma, gzmk和 prf1。本发明将肿瘤患者的肿瘤细胞和对应的tils体外共孵育4-24 h,然后将这些细胞进行单细胞转录组和tcr组测序,根据所述10个标志物的综合表达得分,筛选得分最高的前几位为肿瘤特异t细胞的tcr。
7.具体地,本发明提供一种筛选肿瘤特异tcr的方法,包括以下步骤:(1)将肿瘤患者的自体肿瘤细胞对应的肿瘤浸润t细胞(tils)激活;本领域技术人员能够理解,激活tils所采用的方法均是本领域内常用的方法,比如将肿瘤患者的肿瘤细胞与对应的tils体外共孵育,或者由于肿瘤抗原负载的提呈细胞,比如树突状细胞或b细胞等均可以代替肿瘤患者的肿瘤细胞与患者的tils进行体外共孵育,这样做的目的均是用肿瘤抗原激活tils,进而用于筛选肿瘤特异的t细胞的tcr。在本发明的一个实施例中,是采用肿瘤患者的肿瘤细胞与对应的tils体外共孵育4-24 h,可实现对tils的激活。
8.(2)将这些细胞进行单细胞转录组和tcr组测序,获得每个tils细胞的tcr序列和每个基因的表达数值;(3)对于表达同一tcr的全部tils,以其10种标志物表达值的平均值作为该种tcr的激活得分,根据该tcr激活得分,筛选得分较高者为对应肿瘤特异t细胞的tcr;所述10种标志物分别为:ifng, il2, tnf, il2ra, cd69, tnfrsf9, gzmb, gzma, gzmk和 prf1。
9.步骤(3)的每个tils的tcr激活得分计算方法为:将每个tils的10种标志物正态化表达数值输入seurat addmodulescore,根据软件默认参数计算出tils激活得分,该得分为这10种t细胞标志物正态化表达数值减去背景值后的平均表达值,每个tils可以计算出一个激活得分,然后将具有相同tcr的所有tils的激活得分取平均值,即为该种tcr激活得分的数值。
10.seurat addmodulescore 软件程序计算方法的参考文献和使用说明见上文。
11.本发明提供的方法,步骤(3)还包括筛选激活得分数值较高的前三位tcr即为该肿瘤患者肿瘤特异tcr。发明人经过大量试验验证后发现,激活得分较高的前三位tcr能够被证实其的确为肿瘤特异tcr。
12.本发明提供了上述筛选肿瘤特异tcr方法在制备tcr-t细胞、肿瘤免疫和/或肿瘤治疗中的应用。
13.本发明还提供了上述筛选肿瘤特异tcr方法筛选得到的tcr在制备tcr-t细胞中的应用。本领域技术人员能够理解,上述肿瘤特异tcr可广泛应用于制备tcr-t细胞、肿瘤免疫和/或肿瘤治疗中,其应用属于本发明的保护范围。
14.另一方面,本发明提供了用于筛选肿瘤特异tcr的标志物组合,由以下10种基因或对应蛋白组成:ifng, il2, tnf, il2ra, cd69, tnfrsf9, gzmb, gzma, gzmk和 prf1。
15.本发明提供了所述标志物组合在体外筛选肿瘤特异tcr、和/或在提高肿瘤特异
tcr筛选效率和准确率中的应用。
16.本发明还提供了所述的标志物组合在制备tcr-t细胞中的应用。
17.本领域技术人员应理解,用于检测上述标志物组合中每个标志物表达量的试剂组合在筛选肿瘤特异tcr中的应用以及在制备tcr-t细胞中的应用都属于本发明的保护范围。
18.本发明利用单细胞测序技术,根据10个t细胞标志物的综合得分,建立一套高效筛选肿瘤特异tcr的方法,可以为每位肿瘤患者高效筛选多种肿瘤特异tcr,优于根据单一标志物(ifn-γ或41bb)筛选肿瘤特异tcr的方法,更优于其他多个标志物组合评分筛选肿瘤特异tcr的方法。
附图说明
19.图1为未转染tcr的对照t细胞(un-tranduced)和转染每种tcr的t细胞(p1-ifn-tcr1,p1-ifn-tcr2,p1-ifn-tcr3,p1-41bb-tcr2,p1-41bb-tcr3)与对应的自体肿瘤细胞(atc)体外共孵育12h后分泌ifn-γ的浓度比较图。
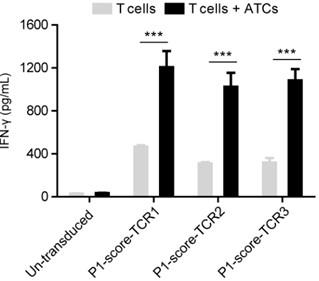
20.图2为未转染tcr的对照t细胞(un-tranduced)和转染每种tcr的t细胞(p1-score-tcr1,p1-score-tcr2,p1-score-tcr3)与对应的自体肿瘤细胞(atc)体外共孵育12h后分泌ifn-γ的浓度比较图。
21.图3为未转染tcr的对照t细胞(un-tranduced)和转染p1-score2-tcr2的t细胞与对应的自体肿瘤细胞(atc)体外共孵育12h后分泌ifn-γ的浓度比较图。
22.图4为未转染tcr的对照t细胞(un-tranduced)和转染p1-score5-tcr3的t细胞与对应的自体肿瘤细胞(atc)体外共孵育12h后分泌ifn-γ的浓度比较图。
23.图5为未转染tcr的对照t细胞(un-tranduced)和转染每种tcr的t细胞(p1-score-tcr1,p1-score-tcr2,p1-score-tcr3)不同效靶比杀伤自体肿瘤细胞(atc)的能力。
24.图6为未转染tcr的对照t细胞(un-tranduced)和转染每种tcr的t细胞(p1-score-tcr1,p1-score-tcr2,p1-score-tcr3)回输移植瘤小鼠后,肿瘤体积的统计结果。
25.*代表p《0.05,***代表p《0.001,未标注*表示不存在统计学差异。
具体实施方式
26.以下实施例用于说明本发明,但不用来限制本发明的范围。慢病毒包装质粒(pmdl,vsv-g,rev)、pcdh-ef1-luc2-t2a-tdtomato质粒购自武汉淼灵生物科技有限公司。xbai和sali内切酶购自newenglandbiolabs(beijing)ltd。pei购自sigma公司。x-vivo15培养基购自lonza公司。il-2,il-7,il-15细胞因子购自peprotech公司。okt3购自acrobiosystems有限公司。cd28抗体购自同立海源公司。293ft细胞购自atcc。cd3,cfse,pi等流式抗体购自bd公司,抗鼠源tcrβ链恒定区流式抗体购自ebioscience公司。ifn-γ检测试剂盒购买自依科赛生物科技有限公司。肺癌患者的tils,移植瘤模型均为本实验构建。nod/scid免疫缺陷小鼠购自北京维通利华实验动物技术有限公司。
27.实施例1tils单细胞转录组和tcr组测序tils培养肿瘤患者的肿瘤组织经手术切除后,将肿瘤组织切碎到1-2毫米的小块,每一小块肿瘤组织分别放置于24孔细胞培养板的一个孔内,然后加入t细胞培养基。t细胞培养基包
含x-vivo 15无血清培养基(lonza, usa) 以及il2 (50u/ml; peprotech, usa), il-7 (10 ng/ml; peprotech, usa), il-15(10 ng/ml, peprotech, usa), okt3 antibody (50ng/ml; acro, usa) and anti-cd28 antibody (1ug/ml; t&l biotechnology, china)。 该小块肿瘤组织在细胞培养箱培养,直到获得最终tils。
28.与对应肿瘤细胞共孵育tils与对应的自体肿瘤细胞在x-vivo15无血清培养基中共孵育12h,然后pbs冲洗后,按着10xgenomic要求,细胞大小:≤30μm;细胞活率≥85%;成团率及碎片率<5%(明场检测);细胞浓度及总量:浓度700-1200个/μl,总量≥10万。建立单细胞转录组和tcr组文库并完成上机测序,由安诺优达基因科技(北京)有限公司完成。
29.单细胞转录组和tcr组测序根据单细胞转录组和tcr组基本质控要求后,同时需要符合下面质控标准。单细胞转录组质控标准:每个细胞基因数目在800-10000,线粒体基因占比小于20%,且cd3d, cd3e 和 cd3g的umi平均值大于 0。单细胞tcr组质控标准:tcr α和β链的umi均大于0。同时符合单细胞转录组和tcr组质控标准的t细胞纳入最终分析,应用seurat的
‘
lognormalize’方法计算每个tils细胞基因正态化的表达数值,应用cell ranger vdj pipeline对tcr α和β链序列进行组装和识别,获得符合要求的tils细胞共6144个。
30.理论上如果某一tcr可以特异识别对应的肿瘤细胞,则表达该tcr的全部tils细胞都应该被对应的肿瘤细胞激活,考虑到具有相同tcr的tils细胞不一定都能与对应肿瘤细胞接触或肿瘤细胞异质性,这些可能会导致一部分t细胞无法被对应肿瘤细胞特异激活,但总体上表达同一肿瘤特异tcr的大部分tils细胞能被特异激活,因此,本发明将表达相同tcr的tils细胞统一分析,即将表达同一tcr的全部tils细胞的标志物表达值的平均值作为该种tcr标志物的表达数值。
31.将每个tils的标志物正态化表达数值输入seurat addmodulescore(软件程序计算方法参见参考文献和使用说明:cell. 2021 nov 11;184(23):5838. cell 177, 1888-1902 e1821 (2019).https://rdrr.io/cran/seurat/man/addmodulescore.html,根据软件默认参数计算tils激活得分,该得分为这些标志物正态化表达数值减去背景值后的平均表达值,每个tils计算出一个激活得分,然后将具有相同tcr的所有tils的激活得分取平均值,即为该种tcr激活得分的数值。
32.以下各试验组,均针对上述符合要求的6144个tils细胞进行的试验。经检测发现6144个tils细胞中共有447种不同的tcr,其中最高频tcr有1116个tils细胞具有该种tcr,最低频tcr只有一个tils细胞具有该tcr,平均约14个tils细胞具有一个相同的tcr。以下各个试验组均利用上述软件直接计算出每个tils细胞的激活得分,然后把表达同一tcr的所有tils细胞的激活得分取平均值,即为该类tcr的激活得分。不同激活得分计算的唯一区别就是一开始纳入的标志物种类的区别,后面计算方法一样。本发明证明在上述6144个tils细胞中,试验组(3)筛选确定的具有特异识别和杀伤对应肿瘤细胞的3种tcr(p1-score-tcr1, p1-score-tcr2, p1-score-tcr3)分别有3,4,3个tils细胞具有该tcr(p1-score-tcr1, p1-score-tcr2, p1-score-tcr3)。分组试验如下:试验组(1)一种标志物ifng本试验组按ifng的表达数值进行排序,按照上述方法计算tils细胞激活得分并选
tcr3和p1-score-tcr2序列一样,所以p1-score4-tcr1和p1-score4-tcr3基因修饰t细胞都是具备特异识别肿瘤细胞能力,但是p1-score4-tcr2和p1-score2-tcr2序列相同,因此p1-score4-tcr2基因修饰t细胞不能特异识别肿瘤细胞。
47.试验组(7),根据激活综合得分5筛选的3种tcr(p1-score5-tcr1,p1-score5-tcr2,p1-score5-tcr3),其中p1-score5-tcr1和p1-score-tcr3的序列一样,所以p1-score5-tcr1基因修饰t细胞是具备特异识别和杀伤肿瘤细胞能力,但是p1-score5-tcr2和p1-score2-tcr2序列相同,因此p1-score5-tcr2基因修饰t细胞不能特异识别肿瘤细胞,而且p1-score5-tcr3基因修饰t细胞也不能特异识别肿瘤细胞(图4)。
48.根据t细胞激活得分筛选出的3种tcr(p1-score-tcr1,p1-score-tcr2,p1-score-tcr3)均可以特异识别对应肿瘤细胞,而且其他组筛选的肿瘤特异tcr均属于上述三个tcr,因此,总共上述三种tcr(p1-score-tcr1,p1-score-tcr2,p1-score-tcr3)可以特异识别对应的肿瘤细胞。进一步,本实施例通过体外杀伤实验评价这3种tcr特异杀伤对应肿瘤细胞的能力,每种tcr-t细胞分别与cfse预标记的自体肺癌肿瘤细胞以30:1,10:1,2:1效靶比共孵育,流式鉴定cfse标记的靶细胞中细胞死亡比例(pi
+
靶细胞百分比)。结果显示,这三种tcr-t可以体外特异杀伤对应肿瘤细胞(图5),而且这三种tcr-t中的每种tcr-t细胞(6
×
106)回输对应肺癌肿瘤细胞构建的移植瘤小鼠模型,结果显示,与未经基因修饰t细胞相比,这三种tcr基因修饰t细胞可以体内杀伤小鼠移植瘤模型(图6)。
49.实施例6其他4例肺癌患者验证上述筛选tcr的策略本实施例再纳入4例肺癌患者,以上述模式获得患者的tils和肿瘤细胞,按照实施例1描述的方法进行单细胞转录组和tcr组测序,获得每个t细胞tcr序列和每个标志物的表达数值,然后分别以上述试验组(1)-(7)种方式筛选肿瘤特异tcr,最终发现按着试验组(3)的ifng,il2,tnf,il2ra,cd69,tnfrsf9,gzmb,gzma,gzmk,和prf1表达数值的平均值(激活综合得分)进行排序,得分最高的3种tcr均能特异识别和杀伤对应的肿瘤细胞,而以单独ifng或tnfrsf9得分筛选的3种tcr以及根据试验组(4)-(7)多种t细胞标志物计算的每种激活得分(激活综合得分2/3/4/5)筛选的3种tcr,最多只有1-2种tcr可以特异识别对应肿瘤细胞;因此可以证明本发明提出的筛选肿瘤特异tcr方法具有比根据单独t细胞标志物的筛选方法、以及其他组合方式的多个t细胞标志物、甚至超过10个t细胞标志物的组合筛选具有更好的筛选效果,本发明方法筛选得到的得分最高的前三位tcr验证其的确是肿瘤特异tcr,为后续制备tcr-t细胞奠定了基础。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1