基于纳米孔测序的进境植物检疫性病毒鉴定方法及应用与流程
1.本发明涉及植物病毒检疫领域,特别涉及一种基于纳米孔测序的进境植物检疫性病毒鉴定方法及应用。
背景技术:
2.植物病毒素有植物“癌症”之称,据不完全统计,全世界每年因植物病毒造成的农业损失占粮食作物总产量的10%,损失高达200亿美元。随着国际间频繁的农业交流,如种子和苗木调运,水果蔬菜贸易等交流,一些检疫性植物病毒很可能会传入我国。目前我国对国门生物安全越来越重视,对植物检疫提出了更高的要求,因而检疫部门迫切需要提高植物病毒的检疫鉴定技术、加强口岸的检疫工作,以减少危险性植物病毒的传入,保护我国的农业生产和生态安全。
3.目前检测植物病毒的传统方法包括血清学检测、电子显微镜观察、指示植物物种、通过pcr或rt-pcr进行的dna扩增、芯片杂交等技术。上述方法使用的一个重要前提是对病原物的基因组特征、血清学特性、生物学特性、理化特性等有预先的了解,而在对未知病原物了解缺乏的情况下,这些检测方法就会出现耗时长、效率低下、容易出现检测误差等问题,因此极大地限制了其在实际工作中的应用。随着测序技术的发展,测序技术也被迅速用于植物病毒的快速检测,目前主流的基于深度测序的植物病毒检测主要以第二代测序 (next-generation sequencing,ngs)技术为主。基于二代测序的ngs测序技术被广泛的应用到动植物病毒的诊断中,具有高效、快速、高通量以及非序列依赖性等特点,可实现单一样品多目标检测,避免一些病毒的漏检或错检,还能发现一些新的病毒或类病毒,这要优于传统的elisa或pcr技术。但是基于ngs 测序技术的病毒检测依然面临很多问题:1.在设备上,ngs测序仪都属于大型仪器,需要在专业的实验室或公司平台;2.在实验操作上,测序文库准备复杂,步骤繁琐,需要专业的实验操作人员;3.在测序流程和检测周期上,实验操作和测序需要2-3天的时间;4.在测序数据质量和数据分析上,由于ngs依赖于 pcr扩增,因此具有一定的测序偏好性,导致基因组覆盖不全;而且由于ngs测序读长不足kb,因此病毒基因组需要组装,在一定程度上结果极度依赖软件和算法的优化和提高。
4.随着各国对生物安全的日益重视,急需一种更加快速、精准以及全面的检测技术来满足植物病毒检疫需求。
技术实现要素:
5.本发明的目的在于提供一种可利用纳米孔测序技术对进境植物检疫性病毒进行鉴定的方案,对植物检疫性病毒进行数据前处理并生成包括比对序列统计、序列总数、测序深度以及不同测序深度被检测病毒基因组覆盖度的检测报告。
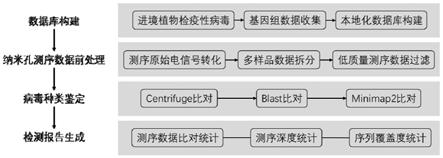
6.为实现以上目的,本技术方案提供一种基于纳米孔测序的进境植物检疫性病毒鉴定方法,包括以下步骤:
7.构建进境植物检疫性病毒基因组数据库,所述进境植物检疫性病毒基因组数据库
包括至少一进境植物检疫性病毒的病毒基因组序列;
8.利用纳米孔测序技术获取待测样品的纳米孔测序数据,并前处理所述纳米孔测序数据得到分析数据;
9.初步分类鉴定所述分析数据得到初步鉴定结果,比对所述初步鉴定结果和所述分析数据并过滤得到一致性比对序列,比对所述一致性比对序列和所述进境植物检疫性病毒基因组数据库并过滤得到目标序列,比对所述目标序列和所述一致性比对序列并过滤得到有效鉴定结果。
10.在一些实施例中,在获取了有效鉴定结果之后还基于所述有效鉴定结果生成鉴定报告,所述鉴定报告包含纳米孔测序数据和分析数据的统计结果,且包含了比对序列统计、序列总数、测序深度以及不同测序深度被检测病毒基因组覆盖度等信息内容。
11.本方案提供的基于纳米孔测序的进境植物检疫性病毒鉴定方法可用于鉴定进境植物检疫性病毒,故本方案需要首先构建进境植物检疫性病毒基因组数据库。本方案涉及的进境植物检疫性病毒包括但不限于:wheat streak mosaicvirus,wsmv小麦线条花叶病毒、sugarcane streak virus,ssv甘蔗线条病毒、potato virus v,pvv马铃薯v病毒、potato virus a,pva马铃薯a病毒、tobacco ringspot virus,trsv烟草环斑病毒、southern bean mosaic virus, sbmv南方菜豆花叶病毒、potato mop-top virus,pmtv马铃薯帚顶病毒、oatmosaic virus,omv燕麦花叶病毒、maize chlorotic mottle virus,mcmv玉米褪绿斑驳毒、maize chlorotic dwarf virus,mcdv玉米褪绿矮缩病毒、cacaoswollen shoot virus,cssv可可肿枝病毒、carnation ringspot virus,crsv 香石竹环斑病毒、bean pod mottle virus,bpmv菜豆荚斑驳病毒、arabis mosaicvirus,armv南芥菜花叶病毒、potato yellow dwarf virus,pydv马铃薯黄矮病毒等。值得说明的是,本方案可适用于任意已知病毒基因组序列的进境植物检疫性病毒。
12.在本方案的一具体实施例中,首先筛选出属于植物检疫性植物病毒的条目,根据植物检疫性病毒条目检索病毒的基因组测序信息及参考基因组序列,下载并统一格式,将收集整理得到的病毒基因组序列构建进境植物检疫性病毒基因组数据库,用于后续流程分析使用。
13.本方案采用纳米孔测序技术对待测样品进行检测,以获取纳米孔测序数据。纳米孔测序技术是近几年逐渐发展成熟的一种全新的测序技术,不同于以往测序技术基于化学信号,它是基于核酸分子通过纳米孔时电势差变化而进行碱基检测的单分子实时测序技术,是目前唯一可以对核酸分子直接进行测序的技术。该技术可实现对dna/cdna/rna的直接测序,具有速度快、读长长、准确度高、数据实时分析、机器便携易用、文库构建简单、无pcr扩展偏好性、平台成本低等其他测序平台不具有的优势。
14.然而纳米孔测序技术产生的纳米孔测序数据为电信号,故需要对其进行转换处理后得到所述分析数据。具体的,在“前处理所述纳米孔测序数据得到分析数据”步骤中,将所述纳米孔测序数据转换为fasta碱基序列文件,过滤低质量测序数据得到所述分析数据。
15.在本方案的实施例中,低质量测序数据为测序质量值小于7以及序列长度小于100bp的测序片段。当然,低质量测序数据的定义内容可自定义设定。
16.另外,若本方案采用对多样本测序的纳米孔测序技术,则在分析每个待测样品的纳米孔测序数据之前需要对混合的待测样品进行拆分,其中同一测序序列两端标签一致的
纳米孔测序数据为同一类样品。
17.在“初步分类鉴定所述分析数据得到初步鉴定结果”步骤中,使用 centrifuge软件及本地数据库对所述分析数据进行分类鉴定,并过滤低鉴定值的分类结果得到所述初步鉴定结果。这步骤的作用是在:初步过滤噪音结果。另外,此处所指的本地数据库指的是:前面收集的以及构建的植物检疫性病毒基因组数据库。
18.在本方案的实施例中,过滤掉centscore低于150的分类结果,剩余的物种分类结果为所述初步鉴定结果。当然,centscore的阈值也是人为设定的。
19.在“比对所述初步鉴定结果和所述分析数据并过滤得到一致性比对序列”步骤中,使用minimap2软件将所述分析数据与经centrifuge初步鉴定后的所述初步鉴定结果比对得到比对值,筛选比对值低于比对阈值的比对信息,剩余的比对信息组成所述一致性比对序列。这步骤的作用是:进一步筛选噪音数据。
20.在本方案的实施例中过滤比对值小于50的比对信息,剩余的比对信息组成一致性比对序列。当然,比对阈值可人为设定。
21.在“比对所述一致性比对序列和所述进境植物检疫性病毒基因组数据库并过滤得到目标序列”步骤中,将所述一致性序列片段通过blast软件与进境植物检疫性病毒基因组数据库比对得到比对值,筛选比对值低于识别阈值的比对信息,剩余的比对信息组成目标序列。这步骤的目的是:进一步筛选过滤。
22.在本方案的实施例中,过滤比对值低于85%的比对信息,剩余的比对信息组成目标序列。当然,识别阈值可人为设定。
23.在“比对所述目标序列和所述一致性比对序列并过滤得到有效鉴定结果”步骤中,根据blast鉴定结果再次使用minimap2软件将所述分析数据与所述目标序列进行比对得到比对值,筛选比对值低于设定阈值以及碱基长度低于设定阈值的分析数据,得到最终的有效鉴定结果。这步骤的目的是:进一步筛选过滤。
24.在本方案的实施例中,比对结果过滤掉比对分值低于50以及分析数据上有效比对上目标序列碱基总数占整个分析数据的长度百分比低于80%的分析数据。
25.相较现有技术,本技术方案具有以下特点和有益效果:
26.首先构建进境植物检疫性病毒基因组数据库,再利用纳米孔测序技术检测待测样品并前处理得到分析数据,基于进境植物检疫性病毒基因组数据库对分析数据进行病毒种类鉴定,并得到针对待测样品的检测报告。
附图说明
27.图1是根据本方案的一实施例的基于纳米孔测序的进境植物检疫性病毒鉴定方法的流程示意图。
28.图2是植物病毒浓度5%基因组参考序列覆盖度曲线图。
29.图3是病毒浓度0.5%基因组参考序列覆盖度曲线图。
30.图4是病毒浓度0.05%基因组参考序列覆盖度曲线图。
31.图5和图6是本方案检测得到的检测报告的示意图。
具体实施方式
32.为了便于理解本发明,下面将参照相关附图对本发明进行更全面的描述。附图中给出了本发明的较佳实施例。但是,本发明可以以许多不同的形式来实现,并不限于本文所描述的实施例。相反地,提供这些实施例的目的是使对本发明的公开内容的理解更加透彻全面。除非另有定义,本文所使用的所有的技术和科学术语与属于本发明的技术领域的技术人员通常理解的含义相同。本文中在本发明的说明书中所使用的术语只是为了描述具体的实施例的目的,不是旨在于限制本发明。本文所使用的术语“和/或”包括一个或多个相关的所列项目的任意的和所有的组合。
33.实施例样品背景:本次实施例选取了15种粮食作物上常见的植物检疫性病毒(wheatstreakmosaicvirus,wsmv小麦线条花叶病毒、sugarcanestreakvirus,ssv甘蔗线条病毒、potatovirusv,pvv马铃薯v病毒、potatovirusa,pva马铃薯a病毒、tobaccoringspotvirus,trsv烟草环斑病毒、southernbeanmosaicvirus,sbmv南方菜豆花叶病毒、potatomop-topvirus,pmtv马铃薯帚顶病毒、oatmosaicvirus,omv燕麦花叶病毒、maizechloroticmottlevirus,mcmv玉米褪绿斑驳毒、maizechloroticdwarfvirus,mcdv玉米褪绿矮缩病毒、cacaoswollenshootvirus,cssv可可肿枝病毒、carnationringspotvirus,crsv香石竹环斑病毒、beanpodmottlevirus,bpmv菜豆荚斑驳病毒、arabismosaicvirus,armv南芥菜花叶病毒、potatoyellowdwarfvirus,pydv马铃薯黄矮病毒),植物病毒基因组通过引物扩增后等量混合,然后分别以0%,、0.05%、0.5%、5%浓度混合到健康无毒的烟草基因组反转录dna提取液,然后通过纳米孔测序,利用本发明一种基于纳米孔测序技术进境植物检疫性病毒鉴定及应用检测鉴定的成效,包括以下步骤:
34.一、数据库构建
35.1、进境植物检疫性病毒条目获取
36.确定目前我国进境植物检疫性病毒包含39种,类别如下:
37.表1.我国进境植物检疫性病毒
38.[0039][0040]
1.病毒基因组序列下载
[0041]
从ncbi网站下载病毒基因组,病毒基因组数据信息如表2所示:
[0042]
表2.病毒基因组数据信息
[0043]
[0044][0045]
2.基因组序列本地化数据库构建
[0046]
分别使用blast、minimap2、centrifuge软件构建本地化分析数据库。
[0047]
二、纳米孔测序数据前处理
[0048]
1.分析第一步是将电信号转换成碱基信号,该步骤使用官方推荐的guppy软件basecaller模块,转换后生成fastq的测序文件,得到的纳米孔测序数据如表3所示:
[0049]
表3纳米孔测序数据统计
[0050]
typetotal seqstotal bases(bp)avg.length(bp)n50(bp)passed6,868,6663,609,211,997525.46635failed772,947368,012,963476.12580
total7,641,6133,977,224,960520.47629
[0051]
2.样品数据拆分
[0052]
表4多样品纳米孔测序拆分情况
[0053]
categorybarcodetagtotal ntotal bases(bp)0%barcode121,033,284406,155,0640.05%barcode111,180,906406,802,0310.50%barcode101,209,778457,299,2215%barcode091,319,984583,599,269
[0054]
3.低质量测序数据过滤
[0055]
分析第二步是过滤掉测序数据中产生的一些接头序列或者低质量片段,使用porechop和nanofilt软件进行质量过滤。porechop主要用于去除纳米孔测序中测序接头序列,nanofilt选择去除平均质量得分小于7且长度小于200 bp的测序片段,得到表5所示的分析数据。
[0056]
表5分析数据
[0057]
categorytotal ntotal bases(bp)clean readsclean bases0%1,033,284406,155,064969,462398,467,6030.05%1,180,906406,802,0311,022,682390,569,1280.50%1,209,778457,299,2211,102,093445,803,3775%1,319,984583,599,2691,262,248576,286,871
[0058]
三、病毒种类鉴定
[0059]
将分析数据与病毒序列数据库比对:使用三代测序数据比对软件 minimap2使用默认参数将去除低质量后的测序数据与本地病毒参考基因组数据库进行比对,筛选比对质量值大于50且测序片段80%部分均可比对上参考序列的比对数据值,按照比对结果,鉴定病毒的种类或者株系类型,得到表6所示的鉴定结果。
[0060]
表6鉴定结果
[0061]
样品病毒来源序列数病毒来源序列碱基总数检测病毒数5%254,923107,657,124150.5%26,82510,996,148150.05%3,4161,385,510150%000
[0062]
四、检测报告生成:
[0063]
生成的报告如图5和图6所示。
[0064]
本发明不局限于上述最佳实施方式,任何人在本发明的启示下都可得出其他各种形式的产品,但不论在其形状或结构上作任何变化,凡是具有与本技术相同或相近似的技术方案,均落在本发明的保护范围之内。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1