与母猪产死胎性状相关的SNP分子标记及其应用
与母猪产死胎性状相关的snp分子标记及其应用
技术领域
1.本发明属于猪分子标记技术领域,具体涉及一种与母猪产死胎性状相关的snp分子标记及其应用。
背景技术:
2.在我国,猪肉产量和销量位居世界前列,受疫病影响导致生猪存栏量及可繁母猪存栏量显著降低,在这种局势下,提高母猪繁殖性能显得尤为重要。繁殖性状是母猪重要经济性状之一,针对影响目标性状的分子标记位点实施分子育种,能够有效提高育种效率及母猪的繁殖性能(王源,2018)。近年来高通量测序技术、基因芯片技术发展迅速,这些相关技术的发展也作为实施高效育种方法来提高母猪繁殖性能的前提和基础(hickey et al,2017),且评估发现用全基因组测序数据填充snp分子标记芯片数据能提高基因组选择等相关分析的准确性(李勇等,2022)。目前全基因组关联分析已成为标记辅助育种的重要手段之一,通过关联分析从全基因组范围内筛选出控制母猪繁殖性状的重要变异位点,为揭示其相关遗传机理提供理论依据,从而达到辅助育种及改善母猪繁殖性能的目的(赵德胜等,2018)。然而,大多数对母猪繁殖性状的研究致力于增加窝产仔数,与此同时容易忽略了仔猪出生时的存活率。ding等人基于基因芯片数据对美系杜洛克和加系杜洛克的窝性状进行全基因组关联分析,发现位于7号染色体上存在一个显著关联的多效性snp,同时影响美系杜洛克的产死胎数和初生仔猪存活率(ding et al,2021)。verardo等人对大白猪产死胎数和乳头数进行全基因组关联分析,分析结果得出有18个snp与产死胎数显著相关,并找到7个影响大白猪产死胎数的候选基因(verardo et al,2016)。
3.本发明使用gmat软件中的uvlmm分析模块进行全基因组关联分析(wang et al,2020),筛选出与母猪产死胎性状显著相关的snp分子标记位点。
技术实现要素:
4.本发明的目的在于克服现有技术的缺陷,利用基因芯片技术进行基因分型,与母猪产死胎性状进行全基因组关联分析,筛选出6个与母猪产死胎性状显著相关的snp,为母猪繁殖性状提供了新的snp分子标记资源。
5.为了实现上述目的,本发明采用以下技术方案:
6.申请人通过全基因组关联分析筛选得到与母猪产死胎性状显著相关的snp分子标记,并依据ensmble数据库猪11.1版本参考基因组得到每个snp上下游100bp的核苷酸序列,具体如下:
7.含有snp1标记的核苷酸序列如seq id no.1和2所示,snp1标记位于所述序列的第101位,对应于猪基因组15号染色体第68424849位,对应的基因为slc4a10,多态性位点为c或t;该位点的基因型为tt时,母猪拥有高的产死胎率;
8.含有snp2标记的核苷酸序列如seq id no.3和4所示,snp2标记位于所述序列的第101位,对应于猪基因组15号染色体第68442233位,对应的基因为slc4a10,在dbsnp数据库
中的登录号为rs341306021,多态性位点为t或c;该位点的基因型为cc时,母猪拥有高的产死胎率;
9.含有snp3标记的核苷酸序列如seq id no.5和6所示,snp3标记位于所述序列的第101位,对应于猪基因组15号染色体第68516011位,对应的基因为slc4a10,多态性位点为g或a;该位点的基因型为aa时,母猪拥有高的产死胎率;
10.含有snp4标记的核苷酸序列如seq id no.7和8所示,snp4标记位于所述序列的第101位,对应于猪基因组15号染色体第68547427位,对应的基因为slc4a10,在dbsnp数据库中的登录号为rs318344759,多态性位点为g或a;该位点的基因型为aa时,母猪拥有高的产死胎率;
11.含有snp5标记的核苷酸序列如seq id no.9和10所示,snp5标记位于所述序列的第101位,对应于猪基因组15号染色体第68585156位,对应的基因为slc4a10,多态性位点为g或a;该位点的基因型为aa时,母猪拥有高的产死胎率;
12.含有snp6标记的核苷酸序列如seq id no.11和12所示,snp6标记位于所述序列的第101位,对应于猪基因组15号染色体第69126372位,对应的基因为slc4a10,多态性位点为a或t;该位点的基因型为tt时,母猪拥有高的产死胎率。
附图说明
13.图1:实施例2制作的曼哈顿图,将gwas分析的结果可视化。阈值线以上共有13个snp,其中5号和7号染色体上各有一个snp为lncrna,18号染色体上有4个snp位于基因间区,属于非编码区。其余7个位于基因编码区的为本发明筛选出的与大白母猪产死胎性状显著相关的snp分子标记。
14.图2:实施例2制作的qq图,通过比较实际的-log(p)值和期望的-log(p)值概率分布的分位数从而对两个概率分布进行比较,用于进一步判断gwas结果的可靠性。
具体实施方式
15.实施例1基因分型检测及数据处理
16.(1)dna提取
17.①
采集1175头大白母猪的耳朵组织,将采集到的耳样组织捣碎置于玻璃匀浆器中,加入等体积的由100mmol/l的tris饱和酚、500mmol/l的乙二胺四乙酸二钠(edta)、20mmol/l的氯化钠(nacl)、10%浓度的十二烷基硫酸钠(sds)、20μg/ml的胰rna酶配置成的细胞裂解缓冲液,再加入10ng/ml的蛋白酶k,混匀,置于65℃恒温水浴锅中水浴30min;
18.缓慢振荡离心管15min,置于离心机中以12000rpm离心5min后,取上清
19.液入另一离心管中;
20.②
加入等体积的苯酚、氯仿、异戊醇(体积比为25:24:1),振荡混匀,置于离心机中以12000rpm离心5min后,取上清液入另一离心管中;
21.③
再加入等体积的苯酚、氯仿、异戊醇(体积比为25:24:1),振荡混匀,置于离心机中以12000rpm离心10min,取上清液入另一离心管中;
22.④
加入2倍体积的预冷无水乙醇,静置待乙醇挥发后,挑出dna沉淀并使用超纯水溶解dna;
23.⑤
用dna浓度测定仪和琼脂糖凝胶电泳检测dna质量。
24.(2)基因分型检测
25.①
根据上述所提取的dna,使用illumina公司研制的50k snp全基因组芯片进行基因分型,该芯片包含超过50000个snp分子标记位点;
26.②
基于152头大白母猪全基因组重测序数据,对测序原始数据进行基因分型处理,并使用plink v1.9软件对得到的snp分子标记位点进行质控,剔除检出率小于90%、最小等位基因频率小于0.05的snp,最终有17985419个snp用于基因型填充。
27.(3)基因型填充
28.使用beagle v5.1软件,以大白猪全基因组重测序数据为参考群,对芯片数据进行填充,筛选出10197628个填充准确性高于0.8的snp分子标记位点用于后续分析。
29.实施例2snp分子标记与大白母猪产死胎性状的全基因组关联分析
30.本发明中的序列和全基因组关联分析结果,是基于猪基因组11.1版本的信息
31.(1)表型定义
32.母猪产死胎视为case-control性状,根据原表型记录中每头母猪每胎产死仔猪的情况,将母猪有产两胎及以上死胎作为case,母猪未产死胎或只产一胎死胎作为control,case-control构建新表型用于后续全基因组关联分析。
33.(2)snp分子标记质控
34.根据上述基因型填充后筛选出的可靠snp分子标记位点,使用plink v1.9软件分别对填充后的case表型和control表型的snp进行质控,针对case表型的snp数据剔除最小等位基因频率小于0.01的snp,针对control表型的snp数据剔除最小等位基因频率小于0.01的snp,合并后取完全重叠的snp分子标记。最终有1175个样本和9409373个snp用于全基因组关联分析。
35.(3)大白母猪产死胎性状全基因组关联分析
36.本实施例全基因组关联分析所用的实验猪群为大白猪,包括1175头母猪。根据实验猪群所记录的产死胎信息,基于混合线性模型的方法,使用gmat软件中的uvlmm分析模块,对snp分子标记与大白母猪的产死胎性状进行全基因组关联分析。具体模型如下所示:
37.y=μ+wα+xβ+zu+e
38.其中,y为表型向量;μ为群体均值;w为固定效应设计矩阵;α为固定效应向量的分类变量;x为snp向量;β为snp效应;z为设计矩阵;u为个体效应;e为随机残差。
39.根据gwas的结果将每个snp位点p值计算为-log(p),根据显著阈值线计算公式-log(1/snp数)计算,本发明计算得每个snp位点的-log(p)值大于等于6.973561时表明该snp与性状之间存在显著关联,并使用r语言的cmplot包绘曼哈顿图和qq图(图1,图2)。
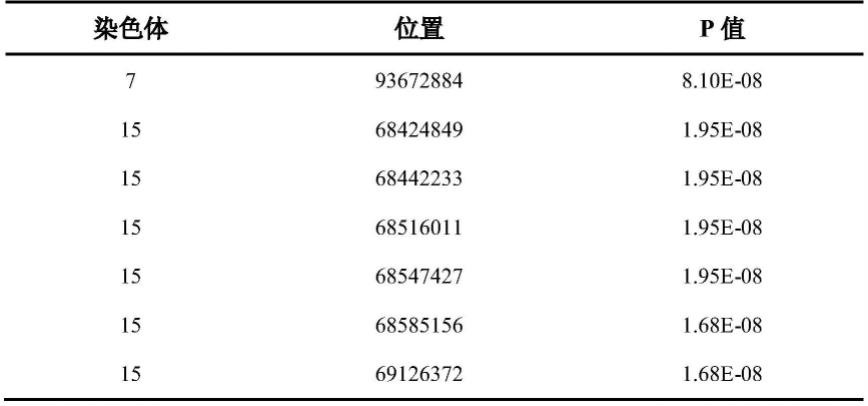
40.表1:基于gwas鉴定的大白母猪产死胎性状的候选snp位点
[0041][0042]
表2:候选snp位点在大白母猪中的基因型频率和产死胎率分布
[0043][0044]
表2说明:括号内为该基因型母猪有2胎及以上产死胎的频率。
[0045]
如表1所述,筛选出7个与母猪产死胎性状显著关联的snp位点,进一步统计每个snp位点不同基因型的个体数以及对应基因型产2胎及以上死胎的个体数,并计算不同基因型的产死胎率(产2胎及以上死胎的个体数/该基因型个体数)。由表2可知,对于15:68424849、15:68442233、15:68516011、15:68547427、15:68585156、15:69126372这6个snp位点,基因型分别为tt、cc、aa、aa、aa、tt时,母猪有2胎及以上产死胎的频率要高于其他基因型的个体;对于7:93672884这个snp位点,由于在1175头母猪中未出现隐性纯合aa基因型的个体,无法判断哪种基因型对母猪产死胎频率的影响。
[0046]
主要参考文献:
[0047]
[1]李勇,杨漫漫,苗泽圃,沈俊燃,陈涛,魏强.低深度全基因组测序在大白猪繁殖性状基因组选择的应用[j].农业生物技术学报,2022,30(02):325-334.
[0048]
[2]王源.猪繁殖性状全基因组关联分析及拷贝数变异检测[d].中国农业大学,2018.
[0049]
[3]赵德胜,张崇志.家畜全基因组关联分析研究进展[j].中国畜牧兽医,2018,45
(02):463-470.
[0050]
[4]ding r,qiu y,zhuang z,ruan d,wu j,zhou s,ye j,cao l,hong l,xu z,zheng e,li z,wu z,yang j.genome-wide association studies reveals polygenic genetic architecture of litter traits in duroc pigs[j].theriogenology.2021,173:269-278.
[0051]
[5]hickey jm,chiurugwi t,mackay i,powell w.genomic prediction unifies animal and plant breeding programs to form platforms for biological discovery[j].nat genet,2017,49:1297-1303.
[0052]
[6]verardo ll,silva ff,lopes ms,madsen o,bastiaansen jw,knol ef,kelly m,varona l,lopes ps,se.revealing new candidate genes for reproductive traits in pigs:combining bayesian gwas and functional pathways[j].genet sel evol,2016,48:9.
[0053]
[7]wang d,tang h,liu jf,xu s,zhang q,ning c.rapid epistatic mixed-model association studies by controlling multiple polygenic effects[j].bioinformatics,2020,36:4833-4837.
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1