一种简化莱茵衣藻叶绿体基因组设计与合成组装的方法及应用
1.本发明涉及生物技术以及合成生物学领域,尤其涉及一种简化莱茵衣藻叶绿体基因组设计与合成组装的方法及应用。
背景技术:
2.莱茵衣藻(chlamydomonas reinhardtii)是一种单细胞真核鞭毛藻类,是研究多种生命活动(如光合作用、鞭毛组装、趋光性和生理节律等)的模式生物,与酵母细胞有诸多共同特征,素有“光合酵母”之称。莱菌衣藻具有细胞核基因组、叶绿体基因组和线粒体基因组3套遗传系统,是少数3套遗传系统均可方便有效地进行遗传转化的模式物种之一。由于莱茵衣藻具有培养条件简单、生长周期短、光合效率高和遗传背景清楚等特点,莱茵衣藻被认为是具有广泛应用前景的转基因生物反应器。
3.合成生物学(synthetic biology)是以工程化设计理念,对生物体进行有目标的设计、改造乃至重新合成。对基因组信息的“编写”将引发以基因组设计与化学合成为标志的第三次生物技术革命,打破了非生命物质与生命的界限,开启了“设计生命、再造生命和重塑生命”的进程,推动生命科学由理解生命向创造生命延伸,可望在人类健康、环境、能源、农业等领域产生革命性发展[cello j,paul a v,wimmere.chemical synthesis of poliovirus cdna:generation of infectious virus in the absence of natural template[j].science,297(5583):1016-8]。叶绿体基因组为合成生物学领域提供了独特的机会。叶绿体基因组在一个相对较小的单独空间里自成一套遗传体系,主要编码与光合作用密切相关的一些蛋白和一些核糖体蛋白,大小范围在150-205kb之间,许多类群的叶绿体基因组已完成测序,并且在植物和藻类中,叶绿体遗传转化和表达技术已非常成熟[smith h o,hutchison c a,pfannkoch c,et al.generating a synthetic genome by whole genome assembly:?x174 bacteriophage from synthetic oligonucleotides[j].proceedings of the national academy of sciences,2003,100(26):15440-15445.]。因此,这些天然的较小的可操控的基因组是食品,能源,医药等多种生物制品领域基因工程改造的理想材料,是合成生物学的理想目标[chan ly,kosuri s,endy d.refactoring bacteriophage t7.mol syst biol.2005;1:2005.0018.]。
[0004]
在探索生命进化和演化的过程中,最小基因组一直是研究人员追寻的目标。适度精简生物的基因组能够优化细胞代谢途径,改善对底物、能量的利用效率,极大的提高预测可控制细胞生理性能的可能性。莱茵衣藻基因组中具有约20%的重复dna序列,这些序列是否具有生物学功能,具有何种生物学功能,之前仅有零星的报道。例如有研究指出,删除peta与petd之间的重复片段,不影响基因的正常生物学功能,但转录出的mrna结构不同;删除atpb、psab基因附近的重复片段,则影响光合作用。然而目前,尚未有研究人员从整个叶绿体基因组出发,全局改造这些重复片段,探索重复片段的功能。因此,现有技术还有待改进。
技术实现要素:
[0005]
鉴于上述现有技术的不足,本发明提供了一种简化莱茵衣藻叶绿体基因组设计与合成组装的方法及应用,旨在通过删减非必要基因,删除冗余重复片段,精简莱茵衣藻叶绿体基因组,为医药、能源、环境、农业等方面的研究与生产提供全新的底盘细胞和优化后的平台载体。
[0006]
本发明的技术方案如下:
[0007]
一种简化莱茵衣藻叶绿体基因组设计与合成组装的方法,其中,所述方法包括简化莱茵衣藻叶绿体基因组的理性设计、合成组装、功能验证以及应用;其中,基因组的理性设计包括删除莱茵衣藻叶绿体基因组中短重复片段,插入noti和asci限制性内切酶位点,插入抗性筛选标记,添加pcr水印标签。
[0008]
所述的简化莱茵衣藻叶绿体基因组设计与合成组装的方法,其中,基因组的理性设计包括删除莱茵衣藻叶绿体基因组中短重复片段59处,插入7个noti和4个asci限制性内切酶位点,插入aada、aphviii和cat抗性筛选标记,添加28对pcr水印标签。
[0009]
所述的简化莱茵衣藻叶绿体基因组设计与合成组装的方法,其中,设计的简化莱茵衣藻叶绿体基因组全长为186,890bp。
[0010]
所述的简化莱茵衣藻叶绿体基因组设计与合成组装的方法,其中,将所述简化莱茵衣藻叶绿体基因组分成65个一级片段进行合成,每个一级片段两端各带有apai限制性酶切位点,两两片段之间依次有80bp的同源臂。
[0011]
所述的简化莱茵衣藻叶绿体基因组设计与合成组装的方法,其中,所述一级片段全部通过化学方法合成。
[0012]
所述的简化莱茵衣藻叶绿体基因组设计与合成组装的方法,其中,所述简化莱茵衣藻叶绿体基因组的合成组装包括如下步骤:
[0013]
6.1将所述简化莱茵衣藻叶绿体基因组分成65个一级片段进行合成;
[0014]
6.2利用酵母将合成的65个一级片段组装成14个二级片段;
[0015]
6.3将14个二级片段组装为三个三级片段质粒;
[0016]
6.4将所述三级片段质粒转入到不同的酵母菌株,并利用酵母杂交分孢的特性将所述三级片段质粒合并到一个酵母菌株中;
[0017]
6.5利用核酸内切酶i-scei诱导体内同源重组,得到所述简化莱茵衣藻叶绿体基因组。
[0018]
所述的简化莱茵衣藻叶绿体基因组设计与合成组装的方法,其中,所述简化莱茵衣藻叶绿体基因组的功能验证包括如下步骤:
[0019]
7.1将所述简化莱茵衣藻叶绿体基因组转入大肠杆菌中扩繁,并提取质粒dna;
[0020]
7.2利用基因枪将所述质粒dna转化进入莱茵衣藻叶绿体中;
[0021]
7.3经转化后的莱茵衣藻细胞在含壮观霉素的培养平板筛选,挑取绿色单克隆;
[0022]
7.4利用预先设计的水印标签对所述绿色单克隆进行检测,选取叶绿体基因组被替换为所述简化莱茵衣藻叶绿体基因组的阳性转化子;
[0023]
7.5检测所述阳性转化子的生物学活性。
[0024]
所述的简化莱茵衣藻叶绿体基因组设计与合成组装的方法,其中,基因枪使用参数为:金粉颗粒直径1.0μm,可裂膜1100psi,轰击距离9厘米,dna浓度1μg/μl。
[0025]
一种简化莱茵衣藻叶绿体基因组的应用,其中,将所述的简化莱茵衣藻叶绿体基因组应用于细胞工程或代谢工程,其中,所述的简化莱茵衣藻叶绿体基因组按照如上任一所述的简化莱茵衣藻叶绿体基因组设计与合成组装的方法制备而来。
[0026]
所述的简化莱茵衣藻叶绿体基因组的应用,其中,将β类胡萝卜素代谢通路应用模块整合到所述的简化莱茵衣藻叶绿体基因组,进行目标产物的生物合成。
[0027]
有益效果:本发明提供了一种简化莱茵衣藻叶绿体基因组设计与合成组装的方法及应用。所述方法包括简化莱茵衣藻叶绿体基因组的理性设计、合成、组装、功能验证以及应用。本发明通过删除莱茵衣藻叶绿体基因组中短重复片段,插入noti和asci限制性内切酶位点,插入抗性筛选标记,添加pcr水印标签,设计得到了简化的莱茵衣藻叶绿体基因组。本发明选择莱茵衣藻叶绿体基因组作为研究对象,基于莱茵衣藻天然的细胞器叶绿体基因组,重新设计和人工构建莱茵衣藻的叶绿体基因组,通过移除冗余序列,改变、添加全新的遗传元件或代谢通路等实现对基因组的全面改造,并对莱茵衣藻叶绿体基因组的设计缺陷和序列缺陷进行修复,实现化学合成与设计序列的精确匹配,验证并评价莱茵衣藻叶绿体基因组的设计原则。本发明首次全叶绿体基因组范围内探索短分散重复序列的功能,并从整个叶绿体基因组出发,全局改造这些重复片段,探索这些重复片段的功能,为阐释这些重复片段的起源、生物学意义以及在进化中的作用提供依据;首次尝试通过删减非必要基因,删除冗余重复片段,精简莱茵衣藻叶绿体基因组;首次尝试在叶绿体基因组中组装代谢通路。本发明提供的方法,可实现叶绿体的定制化构建,为医药、能源、环境、农业等方面的研究与生产提供全新的底盘细胞和优化后的平台载体。
附图说明
[0028]
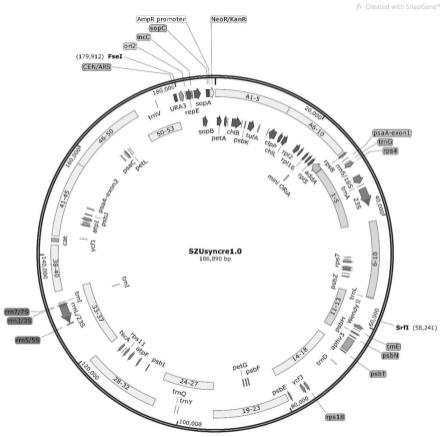
图1为本发明实施例提供的简化莱茵衣藻叶绿体基因组的图谱。
[0029]
图2为本发明实施例提供的原基因组与简化莱茵衣藻叶绿体基因组中sdrs含量对比图。
[0030]
图3为本发明实施例提供的pcr验证酵母组装的简化莱茵衣藻叶绿体基因组序列结果示意图。
[0031]
图4为本发明实施例提供的转基因衣藻删除位置上的酶切位点结果示意图。
[0032]
图5为本发明实施例提供的水印pcr标签检测结果示意图。
[0033]
图6为本发明实施例提供的转化子莱茵衣藻与野生型衣藻生长曲线图。
[0034]
图7为本发明实施例提供的野生型及转化衣藻中的β类胡萝卜素含量图。
具体实施方式
[0035]
本发明提供一种简化莱茵衣藻叶绿体基因组设计与合成组装的方法及应用,为使本发明的目的、技术方案及效果更加清楚、明确,以下对本发明进一步详细说明。应当理解,此处所描述的具体实施例仅仅用以解释本发明,并不用于限定本发明。
[0036]
本发明实施例提供一种简化莱茵衣藻叶绿体基因组设计与合成组装的方法,所述方法包括简化莱茵衣藻叶绿体基因组的理性设计、合成组装、功能验证以及应用。
[0037]
莱茵衣藻叶绿体基因组具有代表性,其基因组结构与小球藻、拟南芥、短柄草及水稻等中的叶绿体基因组结构类似,研究其人工合成装配策略对理解其他物种的叶绿体基因
组人工改造具有较好的参考价值。而且,莱茵衣藻叶绿体基因组又具有其独特的特征,含有自身含量约20%的重复dna序列,大多数基因间区域由多种类型的短分散重复序列(sdr)组成,这些重复序列可能具有结构或进化意义,这将对叶绿体的基因功能及进化研究提供新的证据。因此,本发明实施例选择莱茵衣藻叶绿体基因组作为研究对象,从整个叶绿体基因组出发,首次在全叶绿体基因组范围内探索短分散重复序列的功能,全局改造这些重复片段,探索这些重复片段的功能,为阐释这些重复片段的起源、生物学意义以及在进化中的作用提供依据
[0038]
本发明实施例基于莱茵衣藻天然的细胞器叶绿体基因组,重新设计和人工构建简化版的莱茵衣藻的叶绿体基因组,通过移除冗余序列,改变、添加全新的遗传元件或代谢通路等实现对基因组的全面改造,并对莱茵衣藻叶绿体基因组的设计缺陷和序列缺陷进行修复。采用合成生物学的方法,利用全化学合成设计的简化莱茵衣藻叶绿体基因组片段,在酵母-细菌系统中,实现简化版叶绿体基因组的全化学从头合成及组装。然后将全化学合成的简化版叶绿体基因组转化衣藻细胞,通过多种技术手段,替换叶绿体原基因组,实现全化学合成简化莱茵衣藻叶绿体基因组的生物学功能。
[0039]
在一些实施方式中,基因组的设计包括删除莱茵衣藻叶绿体基因组中短重复片段,插入noti和asci限制性内切酶位点,插入抗性筛选标记,添加pcr水印标签。
[0040]
在一些具体的实施方式中,基因组的设计包括删除莱茵衣藻叶绿体基因组中短重复片段(sdrs)59处,总计碱基数量32485bp;插入特殊酶切位点11个,包括7个noti和4个asci限制性内切酶位点;插入抗性筛选标记3个,包括aada、aphviii和cat抗性筛选标记;添加28对pcr水印标签。最终设计的简化莱茵衣藻叶绿体基因组全长为186,890bp。
[0041]
具体的,本发明实施例基于野生型莱茵衣藻叶绿体基因组(nc_005353.1)进行改进。
[0042]
第一,删减野生型莱茵衣藻叶绿体基因组中段重复片段:利用生物信息学软件vmatch计算野生型莱茵衣藻叶绿体基因组中重复片段位置,参数设置如下:
[0043]
vmatch-d-absolute-l 20cre.cp.mt.genomes.fasta》result
[0044]
一共删减碱基32485bp。
[0045]
第二,插入特殊酶切位点11个,包括7个noti(gcggccgc)酶切位点和4个asci(ggcgcgcc)酶切位点。
[0046]
第三,插入抗性筛选标记表达框3个,包括aada、aphviii、cat。
[0047]
第四,添加pcr水印标签28对。
[0048]
最终,设计合成的简化莱茵衣藻叶绿体基因组大小为186,890bp,图谱如图1所示。设计过程中,一共删减碱基32485bp,添加碱基6099bp,设计完成后,简化莱茵衣藻叶绿体基因组基本不含有短重复片段。
[0049]
本发明实施例基于莱茵衣藻天然的叶绿体基因组,理性设计和人工构建简化版的莱茵衣藻叶绿体基因组,通过移除冗余序列,改变或添加全新的遗传元件或代谢通路等实现对基因组的全面改造。并对莱茵衣藻叶绿体基因组的设计缺陷和序列缺陷进行修复,实现化学合成与设计序列的精确匹配,验证并评价莱茵衣藻叶绿体基因组的设计原则,在此基础上实现叶绿体的定制化构建。本发明实施例首次尝试通过删减非必要基因,删除无用重复片段,精简莱茵衣藻叶绿体基因组,为医药、能源、环境、农业等方面的研究与生产提供
全新的底盘细胞和优化后的平台载体。
[0050]
在一些实施方式中,将所述简化莱茵衣藻叶绿体基因组分成65个一级片段进行合成,每个一级片段两端各带有apai限制性酶切位点,两两片段之间依次有80bp的同源臂。所述一级片段全部通过化学方法合成。
[0051]
目前人工合成碱基性价比较合适的长度为3kb左右,因此将设计的叶绿体基因组片段交由生物合成公司,每个一级片段长度约为2.8k,首尾片段带有酵母大肠杆菌穿梭质粒同源片段约80bp,其余片段含有上下游的同源片段80bp。
[0052]
在一些实施方式中,所述简化莱茵衣藻叶绿体基因组的合成组装包括如下步骤:
[0053]
s10、将所述简化莱茵衣藻叶绿体基因组分成65个一级片段进行合成;
[0054]
s20、利用酵母将合成的65个一级片段组装成14个二级片段;
[0055]
s30、将14个二级片段组装为三个三级片段质粒;
[0056]
s40、将所述三级片段质粒转入到不同的酵母菌株,并利用酵母杂交分孢的特性将所述三级片段质粒合并到一个酵母菌株中;
[0057]
s50、利用核酸内切酶i-scei诱导体内同源重组,得到所述简化莱茵衣藻叶绿体基因组。
[0058]
本发明实施例利用酵母高效的同源重组功能,在酵母体内组装上述构建的简化莱茵衣藻叶绿体基因组一级片段。利用每个片段上预留的同源臂,在酵母体内将65个一级片段组装成14个二级片段,得到14个二级片段质粒,每个二级片段质粒含有5个左右的一级片段。之后,再将二级片段质粒组装成大的三级片段质粒,每5个左右二级片段质粒组装成一个三级片段质粒。14个二级片段质粒经组装后得到3个三级片段质粒。通过酵母杂交,将3个三级片段质粒导入一个酵母之中,诱导预留的限制性内切酶表达,使得3个三级片段质粒在酵母体内完成组装,成功构建简化莱茵衣藻叶绿体基因组。
[0059]
在一些实施方式中,所述简化莱茵衣藻叶绿体基因组的功能验证包括如下步骤:
[0060]
s100、将所述简化莱茵衣藻叶绿体基因组转入大肠杆菌中扩繁,并提取质粒dna;
[0061]
s200、利用基因枪将所述质粒dna转化进入莱茵衣藻叶绿体中;
[0062]
s300、经转化后的莱茵衣藻细胞在含壮观霉素的培养平板筛选,挑取绿色单克隆;
[0063]
s400、利用预先设计的水印标签对所述绿色单克隆进行检测,选取叶绿体基因组被替换为所述简化莱茵衣藻叶绿体基因组的阳性转化子;
[0064]
s500、检测所述阳性转化子的生物学活性。
[0065]
在一些具体的实施方式中,基因枪使用参数为:金粉颗粒直径1.0μm,可裂膜1100psi,轰击距离9厘米,dna浓度1μg/μl。
[0066]
在一些具体的实施方式中,筛选平板培养基中含有150μg/ml的壮观霉素。
[0067]
在一些具体的实施方式中,检测所述阳性转化子的生物学活性包括含简化叶绿体基因组的衣藻生长状况、叶绿体的光合作用效率、叶绿体基因的转录模式以及基因表达情况。
[0068]
本发明实施例利用基因枪将上述质粒dna注入衣藻叶绿体中,通过抗性平板筛选阳性克隆。筛选平板培养基中含有150μg/ml的壮观霉素,如果藻细胞能在筛选平板上长出单克隆藻落,说明简化叶绿体基因组已经转到叶绿体中。再利用设计的水印标签检测单克隆藻落中衣藻叶绿体基因组是否全部替换为人工设计合成的简化莱茵衣藻叶绿体基因组
序列,并选取全部替换的阳性转化子。之后,检测阳性转化子的生物学活性,包括含简化叶绿体基因组叶绿体的光合作用效率、叶绿体基因的转录模式以及基因表达情况。
[0069]
本发明实施例还提供一种简化莱茵衣藻叶绿体基因组的应用,将按照上述方法制备而来的简化莱茵衣藻叶绿体基因组应用于细胞工程或代谢工程。
[0070]
在一些实施方式中,将β类胡萝卜素代谢通路等应用模块整合到所述的简化莱茵衣藻叶绿体基因组,进行目标产物(如β类胡萝卜素等)的生物合成。具体的,将所述的简化莱茵衣藻叶绿体基因组应用于β类胡萝卜素代谢通路的导入,在莱茵衣藻中增加类胡罗卜素的含量。
[0071]
具体的,可利用预留的位点,通过酵母将代谢路径导入简化莱茵衣藻叶绿体基因组。将装好的β类胡萝卜素合成路径基因片段插入本发明实施例合成的一级片段f5中,在一级片段f5中有一个bamhi酶切位点,β类胡萝卜素合成路径基因片段在设计时也在两端插入了bamhi酶切位点,利用酶切连接的方法可以将β类胡萝卜素合成路径基因片段插入到一级片段f5中,形成新的f5序列。然后按照所述的组装方法,可以得到一个含有β类胡萝卜合成路径的改进的简化莱茵衣藻叶绿体基因组。
[0072]
理性设计合成莱茵衣藻叶绿体基因组的一个目的在于能够为医药、能源等领域提供全新的底盘细胞器和设计的代谢途径,因此本发明实施例以β类胡萝卜为例,在叶绿体组装过程中,设计一条β类胡萝卜素的代谢路径,装配至设计的叶绿体基因组中,以此为例实现叶绿体的定制化构建。
[0073]
下面通过具体实施例对本发明一种简化莱茵衣藻叶绿体基因组设计与合成组装的方法及应用做进一步的解释说明:
[0074]
若无特别说明,本发明实施例所用到的材料、试剂等均可以通过商业途径购买;若无特别说明,本发明实施例所采用的方法、流程等均为本领域常规技术或本领域技术人员可以根据公知常识或常规技术进行合理设置。
[0075]
实施例1简化莱茵衣藻叶绿体基因组的设计
[0076]
从ncbi下载野生型莱茵衣藻叶绿体基因组序列(nc_005353.1),大小为205,503bp。在野生型叶绿体基因组序列的基础上对其进行设计,包括删除莱茵衣藻叶绿体基因组中短重复片段,插入noti和asci限制性内切酶位点,插入aada、aphviii和cat抗性筛选标记,添加pcr水印标签。
[0077]
1、删减莱茵衣藻叶绿体基因组中重复片段
[0078]
利用生物信息学软件vmatch计算莱茵衣藻叶绿体基因组中重复片段位置,参数设置如下:
[0079]
vmatch-d-absolute-l 20cre.cp.mt.genomes.fasta》result
[0080]
具体删除的位置信息如表1所示,一共删减碱基32485bp。
[0081]
表1莱茵衣藻叶绿体基因组删除的位置信息表
[0082]
[0083][0084][0085]
2、插入特殊酶切位点
[0086]
共插入特殊酶切位点11个,包括7个noti(gcggccgc)酶切位点,插入位置为基因组(3159-3166;8978-8985;15735-15742;17522-17529;19146-19153;23461-23468;179059-179066)和4个asci(ggcgcgcc)酶切位点,插入位置为基因组(1728-1735;18726-18733;20178-20185;24451-24458)。
[0087]
3、插入抗性筛选标记表达框
[0088]
插入3个抗性筛选标记表达框,包括aada、aphviii、cat,其中,aada的核苷酸序列如seq.id no.1所示,aphviii的核苷酸序列如seq.id no.2所示,cat的核苷酸序列如seq.id no.3所示。
[0089]
4、添加pcr水印标签
[0090]
添加pcr水印标签28对。
[0091]
最终,设计合成的简化莱茵衣藻叶绿体基因组大小为186,890bp,图谱如图1所示。设计过程中,一共删减碱基32485bp,添加碱基6099bp。设计完成后,简化莱茵衣藻叶绿体基因组基本不含有短重复片段,对比结果见图2,以基因组约30kb序列结果展示。
[0092]
实施例2简化莱茵衣藻叶绿体基因组的合成组装
[0093]
1、一级片段的化学合成
[0094]
将设计得到的186,890bp简化莱茵衣藻叶绿体基因组序列,分成65个一级片段进行合成,每个片段长2880bp,两端各带有apai限制性酶切位点,可以通过直接酶切合成质粒得到用于组装的片段,两两片段之间依次有80bp的同源臂。所有一级片段委托商业公司合成,且序列均经过测序和酶切确认。
[0095]
2、二级片段质粒组装
[0096]
鉴于合成的简化莱茵衣藻叶绿体基因组一级片段数目较多,为了确保组装效率,利用酵母将65个一级片段先组装成14个二级片段质粒,每个质粒约14kb。
[0097]
14个二级片段质粒在by4741背景菌株(商业化菌株,购于辉诺生物医药科技有限公司,货号:a226)进行组装,每个二级片段质粒包括4-5个叶绿体基因组一级片段、1个酵母细菌穿梭质粒骨架,其中prs416(商业化质粒,购于acd,货号:518681-c2)作为二级片段质粒的载体,ura3基因提供营养缺陷型筛选标记,具体组装过程如下:
[0098]
(1)片段准备:通过酶切的方法得到合成的一级片段,通过pcr扩增的方法得到酵母细菌穿梭质粒骨架,将酶切产物及pcr扩增产物回收备用;
[0099]
(2)转化:将上述回收产物共转化到by4741酵母细胞中,用sc-ura平板筛选阳性克隆;
[0100]
(3)鉴定:随机挑选单克隆进行酵母菌落pcr初筛和基因组复筛,最终经全部junction引物验证。
[0101]
所有14个二级片段质粒均按照上述方法步骤组装完成。
[0102]
3、三级片段质粒的组装
[0103]
(1)三级片段质粒1的组装
[0104]
三级片段质粒1在by4741背景菌株(商业化菌株,购于辉诺生物医药科技有限公司,货号:a226)进行组装,共7个片段,包括4个上述二级片段质粒的叶绿体基因组合成片段、1个bac片段、ura3筛选基因和a10-f46桥接片段。其中bac作为三级片段质粒1的载体,ura3基因提供营养缺陷型筛选标记,a10-f46桥接片段用于在片段a10和片段a10之间插入
i-scei酶切位点。具体组装过程如下:
[0105]
1)片段准备:通过酶切的方法得到合成片段a1-5、a6-10、f46-50和f50-53,通过pcr扩增的方法得到bac片段、ura3筛选基因和a10-f46桥接片段,将酶切产物及pcr扩增产物回收备用。其中bac片段来源于质粒pbelobac11(商业化质粒,购于普如汀生物技术(北京)有限公司,货号:pbelobac11),所用扩增引物如seq id no.4-seq id no.9所示:
[0106]
seq id no.4:1-bac-1f
[0107]
ggcaacgtccattcctatctgtttacggattattaatgtcctcatgtattcatagcaagcacatgtcggccgcctcggcctc
[0108]
seq id no.5:1-bac-1r
[0109]
cctcagtcacttattatcactagcgctcgccgcagccgtgtaaccgagcatagcgagcgaactggcgaggaagcaaagaa
[0110]
seq id no.6:1-bac-2f
[0111]
gatatcgggggttagttcgtcatcattgatgagggttgattatcacagtttattactctgaattggctatccgcgtgt
[0112]
seq id no.7:1-bac-2r
[0113]
gaatacatgaggacattaataatccgtaaacagataggaatggacgttgcctagaaagaatcggccataatggccactctgcg
[0114]
seq id no.8:1-i-seci-f
[0115]
ttttttaaaacgcaagtccttttttaagatagttattattaaccattatgttgcacatactcctagggataacagggtaat
[0116]
seq id no.9:1-i-seci-r
[0117]
ctaaaaaaccgtaagcaatccatgttgtggtaaattctgcaggtttttcaaaaaaacttgaaatattattaccctgttatccctagg
[0118]
2)转化:将上述回收产物共转化到by4741酵母细胞中,用sc-ura平板筛选阳性克隆。
[0119]
3)鉴定:随机挑选单克隆进行酵母菌落pcr初筛和基因组复筛,最终经全部junction引物验证。
[0120]
(2)三级片段质粒2的组装
[0121]
三级片段质粒2在by4742背景菌株(商业化菌株,购于北京兴华越洋生物科技有限公司,货号nrr01120)进行组装,共7个片段,包括6个上述二级片段质粒的叶绿体基因组合成片段和prs415(商业化质粒,购于普如汀生物技术(北京)有限公司,货号:prs415)载体片段。过程与三级片段质粒1的组装类似,除了使用sc-leu平板筛选阳性克隆。经全部junction引物验证,成功在by4742细胞中组装了三级片段质粒2。所用扩增引物如seq id no.10-seq id no.11所示:
[0122]
seq id no.10:2-rs415-f
[0123]
cgacctctttgcatatctgaatcttacaaagaaattcctacaggagaagttagtatttcttagggataacagggtaatcaattcgccctatagtgagt
[0124]
seq id no.11:2-rs415-r
[0125]
gtctctggtaagatttttgttttattaccaagaaaagataataatacaaaccatatatatattaccct
gttatccctacaccgcggtggagctccag
[0126]
(3)三级片段质粒3的组装
[0127]
三级片段质粒3在by4741背景菌株进行组装,共8个片段,包括7个上述二级片段质粒的叶绿体基因组合成片段和prs411(商业化质粒,购于普如汀生物技术(北京)有限公司,货号:biovector number:87474)载体片段,过程与三级片段质粒1的组装类似,除了使用sc-met平板筛选阳性克隆。经全部junction引物验证,成功在by4741细胞中组装了三级片段质粒。所用扩增引物如seq id no.12-seq id no.13所示:
[0128]
seq id no.12:3-rs411-f
[0129]
tagtcaattattaagttcaaaaaatttagttaaatctttaaagcaagcatcgattaatagtagggataacagggtaatcaccgcggtggagctccag
[0130]
seq id no.13:3-rs411-r
[0131]
atattccgctattagtgttacaagttttattaactttgtttaaagattttaaaaaaaccgattaccctgttatccctacaattcgccctatagtgagt
[0132]
4、3个三级片段质粒利用酵母杂交到一个酵母细胞中
[0133]
得到三个三级片段质粒的酵母菌株后,还需要进行两轮杂交过程将其合并到一个酵母菌株中,具体过程如下:
[0134]
将包含三级片段质粒2的酵母菌株中的met基因利用同源交换原理敲除,用kanmx片段替换基因组上的met基因,kanmx片段及替换位置上下游序列见seq id no.14。以pfa6-kanmx4(商业化质粒,购于上海雅吉生物科技有限公司,货号:yc-14391rj)为模板进行pcr扩增得到kanmx,引物序列如seq id no.15-seq id no.16所示:
[0135]
seq id no.15:met17f:
[0136]
gatatcttcggatgcaagggttcgaatcccttagctctcagacatggaggcccagaatac;
[0137]
seq id no.16:met17r:
[0138]
aagtaggtttatacataattttacaactcattacgcacaccagtatagcgaccagcattc。
[0139]
将25μl pcr产物转化到三级片段质粒中(by4742背景),sc-leu+g418筛选;将转化的板子影印到g418的板子上,挑在两种板子上都能长的单克隆,进行pcr验证,验证正确的菌株作为三级片段质粒2进行后续实验。
[0140]
将上述含有三级片段质粒2和三级片段质粒3的酵母菌株进行杂交,通过sc-leu-met平板筛选同时含有三级片段质粒2(leu2)和三级片段质粒3(met17)的二倍体酵母菌株。随机从sc-leu-met平板上挑选二倍体酵母菌株(#1,#2)进行产孢和分孢过程得到单倍体酵母。
[0141]
将ypd平板分别影印到sc-leu、sc-met和sc-ura平板上,sc-leu和sc-met用于筛选同时含有三级片段质粒2和三级片段质粒3的单倍体酵母菌,sc-ura平板用于确保得到的单倍体不会在sc-ura平板上生长,以进行后续三个三级片段质粒的杂交。最终,得到了同时含有三级片段质粒2和三级片段质粒3的单倍体酵母菌。
[0142]
上述筛选得到的单倍体酵母菌的交配型是未知的。为了后续能够与三级片段质粒1进行杂交,需要从中挑选交配型为alpha的单倍体酵母菌株。szu-jdy19(保藏号:cctcc m 20221034)和szu-jdy20(保藏号:cctcc m 20221033)的交配型分别为a和alpha,与其进行杂交的单倍体菌株只有成功杂交成二倍体才能够在sd平板上生长。将上述ypd分孢的平板
分别与szu-jdy19和szu-jdy20进行杂交,然后影印到sd平板上,与szu-jdy19杂交后能在sd平板上生长,与szu-jdy20杂交后不能在sd平板上生长,说明其交配型为alpha,通过这种方法成功筛选到了目的菌株。
[0143]
将上述筛选到的酵母分别与含有三级片段质粒1的酵母菌进行杂交,通过sc-leu-met-ura平板筛选同时含有3个三级片段质粒的酵母菌,从平板上挑选单克隆进行后续实验。
[0144]
5、简化莱茵衣藻叶绿体基因组的组装
[0145]
三个三级片段质粒中两两各有一个同源片段,三级片段质粒1和三级片段质粒2上都含有片段a10,三级片段质粒2和三级片段质粒3都有片段f23,三级片段质粒3和三级片段质粒1都含有片段f46,且在同源片段的一端都含有i-scei切割位点。i-scei是来源于酿酒酵母线粒体内含子编码的一种核酸内切酶,能够特异性识别约18bp的序列,并在识别位点产生一个双链断裂缺口进而激活酵母细胞的同源重组修复机制。通过诱导i-scei在酵母细胞中的表达,将三级片段质粒1、2、3进行线性化,借助酵母细胞的同源重组修复系统,实现以三级片段质粒1的bac载体为载体的完整的叶绿体基因组的组装。
[0146]
将szu-zlp012质粒(保藏号:cctcc m 20221031)(带有i-scei核酸内切酶和his3营养缺陷筛选标记基因)转化到上一步得到的同时含有3个三级片段质粒的酵母中,用sc-ura-leu-met-his平板筛选得到同时含有3个三级片段质粒和szu-zlp012质粒的酵母菌。挑选单克隆划线到sc-ura-his+半乳糖的平板上,利用半乳糖诱导i-scei的表达。挑选单克隆进行酵母菌落pcr初筛和全junction pcr复筛,最终得到了含有简化莱茵衣藻叶绿体基因组的正确菌株(保藏号:cctcc m 20221032),pcr检测结果见图3,其中,简化质粒junction pcr检测结果:a1-5;a6-10;f1-5;f6-10;f11-13;f14-18;f19-23;f24-27;f28-32;f33-37;f38-40;f41-45;f46-50;f51-53。
[0147]
实施例3简化莱茵衣藻叶绿体基因组的转化
[0148]
上述实施例2中验证正确的简化莱茵衣藻叶绿体基因组,转入大肠杆菌,利用大肠杆菌扩繁该基因组,之后提质粒,得到能用于叶绿体转化的高浓度的质粒。
[0149]
该质粒转化莱茵衣藻的方法如下:
[0150]
首先,莱菌衣藻cc-125在tap培养液中培养至对数期,细胞数约为1~2
×
106细胞/ml,室温(20-25℃)离心收集;用tap液体培养基重悬,调整细胞浓度至2
×
l08细胞/ml;吸取300μl悬浮液涂布于tap固体平板培养基,22℃光照培养箱(光照条件为90μe/m2/s)中培养1-2天以形成细胞层。
[0151]
之后,所述衣藻细胞在无菌条件下用基因枪(bio-rad)轰击。使用伯乐台式基因枪pds-1000/he,具体步骤如下:
[0152]
取50μl金粉悬浮液(60μg/ml),加入5μg(1μg/μl)上述合成的环状质粒和50μl 2mcacl2、20μl 0.1m亚精胺,通过涡轮振荡器振荡1-3分钟以混合;8000rpm离心10秒,弃上清;用无水乙醇清洗,振荡,再8000rpm离心10秒,弃上清,共5次;最后用60μl无水乙醇重悬沉淀。
[0153]
每次轰击取10μl,轰击参数如下:真空度25inches-hg,轰击距离9cm,每孤轰击3次,22℃光照培养箱恢复培养12小时,然后转移到筛选平板上继续培养(22℃,90μe/m2/s)1-2周,至绿色单克隆长出。
identification of six genes.fems microbiol.lett.1991,78,157-161.],5个基因的功能均有文献验证。
[0166]
1、β类胡萝卜素合成路径的设计
[0167]
根据报道的叶绿体转录组数据,选取了莱茵衣藻叶绿体基因组自身存在的多基因共表达序列,将其中原基因orf替换成β类胡萝卜素合成路径基因orf,其余序列不变,然后切割成4段3kb左右的片段,送生物公司合成,序列见seq id no.17。
[0168]
2、β类胡萝卜素合成路径的组装
[0169]
片段在by4741背景菌株进行组装,包括4个β类胡萝卜素合成路径基因片段、1个酵母细菌穿梭质粒骨架,其中prs416作为节选片段质粒的载体,ura3基因提供营养缺陷型筛选标记,具体组装过程如下:
[0170]
(1)片段准备:通过酶切的方法得到合成片段,通过pcr扩增的方法得到酵母细菌穿梭质粒骨架,将酶切产物及pcr扩增产物回收备用;
[0171]
(2)转化:将上述回收产物共转化到by4741酵母细胞中,用sc-ura平板筛选阳性克隆;
[0172]
(3)鉴定:随机挑选单克隆进行酵母菌落pcr初筛和基因组复筛,最终经全部junction引物验证。
[0173]
3、β类胡萝卜素合成路径与简化叶绿体基因组的整合
[0174]
将装好的β类胡萝卜素合成路径基因片段插入实施例2中合成的片段f5中,在片段f5中有一个bamhi酶切位点,β类胡萝卜素合成路径基因片段在设计时也在两端插入了bamhi酶切位点,利用酶切连接的方法可以将β类胡萝卜素合成路径基因片段插入到片段f5中,形成新的f5序列,然后按照实施例3的组装方法,得到一个含有β类胡萝卜合成路径的设计合成的莱茵衣藻叶绿体基因组。
[0175]
4、上述得到的含有β类胡萝卜合成路径的设计合成的莱茵衣藻叶绿体基因组转化莱茵衣藻的方法及筛选检测方法与见实施例3,实施例4中所述。
[0176]
5、转化子β类胡萝卜素的检测
[0177]
(1)培养转化衣藻:100ml atp液体培养基灭菌,加入相应的抗生素(壮观霉素100μg/ml);加入1ml生长至对数期的转化衣藻,22℃光照培养箱(光照条件为90μe/m2/s)养至对数期;4℃,4000rpm离心8min,收集藻体;适量ddh2o洗涤藻体一次,去除多余的培养基;冷冻干燥过夜。
[0178]
(2)转化衣藻色素提取:冻干的藻体称量(约25mg),于2.0ml的离心管,加500μl丙酮涡旋吹打;55℃水浴15min,每5分钟涡旋一次;4℃,12000g离心5min,取上清于新的2.0ml的离心管中;重复一次,沉淀中加500μl丙酮再提取一次,尽量将色素提取完全;真空涡旋干燥,加入300μl甲醇:异丙醇(8:2)定量溶解,用0.2μm滤膜(有机)过滤到1.5ml的棕色离心管中,做好标记。
[0179]
(3)hplc检测:在含有内插管的样品瓶中加50μl的样品,进行液相色谱检测。
[0180]
高效液相色谱:agilent technologies,agilent,usa
[0181]
色谱柱:5μ,150
×
3.0mm,phenomenex inc.,aschaffenburg,germany
[0182]
流动相a:ddh2o
[0183]
流动相b:甲醇:异丙醇(8:2)
[0184]
流速:0.6ml/min
[0185]
分析时间:18min
[0186]
梯度洗脱:
[0187]
0min:15%a,85%b
[0188]
0-10min:100%a,0%b
[0189]
10-12min:100%a,0%b
[0190]
12-18min:15%a,85%b
[0191]
采用紫外检测器在最大吸收值280nm,400nm,440nm,450nm,475nm处测吸收峰面积,再根据标准曲线计算产量。
[0192]
通过上述技术手段可以检测到转化衣藻中的β类胡萝卜素。结果表明(见图7),与野生型衣藻相比,转化衣藻中的β类胡萝卜素含量明显增加,表明设计合成的β类胡萝卜素路径正常工作,这种组装策略能够应用到其他代谢通路的设计与合成。
[0193]
综上所述,本发明提供了一种简化莱茵衣藻叶绿体基因组设计与合成组装的方法及应用。所述方法包括简化莱茵衣藻叶绿体基因组的理性设计、合成、组装、功能验证以及应用。本发明通过删除莱茵衣藻叶绿体基因组中短重复片段,插入noti和asci限制性内切酶位点,插入抗性筛选标记,添加pcr水印标签,设计得到了简化的莱茵衣藻叶绿体基因组。本发明选择莱茵衣藻叶绿体基因组作为研究对象,基于莱茵衣藻天然的细胞器叶绿体基因组,重新设计和人工构建莱茵衣藻的叶绿体基因组,通过移除冗余序列,改变、添加全新的遗传元件或代谢通路等实现对基因组的全面改造,并对莱茵衣藻叶绿体基因组的设计缺陷和序列缺陷进行修复,实现化学合成与设计序列的精确匹配,验证并评价莱茵衣藻叶绿体基因组的设计原则。本发明首次全叶绿体基因组范围内探索短分散重复序列的功能,并从整个叶绿体基因组出发,全局改造这些重复片段,探索这些重复片段的功能,为阐释这些重复片段的起源、生物学意义以及在进化中的作用提供依据;首次尝试通过删除冗余序列,精简莱茵衣藻叶绿体基因组;首次尝试在叶绿体基因组中组装代谢通路。本发明提供的方法,可实现叶绿体的定制化构建,为医药、能源、环境、农业等方面的研究与生产提供全新的底盘细胞和优化后的平台载体。
[0194]
应当理解的是,本发明的应用不限于上述的举例,对本领域普通技术人员来说,可以根据上述说明加以改进或变换,所有这些改进和变换都应属于本发明所附权利要求的保护范围。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1