一种海南黄牛液相育种芯片及其应用
1.本发明属于分子检测技术领域,具体涉及一种海南黄牛液相育种芯片,还涉及其应用。
背景技术:
2.海南黄牛又称高峰黄牛、雷琼牛,是中国最南端的黄牛品种,具有瘤牛血统(bos indicus),是适应热带和亚热带地区气候与环境条件的优良畜种,主产于广东省湛江市和海南岛各县(市)。海南黄牛具有极强的抗逆、抗高温高湿、繁殖性能稳定等特征,但由于其体型小、生长速度缓慢等劣势,与我国地方优良的役肉兼用型黄牛品种存在一定的差距。因此,积极探索海南黄牛种质资源保护和利用的有效途径,对海南黄牛进行种质资源鉴定和选育极为重要。
3.分子标记技术是采用分布在生物基因组上的遗传标记作为靶标,测定目标基因组位点和区域的相关基因型变异,从而在整体上代表全基因组水平的遗传变异。单核苷酸多态性(single nucleotide polymorphism,snp),指的是在基因组水平上的单个核苷酸的变异,是继rflp、rapd、aflp、ssr等分子标记技术之后发展起来的新一代分子标记技术,具有数量多、分布广、较高的遗传稳定性、丰富的多态性、易于检测等优势特点,被广泛应用于性状关联分析、亲缘鉴定等动物分子遗传研究中。
4.目前高通量snp检测技术主要采用测序和固相芯片两种策略。测序策略包括测序基因分型(gbs)和全基因组重测序(wgs),但需要大规模数据积累和强大的生物信息支撑。鉴于随机简化基因组测序存在的种种限制,理想的gbs策略就是从基因组上定点进行测序并获得物理位置相对固定的标记。固相芯片基于探针与dna序列的互补杂交,通过探针标记物的荧光信号进行分型,但也存在分型成本高、不易定制等缺点。目前,市场上已有的牛snp固相芯片,如bovine snp50等,主要是基于illumina和affymetrix基因分型平台的普通牛(bos taurus)snp固相芯片,无法用于海南黄牛品种的遗传多样性研究和遗传评估。为了更好的挖掘和保护热带地区瘤牛品种这一宝贵的种质资源,开发一款适合海南黄牛的snp芯片是有必要的。
5.近年发展起来的基于snp的靶向测序基因型检测的液相芯片具有检测效率高、成本低、适应性广、迭代灵活等特点,适合于所有生物遗传变异和基因型的检测,并广泛应用于种质资源评价、遗传图谱构建、基因定位和克隆、分子标记辅助选择等领域。由此,开发出专属于海南黄牛的snp液相芯片对海南黄牛种质资源保护和利用极为重要。
技术实现要素:
6.本发明的目的是提供一种海南黄牛液相育种芯片,可快速实现海南黄牛的基因型分析及品种鉴定。
7.本发明的另一目的是提供上述海南黄牛液相育种芯片在海南黑山羊育种中的应用。
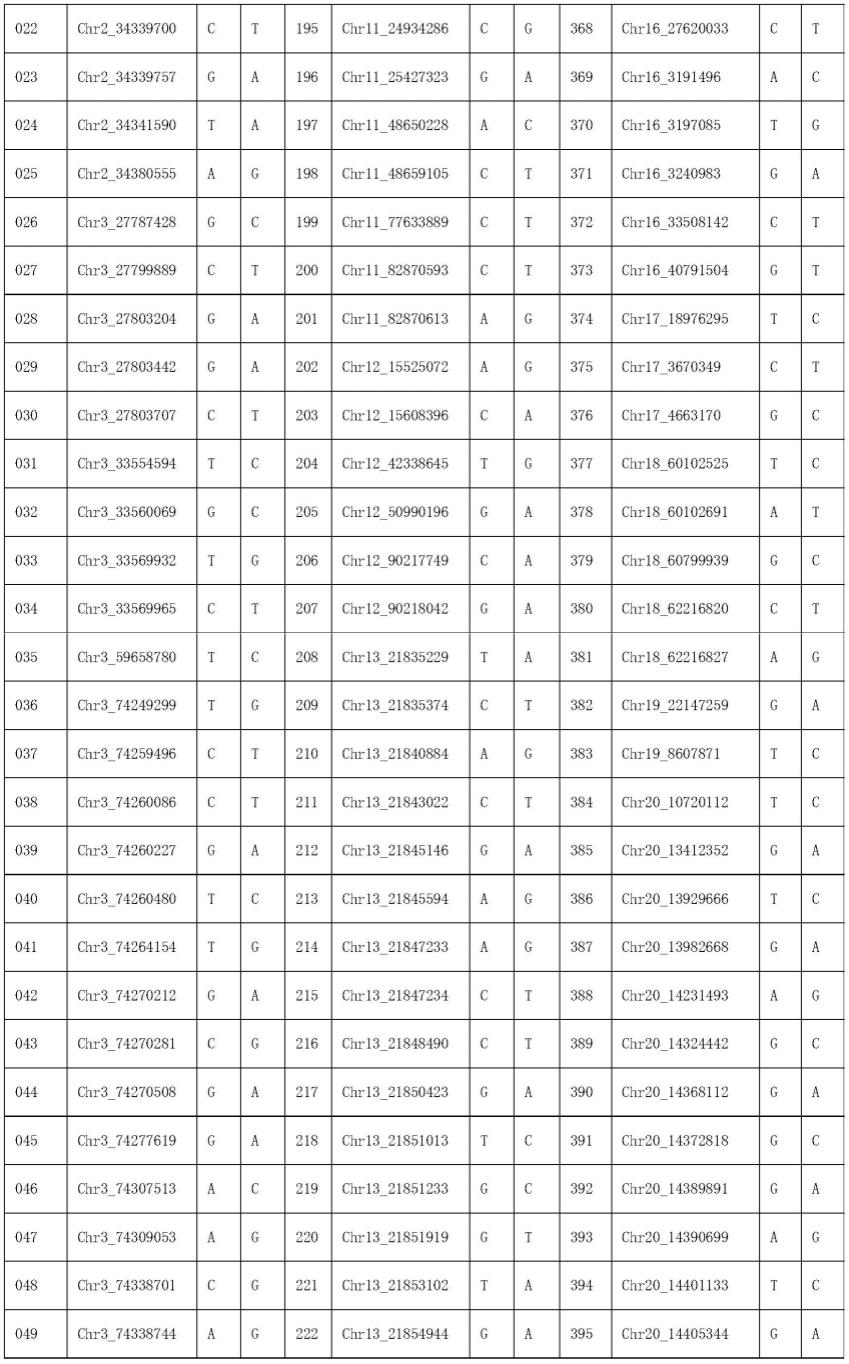
8.本发明所采用的技术方案是,一种海南黄牛液相育种芯片,该芯片的基因分型对象包括定位于ncbi牛参考基因组bos_taurus_umd_3.1.1上的518个snp位点,snp位点的位置信息如下所示;
9.10.11.12.13.14.[0015][0016]
本发明所采用的另一技术方案是,海南黄牛液相育种芯片应用于海南黄牛血统分析、遗传多样性评估、种质资源和亲缘关系鉴定、遗传图谱构建及基因定位、全基因组关联分析、分子标记辅助育种。
[0017]
本发明的有益效果是:第一,检测效率高,简化了生物信息分析过程,可以快速、低成本地实现目标snp位点基因分型;第二,根据靶向测序技术的探针长度不同,同时还可以对目标snp临近一定范围的snp进行分型。第三,对海南黄牛具有专属性,可快速实现海南黄牛的基因型分析及品种鉴定,科学引导海南黄牛杂交改良工作,助力海南黄牛种质资源的保护和开发。第四,应用灵活,可以通过直接增减探针的方式对目标snp位点进行调整,可在海南黄牛育种中的多个领域具有较高的应用价值。五是适应性广,检测不需要借助于特定的昂贵设备,适配多种测序平台。
附图说明
[0018]
图1为实施例中重测序数据的所有牛pca分析的结果图;方框内的圆点代表海南牛个体,方框外的圆点代表非海南牛个体;
[0019]
图2为实施例中海南黄牛液相育种芯片的snp分子标记位点在染色体上的分布情况图(每1mb的窗口内所包含snp的个数);
[0020]
图3为实施例中genobaits靶向捕获技术原理与流程图;
[0021]
图4为实施例中96份样本的海南黄牛液相育种芯片检测结果以及56份重测序结果集合的目标snp基因型pca分析结果图;圆形黑点代表海南牛个体,圆形灰点代表非海南牛个体;
[0022]
图5为实施例中96份样本的海南黄牛液相育种芯片检测结果以及56份的重测序结果集合的目标snp基因型系统发育树聚类分析结果图;弧形线为海南黄牛聚类区。
具体实施方式
[0023]
下面结合附图和具体实施方式对本发明进行详细说明。
[0024]
实施例1获得海南黄牛特异性snp分子标记
[0025]
1.获得牛全基因组重测序数据的snp信息
[0026]
全基因组重测序数据,通过数据分析获得高度可信的snp位点用于液相芯片的位点开发工作。本发明利用13个不同品种共79头牛的全基因组重测序数据,如表1所示,通过数据分析获得高度可信的snp位点用于后续筛选。其中14只海南黄牛全基因组重测序数据已上传至genbank(登录号为prjna774120,未公开)。其余65个牛个体的基因组信息均为ncbi下载的公开可用数据。
[0027]
表1全基因组重测序数据的牛品种及数量
[0028][0029]
重测序的基本流程:
[0030]
首先采用genobaits dna-seq library prep试剂盒对质检合格的dna构建重测序文库,文库构建完成后。文库质量检测合格后,利用华大mgi-t7测序平台进行测序,测序模式为pe150模式。
[0031]
测序得到的原始测序序列(sequenced reads)或者raw reads,里面含有带接头的、低质量的reads。为了保证信息分析质量,必须对raw reads过滤,得到clean reads,使用clean reads进行后续分析。使用软件fastp(version 0.20.0,参数:-n 10-q 20-u 40)对raw reads进行过滤,数据处理的步骤如下:去除接头序列(adapter);当测序read中含有的n的含量超过该条read长度比例的10%时,需要去除此对paired reads;当测序read中含有的低质量(q≤20)碱基数超过该条read长度比例的40%时,需要去除此对paired reads。
[0032]
使用软件bwa(mem比对方式)将进行质控后的clean reads与参考基因组序列进行比对,通过比对可以定位clean reads在参考基因组(bos_taurus_umd_3.1.1)上的位置。
[0033]
根据clean reads在参考基因组的比对结果,使用软件gatk(version v3.5-0-g36282e4)的unifiedgenotyper模块进行变异检测,检测参数为:-dcov 1000000-minindelfrac 0.15-glm both-l info。根据基因组数据情况,使用variantfiltration模块进行过滤,过滤参数为:
‑‑
filterexpression"mq0》=4&&((mq0/(1.0*dp))》0.1)"
‑‑
filtername"hard_to_validate"
‑‑
filterexpression"dp《5||qd《2"
‑‑
filtername"low_read_support"。总共检测出47,243,114个snp。
[0034]
为了获得海南黄牛特异性snp位点,需构建海南黄牛参考种群和非海南黄牛参考种群,对79个牛个体基因型数据进行群体遗传分析,对所有样本进行主成分分析(pca分析),pc1和pc2分别为35.26%和10.62%。pca分析结果,如图1所示,成功构建了海南黄牛与非海南黄牛群体,方框内的圆点代表海南牛个体,方框外的圆点代表非海南牛个体。
[0035]
确定海南黄牛和非海南黄牛参考种群后,将所有样本分为海南黄牛与非海南黄牛两组,使用perl自写的程序根据maf>0.1,缺失率<20%,杂合率<15%进行背景位点的初步过滤,然后计算海南黑山羊与非海南黑山羊组之间每一个snp变异位点fst值,筛选出海南牛特异的位点(即在海南牛群体与除海南牛以外群体之间存在差异的位点),筛选出海南牛特异snp候选位点有619个。
[0036]
基于snp的位置及其两侧序列信息设计一条或多条覆盖目标snp的探针,筛选位点须通过探针设计评估,探针长度在110bp,gc含量比例在30%-70%之间,同源性区域(序列比对相似性高的区域)个数<5。通过探针设计评估的海南牛特异位点518个。以1mb窗口大小在染色体滑动统计窗口内的snp个数,目标snp位点在基因组上的分布情况如图2所示;
[0037]
实施例2海南黄牛snp液相芯片的设计和制备
[0038]
根据snp位点的位置及两侧序列信息设计探针,以及基于sry基因序列设计用于性别鉴定的探针,通过石家庄博瑞迪生物技术有限公司进行探针合成及测试,最终挑出符合要求的位点完成海南黄牛snp标记检测试剂盒开发,从而获得基于液相探针捕获技术genobaits的海南黄牛snp液相芯片。
[0039]
genobaits工作原理:根据dna互补原理,在每个待测位点设计一条或多条覆盖目标snp的探针,这些用生物素(biotin)修饰的探针可以与变性后的重测序文库中目标区域杂交形成双链,利用链霉亲和素包被的磁珠将携带有生物素的分子吸附,经过洗脱、扩增和测序最终获得目标snp的基因型。利用博瑞迪专属技术,多达5万个位点的检测可以在单管中同时完成,大大增加了检测通量,降低检测成本,如图3所示。
[0040]
实施例3海南黄牛液相育种芯片检测分析流程
[0041]
1.dna样本提取与检测
[0042]
采用高通量dna提取试剂盒进行样品dna的提取。提取后的dna样品进行2种检测:(1)使用1%琼脂糖凝胶电泳方法分析dna的纯度和完整性;(2)使用qubit对dna浓度进行准确定量。
[0043]
2.genobaits技术流程
[0044]
取定量质检合格的dna利用超声波破碎仪进行随机物理破碎,破碎片段峰值控制在200-300bp,破碎后的dna经过末端修复后连接a尾。利用连接酶将加a后的dna片段与测序接头连接在一起,然后利用羧基修饰的磁珠对文库进行纯化和片段选择,保留插入片段在200-300bp的连接产物。连接产物加入带有barcode的测序引物和高保真pcr反应体系进行
pcr扩增,不同的barcode用于区分不同的样品。经过羧基磁珠纯化后扩增产物,即可用于探针杂交实验。
[0045]
取500ng完成构建的测序文库,冻干后加入探针与杂交试剂,变性后置于65℃温育2小时即可完成杂交反应。杂交产物进行洗液清洗后,再进行一轮pcr完成杂交捕获文库的构建。
[0046]
3.文库构建及上机测序
[0047]
通过上述dna质检合格的样品,使用相应的产品进行靶向测序文库构建,最终完成该项目全部样品的文库制备。文库构建完成后,先使用qubit2.0进行初步定量,使用qpcr的方法对文库的有效浓度进行准确定量以保证文库质量。文库检合格后,进入上机测序阶段。
[0048]
4.信息分析流程
[0049]
信息分析的内容包括:数据质控(去除接头和低质量数据)、与参考基因组比对、变异检测与注释等分析。
[0050]
实施例4海南黄牛液相育种芯片的应用
[0051]
本发明利用开发的海南黄牛液相育种芯片根据上述液相芯片检测分析流程对96个质检合格的牛dna样本进行检测。为了验证该海南黄牛液相育种芯片的可重复性,选择4个牛dna样(cs02_d1f、cs02_x1f、cs02_x7_f、cs02_x8f)做组内与组间重复。组内重复则用同一液相芯片重复检测一次,检测结果对比显示一致率在99.65%-99.85%之间,组间重复则用不同批次液相芯片检测三次,显示重复率完全一致,说明重复性好,符合检测要求。然后对获得的100份牛样本的数据(96个牛dna样本+4个重复的牛dna样)使用plink软件分析处理,目标snp位点检出率在96.60%-99.07%之间,说明符合海南黄牛液相育种芯片要求。
[0052]
本发明所涉及的海南黄牛液相育种芯片在对牛dna样本进行检测时,基于靶向测序技术可得到snp位点基因分型信息。在进行品种鉴定和聚类分析时,首先要获得96个待测dna样本(海南黄牛样本82个和安格斯牛样本14个)的液相芯片目标snp检测分析结果,以及前期56个非海南黄牛个体重测序数据的目标snp测序分析结果。然后通过bcftools软件集合成一个vcf文件。最后把所有样本分为海南黄牛与非海南黄牛两组,分别进行主成分分析(pca)和系统发育树的构建。主成分分析的前3个主成分(pc1、pc2、pc3)的聚类结果如图4所示,以及系统发育树的聚类结果如图5所示;
[0053]
将上述主成分分析(pca)和系统发育树的结果与实际分组情况对比分析,发现海南黄牛与非海南黄牛有较明显的分簇,基本实现了区分功能。说明在对未知牛进行海南黄牛品种鉴定时,未知牛dna样本可使用本发明海南黄牛液相育种芯片进行基因型检测,未知牛dna样本的检测结果可结合此次的芯片检测结果做聚类分析,可鉴定未知牛是否属于海南黄牛品种。海南黄牛液相育种芯片的snp基因型数据还有助于辨别海南黄牛血统的纯正性,科学引导海南黄牛杂交改良工作,助力海南黄牛种质资源的保护和利用。
[0054]
全基因组选择技术是新一代分子育种技术。本发明既可用于海南黄牛品种鉴定,又可用于海南黄牛育种工作。对芯片snp位点的基因注释,与牛表型性状相关联,将来可在基因定位、gwas、海南黄牛分子标记辅助育种的研究中发挥重要的作用。
[0055]
本发明可以用于海南黄牛低成本且大规模的基因分型。由于设计灵活,后期扩大检测样本数量,可以通过直接增减探针的方式对感兴趣的目标snp位点进行调整。这样的液相芯片更适用于海南黄牛品种的研究,有利于对海南黄牛种质资源的研究与保护。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1