一组具有抗病毒活性的肽类化合物奥米克欣B族化合物的应用
一组具有抗病毒活性的肽类化合物奥米克欣b族化合物的应用
技术领域
1.本发明属于医药生物技术领域,具体而言,涉及一组具有抗病毒活性的肽类化合物奥米克欣b族化合物的应用。
背景技术:
2.烈性病毒性感染是严重危害人类的传染性疾病。其中,新世纪以来由冠状病毒引起的非典型肺炎以其强烈的传染性和高致死性而著称。而每年在世界流行的由流感病毒感染引起的流行性感冒以及禽流感,其感染人群之广,致死数之大早已成为世界公共卫生安全不可忽视的问题,也是需要特别关注的。这些烈性病毒感染性疾病急需安全有效的广谱抗病毒,特别是抗冠状病毒药物对新型冠状病毒的有效治疗。
3.近期,分别在2019年1月24日《新英格兰医学》和1月29日《柳叶刀》发表的由我国ccdc等单位发表的有关2019-ncov冠状病毒的全基因测序显示,与sars冠状病毒只有79%的同源,其属于不同于sars冠状病毒的新冠状病毒。2019-ncov冠状病毒基因组可编码蛋白区的碱基长度为29844个碱基,编码12个蛋白:1ab,s,3,e,m,7,8,9,10b,n,13,和14。其中,1ab是编码一个由7096个氨基酸组成的非结构蛋白前体多聚蛋白的基因,s为编码刺突蛋白,还含有e、m和n蛋白等其它囊膜蛋白。一般来讲1ab编码的7096氨基酸的非结构前体多聚蛋白会被由病毒编码的蛋白酶3clpro和plpro切割形成16个非结构性蛋白(nonstructural protein,nsp),大多数nsps参与病毒复制复合物的形成[1,2]。根据对sars冠状病毒和中东呼吸综合征冠状病毒mers等相关药物靶标研究,由2019-ncov冠状病毒基因组同源比对研究可知,2019-ncov可被利用的关键药物靶标有刺突蛋白与人的细胞膜上的血管紧张素转换酶2(ace2)的相互作用,即入胞机制,rna依赖的rna聚合酶rdrp,和负责水解由7096氨基酸组成的多聚蛋白为功能蛋白的半胱氨酸蛋白酶3clpro和plpro[3]。
[0004]
流感病毒属正粘病毒科甲型流感病毒属。禽甲型流感病毒颗粒呈多形性,其中球形直径80~120nm,有囊膜。基因组为分节段单股负链rna。依据其外膜血凝素(h)和神经氨酸酶(n)蛋白抗原性不同,目前可分为16个h亚型(h1~h16)和9个n亚型(n1~n9)[4]。其中,流感病毒表面血凝素(hemagglutinin,ha)在被宿主细胞内蛋白酶剪切成ha1和ha2之前是以前体蛋白ha0的形式存在,因此,宿主细胞蛋白酶对病毒感染至关重要[5]。
[0005]
冠状病毒、流感病毒、副流感病毒等呼吸道相关病毒在进入呼吸道上皮细胞时,都需要利用宿主细胞的蛋白酶来切割、活化病毒蛋白,才能进入细胞进行复制[6]。因此这些宿主细胞编码的蛋白酶的抑制剂可能对这些呼吸道相关病毒具有广谱的抗病毒活性。更重要的是,针对宿主细胞的蛋白酶为靶点的抗病毒药物还可有效地避免病毒的逃逸变异。
[0006]
微生物来源天然产物是新型抗感染抗生素的主要来源,据统计,在现有25000个具有一定生物活性的微生物次级代谢产物中,大约10%的微生物代谢产物具有抗各种病毒活性,如抗合胞病毒药物利巴韦林就来自于微生物次级代谢产物病毒唑,还有广谱抗病毒抗生素海绵尿核苷、阿糖尿苷和阿糖腺苷(vidarabine)以及磷霉素和formycin等也来自微生
物天然产物或对微生物天然产物的改造等[7]。
[0007]
从天然产物中寻找新的药物先导化合物一直是研究热点,相对于动植物的次级代谢产物,微生物次级代谢产物更具有资源可持续性、不破坏生态环境等特点,因此也拥有更大的开发利用价值。
[0008]
早在上世纪60年代,医药生物技术研究所的科研前辈们从采自我国南方的土壤样品中,分离并筛选获得一株具有良好抗病毒活性的streptomyces sp.cpcc 200451,并使用经典的筛选方法与离子交换树脂柱层析法从cpcc 200451菌株发酵液中得到了其有效组分。曾作为抗人类流感病毒药物试用于临床试验,以提取物溶液制剂滴鼻的方式治疗流感病人,降低高烧,具有优良的效果。研究中还发现该有效组分对多种病毒,如流感病毒、冠状病毒、新城疫病毒等都表现出高度的敏感性,但是碍于当时的实验条件以及分离手段,且在分离过程中使用了强酸等剧烈条件,因此,一直不能得到streptomyces sp.cpcc 200451稳定而确切的药效成分,也就不能确定抗病毒活性成分的结构。
[0009]
随着微生物全基因组dna测序技术的快速发展,越来越多的微生物基因组完成测序并已实现信息共享,加之,生物信息学和分子生物学等一系列前沿技术广泛应用于基因组研究领域,极大地加速了科研人员挖掘微生物基因资源的进程[8]。研究发现,微生物次级代谢产物的生物合成基因往往成簇排列,且具有高度的保守性。通过微生物基因组的生物信息学分析,发现与解析有关次级代谢基因簇,能够推测产物的结构及其理化性质,也可以指导目标化合物的分离纯化[9]。生物信息学的兴起,不仅为微生物药物的发展提供了新契机,而且在微生物新次级代谢产物的发现中发挥着十分重要的作用,更为我们解决cpcc200451产生的抗病毒活性化合物的鉴定这一难题提供了新的科研思路。
[0010]
伴随“后基因组时代”的到来,以高通量测序为基础的转录组学、代谢组学等组学技术相继出现并得到广泛应用[10]。转录组学(transcriptomics)是研究生物体在某一功能状态下产生的所有转录本的集合,目前原核生物转录组测序的研究对象主要为mrna,通过比较不同发酵条件下微生物的转录组可以得到基因表达谱的变化情况,从而找到导致表达差异的生物合成基因信息[11,12]。结合基因组序列、生物信息学等分析技术,有助于定位目标代谢物的生物合成基因簇。代谢组学(metabonomics)是指生物体内源性代谢物质的动态整体,由于微生物在不同的发酵条件下可以产生不同的次级代谢产物,而代谢产物的多样性归功于生物合成基因的多样性,联合应用基因组学、转录组学以及代谢组学进行分析,不仅可以从现象中检测出差异的代谢产物,更从基因水平上解释了代谢产物变化的原因[13]。因此,通过对微生物在有活性的发酵条件下的转录组和代谢组的变化进行分析,并与无活性的发酵条件下的基因表达情况和代谢产物进行比较,即活性导向的比较转录组和比较代谢组分析,可以帮助我们找到活性相关的生物合成基因簇和代谢产物,并指导目标化合物的分离纯化与结构解析。
[0011]
为了探明streptomyces sp.cpcc 200451中抗病毒活性的有效组分,我们以streptomyces sp.cpcc200451的全基因组信息作为研究的出发点,通过基因组生物信息学的分析,寻找次级代谢相关基因簇;利用活性导向的比较转录组数据分析,锁定cpcc 200451活性相关物质的生物合成基因簇;结合分子生物学等技术手段,对目标基因簇的关键基因进行敲除和过表达等遗传操作,从而确定cpcc 200451活性组分所在的生物合成基因簇。并且,通过进行活性导向的比较代谢组数据分析,获得活性物质相关的结构特点,以
帮助目标产物分离及结构确证。综合运用生物信息学与化学分离等技术方法,分离获得活性物质单体,最终明确了streptomyces sp.cpcc 200451抗病毒活性的药效成分。目前我们发现的streptomyces sp.cpcc200451产生的抗病毒活性化合物中有些为已知的蛋白酶抑制剂,如antipain和chymostatin等,说明奥米克欣系列化合物可以靶向病毒和宿主细胞的蛋白酶,这可能是其具有针对多种呼吸道相关病毒尤其是冠状病毒和流感病毒的抗病毒活性的原因。[参考文献]
[0012]
[1]na zhu,dingyu zhang,wenling wang,et al.,a novel coronavirus from patients with pneumonia in china,the new england journal of medicine,january 24,2020,doi:10.1056/nejmoa2001017.
[0013]
[2]nanshan chen,min zhou,xuan dong,et al.,epidemiological and clinical characteristics of 99 cases of 2019 novel coronavirus pneumonia in wuhan,china:a descriptive study,the lancet,january 30,2020,doi:10.1016/s0140-6736(20)30211-7.
[0014]
[3]guangdi li&erik de clercq,therapeutic options for the 2019 novel coronavirus(2019-ncov),nature,10th february,2020,doi:10.1038/d41573-020-00016-0.
[0015]
[4]bernadeta dadonaite,brad gilbertson,et al.the structure of the influenza a virus genome,nature microbiology|vol 4|november 2019|1781
–
1789
[0016]
[5]陈伟,王莉莉,抗流感病毒药物靶标研究进展[j],国际药学研究杂志,2013,40(1):1-7
[0017]
[6]manon laporte,lieve naesens,et al.,airway proteases:an emerging drug target for influenza and other respiratory virus infections.current opinion in virology,2017,24,16-24.
[0018]
[7]naoki takizawa and manabu yamasaki,current landscape and future prospects of antiviral drugs derived from microbial products,the journal of antibiotics(2018)71,45
–
52.
[0019]
[8]陈铭.后基因组时代的生物信息学[j].生物信息学,2004,21(2):29-34.
[0020]
[9]kim e s j.midostaurin:first global approval[j].drugs,2017,77(11):1-9.
[0021]
[10]lockhart d j,winzeler e a.genomics,gene expression and dna arrays[j].nature,2000,405(6788):827-836.
[0022]
[11]velculescu v e,zhang l,zhou w,et al.characterization of the yeast transcriptome[j].cell,1997,88(2):243-251.
[0023]
[12]wang z,gerstein m,snyder m.rna-seq:a revolutionary tool for transcriptomics[j].nature reviews genetics,2009,10(1):57-63.
[0024]
[13]tang j.microbial metabolomics[j].current genomics,2011,12(6):391-403.
[0025]
[14]benson d a,cavanaugh m,clark k,et al.genbank[j].nucleic acids research,2017,45(d1):d37-d42.
[0026]
[15]finn r d,coggill p,eberhardt r y,et al.the pfam protein families database:towards a more sustainable future[j].nucleic acids research,2016,44(d1):d279-285.
[0027]
[16]eddy s r.accelerated profile hmm searches[j].plos computational biology,2011,7(10):e1002195.
[0028]
[17]王世媛.非核糖体肽合成酶(nrpss)作用机理与应用的研究进展[j].微生物学报,2007,47(4):734-737.
[0029]
[18]suda h,aoyagi t,hamada m,et al.antipain,a new protease inhibitor isolated from actinomycetes[j].journal of antibiotics(tokyo),1972,25(4):263-266.
[0030]
[19]umezawa h,aoyagi t,morishima h,et al.chymostatin,a new chymotrypsin inhibitor produced by actinomycetes[j].journal of antibiotics(tokyo),1970,23(8):425-427.
[0031]
[20]umezawa h,aoyagi t,okura a,et al.letter:elastatinal,a new elastase inhibitor produced by actinomycetes[j].journal of antibiotics(tokyo),1973,26(12):787-789.
[0032]
[21]maxson t,tietz j i,hudson g a,et al.targeting reactive carbonyls for identifying natural products and their biosynthetic origins[j].journal of the american chemical society,2016,138(46):15157-15166.
[0033]
[22]imker h j,walsh c t,wuest w m.sylc catalyzes ureido-bond formation during biosynthesis of the proteasome inhibitor syringolin a[j].journal of the american chemical society,2009,131(51):18263-18265.
[0034]
[23]chen y,mcclure r a,zheng y,et al.proteomics guided discovery of flavopeptins:anti-proliferative aldehydes synthesized by a reductase domain-containing non-ribosomal peptide synthetase[j].journal of the american chemical society,2013,135(28):10449-10456.
[0035]
[24]yan n.structural biology of the major facilitator superfamily transporters[j].annual review of biophysics,2015,44:257-283.
技术实现要素:
[0036]
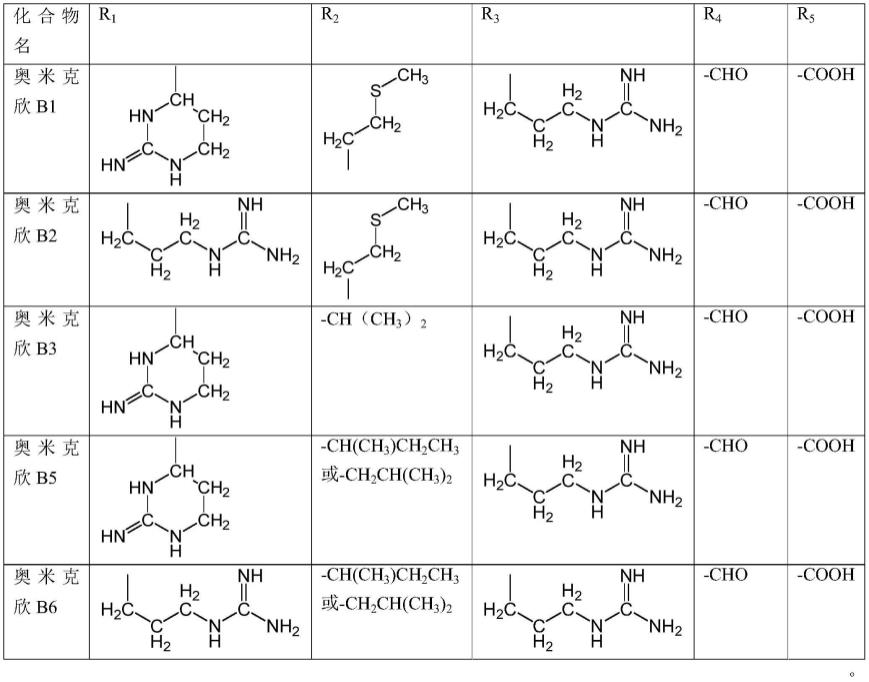
本发明首先涉及一组肽类化合物(奥米克欣)在制备抗病毒的药物中的应用,所述的肽类化合物的结构为式(1)所示:
[0037][0038]
其中,r1~r5可选的各种取代基种类如下表所示:
[0039]
[0040][0041]
优选的,所述的病毒为呼吸道病毒;
[0042]
最优选的,所述的病毒为流感病毒、冠状病毒。
[0043]
本发明还涉及所述的肽类化合物(奥米克欣)的制备方法,所述方法包括如下步骤,
[0044]
(1)发酵链霉菌(streptomyces sp.)cpcc 200451,发酵液离心(4000rpm/min,
15min)后收取上清液;
[0045]
(2)采用大孔吸附树脂级联反相c18层析柱hplc法收集活性组分;
[0046]
(3)将步骤(2)获得的活性组分,经半制备rp-hplc分离,即得所述肽类化合物奥米克欣。
[0047]
步骤(1)所述的发酵为,
[0048]
以10%的接种量转种于a3发酵培养基中,在28℃、200rpm下培养3~10d后,收集发酵液;
[0049]
所述a3发酵培养基各成分含量为(单位为g/l):甘油20,糊精20,蛋白胨10,酵母粉5,硫酸铵2,碳酸钙2;ph 7.2-7.4;
[0050]
步骤(2)的大孔吸附树脂级联反相hplc法的步骤参数为:
[0051]
使用hp20大孔吸附树脂和c18反相hplc层析柱,
[0052]
分离步骤为:
[0053]
1)上清液经大孔吸附树脂diaion hp20进行吸附后,使用两倍柱体积的去离子水冲洗;
[0054]
2)采用乙醇-水进行梯度洗脱(20%、50%和100%乙醇依次洗脱),每个梯度洗脱至流出液无颜色或颜色不变,收集50%乙醇梯度的洗脱液,经过减压浓缩后备用;
[0055]
3)步骤2)得到的50%乙醇梯度的洗脱液浓缩后进行c18柱层析,以乙腈-水梯度洗脱(10%、12%、15%、20%、25%、30%、40%、50%、80%和100%乙腈),收集15%~80%的梯度洗脱液,优选的,收集15%~20%乙腈-水洗脱液;
[0056]
步骤(3)所述的半制备rp-hplc参数及方法为:色谱柱:shiseido capcell-pak pfp 5μm,10
×
250mm;流动相:
[0057]
奥米克欣a系列化合物:25%acn/含0.1%tfa的h2o;
[0058]
奥米克欣b系列化合物:20%acn/含0.1%hcooh的h2o;
[0059]
奥米克欣c系列化合物:40%acn/含0.1%tfa的h2o;
[0060]
流速:1.5ml/min。
[0061]
本发明还涉及一组新的肽类化合物(奥米克欣),所述肽类化合物分别为奥米克欣a1、a2、a6、b1、b2、b3、b5、b6、c1、c2、c6,其结构通式为式(1)所示,各个化合物的r1~r4的取代基分别如下表所示:
[0062][0063][0064]
本发明还涉及微生物生物合成所述肽类化合物的基因簇,
qpcr
[0090]
图20、活性化合物的分离流程与技术路线
[0091]
图21、奥米克欣a(omicsynin a)类化合物(omicsynin a1、omicsynin a2)的化学结构
[0092]
图22、奥米克欣a1的1h(22a)、
13
c(22b)、dept(22c)、1h-1
h cosy(22d)、hsqc(22e)、hmbc(22f)、noesy(22g)谱图及hrms(22h)分析数据
[0093]
图23、奥米克欣a2的1h(23a)、
13
c(23b)、dept(23c)、1h-1
h cosy(23d)、hsqc(23e)、hmbc(23f)、noesy(23g)谱图及hrms(23h)分析数据图
[0094]
图24、奥米克欣a6的hrms分析数据图
[0095]
图25、奥米克欣b1的hrms分析数据图
[0096]
图26、奥米克欣b2的hrms分析数据图
[0097]
图27、奥米克欣b3的hrms分析数据图
[0098]
图28、奥米克欣b5的hrms分析数据图
[0099]
图29、奥米克欣b6的hrms分析数据图
[0100]
图30、奥米克欣c1的hrms分析数据图
[0101]
图31、奥米克欣c2的hrms分析数据图
[0102]
图32、奥米克欣c6的hrms分析数据图
具体实施方式
[0103]
材料及方法
[0104]
菌株:
[0105]
streptomyces sp.cpcc 200451,中国药学微生物菌种保藏管理中心(china pharmaceutical culture collection),保藏号cpcc 200451;
[0106]
大肠杆菌dh5α;
[0107]
大肠杆菌et12567/puz8002;
[0108]
质粒:
[0109]
pset152,链霉菌整合型载体;
[0110]
poj260,链霉菌基因敲除质粒;
[0111]
引物:
[0112][0113]
[0114]
[0115][0116]
发酵培养基:
[0117]
a1培养基(g/l):葡萄糖5,麦芽膏10,棉籽饼粉10,可溶性淀粉20,酵母膏5,磷酸氢二钾0.5,硫酸铵5,碳酸钙3,氯化钠1,ph 7.2-7.4。
[0118]
a2培养基(g/l):葡萄糖5,酵母膏5,蛋白胨5,牛肉膏5,玉米浆4,黄豆饼粉10,碳酸钙4,二氯化钴0.02,可溶性淀粉20,ph 7.2-7.4。
[0119]
a3培养基(g/l):甘油20,糊精20,蛋白胨10,酵母粉5,硫酸铵2,碳酸钙2,ph 7.2-7.4。
[0120]
a4培养基(g/l):可溶性淀粉30,黄豆饼粉15,硫代硫酸钠20μ,硫酸亚铁0.5,磷酸氢二钾0.5,氯化钾0.3,ph 7.2-7.4。
[0121]
b1葡萄糖天门冬素培养基(g/l):葡萄糖10,天门冬素0.5,磷酸氢二钾0.5,ph 7.2-7.4。
[0122]
b2合成5号培养基(g/l):硝酸钾1,氯化钠0.5,磷酸氢二钾0.5,硫酸亚铁0.01,硫酸镁0.5,可溶性淀粉20,ph 7.0。
[0123]
b3丙酸钠培养基(g/l):丙酸钠2,硝酸铵0.1,氯化钾0.1,硫酸镁0.05,硫酸亚铁0.05,ph 7.2。
[0124]
b4培养基(g/l):琥珀酸钠0.9,磷酸二氢铵0.5,硫酸镁0.1,硫酸亚铁0.01,ph 7.2。
[0125]
b5 waksman培养基(g/l):硫酸铵0.2,磷酸氢二钾3,硫酸镁0.5,氯化钙0.126,ph 7.2。
[0126]
b6 twye培养基(g/l):酵母粉0.25,磷酸氢二钾0.5,ph 7.2。
[0127]
b7克氏合成1号培养基(g/l):磷酸氢二钾1,碳酸镁0.3,氯化钠0.2,硝酸钾1,硫酸亚铁0.01,碳酸钙0.5,葡萄糖20,ph 7.0。
[0128]
b8察氏培养基(g/l):蔗糖30,硝酸钾2,磷酸氢二钾1,氯化钾0.5,硫酸镁0.5,硫酸亚铁0.01,ph 7.2-7.4。
[0129]
b9 isp7培养基(g/l):酪氨酸0.5,甘油15,天门冬素1,磷酸氢二钾0.5,硫酸镁0.5,氯化钠0.5,硫酸亚铁0.01,ph 7.2-7.4。
[0130]
b10培养基(g/l):可溶性淀粉2,硫酸亚铁0.01,硫酸镁0.5,硝酸钾1,氯化钠0.4,磷酸氢二钾0.5,ph 7.2。
[0131]
其他培养基为本领域常规标准化培养基。
[0132]
其他常用试剂为国产分析纯或色谱纯。
[0133]
实施例1、链霉菌cpcc 200451的培养及测序
[0134]
1、链霉菌(streptomyces sp.)cpcc 200451的培养与菌种保藏
[0135]
使用ymg或tsb液体培养基培养cpcc 200451的菌丝体,在28℃、200rpm摇床中培养36-72h;固体培养链霉菌cpcc 200451时采用ymg固体培养基,在28℃、培养箱中培养5-7d,产孢固体培养基使用ms培养基。
[0136]
实验中所用到的菌种全部采用低温甘油保藏方法,于-20℃、或-80℃冷冻保存。
[0137]
2、链霉菌(streptomyces sp.)cpcc 200451的发酵培养
[0138]
将cpcc 200451接种于ymg固体培养基表面,28℃恒温箱培养7d;转接种于含有100ml ymg液体培养基的500ml摇瓶中,28℃、200rpm摇床上培养48h后,以10%的接种量转种于100ml/500ml的发酵培养基中,继续在28℃、200rpm摇床培养5d后,收集发酵液。
[0139]
tsb培养基(g/l):蛋白胨2,氯化钠5,葡萄糖2.5,磷酸氢二钾2.5。
[0140]
ymg培养基(g/l):葡萄糖10,麦芽浸膏10,酵母浸粉10,琼脂15,ph 7.0。
[0141]
ms培养基(g/l):甘露醇20,黄豆粉20,琼脂20,采用自来水配置。使用时加入终浓度为10mm的氯化镁。
[0142]
其他培养基:
[0143]
ys培养基(g/l):酵母浸粉2,可溶性淀粉10,琼脂15,ph 7.2-7.4。
[0144]
pyg培养基(g/l):蛋白胨3,酵母浸粉5,甘油10,琼脂15,ph 7.2-7.4。
[0145]
isp4培养基(g/l):可溶性淀粉10,磷酸氢二钾1,氯化钠1,硫酸铵2,硫酸镁1,碳酸钙2,琼脂15,ph 7.0-7.4。
[0146]
3、cpcc 200451全基因组测序
[0147]
为了探明streptomyces sp.cpcc 200451中抗病毒活性的有效组分,揭示其生物合成基因簇以及生物合成机制,我们首先对中国药学微生物菌种保藏管理中心提供的streptomyces sp.cpcc 200451进行了全基因组dna测序。
[0148]
由北京华大基因(beijing genomics institute)完成该高通量测序工作。采用全新的三代测序pacbio rsii平台,并结合二代测序illumina hiseq 4000平台对cpcc 200451的全基因组dna进行测序,拼接组装获得其基因组精细图。cpcc 200451的基因组是一个长度为8,918,347bps的线性染色体,(g+c)mol百分含量为73.6%,除染色体外,未发现游离质粒的存在。
[0149]
cpcc 200451基因组中含串联重复序列共计316个,总长为151,923bp,占基因组全长的1.7%。经基因注释分析后发现,基因组预测含有8,151个蛋白编码基因;通过对rrna库进行比对,并利用rrnammer software软件预测到11个rrna操纵子;采用trnascan-se软件进行预测,共找到73个trna编码基因。
[0150]
实施例2、分析及确定cpcc 200451中抗流感病毒的次级代谢产物生物合成基因簇
[0151]
转录水平的调控是原核生物最为重要的调控方式之一,本实施例通过比较分析具有不同抗病毒活性(高活性、低活性、无活性)的发酵液样品中streptomyces sp.cpcc 200451全基因组的转录水平差异,从而初步锁定cpcc 200451抗病毒有效组分所在的生物合成基因簇
[0152]
1、发酵条件的筛选
[0153]
为了获得具有不同抗流感病毒活性的发酵液样品,我们首先对cpcc 200451的发酵条件进行了筛选,尝试了14种发酵培养基和4个发酵时间点,并对发酵液的抗流感病毒活性进行测定。
[0154]
(1)发酵培养基的筛选
[0155]
将链霉菌cpcc 200451的孢子悬液分别涂布于ymg固体培养基表面,28℃恒温箱中培养7d后,使用接种铲挖取相同大小的菌体打散后,接种于14种不同的发酵培养基,于28℃、200rpm摇床中培养。
[0156]
(2)发酵时间的选择
[0157]
分别在3d、5d、7d和10d,收集上述不同培养基的发酵液样品,离心后收取上清液部分测定其抗流感病毒活性。
[0158]
2、抗流感病毒活性测定
[0159]
抗流感病毒活性的测定由本研究所病毒室完成,病毒株为流感病毒a/fm/1/47(h1n1)和a/汉防/359/95(h3n2)。
[0160]
测试方法:
[0161]
(1)使用96孔培养板接种犬肾细胞mdck细胞,静置于37℃、5%co2培养;
[0162]
(2)24h后感染流感病毒,吸附2h后弃病毒液,加入含样品及阳性对照药的维持液,设立细胞对照孔和病毒对照孔,继续培养;
[0163]
(3)根据病毒对照组的病变程度观察各组细胞病变程度,采用reed-muench方法分别计算出不同样品对于细胞的半数致死浓度(tc
50
)、对于病毒的半数抑制浓度(ic
50
),并计算选择指数(si=tc
50
/ic
50
)。
[0164]
活性测试结果显示,a3培养基来源streptomyces sp.cpcc 200451发酵液样品抗流感病毒a/汉防/359/95(h3n2)活性最高,其次为a1发酵培养基和a2发酵培养基,并且在发酵时间为5d时的发酵液样品显著高于其它3个时间点(表1)。此外,根据菌株的生长状态和抗流感病毒活性测定结果,选择b7为无活性的发酵培养基,作为后续比较转录组分析的阴性结果对照,以帮助缩小目标生物合成基因簇的筛选范围。
[0165]
进一步的测定结果表明,a3培养基来源streptomyces sp.cpcc 200451发酵液不仅对流感病毒a/汉防/359/95(h3n2)具有较好活性,而且对流感病毒a/fm/1/47(h1n1)也表现出一定活性(表2)。
[0166]
表1、cpcc 200451发酵液抗流感病毒活性的测定
[0167][0168]
表2、cpcc 200451发酵液抗病毒活性的测定
[0169][0170]
3、转录组测序分析rt-qpcr验证
[0171]
根据抗流感病毒活性测定结果,选定a3作为高活性发酵培养基,b7作为无活性的发酵培养基,对cpcc 200451进行发酵,并在发酵初期收集菌体,提取总rna,进行转录组测序(rna-seq)和数据分析。
[0172]
(1)rna样品的制备
[0173]
依据发酵液的抗流感病毒活性测定结果,选择a3和b7分别作为链霉菌cpcc 200451的高活和无活性的发酵培养基,并在发酵初期12h、24h和48h分别收集菌体,使用改进后的trizol法进行链霉菌cpcc200451的总rna的提取,样品名称分别为a3-24、a3-48、a3-72、b7-24、b7-48和b7-72。
[0174]
经检测后共6个rna样品的提取质量较好,没有明显的基因组dna污染和严重降解的现象,基本符合高通量测序要求。
[0175]
(2)rna-seq转录组测序及数据分析
[0176]
由北京华大基因公司针对上述6个rna样品进行建库,并且采用二代测序平台bgiseq-500进行高通量测序。转录组的可视化视图如图1所示。结合antismash预测的次级
代谢产物生物合成基因簇信息,我们找到了3个在a3和b7中呈现出显著差异的生物合成基因簇,分别是cluster 27、cluster 28和cluster 36。
[0177]
第一个差异表达的基因簇是cluster 27,它是一个nrps型铁载体(siderophore)的生物合成基因簇,转录组可视化视图如图2所示。可以看出,相比于b7低活性发酵培养基,cluster 27在a3高活性发酵培养基中呈现显著的高表达。
[0178]
cluster 28也是siderophore的生物合成基因簇,转录组可视化视图(图3)结果显示,cluster 28在高活和低活发酵条件下的转录情况呈现显著差异,同样在a3发酵培养基中显著高表达。
[0179]
第三个在转录水平呈现出显著差异的基因簇是cluster 36,它是nrps型生物合成基因簇,在a3高活性发酵培养基中呈现出显著的高表达,转录组可视化视图如图4所示。
[0180]
(2)rt-qpcr验证转录组数据分析结果
[0181]
采用了荧光定量rt-qpcr分别对这些基因簇的转录水平进行了验证。
[0182]
鉴于cluster 27和cluster 28均为铁载体的生物合成基因簇,以cluster 27为例进行介绍。本实验中选取了cluster 27中3个铁载体生物合成相关基因,分别是c27_5814(dhb)、c27_5819(nrps)和c27_5821(transporter),进行rt-qpcr测定,结果如图5所示。streptomyces sp.cpcc 200451中cluster 27在a3高活性发酵条件下显著高表达,即rt-qpcr实验结果与转录组分析结果一致,表明转录组测序数据可信。
[0183]
选择cluster 36生物合成基因簇中3个功能基因,c36_7094基因(nrps)、c36_7097基因(nrps)和c36_7098基因(nrps),结果如图6所示,rt-qpcr验证结果与转录组测序结果一致。
[0184]
实施例3、三个关键基因簇的生物信息学分析及功能分析
[0185]
antismash预测,基因簇cluster 27和cluster 28为铁载体的生物合成基因簇。生物信息学分析显示,cluster 27和cluster 28均含有多个铁阻遏蛋白的结合位点,提示其表达受到了铁离子浓度的调控,即在寡铁培养基中高表达,在富铁培养基中不能表达。
[0186]
因此,我们在a3高活性发酵培养基中加入0.05%的铁离子,即a3-fe
3+
发酵培养基。将streptomyces sp.cpcc 200451同时采用a3和a3-fe
3+
培养基,在同样的条件下进行发酵培养,在发酵初期48h收集菌体提取rna,进行rna-seq测序与数据分析,以及rt-qpcr验证。结果显示,相比于a3培养基,a3-fe
3+
发酵条件下收集的streptomyces sp.cpcc 200451,cluster 27和cluster 28均不再表达。
[0187]
然而,抗流感病毒活性测定结果表明,加入铁离子后的streptomyces sp.cpcc 200451发酵液仍然存在很高的活性,因此,证明这两个siderophore基因簇(cluster 27和cluster 28)的合成产物并不是链霉菌cpcc 200451主要的抗流感病毒活性物质。
[0188]
排除了前两个转录表达差异的基因簇后,我们把注意力转向第三个呈现显著表达差异的基因簇——cluster 36。比较了streptomyces sp.cpcc 200451在a3和a3-fe
3+
发酵条件下cluster 36的转录组数据,并以a1发酵培养基作为对照进行分析后发现,基因组中生物合成基因簇cluster 36在a3-fe
3+
培养基中仍然处于高表达状态(图7)。采用rt-qpcr进行验证,结果显示cluster 36在a3和a3-fe
3+
培养基中的表达显著高于a1培养基,与转录组比较结果一致(图8)。结合加入过量铁离子后的抗流感病毒活性测定结果,推测cluster 36可能为streptomyces sp.cpcc 200451抗病毒活性成分所在的生物合成基因簇。
[0189]
cluster 36位于streptomyces sp.cpcc 200451基因组chromosome 1:7,822,964-7,875,615,全长为52.6kb,生物信息学分析预测该基因簇中共含有50个开放阅读框,基因簇核心区域为基因7092-7102(chromosome 1:7,841,516-7,857,514)。
[0190]
采用genbank数据库
[14-16]
中的blastp功能对cluster 36内包含的50个开放阅读框所编码蛋白的氨基酸序列进行了同源性分析,结果显示,cluster 36属于nrps类的生物合成基因簇,nrps(nonribosomal peptide synthetases)在非核糖体多肽合成时起到关键作用,是由多个相互独立的模块按照特定的空间顺序串联而成的多功能蛋白复合体,它能够特异性识别、活化并转运特定的氨基酸底物,并按一定的顺序缩合形成肽链,合成并释放非核糖体多肽。nrps中每个模块至少包含3个核心结构域,包括腺苷酰化结构域(a结构域)、肽酰基载体蛋白结构域(pcp结构域)和缩合结构域(c结构域)
[17]
。nrps的最后一个组件中还含有一个特别的结构域,它处于合成酶肽链的最下游,称为硫酯酶结构域(te结构域),负责从nrps模块上释放肽链。此外,还可能包括其他特异性结构域,如差向异构化结构域(epimerization,e结构域)、甲基化结构域(methyltransferase,m结构域)等对底物氨基酸进行相应的修饰。
[0191]
antismash预测,cluster 36与deimino-antipain的生物合成基因簇拥有最高相似性为66%,相似基因位于cluster 36的核心区域,对cluster 36与deimino-antipain中的相似基因所编码的蛋白序列采用blastp工具进行比对,结果如图9所示。这种蛋白酶抑制剂家族已有40多年的历史,通常具有相对低的分子量、疏水性、c-末端醛的存在和内部脲基键等特征,结构的高度相似性暗示着它们具有共同的生物合成途径,因此,进化形成相关的生物合成基因簇
[18-20]
。
[0192]
2016年,maxson等从streptomyces albulus nrrl b-3066中采用探针检测并分离得到deimino-antipain,并且对其生物合成基因簇进行了解析
[21]
,如图10所示,由基因anpc-g构成了nrps,其中,基因anpd、anpe和anpf的a结构域分别负责组装arg、phe和val。然而,负责组装arg(或cit)的第四个a结构域,一种推测是phe加载模块(anpe)也安装arg/cit,类似于syringolin的生物合成或arg特定模块(anpd)以非连续的方式执行两次功能
[22]
;另一种情况是anpd以特定的、非连续的方式安装cit(或arg随后被cit取代),然后是arg。anpc基因仅含有c结构域,而anpg含有pcp结构域和c结构域,以及可能负责释放的nad还原(r)结构域(而不是传统的硫酯酶),最终产生c-末端醛的产物
[23]
。在nrps基因之外,anpa基因可能编码水解酶,其可能作用于arg的组装之前或之后,在cit的形成中起作用。anpb属于mfs转运蛋白超家族(major facilitator superfamily)
[24]
,而anph的编码产物是起调节作用的组氨酸激酶。随后,maxson等研究者采用异源表达的方法证明deimino-antipain中cit的组装需要该生物合成基因簇以外的其它基因行使功能。
[0193]
此外,文献报道,负责这类肽醛化合物合成的生物合成基因簇,anpb-g基因方向和顺序均保持一致,按照编码acyl-coa脱氢酶的基因anpi的存在与否及其排列位置的不同,大概分为三类:第一类是不含有基因anpi的生物合成基因簇,如deimino-antipain;第二类基因anpi位于anpd与anpe之间,并且大部分属于这种排列方式的基因簇占绝大多数;只有少数基因簇的anpi位于anpg基因之后,也就是第三种anp类的生物合成基因簇
[21]
。因此,streptomyces sp.cpcc 200451中cluster 36属于比较常见的第二种anp类的基因簇,不同之处在于还多出一个sdr还原酶的编码基因,其功能还需要进一步的研究与确证。
[0194]
综上所述,我们可以推测streptomyces sp.cpcc 200451中cluster 36的编码产物丰富多样,并且可能与deimino-antipain、chymostatin、elastatinal和mapi等化合物结构相似。为了进一步确证cluster 36是否为链霉菌cpcc 200451抗流感病毒活性组分所在的生物合成基因簇,我们进行了该菌株敲除和过表达遗传操作体系的构建。
[0195]
实施例4、基因敲除法验证链霉菌cpcc 200451中cluster 36基因簇的功能
[0196]
cpcc 200451在ms培养基中培养96-120h时,孢子的形态和数量达到最佳状态,选择120h为cpcc200451的孢子收集时间。同时,由于cpcc 200451对阿泊拉霉素(apramycin)敏感,因此,选择阿泊拉霉素作为cpcc 200451的筛选标记,选择氨曲南(aztreonam)作为接合转移实验中大肠杆菌的抑制剂。
[0197]
cpcc 200451敲除遗传操作系统的建立
[0198]
选择cluster 36内2个nrps型的功能基因,分别为基因7094(chromosome 1:7,844,718-7,847,825)和基因7098(chromosome 1:7,850,958-7,852,772),进行敲除遗传操作系统的构建。
[0199]
设计引物,分别扩增出包含目标基因上游和下游的两个片段(即前臂和后臂),将其连接到自杀型质粒poj260的多克隆位点,以接合转移的方式将重组质粒导入streptomyces sp.cpcc 200451中。利用阿泊拉霉素抗性标记筛选单交换菌株,鉴定正确后,在没有阿泊拉霉素的ms固体培养基上传代培养约5代后,影印筛选丧失了阿泊拉霉素抗性的双交换突变菌株,并通过pcr技术进行结果验证,最终获得缺失目标基因的阻断株,具体步骤大致如下。
[0200]
(1)阻断质粒的构建
[0201]
以cpcc 200451基因组dna为模板,分别在7094基因和7098基因的左右两侧各2000bp左右处设计两对引物,采用pcr技术分别扩增出用于双交换的左、右同源臂。7094基因两臂的长度分别为2129bp和2215bp;7098基因两臂的长度分别为2056bp和2173bp。左臂的两端分别引入hindiii和ecori酶切位点,右臂两端则分别引入ecori和hindiii酶切位点。
[0202]
选择poj260自杀型质粒进行阻断株的构建,首先分别将pcr扩增得到的左、右同源臂连接于t载体上,并转化至e.coli感受态细胞后,通过提取重组质粒进行测序验证,并将测序正确的重组质粒使用ecori和hindiii进行酶切。同时将质粒poj260采用hindiii进行酶切,回收载体dna大片段后,与上述经酶切处理的左、右同源臂进行三片段连接,连接产物转化至e.coli dh5α感受态细胞,利用质粒poj260的阿泊拉霉素抗性标记筛选阳性转化子,通过提取质粒及酶切验证(图11和图12),从而获得正确的重组质粒,分别命名为poj7094lr和poj7098lr。
[0203]
lanes 1-3,poj7094lr/hindiii;lanes 4-6,poj7094lr/ecori;lanes 7-9,poj7094lr/psti;lanes 10-12,poj7094lr/kpni.
[0204]
lanes 1-3,poj7098lr/hindiii;lanes 4-6,poj7098lr/ecori;lanes 7-9,poj7098lr/psti;lanes 10-12,poj7098lr/ncoi.
[0205]
(2)单交换突变株的筛选
[0206]
分别将重组质粒poj7094lr和poj7098lr转化至e.coli et12567/puz8002感受态细胞中,然后以接合转移的方式导入cpcc 200451中,采用阿泊拉霉素和氨曲南进行抗性筛
选。抗生素覆盖3-5d后,平板上长出具有阿泊拉霉素抗性的接合子,挑取单菌落影印至含有阿泊拉霉素的平板上,该菌株为可能的单交换突变株。
[0207]
通过提取菌株基因组总dna,采用pcr技术鉴定单交换突变株。设计3对引物(p1p2、p3p4和p5p6),分别扩增左同源臂及其侧翼区、右同源臂及其侧翼区和目标基因的片段(图13)。如果是左单交换突变株,当使用引物p1p2进行pcr时,可扩增除产物片段大小约2kb的左同源臂。反之,当使用引物p3p4扩增出产物片段大小约2kb的右同源臂时,则为右单交换菌株。
[0208]
(3)双交换突变株的筛选
[0209]
将pcr验证后正确的单交换菌株在未添加阿泊拉霉素的ms平板上传代培养大约5代后,影印筛选丢失阿泊拉霉素抗性的菌株,采用pcr技术进行双交换突变株的鉴定。当使用位于左同源臂右侧边缘的p3引物,与位于右臂左侧边缘的p2引物进行pcr验证时,只可以扩增出小片段目的条带,而不能扩增出与被敲除基因相当的大片段;使用p5p6扩增不到基因内部片段,并且引物p1p4可扩增出约4kb的左同源臂和右同源臂的连接产物,并将此4kb的pcr产物进行测序验证,若序列为左、右同源臂无误,则证明该菌株为双交换突变株。
[0210]
共获得基因7094的12个单交换接合子,选择左单交换和右单交换突变株各1株,在无阿泊拉霉素抗性的ms平板上传代培养5代后,收集孢子进行稀释涂布平板,从右单交换突变株中筛选到两株疑似的双交换突变株(名称以7094-ko-10和7094-ko-33表示),并进行pcr验证,如图14所示。
[0211]
7098基因在含有阿泊拉霉素抗性的平板上仅筛选得到的1个接合子,并经pcr验证。同样,将该菌株在无阿泊拉霉素抗生素的ms平板上经过传代培养5代后,我们筛选获得丢失了阿泊拉霉素的双交换突变株(名称为7098-ko-37和7098-ko-47),并进行了pcr鉴定,如图15所示。
[0212]
(4)rt-qpcr对阻断株的验证
[0213]
将上述筛选得到的阻断株7094-ko、7098-ko与cpcc 200451野生型菌株,同时使用加入过量铁离子的a3-fe
3+
培养基,在同等条件下进行发酵。并且在发酵初期48h,收集菌体并提取rna,反转录为cdna后,对cluster 36的相关基因进行qrt-pcr验证(图16和17)。结果显示,已成功将目标基因敲除;同时还发现,当敲除7094基因时,7098基因也不再表达,并且7097基因和7099基因的表达也受到了影响;而敲除7098基因后,7094基因的表达并未受影响。
[0214]
(5)回补菌株的构建
[0215]
以野生型streptomyces sp.cpcc 200451的基因组dna为模板,分别设计引物7094_f(含ndei酶切位点)和7094_r(含xbai酶切位点),以及7098_f(含ndei酶切位点)和7098_r(含bamhi酶切位点),利用pcr技术分别扩增出7094基因和7098基因,并克隆到pset152质粒(含有红霉素强启动子及噬菌体φc31整合位点,具有阿泊拉霉素抗性的筛选标记)的相应酶切位点上,分别构建7094基因和7098基因遗传互补重组质粒。经酶切和测序验证无误后,得到回补质粒pl-7094和pl-7098。
[0216]
以接合转移的方式将验证正确的回补质粒分别导入到阻断株7094-ko和7098-ko中,以质粒pset152的阿泊拉霉素抗性作为筛选标记,每个基因分别3个挑取接合子,分别使用阿泊拉霉素抗性、pset152整合位点和回补基因共3对引物,进行pcr验证,结果证明遗传
互补菌株7094-koc和7098-koc构建成功。
[0217]
(6)rt-qpcr对各个菌株的验证
[0218]
将上述筛选得到的阻断株7094-ko、7098-ko,回补菌株7094-koc、7098-koc和streptomyces sp.cpcc 200451野生型菌株,同时使用a3-fe
3+
发酵培养基,并且在同等条件下进行发酵。在发酵初期48h,收集菌体并提取rna,反转录为cdna后,对7094基因和7098基因进行rt-qpcr验证。结果显示,将7094基因成功回补到了敲除株7094-ko中;成功将7098基因回补到阻断株7098-ko中。
[0219]
(7)阻断株与回补株抗流感病毒活性的测定
[0220]
对阻断株7094-ko、7098-ko,回补菌株7094-koc、7098-koc和cpcc 200451野生型菌株的发酵液进行了抗流感病毒活性测定,结果表明,敲除该基因簇结构基因7094或7098基因后,链霉菌cpcc200451抗流感病毒活性消失;回补7094基因后并不能恢复其抗流感病毒能力,而回补7098基因后的菌株又具有了较好的抗流感病毒活性(表3)。因此证明,cluster 36的表达与streptomyces sp.cpcc 200451的抗流感病毒活性密切相关。
[0221]
表3、cpcc 200451野生型菌株与阻断株、回补株的发酵液抗流感病毒活性测定结果
[0222][0223]
实施例5、cpcc 200451过表达遗传操作系统的建立
[0224]
为了进一步确证cluster 36为streptomyces sp.cpcc 200451抗流感病毒活性组分所在的生物合成基因簇,我们选择该基因簇内的5个调节基因,以质粒pset152为基础,分别构建过表达质粒,并导入streptomyces sp.cpcc 200451中进行过表达。采用a1发酵培养基对重组菌株进行同等条件下的发酵,通过检测和比较抗流感病毒活性的变化,从而确定cluster 36中抗流感活性成分表达的调控基因及其调控作用。
[0225]
1、调节基因过表达质粒的构建
[0226]
首先以streptomyces sp.cpcc 200451的基因组dna为模板,选择cluster 36内的5个表达调控基因7081、基因7082、基因7083、基因7089和基因7102,分别设计引物(引物序
列详见材料),采用pcr技术扩增出这5个调控基因的dna片段。
[0227]
利用ndei和bamhi双酶切整合型质粒pset152,由于调节基因7081和7089内部含有bamhⅰ位点,因此在它们的两端分别引入ndei和xbai酶切位点,同时对质粒pset152进行ndei和xbai双酶切。
[0228]
而在7082、7083和7102调节基因的两端引入ndei和bamhi酶切位点。
[0229]
首先将pcr产物连接到peasy-t载体上,测序验证序列正确后,再使用相应的酶切位点对调节基因进行酶切与产物回收,然后将其连接在同样酶切后的质粒pset152载体上,从而获得重组质粒。
[0230]
2、以电转化的方式导入streptomyces sp.cpcc 200451
[0231]
以上重组质粒以电转化的方法导入到streptomyces sp.cpcc 200451野生型菌株中,利用阿泊拉霉素抗性进行筛选,获得过表达的重组菌株。同时将空载体pset152导入到streptomyces sp.cpcc 200451野生型菌株中,以此作为对照菌株。pcr验证引物为pset152和attb-streptomyces,若重组质粒正确的整合到streptomyces sp.cpcc 200451基因组中,则pcr可扩增出1.6kb的目的条带,经过pcr验证,调节基因7081、7082、7083和7102分别得到3个过表达菌株,调节基因7089获得2个重组菌株,分别命名为200451/pl-7081、200451/pl-7082、200451/pl-7083、200451/pl-7089和200451/pl-7102。
[0232]
3、过表达菌株的抗流感病毒活性的测定
[0233]
为了进一步探讨cluster 36内调节基因上调后对streptomyces sp.cpcc 200451抗流感病毒活性的影响,我们选择了a1、a3和b7共3种发酵培养基,对上述不同调节基因的过表达菌株进行同等条件下发酵,并收取发酵液样品进行抗流感病毒活性的测定(表4)。结果显示,当过表达调节基因7102时,来自a1和a3培养基的发酵液样品抗流感病毒活性均呈现出一定程度的提高;过表达其他4个调节基因后,发酵液的抗病毒活性无明显变化;而使用b7培养基的发酵产物并未出现明显提高的抗流感病毒活性,考虑主要是因为b7为寡营养培养基,链霉菌的生长在一定程度上受到了限制,因此不能合成丰富的次级代谢产物。
[0234]
表4过表达菌株的抗流感病毒活性测定
[0235][0236][0237]
4、过表达菌株的转录组分析
[0238]
鉴于过表达调节基因7102时,a1培养基的发酵液样品的抗流感病毒活性由原来的较低水平变为较高水平,因此我们对此变化进行了转录水平差异的进一步研究。通过使用a1发酵培养基对streptomyces sp.cpcc 200451野生型菌株与调节基因7102的过表达菌株
同时发酵,并在发酵初期48h收集菌体,提取rna,进行转录组测序(rna-seq)及数据分析。使用可视化工具对cluster 36的转录情况进行了可视化查看(图18)。结果表明,调节基因7102过表达后,cluster 36核心区(chromosome 1:7,841,516-7,858,166)基因呈现显著上调。
[0239]
5、rt-qpcr对过表达菌株的验证
[0240]
为了验证转录组数据的可靠性,我们还将上述提取到的rna样品,反转录成cdna进行荧光定量rt-qpcr验证。结果如图19所示,使用a1发酵培养基收集的菌体,相比于streptomyces sp.cpcc 200451野生型菌株,过表达调节基因7102后,cluster 36核心区基因呈现出2~8倍的上调,此变化与转录组结果相一致,因此,我们推论调节基因7102调控cluster 36的核心区基因的表达;并且上调基因7102会引起streptomyces sp.cpcc 200451抗流感病毒活性的升高,即调节基因7102起到正调控作用。
[0241]
综上,本研究选择2个功能基因,进行了敲除遗传操作系统的构建,rt-qpcr验证结果显示,已成功将两个功能基因敲除,并对敲除株与streptomyces sp.cpcc 200451野生型菌株进行发酵及抗流感病毒活性测定,结果显示,敲除该基因簇结构基因后,链霉菌cpcc 200451抗流感病毒活性消失;选择5个调节基因,成功构建了目标基因簇的过表达菌株,转录组分析结果与rt-qpcr验证结果共同显示,当过表达调节基因7102会引起该基因簇核心区基因表达上调。因此,最终锁定cluster 36为streptomyces sp.cpcc 200451抗流感病毒活性物质所在的基因簇。
[0242]
实施例6、抗病毒活性成分的化学分离与纯化
[0243]
前期实验结果已表明,在a3高活培养基中加入过量的铁离子,可以使siderophore的生物合成基因簇不再表达,而且目标基因簇cluster 36可以正常表达,因此,本研究采用a3-fe
3+
培养基对streptomyces sp.cpcc 200451野生型菌株进行了大量发酵,通过离心收集上清液,同时以cluster 36中基因7094、基因7098的阻断株7094-ko和7098-ko(无抗流感病毒活性)作为负对照,在同等条件下进行发酵,收取发酵液样品的上清部分,与活性发酵液进行同样的处理。综合活性测定结果和hplc分析结果,追踪具有抗流感病毒活性的组分,通过hplc进行样品的制备与纯化。活性化合物的分离流程与技术路线如图20所示。
[0244]
将收集到streptomyces sp.cpcc 200451野生型菌株的发酵液上清部分共14l,经大孔吸附树脂hp20进行吸附后,收集流穿液样品;使用两倍柱体积的去离子水冲洗,收集水洗液样品;采用乙醇-水进行梯度洗脱(20%、50%和100%乙醇依次洗脱),每个梯度洗脱至流出液无颜色或颜色不变,分别收集各个梯度的洗脱液,经过减压浓缩后,冻干备用。将上述发酵原液、流穿液、水洗液、20%乙醇、50%乙醇和100%乙醇洗脱液分别进行抗流感病毒活性的测定,结果表明,抗病毒活性成分主要集中在50%乙醇洗脱液部分,此外,100%乙醇洗脱部分也具有一定的活性(表5)。
[0245]
表5发酵样品抗流感病毒活性测定结果
[0246][0247][0248]
将上述抗流感病毒活性最好的50%乙醇部分(200451-50e),进一步采用反相c18(ods-a-hg)开放柱层析,以乙腈-水进行梯度洗脱(10%、12%、15%、20%、25%、30%、40%、50%、80%和100%乙腈),使用agilent-c18-aq色谱分析柱(5μm,4.6
×
150mm)进行hplc分析,流动相为乙腈和水(含有0.1%的tfa),分析条件为0-30min(0-30%乙腈),30-60min(30-100%乙腈)。根据各馏份中主成分的hplc分析结果进行合并后共获得10个组分(a-j),分别表示为50e-c18-a~j,并进行抗流感病毒活性测试(表6)。结果表明,50e-c18-c~i组分均具有一定的抗流感病毒活性,其中,50e-c18-e和50e-c18-f组分的抗病毒活性显著高于其他各组分。
[0249]
表6、50e-c18各组分的抗流感病毒活性测定
[0250][0251][0252]
实施例7、次级代谢产物的分离纯化及结构鉴定
[0253]
将活性组分50e-c18-g直接经rp-hplc半制备(shiseido capcell-pak pfp 5μm,10
×
250mm,25%acn/含0.1%tfa的h2o,1.5ml/min)得到一组化合物,命名为奥米克欣a(omicsynin a)。
[0254]
将活性组分50e-c18-e与50e-c18-f直接经rp-hplc半制备(shiseido capcell-pak pfp 5μm,10
×
250mm,20%acn/含0.1%hcooh的h2o,1.5ml/min)得到一组化合物,命名为奥米克欣b(omicsynin b)。
[0255]
将活性组分200451-100e直接经rp-hplc半制备(shiseido capcell-pak pfp 5μm,10
×
250mm,40%acn/含0.1%tfa的h2o,1.5ml/min)得到一组化合物,命名为奥米克欣c(omicsyninc)。
[0256]
综合运用现代波谱学技术手段,包括hresims、1h-nmr、
13
c-nmr、dept、1h-1
h cosy、hsqc、hmbc和noesy鉴定与解析所收集的化合物的化学结构,并通过hresims/ms提供的碎片离子特征,推导各个化合物的化学结构,如下表7所示
[0257]
表7、奥米克欣a-c的结构鉴定
[0258]
[0259]
[0260][0261]
其中。omicsynin a类新化合物(omicsynin a1、omicsynin a2)的化学结构如图21
所示,nmr数据如表8所示,1h-nmr、
13
c-nmr、dept、1h-1
h cosy、hsqc、hmbc、noesy谱图及hrms分析数据如图22、图23所示。
[0262]
化合物a6、b1、b2、b3、b5、b6、c1、c2、c6的hrms分析数据如图24~32所示
[0263]
表8化合物omicsynin a1和omicsynin a2的nmr数据(600mhz,dmso-d6)
[0264]
[0265][0266]
实施例8、奥米克欣(omicsynin)类化合物抑制冠状病毒活性测定
[0267]
1、cpe法抗病毒药效实验方法及步骤:
[0268]
(1)实验在传代肝细胞huh7.5细胞中进行,细胞1
×
104个/孔接种于96孔板中,过夜培养;
[0269]
(2)以100tcid50的冠状病毒液感染细胞,待测药物用培养液稀释,分别于感染同时给药和感染后2h给药两种给药方案进行测定,待测药物以三倍稀释8个剂量的样品进行实验,阳性对照药利巴韦林注射液购自天津金耀集团湖北天药药业股份有限公司,用时稀释至所需浓度;
[0270]
(3)每个剂量设2个平行孔,待病毒对照组病变达cpe评价标准4+号时观察结果,记录并用reed-muench法计算药物对病毒的半数抑制浓度(ic50,公式如下)并计算选择指数(si=tc50/ic50);
[0271][0272]
其中:a=累积抑制率《50%的药物浓度,b=累积抑制率》50%的抑制率,c=累积抑制率《50%的抑制率,d=log稀释倍数
[0273]
cpe评价标准:以细胞死亡比例分别标记为4+(细胞死亡比例75%~100%)、3+(细胞死亡比例50%~75%)、2+(细胞死亡比例25%~50%)、1+(细胞死亡比例0~25%)、0+(细胞全部存活)。
[0274]
重复实验2次以上,给出代表性结果。
[0275]
2、对hcov-229e毒株的药效
[0276]
(1)感染同时给药:在huh7.5细胞中,cpe法测定样品0h给药对hcov-229e毒株的抑制效果,同时测定利巴韦林(rbv)的活性。
[0277][0278][0279]
样品说明:
[0280]
rbv,利巴韦林注射液购自天津金耀集团湖北天药药业股份有限公司,批号为31712252,规格为100mg/ml;
[0281]
antipain(#37682-72-7,5mg)购自上海一飞生物科技有限公司,等同于本发明所述的从链霉菌cpcc200451发酵样品中分离获得的单体化合物omicsynin b4;
[0282]
chymostatin(#9076-44-2,5mg)购自sigma-aldrich公司,该试剂包含三个化合物chymostatin a、b、c,等同于本发明中所述的化合物omicsynin c3和c5;
[0283]
测试样品7094-ko-20e、7094-ko-50e、7094-ko-100e分别为阻断株7094-ko(敲除cluster36中基因7094的突变株)发酵液经大孔吸附树脂柱层析后所得的20%乙醇、50%乙醇和100%乙醇洗脱液样品;
[0284]
测试样品200451-20e、200451-50e、200451-100e分别为链霉菌cpcc 200451野生型菌株发酵液经大孔吸附树脂柱层析后所得的20%乙醇、50%乙醇和100%乙醇洗脱液样品;
[0285]
ods 24与ods 26为200451-50e样品经反相c18柱层析后使用30%乙腈-水洗脱所收集的第24和第26份馏分,均归属于本发明实施例6中所述的50e-c18-g组分,第24馏分主要含有化合物omicsynin a3和a4,第26份馏分主要含有化合物omicsynin a1、a2;
[0286]
测试样品200451-a3与200451-a3fe分别为链霉菌cpcc 200451采用a3、a3-fe
3+
(0.05%fe
3+
)发酵培养基所得的发酵上清液,经冷冻干燥处理后的固体样品。
[0287]
(2)感染后2h给药:在huh7.5细胞中,cpe法测定样品2h给药对hcov-229e毒株的抑制效果,同时测定利巴韦林(rbv)的活性。
[0288][0289]
样品说明:同上
[0290]
上述结果显示,含有奥米克欣的链霉菌cpcc 200451发酵液及由发酵液提纯的奥米克欣类化合物,对冠状病毒同样具有很好的抑制效果。
[0291]
最后需要说明的是,以上实施例仅用作帮助本领域技术人员理解本发明的实质,不用来限定本发明的保护范围。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1