一种结直肠癌肠道微生物标志物及其应用与构建结直肠癌检测模型的方法
本发明涉及生物医学,更具体的说是涉及一种结直肠癌肠道微生物标志物及其应用与构建结直肠癌检测模型的方法。
背景技术:
1、结直肠癌是一种恶性肿瘤,起源于结肠或直肠的内膜上皮细胞。它是全球最常见的癌症之一,发病率和死亡率逐年上升。结直肠癌早期症状不明显,常常被忽视或误诊;随着病情进展,患者可能会出现腹痛、腹泻、便秘、便血、贫血等症状。如果癌细胞扩散到其他部位,还可能出现肝、肺等器官的转移症状。
2、结直肠癌的诊断包括肠镜检查、组织活检和影像学检查等。目前,结直肠癌的诊断方法还存在一些不足之处,如结肠镜检查需要患者进行肠道准备,且有一定的创伤性和风险性;ct扫描可以提供高分辨率的影像,帮助医生确定肿瘤的位置、大小、形态等信息,但是放射线可能会对患者造成一定的辐射损伤;血液中的肿瘤标志物如cea、ca19-9等虽然可以用于筛查结直肠癌,但其敏感性和特异性并不高,不能作为确诊的依据,对于结直肠癌的预后评估也不够准确,无法根据患者的具体情况制定个性化的治疗方案。
3、早期发现和治疗可以显著提高患者的生存率和生活质量;研究表明,肠道微生物群落的失调与结直肠癌的发生和发展密切相关。因此,迫切需要一种微生物标志物,作为结直肠癌的早期诊断和预后评估依据,通过分析患者肠道微生物的组成和功能变化,更早地发现癌症迹象,并监测治疗效果。
技术实现思路
1、本发明的目的是提供一种结直肠癌肠道微生物标志物及其应用与构建结直肠癌检测模型的方法,通过分析患者肠道微生物的组成和功能变化,更早地发现癌症迹象,并监测治疗效果。
2、为实现上述目的,本发明提供如下技术方案:
3、本发明提到了一种结直肠癌肠道微生物标志物,所述肠道微生物标志物包括以下菌种:tm7phylum_sp_oral_taxon_348、科氏梭菌(clostridium_sp_nsj_42)、狄氏副拟杆菌(parabacteroides_distasonis)、血链球菌(streptococcus_sanguinis)、产孢肠道细菌(turicibacter sanguinis)、candidatus_nanosynsacchari_sp_tm7_anc_38_39_g1_1、华德萨特菌(sutterella_wadsworthensis)、齿放线菌(actinomyces_dentalis)、非典型韦荣菌(veillonella_atypica)、放线菌属(actinomyces_sp_icm47)、gemmiger_formicilis、大肠杆菌(escherichia_coli)、blautia_faecicola、沟迹优杆菌(eubacterium_sulci)、变异白蚁菌(isoptericola_variabilis)、sellimonas_intestinalis、瘤胃球菌属(ruminococcus_sp_af41_9)、多尔氏菌(dorea_longicatena)、普通拟杆菌(phocaeicola_vulgatus)、粪肠球菌(enterococcus_faecium)、灵巧粪球菌(coprococcus_catus)、直肠真杆菌(eubacterium_rectale)、梭状芽孢杆菌(clostridium_sp_af20_17lb)、罗姆布茨菌(romboutsia_timonensis)、产丁酸无杆菌(agathobaculum_butyriciproducens)、多形拟杆菌(bacteroides_thetaiotaomicron)、阿德勒克罗伊茨菌属(adlercreutzia_equolifaciens)、口腔消化链球菌(peptostreptococcus_stomatis)、candidatus_cibiobacter_qucibialis、卵形拟杆菌(bacteroides_ovatus)、脆弱拟杆菌(bacteroides_fragilis)、普拉梭菌(faecalibacterium_prausnitzii)、fusicatenibacter_saccharivorans。
4、本发明还提到了一种结直肠癌肠道微生物标志物的应用,所述应用包括用于制备诊断患者是否患有结直肠癌或结直肠癌风险的产品、作为靶点用于结直肠癌的病情评估和治疗以及构建结直肠癌检测模型;所述产品包括检测试剂、检测试剂盒和检测芯片中的任意一种。
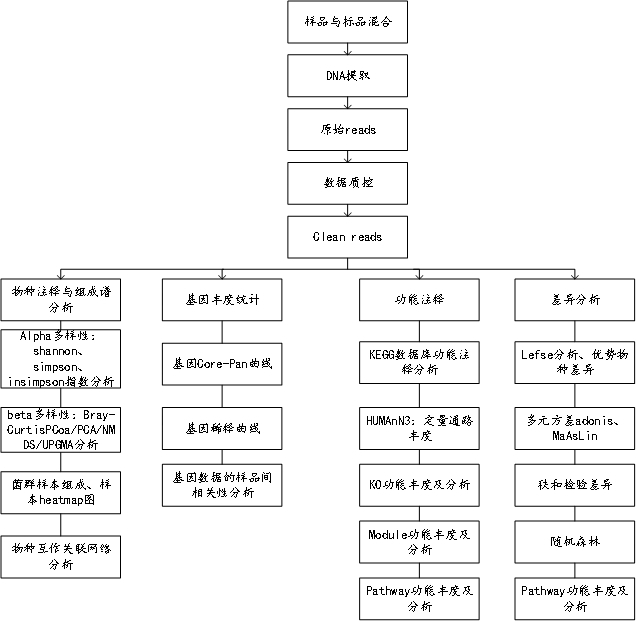
5、本发明还提供了一种应用结直肠癌肠道微生物标志物构建结直肠癌检测模型的方法,包括以下步骤:
6、(1)分别提取结直肠癌患者和健康人群的粪便中总dna,预处理后进行高通量测序,对测序结果进行物种注释与组成谱分析、基因丰度统计、功能注释以及差异分析,筛选出直肠癌肠道微生物标志物;
7、(2)通过结直肠癌肠道微生物标志物建立鉴定结直肠癌和健康人群的随机森林模型,评估,得到结直肠癌检测模型;
8、(3)将待测患者的宏基因组及其丰度信息输入结直肠癌检测模型进行识别。
9、进一步地,所述步骤(1)中,所述物种注释与组成谱分析包括:
10、alpha多样性:shannon、simpson、insimpson指数分析;
11、beta多样性:bray-curtispcoa、pca、nmds、upgma分析;
12、菌群样本组成、样本heatmap图;
13、物种互作关联网络分析。
14、进一步地,所述步骤(1)中,所述基因丰度统计包括:基因core-pan曲线、基因稀释曲线、基因数目的样品间相关性分析。
15、进一步地,所述步骤(1)中,所述功能注释包括:kegg数据率功能注释分析、humann3:定量通路丰度、ko功能丰度及分析、module功能丰度及分析、pathway功能丰度及分析。
16、进一步地,所述步骤(1)中,所述差异分析包括:lefse分析、优势物种差异、多元方差adonis、maaslin、秩和检验差异、随机森林、pathway功能丰度及分析。
17、进一步地,所述步骤(1)中进行高通量测序,包括:用fastp进行去除接头、用bowtie2去除宿主序列、用metaphlan4进行基于marker的物种丰度分析、用humann33进行功能分析。
18、进一步地,所述步骤(2)中,通过结直肠癌肠道微生物标志物建立鉴定结直肠癌和健康人群的随机森林模型,评估,得到结直肠癌检测模型,具体为:
19、将结直肠癌患者和健康人群分为训练集和测试集,利用python中的sklearn.ensemb1e.randomforestclassifier对所述训练集进行随机森林模型训练;
20、所述评估的方法为:通过绘制学习曲线衡量随机森林模型对数据的拟合程度,并通过十折交叉验证来评估随机森林模型中每一组参数的性能,选出最优参数组合。
21、进一步地,所述步骤(2)中随机森林模型的训练参数为:基学习器的数据nesfimators={100,500,1000},树的最大深度max_depth={1,2,3,7,9},最大特征数max_features=[″log2″,″sqrt″],使用gridsearchcv从python的sci kit-learn库中进行参数调优。
22、根据本发明提供的具体实施例,本发明公开了以下技术效果:
23、(1)本发明从宏基因组数据中提取与结直肠癌发病机制和进程相关的生物标志物;采用随机森林算法来构建结直肠癌检测模型具体为:构建随机森林模型,并进行评估得到结直肠癌检测模型,增加了结直肠癌检测模型的泛化能力和抗过拟合性,能够更准确地预测和评估结直肠癌的病情,有助于医生制定更有效的治疗方案,提高患者的生存率和生活质量。
24、(2)本发明提供的方法具有非侵入性、操作简便等优点,能够为结直肠癌的诊断提供新的途径,筛选出的结直肠癌微生物标志物和构建的结直肠癌检测模型具有较高的准确性和特异性,有助于提高结直肠癌的诊断效率。本发明的应用能够为结直肠癌的早期筛查、个性化治疗以及疾病监测提供有力的支持。
25、(3)本发明中识别结直肠癌的特异性肠道微生物标志物的技术方案,有助于发现新的生物标志物,发现新的生物标志物或治疗靶点。
- 还没有人留言评论。精彩留言会获得点赞!