基于Caffe的堆栈降噪自编码基因信息特征提取的方法与流程
本发明涉及生物信息技术领域,具体为基于Caffe的堆栈降噪自编码基因信息特征提取的方法。
背景技术:
人类基因组计划奠定了从基因切入研究复杂疾病的基础,人们希望找到人类发病与基因之间的关系。全基因组关联研究(Genome-wide association study)是指在人类全基因组范围内找出存在的序列变异,即单核苷酸多态性(SNP),从中筛选出与疾病相关的SNPs。这一研究方法的引入,使对遗传流行病的发病预测不再停留在传统的年龄、家族史等“环境性”因素分析,而是通过对人体的全基因组的分析,找出可能导致今后发病的基因,并结合“环境性”因素,得出包括癌症在内的多种流行病的发病率。虽然,GWAS已经发现了很多与复杂疾病相关的SNP位点,但是GWAS仍然存在很多问题,其成果与人们的预期差距甚远。
BLAST(Basic Local Alignment Search Tool)是一套在DNA数据库或蛋白质数据库中进行相似性比较的分析工具。BLAST程序能迅速使目标基因序列与公开数据库进行相似性序列比较。BLAST采用一种局部的算法获得两个序列中具有相似性的序列,并且对一条或多条序列(可以是任何形式的序列)在一个或多个核酸或蛋白序列库中进行比对。然而,BLAST更多的表现为一种查询比对工具,并不能通过深度学习算法进行自动的特征提取和基因性状识别。
Caffe是一个清晰而高效的深度学习框架,它是纯粹的C++/CUDA架构,支持命令行、Python和MATLAB接口,可以在CPU和GPU直接无缝切换,用于研究机器视觉的开源卷积神经网络框架,并且主要应用于图像领域处理。它的特点是:快速搭建网络结构,代码可扩展,计算速度快等。利用Caffe开源深度学习框架,搭建基于Caffe的堆栈降噪自编码基因特征提取模型,然后构建基于该模型的基因性状识别系统,并使用该系统对待测基因序列进行精确、高效的特征提取与自动识别基因的对应性状。
本发明基于深度学习算法,提供了基于Caffe的堆栈降噪自编码基因信息特征提取的方法,能够在以机器学习的方式,通过训练特征提取模型与构建自动识别系统,更加精确、高效的实现基因序列分析与分类识别,为基因信息的比对研究、分类识别等提供了一种新的技术方案。
技术实现要素:
本发明的目的在于提供基于Caffe的堆栈降噪自编码基因信息特征提取的方法,以解决上述背景技术中提出的问题。
为实现上述目的,本发明提供如下技术方案:基于Caffe的堆栈降噪自编码基因信息特征提取的方法,该基于Caffe的堆栈降噪自编码基因信息特征提取的方法具体步骤如下:
S1:数据准备,基因碱基序列可视化,图像数据预处理,首先将基因碱基序列进行可视化转换,即先由序列信息转换成二进制数字节流表示,再利用颜色空间转换为二维的图像格式,然后使用Caffe提供的工具将图像数据调整到固定大小,最后使用Caffe的工具将全部基因图像转换为适用于Caffe的数据库文件,格式为leveldb或者lmdb;
S2:搭建基于Caffe的堆栈降噪自编码基因特征提取模型,第一步:定义堆栈降噪自编码基因特征提取模型,第二步:编写基于caffe的堆栈降噪自编码模型的模型描述文件;
S3:逐层训练堆栈降噪自编码基因特征提取模型,采用逐层贪婪的训练方法,编写基于Caffe的模型求解文件,使用步骤S1准备好的基因信息数据,对步骤S2模型描述文件定义的堆栈降噪自编码基因特征提取模型进行训练,求解特征中各层的参数;
S4:使用有标签基因数据微调堆栈降噪自编码基因特征提取模型,在堆栈降噪自编码模型的基础上,构建一个分类模型,使用有标签(已知性状分类)的基因信息数据对该分类模型进行训练;
S5:使用训练好的基因特征提取模型搭建基因性状识别系统,并使用该系统对基因性状进行识别,保留经过微调之后的堆栈降噪自编码基因特征提取模型,并以大量的基因信息数据对该基因特征提取模型进行特征提取训练。利用微调的基因特征提取模型,构建基因性状识别模型,对待测基因信息数据进行对应性状的分类识别。
进一步地,所述S2中模型描述文件是Caffe用来定义堆栈降噪自编码模型的网络结构(网络连接方式)以及网络中各层的参数。
进一步地,所述步骤S4中所谓有标签的基因信息数据是指带有该基因对应性状标签的数据。
本发明的技术方案采用深度学习的算法框架,在图像特征提取的基础上,通过训练分类识别模型,以此可进行对待测基因的特征提取和进一步的分类识别。
附图说明
图1为本发明技术方案实施流程图;
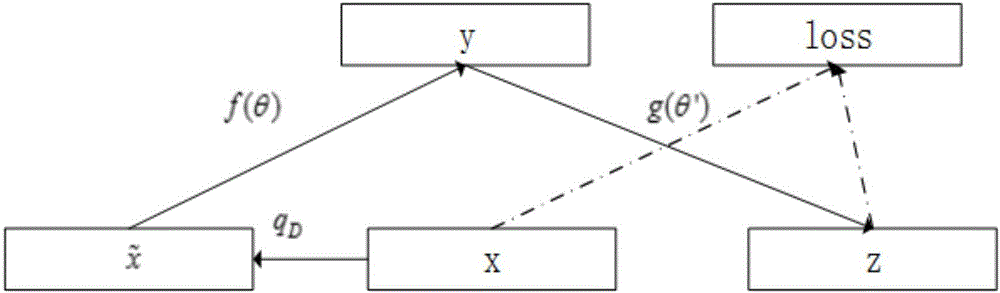
图2为本发明降噪自编码器示意图;
图3为本发明堆栈式降噪自编码器示意图;
图4为本发明堆栈降噪自编码基因信息识别模型微调示意图;
图5为本发明堆栈降噪自编码基因特征提取模型搭建基因性状识别系统图。
具体实施方式
为了使本发明的目的、技术方案及优点更加清楚明白,以下结合具体实施例,对本发明进行进一步详细说明。应当理解,此处所描述的具体实施例仅仅用以解释本发明,并不用于限定本发明。
实施例
步骤S1的数据准备:首先准备用于训练模型的基因信息数据,这里的基因信息数据采用不同个体的同名基因,假设有n个不同人的基因样本数据。将这n个人的该基因碱基序列进行可视化操作,可视化结果为n个图像格式的基因数据;
假设图像像素大小为p0×q0,然后,将这n张图像像素设置到固定大小p×q。使用Caffe提供的convert_imageset工具,将这n张图像样本转换为适用于Caffe的数据库文件,数据库文件格式为leveldb或者lmdb,优选lmdb。
步骤S2是基于Caffe,搭建一个堆栈降噪自编码基因特征提取模型。模型的基础单元是一个降噪自编码模型,一个基因特征提取模型是由若干个降噪自编码模型的基础单元逐层堆叠在一起构成一个深度模型;降噪自编码模型是在原始输入基础上加入随机噪声,生成新的输入。然后,一个编码器将新的输入进行编码,一个解码器将编码的结果进行解码;降噪自编码模型的目的是使经过解码器解码出来的结果尽可能地接近原始输入。这个模型用下面的公式表示:
这里,x是原始输入,qD是加入噪声操作,表示加入噪声之后新的输入;
编码器为:
这里,W为权重,b为偏置,s是非线性Sigmoid函数,y为编码器的输出,θ=(W,b)为编码器的参数;
解码器为:
z=gθ(y)=s(W′y+b′)
这里,W′为权重,b′为偏置,y为编码器的输出,s是非线性Sigmoid函数,z是解码器的输出,θ′=(W′,b′)为解码器的参数;
Loss函数为:
L(x,z)=L2(x,z)=C(σ2)||x-z||2
这里,C(σ2)为常数项,并且只与σ2有关;
堆栈降噪自编码模型由若干个降噪自编码基础单元逐层堆叠构成的深度模型。前一个降噪自编码模型的编码器输出作为后一个降噪自编码模型的输入;
再根据上述定义的模型结构,编写适用于Caffe的模型描述文件。
步骤S3逐层训练堆栈降噪自编码基因特征提取模型,是采用逐层贪婪算法来训练堆栈降噪自编码基因特征提取模型;
所谓逐层贪婪算法指的是,首先训练第一个降噪自编码模型,固定其它降噪自编码模型。训练完第一个降噪自编码模型后,丢弃它的解码器,将该降噪自编码模型的输出作为第二个降噪自编码模型的输入,训练第二个降噪自编码模型。接下来采用同样的方法,逐一训练各个降噪自编码模型,直到训练结束;
按照逐层贪婪算法,编写适用于Caffe的模型求解文件,结合步骤S1准备的基因数据,训练步骤S2定义的堆栈降噪自编码基因特征提取模型。
步骤S4是在训练好的堆栈降噪自编码基因特征提取模型的基础上,添加一个softmax分类器,构成一个有监督分类模型。然后按照步骤S1准备的具有对应性状标签的基因数据,利用该有标签的基因数据训练该监督分类模型;
监督分类模型的权重初始值为步骤S3逐层贪婪训练得到的模型参数;
编写基于Caffe的有监督分类模型的描述文件和基于Caffe的有监督分类模型的微调求解文件。保存训练结果中堆栈降噪自编码各层参数作为堆栈降噪自编码基因信息特征提取模型的参数。
步骤S5是结合步骤S2定义的堆栈降噪自编码基因信息特征提取模型和步骤S4训练得到的经微调的特征提取模型参数,构造一个新的基因信息特征提取模型;
利用该新建的基因特征提取模型可搭建基因对应性状识别系统,该基因性状识别系统核心部分为经过监督分类模型微调参数结合的堆栈降噪自编码基因信息特征提取模型,因此将待检测的基因序列信息,转化为可视化的无标签基因图像数据,利用以上基因信息特征体模型进行基因特征提取并分类识别,输出即为具有对应性状标签的基因识别结果。
以上所述,仅为本发明较佳的具体实施方式,但本发明的保护范围并不局限于此,任何熟悉本技术领域的技术人员在本发明揭露的技术范围内,根据本发明的技术方案及其发明构思加以等同替换或改变,都应涵盖在本发明的保护范围之内。
- 还没有人留言评论。精彩留言会获得点赞!