基于影像组学的脑胶质瘤分子标记物无损预测方法和预测系统与流程
本发明属于计算机辅助诊断技术领域,具体为基于影像组学(Radiomics)的脑胶质瘤分子标记物无损预测方法和预测系统。
背景技术:
胶质瘤是最常见的脑部恶性肿瘤,其中约30%为低级别胶质瘤(LGG,WHO分级I和II级)。尽管低级别胶质瘤有相对较好预后,但几乎所有的低级别胶质瘤都会发展为具有高死亡率的高级别胶质瘤。与胶质母细胞瘤(GBM,WHO IV级)相比,对LGG的基因表达分析和理解的研究相对欠缺。
IDH1(异柠檬酸脱氢酶1)具有显著的诊断、预后、预测价值,是胶质瘤中最重要的分子标记物[1]。大多数的较低级别胶质瘤(WHO II级和III级)及继发性的GBM都存在IDH1突变,而原发性的GBM中较少观察到IDH1突变;IDH1独立于常规预后指标与较长无进展生存期有关,含有IDH基因突变的高级别胶质瘤有显著较好预后;IDH1突变和1p/19q共缺失的低级别胶质瘤对放化疗敏感;IDH1野生型的低级别胶质瘤在分子及临床表现上与胶质母细胞瘤相似;IDH1突变的病人实施最大化切除可获得生存期的增加,因此IDH1突变可指导最大化肿瘤切除。正因为IDH1的重要临床价值,胶质瘤IDH1状态的评估无疑具有重要意义。现在临床上IDH1状态评估主要是通过获取肿瘤组织后进行基因测序得到。
无损IDH1状态的预测将极大的帮助临床进行胶质瘤的早期诊断和治疗方案制定。作为新兴的医学影像处理技术,影像组学通过从医学影像中自动提取高通量的图像特征,挖掘和建立图像特征和基因、蛋白、代谢、生理等指标的关联[2]。近期,影像组学已经应用在肺癌、乳腺癌、前列腺癌、头颈癌的分子分型、肿瘤异质性、肿瘤检测等的研究中,并取得了初步的成功。本发明的目的是设计一套从常规磁共振图像中提取高通量图像特征,进而得到以IDH1为例的分子标记物状态的标准化无损预测方法。
技术实现要素:
本发明的目的是提出一种自动化、规范化的基于影像组学的脑胶质瘤分子标记物IDH1无损预测方法。
本发明构造的影像组学方法,框架如图1所示,包含了图像分割、配准、特征提取、特征筛选、分类决策等环节。
本发明提出的基于影像组学的胶质瘤分子标记物IDH1无损预测方法,其步骤如下:
步骤一.图像分割
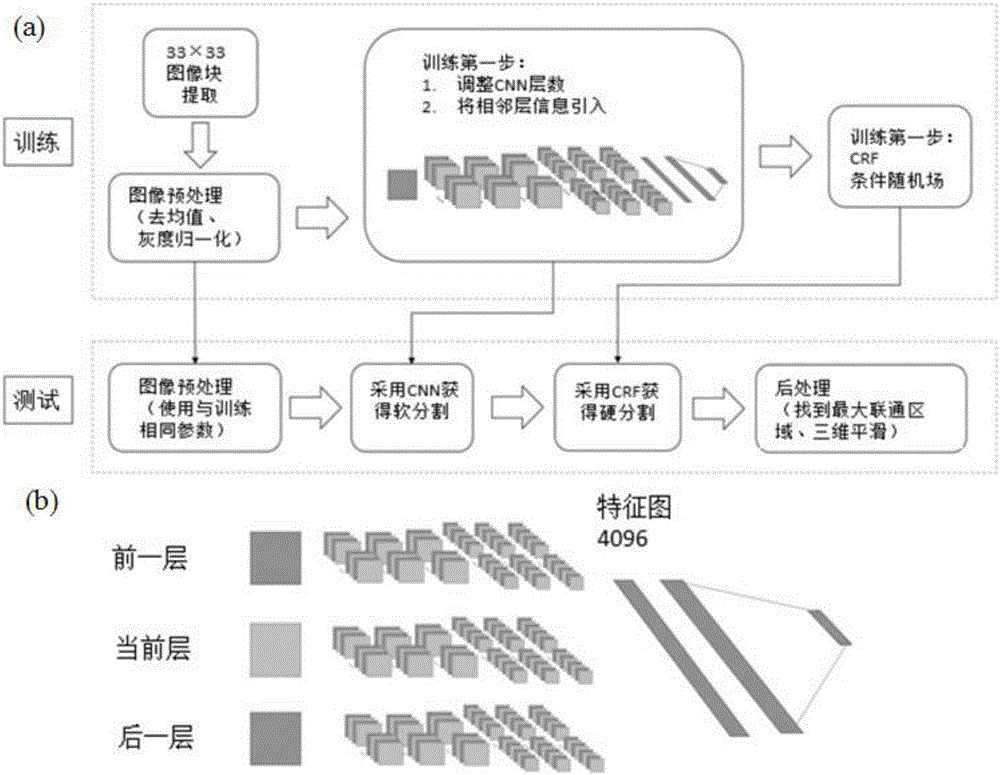
图像分割是影像组学中的关键和瓶颈问题,随着深度学习在图像处理领域的广泛应用,基于深度学习的医学影像自动分割显示出比传统方法更好的分割精度和鲁棒性。本发明中,我们采用了基于卷积神经网络的磁共振影像分割方法,在文献[3]报道方法的基础上进行了网络结果的调整,将磁共振图像的三维信息引入到传统二维CNN图像分割中,将全连接的条件随机场(CRF)被作为后处理环节加入到分割方法中,使得网络对对比度低的低级别胶质瘤也有很好的分割效果。
本发明设计的CNN胶质瘤磁共振影像分割方法,具有如图2(a)所示的结构。CNN网络中包括4层卷积层,2层池化层,2层全连接层。文献[3]中的CNN的输入为二维图像,即为二维CNN。由于低级别胶质瘤较高级别胶质瘤具有更小的尺寸、更低的图像对比度,直接用[3]的方法分割结果欠理想,因此针对低级别胶质瘤的分割,对CNN结构做了如下调整。一是将临近层的磁共振图像信息送入CNN网络中,见图2(b),即将相邻层的信息引入到当前层的训练中,实现了输入信息的三维化,使得网络对体积较小的胶质瘤也能得到较好的分割结果;二是将全连接的条件随机场(CRF)被作为后处理环节加入到胶质瘤的图像分割中,使得网络对对比度低的低级别胶质瘤也有很好的分割效果。
通过前后两个环节的改进,使分割效果的Dice相似系数从原始CNN的0.76提高到0.85。
我们使用卷积神经网络和条件随机场结合来改进脑胶质瘤的图像分割。首先将包含肿瘤区域的脑部磁共振图像划分为若干个小块,以中心点的类别作为目标输入到卷积神经网络进行训练,使用随机梯度下降法使网络的权值反向传递,获得稳定的网络。
在测试的阶段,我们将整幅图像输入到网络之中,将最后一个全连接层之后的特征图进行上采样至输入的大小,以作为条件随机场的单元势函数。条件随机场利用单元势函数的信息进行若干次循环,获得最后准确的肿瘤区域。
条件随机场(CRF)后处理环节的具体实现为,先计算如下每个像素点的单元势函数:
θu(xi)=-log P(xi) (1)
其中,P(xi)是最后一个全连接层的特征图经过上采样,θu(xi)是获得能单元势函数:
其中,E(x)是条件随机场的能量函数,θp(xi+xj)是任意两个像素点i和j之间的势函数,通过以下计算获得:
其中,μp(xi+xj)是判断两个点是不是同一个点,不是得话则是1。之后为定义的势函数的核,可以由以下计算所得
式中,pi和Ii代表像素i在CNN网络中的位置和灰度。可以看出,前一项为两个像素点位置和灰度值之间差别的权值,后一项为对于位置的模糊项。ω为两项的权值,σ为两项的方差,这5个参数和条件随机场的循环次数会影响分割的准确性,需要从训练集训练获得。在CNN分割结果的基础上,每层中具有最大相似度的区域被标定为肿瘤区域。
步骤二.特征提取
位置特征提取。首先将分割后的肿瘤配准到标准脑图集,标准脑图集采用MN152(Montreal Neurological Institute(MNI)),配准方法采用MNI提供的SPM12软件;采用Anatomical Automatic Labeling(AAL)方法将标准脑图集划分为116个感兴趣区域AVOI(anatomical volumes of interest);对配准到MN152的胶质瘤,统计其落于116个AVOIs的情况,若肿瘤落在某个AVOI的体素量大于10则认为该胶质瘤在此AVOI,记为1,否则记为0。因此对每个病例可得到116个0和1相间的字符串表示该胶质瘤在全脑的分布情况;
对IDH1突变型和野生型胶质分别统计在全脑发生的情况,通过独立样本T检验和U检验(Independent-samples T test and Mann-Whitney U test)统计两类胶质瘤位置分布的统计差异;将每个病例的位置分布表作为116个位置特征用于后继的影像组学(Radiomics)分析。
除了116个位置特征外,还提取了灰度特征21个,形状特征15个,纹理特征39个(这些特征的提取方法详见附录1)对灰度和纹理等60个特征进行三维小波分解,得到480个小波特征;共计671个特征。特征列表见表1。其中,各个特征的计算方法可参见[2],[4]-[6]。
上述高通量特征,共计671个,具体列表如下:
位置特征,共11个,统计肿瘤出现在AAL共116个分区的出现情况;
灰度特征,共21个,具体为:1)能量,2)直方图的熵,3)峰值,4)最大值,5)平均绝对误差,6)平均,7)中值,8)最小值,9)灰度范围,10)均方根,11)歪斜度,12)标准差,13)直方图均匀度,14)方差,15)高斯拟合的参数a,16)高斯拟合的参数b,17)高斯拟合的参数c,18)直方图均值,19)直方图方差,20)直方图歪斜度,21)直方图峰值。
形状特征,共15个,具体为:1)紧密度1,2)紧密度,3)最长距离,4)不对称度,5)类球度,6)表面积,7)表面积体积比,8)体积,9)面积边界框比,10)最长的椭圆长轴,11)最短的椭圆短轴,12)离心率,13)方向,14)紧致度,15)傅里叶描述子。
纹理特征,共39个,其中:
灰度共生矩阵,有8个,具体为:1)能量,2)对比度,3)相关度,4)同质性,5)方差,6)平均值之和,7)熵,8)不同度;
灰度行程矩阵,有13个,具体为:11)灰度不均匀性,12)长线不均匀性,13)长线百分比,14)低灰度值的线度量,15)高灰度值的线长度,16)短线的低灰度值的线度量,17)短线的高灰度值的线度量,18)长线的低灰度值的线度量,19)长线的高灰度值的线度量,20)灰度值方差,21)长线方差;
灰度区域大小矩阵,有13个,具体为:22)小区块度量,23)大区块度量,24)灰度不均匀性,25)区块不均匀性,26)区块百分比,27)低灰度值的区块度量,28)高灰度值的区块度量,29)小区域的低灰度值的区块度量,30)小区域的高灰度值的区块度量,31)大区域的低灰度值的区块度量,32)大区域的高灰度值的区块度量,33)灰度值方差,34)区块大小方差;
领域灰度矩阵,有5个,具体为:35)粗糙度,36)对比度,37)忙碌度,38)复杂度,39)强度;
小波,共480个,为小波三个方向的8个高频低频分量。
步骤三.特征筛选
671个高通量特征中,许多特征间是高度相关的冗余特征,若将这些特征直接用于IDH1的预测将会造成分类器的过敏感。本发明中,采用两步特征筛选法进行特征选择。第一步,基于独立样本t检验,选出p<0.05即有统计差异的特征,此步骤中共选择特征197个;第二步,采用改进的遗传算法对197个特征进行进一步筛选,得到110个特征。遗传算法[7]从随机产生的一群初始解开始搜索,种群中的个体称为染色体,每个染色体都是优化问题的一个解。通过模拟染色体的选择、交叉、变异,按照优胜劣汰的机制不断产生后代种群。选择策略的优劣直接决定了后代种群的性能,通常需要一个适应度函数对染色体的优劣程度进行评价,传统的适应度函数将分类的准确率作为标准。若仅采用分类准确率作为适应度函数,则会由于特征间的强相关性、冗余性造成搜索不到最优解。为了解决这个问题,本发明中提出一种基于最小冗余-最大相关(minimal-redundancy-maximal-relevance,mRMR)准则的适应度函数。在特征空间Ω内,已选特征子集S内特征间的最小冗余定义为:
其中,S代表特征子集的集合,I(di;dj)表示特征di与特征dj之间的互信息,m为特征子集的大小。S与目标类别c间的最大相关定义为:
其中,c为目标类别,I(di;c)为特征di与类别c之间的互信息。
则mRMR准则定义为:
即用选择最小冗余减去最大相关的最大值作为选择标准,将特征空间的若干个特征进行排序。
基于mRMR准则的适应度函数定义为:
其中,Accuracy为使用S所获得的分类准确率,Rank表示所选择特征的mRMR排序值之和。
步骤四.分类判决
将遗传算法选出的110个特征送入分类器进行IDH1状态预测,分类器采用经典的支持向量机和AdaBoost算法进行分类。
本发明采用留一法交叉验证(Leave-one-out cross-validation,LOOCV)作为验证模型,为全面评价Radiomics方法的IDH1预测性能,奔放没采用七个量化指标对预测性能进行定量化评价,分别为:准确度(accuracy,ACC)、敏感度(sensitivity,SENS)、特异度(specificity,SPEC)、阳性预测值(positive predictive value,PPV)、阴性预测值(negative predictive value,NPV)、Matthew相关系数(Matthew’s correlation coefficient,MCC);另外,ROC曲线下的面积(area under the ROC curve,AUC)作为整体评估准则。各指标计算方法见附录2。
对应于上述胶质瘤分子标记物IDH1无损预测方法,本发明还构建了基于影像组学的胶质瘤分子标记物IDH1无损预测系统,其包括四个模块:图像分割模块,特征提取模块,特征筛选模块,分类判决模块,分别用于执行预测方法的图像分割、特征提取、特征筛选、分类判决四个步骤的操作运算;其中,图像分割模块包括上述公式(1)-(4)等的运算;特征提取模块分为位置提取子模块,灰度特征提取子模块,形状特征提取子模块,纹理特征提取子模块,小波分解计算子模块,分别用于执行置特征提取、灰度特征提取、形状特征提取、纹理特征提取、小波分解计算的操作运算;这些子模块中包含附录1中对应的计算公式。
本发明设计了一套从常规磁共振图像中提取高通量图像特征,进而得到分子标记物状态的标准化方法,从图像分割、特征提取、特征筛选、分类判决四个步骤设计了行之有效的解决方案,最终以IDH1预测为例获得了胶质瘤分子标记物的无损预测。
附图说明
图1、本发明提出Radiomics方法的流程图。
图2、(a)图像分割所用的改进卷积神经网络算法框图,(b)三维信息引入CNN示意图。
图3、76例IDH1突变和34例IDH1野生型的肿瘤在标准脑图集上叠加的结果。
图4、两类肿瘤位置分布差异显著区域。
图5、110例病例671个特征的无监督聚类热图。
图6、两分类器IDH1预测的ROC曲线图。
具体实施方式
以下是整个算法的具体实现步骤:
1、首先对原始图像进行去脑壳、灰度归一化等操作,对30例240片次公正图像进行手工标注作为CNN的训练集,将图像划分我32*32的小块送入网络进行训练。
2、采用图2所示的CNN对图像进行分割,随后用CRF能量随机场对分割结果进行调整。
3、将分割好的肿瘤用SPM12映射到标准脑图集MN152,对76例IDH1突变和34例IDH1野生型肿瘤在标准脑图集上进行分别叠加,将叠加结果划分为AAL116个分区,统计两类肿瘤在116个分区上的分布作为116个位置特征。
4、分别提取表一中所示的灰度、形状、纹理、小波特征共555个,加上116个位置特征,对每个病例共计提取671个特征。
5、对671个特征进行独立样本t检验,去除p>0.05的不显著特征;用改进遗传算法对筛选出的197个特征再进行筛选,最终得到110个特征。
6、基于110个特征,用SVM和Adaboost算法对IDH1进行预测,采用LOOCV留一法作为交叉验证法,统计预测准确度、灵敏度、特异性等8个指标。
结果分析
图3给出了76例IDH1突变和34例IDH1野生型的肿瘤在标准脑图集上叠加的结果,可见两类肿瘤在位置分布上有明显不同。将标准脑图划分为116个AAL分区后,两类肿瘤在第14,40,68,70和88个分区上有明显差异,五个区域的示意图见图4。
图5给出了110例病例671个特征的无监督聚类热图,图顶部蓝色和黄色分别表示特征无监督聚类效果,红色和绿色分别表示真实的IDH1状态,可见高通量特征与IDH1状态有强相关性。
表2给出了SVM和Adaboost分类器在不同特征数量下用留一法交叉验证得到对IDH1预测的结果。可见本发明提出的Radiomics方法能得到准确率为80%的IDH1预测,ROC曲线下面积达到86%。图6给出了两个分类器的ROC曲线图。
表2.两种分类算法在不同特征数量下的预测效果
附录1:
灰度特征:
1)能量
其中,N为图像体素的全部,X为像素点的灰度值。
2)直方图的熵
其中P为灰度值分布在直方图的区间Nl的数目。
3)峰值
其中,为灰度值的平均值。
5)均值
10)均方根
11)歪斜度
12)标准差
13)直方图的均匀度
14)方差
形状特征:
1)紧密度1
其中,V为肿瘤的体积,A为肿瘤的表面积。
2)紧密度2
4)不对称度
其中,R为肿瘤拟合的椭圆。
5)类球度
7)表面积体积比
纹理特征
灰度共生矩阵
1)能量
其中,p(i,j)为灰度共生矩阵,由以下定义:
其中,P(i,j)为灰度值i和灰度值j在三维空间的连接数,Ng为灰度值的总数。
2)对比度
3)相关度
其中,μi和μj为i和j的加权和。
4)同质性
5)方差
6)平均值之和
7)熵
8)不同度
灰度行程矩阵
9)短线度量
其中,p(i,j)为灰度行程矩阵,由以下定义:
其中,P(i,j)为长度为j的灰度值i的数目,Ng为灰度值的总数,Lr为长度的总数。
10)长线度量
11)灰度不均匀性
12)长线不均匀性
13)长线百分比
14)低灰度值的线度量
15)高灰度值的线长度
16)短线的低灰度值的线度量
17)短线的高灰度值的线度量
18)长线的低灰度值的线度量
19)长线的高灰度值的线度量
20)灰度值方差
其中,μi为i的加权和,类似的
21)长线方差
其中,μj为j的加权和,
灰度区域大小矩阵
22)小区块度量
其中,p(i,j)为灰度区域大小矩阵,由以下定义:
其中,P(i,j)为面积为j的灰度值i的数目,Ng为灰度值的总数,Lr为面积的总数。
23)大区块度量
24)灰度不均匀性
25)区块不均匀性
26)区块百分比
27)低灰度值的区块度量
28)高灰度值的区块度量
29)小区域的低灰度值的区块度量
30)小区域的高灰度值的区块度量
31)大区域的低灰度值的区块度量
32)大区域的高灰度值的区块度量
33)灰度值方差
其中,μi为i的加权和,类似的
34)区块大小方差
其中,μj为j的加权和,
领域灰度矩阵
35)粗糙度
其中,ε是一个很小的值,P(i)是灰度值i在三维空间的总和,
其中,Ni是肿瘤区域所有灰度值的总数,是三维连接体素灰度值的平均值。
36)对比度
37)忙碌度
38)复杂度
39)强度
附录2:
其中,TP,FP,TN和FN分别代表了正确阳性,错误阳性,正确阴性和错误阴性代表的数目。
参考文献
[1]Weller M,Pfister SM,Wick W,Hegi ME,Reifenberger G,Stupp R(2013)Molecular neuro-oncology in clinical practice:a new horizon.Lancet Oncol.DOI:10.1016/S1470-2045(13)70168-2.
[2]Aerts HJ,Velazquez ER,Leijenaar RT,et al(2014)Decoding tumour phenotype by noninvasive imaging using a quantitative radiomicsapproach.NatCommun.DOI:10.1038/ncomms5006.
[3]Pereira S,Pinto A,Alves V,Silva CA(2016)Brain Tumor Segmentation using Convolutional Neural Networks in MRI Images.IEEE Trans Med Imaging.DOI:10.1109/TMI.2016.2538465.
[4]Vallières M,Freeman CR,Skamene SR,ElNaqa I(2015)A radiomics model from joint FDG-PET and MRI texture features for the prediction of lung metastases in soft-tissue sarcomas of the extremities.Phys Med Biol.DOI:10.1088/0031-9155/60/14/5471.
[5]Collewet G,Strzelecki M,Mariette F(2004)Influence of MRI acquisition protocols and image intensity normalization methods on texture classification.MagnReson Imaging.DOI:10.1016/j.mri.2003.09.001.
[6]Haralick RM,Shanmugam K,Dinstein I(1990)Textural features for image classification.IEEE Trans Syst Man Cybern B Cybern.DOI:10.1109/TSMC.1973.4309314.
[7]DebK,Pratap A,AgarwalS,Meyarivan T.A fast and elitist multiobjective genetic algorithm:NSGA-II.IEEE T.Evolutionary Computation,2002,6:182-197.。
- 还没有人留言评论。精彩留言会获得点赞!