一种基于深度学习的肠道病变识别方法与流程
本发明涉及病变识别技术领域,特别是涉及一种基于深度学习的肠道病变识别方法。
背景技术:
结肠癌是我国常见消化系肿瘤之一,随着生活水平的不断提高,结肠癌发病率在不断增加。腺瘤性息肉与结肠癌的发生、发展密切相关,目前腺瘤性息肉可能发生癌变已经得到公认,腺瘤性息肉属于癌前病变,如果可能会导致癌症的息肉能够被检测出来并且切除掉,那么就可以极大地防止大肠癌的发生。
电子肠镜是目前最主要的肠道病变检查方法。检查时着重观察有利于肠道病变的诊断和治疗。然而,就结肠息肉这一常见病变来说,目前的结肠镜检查手术中仍然存在着10%-30%的漏诊率,因此提高肠道病变的识别率具有很高的临床应用价值。
深度学习技术是近十年来人工智能领域取得的最重要突破之一,它在语音识别、自然语言处理、计算机视觉、图像与视频分析等诸多领域都取得了巨大成功。到目前为止,针对肠镜图像与视频中病变的自动检测已经进行了许多研究,比如使用svm、rf、knn等利用形状、颜色、背景、纹理等信息来检测肠道病变,但是这些现有的技术方案在检测速度以及检测精度上都达不到理想的效果。
技术实现要素:
本发明的目的是针对现有技术中存在的技术缺陷,而提供一种基于深度学习的肠道病变识别方法,以解决现有检测方法检测速度慢,难以达到实时检测标准的问题。
为实现本发明的目的所采用的技术方案是:
一种基于深度学习的肠道病变识别方法,包括以下步骤;
步骤101,搭建深度学习框架;
步骤102,各类常见肠道病变图片收集;
步骤103,图片数据预处理:
步骤104,使用yolov3目标检测算法构建肠道病变识别模型;
步骤105,使用图片数据预处理后形成的数据集在深度学习框架中进行训练,得到包含多类肠道病变目标检测相关模型;
步骤106,通过前端web界面通过发送数据到后端,调用后端的肠道病变目标检测相关模型开始检测,将检测结果返回给前端web界面。
所述图片数据预处理的步骤如下:
步骤201,按照病变类型对图片进行分类;
步骤202,将所有图片按照固定顺序重命名;
步骤203,使用图像标注工具对原始图片进行标注,使用矩形框标出图片中的病变组织,根据图像尺寸、矩形框坐标、病症种类信息生成xml格式文档,然后使用python编写脚本将xml文件转换为txt格式文件,转换完的txt文件记录着每张图片中的病变类别信息以及坐标信息。
步骤204,根据图片与标注文件制作voc数据集。
所述图像标注工具可以为labelimg、labelme、vatic、sloth的一种。
所述深度学习框架可以为darknet、tensorflow、caffe中一种。
与现有的肠道病变识别方法相比,本发明的有益效果是:
本发明通过使用yolov3目标检测算法构建肠道病变识别模型,具有较快的检测速度。经过测试,平均测试每一幅图片的时间为31.959ms,即检测速度可以达到31fps以上,能够达到实时检测的水平。可以在临床结肠镜手术中辅助医生完成检查,尽可能地避免息肉、憩室、出血等常见病变的漏检和误检。
附图说明
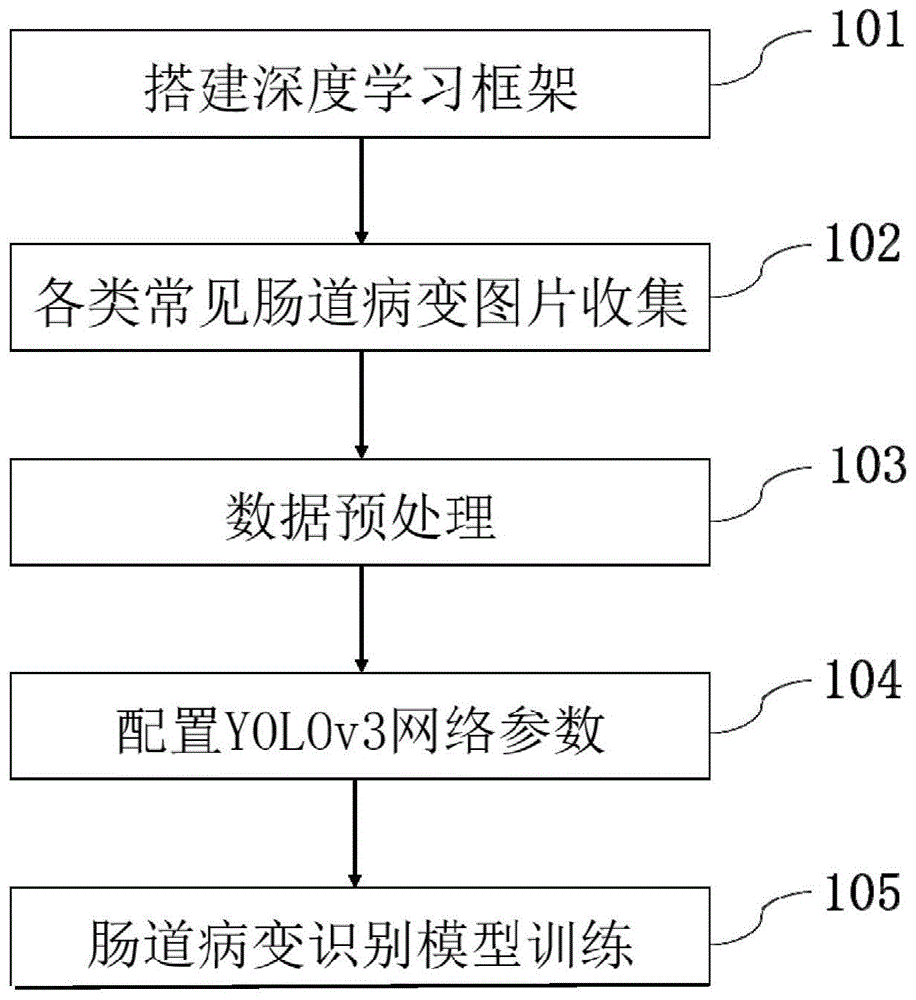
图1为本发明基于深度学习的肠道病变识别方法中病变识别模型构建的流程图;
图2为本发明基于深度学习的肠道病变识别方法中数据预处理的流程图;
图3为本发明基于深度学习的肠道病变识别方法中前端web界面与后端肠道病变识别模型通信示意图。
具体实施方式
以下结合附图和具体实施例对本发明作进一步详细说明。应当理解,此处所描述的具体实施例仅仅用以解释本发明,并不用于限定本发明。
本发明是利用深度学习框架可以对处理后的图像数据进行训练,然后使用yolov3目标检测算法建立肠道病变识别模型以进行深度学习,识别肠道病变的。
如图1所示,本发明基于深度学习的肠道病变识别方法,包括步骤:
步骤101,搭建深度学习框架。如在linux系统下,安装nvidia驱动以及必备依赖库和相关软件:python2.7,cuda8.0,cudnn6.0.21,opencv3.4.0,最后安装深度学习框架;
本实施例中,上述的深度学习框架可以为darknet、tensorflow、caffe等,本实施例对此不做限定,利用深度学习框架可以对处理后的图像数据进行训练。
步骤102,各类常见肠道病变图片收集。从医院的多位经验丰富的内窥镜专家在医院进行的多年内窥镜临床手术中获取各类肠道病变图片,图片格式为jpeg。
步骤103,数据预处理。请参见图2所示,包括以下步骤:
步骤201,按照病变类型对图片进行分类。可以按照常见肠道病变如结肠息肉、结肠憩室、肠道出血等进行分类。
步骤202,将所有图片按照固定顺序重命名。
步骤203,使用图像标注工具对原始图片进行标注。使用矩形框标出图片中的病变组织,根据图像尺寸、矩形框坐标、病症种类等信息生成xml格式文档。然后使用python编写脚本将xml文件转换为txt格式文件,转换完的txt文件记录着每张图片中的病变类别信息以及坐标信息。
本实施例中,上述图像标注工具可以为labelimg、labelme、vatic、sloth等可用于目标检测任务数据集制作的软件,本实施例对此不做限定。
步骤204,根据图片与标注文件制作voc数据集。
步骤104,配置yolov3网络相关参数,构建肠道病变识别模型。根据voc数据集配置batch/subdivisions=64/16、learning_rate=0.001、max_batches=30000、steps=2500,3500。然后根据已经标注完成的训练样本,使用k-means聚类计算出anchors参数的最优初始值,设置扩充数据集相关参数:angle=5,saturation=1.5,exposure=1.5,hue=0.1。
步骤105,肠道病变识别模型的训练。根据病变种类数量修改classes相关参数,执行语句使机器在gpu中开始训练肠道病变识别模型,得到包含多类肠道病变目标检测相关模型。
待上述步骤执行到105之后,可得到包含多类肠道病变目标检测相关模型,随后使用vue.js构建用户界面的渐进式框架进行前端web界面301的开发,采用自底向上增量开发的设计,并且使用websocket通信协议,与后端服务器302通信,完成前后端通信功能。
图3为本申请基于深度学习的肠道病变识别方法中前端web界面与后端肠道病变识别模型通信示意图:前端web界面301与后端服务器302通过websocket通信协议实现相互通信,后端服务器部分302包含了肠道病变识别模型。前端web界面通过websocket发送数据到后端,调用后端的肠道病变识别模型开始检测,然后通过websocket数据接收将检测结果返回给前端web界面。
以上所述仅是本发明的优选实施方式,应当指出的是,对于本技术领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以做出若干改进和润饰,这些改进和润饰也应视为本发明的保护范围。
- 还没有人留言评论。精彩留言会获得点赞!