基于CUDA的基因数据处理方法、装置和CUDA构架与流程
基于cuda的基因数据处理方法、装置和cuda构架
技术领域
1.本技术涉及基因数据技术领域,具体涉及一种基于cuda的基因数据处理方法、装置、cuda构架和存储介质。
背景技术:
2.随着基因测序技术的不断发展和基因检测服务的持续推广,该方法被广泛地应用于新物种、病毒以及疾病的研发分析中;与此同时大量的基因测序数据大量涌出,如何高效地完成对这些数据进行分析处理就显得尤为重要。
3.目前,常用的基因数据分析过程为:数据分析人员在x86构架的cpu在(ccentral processing unit,中央处理器)运行一些基因分析相关算法从而完成对基因数据分析。然而,该过程繁琐,处理效率低,并且cpu和内存等的利用率也低,无法使单位时间内执行任务最大化,导致出现无法充分利用系统资源的问题。
技术实现要素:
4.有鉴于此,本技术实施例中提供了一种基于cuda的基因数据处理方法、装置、设备和存储介质。
5.第一方面,本技术实施例提供了一种基于cuda的基因数据处理方法,所述cuda包括主机端和设备端,所述方法应用于所述设备端;所述方法包括:
6.从所述主机端的内存中获取待处理的基因测序数据;
7.将所述待处理的基因测序数据存储于资源池;
8.采用graph方法从所述资源池中获取所述待处理的基因测序数据,并采用多线程并行方式对所述待处理的基因测序数据进行分析,得到相应的分析结果;
9.将所述相应的分析结果发送至所述主机端,以供所述主机端根据所述相应的分析结果得到基因数据分析结果。
10.第二方面,本技术实施例提供了一种基于cuda的基因数据处理装置,该装置包括:
11.所述cuda包括主机端和设备端,所述装置应用于所述设备端;所述装置包括:
12.数据获取模块,用于从所述主机端的内存中获取待处理的基因测序数据;
13.数据存储模块,用于将所述待处理的基因测序数据存储于资源池;
14.数据分析模块,用于采用graph方法从所述资源池中获取所述待处理的基因测序数据,并采用多线程并行方式对所述待处理的基因测序数据进行分析,得到相应的分析结果;
15.结果发送模块,用于将所述相应的分析结果发送至所述主机端,以供所述主机端根据所述相应的分析结果得到基因数据测试结果。
16.第三方面,本技术实施例提供了一种cuda构架,包括:包括主机端和设备端;所述设备端包括资源池和多个gpu;
17.所述主机端用于在内存空闲时存储待处理的基因测序数据,并在所述设备端的资
源池空闲时将所述待处理的基因测试数据发送至所述设备端;
18.空闲状态的所述gpu用于执行上述第一方面提供的基于cuda的基因数据处理方法。
19.第四方面,本技术实施例提供了一种计算机可读取存储介质,计算机可读取存储介质中存储有程序代码,程序代码可被处理器调用执行上述第一方面提供的基于cuda的基因数据处理方法。
20.本技术实施例提供的基于cuda的基因数据处理方法、装置、设备和存储介质,其中cuda包括主机端和设备端,主机端可以将存储于内存中的待处理的基因测序数据发送至设备端,设备端将待处理的基因测序数据存储于资源池,然后设备端采用graph方法从资源池中获取待处理的基因测序数据,并采用多线程并行方式对待处理的基因测序数据进行分析,得到相应的分析结果,并将相应的分析结果发送至主机端,以供主机端根据相应的分析结果得到基因数据分析结果。
21.该基因数据处理方法采用基于cuda的graph技术来对基因数据进行处理分析,可以实现gpu高效比对以及降低了gpu的延时,从而能大大提高基因数据处理的效率。
附图说明
22.为了更清楚地说明本技术实施例或现有技术中的技术方案,下面将对实施例或现有技术描述中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本技术的实施例,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据提供的附图获得其他的附图。
23.图1为本技术实施例提供的基于cuda的基因数据处理方法的应用场景(即cuda构架)示意图;
24.图2为本技术一个实施例提供的基于cuda的基因数据处理方法的流程示意图;
25.图3为本技术一个实施例提供环形缓存的结构示意图;
26.图4为本技术一个实施例提供的基于cuda的基因数据处理装置的结构图;
27.图5为本技术一个实施例中提供的计算机可读存储介质的结构示意图。
具体实施方式
28.下面将对本技术实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例仅仅是本技术一部分实施例,而不是全部的实施例。基于本技术中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本技术保护的范围。
29.名词解释:
30.基因(gene,mendelian factor)是指携带有遗传信息的dna或rna序列(即基因是具有遗传效应的dna或rna片段),也称为遗传因子,是控制性状的基本遗传单位。基因通过指导蛋白质的合成来表达自己所携带的遗传信息,从而控制生物个体的性状表现。基因测序是一种新型基因检测技术,从血液或唾液中分析测定基因全序列,从而预测罹患多种疾病的可能性,个体的行为特征及行为合理等。
31.短序列(read):是一小段短的测序片段,是高通量测序仪产生的测序数据,对整个
基因组进行测序,就会产生成百上千万的read,然后将这些read拼接起来就能获得基因组的全序列。
32.比对分析:ngs测序下来的短序列(read)存储于fastq文件里面,虽然它们原本都来自于有序的基因组,但在经过dna建库和测序之后,文件中不同read之间的前后顺序关系就已经全部丢失了。因此,fastq文件中紧挨着的两条read之间没有任何位置关系,它们都是随机来自于原本基因组中某个位置的短序列而已。因此,我们需要先把这一大堆的短序列捋顺,一个个去跟该物种的参考基因组比较,找到每一条read在参考基因组上的位置,然后按顺序排列好,这个过程就称为测序数据的比对。
33.排序分析:为什么bwa比对后输出的bam文件是没顺序的?原因是fastq文件里面这些被测序下来的read是随机分布于基因组上面的,第一步的比对是按照fastq文件的顺序把read逐一定位到参考基因组上之后,随即就输出了,它不会也不可能在这一步里面能够自动识别比对位置的先后位置重排比对结果。因此,比对后得到的结果文件中,每一条记录之间位置的先后顺序是乱的,我们后续去重复等步骤都需要在比对记录按照顺序从小到大排序下来才能进行,所以这才是需要进行排序的原因。
34.去除重复:在排序完成之后执行去除重复(即去除pcr重复序列)。什么是重复序列?它是如何产生的,以及为什么需要去除掉?这跟实验过程建库和测序相关。在ngs测序之前都需要先构建测序文库:通过物理(超声)打断或者化学试剂(酶切)切断原始的dna序列,然后选择特定长度范围的序列去进行pcr扩增并上机测序。因此,这里重复序列的来源实际上就是由pcr过程中所引入的。
35.碱基质量值校正:是为了(尽可能)校正测序过程中的系统性错误,因为变异检测是一个极度依赖测序碱基质量值的步骤。因为这个质量值是衡量我们测序出来的这个碱基到底有多正确的重要(甚至是唯一)指标。没办法直接测得,可通过统计学的技巧获得极其接近的分布结果。根据一个群体中发现的已知变异,在某个人身上也很可能是同样存在的。因此,可以对比对结果进行直接分析,先排除掉所有的已知变异位点,然后计算每个(报告出来的)质量值下面有多少个碱基在比对之后与参考基因组上的碱基是不同的,这些不同碱基就被我们认为是错误的碱基,它们的数目比例反映的就是真实的碱基错误率,换算成phred score,把这些信息输出为一份校准表文件,重新调整原来bam文件中的碱基质量值,并使用这个新的质量值重新输出一份新的bam文件。
36.变异检测分析:变异检测分析的目的是准确检测出每个样本(如人类)基因组中的变异集合,也就是人与人之间存在差异的那些dna序列。
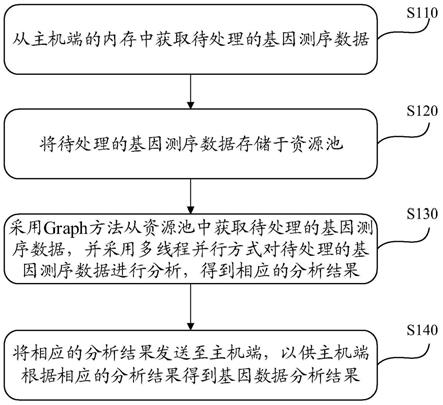
37.为了更详细说明本技术,下面结合附图对本技术提供的一种基于cuda的基因数据处理方法、装置、终端设备和计算机存储介质,进行具体地描述。请参考图1,图1示出了本技术实施例提供的基于cuda的基因数据处理方法的应用场景(即cuda构架)的示意图。cuda(compute unified device architecture)的中文全称为计算统一设备架构,是显卡厂商nvidia推出的运算平台。该应用场景包括本技术实施例提供的主机端(即host)100和设备端(即device)200。其中主机端(即host)中包括cpu102、芯片组104(即chipset)和存储器106(即dram),存储器106又称为资源池。设备端200包括存储器202(即dram)和gpu204,存储器202又称为资源池,包括局部内存(即local memory)和全局内存(即global memory)。
38.cpu(central processing unit,即中央处理器),是专为顺序串行处理而优化的
几个核心组成,cpu虽然每个核心自身能力极强,处理任务上非常强悍,无奈他核心少,在并行计算上表现不佳,擅长流程控制和逻辑处理,不规则数据结构,不可预测存储结构,单线程程序,分支密集型算法。因此,cpu是计算机的运算和控制的核心。
39.gpu(graphic processing unit),即中文名称图形处理器)由数以千计的更小、更高效的核心组成,这些核心专门为同时处理多任务而设计,可高效地处理并行任务,虽然他的每个核心的计算能力不算强,但他胜在核心非常多,可以同时处理多个计算任务,在并行计算的支持上做得很好。gpu擅长数据并行计算,规则数据结构,可预测存储模式。因此,gpu主要用作图形图像处理。图像在计算机呈现的形式就是矩阵,我们对图像的处理其实就是操作各种矩阵进行计算,而很多矩阵的运算其实可以做并行化,这使得图像处理可以做得很快,因此gpu在图形图像领域也有了大展拳脚的机会。下图表示的就是一个多gpu计算机硬件系统,可以看出,一个gpu内存就有很多个sp和各类内存,这些硬件都是gpu进行高效并行计算的基础。另外,gpu技术的发展是一个进化过程。20世纪70年代和80年代的早期gpu被用来加载和渲cpu中的2d图形计算,到20世纪90年代末,对3d渲染的支持就已经很普遍(buck,2010年)。随着2001年“可编程管道(pipeline)”的出现,通过使用在gpu上执行的相关的程序代码,可编程管道使程序员能够实现对“着色器(shader)”的自定义呈现操作。在着色器语言和可编程gpu的支持下,研究人员和开发人员开始利用可编程管道的通用性来解决与图形无关的问题。
40.对于gpu而言,其包括sp和sm(流处理器)。sp:最基本的处理单元,streaming processor,也称为cuda core。最后具体的指令和任务都是在sp上处理的。gpu进行并行计算,也就是很多个sp同时做处理。sm:多个sp加上其他的一些资源组成一个streaming multiprocessor。sm可以看做gpu的心脏(对比cpu核心)。cuda将这些资源分配给所有驻留在sm中的threads。因此,这些有限的资源就使每个sm中active warps有非常严格的限制,也就限制了并行能力。需要指出,每个sm包含的sp数量依据gpu架构而不同,fermi架构f100是32个,gf10x是48个,kepler架构都是192个,maxwell都是128个。简而言之,sp是线程执行的硬件单位,sm中包含多个sp,一个gpu可以有多个sm(比如16个),最终一个gpu可能包含有上千个sp。这么多核心“同时运行”,速度可想而知,这个引号只是想表明实际上,软件逻辑上是所有sp是并行的,但是物理上并不是所有sp都能同时执行计算(比如我们只有8个sm却有1024个线程块需要调度处理),因为有些会处于挂起,就绪等其他状态,这有关gpu的线程调度。
41.本技术实施例对设备端中gpu的数量和类型不加以限定。设备端中的gpu可以用来执行本技术实施例中提供的一种基于cuda的基因数据处理方法。
42.此外,主机端通过kernel来控制设备端中gpu的操作。kernel在gpu上执行的核心程序,这个kernel是运行在gpu的某一组线程块上。要理解kernel,必须要对kernel线程层次结构有一个清晰的认识。首先gpu上很多并行化的轻量级线程。kernel在device上执行时实际上是启动很多线程,一个kernel所启动的所有线程称为一个网格(grid),同一个网格上的线程共享相同的全局内存空间,grid是线程结构的第一层次,而网格又可以分为很多线程块(block),一个线程块里面包含很多线程,这是第二个层次。这就是cuda的线程组织结构。
43.基于此,那么在本实施例中,cpu主要工作为:(1)获取主机端中的空闲状态,空闲
则读取基因数据(即待处理的基因测序数据),当基因数据装载到设备端资源池后,将结果返回给主机端,主机端的内存成为空闲状态;(2)设备端完成对待处理的基因测序数据计算或处理后,会通过主机端计算资源流将设备端的计算结果拷贝到主机端的资源池中,cpu会获取主机端中的结果内容,通过多线程处理,完成结果的写出,然后将资源返回给主机端。
44.gpu的主要工作为:(1)获取设备端的计算资源,判断是否有空闲的设备端资源可用于计算分配;(2)完成gpu的多卡调度,可以是多个(例如2个)gpu卡为一组,有一个调度中心,其中每个gpu只负责管理自己辖区的gpu的设备端资源,每组内部通信,任务自己管理,可无限扩展,多个组之间会互相抢数据来完成工作;(3)获取基因数据(即基因数据资源)用于计算,且根据资源空余情况自动关闭数据拷贝。(4)启动cudagraph用于基因数据(即基因数据资源)计算。
45.基于此,本技术实施例中提供了一种基于cuda的基因数据处理方法。请参阅图2,图2示出了本技术实施例提供的一种基于cuda的基因数据处理方法的流程示意图,以该方法应用于图1中的设备端(即gpu)为例进行说明,包括以下步骤:
46.步骤s110,从主机端的内存中获取待处理的基因测序数据。
47.步骤s120,将待处理的基因测序数据存储于资源池。
48.其中,待处理的基因测序数据是对某一个或多个样本进行基因测序后产生的并需要进行生物信息学分析的数据,通常是一些fastq数据。由于一次测试可能产生的数据量比较大,在对数据进行生物信息学分析时可以分批次进行,因此待处理的基因测试数据可以是一次测序产生的一部分数据。
49.在本实施例中,主机端(即cpu)在空闲时,可以从测序仪中读取待处理的基因测序数据,并存储在内存中。然后当设备端空闲时,可以将主机端的内存中存储的待处理的基因测序数据发送至设备端,然后存储在设备端的资源池中。
50.步骤s130,采用graph方法从资源池中获取待处理的基因测序数据,并采用多线程并行方式对待处理的基因测序数据进行分析,得到相应的分析结果。
51.其中,graph是一种定义的计算方式,是一些加减乘除等运算的组合,类似于一个函数。它本身不会进行任何计算,也不保存任何中间计算结果。graph是tensorflow的一部分。tensorflow是一种符号式编程框架,首先要构造一个图(graph),然后在这个图上做运算。打个比方,graph就像一条生产线,session就像生产者。生产线具有一系列的加工步骤(加减乘除等运算),生产者把原料投进去,就能得到产品。不同生产者都可以使用这条生产线,只要他们的加工步骤是一样的就行。同样的,一个graph可以供多个session使用,而一个session不一定需要使用graph的全部,可以只使用其中的一部分。
52.计算图graph我们可以简单理解成一个电路板,我们在电路板定义好电路(定义计算和tensor),然后通过插头进行通电(通过session进行计算),整个电路就开始运作了。在本实施例中,采用graph方法可以为设备端提供计算资源管理和存储,用于gpu多卡的资源分配和任务调度。
53.另外,在对待处理的基因测序数据采用多线程并行方式进行分析,得到相应的分析结果。对待处理的基因测序数据进行分析包括但不限于比对分析、变异分析、注释分析、排序分析、去重分析以及质控分析等。
54.步骤s140,将相应的分析结果发送至主机端,以供主机端根据相应的分析结果得
到基因数据分析结果。
55.设备端对待处理的基因测序数据进行分析处理后,可以将分析结果发送至主机端,主机端根据分析结果可以得到最终的基因数据分析结果。主机端在得到最终的基因数据分析结果后可以生成相应的分析报告等。
56.本技术实施例提供的基于cuda的基因数据处理方法、装置、设备和存储介质,其中cuda包括主机端和设备端,主机端可以将存储于内存中的待处理的基因测序数据发送至设备端,设备端将待处理的基因测序数据存储于资源池,然后设备端采用graph方法从资源池中获取待处理的基因测序数据,并采用多线程并行方式对待处理的基因测序数据进行分析,得到相应的分析结果,并将相应的分析结果发送至主机端,以供主机端根据相应的分析结果得到基因数据分析结果。
57.该基因数据处理方法采用基于cuda的graph技术来对基因数据进行处理分析,可以实现gpu高效比对以及降低了gpu的延时,从而能大大提高基因数据处理的效率。
58.在一个实施例中,设备端包括多个gpu;采用graph方法从资源池中获取待处理的基因测序数据,并采用多线程并行方式对待处理的基因测序数据进行分析,得到相应的分析结果,包括:任一个空闲状态的gpu采用graph方法确定各空闲状态的gpu的数据资源分配方案;各空闲状态的gpu根据数据资源分配方案从待处理的基因测序数据获取相应的基因数据资源,并对所基因数据资源采用多线程并行方式进行分析,得到相应的分析结果;其中基因数据资源为待处理的基因测序数据中的一部分基因测试数据。
59.具体而言,设备端通常包括多个gpu,每一个处于空闲状态的gpu都可以用来对待处理的基因测序数据进行分析。但为了提高数据处理的效率可以让多个gpu来对待处理的基因测序数据进行分析。因此,任一个空闲状态的gpu采用graph方法确定各空闲状态的gpu的数据资源分配方案,然后每一个空闲状态的gpu可以按照数据资源分配方案去资源库中抢取相应的基因数据资源,抢到基因数据资源的gpu就可以对基因数据资源进行分析处理,从而得到相应的分析结果。
60.需要说明的是基因数据资源是待处理的基因测试数据的一部分,对于每一个空闲状态的gpu来说,其抢取的基因数据资源大小或数量可能不相同,具体可以根据gpu的处理能力确定。
61.另外,空闲状态的gpu在对基因数据资源进行处理时通常是根据kernel函数进行计算。当空闲状态的gpu将基因数据资源处理完成后,可以将处理结果返回至设备端的资源库中,然后再去资源库中抢取基因数据资源。
62.在本实施例中,采用多个gpu对待处理的基因测序数据进行并行处理,能进一步地提高基因测序数据的处理效果。
63.进一步地,给出了一种对基因数据资源处理的具体实施方式,描述如下:
64.在一个实施例中,对所基因数据资源采用多线程并行方式进行分析,包括:对基因数据资源采用多线程并行方式依次进行质控分析和变异检测分析。
65.具体而言,对基因数据资源进行分析可以包括质控分析和变异检测分析。其中,质控分析是指对基因数据资源进行质量控制,从而确定基因测序数据是否准确有效。变异检测分析时对基因数据资源中的序列进行基因突变检测,从而确定突变结果等。
66.在一个实施例中,对基因数据资源采用多线程并行方式进行质控分析,包括:对基
因数据资源进行切分处理,以得到各短序列;对已有的变异位点数据库进行统计分析,得到变异分布;判断各短序列的碱基质量值,当任一短序列的碱基质量值大于或等于预设值,且任一短序列锚定到变异分布的密集区域时,采用可变k-mer对任一短序列进行比对匹配;或且任一短序列锚定到变异分布的稀疏区域时,采用恒定k-mer对任一短序列进行比对匹配;当任一短序列的碱基质量值小于预设值,对任一短序列进行过滤,并对过滤后的短序列进行比对;对比对结果进行排序和去重;判断去重后的比对结果的碱基质量值,当碱基质量值小于预设值时,对去重后的比对结果进行碱基质量值校正。
67.具体地,可以将基因数据资源进行切分,生成各短序列。然后对各短需要进行质控分析,例如变异位点检测、比对、排序、去重、重比对、碱基质量值校正等。其中具体过程为:可以先对参考序列(即reference)建索引,然后将基因数据资源(即read)切成短序列,例如小片段。然后将各短序列已有的变异位点数据库进行比对,得到变异分布,即将各短序列比对到参考序列的特异位置,根据变异位点之间的碱基长度的概率分布函数。根据概率分布在不同区域高效获得不同长度的精确匹配。之外做对比分析,通常是采用kernel函数做比对分析,在比对分析的过程中:1)判断各短序列(即fastq)的碱基质量值,当碱基质量值小于预设值,说明短序列的碱基质量值较低,此时则先对短序列进行过滤,再做后续计算;2)当碱基质量值大于等于预设值,说明短序列的碱基质量值较高,且锚定到变异分布较密集的区域,则取可变kmer进行匹配,提高匹配精度;3)若碱基质量值较高,且锚定到变异分布较稀疏的区域,则取较恒定的适配reads长度的kmer,提高匹配效率,即不整体先做过滤,判断测序质量没问题就做比对,有问题再过滤;比对的时候根据比对到的不同变异位置区域,采用不同kmer的匹配方法。
68.接下来对比对结果排序、去重以及对碱基质量值小于预设值的短序列进行碱基质量值校正;之后在质量值较高的区域进行变异检测,在质量值较低的区域进行碱基质量值校正,再做后续的变异检测。
69.采用该方式可以能快速且方便地对基因数据资源进行质控处理,从而得到质量良好的短序列,以便后续进行变异检测分析。
70.在一个实施例中,对基因数据资源采用多线程并行方式进行变异检测分析,包括:采用多线程并行方式查找各短序列的活性区域;对各活性区域进行重比对和局部组装;对重比对和局部组装后的各活性区域进行相似度计算和注释,以得到变异结果。
71.具体地,检测分析变异的过程中采用多线程模式构成流水线,查找active各短序列的活性区域(即active region),对确定的active region进行重比对和局部组装,然后进行相似度计算,最后计算注释的各种统计量,输出变异结果。其中,相似度是指计算两条序列的比对情况的似然概率,输入是一条参考基因序列称为halotype以及一条实际基因序列称为read。
72.在本实施例中,采用所线程模式的变异检测分析方法能快速地得到分析结果,大大提高了基因数据分析的效率。
73.接下来,还给出了一种待处理的基因测序数据的存储实施方式,详细描述如下:
74.在一个实施例中,将待处理的基因测序数据存储于资源池,包括:采用环形缓存的方式将待处理的基因测序数据存储于资源池。
75.具体地,可以待处理的基因测序数据在资源池存储方式可以是环形缓存方式。其
中环形缓存结构如图3所示,其中环形缓存结构中包括多个存储区域(即图中的每一个小格子表达一个存储区域),在需要对待处理的基因测试数据存储时可以根据环形缓存结构按照顺时针或逆时针方向依次将待处理的基因测试数据存入存储区域中,通常情况下将一个基因数据资源存储于一个存储区域中;在gpu去抢取基因数据资源时,也可以按照顺时针或逆时针方向依次抢取基因数据资源。
76.由于设备端中的gpu属于对显存资源的管理,释放资源本身也耗时knernel部分。在graph启动的计算任务,运行前后有分配函数负责拷贝和分配资源。总任务运行完成后,进行内存替换和释放,此处通过环形缓冲,实现内存的替换,减少了释放过程。
77.可选地,主机端在存储待处理的基因测序数据时也可以采用此环形缓冲结构,从读文件到输出不再需要或可以减少内存拷贝。
78.在一个实施例中,从主机端的内存中获取待处理的基因测序数据,包括:获取资源池的空闲情况;根据资源池的空闲情况主机端的内存中获取相应数量的待处理的基因测序数据。
79.其中,待处理的基因测序数据的数量或大小可以不固定。具体可以根据设备端中资源池的空闲情况确定,如资源池的内存占用较多,则表示不空闲,此时能存储待处理的基因测序数据的数量很小,那么可以从主机端获取数量较小的待处理的基因测序数据;若当资源池的内存占用较少,则表示空闲,此时能存储待处理的基因测序数据的数量比较多,那么可以从主机端获取数量较多的待处理的基因测序数据。采用该方式可以实时调整资源库中存储的待处理的基因测序数据,以避免数据量过大无法存储以及数据量过小造成空闲的gpu没有数据处理的情况,从而最大程度地提高数据处理效率。
80.应该理解的是,虽然图2的流程图中的各个步骤按照箭头的指示依次显示,但是这些步骤并不是必然按照箭头指示的顺序依次执行。除非本文中有明确的说明,这些步骤的执行并没有严格的顺序限制,这些步骤可以以其它的顺序执行。而且图2中的至少一部分步骤可以包括多个子步骤或者多个阶段,这些子步骤或者阶段并不必然是在同一时刻执行完成,而是可以在不同的时刻执行,这些子步骤或者阶段的执行顺序也不必然是依次进行,而是可以与其它步骤或者其它步骤的子步骤或者阶段的至少一部分轮流或者交替地执行。
81.上述本技术公开的实施例中详细描述了一种基于cuda的基因数据处理方法,对于本技术公开的上述方法可采用多种形式的设备实现,因此本技术还公开了对应上述方法的基于cuda的基因数据处理装置,下面给出具体的实施例进行详细说明。
82.请参阅图4,为本技术实施例公开的一种基于cuda的基因数据处理装置,cuda包括主机端和设备端,装置应用于设备端;装置包括:
83.数据获取模块410,用于从主机端的内存中获取待处理的基因测序数据。
84.数据存储模块420,用于将待处理的基因测序数据存储于资源池。
85.数据分析模块430,用于采用graph方法从资源池中获取待处理的基因测序数据,并采用多线程并行方式对待处理的基因测序数据进行分析,得到相应的分析结果。
86.结果发送模块440,用于将相应的分析结果发送至主机端,以供主机端根据相应的分析结果得到基因数据测试结果。
87.在一个实施例中,设备端包括多个gpu;其中,gpu中包括数据分析模块430。数据分析模块430,用于采用graph方法确定各空闲状态的gpu的数据资源分配方案;根据数据资源
分配方案从述待处理的基因测序数据获取相应的基因数据资源,并对所基因数据资源采用多线程并行方式进行分析,得到相应的分析结果;其中基因数据资源为待处理的基因测序数据中的一部分基因测试数据。
88.在一个实施例中,数据分析模块430,用于对基因数据资源采用多线程并行方式依次进行质控分析和变异检测分析。
89.在一个实施例中,数据分析模块430,用于对所述基因数据资源进行切分处理,以得到各短序列;对已有的变异位点数据库进行统计分析,得到变异分布;判断各短序列的碱基质量值,当任一短序列的碱基质量值大于或等于预设值,且任一短序列锚定到变异分布的密集区域时,采用可变k-mer对任一短序列进行比对匹配;或且任一短序列锚定到变异分布的稀疏区域时,采用恒定k-mer对任一短序列进行比对匹配;当任一短序列的碱基质量值小于预设值,对任一短序列进行过滤,并对过滤后的短序列进行比对;对比对结果进行排序和去重;判断去重后的比对结果的碱基质量值,当碱基质量值小于预设值时,对去重后的比对结果进行碱基质量值校正。
90.在一个实施例中,数据分析模块430,用于采用多线程并行方式查找各短序列的活性区域;对各活性区域进行重比对和局部组装;对重比对和局部组装后的各活性区域进行相似度计算和注释,以得到变异结果。
91.在一个实施例中,数据存储模块420,用于采用环形缓存的方式将待处理的基因测序数据存储于资源池。
92.在一个实施例中,数据获取模块410,用于获取资源池的空闲情况;根据资源池的空闲情况主机端的内存中获取相应数量的待处理的基因测序数据。
93.关于基于cuda的基因数据处理装置的具体限定可以参见上文中对于方法的限定,在此不再赘述。上述装置中的各个模块可全部或部分通过软件、硬件及其组合来实现。上述各模块可以硬件形式内嵌于或独立于终端设备中的处理器中,也可以以软件形式存储于终端设备中的存储器中,以便于处理器调用执行以上各个模块对应的操作。
94.请参考图1,图1其示出了本技术实施例提供的一种cuda构架的结构框图。cuda构架包括主机端100和设备端200;设备端100包括资源池和多个gpu;主机端用于在内存空闲时存储待处理的基因测序数据,并在设备端200的资源池空闲时将待处理的基因测试数据发送至设备端;空闲状态的gpu用于执行上述基于cuda的基因数据处理方法实施例中所描述的方法。
95.本领域技术人员可以理解,图1中示出的结构,仅仅是与本技术方案相关的部分结构的框图,并不构成对本技术方案所应用于其上的终端设备的限定,具体的终端设备可以包括比图中所示更多或更少的部件,或者组合某些部件,或者具有不同的部件布置。
96.综上,本技术实施例提供的终端设备用于实现前述方法实施例中相应的基于cuda的基因数据处理方法,并具有相应的方法实施例的有益效果,在此不再赘述。
97.请参阅图5,其示出了本技术实施例提供的一种计算机可读取存储介质的结构框图。该计算机可读取存储介质50中存储有程序代码,程序代码可被处理器调用执行上述基于cuda的基因数据处理方法实施例中所描述的方法。
98.计算机可读取存储介质50可以是诸如闪存、eeprom(电可擦除可编程只读存储器)、eprom、硬盘或者rom之类的电子存储器。可选地,计算机可读取存储介质50包括非瞬时
性计算机可读介质(non-transitory computer-readable storage medium)。计算机可读取存储介质50具有执行上述方法中的任何方法步骤的程序代码52的存储空间。这些程序代码可以从一个或者多个计算机程序产品中读出或者写入到这一个或者多个计算机程序产品中。程序代码52可以例如以适当形式进行压缩。
99.在本说明书的描述中,参考术语“一个实施例”、“一些实施例”、“示例”、“具体示例”、或“一些示例”等的描述意指结合该实施例或示例描述的具体特征、结构、材料或者特点包含于本技术的至少一个实施例或示例中。在本说明书中,对上述术语的示意性表述不必须针对的是相同的实施例或示例。而且,描述的具体特征、结构、材料或者特点可以在任一个或多个实施例或示例中以合适的方式结合。此外,在不相互矛盾的情况下,本领域的技术人员可以将本说明书中描述的不同实施例或示例以及不同实施例或示例的特征进行结合和组合。
100.对所公开的实施例的上述说明,使本领域专业技术人员能够实现或使用本技术。对这些实施例的多种修改对本领域的专业技术人员来说将是显而易见的,本文中所定义的一般原理可以在不脱离本技术的精神或范围的情况下,在其它实施例中实现。因此,本技术将不会被限制于本文所示的这些实施例,而是要符合与本文所公开的原理和新颖特点相一致的最宽的范围。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1