一种基于多层次聚类的脑部肿瘤图像分割方法
1.本发明属于医疗图像分割领域,具体涉及一种基于多层次聚类的脑部肿瘤图像分割方法。
背景技术:
2.脑部肿瘤发病率近些年不断提高,对相应的医疗技术要求也越来越高。治疗过程中,脑部肿瘤图像的精确分割是疾病筛查、制定手术计划、构建肿瘤模型等实际应用的先决条件,具有重大的研究意义。研究人员针对脑部医学图像分割进行了大量的研究,取得了很多的研究成果。
3.临床应用中对脑部肿瘤图像的分割主要依靠经验丰富的专家根据自身的经验和病理学知识,借助计算机上的辅助画图软件在原始的mri图像中手动勾勒出肿瘤的病灶区位置,耗时耗力、效率低下、缺乏实时性、分割结果也有很大的差异性,不能满足批量处理脑部肿瘤图像分割工作的需求。聚类方法不需要高精度的标注信息,简单高效,易于实现,因此很多研究者进行了这一方面的研究探索。
4.目前基于k-means、fcm、ap、dp等聚类算法的改进型以及互相组合的聚类算法很多。而脑部肿瘤图像分割的精度要求高,各个聚类算法本身存在缺陷,分割效果具有较大的提升空间,ap算法和dp算法作为比较新的聚类算法虽然具有分割效果好、不需要人工指定过多参数等优势,但是受限于两个算法都需要针对数据量构建大规模的相似度矩阵,难以直接应用在数据点比较多的脑部肿瘤图像处理上。
技术实现要素:
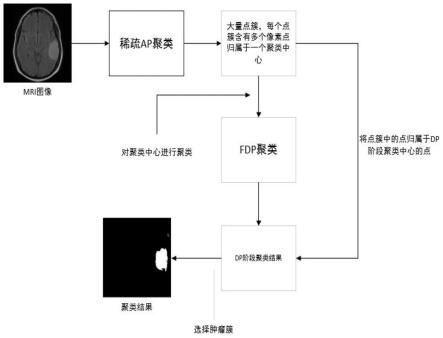
5.针对脑部肿瘤图像分割难度大,以及大规模标注信息获取困难等特点,本发明提出一种基于多层次聚类的脑部肿瘤图像分割方法。该分割方法引入了tamura纹理特征结合glcm 特征与灰度信息,改善了分割结果,用标准化欧氏距离优化聚类算法中常用的经典欧式距离,降低了极值特征对分割结果的过度影响,这是一个完整的脑部肿瘤图像分割方案,包括了预处理,聚类分割和阈值法选取肿瘤区域三部分,其中分割部分是优化改进的sapdp聚类,这是一种将稀疏近邻传播聚类(sap)和密度峰值聚类(dp)结合在一起的多层次聚类算法。
6.本发明的技术方案是:
7.第一方面,本发明提供一种基于多层次聚类的脑部肿瘤图像分割方法,其特征在于,该图像分割方法包含以下内容:
8.采集有肿瘤的脑部肿瘤核磁共振图像数据,作为脑部肿瘤原始图像,同时获取该图像的肿瘤区域掩膜,构成算法测试数据集;
9.利用多层次聚类分割图像,包括预处理模块、特征提取模块、层次聚类分割模块、肿瘤区域选取模块,
10.所述预处理模块选用均值滤波,输入脑部肿瘤原始图像,输出滤波后的脑部肿瘤
图像,对算法测试数据集中的原始图像的每个像素点用该像素点3
×
3邻域的均值代替该像素点,对各个像素点完成平均平滑滤波,获得滤波后的脑部肿瘤图像;
11.所述特征提取模块对滤波后的脑部肿瘤图像进行特征提取,以滤波后的脑部肿瘤图像本身像素值、3
×
3邻域像素均值、5
×
5邻域像素均值、灰度共生矩阵的能量、灰度共生矩阵的均值、tamura中的对比度与粗糙度这七个指标为滤波后的脑部肿瘤图像的每个像素点构建特征向量,所有像素的特征向量构成一个特征矩阵:
12.所述层次聚类分割模块的输入为特征提取模块获得的特征矩阵,输出最终的聚类中心以及每个点所属的类别,具体过程是:将粗分阶段的属于每个代表点的数据点,分配给划分阶段的该代表点属于的类别,即fdp阶段新生成的聚类中心获得包含的sap阶段聚类中心的所有归属点,完成分割;
13.所述的肿瘤区域选取模块采用阈值分割法实现,通过对聚类分割结果进行阈值选取,滤除非肿瘤簇的聚类簇,留下肿瘤区域掩膜二值图,完成脑部肿瘤图像的高精度分割。
14.第二方面,本发明提供一种计算机可读存储介质,所述存储介质中存储有计算机程序,所述计算机程序适用于被计算机加载时执行上述的图像分割方法。
15.第三方面,本发明提供一种基于多层次聚类的脑部肿瘤图像分割装置,其特征在于,包括存储器、处理器,存储器用于存储计算机程序,所述计算机程序包括预处理模块、特征提取模块、层次聚类分割模块、肿瘤区域选取模块,
16.所述预处理模块选用均值滤波,输入脑部肿瘤原始图像,输出滤波后的脑部肿瘤图像,对原始图像的每个像素点用该像素点3
×
3邻域的均值代替该像素点,对各个像素点完成平均平滑滤波,获得滤波后的脑部肿瘤图像;
17.所述特征提取模块对滤波后的脑部肿瘤图像进行特征提取,以滤波后的脑部肿瘤图像本身像素值、3
×
3邻域像素均值、5
×
5邻域像素均值、灰度共生矩阵的能量、灰度共生矩阵的均值、tamura中的对比度与粗糙度这七个指标为滤波后的脑部肿瘤图像的每个像素点构建特征向量,所有像素的特征向量构成一个特征矩阵:
18.所述层次聚类分割模块输入为特征提取模块获得的特征矩阵,输出最终的聚类中心以及每个点所属的类别,具体过程是:将粗分阶段的属于每个代表点的数据点,分配给划分阶段的该代表点属于的类别,即fdp阶段新生成的聚类中心获得包含的sap阶段聚类中心的所有归属点,完成分割;
19.所述的肿瘤区域选取模块采用阈值分割法实现,通过对聚类分割结果进行阈值选取,滤除非肿瘤簇的聚类簇,留下肿瘤区域掩膜二值图,完成脑部肿瘤图像的高精度分割。掩膜二值图标示了肿瘤的位置、大小、形状、边界等信息。
20.与现有技术相比,本发明具有以下优点:
21.本发明可以高效精确的实现脑部肿瘤图像的分割,结合了sap聚类方法和dp聚类优点,在运算速度上超过了ap算法。稀疏ap算法引入,通过大量实验来确定邻域像素的最佳数目 t,为了兼顾算法运算速度和分割精度,设置t值为32
×
32邻域内的所有像素点的数量,这样相似矩阵的规模为2562×
322=67108864,比原先2564=4294967296减少了64倍的复杂度。
22.本发明不需要人工指定聚类中心就可以实现图像分割,脑部肿瘤图像分割任务往往不知道聚类中心的确切数目,这样就避免了随机指定聚类中心数对聚类分割结果的干
扰。需要人为确定的参数少,包括粗化阶段的偏置参数p、阻尼因子λ以及划分阶段的截断距离dc,这些参数会影响聚类分割的结果,为了适应脑部肿瘤图片灰度值差别大,聚类簇的边界不明显,聚类中心多的特点,通过实验调整可以得到适应脑部肿瘤图像分割的参数组合,对于这类数据,设置p为相似矩阵s中所有元素的中值;λ选取0.8;dc选择使每个点的平均邻居数在2%,不需要针对每一张图像进行调整。
23.本发明引入了像素点邻域的tamura纹理特征,结合像素点的邻域灰度值以及邻域灰度共生矩阵特征为每个像素点构建了特征矩阵,并针对每个像素的多维的特征向量进行聚类。引入了多维度的特征进行聚类,相比于仅利用灰度信息聚类,图像分割效果有了提升实验过程中平均获得了0.03的dice值提升。
24.本发明引入了标准化欧氏距离来代替常应用在聚类算法中的欧氏距离,这样可以有效的削弱特征度量中存在的数值过大的分量对分割结果的不利影响。如果像素点x
ij
某个特征度量数值比较大,那么这一特征将使得其他特征对该像素点的聚类结果几乎失去影响,而加权思想的引入,可以削弱这种不利影响,使每组特征分量都具有对聚类结果的影响。
25.本发明所述算法进行图像分割并与真实分割结果进行对比,计算dice系数,结果显示平均dice值可以达到0.89高于k-means的0.80和sifcm的0.82。本发明提出了改进的多层次聚类算法在可以分割大型图像的基础上,对关键参数不敏感,而且分割效果更好。
附图说明
26.图1为数据示例图,图中左侧为原始脑部肿瘤核磁共振图像,右侧为这张图像的肿瘤区域掩膜二值图。
27.图2为sapdp算法流程图。
28.图3实验流程。
29.图4分割过程及结果对比图,显示了输入图像、金标准掩膜图像、空间模糊c均值聚类分割结果、k-means分割结果、经典ap分割结果以及本文算法的分割结果。
30.图5是对同一张图像,比较不同数量的聚类特征对聚类结果的影响图。
具体实施方式
31.以下结合本发明中实施例附图对本发明的具体实施方案进行更完整更清楚地描述。显然,所描述的实施例仅仅是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有任何创造性劳动前提下所获得的所有其他实施例,都属于本发明的范畴。
32.本发明为一种基于多层次聚类的脑部肿瘤图像分割方法,该方法用于脑部肿瘤图像分割任务中,具体包括以下步骤:
33.⑴
数据集准备
34.本发明原始数据为包含位于癌症基因组图谱(tcga)上的110名低级胶质瘤患者的脑部肿瘤核磁共振图像和手动分割结果(即该医疗影像对应的肿瘤区域掩膜)其中,原始数据中包括表示肿瘤区域的掩膜图像和未经分割处理的原始医疗影像两种图像。实验过程中选取了有肿瘤的脑部肿瘤核磁共振图像和手动分割结果的数据构建算法测试数据集36组
病例,每组选择五到十张图像不等,共计303张构成算法测试数据集。原始数据由网络收集而来,结合一部分现实医院收集来的图像共同用于本发明所提出的算法的效果验证。如图1所示,图中左侧为原始脑部肿瘤核磁共振图像,右侧为这张图像的肿瘤区域掩膜二值图。实际处理过程中只对左侧的原始图像进行处理,右侧的标注信息用于与分割结果进行比较并得出dice值。
35.⑵
多层次聚类分割图像,包括预处理模块、特征提取模块、层次聚类分割模块、肿瘤区域选取模块,
36.预处理模块选用均值滤波,输入脑部肿瘤原始图像,输出滤波后的脑部肿瘤图像,对算法测试数据集中的原始图像的每个像素点用该像素点3
×
3邻域的均值代替该像素点,对各个像素点完成平均平滑滤波,获得滤波后的脑部肿瘤图像。平滑效果与滤波窗口有关,窗口越大,平滑效果越强,但细节和边缘也越加模糊。为了适应脑部肿瘤图像的灰度特征,经过实验比较,本发明选用的3
×
3滤波窗口。
37.特征提取模块实现过程是:
38.对滤波后的脑部肿瘤图像进行特征提取,以滤波后的脑部肿瘤图像本身像素值、3
×
3邻域像素均值、5
×
5邻域像素均值、灰度共生矩阵的能量、灰度共生矩阵的均值、tamura中的对比度与粗糙度这七个指标为滤波后的脑部肿瘤图像的每个像素点构建特征向量,是一个多维的像素信息向量,所有像素的特征向量构成一个特征矩阵:
39.各个指标的计算过程是:首先计算滤波后的脑部肿瘤图像每个像素点的3
×
3邻域像素均值和5
×
5邻域像素均值;
40.同时,对滤波后的脑部肿瘤图像计算像素周边区域的glcm特征阶段,先采用边缘像素的倒序的方式为每个方向扩充4个像素值,实现图像边界扩充,通过实验,确定选用9
×
9的滑块计算每个像素周边的灰度共生矩阵信息能够最好的保证每个像素点周边灰度共生矩阵特征能够代表该点的特征,某像素点的纹理特征与周边像素点的纹理特征有所区分,再求取包括灰度共生矩阵的能量、灰度共生矩阵的均值这两个指标在内的特征。
41.通过实验,以每个像素周边的灰度共生矩阵信息能够最好的保证每个像素点周边灰度共生矩阵特征能够代表该点的特征,并与附近的像素点的灰度共生矩阵特征有所区分为要求,选用7
×
7的计算滑块对滤波后的脑部肿瘤图像求取每个像素点周围7
×
7范围内的tamura特征值,计算tamura图像纹理分析中的对比度和粗糙度这两个指标。
42.层次聚类分割模块输入为特征提取模块获得的特征矩阵,输出最终的聚类中心以及每个点所属的类别,实现过程:
43.粗化阶段:由稀疏ap算法实现,传统ap算法针对全体数据构建相似度矩阵,计算每两个点之间的距离作为相似度,每个点与自身的距离定义为偏置参数p,若数据量总数为n则相似度矩阵的大小为n
×
n。本实施例稀疏ap算法只考虑指定数量的最邻近的部分点,这就大大减小了计算量和内存占用。稀疏ap算法输入ap数据集x={x1,x2,......,xn},x是特征提取模块得到的特征矩阵,xi是第i个像素点对应的特征向量,简称数据点xi,n是总的像素点数,n=256
×
256,输出聚类中心作为下一步划分阶段的聚类数据,以及每个数据点归属于哪一个聚类中心的对应关系,操作如下:
44.1.1)利用公式(1)计算ap数据集x={x1,x2,......,xn}的稀疏相似度矩阵s,s的大小为n
×
t,t是选择的近邻数据点个数(t=322)。式(1)中偏置参数p设定为稀疏相似矩阵 s
中所有元素的中值,距离公式选用标准化欧氏距离,吸引度矩阵r和归属度矩阵a都初始化为n
×
t的零矩阵。
[0045][0046]
1.2)利用公式(2)计算r
t
(i,j)和a
t
(i,j):
[0047][0048][0049]rt
(i,j)是指第t次迭代后吸引度矩阵在(i,j)位置上的吸引度值,表示数据点xj对数据点xi的吸引程度,即数据点j适合代表数据点i的程度;
[0050]at
(i,j)是指第t次迭代后归属度矩阵在(i,j)位置上的归属度值,表示数据点xi选择数据点xj作为聚类中心的适合程度,当i=j时,计算a
t
(i,i)代表a
t
(i,j)。
[0051]
1.3)利用公式(3)在迭代过程中更新r
t
,a
t
的值,式(2)和式(3)中有(i,j)代表是矩阵某一位置的数值,无(i,j)代表的是矩阵本身。
[0052]rt
=r
t
(1-λ)+r
t-1
λ
[0053]at
=a
t
(1-λ)+a
t-1
λ
ꢀꢀꢀ
(3)
[0054]
式中λ表示阻尼因子,可以防止迭代过程中r与a中的值产生振荡,迭代更平稳一般取λ∈[0.5,1],本发明申请中取λ为0.8,当取值小的时候,迭代次数会减少,算法收敛加快,但是会增加聚类过程中的不稳定因素,影响聚类效果。
[0055]
1.4)若吸引度矩阵r和归属度矩阵a随着迭代已经不再更新数值,或者迭代次数t已经达到了最大迭代次数限制,则执行步骤1.5)的判断操作,若是两个条件都不满足,则继续循环执行步骤1.2)-1.3),更新r与a的值。
[0056]
1.5)若点xk的r
tk
+a
tk
>0,(r
tk
为点xk对应的第t次迭代的吸引度矩阵,a
tk
为点xk对应的第t次迭代的归属度矩阵)则点xk是一个聚类中心即粗化阶段选出来的代表点,所有的聚类中心构成代表点集。将每个非聚类中心点(即非代表点)分配到与其相似度最大的聚类中心(即代表点)所在的聚类簇当中。在稀疏ap算法中,偏置参数p被设置为稀疏相似矩阵s中所有元素的中值,也可以根据需求进行调整,如也可以选择设置为相似矩阵中所有值的平均值或最小值,p值越大,产生的聚类中心数越多。
[0057]
划分阶段:采用fdp算法实现,fdp算法输入dp数据集y={y1,y2,......,ym},y是粗分阶段得到的聚类中心的像素所具有的特征向量的集合,y
p
是dp数据集中的第p个数据,m 是粗分阶段聚类后的聚类中心点的数量。输出划分阶段的聚类中心和每个聚类中心的归属点,操作如下。
[0058]
2.1)使用标准化欧氏距离计算dp数据集y中每两个点之间的距离,若y中共有m个数据,则将这些距离数值存储在一个m
×
m的矩阵中,构成fdp阶段的距离矩阵,用于下一步计算局部密度和距离。一些特征值因为数值较大而对聚类结果有着过大的影响,使用标准化欧氏距离可以避免这种情况。
[0059]
2.2)局部密度ρ公式选用了高斯核函数,相比于截断核函数,高斯核函数虽然增加了计算量,却可以更好的减少错误聚类。
[0060][0061]
式(4)中d
pq
为点y
p
与点yq之间的距离,即距离矩阵(p,q)位置上的值,p、q取值范围为1~m的整数。dc为截断距离,然后通过自动搜索得到使每个点的平均邻居数在2%的dc值,取这个dc值为本方法使用的截断距离。将ρ降序排列,即排序后,δs的计算方法如式(5)所示:
[0062][0063]
2.3)确定聚类中心,δ和ρ都大的点被选择成为聚类中心。求取局部密度最大值与局部密度最小值的平均值作为密度阈值,求取距离最大值和距离最小值的平均值作为距离阈值,遍历所有的点,将局部密度与距离都大于各自设定的阈值的点选取成为聚类中心点,剩下的点距离最近且局部密度高的点为所选取出的聚类中心点所在的类中。
[0064]
将粗分阶段的属于每个代表点的数据点,分配给划分阶段的该代表点属于的类别,即fdp 阶段新生成的聚类中心获得包含的sap阶段聚类中心的所有归属点,完成分割。
[0065]
本发明中特征提取部分加入了在医学领域表现好的tamura特征纹理以及邻域灰度值等七个维度指标的信息构建特征矩阵,信息维度更丰富,采用了标准化欧氏距离,并引入加权因素,解决各个特征分量数值差异大的问题,成功将sapdp算法应用在脑部肿瘤图像分割中。用标准化欧氏距离来计算像素点距离,可以改善部分因特征度量值太大导致该度量对分割结果影响过大的问题。
[0066]
所述的肿瘤区域选取模块采用阈值分割法实现,通过对聚类分割结果进行阈值选取,滤除非肿瘤簇的聚类簇,留下肿瘤区域掩膜二值图,完成脑部肿瘤图像的高精度分割,实现了肿瘤区域的定位和定形。掩膜二值图标示了肿瘤的位置、大小、形状、边界等信息。
[0067]
本发明方法进行图像分割与真实分割结果进行对比,计算dice系数,结果显示本发明方法平均dice值可以达到0.89,明显高于k-means的0.80和sifcm的0.82。
[0068]
原始图像经过上述分割方法得到分割结果,将分割得到的肿瘤区域与数据集自带的掩膜信息做对比,计算dice参数,与传统k-means、空间模糊c均值、经典ap聚类进行对比。在对比过程中,传统k-means和空间模糊c均值算法都有需要指定的参数,如k-means的聚类数k,sifcm的聚类数k与模糊系数m,为了获得更好的分割效果,更合理的与本文方法对比,传统算法的相关参数进行了多次实验调整,选取最优参数组合的分割结果进行对比。经典ap算法由于内存溢出,无法直接分割数据集中的图像,因此对部分脑部mri图像进行了缩小来适应经典ap算法。评价指标是分割结果与手动分割的真实结果的dice值。dice系数是像素级别的比较关系,真实目标出现在某片区域a,模型预测结果的目标区域为b,那么dice系数公式如式(6)。
[0069][0070]
dice系数的取值范围为0-1,越接近1说明分割的结果越好。实验流程如图3所示。
[0071]
⑶
测试改进型sapdp图像分割效果
[0072]
如图4所示,对比图显示了输入图像、金标准掩膜图像、空间模糊c均值聚类分割结
果、 k-means分割结果、经典ap分割结果以及本文算法的分割结果。本次分割任务的图像具有 65536个像素,尺寸为256
×
256,直接使用经典ap算法进行分割出现了内存不足的现象,因此将原始图像缩小成64
×
64的新图像并对其进行分割,得到的分割结果会出现模糊现象,丢失了大量细节信息,但是通过计算dice系数,发现依然有0.72的准确率。ap算法准确率偏低并不是算法本身聚类效果差,而是在缩小图像的操作过程中,丢失大量信息导致。sifcm 与k-means作为划分法的聚类在肿瘤边界的界定上有着不错的效果,但是对于不属于肿瘤区域但灰度信息接近的颅骨部分难以辨别。处理结果都没有进行形态学操作,可以看到我们的算法在取得了良好的肿瘤边界分割的同时,对颅骨具有最好的剔除能力。本文的方法对这一图像实现了最优的分割,可以从中判断肿瘤的位置、大小和形状,肿瘤边界上也清晰的展现出来。本组数据处理结果的dice系数如表1所示。
[0073]
表1单一组别图像dice值对比
[0074][0075]
本文还研究了只利用灰度信息进行多层次聚类的图像分割效果。图5是对同一张图像,比较不同数量的聚类特征对聚类结果的影响,图5中第二张图像是只采用灰阶值进行分割的结果,算出的dice系数是0.93,而加上tamura特征度量和glcm特征度量之后再进行聚类分割,dice系数提升到了0.97。通过大量的实验对比,增加本方法所采用的纹理特征度量用来聚类可以有效地提升聚类的效果,在36组图像的实验过程中,平均提升了超过0.03的 dice值。
[0076]
表2是全部实验组中各个方法dice值的部分示例,计算每个实验组中多张图像的平均 dice值作为该组图像的评价指标。由表可见,部分图像(如第35组)由于图像自身特点,较难精确的分割出肿瘤区域,但是我们的算法在分割效果上依然优于其他算法。
[0077]
表2部分组别分割结果dice
[0078][0079][0080]
实验过程共对36组脑部mri图像进行了分割比对,36组303张图像整体的dice平均值由表3所示。在计算过程中,对k-means和sifcm算法进行了大量的参数调整,对于本文算法,粗化阶段的偏置参数p与细化阶段的截断距离dc确定之后不需要调整,也不需要额外指定其他参数。
[0081]
表3全部组别图像处理平均dice值
[0082][0083]
由表3可见,本发明算法与经典的算法相比切实提高了肿瘤分割的准确率,而且对ap 算法有着计算复杂度更低的优势,需要说明的是使用sifcm与k-means对缩小尺寸的图像进行处理结果的平均dice系数分别是0.62(k-means)和0.65(sifcm),本发明算法未对缩小图像进行处理。
[0084]
本发明基于多层次聚类的脑部肿瘤图像分割方法包括图像预处理模块、图像分割模块、肿瘤区域选取模块三个部分。在分割之前先对原始图像进行滤波处理,削弱图像中的噪声影响。接下来对滤波操作后的图像进行tamura纹理特征提取和glcm特征提取,得到图像的部分纹理特征度量并与滤波后的像素灰度信息进行组合,得到每个像素点的特征向量,所有像素点的特征向量构成特征矩阵。对特征矩阵进行聚类,粗化阶段采取稀疏ap聚类得到大量点簇;划分阶段采用密度峰值聚类算法对第一阶段的点簇中心进行聚类,得到新的聚类中心;最终在细化阶段将所有点划分给划分阶段的聚类中心,得到图像分割结果。将同一类别目标用相同的颜色表示,并通过阈值分割算法制作掩膜二值图,将聚类结果中的肿瘤部分提取出来。与传统的k-means聚类、改进型模糊c均值聚类和近邻传播聚类等算法相比可以实现更好的分割,tamura纹理特征的引入对方法的正面影响显著,实验证明相比于不加纹理特征的组别效果更好。
[0085]
本发明未述及之处适用于现有技术。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1