一种基于STRs先验信息的DNA调控元件设计方法
本发明属于合成生物学调控元件设计领域,具体涉及基于侧翼序列先验知识的深度生成网络方法及其应用。
背景技术:
1、dna调控元件是通过与蛋白质、rna等分子相互作用,调控基因表达的重要dna区域。它们包括启动子、增强子、终止子等,通常由顺式调控序列和转录因子结合位点(tfbss)组成。启动子是调控基因表达的核心元件,设计具有理想特性的合成启动子对生物合成工程和基因治疗至关重要。目前,启动子设计主要采用传统设计方法和智能计算设计方法。
2、传统方法包括:基于天然元件随机突变,即对一个功能启动子进行突变,筛选出具有所需功能的序列;基于完全随机生成,即随机生成大量dna序列,筛选出具有功能的启动子序列;基于转录因子结合位点设计,即在基本启动子骨架上,根据专家知识确定转录因子结合位点的摆放方式。这些方法依赖大量实验数据,设计效率低,难以针对特定需求进行精确优化。智能计算设计方法则基于深度学习模型,结合生成模型和预测模型,通过梯度传播、筛选、遗传算法等方式优化序列。这种方法提高了设计效率和可靠性,无需大量实验数据即可实现定制化设计。
3、尽管如此,现有的基因序列生成和分析方法仍存在问题,例如生成序列忽略重要生物特征、预测模型精度不高等,在设计序列过程没有先验知识约束等。因此,开发一种能够高效生成具有高度多样性且保留重要生物特征的基因序列的新方法具有重要意义。
技术实现思路
1、本发明旨在解决现有技术中的问题,提供一种基于strs先验信息的dna调控元件设计方法,以期能提升生成序列的生物功能性,并能显著提高预测模型的准确性和可靠性,使设计出的dna调控元件在实际应用中具有有效性,从而改善生成序列忽略重要生物特征、预测模型精度不高的问题。
2、本发明为达到上述发明目的,采用如下技术方案:
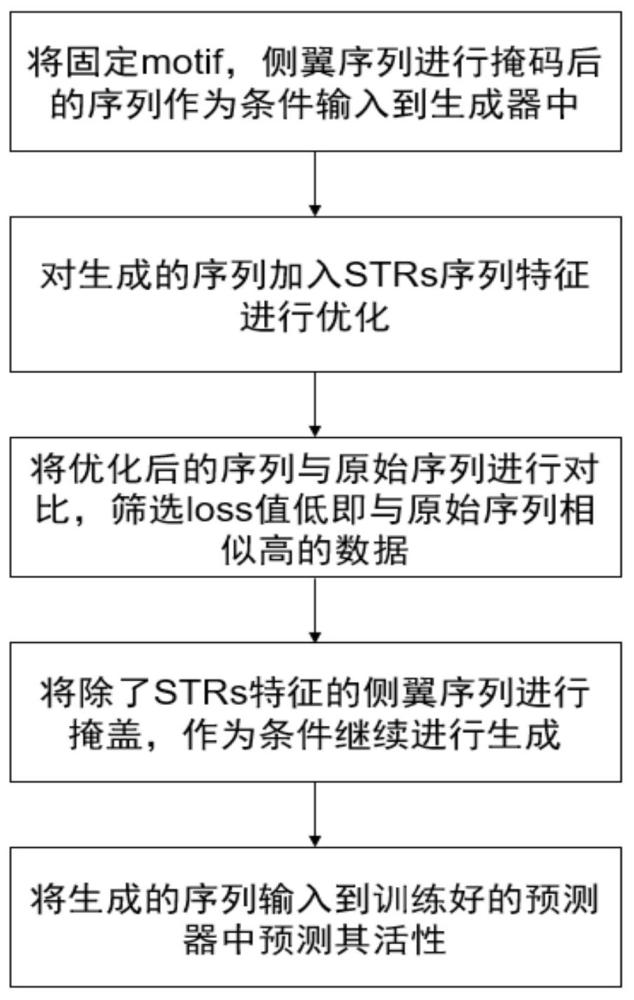
3、本发明一种基于strs先验信息的dna调控元件设计方法的特点在于,是按如下步骤进行:
4、步骤1: 获取原始原件序列以及每条序列对应的基序位置;
5、步骤1.1:获取原始元件序列集合nat={n1,n2,…,ni,…,nn}及其活性值exp={e1,e2,…,ei,…,en},其中,ni表示第i个原始元件序列,ei表示ni的活性值,n表示原始元件序列的数量,i∈[1,n];
6、将ni中y个基序位置所组成序列记为mpi={(ai,1,bi,1), (ai,2,bi,2), …,(ai,y,bi,y),…, (ai,y,bi,y)},其中,(ai,y,bi,y)表示第i个原始元件序列ni的第y个基序区间,ai,y表示第i个原始元件序列ni的第y个基序起始位置,bi,y表示第i个原始元件序列ni的第y个基序结束位置, y∈[1,y];
7、步骤1.2:将nat中基序位置以外的序列作为侧翼序列,则将第i个原始元件序列ni的第y个基序位置的左侧翼序列的区间记为(bi,y-1,ai,y),将ni的第y个基序位置的右侧翼序列的区间记为(bi,y,ai,y+1);
8、步骤2:设置随机噪声序列={z1,z2,…,zi,…,zn},其中,zi表示第i个随机噪声;
9、对nat的侧翼序列进行掩码,得到x={x1,x2,…,xi,…,xn},其中,xi表示ni 的侧翼序列的掩码;
10、将x和z输入生成器中进行处理,得到生成序列v={v1,v2,…,vi,…,vn},其中,vi表示第i个原始元件序列ni 对应的生成序列;
11、步骤3:设计含短串联重复序列特征的元件生成序列s:
12、步骤4:构建相似度计算模型l,用于计算nat和s的相似性得分t={ t1,t2, …,ti,…,tn },其中,ti表示ni和si的相似度得分;
13、对t进行降序排序,筛选前k个相似性得分所对应的生成序列组成与nat相似的生成序列集合tf={ tf1,tf2, …,tfk,…,tfk },其中,tfk为第k个与nat相似的生成序列,k∈[1,k];
14、步骤5:构建条件对抗生成网络,并利用生成器生成tf中除基序以及短串联重复序列外的其余侧翼序列的生成序列h;
15、步骤5.1:将tf中除基序以及短串联重复序列外的其余侧翼序列进行掩码处理后,得到掩码后的侧翼序列m={m1,m2, …,mk,…,mk};其中,mk表示tf中第k条侧翼掩码序列;
16、步骤5.2:将掩码后的侧翼序列m作为条件,并与随机噪声z一起输入条件对抗生成网络中的生成器进行处理,得到除基序以及短串联重复序列外的其余侧翼序列的生成序列h={ h1,h2, …,hk,…,hk },其中,hk表示tf中第k条侧翼掩码序列mk对应的生成序列;
17、步骤6:构建基于多尺度卷积注意力机制的预测器,并用于对nat进行处理,输出预测的活性值p,从而基于p和exp,训练预测器,得到元件序列活性预测模型;
18、步骤7:对条件对抗生成网络中判别器的反馈操作进行判别,以优化生成序列h的活性值;
19、步骤7.1:使用式(7)计算mk与hk的保守值uk,从而得到m与h的保守值序列u={u1,u2,…,uk,…,uk};
20、 (7)
21、式(7)中,表示mk和hk中所有非掩码字符总数,表示mk和hk中在所有非掩码字符位置上字符相同的总数;
22、步骤7.2:当uk为100%时,判断是否对判别器进行反馈操作,若是,则将hk与mk拼接后,形成第k个假数据对fk,将hk与mk拼接后,形成第k个真实数据对rk;否则,将hk与mk拼接后,形成第k个假数据对fk,将tfk与mk拼接后,形成第k个真实数据对rk;
23、步骤7.3:将fk以及rk输入到判别器中进行处理,并输出fk 被判别为真实样本的概率pfk以及rk被判别为真实样本的概率prk;
24、步骤7.4:利用式(2)构建判别器的损失函数ld,用于识别tfk与hk的差异:
25、 (2)
26、步骤7.5:根据损失函数ld,将判别器的识别结果反馈到生成器中进行优化训练,从而得到训练后的条件对抗生成网络。
27、本发明所述的一种基于strs先验信息的dna调控元件设计方法的特点也在于,所述步骤3是按如下步骤进行:
28、步骤3.1:获取碱基集合b,并由四种碱基组成,包括:鸟嘌呤、胞嘧啶、腺嘌呤、胸腺嘧啶;
29、步骤3.2:令con表示b的碱基子集中的碱基个数,并随机初始化con;
30、步骤3.3:从b中随机选择con个碱基组成一个碱基单元q,,,表示q中的元素个数;
31、步骤3.4:按照步骤3.3的过程重复rep次,从而得到rep个碱基单元并拼接为一个短串联重复序列;
32、步骤3.5:按照步骤3.4的过程,得到j个短串联重复序列组成的集合stri={stri,1, stri,2,…,stri,j , …,stri,j },其中,stri,j 表示ni 对应的生成序列vi的第j个短串联重复序列,j∈[1,j];
33、步骤3.6:检验stri,j的长度是否小于vi的第j个侧翼序列的长度,若是,则通过式(1)将stri,j插入到vi的侧翼序列中,得到第i个含有短串联重复序列特征的生成序列si,从而得到含有短串联重复序列特征的生成序列集合s={s1,s2, …,si,…,sn};若否,则返回步骤3.3和步骤3.4重新生成stri,j;
34、<mstyle displaystyle="true" mathcolor="#000000"><mstyle displaystyle="true" mathcolor="#000000"><mi>s</mi></mstyle><mstyle displaystyle="true" mathcolor="#000000"><msub><mrow /><mi>i</mi></msub></mstyle><mstyle displaystyle="true" mathcolor="#000000"><mi>=</mi></mstyle><mstyle displaystyle="true" mathcolor="#000000"><mi>[</mi></mstyle><mstyle displaystyle="true" mathcolor="#000000"><mi>(</mi></mstyle><mstyle displaystyle="true" mathcolor="#000000"><mi>b</mi></mstyle><mstyle displaystyle="true" mathcolor="#000000"><msub><mrow /><mrow><mi>i</mi><mi>,</mi><mi>y</mi><mi>−</mi><mi>1</mi></mrow></msub></mstyle><mstyle displaystyle="true" mathcolor="#000000"><mi>,</mi></mstyle><mstyle displaystyle="true" mathcolor="#000000"><mi>s</mi></mstyle><mstyle displaystyle="true" mathcolor="#000000"><mi>t</mi></mstyle><mstyle displaystyle="true" mathcolor="#000000"><mi>r</mi></mstyle><mstyle displaystyle="true" mathcolor="#000000"><msub><mrow /><mrow><mi>i</mi><mi>,</mi><mi>j</mi></mrow></msub></mstyle><mstyle displaystyle="true" mathcolor="#000000"><mi>,</mi></mstyle><mstyle displaystyle="true" mathcolor="#000000"><mi>a</mi></mstyle><mstyle displaystyle="true" mathcolor="#000000"><msub><mrow /><mrow><mi>i</mi><mi>,</mi><mi>y</mi></mrow></msub></mstyle><mstyle displaystyle="true" mathcolor="#000000"><mi>)</mi></mstyle><mstyle displaystyle="true" mathcolor="#000000"><mi>,</mi></mstyle><mstyle displaystyle="true" mathcolor="#000000"><mi>(</mi></mstyle><mstyle displaystyle="true" mathcolor="#000000"><mi>b</mi></mstyle><mstyle displaystyle="true" mathcolor="#000000"><msub><mrow /><mrow><mi>i</mi><mi>,</mi><mi>y</mi></mrow></msub></mstyle><mstyle displaystyle="true" mathcolor="#000000"><mi>,</mi></mstyle><mstyle displaystyle="true" mathcolor="#000000"><mi>s</mi></mstyle><mstyle displaystyle="true" mathcolor="#000000"><mi>t</mi></mstyle><mstyle displaystyle="true" mathcolor="#000000"><mi>r</mi></mstyle><mstyle displaystyle="true" mathcolor="#000000"><msub><mrow /><mrow><mi>i</mi><mi>,</mi><mi>j</mi><mi>+</mi><mi>1</mi></mrow></msub></mstyle><mstyle displaystyle="true" mathcolor="#000000"><mi>,</mi></mstyle><mstyle displaystyle="true" mathcolor="#000000"><mi>a</mi></mstyle><mstyle displaystyle="true" mathcolor="#000000"><msub><mrow /><mrow><mi>i</mi><mi>,</mi><mi>y</mi><mi>+</mi><mi>1</mi></mrow></msub></mstyle><mstyle displaystyle="true" mathcolor="#000000"><mi>)</mi></mstyle><mstyle displaystyle="true" mathcolor="#000000"><mi>]</mi></mstyle></mstyle> (1)。
35、所述步骤6中的预测器包括:编码层、卷积神经网络层、注意力机制模块、门控机制模块和预测模块;
36、步骤6.1:所述编码层对nat进行one-hot编码处理,得到编码后的编码矩阵,再输入不同膨胀率的卷积神经网络层中进行处理,得到nat的局部结构特征f;
37、步骤6.2:所述注意力机制模块来捕捉f中的全局结构特征:
38、步骤6.3:所述门控机制模块利用gru层捕捉的上下文依赖关系,输出最后一个时间步的隐藏状态;
39、步骤6.4:所述预测模块将隐藏状态依次经过全连接层、relu激活函数和dropout操作后,再通过另一全连接层的处理后,输出活性预测值p={p1,p2,…,pi,…,pn},其中,pi表示第i个原始元件序列ni的活性预测值;
40、步骤6.5:利用式(6)构建损失函数,用于对预测器进行参数优化,从而得到元件序列活性预测模型;
41、 (6)。
42、所述步骤6.2包括:
43、步骤6.2.1:f经过三层卷积神经网络层分别映射为不同的特征张量 a、b、c后,计算点积矩阵,从而利用式(3)得到注意力权重矩阵wab中第行第列权重:
44、 (3)
45、式(3)中,表示o中第行第列元素,代表wab的列数;
46、步骤6.2.2:利用式(4)得到注意力特征fatt:
47、 (4)
48、步骤6.2.3:fatt 通过另一个卷积层进行映射后,生成输出特征fout;
49、步骤6.2.4:利用式(5)得到nat的全局结构特征:
50、 (5)
51、所述步骤7.2中是按如下过程判别是否对判别器进行反馈操作:
52、步骤a:获取tf对应的原始元件序列ntf={ntf1,ntf2,…,ntfk,…,ntfk}及其活性值etf={etf1,etf2,…,etfk,…,etfk},其中,ntfk代表tfk对应的原始元件序列,etfk代表tfk对应的原始序列的活性值;
53、步骤b:将h输入到元件序列活性预测模型中进行处理,得到h的最优活性预测值p*={p*1,p*2,…,p*k,…,p*k},其中,p*k代表hk的最优活性预测值;
54、步骤c:若p*k> etfk, 则表示对判别器进行反馈操作,用于指导生成器生成高活性序列;否则,表示不对判别器进行反馈操作。
55、本发明一种电子设备,包括存储器以及处理器的特点在于,所述存储器用于存储支持处理器执行所述dna调控元件设计方法的程序,所述处理器被配置为用于执行所述存储器中存储的程序。
56、本发明一种计算机可读存储介质,计算机可读存储介质上存储有计算机程序的特点在于,所述计算机程序被处理器运行时执行所述dna调控元件设计方法的步骤。
57、与现有技术相比,本发明的有益效果在于:
58、1、本发明通过在使用cgan生成新的基因序列后,将生成的序列加入特定的strs序列特征,再计算此序列与自然序列的相似度后进行筛选。此过程不仅引入了strs特征,还确保了生成序列在生物结构特征上与自然序列保持高度一致,克服了现有技术中生成序列与自然序列相似度低的问题,从而提高了生成序列的生物学可信度。
59、2、本发明将筛选得到的元件序列固定侧翼strs区域,其余侧翼序列进行掩码操作,并将预处理后的序列作为条件输入到cgan中。这种方法能够在保留重要生物特征的同时,生成具有高度多样性的基因序列,克服了现有技术中生成序列单一的问题,从而提高了序列的生物学多样性。
60、3、本发明基于多尺度卷积注意力机制构建元件序列活性预测器。首先提取序列的不同尺度特征,结合注意力机制识别和强化对序列活性有重要影响的关键区域,构建预测器以预测生物序列的活性值。该过程能够捕捉序列中的短程和长程依赖关系,克服了现有技术中预测准确性不高的问题,从而大幅提高了预测的准确性和可靠性。
- 还没有人留言评论。精彩留言会获得点赞!