一种植物成对NLR抗性基因数据库的构建方法及其多物种成对NLR基因数据库
一种植物成对nlr抗性基因数据库的构建方法及其多物种成对nlr基因数据库
技术领域
1.本发明涉及一种基因序列数据处理方法,具体涉及植物成对nlr基因数据库的构建方法和多物种成对nlr基因数据库。
背景技术:
2.植物是地球生态的重要成员,也是人类生活中不可或缺的重要组成部分。人类的衣、食、住、行往往都离不开植物直接或间接的参与,但植物的生长发育过程并不是一帆风顺的,往往伴随着各种生物胁迫的发生,其中绝大部分的生物胁迫源自于微生物的侵染,它们往往会损害植物生长繁殖,严重时更有可能造成植物的大量减产。
3.目前应对病菌侵害的防治措施仍然是以施用农药为主,但农药的使用会对生态造成很大的伤害,一些农药含有持久性有机污染物,这些污染物难以降解,会在土壤中保留数十年,对土壤的质量和生物多样性产生不利影响(jacobsen and2014)。而农药的残留以及其通过食物链的进一步浓缩则会危害到我们人体的生命健康,尤其是儿童、孕妇和老人等高危人群(kim et al.,2017)。一些关于农药对人体健康的影响研究表明,农药可能与多种疾病有关,包括癌症、白血病和哮喘等等。因此,为了能够有效的、可持续发展的进行病菌防治,我们必须综合使用多种防治手段,而利用植物自身的抗性控制病害是已知最经济有效且安全的途径之一,植物抗病的分子机制研究、抗病基因的鉴定、克隆和转基因因此成为植物学领域的研究重点。
4.植物利用自身抗性抵御病原菌的方式有两种(jones and dangl,2006),首先在病原菌侵染时启动病原菌相关分子模式介导免疫(pamp
‑
triggered immunity,pti),这一过程涉及对微生物共有的保守特征(pathogen
‑
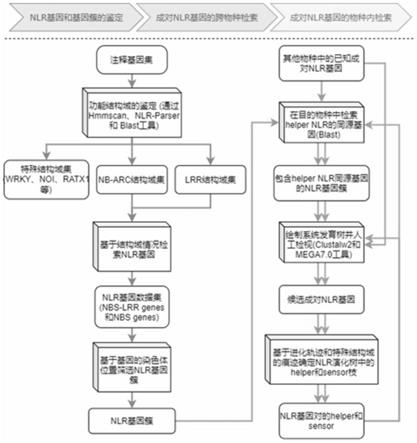
associated molecular pattern,pamps)的识别。一旦病原菌无法得到pti的有效抑制,效应因子介导的免疫反应(effector
‑
triggered immunity,eti)就会被激发。eti过程需要植物抗病基因(resistance gene)参与,r基因与病原菌avr效应因子结合后会诱发感染部位产生超敏性细胞死亡(hypersensitive cell death),造成组织坏死以避免病原菌进一步侵染。
5.nbs
‑
lrr基因(又称nlr基因)是植物抗病基因中数量最多分布最广的抗病基因群,一直是植物抗病研究中的关键基因。大部分nlr蛋白都是典型的模块化多结构域蛋白,其核心元件为中央的核苷酸结合位点(nucleotide binding site,nbs)以及c端的富亮氨酸重复区(leucine
‑
rich repeat,lrr),nbs结构域是nlr蛋白受体活性的开关,通过构象变化控制信号传导(moffett et al.,n.d.),而lrr结构域则通常在直接识别病原体效应因子的过程中参与特异性识别(jia et al.,2000)。除此之外nlr蛋白还包含一个n端的可变结构域,根据该可变结构域的不同又可以将nb
‑
lrr基因分为两大类:tnl(tir
‑
nbs
‑
lrr),在n端具有tir(tolland inter leukin
‑
1receptor)结构域;cnl(cc
‑
nbs
‑
lrr),在n端具有一个卷曲螺旋结构(coiled
‑
coil,cc)。然而这些nlr基因却具有如下这些演化特征:其在全基因组分布并不均匀,以集中分布为主,大多成簇分布,同一簇中以串联重复为主;同一nbs
‑
lrr基因在
不同物种间甚至是同一物种的不同品系之间都存在着很大的核苷酸差异;植物的不同品系间,nlr基因的复制和丢失十分频繁。以上这些特征为植物抗病基因的克隆、分析以及应用均制造了较大的阻碍。
6.目前针对植物抗病基因的研究主要集中于重要农作物上的抗性基因单体,对于非被子植物和多基因协作的研究相较之下要少得多。“基因对基因”是关于nlr基因抗性机制最早的假说,即一个基因负责一种病原菌的识别免疫,但越来越多的研究发现这一假说并不完备(lee et al.,2009;narusaka et al.,2009;peart et al.,2005)。许多研究结果表明植物对某些病原菌的免疫需要两个遗传连锁的基因协作才能完成,例如拟南芥中的rrs1和rps4(narusaka et al.,2009),水稻中的pi5
‑
1和pi5
‑
2(lee et al.,2009),烟草中的n和nrg1(peart et al.,2005)等等。它们被统称为成对nlr基因,在基因组上往往头对头排列。一些研究发现成对nlr基因在转基因育种后不仅具有更广谱持久的抗性,同时能降低抗性提高所造成的抗性成本增加(deng et al.,2017),是重要的转基因育种资源。joshua等人在扫描了十三种水稻亚种的nlr基因后发现有近50%的相邻nlr基因呈头对头排列,显著区别于基因簇内duplication串联重复造成的头对尾排列偏好(stein et al.,2018),以上的这些结果暗示nlr基因的成对抗性特征可能是植物抗性机制中的一种重要且普遍的模式,因此对成对nlr基因的深入研究是突破植物抗病研究瓶颈至关重要的一步。
7.目前已报道的成对nlr大多数具有以下几点特征:1.基因结构上同时具有nbs和lrr结构域,sensor常常包含一个特殊结构域如wrky、noi等(nishimura et al.,2015);2.染色体位置上helper与sensor头对头紧邻分布;3.演化上helper比sensor更加保守;4.系统发育特征上helper与sensor分属进化树的不同枝,具有镜像分布的拓扑关系。本发明基于以上这些成对nlr基因的演化特征,通过生物信息学方法,实现了植物中成对nlr基因数据库的构建。
技术实现要素:
8.针对国内外迄今尚缺乏有效鉴定成对nlr基因数据的现状,本发明的目的是提供一种植物成对nlr基因数据库的构建方法。
9.为了解决现有技术的问题,本发明提供了如下技术方案:本发明的一种植物成对nlr抗性基因数据库的构建方法,包括如下步骤:
10.(1)基础数据的收集:从jgi或ensembl收集目的物种的基因组序列、蛋白序列以及基因注释信息;
11.(2)nlr基因和基因簇的鉴定:首先通过hmmscan和nlr
‑
parser工具鉴定nlr基因,然后结合nlr基因的基因组注释位置,筛选其中的成簇nlr基因;
12.(3)特殊结构域的鉴定:通过hmmscan和blast工具,分析nlr基因的特殊结构域情况;
13.(4)成对nlr基因的鉴定:首先基于已报道的成对nlr基因,通过blast工具在目的物种的成簇nlr基因中搜索helper同源基因,然后将包含helper同源基因的nlr基因簇筛选出来通过clustalw2工具构建系统发育树并通过mega7.0工具进行人工检视,最终基于成对nlr基因的演化特征筛得目的物种的成对nlr基因;
14.(5)成对nlr基因的再鉴定:基于(4)搜索到的候选成对nlr基因,重复步骤(4)中的
分析检索流程,直至没有新得成对nlr基因被检索出来。
15.进一步地,在步骤(3)中,在通过hmmscan和nlr
‑
parser检测完nb
‑
arc结构域以及lrr结构域之后,将同时拥有nb
‑
arc结构域以及lrr结构域的基因归为主要nlr基因,而只有nb
‑
arc结构域的基因则归为候选nlr基因。
16.进一步地,在步骤(4)中,在检索nlr基因簇的过程中,将同一染色体上物理距离200kb以内或基因编号距离两个及以内的nlr基因视为同一基因簇内的基因。
17.更进一步地,在步骤(4)中,通过多种途径挖掘nlr基因的特殊结构域痕迹,包括对目的蛋白进行hmmscan搜索,对目的基因前后蛋白进行hmmscan搜索以及对目的基因前后5k以内核苷酸序列使用tblastn搜索,并以此作为sensor的判定依据之一。
18.进一步地,在步骤(4)中,将包含helper同源基因的nlr基因簇筛选出来后,使用nb
‑
arc结构域蛋白序列进行系统发育树的绘制,基于nlr基因的系统发育关系和特殊结构域痕迹确定系统发育树中的helper与sensor枝,候选helper和sensor在进化树上需要与已鉴定序列的helper和sensor聚集于同一枝中,与已鉴定helper聚集于一枝即为helper,其对应的基因即为sensor。
19.进一步地,在步骤(5)中,所述的成对nlr基因的特征值数据包括染色体上的基因位置、基因编号、链向、特殊结构域、基因类型、pair
‑
id、tir类型、nbs位置。
20.本发明所述的植物成对nlr抗性基因数据库的构建方法构建的多物种成对nlr基因数据库,其特征在于:以成对nlr基因对应的特征值作为数据项标识建立成对nlr基因数据库:https://figshare.com/articles/dataset/database_of_paired_nlr_genes/15096966。
21.进一步地,多物种为二穗短柄草、大麦、高粱、狗尾草或拟南芥。
22.有益效果:利用本发明提供的方法,可以对植物物种进行成对nlr基因的高效检索和分析,一方面为该物种的抗病功能研究提供了大量可靠的候选基因对,从而加速植物的抗病育种;另一方面基于多物种成对nlr基因数据库的构建,我们可以组合研究不同物种的nlr基因对,从而找到同时具备广谱性和高效性的nlr基因对组合并应用于不同的经济作物,减少病菌侵害对人民的生产生活的影响。
23.与现有技术相比,本发明具有如下优点:本发明提供的成对nlr基因数据库构建方法具有重要的科学价值和应用价值。近年来,成对nlr基因方面的研究进展并不理想:(1)尚没有能够高效鉴定全基因组成对nlr基因的生物信息学方法,因此后续的功能鉴定无法顺利开展;(2)成对nlr基因的抗病功能验证,存在周期长、病菌特异、功能冗余等问题。而本发明的出现,很好的弥补了这一方面研究方法的缺失,为不同物种中成对nlr基因的快速鉴定提供了非常好的途径。使用本发明提供的方法,可以对测序物种的成对nlr基因进行全基因组范围的检测和分析,既推动了作物的抗病研究,又为研究和探索作物的抗病演化提供了分子证据。
附图说明
24.图1为本发明流程示意图。
25.图2为二穗短柄草成对nlr系统发育特征示意图,特殊结构域已在图中标注。图a为二穗短柄草跨物种鉴定代表枝,红色正方形表示二穗短柄草成对nlr,蓝色圆形代表水稻成
对nlr种子序列,左列为helper,右列为sensor,clade标注与跨物种大树中一致。b:二穗短柄草成对nlr重建进化树,深色表示在跨物种中鉴定得到的基因对,浅色表示在自循环中鉴定得到的结果。
26.图3为大麦成对nlr系统发育特征示意图,特殊结构域已在图中标注。图a为大麦跨物种鉴定代表枝,黑色三角形表示大麦成对nlr,蓝色圆形代表水稻成对nlr种子序列,左列为helper,右列为sensor,clade标注与跨物种大树中一致;图b为大麦成对nlr重建进化树。
27.图4为高粱成对nlr系统发育特征示意图,特殊结构域已在图中标注。图a为高粱跨物种鉴定代表枝,深蓝色倒三角形表示高粱成对nlr,蓝色圆形代表水稻成对nlr种子序列,左列为helper,右列为sensor,clade标注与跨物种大树中一致;图b为高粱成对nlr重建进化树。
28.图5为狗尾草成对nlr系统发育特征示意图,特殊结构域已在图中标注。图a为狗尾草跨物种鉴定代表枝,棕色菱形表示狗尾草成对nlr,蓝色圆形代表水稻成对nlr种子序列,左列为helper,右列为sensor,clade标注与跨物种大树中一致;图b为狗尾草成对nlr重建进化树。
29.图6为拟南芥成对nlr基因的系统发育树。其中clade i为拟南芥通过已报道基因的拓扑关系检索到的成对nlr基因,clade ii为拟南芥通过自循环绘树检测到的成对nlr基因,helper枝和sensor枝标注在右侧。
具体实施方式
30.以下通过实例进一步对本发明进行描述,但不用来限制本发明的应用范围。
31.本发明的一种植物成对nlr抗性基因数据库的构建方法,包括如下步骤:
32.(1)基础数据的收集:从jgi或ensembl收集目的物种的基因组序列、蛋白序列以及基因注释信息;
33.(2)nlr基因和基因簇的鉴定:首先通过hmmscan和nlr
‑
parser工具鉴定nlr基因,然后结合nlr基因的基因组注释位置,筛选其中的成簇nlr基因;
34.(3)特殊结构域的鉴定:通过hmmscan和blast工具,分析nlr基因的特殊结构域情况;在通过hmmscan和nlr
‑
parser检测完nb
‑
arc结构域以及lrr结构域之后,将同时拥有nb
‑
arc结构域以及lrr结构域的基因归为主要nlr基因,而只有nb
‑
arc结构域的基因则归为候选nlr基因。
35.(4)成对nlr基因的鉴定:首先基于已报道的成对nlr基因,通过blast工具在目的物种的成簇nlr基因中搜索helper同源基因,然后将包含helper同源基因的nlr基因簇筛选出来通过clustalw2工具构建系统发育树并通过mega7.0工具进行人工检视,最终基于成对nlr基因的演化特征筛得目的物种的成对nlr基因;
36.通过多种途径挖掘nlr基因的特殊结构域痕迹,包括对目的蛋白进行hmmscan搜索,对目的基因前后蛋白进行hmmscan搜索以及对目的基因前后5k以内核苷酸序列使用tblastn搜索,并以此作为sensor的判定依据之一。
37.将包含helper同源基因的nlr基因簇筛选出来后,使用nb
‑
arc结构域蛋白序列进行系统发育树的绘制,基于nlr基因的系统发育关系和特殊结构域痕迹确定系统发育树中的helper与sensor枝,候选helper和sensor在进化树上需要与已鉴定序列的helper和
sensor聚集于同一枝中,与已鉴定helper聚集于一枝即为helper,其对应的基因即为sensor。
38.在检索nlr基因簇的过程中,将同一染色体上物理距离200kb以内或基因编号距离两个及以内的nlr基因视为同一基因簇内的基因。
39.(5)成对nlr基因的再鉴定:基于(4)搜索到的候选成对nlr基因,重复步骤(4)中的分析检索流程,直至没有新得成对nlr基因被检索出来。
40.所述的成对nlr基因的特征值数据包括染色体上的基因位置、基因编号、链向、特殊结构域、基因类型、pair
‑
id、tir类型、nbs位置。
41.本发明所述的植物成对nlr抗性基因数据库的构建方法构建的多物种成对nlr基因数据库,其特征在于:以成对nlr基因对应的特征值作为数据项标识建立成对nlr基因数据库:https://figshare.com/articles/dataset/database_of_paired_nlr_genes/15096966。
42.多物种为二穗短柄草、大麦、高粱、狗尾草或拟南芥。
43.实施例1
44.二穗短柄草(brachypodium distachyon)中成对nlr基因的鉴定
45.1、基础数据的收集:从jgi公共数据库中收集二穗短柄草的基因组序列、蛋白序列以及基因注释信息(https://phytozome.jgi.doe.gov);
46.2、nlr基因和基因簇的鉴定:(1)首先基于全基因组的蛋白序列信息,通过hmmscan工具获取所有蛋白序列的结构域信息,并从中筛选包含nb
‑
arc结构域(pf00931)的蛋白,参数设置为e
‑
value≤1e
‑
4;(2)然后再通过nlr
‑
parser工具,对lrr结构域进行检测,筛选具有motif 9,11或19的蛋白,同时拥有nb
‑
arc结构域以及lrr结构域的基因即主要nlr基因,而只有nb
‑
arc结构域的基因为候选nlr基因;(3)接着基于基因注释中nlr基因在染色体上的物理距离和基因编号,我们将同一染色体上物理距离200kb以内(holub,2001)或基因编号距离两个及以内的nlr基因视为同一基因簇内的基因,鉴定得到所有的nlr基因簇;(4)最终我们在短柄草中鉴定得到了336个nbs基因,272个nlr基因,并最终筛得了69个基因簇;
47.3、特殊结构域的鉴定:(1)基于pfam数据库在本地使用hmmscan对所有nlr蛋白序列进行结构域搜索,对wrky、noi、ratx1、srp54等已报道的特殊结构域进行标注;(2)对nlr基因的前后基因同样进行特殊结构域的搜索,如果前后基因存在特殊结构域,则该nlr基因的祖先基因也可能具有特殊结构域;(3)从pfam官网上搜索已报道的sensor基因的特殊结构域,并从alignments界面中下载该结构域蛋白序列的fasta文件,然后通过formatdb工具构建蛋白序列数据库,随后使用tblastn工具搜索成簇nlr基因前后5k以内核苷酸序列中的特殊结构域痕迹,e
‑
value<1e
‑
10;(4)最后整合以上三种方法获得的特殊结构域信息,以作为sensor的判定依据之一。
48.4、成对nlr基因的鉴定:(1)基于水稻已报道的成对nlr基因(wang et al.,2019),通过blast(2.3.0+)工具在成簇nlr基因中搜索helper同源基因,e
‑
value<10;(2)将包含helper同源基因的nlr基因簇筛选出来,使用nb
‑
arc结构域蛋白序列通过clustalw2工具绘制系统发育树(bootstrap值设置为1000,其余采用默认值);(3)通过mega7.0工具进行人工检视,去除duplication和不具有保守系统发育特征的nlr;(4)通过系统发育关系和特殊结构域(如wrky、noi等)确定helper与sensor枝,候选helper和sensor在进化树上需要与已鉴
定序列的helper和sensor聚集于同一枝中,与已鉴定helper聚集于一枝即为helper,其对应的基因即为sensor,反之亦然,最终筛得成对nlr基因16对(图2a);
49.5、成对nlr基因的再鉴定:基于步骤4搜索到的16对成对nlr基因,重复步骤4中的分析检索流程,直至没有新得成对nlr基因被检索出来,最终得到22对成对nlr基因(图2b)。
50.6、以成对nlr基因对应的特征值作为数据项标识通过mysql建立二穗短柄草成对nlr基因数据库。
51.实施例2
52.大麦(hordeum vulgare)中成对nlr基因的鉴定
53.1、基础数据的收集:从ensembl公共数据库中收集大麦的基因组序列、蛋白序列以及基因注释信息(http://plants.ensembl.org);
54.2、nlr基因和基因簇的鉴定:(1)首先基于全基因组的蛋白序列信息,通过hmmscan工具获取所有蛋白序列的结构域信息,并从中筛选包含nb
‑
arc结构域(pf00931)的蛋白,参数设置为e
‑
value≤1e
‑
4;(2)然后再通过nlr
‑
parser工具,对lrr结构域进行检测,筛选具有motif 9,11或19的蛋白,同时拥有nb
‑
arc结构域以及lrr结构域的基因即主要nlr基因,而只有nb
‑
arc结构域的基因为候选nlr基因;(3)接着基于基因注释中nlr基因在染色体上的物理距离和基因编号,我们将同一染色体上物理距离200kb以内(holub,2001)或基因编号距离两个及以内的nlr基因视为同一基因簇内的基因,鉴定得到所有的nlr基因簇;(4)最终我们在大麦中鉴定得到了405个nbs基因,318个nlr基因,并最终筛得了74个基因簇;
55.3、特殊结构域的鉴定:(1)基于pfam数据库在本地使用hmmscan对所有nlr蛋白序列进行结构域搜索,对wrky、noi、ratx1、srp54等已报道的特殊结构域进行标注;(2)对nlr基因的前后基因同样进行特殊结构域的搜索,如果前后基因存在特殊结构域,则该nlr基因的祖先基因也可能具有特殊结构域;(3)从pfam官网上搜索已报道的sensor基因的特殊结构域,并从alignments界面中下载该结构域蛋白序列的fasta文件,然后通过formatdb工具构建蛋白序列数据库,随后使用tblastn工具搜索成簇nlr基因前后5k以内核苷酸序列中的特殊结构域痕迹,e
‑
value<1e
‑
10;(4)最后整合以上三种方法获得的特殊结构域信息,以作为sensor的判定依据之一。
56.4、成对nlr基因的鉴定:(1)基于水稻已报道的成对nlr基因(wang et al.,2019),通过blast(2.3.0+)工具在成簇nlr基因中搜索helper同源基因,e
‑
value<10;(2)将包含helper同源基因的nlr基因簇筛选出来,使用nb
‑
arc结构域蛋白序列通过clustalw2工具绘制系统发育树(bootstrap值设置为1000,其余采用默认值);(3)通过mega7.0工具进行人工检视,去除duplication和不具有保守系统发育特征的nlr;(4)通过系统发育关系和特殊结构域(如wrky、noi等)确定helper与sensor枝,候选helper和sensor在进化树上需要与已鉴定序列的helper和sensor聚集于同一枝中,与已鉴定helper聚集于一枝即为helper,其对应的基因即为sensor,反之亦然,最终筛得成对nlr基因16对(图3a);
57.5、成对nlr基因的再鉴定:基于步骤4搜索到的成对nlr基因,重复步骤4中的分析检索流程,直至没有新得成对nlr基因被检索出来,最终得到16对成对nlr基因(图3b)。
58.6、以这些成对nlr基因对应的特征值作为数据项标识建立大麦成对nlr基因数据库。
59.实施例3
60.高粱(sorghum bicolor)中成对nlr基因的鉴定
61.1、基础数据的收集:从jgi公共数据库中收集高粱的基因组序列、蛋白序列以及基因注释信息(https://phytozome.jgi.doe.gov);
62.2、nlr基因和基因簇的鉴定:(1)首先基于全基因组的蛋白序列信息,通过hmmscan工具获取所有蛋白序列的结构域信息,并从中筛选包含nb
‑
arc结构域(pf00931)的蛋白,参数设置为e
‑
value≤1e
‑
4;(2)然后再通过nlr
‑
parser工具,对lrr结构域进行检测,筛选具有motif 9,11或19的蛋白,同时拥有nb
‑
arc结构域以及lrr结构域的基因即主要nlr基因,而只有nb
‑
arc结构域的基因为候选nlr基因;(3)接着基于基因注释中nlr基因在染色体上的物理距离和基因编号,我们将同一染色体上物理距离200kb以内(holub,2001)或基因编号距离两个及以内的nlr基因视为同一基因簇内的基因,鉴定得到所有的nlr基因簇;(4)最终我们在高粱中鉴定得到了326个nbs基因,275个nlr基因,并最终筛得了55个基因簇;
63.3、特殊结构域的鉴定:(1)基于pfam数据库在本地使用hmmscan对所有nlr蛋白序列进行结构域搜索,对wrky、noi、ratx1、srp54等已报道的特殊结构域进行标注;(2)对nlr基因的前后基因同样进行特殊结构域的搜索,如果前后基因存在特殊结构域,则该nlr基因的祖先基因也可能具有特殊结构域;(3)从pfam官网上搜索已报道的sensor基因的特殊结构域,并从alignments界面中下载该结构域蛋白序列的fasta文件,然后通过formatdb工具构建蛋白序列数据库,随后使用tblastn工具搜索成簇nlr基因前后5k以内核苷酸序列中的特殊结构域痕迹,e
‑
value<1e
‑
10;(4)最后整合以上三种方法获得的特殊结构域信息,以作为sensor的判定依据之一。
64.4、成对nlr基因的鉴定:(1)基于水稻已报道的成对nlr基因(wang et al.,2019),通过blast(2.3.0+)工具在成簇nlr基因中搜索helper同源基因,e
‑
value<10;(2)将包含helper同源基因的nlr基因簇筛选出来,使用nb
‑
arc结构域蛋白序列通过clustalw2工具绘制系统发育树(bootstrap值设置为1000,其余采用默认值);(3)通过mega7.0工具进行人工检视,去除duplication和不具有保守系统发育特征的nlr;(4)通过系统发育关系和特殊结构域(如wrky、noi等)确定helper与sensor枝,候选helper和sensor在进化树上需要与已鉴定序列的helper和sensor聚集于同一枝中,与已鉴定helper聚集于一枝即为helper,其对应的基因即为sensor,反之亦然,最终筛得成对nlr基因19对(图4a);
65.5、成对nlr基因的再鉴定:基于步骤4搜索到的成对nlr基因,重复步骤4中的分析检索流程,直至没有新得成对nlr基因被检索出来,最终得到19对成对nlr基因(图4b)。
66.6、以成对nlr基因对应的特征值作为数据项标识建立成对nlr基因数据库。
67.实施例4
68.狗尾草(setaria viridis)中成对nlr基因的鉴定
69.1、基础数据的收集:从jgi公共数据库中收集狗尾草的基因组序列、蛋白序列以及基因注释信息(https://phytozome.jgi.doe.gov);
70.2、nlr基因和基因簇的鉴定:(1)首先基于全基因组的蛋白序列信息,通过hmmscan工具获取所有蛋白序列的结构域信息,并从中筛选包含nb
‑
arc结构域(pf00931)的蛋白,参数设置为e
‑
value≤1e
‑
4;(2)然后再通过nlr
‑
parser工具,对lrr结构域进行检测,筛选具有motif 9,11或19的蛋白,同时拥有nb
‑
arc结构域以及lrr结构域的基因即主要nlr基因,而只有nb
‑
arc结构域的基因为候选nlr基因;(3)接着基于基因注释中nlr基因在染色体上
的物理距离和基因编号,我们将同一染色体上物理距离200kb以内(holub,2001)或基因编号距离两个及以内的nlr基因视为同一基因簇内的基因,鉴定得到所有的nlr基因簇;(4)最终我们在狗尾草中鉴定得到了388个nbs基因,291个nlr基因,并最终筛得了74个基因簇;
71.3、特殊结构域的鉴定:(1)基于pfam数据库在本地使用hmmscan对所有nlr蛋白序列进行结构域搜索,对wrky、noi、ratx1、srp54等已报道的特殊结构域进行标注;(2)对nlr基因的前后基因同样进行特殊结构域的搜索,如果前后基因存在特殊结构域,则该nlr基因的祖先基因也可能具有特殊结构域;(3)从pfam官网上搜索已报道的sensor基因的特殊结构域,并从alignments界面中下载该结构域蛋白序列的fasta文件,然后通过formatdb工具构建蛋白序列数据库,随后使用tblastn工具搜索成簇nlr基因前后5k以内核苷酸序列中的特殊结构域痕迹,e
‑
value<1e
‑
10;(4)最后整合以上三种方法获得的特殊结构域信息,以作为sensor的判定依据之一。
72.4、成对nlr基因的鉴定:(1)基于水稻已报道的成对nlr基因(wang et al.,2019),通过blast(2.3.0+)工具在成簇nlr基因中搜索helper同源基因,e
‑
value<10;(2)将包含helper同源基因的nlr基因簇筛选出来,使用nb
‑
arc结构域蛋白序列通过clustalw2工具绘制系统发育树(bootstrap值设置为1000,其余采用默认值);(3)通过mega7.0工具进行人工检视,去除duplication和不具有保守系统发育特征的nlr;(4)通过系统发育关系和特殊结构域(如wrky、noi等)确定helper与sensor枝,候选helper和sensor在进化树上需要与已鉴定序列的helper和sensor聚集于同一枝中,与已鉴定helper聚集于一枝即为helper,其对应的基因即为sensor,反之亦然,最终筛得成对nlr基因20对(图5a);
73.5、成对nlr基因的再鉴定:基于步骤4搜索到的成对nlr基因,重复步骤4中的分析检索流程,直至没有新得成对nlr基因被检索出来,最终得到20对成对nlr基因(图5b)。
74.6、以成对nlr基因对应的特征值作为数据项标识建立成对nlr基因数据库。
75.实施例5
76.拟南芥(arabidopsis thaliana)中成对nlr基因的鉴定
77.1、基础数据的收集:从jgi公共数据库中收集拟南芥的基因组序列、蛋白序列以及基因注释信息(https://phytozome.jgi.doe.gov);
78.2、nlr基因和基因簇的鉴定:(1)首先基于全基因组的蛋白序列信息,通过hmmscan工具获取所有蛋白序列的结构域信息,并从中筛选包含nb
‑
arc结构域(pf00931)的蛋白,参数设置为e
‑
value≤1e
‑
4;(2)然后再通过nlr
‑
parser工具,对lrr结构域进行检测,筛选具有motif 9,11或19的蛋白,同时拥有nb
‑
arc结构域以及lrr结构域的基因即主要nlr基因,而只有nb
‑
arc结构域的基因为候选nlr基因;(3)接着基于基因注释中nlr基因在染色体上的物理距离和基因编号,我们将同一染色体上物理距离200kb以内(holub,2001)或基因编号距离两个及以内的nlr基因视为同一基因簇内的基因,鉴定得到所有的nlr基因簇;(4)最终我们在短柄草中鉴定得到了165个nbs基因,其中142个nlr基因,并最终筛得了23个基因簇;
79.3、特殊结构域的鉴定:(1)基于pfam数据库在本地使用hmmscan对所有nlr蛋白序列进行结构域搜索,对wrky、noi、ratx1、srp54等已报道的特殊结构域进行标注;(2)对nlr基因的前后基因同样进行特殊结构域的搜索,如果前后基因存在特殊结构域,则该nlr基因的祖先基因也可能具有特殊结构域;(3)从pfam官网上搜索已报道的sensor基因的特殊结
构域,并从alignments界面中下载该结构域蛋白序列的fasta文件,然后通过formatdb工具构建蛋白序列数据库,随后使用tblastn工具搜索成簇nlr基因前后5k以内核苷酸序列中的特殊结构域痕迹,e
‑
value<1e
‑
10;(4)最后整合以上三种方法获得的特殊结构域信息,以作为sensor的判定依据之一。
80.4、成对nlr基因的鉴定:(1)基于拟南芥已报道的成对nlr基因rps1/rps4(narusaka et al.,2009)以及rpp2a/rpp2b(sinapidou et al.,2004),通过blast(2.3.0+)工具在成簇nlr基因中搜索helper同源基因,e
‑
value<10;(2)将包含helper同源基因的nlr基因簇筛选出来,使用nb
‑
arc结构域蛋白序列通过clustalw2工具绘制系统发育树(bootstrap值设置为1000,其余采用默认值);(3)通过mega7.0工具进行人工检视,去除duplication和不具有保守系统发育特征的nlr;(4)通过系统发育关系和特殊结构域(如wrky、noi等)确定helper与sensor枝,候选helper和sensor在进化树上需要与已鉴定序列的helper和sensor聚集于同一枝中,与已鉴定helper聚集于一枝即为helper,其对应的基因即为sensor,反之亦然,最终筛得成对nlr基因12对(图6clade i);
81.5、成对nlr基因的再鉴定:基于步骤4搜索到的成对nlr基因,重复步骤4中的分析检索流程,直至没有新得成对nlr基因被检索出来,最终得到18对成对nlr基因(图6clade ii)。
82.6、以成对nlr基因对应的特征值作为数据项标识建立成对nlr基因数据库。
83.以上显示和描述了本发明的基本原理、主要特征和本发明的优点。本行业的技术人员应该了解,本发明不受上述实施例的限制,上述实施例和说明书中描述的只是说明本发明的原理,在不脱离本发明精神和范围的前提下,本发明还会有各种变化和改进,本发明要求保护范围由所附的权利要求书、说明书及其等效物界定。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1