本发明涉及一种黄病毒人源单克隆抗体及应用,属于医药技术领域。
背景技术:
2015年中南美洲暴发寨卡疫情,目前已经造成69个国家数百万人感染,我国也出现数例输入性病例。随着人口的流动及气候变化,寨卡病毒在我国存在较高的流行风险。然而,由于寨卡病毒感染时,80%患者无明显症状,20%患者症状轻微,不会引起患者重视。寨卡病毒(zikavirus,zikv)属于黄病毒科,主要由蚊虫叮咬传播。同时,寨卡病毒可以通过母胎屏障,造成新生儿小头症。除此之外,寨卡病毒可以通过性传播。临床数据显示寨卡病毒既可以通过男性患者传染女性,也可以通女性患者传染男性。并且,研究表明男性精液中的寨卡病毒可存活62天,在他发病42天后仍能将寨卡病毒通过性接触传染给女性伴侣。越来越多的证据表明寨卡病毒感染的危害超出我们的认识:如引起新生儿小头症和神经症状(“格林-巴利综合征”等)。最近的研究揭示寨卡病毒感染雄性小鼠可造成睾丸损伤,并最终导致不育。因此必须要对寨卡病毒持续监控及检测,尤其对于育龄人群的检测更为重要。另外,为了研究寨卡病毒及其它黄病毒的致病机制、评估疫苗和治疗性药物效果等,动物模型,如小鼠模型的研究必不可少,这也迫切需要亲和力高,背景低的抗体用于实验研究。
寨卡病毒是有囊膜的正链rna病毒。囊膜上有e蛋白。e蛋白负责受体识别、结合及膜融合,含有重要的中和抗体表位。同时,e蛋白也是检测病毒的理想抗原。x-射线晶体学结构显示黄病毒e蛋白有三个结构域:di,dii和diii。dii头部(98-110位氨基酸)包含一个高度保守的融合区(fusionloop,fl),在病毒入侵的膜融合过程中起关键作用。不同黄病毒fl区域保守性非常高,在病毒感染过程中,免疫细胞会产生大量的针对fl的抗体。
已有研究发现有一些针对黄病毒的广谱抗体,如:2a10g6、4g2,这些抗体都是通过鼠杂交瘤融合产生的针对fl的抗体,其中2a10g6与e蛋白的亲和力kd=2.7nm,4g2与e蛋白的亲和力未知,未见文献报道,因此在基础研究和临床检测中,可能存在亲和力较低,特异性不好,背景高的问题,一定程度上限制了抗体的使用及灵敏度。
本发明的目的是鉴定高亲和力的人源抗体,用于基础研究和临床检测。
技术实现要素:
为了解决上述问题,本发明以大肠杆菌表达的寨卡e蛋白作为抗原,从一例康复期寨卡患者的pbmcs中筛选到可以结合寨卡e蛋白的记忆b细胞。然后对筛选的单一b细胞进行rt-pcr,获得抗体的可变区序列与片段,并进一步与恒定区连接至表达载体中。经哺乳动物细胞表达、纯化后,检测与zike-e蛋白的结合力。本发明获得了3株高亲力结合e蛋白的人源单克隆抗体。
本发明的单克隆抗体,或抗原结合片段的重链可变区的氨基酸序列和/或轻链可变区的氨基酸序列,包含至少一个(比如三个)克隆z5、克隆z6或克隆克隆z7的重链和/或轻链可变区的互补决定区(cdr)。
本发明的第一个目的是提供一种抗体,或抗原结合片段,其包含(a)或(b)或(c)或(d):
(a)抗体的重链可变区含有seqidno:1的26-35位,53-59位,和/或98-105位的氨基酸残基,且抗体的轻链可变区含有seqidno:2的26-34位,52-54位,和/或91-100位的氨基酸残基;
(b)抗体的重链可变区含有seqidno:11的26-35位,53-59位,和/或98-107位氨基酸残基,及抗体的轻链可变区含有seqidno:12的27-32位,50-52位,和/或89-97位氨基酸残基;
(c)抗体的重链可变区含有seqidno:21的26-35位,53-59位,和/或98-111位氨基酸残基,及抗体的轻链可变区含有seqidno:22的26-33位,51-53位,和/或90-100位氨基酸残基;
(d)在上述(a)、(b)、(c)中限定的位点处的氨基酸进行任何修饰或者替换而得到的抗体。
其中,所述的抗体或抗原结合片段特异性结合zike-e蛋白。
在本发明的一种实施方式中,所述抗体或抗原结合片段,其重链可变区的氨基酸序列与seqidno:1的序列具有至少90%的序列相似度且轻链可变区氨基酸序列与seqidno:2的序列具有至少90%的序列相似度,或者其重链可变区的氨基酸序列与seqidno:11的序列具有至少90%的序列相似度且轻链可变区氨基酸序列与seqidno:21的序列具有至少90%的序列相似度,或者其重链可变区的氨基酸序列与seqidno:31的序列具有至少90%的序列相似度且轻链可变区氨基酸序列与seqidno:32的序列具有至少90%的序列相似度。
在本发明的一种实施方式中,所述抗体或抗原结合片段,其重链可变区的氨基酸序列为seqidno:1且轻链可变区氨基酸序列为seqidno:2,或者其重链可变区的氨基酸序列为seqidno:11且轻链可变区氨基酸序列为seqidno:21,或者其重链可变区的氨基酸序列为seqidno:31且轻链可变区氨基酸序列为seqidno:32。
在本发明的一种实施方式中,所述抗体命名为z5;其重链可变区的核苷酸序列为seqidno:3、轻链可变区的核苷酸序列为seqidno:4。
在本发明的一种实施方式中,所述抗体命名为z6;其重链可变区的核苷酸序列为seqidno:13、轻链可变区的核苷酸序列为seqidno:14。
在本发明的一种实施方式中,所述抗体命名为z7;其重链可变区的核苷酸序列为seqidno:23、轻链可变区的核苷酸序列为seqidno:24。
在本发明的一种实施方式中,所述抗体的重链,包括重链可变区和重链恒定区,其中重链恒定区的氨基酸序列如seqidno:31所示(核苷酸序列如seqidno:32所示)。
在本发明的一种实施方式中,所述抗体z6的轻链为κ链,z5和z7的轻链为λ链;轻链包括轻链可变区和轻链恒定区;κ链的轻链恒定区的氨基酸序列如seqidno:33所示(核苷酸序列如seqidno:34所示),λ链的轻链恒定区的氨基酸序列如seqidno:35所示(核苷酸序列如seqidno:36所示)。
本发明的三个抗体,来源于同一病患者,并且均靶向寨卡病毒特有的囊膜蛋白-e蛋白,通过结合e蛋白的dii区域的fusionloop与e蛋白高亲和力结合。
本发明的第二个目的是提供编码所述抗体片段或者抗原结合片段的核苷酸序列、含有所述核苷酸序列的质粒、含有所述质粒的宿主细胞等。
在本发明的一种实施方式中,编码所述抗体的重链的序列,依次包括cmv启动子序列、ecori酶切位点序列、前导序列、编码重链可变区的序列、编码重链恒定区的序列、xhoi酶切位点序列。
在本发明的一种实施方式中,所述抗体的轻链,为κ链和/或λ链;轻链包括轻链可变区和轻链恒定区。
在本发明的一种实施方式中,编码所述抗体的轻链的序列,依次包括cmv启动子序列、第一酶切位点序列、前导序列、编码轻链可变区的序列、编码轻链恒定区的序列、酶切位点序列xhoi。
在本发明的一种实施方式中,所述抗体的轻链为κ链,第一酶切位点为saci。
在本发明的一种实施方式中,所述抗体的轻链为λ链,第一酶切位点为ecori。
在本发明的一种实施方式中,所述前导序列的氨基酸序列如seqidno:37所示(核苷酸序列如seqidno:38所示)。
在本发明的一种实施方式中,所述质粒为病毒质粒。
本发明的第三个目的是提供所述抗体或者抗原结合片段、编码所述抗体片段或者抗原结合片段的核苷酸序列、含有所述核苷酸序列的质粒、含有所述质粒的细胞在制备治疗和/或预防黄病毒病毒的药物方面的应用。
在本发明的一种实施方式中,所述黄病毒病毒为寨卡病毒、登革1-4型或者黄热病毒。
本发明的第四个目的是提供一种药物组合物,所述药物组合物含有有效预防剂量的本发明所述的抗体或者抗原结合片段。
在本发明的一种实施方式中,所述药物组合物还含有医药学上可接受的载体。
本发明的第五个目的是提供一种试剂盒,所述试剂盒中含有本发明所述的抗体或者抗原结合片段,或者编码所述抗体或者抗原结合片段的核苷酸序列,或者表达所述抗体或者抗原结合片段的重组载体/表达盒/转基因细胞系/重组菌。
本发明的有益效果:
本发明获得了3个可以与zikv-e蛋白结合的抗体:z5、z6和z7,并确定了3个抗体的结合位点。本发明的3株抗体与已经报道的zikv抗体序列完全不同,是3个新发现的抗体。这三株抗体与zikv-e的结合常数分别为39.9pm(z5),44.7pm(z6)和200pm(z7),说明三株人源抗体均有很强的寨卡病毒e蛋白结合能力。通过竞争实验,三株抗体结合的位点都与2a10g6存在竞争关系,说明三株抗体结合位点在fl附近,而黄病毒科的e蛋白在fl处高度保守。本发明的抗体有效检测常见的黄病毒科的病毒:寨卡病毒,登革1-4型以及黄热病毒的感染,有巨大的临床检测及基础研究的应用价值。
附图说明
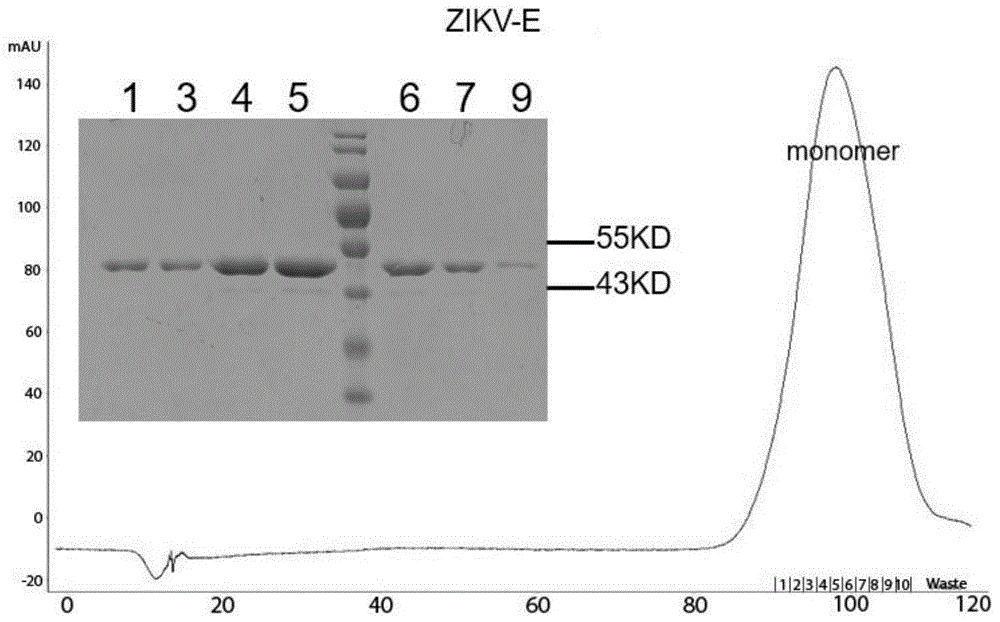
图1:zikv-e蛋白纯化分子筛与sds-page结果;
图2:z5纯化的分子筛层析结果;
图3:z6纯化的分子筛层析结果;
图4:z7纯化的分子筛层析结果;
图5:z5、z6、z7与zikv-e的动力学曲线;
图6:z5、z6、z7与2a10g6的竞争性试验;
图7:z6检测寨卡病毒感染;
图8:z6检测登革-1感染;
图9:z6检测登革-2感染;
图10:z6检测登革-3感染;
图11:z6检测登革-4感染;
图12:z6检测黄热病毒疫苗株yf-17d感染。
具体实施方式
实施例1:寨卡e蛋白的表达与纯化
zikve(氨基酸序列如seqidno:39所示、核苷酸序列如seqidno:40所示)胞外区dna片段通过ndei和xhoi酶切后,连接到pet21a载体上。其中zikve蛋白编码区的3’端连上6个组氨酸标签(hexa-his-tag)的编码序列及翻译终止密码子。再将连接产物转化到bl21大肠杆菌感受态细胞。单克隆接种到40mllb培养基中,培养6-8小时。接种到4l的lb培养基中,37℃培养至od600=0.4-0.6,加入iptg至终浓度1mm,37℃继续培养4-6小时。收获包涵体,通过在稀释法复性包涵体。复性液浓缩后换成20mmtris,150mmnacl,ph8.0缓冲液。将浓缩后的蛋白溶液进一步以分子排阻层析纯化,使用akta-purifier(ge)和superdex200hiload16/60柱子(ge),使用缓冲液a(20mmtris,150mmnacl,ph8.0,同时监测280nm的紫外吸收值,收取目的蛋白,并通过sds-page鉴定蛋白纯度。结果如图1。
实施例2:zikv-e蛋白特异性记忆b细胞的分离
在病人的知情同意下,采集15ml的血液,分离pbmcs。将分离的pbmcs以107/ml的密度与终浓度是100nm的zikv-e蛋白冰上孵育结合半小时,然后用pbs洗2次,再与下列抗体孵育:anti-humancd3/pe-cy5,anti-humancd16/pe-cy5,anti-humancd235a/pe-cy5,anti-humancd19/apc-cy7,anti-humancd27/pacificblue,anti-humancd38/apc,anti-humanigg/fitc,以及anti-his/pe。抗体冰上孵育半小时后,用pbs洗2次。
经facsariaiii分选收集pe-cy5-apc-apc-cy7+pacificblue+fitc+pe+的细胞,直接收集到96孔板内,1细胞/孔。
实施例3:单一b细胞pcr及序列分析
将实施例2获得的细胞通过superscriptiiireversetranscriptase(invitrogen)逆转录,逆转录引物如表1,55℃反应60min。将此逆转录产物作为模板,用hotstartapplus酶(qiagen)进行pcr,扩增抗体可变区序列(pcra)。引物如表2和3,反应条件如下:95℃,5min;95℃30s,55℃(重链/κ链)/50℃(λ链)30s,72℃90s,35个循环,72℃7min。将此作为模板再进行1轮pcr(pcrb),条件如下:95℃5min;95℃30s,58℃(重链)/60℃(κ链)/64℃(λ链)30s,72℃90s,35个循环,72℃7min。
1.2%的琼脂糖凝胶电泳,分离pcr产物。条带大小在400-500bp的切胶回收后送测序公司测序。测序结果用imgt在线软件进行分析。
分析正确的可变区序列与相应的重链/κ链/λ链的恒定区通过搭桥pcr连接,克隆至表达载体pcaggs中。其中重链与λ链以ecori和xhoi连接,κ链以saci与xhoi连接。表达质粒构建如下:
人源抗体设计策略如下:
重链:cmvpromoter-ecori-leadersequences-重链可变区-ch-xhoi;
轻链(κ):cmvpromoter-saci-leadersequences-轻链可变区-cl(κ)-xhoi;
轻链(λ):cmvpromoter-ecori-leadersequences-轻链可变区-cl(λ)-xhoi。
其中,leadersequences的氨基酸序列如seqidno:37所示(核苷酸序列如seqidno:38所示)。
得到了3个抗体z5、z6、z7,其轻、重链cdr序列如表1所示。
表1抗体z5、z6、z7的轻、重链cdr序列
注:h代表重链、l代表轻链。
其中z5的重链可变区的氨基酸序列为seqidno:1(核苷酸序列如seqidno:3),seqidno.5~seqidno.7的cdr-h序列分别位于seqidno:1序列的第26-35位、53-59位、98-105位;z5的轻链可变区氨基酸序列为seqidno:2(核苷酸序列如seqidno:4),seqidno.8~seqidno.10的cdr-l序列分别位于seqidno:2序列的第26-34位、52-54位、91-100位。
其中z6的重链可变区的氨基酸序列为seqidno:11(核苷酸序列如seqidno:13),seqidno.15~seqidno.17的cdr-h序列分别位于seqidno:11序列的第26-35位、53-59位、98-107位;z6的轻链可变区氨基酸序列为seqidno:12(核苷酸序列如seqidno:14),seqidno.18~seqidno.20的cdr-l序列分别位于seqidno:12序列的第27-32位、50-52位、89-97位。
其中z7的重链可变区的氨基酸序列为seqidno:21(核苷酸序列如seqidno:23),seqidno.25~seqidno.27的cdr-h序列分别位于seqidno:21序列的第26-35位、53-59位、98-111位;z7的轻链可变区氨基酸序列为seqidno:22(核苷酸序列如seqidno:24),seqidno.28~seqidno.30的cdr-l序列分别位于seqidno:22序列的第26-33位,51-53位,和/或90-100位。
所述抗体z5、z6、z7的重链恒定区ch的氨基酸序列为seqidno:31(核苷酸序列如seqidno:32)。
z6轻链均为κ型,轻链恒定区cl(κ)的氨基酸序列为seqidno:33(核苷酸序列如seqidno:34)
z5和z7的轻链为λ型,轻链恒定区cl(λ)的氨基酸序列为seqidno:35(核苷酸序列如seqidno:36)
实施例4:抗体的表达及纯化
用含10%fbs的dmem培养293t细胞。用含有特定抗体轻、重链编码基因的质粒共转染293t。转染4-6小时后给细胞换液成无血清的dmem继续培养3天,收集上清,并补加dmem,再培养4天,收集上清。
收集的上清经过5000rpm离心30min后,与含有20mm磷酸钠(ph8.0)等体积混合,经过0.22μm滤膜过滤后,与proteina预装柱结合(5ml,gehealthcare)。以10mm甘氨酸(ph3.0)洗脱结合的蛋白。收集此蛋白浓缩后进行分子筛层析。目的峰通过sds-page确定,结果如图2、3以及4。
实施例5:抗体的性能检测
(1)表面等离子共振技术检测与zikv-e的结合能力
表面等离子共振分析利用biacoret100(biacoreinc.)进行。具体步骤如下:
首先,将anti-humanigg的抗体以氨基偶联的方式固定在cm5芯片的通道(flowcell,fc)1与fc2。固定量控制在10,000响应值(responseunits,ru)左右。将通道调至fc2,然后以抗体捕获的方式结合纯化的抗体,此时,液流速度控制在10μl/min,进样1min,抗体捕获量在100ru左右。再以10mmhepes,150mmnacl,ph7.4溶液倍比稀释zikv-e蛋白,流速调至30μl/min,通道调至fc2-fc1的模式后,从低浓度开始逐一上样zika-e蛋白。曲线组成图如图5所示的动力学曲线。结果如表2所示。结合动力学常数的计算是利用biaevaluationsoftwaret100(biacore,inc.)软件进行。
表2人源抗体与zikve蛋白结合的动力学常数
结果显示,三株抗体均可以与zikv-e蛋白结合。
(2)z5、z6与z7结合位点的确定
确认3个抗体可以与zikv-e蛋白结合后,发明人通过竞争实验确认3个抗体的结合位点。2a10g6与zikv-e蛋白的结合位点已经确定(pdb:5jhl),靶向fl。发明人通过与2a10g6竞争实验检测3个抗体的结合位点与fl的关系。ni-nta探针首先与zikv-e(c端含有6*his)蛋白结合,用buffer洗去未结合的e蛋白,然后分别与z5、z6和z7结合,当抗体达到饱和后,再与z5/z6/z7+2a10g6结合,检测后者是否有额外的响应值的上升。结果如图6所示,2e6是mers-cov的中和抗体,作为阴性对照,2e6不能结合zikv-e蛋白,在第一相不能引起响应值的增高,再与2a10g6结合后,响应值提高。当3个抗体分别与含有e蛋白的探针结合并达到饱和后,再结合2a10g6并不能引起额外的响应值的提高,说明三个抗体都与2a10g6存在竞争关系,也提示了z5、z6和z7都是结合fl的抗体。
(3)z6抗体用于黄病毒科病毒感染检测
由于不同的黄病毒科的病毒在fl处高度保守,因此发明人检测了z6对常见的几种黄病毒科的病毒,如寨卡病毒,登革1-4型病毒以及黄热病毒的检测能力。用不同的稀释倍数的病毒感染vero细胞,检测e蛋白阳性细胞的比例,判断病毒感染情况。具体操作如下:
首先,提前24小时传代vero细胞至24孔板(1*105/孔);病毒感染前用无血清的dmem洗细胞1-2次,然后将病毒母液用dmem按如下各图所示稀释,再加入300μl/孔感染vero,37℃孵育,期间每隔15分钟轻轻摇晃平板;1小时后补加正常培养基dmem+10%fbs至1ml。细胞培养一定时间后检测阳性细胞比例:寨卡病毒(zika-smgc-1,genbankaccessionnumber:kx266255):48小时;登革1-4型(登革1:denv1/cn/gz27/2014strain,genbankaccessionnumber:kp723473;登革2:denv-2strain43,genbankaccessionnumber:af204178;登革3:denv-3yn02strain,genbankaccessionnumber:kf824903;登革4:denv-4chinaguangzhoub5strain,genbankaccessionnumber:af289029;):62小时;黄热病毒(yf-17d疫苗株):60小时。
培养结束时,先用pbs洗2次后,以0.25%胰酶消化2分钟,加入fbs中和胰酶作用,然后轻轻吹打细胞,并转移至圆底96孔板。以300g离心后取点上清,用固定液/通透液(bdcytofix/cytopermtm)处理细胞,冰上30分钟后,同样条件下离心,去除固定液/通透液,再用洗液(bdcytofix/cytopermtm)重悬细胞,离心2次。加入洗液稀释的终浓度2μg/ml的z6抗体,冰上孵育30分钟后,再离心去除染色液;以洗液洗2次,再加入anti-higg/fitc(santacruz)抗体(1:200),冰上避光孵育30分钟后,离心去除抗体,再以洗液洗两次后,转移至流式管内,以bdfacscanto分析fitc阳性的细胞比例。结果如图7-12所示,说明z6抗体可以有效检测寨卡病毒,登革1-4型以及黄热病毒。此外,z5、z7抗体也能有效检测上述病毒。
(4)z6抗体用于小鼠组织免疫荧光染色
确定z6可以用于细胞感染的检测后,发明人又尝试了检测小鼠感染模型中的寨卡病毒。用103pfu寨卡病毒感染ifnαr-/-小鼠(4-6周),8天后取不同部位的雄性生殖系统做冷冻切片,pbs洗3次后,用含1%bsa和0.3%triton的pbs37℃封闭30分钟。然后样品与20μg/ml的z6冰上过夜孵育。pbs洗后,再用1:100稀释的anti-higg/fitc(zsgb-bio)37℃染色1小时。pbs洗后,再用dapi(santacruz)染色。荧光显微镜下观察染色情况。结果显示,z6蛋白特异性地与寨卡病毒e蛋白结合,并且通过染色可以确认寨卡病毒对小鼠雄性生殖系统的感染主要是集中在睾丸和附睾,而对输精管和前列腺则没有感染能力。此外,z5、z7抗体也有类似效果。
综上,本发明通过筛选康复病人的zikv-e特异结合的记忆b细胞,获得3株人源高中和活性的zikv抗体:z5、z6与z7。这3株抗体与已经报道的寨卡抗体序列完全不同,是3株新发现的抗体。三株人源抗体有着临床治疗和预防寨卡的应用价值。
虽然本发明已以较佳实施例公开如上,但其并非用以限定本发明,任何熟悉此技术的人,在不脱离本发明的精神和范围内,都可做各种的改动与修饰,因此本发明的保护范围应该以权利要求书所界定的为准。
<110>中国科学院微生物研究所
<120>一种黄病毒人源单克隆抗体及应用
<160>40
<170>patentinversion3.3
<210>1
<211>116
<212>prt
<213>人工序列
<400>1
glnvalglnleuglngluserglyhisargleuvallysproserglu
151015
thrleuserleuthrcysthrvalserglyglyserileserserasp
202530
protyrleutrpglytrpileargglnproproglylysglyleuglu
354045
trpileglyseriletyrhisserglyseralatyrtyrasnproser
505560
leulysserargvalthrleuservalaspthrserargasnglnphe
65707580
serleulysvalasnservalthralathraspthralavaltyrtyr
859095
cysvalgluvalalaproproglulystrpglyargglyileleuval
100105110
thrvalserser
115
<210>2
<211>115
<212>prt
<213>人工序列
<400>2
glnseralaleuthrglnproproseralaserglyserproglygln
151015
servalthrilesercysthrglythrserseraspvalglyglytyr
202530
asntyrvalsertrptyrglnglnhisproglylysalaprolysleu
354045
metiletyralavalasnlysargproserglyvalproaspargphe
505560
serglyserlysserglyasnthralaserleuthrvalserargleu
65707580
glnaspaspaspglualaasptyrtyrcyssersertyrglyglyser
859095
asnasnileleupheglyglyglythrlysleuthrvalleuglygln
100105110
prolysala
115
<210>3
<211>348
<212>dna
<213>人工序列
<400>3
caggtgcagctgcaggagtcgggccaccggctggtgaagccttcggagaccctgtccctc60
acctgcactgtctctggtggctccatcagtagtgacccttatttatggggctggatccgc120
cagcccccagggaaggggctggagtggattggcagtatttatcatagtgggagtgcctac180
tacaatccgtccctcaagagtcgagtcaccttatccgtagacacgtccaggaaccagttc240
tccctgaaggtgaactctgtgaccgccacagacacggctgtctactattgcgtcgaagtt300
gcaccacctgaaaagtggggccggggaatcctagtcaccgtctcctca348
<210>4
<211>345
<212>dna
<213>人工序列
<400>4
cagtctgccctgactcagcctccctccgcgtccgggtctcctggacagtcagtcaccatc60
tcctgcactggaaccagcagtgacgttggtggctataactatgtctcctggtatcaacaa120
cacccaggcaaagcccccaaattaatgatttatgcggtcaataagcggccctcaggggtc180
cctgatcgcttctctggctccaagtctggcaacacggcctccctgaccgtgtctcggctc240
caggatgatgatgaggctgattattactgcagctcgtatggaggcagcaacaatatacta300
ttcggcggagggaccaagctgaccgtcctaggtcagcccaaggct345
<210>5
<211>10
<212>prt
<213>人工序列
<400>5
glyglyserileserseraspprotyrleu
1510
<210>6
<211>7
<212>prt
<213>人工序列
<400>6
iletyrhisserglyserala
15
<210>7
<211>8
<212>prt
<213>人工序列
<400>7
valgluvalalaproproglulys
15
<210>8
<211>9
<212>prt
<213>人工序列
<400>8
serseraspvalglyglytyrasntyr
15
<210>9
<211>3
<212>prt
<213>人工序列
<400>9
alavalasn
1
<210>10
<211>10
<212>prt
<213>人工序列
<400>10
sersertyrglyglyserasnasnileleu
1510
<210>11
<211>118
<212>prt
<213>人工序列
<400>11
glnvalglnleuglngluserglyproglyleuvallysproserglu
151015
thrleuserleuilecysthrvalserglyglyserileserthrarg
202530
asptyrtyrtrpglytrpileargglnthrproglylysglyleuglu
354045
trpileglyseriletyrtyrserglyglythrtyrtyrserproser
505560
leulysserargvalthrileservalaspthrserlysasnglnphe
65707580
serleulysleuargservalthralaalaaspthralavaltyrtyr
859095
cysalaargglntrpglyasntyrpheasphistrpglyglnglyser
100105110
leuvalthrvalserser
115
<210>12
<211>110
<212>prt
<213>人工序列
<400>12
aspilevalleuthrglnserproserpheleuseralaservalgly
151015
aspargvalthrilethrcysargalaserglnglyileaspthrtyr
202530
leualatrptyrglnglnlysproglylysalaprolysleuleuile
354045
tyrglyalaserthrleuglnserglyvalproserargphesergly
505560
serglyserglythrgluphethrleuthrileserserleuglnpro
65707580
gluaspphealathrtyrtyrcysglnglnleuasnasntyrhisphe
859095
thrpheglyproglythrlysvalaspilelysargthrval
100105110
<210>13
<211>354
<212>dna
<213>人工序列
<400>13
caggtgcagctgcaggagtcgggcccaggactggtgaagccttcggagaccctgtccctc60
atctgcactgtctctggtggctccatcagcactagggattactattggggctggatccgc120
cagaccccagggaaggggctggagtggattgggagtatatattatagtggaggtacgtac180
tacagtccgtccctcaagagtcgagtcaccatatccgtagacacgtccaagaatcagttc240
tccctgaagctgcgctctgtgaccgccgcagacacggctgtttattactgtgcgcgacaa300
tggggcaactactttgaccactggggccagggatccctggtcaccgtctcctca354
<210>14
<211>330
<212>dna
<213>人工序列
<400>14
gacattgtgctgacccagtctccatccttcctgtctgcatctgtaggagacagagtcacc60
atcacttgccgggccagtcaaggcattgacacttatttagcctggtatcagcaaaaacca120
gggaaagcccctaagctcctgatctatggtgcatccactttgcaaagtggggtcccatca180
aggttcagcggcagtggatctgggacagaattcactctcacaatcagcagcctgcagcct240
gaagattttgcaacttattactgtcaacagcttaataattaccatttcactttcggccct300
gggaccaaagtggatatcaaacgcaccgtg330
<210>15
<211>10
<212>prt
<213>人工序列
<400>15
glyglyserileserthrargasptyrtyr
1510
<210>16
<211>7
<212>prt
<213>人工序列
<400>16
iletyrtyrserglyglythr
15
<210>17
<211>10
<212>prt
<213>人工序列
<400>17
alaargglntrpglyasntyrpheasphis
1510
<210>18
<211>6
<212>prt
<213>人工序列
<400>18
glnglyileaspthrtyr
15
<210>19
<211>3
<212>prt
<213>人工序列
<400>19
glyalaser
1
<210>20
<211>9
<212>prt
<213>人工序列
<400>20
glnglnleuasnasntyrhisphethr
15
<210>21
<211>122
<212>prt
<213>人工序列
<400>21
glnvalglnleuglngluserglyproglyleuvallysproserglu
151015
thrleuserleuthrcysthrvalpheglyglyservalasnasnpro
202530
thrasntyrtrpglytrpileargglnserproglylysglyleuglu
354045
trpileglyservaltyrtyrthrglyileasntyrtyrasnproser
505560
leugluserargvalthrilealavalvalthrserlysasnglnphe
65707580
serleulysleuthrservalthralathraspthralavaltyrtyr
859095
cysalaarghisproargprogluthrglytyrasppheasptyrtrp
100105110
glyglnglythrleuvalthrvalserser
115120
<210>22
<211>115
<212>prt
<213>人工序列
<400>22
glnservalleuthrglnproproservalseralaalaproglygln
151015
lysvalthrilesercysserglyserserserasnileglyasnasn
202530
tyrvalsertrptyrglnglnleuproglythralaprolysleuleu
354045
leutyraspasnasnlysargproserglythrproaspargpheser
505560
glyserlysserglythrseralathrleuglyilealaglyleugln
65707580
thrglyaspglualavaltyrhiscysalathrtrpaspserserleu
859095
seralagluvalpheglyglyglythrglnvalthrvalleuglygln
100105110
prolysala
115
<210>23
<211>366
<212>dna
<213>人工序列
<400>23
caggtgcagctgcaggagtcgggcccaggactggtgaagccttcggagaccctgtccctc60
acttgcactgtctttggtggctccgtcaacaatcctactaattactggggctggatccga120
cagtccccaggcaagggactggagtggattgggagtgtctactatactgggatcaactac180
tacaacccgtccctggagagtcgagtcaccatagccgtggtcacgtccaagaaccagttc240
tccctgaagctgacctctgtgaccgccacagacacggctgtgtattactgtgcgagacat300
ccccggccggaaactggctatgattttgactactggggccagggaaccctggtcaccgtc360
tcctca366
<210>24
<211>345
<212>dna
<213>人工序列
<400>24
cagtctgtgytgackcagccgccctcagtgtctgcggccccaggacagaaggtcaccatc60
tcctgctctggaagcagctccaatattgggaataattatgtatcctggtaccagcagctc120
ccaggaacagcccccaaactcctcctttatgataataataagcgaccctcagggactcct180
gaccgattctctggctccaagtctggcacgtcagccaccctgggcatcgccggactccag240
actggggacgaggccgtttatcactgcgcaacatgggatagtagcctgagtgctgaggtt300
ttcggcggagggactcaggtgaccgtcctaggtcagcccaaggct345
<210>25
<211>10
<212>prt
<213>人工序列
<400>25
glyglyservalasnasnprothrasntyr
1510
<210>26
<211>7
<212>prt
<213>人工序列
<400>26
valtyrtyrthrglyileasn
15
<210>27
<211>14
<212>prt
<213>人工序列
<400>27
alaarghisproargprogluthrglytyrasppheasptyr
1510
<210>28
<211>8
<212>prt
<213>人工序列
<400>28
serserasnileglyasnasntyr
15
<210>29
<211>3
<212>prt
<213>人工序列
<400>29
aspasnasn
1
<210>30
<211>11
<212>prt
<213>人工序列
<400>30
alathrtrpaspserserleuseralagluval
1510
<210>31
<211>330
<212>prt
<213>人工序列
<400>31
alaserthrlysglyproservalpheproleualaproserserlys
151015
serthrserglyglythralaalaleuglycysleuvallysasptyr
202530
pheprogluprovalthrvalsertrpasnserglyalaleuthrser
354045
glyvalhisthrpheproalavalleuglnserserglyleutyrser
505560
leuserservalvalthrvalproserserserleuglythrglnthr
65707580
tyrilecysasnvalasnhislysproserasnthrlysvalasplys
859095
argvalgluprolyssercysasplysthrhisthrcysproprocys
100105110
proalaprogluleuleuglyglyproservalpheleuphepropro
115120125
lysprolysaspthrleumetileserargthrprogluvalthrcys
130135140
valvalvalaspvalserhisgluaspprogluvallyspheasntrp
145150155160
tyrvalaspglyvalgluvalhisasnalalysthrlysproargglu
165170175
gluglntyrasnserthrtyrargvalvalservalleuthrvalleu
180185190
hisglnasptrpleuasnglylysglutyrlyscyslysvalserasn
195200205
lysalaleuproalaproileglulysthrileserlysalalysgly
210215220
glnproarggluproglnvaltyrthrleuproproserargaspglu
225230235240
leuthrlysasnglnvalserleuthrcysleuvallysglyphetyr
245250255
proseraspilealavalglutrpgluserasnglyglnprogluasn
260265270
asntyrlysthrthrproprovalleuaspseraspglyserphephe
275280285
leutyrserlysleuthrvalasplysserargtrpglnglnglyasn
290295300
valphesercysservalmethisglualaleuhisasnhistyrthr
305310315320
glnlysserleuserleuserproglylys
325330
<210>32
<211>993
<212>dna
<213>人工序列
<400>32
gcgagcaccaaaggcccgagcgtgtttccgctggcgccgagcagcaaaagcaccagcggc60
ggcaccgcggcgctgggctgcctggtgaaagattattttccggaaccggtgaccgtgagc120
tggaacagcggcgcgctgaccagcggcgtgcatacctttccggcggtgctgcagagcagc180
ggcctgtatagcctgagcagcgtggtgaccgtgccgagcagcagcctgggcacccagacc240
tatatttgcaacgtgaaccataaaccgagcaacaccaaagtggataaacgcgtggagccc300
aaatcttgtgacaaaactcacacatgcccaccgtgcccagcacctgaactcctgggggga360
ccgtcagtcttcctcttccccccaaaacccaaggacaccctcatgatctcccggacccct420
gaggtcacatgcgtggtggtggacgtgagccacgaagaccctgaggtcaagttcaactgg480
tacgtggacggcgtggaggtgcataatgccaagacaaagccgcgggaggagcagtacaac540
agcacgtaccgtgtggtcagcgtcctcaccgtcctgcaccaggactggctgaatggcaag600
gagtacaagtgcaaggtctccaacaaagccctcccagcccccatcgagaaaaccatctcc660
aaagccaaagggcagccccgagaaccacaggtgtacaccctgcccccatcccgggatgag720
ctgaccaagaaccaggtcagcctgacctgcctggtcaaaggcttctatcccagcgacatc780
gccgtggagtgggagagcaatgggcagccggagaacaactacaagaccacgcctcccgtg840
ctggactccgacggctccttcttcctctacagcaagctcaccgtggacaagagcaggtgg900
cagcaggggaacgtcttctcatgctccgtgatgcatgaggctctgcacaaccactacacg960
cagaagagcctctccctgtctccgggtaaatga993
<210>33
<211>105
<212>prt
<213>人工序列
<400>33
alaalaproservalpheilepheproproseraspgluglnleulys
151015
serglythralaservalvalcysleuleuasnasnphetyrproarg
202530
glualalysvalglntrplysvalaspasnalaleuglnserglyasn
354045
serglngluservalthrgluglnaspserlysaspserthrtyrser
505560
leuserserthrleuthrleuserlysalaasptyrglulyshislys
65707580
valtyralacysgluvalthrhisglnglyleuserserprovalthr
859095
lysserpheasnargglyglucysser
100105
<210>34
<211>318
<212>dna
<213>人工序列
<400>34
gctgcccccagcgtgtttatcttccctcccagcgacgagcagctgaagagcggcaccgcc60
agcgtggtctgtctcctgaacaacttctatcccagggaggccaaggtccagtggaaagtg120
gacaacgccctgcaaagcggcaatagccaggagtccgtcacagagcaggacagcaaggac180
agcacctacagcctgtccagcaccctgaccctcagcaaggccgactacgagaagcacaag240
gtgtacgcttgcgaggtgacccatcagggcctgtccagccccgtgaccaagtccttcaac300
aggggcgaatgcagctaa318
<210>35
<211>101
<212>prt
<213>人工序列
<400>35
alaproservalthrleupheproproserserglugluleuglnala
151015
asnlysalathrleuvalcysleuileseraspphetyrproglyala
202530
valthrvalalatrplysalaaspserserprovallysalaglyval
354045
gluthrthrthrproserlysglnserasnasnlystyralaalaser
505560
sertyrleuserleuthrprogluglntrplysserhisargsertyr
65707580
sercysglnvalthrhisgluglyserthrvalglulysthrvalala
859095
prothrglucysser
100
<210>36
<211>306
<212>dna
<213>人工序列
<400>36
gcccctagcgtgacactgttccctccatctagcgaagaactgcaagctaacaaagccaca60
ctcgtgtgcctcattagcgacttctaccctggcgccgtgaccgtggcctggaaagcggac120
tcctctccagtgaaggccggcgtggagacaaccaccccatccaagcagtctaacaacaag180
tacgccgcctcttcctacctgagcctcacacctgagcagtggaagtctcacaggtcctac240
tcttgccaggtgacccacgagggctccacagtggaaaaaaccgtggccccaaccgagtgc300
agctga306
<210>37
<211>21
<212>prt
<213>人工序列
<400>37
metgluthraspthrleuleuleutrpvalleuleuleutrpvalpro
151015
glyserthrglyasp
20
<210>38
<211>63
<212>dna
<213>人工序列
<400>38
atggagacggatacgctgctcctgtgggttttgctgctgtgggttccaggttccactggt60
gac63
<210>39
<211>409
<212>prt
<213>人工序列
<400>39
ileargcysileglyvalserasnargaspphevalgluglymetser
151015
glyglythrtrpvalaspvalvalleugluhisglyglycysvalthr
202530
valmetalaglnasplysprothrvalaspilegluleuvalthrthr
354045
thrvalserasnmetalagluvalargsertyrcystyrglualaser
505560
ileseraspmetalaseraspserargcysprothrglnglygluala
65707580
tyrleuasplysglnseraspthrglntyrvalcyslysargthrleu
859095
valaspargglytrpglyasnglycysglyleupheglylysglyser
100105110
leuvalthrcysalalysphealacysserlyslysmetthrglylys
115120125
serileglnprogluasnleuglutyrargilemetleuservalhis
130135140
glyserglnhisserglymetilevalasnaspthrglyhisgluthr
145150155160
aspgluasnargalalysvalgluilethrproasnserproargala
165170175
glualathrleuglyglypheglyserleuglyleuaspcysglupro
180185190
argthrglyleuasppheseraspleutyrtyrleuthrmetasnasn
195200205
lyshistrpleuvalhislysglutrpphehisaspileproleupro
210215220
trphisalaglyalaaspthrglythrprohistrpasnasnlysglu
225230235240
alaleuvalgluphelysaspalahisalalysargglnthrvalval
245250255
valleuglyserglngluglyalavalhisthralaleualaglyala
260265270
leuglualaglumetaspglyalalysglyargleuserserglyhis
275280285
leulyscysargleulysmetasplysleuargleulysglyvalser
290295300
tyrserleucysthralaalaphethrphethrlysileproalaglu
305310315320
thrleuhisglythrvalthrvalgluvalglntyralaglythrasp
325330335
glyprocyslysvalproalaglnmetalavalaspmetglnthrleu
340345350
thrprovalglyargleuilethralaasnprovalilethrgluser
355360365
thrgluasnserlysmetmetleugluleuasppropropheglyasp
370375380
sertyrilevalileglyvalglyglulyslysilethrhishistrp
385390395400
hisargserglyserthrileglylys
405
<210>40
<211>1227
<212>dna
<213>人工序列
<400>40
attcgctgcattggcgtgagcaatcgcgactttgttgaaggcatgagcggtggcacctgg60
gtggatgtggtgctggaacacggcggttgcgtgaccgttatggcccaggataagccgaca120
gtggacatcgaactggttaccaccacagtgagcaacatggccgaggttcgtagctactgc180
tatgaggccagcatcagcgacatggccagtgacagtcgctgcccgacacagggcgaggcc240
tatctggacaaacagagcgacacccagtacgtttgtaaacgcaccctggtggaccgtggt300
tggggcaatggttgtggtctgtttggtaagggcagcctggtgacctgcgccaaattcgcc360
tgcagcaagaaaatgaccggcaagagcatccagccggaaaacctggagtaccgcattatg420
ctgagcgtgcatggcagccagcatagcggcatgattgtgaacgacaccggtcatgagacc480
gatgaaaaccgcgccaaagtggaaatcaccccgaatagtcctcgtgcagaagccaccctg540
ggcggttttggtagcctgggcctggattgcgagcctcgtaccggtctggattttagtgat600
ctgtattacctgaccatgaataacaaacattggctggttcacaaggaatggttccacgac660
atccctctgccgtggcatgcaggtgcagataccggcacaccgcattggaacaacaaagag720
gccctggtggagttcaaagatgcccacgcaaaacgccagaccgttgtggttctgggtagt780
caggaaggtgccgttcataccgcactggccggtgccctggaagccgaaatggacggcgcc840
aaaggccgcctgagcagtggtcatctgaaatgccgtctgaagatggacaagctgcgcctg900
aagggcgtgagttacagtctgtgtaccgccgccttcaccttcaccaagattcctgccgag960
accctgcatggtacagtgaccgtggaggtgcagtatgcaggtaccgatggtccgtgcaaa1020
gtgccggcccagatggccgtggacatgcagaccttaaccccggtgggccgcctgattacc1080
gccaatccggttattaccgaaagcaccgaaaacagcaaaatgatgctggaactggaccct1140
ccgtttggcgatagctacatcgtgattggcgtgggtgagaagaagatcacacaccactgg1200
caccgtagcggcagtaccatcggtaag1227
- 该技术已申请专利。仅供学习研究,如用于商业用途,请联系技术所有人。
- 技术研发人员:王奇慧;严景华;高福;
- 技术所有人:中国科学院微生物研究所;
- 我是此专利的发明人
- 该领域下的技术专家
- 如您需求助技术专家,请点此查看客服电话进行咨询。
- 1、薛老师:1.CRISPR-Cas系统 2.基因编辑 3.基因修复 4.天然产物合成 5.单分子技术开发与应用
- 2、张老师:1.探索新型氧化还原酶结构-功能关系,电催化反应机制 2.酶电催化导向的酶分子改造 3.纳米材料、生物功能多肽对酶-电极体系的影响4. 生物电化学传感和生物电合成体系的设计与应用。
- 3、豆老师:1.环境纳米材料及挥发性有机化合物(VOCs) 2.CO污染物的催化氧化 3.低温等离子体 4.吸脱附等控制技术
- 4、赵老师:1.高分子材料改性及加工技术 2.微孔及过滤材料 3.环境友好高分子材料
- 5、邬老师:1.高分子材料的共混与复合 2.涉及材料功能化及结构与性能的研究; 高分子热稳定剂的研发
- 如您是高校老师,可以点此联系我们加入专家库。
- 还没有人留言评论。精彩留言会获得点赞!