CasF2蛋白、CRISPR/Cas基因编辑系统及其在植物基因编辑中的应用
casf2蛋白、crispr/cas基因编辑系统及其在植物基因编辑中的应用
技术领域
1.本发明涉及基因编辑技术领域,尤其是涉及casf2蛋白、crispr/cas基因编辑系统及其在植物基因编辑中的应用。
背景技术:
2.基因编辑技术是近年来新兴的一种科学技术,可用于对基因进行靶向沉默和改造,创制非转基因新材料。crispr/cas(clustered regularly interspaced short palindromic repeatsand crispr-associated protein)系统构建简单、花费少和精确度高,而且能够实现同时多个基因进行编辑,效率高,是一项具有良好应用前景的基因靶向修饰技术。
3.目前,crispr/cas系统中主要是cas9、cas12和cas13。然而,这些cas基因的分子量都很大,且pam识别序列范围很窄。因此,开发新的cas基因应用于植物中的基因编辑迫在眉睫。研发新的基因编辑系统可以为作物基因编辑育种提供有效的工具,以实现人们预期的目标。
4.cas基因家族中,目前还没有详细地关于利用casf2基因构建crispr基因编辑载体在作物例如拟南芥、烟草、大豆和玉米中进行应用的相关报道。
5.鉴于此,特提出本发明。
技术实现要素:
6.本发明的目的在于提供casf2蛋白、crispr/cas基因编辑系统及其在植物基因编辑中的应用;本发明提供了一种新的高效、稳定的基因编辑系统,可使用更短的sgrna引导casf2进行靶点序列的编辑,产生dna序列的插入或缺失。
7.本发明提供的技术方案如下:
8.在一个方面,本发明提供了casf2蛋白,所述casf2蛋白的氨基酸序列如seq id no.2所示;或者所述casf2蛋白的氨基酸序列为与seq id no.2所示的序列相比具有一个或多个氨基酸的置换、缺失或添加,且具有相同或相似生物学功能。
9.seq id no.2所示的氨基酸序列如下所示:
10.mdykdhdgdykdhdidykddddkmapkkkrkvgihgvpaapkpavesefskvlkkhfpgerfrssymkrggkilaaqgeeavvaylqgkseeeppnfqppakchvvtksrdfaewpimkaseaiqryiyalstteraackpgksseshaawfaatgvsnhgyshvqglnlifdhtlgrydgvlkkvqlrnekararlesinasradeglpeikaeeeevatnetghllqppginpsfyvyqtispqayrprdeivlppeyagyvrdpnapiplgvvrnrcdiqkgcpgyipewqreagtaispktgkavtvpglspkknkrmrrywrsekekaqdallvtvrigtdwvvidvrgllrnarwrtiapkdislnalldlftgdpvidvrrnivtftytldacgtyarkwtlkgkqtkatldkltatqtvalvaidlgqtnpisagisrvtqengalqcepldrftlpddllkdisayriawdrneeelrarsvealpeaqqaevraldgvsketartqlcadfgldpkrlpwdkmssnttfiseallsnsvsrdqvfftpapkkgakkkapvevmrkdrtwaraykprlsveaqklknealw
alkrtspeylklsrrkeelcrrsinyviektrrrtqcqivipviedlnvrffhgsgkrlpgwdnfftakkenrwfiqglhkafsdlrthrsfyvfevrpertsitcpkcghcevgnrdgeafqclscgktcnadldvathnltqvaltgktmpkreeprdaqgtaparktkkaskskappaeredqtpaqepsqtskrpaatkkagqakkkk。
11.所述casf2蛋白为rna核酸酶,具有以下活性:与靶序列特定位点结合并切割核酸的活性或识别pam位点以及核酸内切酶活性。在一个实施方案中,所述casf2蛋白的氨基酸序列可以为与seq id no.2所示的氨基酸序列相比,其序列相似性在95%以上,优选在97%以上,更优选在98%以上,最优选在99%以上,只要与seq id no.2所示的氨基酸序列具有相同的功能即可。
12.本发明还提供了所述casf2蛋白的编码基因,所述编码基因的核苷酸序列如以下seq id no.1所示:
13.atggattacaaggatcacgacggtgattacaaggaccatgatatcgactacaaggatgacgatgacaagatggcgcctaagaagaagagaaaagtgggaattcacggtgttcctgctgccccaaagccggccgtggagtcagagttctctaaggttcttaagaagcatttcccgggcgagcgcttcagatcttcctacatgaagaggggcggaaagatcctggcggctcagggagaggaggctgtggttgcctacctccagggcaagagcgaggaggagcctccaaacttccagccgcctgctaagtgccacgtcgtgactaagtcaagagatttcgccgagtggccaattatgaaggcgtcagaggctatccagcgctacatttacgctctgtctactaccgagagagccgcgtgcaagccgggcaagtcttctgagagccacgctgcttggttcgctgctacaggcgtctcaaaccacggatactctcatgtgcagggactcaatcttatcttcgatcatactctcggaagatacgacggcgttctgaagaaggtccagctccgcaacgagaaggctagggcccgccttgagtcaatcaatgcttctagggctgacgagggcctgcctgagattaaggctgaggaggaggaggtcgccaccaacgagacaggtcatctccttcagccaccgggcatcaatccatccttctacgtgtaccagacaattagccctcaggcttacagaccaagggatgagatcgtgctccctccagagtacgcgggttacgttagagatcctaacgctcctattccacttggcgttgtccgcaatagatgcgatatccagaagggctgcccaggatacattccggagtggcagagggaggctggaactgccatttccccgaagactggaaaggccgtcaccgtgcctggcctcagcccaaagaagaacaagaggatgagaaggtactggcgctcagagaaggagaaggcgcaggacgctctgctcgttacagtcagaatcggaactgattgggtggttattgacgtgcgcggtcttctgagaaacgcgaggtggcgcactatcgctccaaaggatatttctcttaatgccctccttgacctgttcaccggagatccggtcatcgacgtgcgcagaaatattgttaccttcacatacactctcgatgcttgcggcacctacgccaggaagtggacacttaagggaaagcagactaaggctacccttgataagctgaccgccacacagactgttgcccttgtcgcgatcgacctgggccagaccaaccctatctccgccggaattagccgcgtcacacaggagaatggtgcgctccagtgcgagccacttgatagattcactctgccggatgacctgctcaaggatatctctgcgtacagaattgcttgggacaggaacgaggaggagcttagagcgaggtccgttgaggctctgccagaggctcagcaggccgaagttagggctctcgatggcgtcagcaaggagacagccaggactcagctctgcgcggatttcggactcgacccgaagaggcttccttgggataagatgtcttccaacacaactttcatctcagaggctcttctgagcaattcagtctctcgcgaccaggtgttcttcaccccggcccctaagaagggtgctaagaagaaggcgcctgtggaggttatgcgcaaggatagaacatgggcgcgcgcttacaagccaagactctctgtggaggcccagaagcttaagaatgaggcgctctgggctcttaagagaacctccccagagtacctgaagctcagcaggcgcaaggaggagctttgcagaaggtctatcaactacgttattgagaagacacgcagaaggactcagtgccagatcgtgattccggttattgaggatcttaatgtcagattcttccacggttccggcaagaggctgcctggttgggacaacttcttcactgccaagaaggagaatagatggttcatccagggcctgcacaaggcgttctcagatctcaggacccatcgctctttctacgtcttcgaagtgaggcctgagaggacctctattacatgcccaaagtgcggacactgcgaggttggaaacagggacggagaggctttccagtgcctctcctgcggaaagacctgcaac
gccgatcttgacgttgcgacccataatctgacacaggtcgccctcactggaaagaccatgccgaagagggaggagcctagggatgctcagggtactgctccagctaggaagaccaagaaggcttccaagagcaaggctccacctgctgagagggaggaccagacaccagctcaggagccgtcccagacaagcaagaggcctgccgcgactaagaaggctggacaggctaagaagaagaagtga。
14.casf2基因是cas12j类型的cas基因,具有长片段dna序列删除的潜力。casf2基因的pam识别序列为:5
’‑
tbn-3’(b=g/t/c),并且casf2对识别的靶序列位点特异性很高。casf2编辑系统的靶序列为18bp。casf2在基因组dna上可编辑的位点更多和具有更加广泛的潜在目标靶点。
15.本发明还可包括与seq id no.1所示的casf2蛋白的核苷酸序列反向互补的核苷酸序列。
16.在另一个方面,本发明还提供了一种表达载体,所述表达载体包含前述的核苷酸序列。表达载体可以在目标细胞中表达casf2蛋白,从而在目标细胞中进行相应的基因编辑。常用的载体可以是例如pbi121载体和pcambia1300载体等。
17.本发明也可包括含有所述表达载体的重组细胞,优选地,所述重组细胞为真核细胞,更优选地,所述重组细胞为植物细胞。
18.在另一个方面,本发明还提供了所述casf2蛋白、所述casf2蛋白的编码基因或权所述表达载体在基因编辑中的应用。
19.在另一个方面,本发明还提供了一种crispr/cas基因编辑系统,所述基因编辑系统包含前述的casf2蛋白或表达所述casf2蛋白的质粒。
20.在一个实施方案中,所述基因编辑系统还包含sgrna或表达sgrna的质粒,例如所述表达质粒含有所述sgrna的编码基因。
21.上述两个sgrna都能用于引导casf2蛋白在受体材料中靶基因序列位置进行基因编辑,当使用seq id no.3所示的sgrna3序列时,casf2蛋白具有sgrna序列加工的功能,会把sgrna3剪切成dgrna1序列,从而引导casf2在靶点位点处进行基因编辑。所述sgrna可以为一个或多个sgrna,用于靶向一个或多个靶基因。所述sgrna包括含有其对应识别区序列的核苷酸。
22.在另一个方面,本发明提供了所述crispr/cas基因编辑系统在植物基因编辑中的应用;优选地,所述植物为单子叶植物或双子叶植物;更优选地,所述植物选自拟南芥、烟草、大豆和玉米中的任一种。
23.在一个实施方案中,所述应用包括以下步骤:
24.(a)根据所述casf2蛋白的pam识别位点确定待编辑基因的sgrna;
25.(b)将所述casf2蛋白的基因序列和相应的sgrna克隆到表达载体中;
26.(c)将所述表达载体转化到农杆菌中,通过农杆菌介导的遗传转化方法,将所述表达载体导入目标植物,获得定向基因编辑植株;
27.优选地,在所述遗传转化中,使用mgcl2激活基因编辑活性。
28.casf2基因的编辑活性需要一定浓度的mg
2+
才能激活。casf2在植物体内存在时,没有额外添加一定浓度的mg
2+
,casf2不对细胞中的靶位点进行编辑。
29.在一个实施方案中,所述植物为拟南芥,所述sgrna对应的核苷酸序列(sgrna3序列)为seq id no.3所示。具体地,seq id no.3:
30.taatgtcggaacgctcaacgattgcccctcacgaggggac-n
18
(靶点序列)。
31.在一个实施方案中,所述植物为烟草,所述sgrna对应的核苷酸序列(sgrna1序列)为如seq id no.4所示。具体地,seq id no.4:
32.cgctcaacgattgcccctcacgaggggac-n
18
(靶点序列)。
33.在一个实施方案中,本发明提供了植物拟南芥bri1基因的sgrna靶点序列;所述sgrna靶点序列的核苷酸序列如seq id no.5所示。
34.具体地,seq id no.5:ctgcgaattcaatctccg。
35.在一个实施方案中,本发明提供了植物烟草pds基因的sgrna靶点序列;所述sgrna靶点序列的核苷酸序列如seq id no.6所示。
36.具体地,seq id no.6:gtagtagcgactccatgg。
37.基于本发明所述crispr/cas基因编辑系统在植物基因编辑中的应用,本发明同时也提供了一种利用本发明所述的基因编辑载体系统进行对植物进行基因编辑的方法。
38.在本发明中,crispr/cas系统中的casf2蛋白以及sgrna,二者可以在同一表达载体中也可以在不同的表达载体中。
39.在本发明中,简写“crispr”或者“crispr”均指规律成簇间隔短回文重复,即clustered regularly interspaced short palindromic repeats的首字母缩写。在核苷酸序列中,表示碱基时,如无特别说明,字母n所代表的碱基具有本领域通常的含义,即n代表随机或者任意碱基a、t、c或者g。
40.本文中使用的“含有”、“具有”或“包括”包括了“包含”、“主要由
……
构成”、“基本上由
……
构成”和“由
……
构成”。
41.有益效果:
42.本发明提供了一种新的casf2蛋白和一种高效、稳定的cas介导的基因编辑载体,可以在植物细胞中稳定、高效的表达,从而更好的调控靶基因的表达。casf2蛋白氨基酸数目少,casf2体积小为传递进入植物细胞中提供了优势(便于包装和递送),且casf2使用单一活性位点进行crrna处理和对dna序列的切割。casf2具有广泛的pam识别位点,可以构建多个基因编辑位点载体,实现对更多的基因进行敲除。本发明可用于拟南芥、烟草、大豆和玉米中dna序列的基因编辑。
附图说明
43.为了更清楚地说明本发明具体实施方式或现有技术中的技术方案,下面将对具体实施方式或现有技术描述中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图是本发明的一些实施方式,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他的附图。
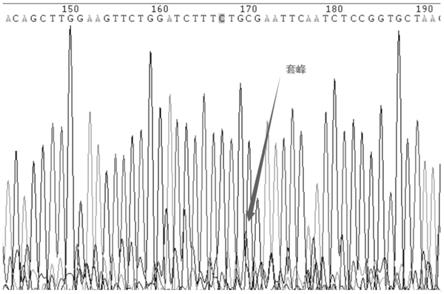
44.图1为本发明实施例提供的pcr测序结果;
45.图2为本发明实施例提供通过crispr/casf2基因编辑系统获得的拟南芥bri1基因编辑植株的表型图;
46.图3为本发明实施例提供的sgrna3 t质粒载体的示意图;
47.图4为本发明实施例提供的phcasf2载体的示意图;
48.图5为本发明实施例提供的sgrna1t质粒的示意图;
49.图6为本发明实施例提供的烟草组培结果图;
50.图7为本发明实施例提供的烟草pds靶点序列测序图。
具体实施方式
51.下面将结合实施例对本发明的技术方案进行清楚、完整地描述,显然,所描述的实施例是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
52.实施例1
53.1.拟南芥靶基因bri1打靶策略:
54.以拟南芥(arabidopsis thaliana)油菜素内酯受体bri1为目的基因,利用crispr/casf2定向编辑拟南芥bri1。
55.casf2/sgrna设计:
56.根据casf2识别的pam序列,选择拟南芥bri1的sgrna靶点的核苷酸序列(靶点序列seq id no.5)(5
’‑3’
):ctgcgaattcaatctccg。
57.2.拟南芥bri1基因的编辑
58.构建拟南芥bri1基因的sgrna,选择拟南芥atu6-26启动子驱动sgrna序列的表达;同时,在sgrna序列后面添加atu6-26终止子序列。以拟南芥u6启动子驱动sgrna的crispr/casf2基因编辑载体。
59.利用kod-plus neo,以sgrna3 t质粒(图3为质粒载体的示意图)作为模板扩增打靶sgrna序列。第1轮pcr,扩增体系(20μl):
[0060][0061]
引物对:u6-26p-f+f2bri1-grna-r扩增1管,引物对:f2bri1-u6-26t-f+u6-26t-r扩增1管。
[0062]
u6-26p-f(seq id no.7):
[0063]
ttcagaggtctcttagtcgacttgccttccgcacaatac;
[0064]
u6-26t-r(seq id no.8):
[0065]
agcgtgggtctcgcgcctattggtttatctcatcggaac;
[0066]
f2bri1-u6-26t-f(seq id no.9):
[0067][0068]
(其中,加粗下划线部分为靶点序列)
[0069]
f2bri1-grna-r(seq id no.10):
[0070]
cggagattgaattcgcaggtcccctcgtgaggggcaatc。
[0071]
pcr反应程序:
[0072][0073]
pcr扩增结束后,分别取2管的pcr产物各2μl,作为进行第2轮pcr的模板。
[0074]
扩增体系:
[0075][0076]
反应程序:
[0077][0078]
第2轮pcr结束后,取pcr产物1μl作为第3轮pcr的模板。反应体系和扩增体系同第1轮。扩增结束后,进行琼脂糖凝胶电泳。
[0079]
pcr产物进行胶回收后,与phcasf2载体(图4为载体示意图)连接。
[0080]
连接体系:
[0081][0082][0083]
连接反应:37℃5min,10℃5min;20℃5min;15个循环,最后37℃5min。
[0084]
连接产物转化e.coli dh5α感受态,感受态涂布在lb(kan
+
)平板上。从平板上挑选菌落送公司测序验证。
[0085]
构建的bri1基因打靶表达载体phcasf2-bri1转化农杆菌gv3101。
[0086]
挑取农杆菌对处于盛花期的拟南芥进行侵染。农杆菌法侵染拟南芥的步骤如下:农杆菌菌种吸取20μl加于3ml酵母液体培养基(yeast extract broth,yeb)(kan
+
、rif
+
)中;28℃,200rpm,摇24h;
[0087]
取摇好的菌,12,000rpm,1min离心收集菌株,去上清,将沉淀用1ml5%蔗糖1/2ms溶液重悬,再次收集菌株;最后仍用1ml 5%蔗糖1/2ms溶液重悬,同时加入0.2μl silwet77和终浓度为10mm mgcl2,充分混匀后,即可侵染拟南芥。
[0088]
筛选抗性苗并进行测序:
[0089]
待拟南芥种子成熟后,收获种子。种子播种在含有50mg/l潮霉素的1/2ms培养基平板上,挑取抗性苗。
[0090]
pcr扩增打靶位点附近的dna序列,pcr产物送公司测序。对bri1基因被编辑的拟南芥植株进行拍照。
[0091]
用于靶点序列附近dna扩增的pcr扩增引物:
[0092]
f2bri1crispr-identify-f(seq id no.11):
[0093]
ctggcttcaagtgctctgcttc;
[0094]
f2bri1crispr-identify-r(seq id no.12):
[0095]
gtaattcgccggaaaactcg。
[0096]
扩增体系同第1轮pcr。
[0097]
实验结果分析:
[0098]
(1)在侵染拟南芥的侵染液中不加入终浓度为10mm mgcl2没有发现bri1基因被编辑的植株,只有在加入了10mm mgcl2的侵染液侵染的拟南芥植株,才出现bri1基因被编辑的植株。
[0099]
(2)扩增bri1靶点附近的dna序列,通过测序发现,靶点出现了套峰(图1中示出);
并且拟南芥植株出现了不育表型,与报道的文献结果一致(图2中示出)。
[0100]
结果表明:casf2蛋白在拟南芥中进行基因编辑需要mgcl2的存在,并且能对拟南芥基因进行基因编辑,产生dna序列的插入、缺失。
[0101]
实施例2
[0102]
1.本氏烟pds基因打靶策略:
[0103]
以本氏烟草(nicotiana benthamiana)八氢番茄红素脱氢酶pds为目的基因,利用crispr/casf2定向编辑本氏烟pds。
[0104]
casf2/sgrna设计:
[0105]
根据casf2识别的pam序列,选择本氏烟pds的sgrna靶点的核苷酸序列(靶点序列seq id no.6)(5
’‑3’
):gtagtagcgactccatgg。
[0106]
2.拟南芥bri1基因的编辑
[0107]
本氏烟pds基因的编辑
[0108]
构建烟草pds基因的sgrna,构建方法同拟南芥一样,把拟南芥bri1靶点引物更换成烟草的pds靶点引物,扩增sgrna序列的模板换成sgrna1t质粒(图5为载体示意图)。烟草pds基因的靶点引物如下:
[0109]
f2ntpds-u6-26t-f(seq id no.13):
[0110][0111]
(其中,加粗下划线部分为靶点序列)
[0112]
f2ntpds-grna-r(seq id no.14):
[0113]
ccatggagtcgctactacgtcccctcgtgaggggcaatc。
[0114]
构建的烟草打靶pds基因质粒phcasf2-pds转化农杆菌gv3101,叶盘法转化烟草,具体步骤如下:
[0115]
1、叶片消毒:
[0116]
1)选生长1-2月本氏烟的叶片放入一个烧杯中,用无菌水浸泡20-30min;
[0117]
2)倒弃无菌水,用0.6%次氯酸钠浸泡叶片(每50ml溶液中加入1滴tween-20),让叶片在水中浸泡10min;
[0118]
3)用无菌水冲洗5次,
[0119]
2、叶片侵染:
[0120]
1)用灭菌剪子或者打孔器把叶片边缘切掉,把叶片剪成1cm2大小,避开叶片主脉,将叶片放到液体共培养培养基中;
[0121]
2)把所有的剪好的叶片转入到侵染液中,侵染8min;然后用无菌滤纸吸去叶片表面的侵染液;
[0122]
3)将叶片迅速放到固体共培养基上,叶片背面朝下,25℃暗培养48h;
[0123]
4)将叶片转移到筛选培养基上,25℃,16h光照/8h黑暗培养,每10天更换一次新鲜的筛选培养基,直至转化细胞分化出不定芽。
[0124]
5)选择白化苗进行照相,并提取白化苗基因组dna,用引物扩增靶点附近的dna序列,pcr产物送公司测序。
[0125]
用于靶点序列附近dna扩增的pcr扩增引物:
[0126]
ntpds-seq-f(seq id no.15):atgccccaaattggacttgtttc;
[0127]
ntpds1-23-r(seq id no.16):ctggagcgtggacaacataaat。
[0128]
构建烟草sgrna靶点的方法与构建拟南芥体系相同。
[0129]
实验结果分析:
[0130]
根据拟南芥的基因编辑结果,casf2蛋白发挥基因编辑需要mgcl2的存在,才会具有活性,在sgrna的引导,对靶点dna序列进行编辑。因此,在烟草pds基因组培中,筛选培养基中加入终浓度为100nm mgcl2;通过农杆菌侵染后,经过2轮的筛选培养基后,产生了白化苗(见图6)。在没有加入mgcl2的筛选培养基中,产生的是绿色分化苗。对白化苗的靶点序列进行测序发现,在靶点序列发生了碱基缺失。烟草pds靶点序列测序图7。
[0131]
同时,在拟南芥中使用sgrna3序列作为sgrna序列,在烟草中使用sgrna1序列作为sgrna序列。通过对构建的拟南芥sgrna3引导序列和烟草中sgrna1引导序列分析发现,casf2蛋白能对pre-crrna进行处理,可以使用序列更短的sgrna1引导casf2进行靶点序列的切割。这样可以同时构建多个sgrna进行多基因的打靶。
[0132]
最后应说明的是:以上各实施例仅用以说明本发明的技术方案,而非对其限制;尽管参照前述各实施例对本发明进行了详细的说明,本领域的普通技术人员应当理解:其依然可以对前述各实施例所记载的技术方案进行修改,或者对其中部分或者全部技术特征进行等同替换;而这些修改或者替换,并不使相应技术方案的本质脱离本发明各实施例技术方案的范围。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1