let-7f-5p及其靶基因在制备用于治疗骨质疏松症药物中的应用的制作方法
本发明涉及生物医学技术领域,特别是涉及一种let-7f-5p及其靶基因在制备用于治疗骨质疏松症药物中的应用。
背景技术:
骨质疏松症(osteoporosis,op)是单位体积骨量减少,骨组织微结构发生改变,骨脆性增加,以致易于发生骨折的代谢性疾病。随着人类进入老年化社会,op在世界各国发病逐年增高,且易罹患骨折等严重并发症,严重威胁人类健康,同时也给社会及家庭造成了沉重的经济负担。
研究op的发病机制,寻找op的防治靶点已成为临床的迫切需求。然而,目前提供有效的op治疗仍然是一项挑战。治疗op的推荐药物,如双膦酸盐和特立帕肽,存在较为严重的副作用,如恶心、上腹痛、消化不良、胃炎、背痛和关节痛等;且特立帕肽每天均需要注射,终身仅可接受一次为期24个月的治疗。
因此,研究op的病理机制和发现更多新的治疗靶点,仍然是一项重要的课题。
技术实现要素:
基于此,有必要针对上述问题,提供一种let-7f-5p及其靶基因作为靶点在制备用于治疗骨质疏松症药物中的应用,以mirnalet-7f-5p及其靶基因为op的治疗提供新的靶点,并以mirnalet-7f-5p为op的治疗提供新的治疗药物。
本发明公开了let-7f-5p及其靶基因作为靶点在制备用于治疗骨质疏松症药物中的应用。
微小rna(mirna)是一类小的非编码rna,通过与靶mrna的互补位点结合来调节转录物的降解或翻译。本发明人在前期应用高通量测序,筛查了giop患者与对照组中椎体组织的差异mirnas并对其靶基因进行了预测和验证。并且在miranda、mirbase及targetscan数据库中预测差异mirnas及其靶基因,发现mirnalet-7f-5p存在差异表达,而且let-7f-5p靶向tgfbr1基因;并且进一步通过实验证实了let-7f-5pmimics可以逆转gc诱导下的成骨分化的降低;体内实验亦证实let-7f-5p尾静脉注射可以减少giop小鼠骨量丢失。
即可将let-7f-5p及其靶基因作为治疗骨质疏松症的靶点应用。
在其中一个实施例中,所述靶基因为tgfbr1基因。本发明通过荧光素酶报告基因技术验证了let-7f-5p对tgfbr1基因的靶向关系,可以此tgfbr1基因为靶基因,继续探寻对该tgfbr1基因有促进作用对象,均有可能作为制备治疗骨质疏松症的药物。
本发明还公开了let-7f-5p在制备用于治疗骨质疏松症药物中的应用。
本发明通过实验证实let-7f-5p在op的治疗中发挥了重要作用,可以用于制备治疗op的药物。
在其中一个实施例中,所述let-7f-5p包括seqidno.1所示序列。
在其中一个实施例中,所述let-7f-5p还包括与seqidno.1所示序列进行一个或多个碱基的取代、缺失、添加修饰的等同序列,所述等同序列可与let-7f-5p的靶mrna序列互补结合。
可以理解的,所述let-7f-5p序列可以与seqidno.1一致,也可以采用与seqidno.1等同的序列,仅需能够与该mirnalet-7f-5p的靶mrna互补结合即可。
在其中一个实施例中,所述骨质疏松症的表现形式为成骨分化降低。
在其中一个实施例中,所述骨质疏松症的表现形式为骨量丢失。
本发明还公开了一种用于治疗骨质疏松症药物,包括let-7f-5p。
在其中一个实施例中,所述药物的给药途径为静脉注射液。
与现有技术相比,本发明具有以下有益效果:
本发明人在前期应用高通量测序,筛查了giop患者与对照组中椎体组织的差异mirnas并对其靶基因进行了预测和验证。并且在miranda、mirbase及targetscan数据库中预测差异mirnas靶基因发现mirnalet-7f-5p存在差异表达,并且进一步通过实验证实了let-7f-5pmimics可以逆转gc诱导下的成骨分化的降低;体内实验亦证实let-7f-5p尾静脉注射可以减少giop小鼠骨量丢失。同时通过荧光素酶报告基因技术验证了let-7f-5p对tgfbr1基因的靶向关系。即可将let-7f-5p及其靶基因作为治疗骨质疏松症的治疗靶点,以mirnalet-7f-5p及其靶基因为op的治疗提供新的靶点,并以mirnalet-7f-5p为op的治疗提供新的治疗药物。
附图说明
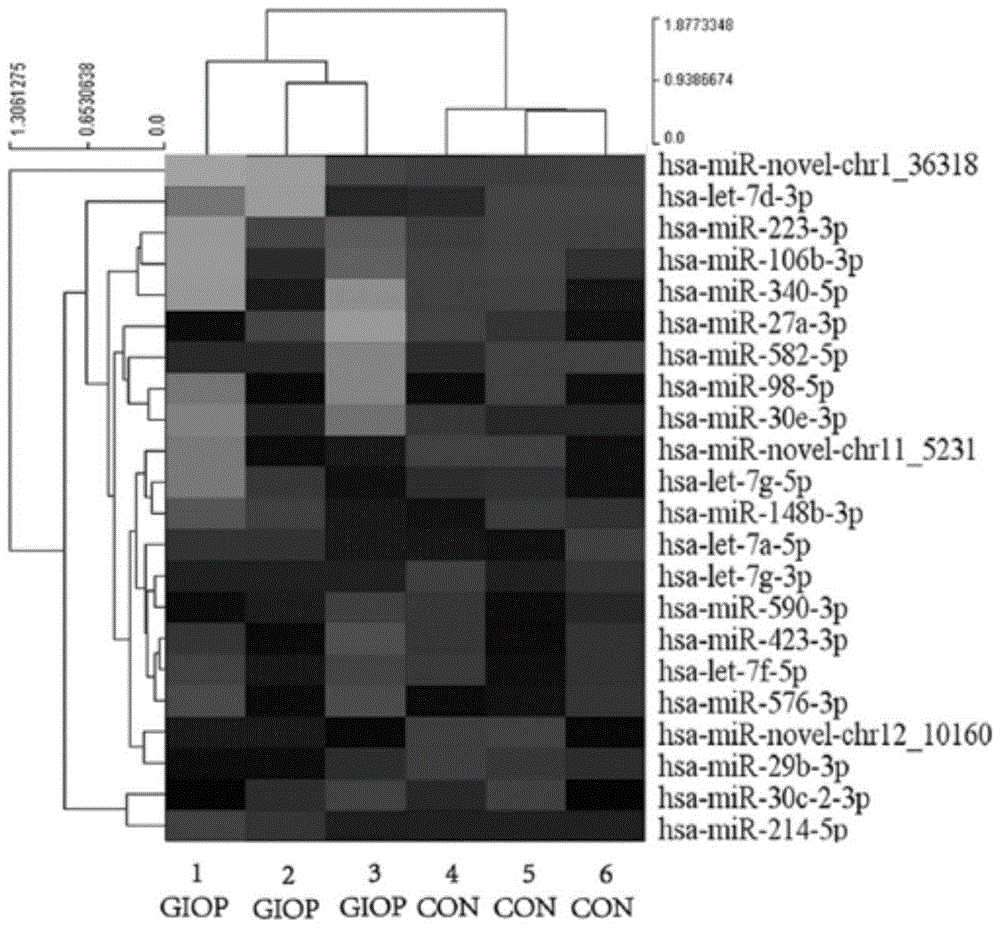
图1为实施例1中giop患者组与对照组的mirna的聚类分析示意图;
图2为实施例1中giop患者组与对照组的let-7f-5p表达量对比示意图;
图3为实施例2中let-7f-5pmimics促进成骨分化示意图;
图4为实施例2中let-7f-5pmimics促进成骨矿化示意图;
图5为实施例2中let-7f-5pmimics促进bmscs成骨alp活性增加示意图;
图6为实施例2中let-7f-5pmimics促进地塞米松诱导下的成骨分化和矿化示意图;
图7为实施例2中let-7f-5pmimics促进地塞米松诱导下的bmscs成骨alp活性增加示意图;
图8为实施例2中let-7f-5pmimics转染的mirna表达示意图;
图9为实施例2中let-7f-5pmimics转染的成骨基因runx2、osx、alp、ocn表达示意图(β-actin为内参基因);
图10为实施例2中小鼠尾静脉注射let-7f-5p增加地塞米松诱导下骨中let-7f-5pmirna表达示意图;
图11为实施例2中l4椎体micro-ct扫描三维图像显示let-7f-5p减少地塞米松诱导下的骨丢失情况;
图12为实施例2中l4椎体micro-ct扫描结果参数显示let-7f-5p减少地塞米松诱导下的骨丢失情况;
图13为实施例2中l4椎体形态计量学显示let-7f-5p减少地塞米松诱导下的骨丢失情况;
图14为实施例2中血清elisa显示let-7f-5p改善血清成骨、破骨标志物情况。
图15为实施例3中根据targetscan数据库,比较了let-7f-5p与野生型或突变型tgfbr13’utr假定靶点的碱基对。
图16为实施例3中荧光素酶报告基因实验,表明let-7f-5p对野生型tgfbr1-3'utr有明显的抑制作用。
具体实施方式
为了便于理解本发明,下面将参照相关附图对本发明进行更全面的描述。附图中给出了本发明的较佳实施例。但是,本发明可以以许多不同的形式来实现,并不限于本文所描述的实施例。相反地,提供这些实施例的目的是使对本发明的公开内容的理解更加透彻全面。
除非另有定义,本文所使用的所有的技术和科学术语与属于本发明的技术领域的技术人员通常理解的含义相同。本文中在本发明的说明书中所使用的术语只是为了描述具体的实施例的目的,不是旨在于限制本发明。
以下实施例所用原料,如非特别说明,均为市售购得。
实施例1
高通量测序筛查。
1、高通量测序
1.1方法。
应用高通量测序,筛查了来自于广州中医药大学第一附属医院的giop患者组与对照组(非giop患者)中椎体组织的差异mirnas,并对其靶基因进行了预测和验证。同时在miranda、mirbase及targetscan数据库中预测差异mirnas靶基因。
1.2结果。
结果如图1所示,图1为giop患者组与对照组的mirna的聚类分析图,其中:giop为giop患者组,con为对照组。结果发现mirnalet-7f-5p存在差异表达。
2、qrt-pcr验证
1.1方法。
采用qrt-pcr的方法验证对比giop患者组与对照组的mirnalet-7f-5p表达量。
1.2结果。
结果如图2所示,图2为giop患者组与对照组的let-7f-5p表达量对比,其中:giop为giop患者组,control为对照组。结果证实giop患者存在let-7f-5p下调,且与对照组相比具有统计学差异(p<0.001)。
上述实验结果表明,对照人群和giop患者相比,giop患者的mirnalet-7f-5p显著降低。
实施例2
mirnalet-7f-5p作为治疗骨质疏松症的药物的动物学实验验证。
一、let-7f-5p促进成骨分化。
1方法。
将8周龄小鼠的长骨(胫骨和股骨)无菌取出,用含有20%胎牛血清的α-mem冲洗骨髓。用40μm细胞滤网过滤,得到小鼠骨髓间充质干细胞(bonemarrowderived-mesenchymalstemcells,bmscs),并在35mm培养皿中以4×104/cm2的密度在37℃、5%co2中培养4天。
将上述培养的bmscs细胞种至12孔板,使用成骨诱导培养基(含有25mg/mll-抗坏血酸、5mmβ-甘油磷酸二钠、10%fbs的a-mem中)诱导成骨分化,并用let-7f-5pmimics(单链化学增强的寡核苷酸,let-7f-5p模拟物,购自广州市锐博生物科技有限公司)干预,即以let-7f-5pmimics(实现let-7f-5p过表达)或mir-nc(阴性对照)转染bmscs24小时。
转染方法为:bmscs铺板36h后,sirna储备液(20μm)5μl加入到200μl无血清的mem中,轻轻混匀,室温静置5min;然后sirna-mate10μl加入上述混合液,立即充分混匀(振荡器震荡或加样枪吹打10次以上),轻微离心,室温静置15min;6孔板细胞弃去原培养液,加入2ml新培养液,再将上述sirnaoligo-sirna-mate混合液逐滴滴加到孔中,轻轻前后摇动培养板混匀。
5、37℃培养3d后,进行相关基因和蛋白检测。
随后在成骨诱导培养基至7天,将上述细胞用于碱性磷酸酶(alp)染色和alp活性测定。矿化通常在培养10至14天后发生,并且用vonkossa方法染色以测量矿化结节形成。
上述alp染色及alp活性测定方法为:在第7天,用4%甲醛(sigma)固定细胞,并与alp底物bcip/nbt(thermoscientificwaltham)一起孵育。为了测量alp活性,用放射免疫沉淀测定法(ripa,beyotime,shanghai,china)裂解细胞。然后通过使用alp活性测定(beyotime)测定alp活性。
vonkossa染色:在第14天,为了测量矿化的结节形成,将细胞用4%甲醛在室温下固定15分钟,并用pbs洗涤3次。接下来,将细胞与5%硝酸银溶液一起温育并暴露于紫外线辐射30分钟。最后,使用5%硫代硫酸钠去除非特异性染色5分钟。
2结果。
bmscs成骨分化结果如图3所示,从图中可以看出,let-7fmimics组有较多的棕色沉淀形成,提示成骨分化增强。
bmscs成骨矿化结果如图4所示,从图中可以看出,let-7fmimics组有较多的黑色成骨钙化结节形成,提示成骨矿化能力增强。
bmscs成骨alp活性测定结果如图5所示,从图中可以看出,let-7f-5pmimics组alp活性较高,且与阴性对照组mir-nc相比具有统计学差异(p<0.01),即let-7f-5p可促进bmscs成骨alp活性增加。
二、let-7f-5p促进激素诱导下的bmscs成骨分化。
1方法。
取上述得到的bmscs(骨髓间充质干细胞),将细胞种至12孔板,待细胞长满至80%后,通过每2-3天添加新鲜成骨诱导培养基和地塞米松(dex)来维持,同时参照上述方法予let-7f-5pmimics干预。具体分组为:mir-nc组(空载体组)、let-7f-5pmimics组。
1.1分化和矿化。
在成骨诱导培养基中5至7天后,将细胞用于碱性磷酸酶(alp)染色和alp活性测定。矿化通常在培养10至14天后发生,并且细胞用vonkossa方法染色以测量矿化结节形成。
1.2成骨基因runx2、osx、alp、ocn表达。
以qpcr检测mir-nc组和let-7f-5pmimics组runx2、osx、alp、ocn成骨基因的表达,具体如下:
用trizolreagent(invitrogen)从bmscs或骨组织中提取总rna,并通过nanodrop2000分光光度计(thermoscientificwaltham,ma)测定rna浓度。将1μgrna稀释到10ml不含dnase和rnase的超纯水中,并加入1μl50mm下述引物。将4μl5×第一链缓冲液,1μl10mmdntp,1μlrnaseout,2μl0.1m二硫苏糖醇和1mlmmlv逆转录酶在不同温度和时间点(25℃,10分钟;45℃,60分钟;75℃,5分钟)依次温育。
另外,对于mirna定量rt-pcr测定,分离总rna并使用mirvanamirna分离试剂盒(thermofisherscientific)富集小rna组分。接下来,用taqmanmirna逆转录试剂盒(thermofisherscientific)制备cdna,并进行rt-pcr。最后,通过taqmanu6snrna定量测定(thermofisherscientific)let-7f-5p表达水平,其中目标序列let-7f-5p如seqidno.1所示,其余检测引物对如seqidno.2-seqidno.13所示:
let-7f-5p:5’-tgaggtagtagattgtatagtt-3’(seqidno.1);
let-7f-5p-r:5’-tctcaactggtgtcgtggagtcggcaattcagttgagaactata-3’(seqidno.2);
let-7f-5p-f:5’-acactccagctgggtgaggtagtagattgtat-3’(seqidno.3);
mrunx2-f:5’-gaccagtcttacccctccta-3’(seqidno.4)
mrunx2-r:5’-ggcagtgtcatcatctgaaa-3’(seqidno.5)
mosx-f:5’-aaaggaggcacaaagaagc-3’(seqidno.6)
mosx-r:5’-caggaaatgagtgagggaag-3’(seqidno.7)
malp-f:5’-gctttaaacccagacacaag-3’(seqidno.8)
malp-r:5’-aagaagaagcctttgaggtt-3’(seqidno.9)
mocn-f:5’-ctctctctgctcactctgct-3’(seqidno.10)
mocn-r:5’-gactgaggctccaaggtag-3’(seqidno.11)
mβ-actin-f:5’-acccagatcatgtttgagac-3’(seqidno.12)
mβ-actin-r:5’-gtcaggatcttcatgaggtagt-3’(seqidno.13)
上述mβ-actin为检测内参基因。
2结果。
2.1分化和矿化结果。
bmscs成骨分化和矿化结果如图6所示,其中alpstaining为分化情况,vonkossastaining为矿化情况,从图中可以看出,let-7fmimics组有较多的棕色沉淀和黑色钙化结节形成,提示let-7fmimics组成骨分化和矿化能力增强。
bmscs成骨alp活性测定结果如图7所示,从图中可以看出,let-7f-5pmimics组alp活性较高,且与阴性对照组mir-nc相比具有统计学差异(p<0.01),即let-7f-5p可促进地塞米松诱导下的bmscs成骨alp活性增加和成骨分化。
2.2runx2、osx、alp基因表达结果。
结果如图8所示,qpcr显示转染let-7f-5pmimics的bmscs中let-7f-5pmirna表达增加;如图9所示,qpcr显示转染let-7f-5pmimics的bmscs中成骨基因runx2、osx、alp、ocn表达增加。
三、let-7f-5p治疗giop的体内实验。
1、方法。
1.1giop动物模型的制作。
8周龄雄性c57bl/6小鼠(每组6只)购自广州中医药大学实验动物中心,腹腔注射地塞米松(dex)(购自广州中医药大学附属第一医院),每日剂量为1mg/kg体重,治疗8周后,解剖椎体骨并用于micro-ct和组织形态学分析。
1.2micro-ct分析。
使用skyscan1172microct成像系统(skyscan,kontich,belgium)进行micro-ct分析,空间分辨率为12mm(x射线源80kv/100μa)。使用内置软件nrecon1.6和ctan1.8分别进行体积重建和分析。覆盖骨小梁的圆柱形区域定义为感兴趣区域(voi)。在限定的voi内计算骨小梁体积分数(bv/tv,%),骨小梁数量(tb.n,mm),骨小梁厚度(tb.th,mm)和骨小梁分离度(tb.sp,mm)。
1.3骨组织形态学分析。
在小鼠安乐死前8天和2天给小鼠注射25mg/kg钙黄绿素。将椎体骨在10%福尔马林中固定过夜,在4℃下储存在70%乙醇中,然后加工并包埋在甲基丙烯酸甲酯中。通过使用切片机制备5mm厚的切片。l4椎骨的组织形态学测量主要包括骨形成率(bfr/bs),骨矿沉积率(mar),矿化表面面积(ms/bs)和成骨细胞数量(n.ob/b.pm)。
1.4酶联免疫吸附检测(elisa)。
禁食小鼠4h后,通过穿刺小鼠颊囊收集血液,使用ids(fountainhills,az)的酶联免疫吸附测定(elisa)试剂盒测量前胶原1型n-末端前肽(p1np)和骨钙蛋白(ocn)的血清浓度。
2、结果。
let-7f-5p抑制地塞米松诱导下的骨丢失情况如图10-14所示。
其中,图10为小鼠尾静脉注射let-7f-5p增加地塞米松诱导下骨中let-7f-5pmrna表达,其中sham表示假手术组,dex表示地塞米松诱导组,pbs为空白对照,mir-nc为阴性对照,agomir为let-7f-5p激动剂组。从图中可以看出,sham和dex条件下,小鼠尾静脉注射let-7f-5p后,其骨中let-7f-5pmirna表达显著增加,与空白对照组和阴性对照组mir-nc相比具有统计学差异(其中sham条件下,p<0.01;dex条件下,p<0.05)。
图11为l4椎体micro-ct扫描三维图像显示let-7f-5p减少地塞米松诱导下的骨丢失情况,从图中可以看出,agomir(let-7f-5p激动剂组)骨小梁增加,骨微细结构改善。
图12为l4椎体micro-ct扫描结果参数显示let-7f-5p减少地塞米松诱导下的骨丢失情况,提示agomir(let-7f-5p激动剂组)骨小梁体积分数(bv/tv)增加,骨小梁数量(tb.n)增加,骨小梁厚度(tb.th)增加,骨小梁分离度(tb.sp)降低,上述参数显示let-7f-5p减少地塞米松诱导下的骨丢失。
图13为l4椎体形态计量学显示let-7f-5p减少地塞米松诱导下的骨丢失,提示agomir(let-7f-5p激动剂组)骨形成率(bfr/bs)增加,骨矿沉积率(mar)增加,矿化表面面积(ms/bs)增加,成骨细胞数量(n.ob/b.pm)增加,显示let-7f-5p减少地塞米松诱导下的骨丢失。
图14为血清elisa显示let-7f-5p改善血清成骨、破骨标志物,前胶原1型n-末端前肽(p1np)和骨钙蛋白(ocn)的血清浓度均有提升。
四、let-7f-5p靶向tgfbr1实验研究。
1.方法。
(1)mirwalk、targetscan等数据库及多种生物信息学平台预测let-7f-5p靶基因
利用mirwalk数据库,结合targetscan7.2(http://targetscan.org)、mirbase、rna22、diana-mt、mirdb、pictar5、pita、miranda等多种生物信息学平台,预测let-7f-5p靶基因及其结合位点。
(2)双荧光素酶报告基因验证let-7f-5p与tgfbr1的靶向关系。
使用小鼠基因组dna作为模板,通过逆转录pcr(rt-pcr)扩增let-7f-5p前体的基因组片段。将扩增产物克隆到限制性位点noti和xhoi的plas2-rfp载体中。然后用修饰的载体病毒感染bmscs,并通过定量rt-pcr(abistep-oneplus)检测let-7f-5p表达。另外,将tgfbr1中let-7f-5p的结合位点和整个tgfrb13'utr序列克隆到限制性位点pmei和xhoi的pmirglo荧光素酶载体中。另外,含有突变序列的一对引物用于产生tgfrb13'utr的突变构建体。
2.结果。
let-7f-5p与tgfbr1靶向关系实验研究如图15-16所示。
其中,图15根据targetscan数据库,比较了let-7f-5p与野生型或突变型tgfbr13’utr假定靶点的碱基对。提示let-7f-5p与tgfbr1存在靶向关系。
图16荧光素酶报告基因实验,表明let-7f-5p对tgfbr1-3'utr的抑制作用。其中,mir-nc表示空载体mirna,tgfbr13’utr表示野生型tgfbr13’utr,mtoftgfbr13’utr表示突变体tgfbr13’utr。let-7f-5pmimics干预后,野生型tgfbr13’utr降低,与空载体mirna(mir-nc)相比具有具有统计学差异(p<0.001),而突变tgfbr13’utr中,mir-nc与let-7f-5pmimics无统计学差异。提示let-7f-5p抑制tgfbr1-3'utr,进一步验证let-7f-5p与tgfbr1存在靶向关系。
通过上述实验可以看出,let-7f-5p具有促进成骨分化的作用(图3-5);同时let-7f-5p促进地塞米松诱导下bmscs成骨分化(图6-7);体内实验证实let-7f-5p减少地塞米松诱导下的骨丢失(图10-14)。同时,let-7f-5p与tgfbr1基因存在靶向关系。因此let-7f-5p及其靶基因的制备可以作为防治op的有价值的药物。
以上所述实施例的各技术特征可以进行任意的组合,为使描述简洁,未对上述实施例中的各个技术特征所有可能的组合都进行描述,然而,只要这些技术特征的组合不存在矛盾,都应当认为是本说明书记载的范围。
以上所述实施例仅表达了本发明的几种实施方式,其描述较为具体和详细,但并不能因此而理解为对发明专利范围的限制。应当指出的是,对于本领域的普通技术人员来说,在不脱离本发明构思的前提下,还可以做出若干变形和改进,这些都属于本发明的保护范围。因此,本发明专利的保护范围应以所附权利要求为准。
序列表
<110>广州中医药大学第一附属医院
<120>let-7f-5p及其靶基因在制备用于治疗骨质疏松症药物中的应用
<160>13
<170>siposequencelisting1.0
<210>1
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>1
tgaggtagtagattgtatagtt22
<210>2
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>2
tctcaactggtgtcgtggagtcggcaattcagttgagaactata44
<210>3
<211>32
<212>dna
<213>人工序列(artificialsequence)
<400>3
acactccagctgggtgaggtagtagattgtat32
<210>4
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>4
gaccagtcttacccctccta20
<210>5
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>5
ggcagtgtcatcatctgaaa20
<210>6
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>6
aaaggaggcacaaagaagc19
<210>7
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>7
caggaaatgagtgagggaag20
<210>8
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>8
gctttaaacccagacacaag20
<210>9
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>9
aagaagaagcctttgaggtt20
<210>10
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>10
ctctctctgctcactctgct20
<210>11
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>11
gactgaggctccaaggtag19
<210>12
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>12
acccagatcatgtttgagac20
<210>13
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>13
gtcaggatcttcatgaggtagt22
- 还没有人留言评论。精彩留言会获得点赞!